Method Article
Système onePot PURE sans cellule
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Nous présentons une méthode rapide et rentable pour produire le système TX-TL recombinant PURE sans cellule à l’aide d’équipements de laboratoire standard.
Résumé
Le système de transcription-traduction PURE (synthèse de protéines à l’aide d’éléments recombinants) défini fournit un châssis attrayant pour la biologie synthétique sans cellules. Malheureusement, les systèmes disponibles dans le commerce sont coûteux et leur accordabilité est limitée. En comparaison, une approche maison peut être personnalisée en fonction des besoins des utilisateurs. Cependant, la préparation de systèmes faits maison prend du temps et est ardue en raison du besoin de ribosomes ainsi que de 36 purifications de protéines à moyenne échelle. La rationalisation de la purification des protéines par coculturation et co-purification permet de minimiser les besoins en temps et en main-d’œuvre. Ici, nous présentons une méthode simple, réglable, rapide et rentable pour produire tous les composants du système PURE en 1 semaine, en utilisant un équipement de laboratoire standard. De plus, les performances du OnePot PURE sont comparables à celles des systèmes disponibles dans le commerce. La méthode de préparation OnePot PURE étend l’accessibilité du système PURE à un plus grand nombre de laboratoires en raison de sa simplicité et de sa rentabilité.
Introduction
Les systèmes de transcription-traduction sans cellules (TX-TL) constituent une plate-forme prometteuse pour l’étude et l’ingénierie des systèmes biologiques. Ils fournissent des conditions de réaction simplifiées et accordables, car ils ne reposent plus sur des processus de maintien de la vie, y compris la croissance, l’homéostasie ou les mécanismes de régulation1. Ainsi, il est prévu que les systèmes sans cellules contribueront à l’étude des systèmes biomoléculaires, offriront un cadre pour tester des stratégies de bioconceptionrationnelles 2, et fourniront un châssis pour une future cellule synthétique3,4. Le système PURE entièrement recombinant offre un châssis particulièrement attrayant en raison de sa composition définie et minimale, ainsi que de son ajustabilité et de sa réglabilité5.
Depuis que le premier système PURE fonctionnel et entièrement recombinant a été mis en place en 20015, des efforts ont été faits pour élargir les limites du système et optimiser la composition du système afin d’améliorer les rendements du système6,7,8, permettre la régulation transcriptionnelle9, membrane 10,11et la synthèse des protéines sécrétoires12, et faciliter le repliement des protéines13,14 . De nos jours, il existe trois systèmes disponibles dans le commerce : PUREfrex (GeneFrontier), PURExpress (NEB) et Magic PURE (Creative Biolabs). Cependant, ces systèmes sont coûteux, leur composition exacte est propriétaire et donc inconnue, et l’adaptabilité est limitée.
Les systèmes PURE préparés en interne se sont avérés être l’option la plus rentable et la plus réglable15,16. Cependant, les 37 étapes de purification requises pour les fractions de protéines et de ribosomes prennent beaucoup de temps et sont fastidieuses. Plusieurs tentatives ont été faites pour améliorer l’efficacité de la préparation du système PURE17,18,19. Nous avons récemment démontré qu’il est possible de coculturer et de co-purifier toutes les protéines non ribosomiques requises présentes dans le système PURE. Cette méthode OnePot s’est avérée rentable et rapide, réduisant le temps de préparation de plusieurs semaines à 3 jours ouvrables. L’approche génère un système PURE avec une capacité de production de protéines comparable au système PURExpress disponible dans le commerce20. Contrairement aux approches précédentes pour simplifier la préparation PURE17,18,19, dans l’approche OnePot, toutes les protéines sont encore exprimées en souches séparées. Cela permet à l’utilisateur d’ajuster la composition du système OnePot PURE en omettant ou en ajoutant simplement des souches spécifiques ou en ajustant les volumes d’inoculation, générant ainsi des systèmes PURE de décrochage ou modifiant les rapports protéiques finaux, respectivement.
Le protocole présenté ici fournit une méthode détaillée pour créer le système OnePot PURE tel que décrit précédemment20, bien que β-mercaptoéthanol ait été remplacé par de la tris(2-carboxyéthyl)phosphine (TCEP). De plus, deux méthodes de purification des ribosomes sont décrites : la purification traditionnelle des ribosomes sans étiquette par interaction hydrophobe et coussin de saccharose, adaptée de Shimizu et al.15,et la purification des ribosomes Ni-NTA basée sur Wang et al.18 et Ederth et al.21 mais significativement modifiée. Cette dernière méthode facilite davantage la préparation du système PURE et le rend accessible à un plus grand nombre de laboratoires, car seul un équipement de laboratoire standard est requis.
Le protocole expérimental résume la préparation d’un système TX-TL polyvalent pure sans cellule pour fournir une plate-forme simple, accordable et rentable sans cellule, qui peut être préparée à l’aide d’un équipement de laboratoire standard en une semaine. En plus d’introduire la composition PURE standard, nous indiquons comment et où elle peut être ajustée, en mettant l’accent sur les étapes critiques du protocole pour assurer la fonctionnalité du système.
Protocole
REMARQUE: Ce protocole décrit la préparation du système TX-TL sans cellule à partir de composants recombinants. Pour plus de commodité, l’œuvre est séparée en cinq parties. La première partie décrit les étapes de préparation, qui doivent être effectuées avant de commencer le protocole. La deuxième partie décrit la préparation de la solution protéique OnePot. La troisième partie décrit les purifications de ribosomes, la quatrième partie détaille la préparation de la solution énergétique et la dernière partie fournit un manuel pour la mise en place d’une réaction PURE. Pour plus de commodité, les protocoles sont divisés en jours et résumés dans les horaires quotidiens du tableau 1. En suivant le calendrier, l’ensemble du système peut être préparé en 1 semaine par une seule personne.
1. Travaux préliminaires
- Préparer les milieux de culture bactériens et les suppléments de milieux comme décrit dans le tableau supplémentaire 1. Préparer et stériliser les matériaux requis, y compris les embouts de pipette, 96 plaques de puits profond.
- Préparation de la souche
- Transformez les déformations d’expression indiquées dans le tableau 2 avec les vecteurs d’expression correspondants à l’aide de la méthode du choc thermique.
- Ajouter le plasmide purifié aux bactéries chimiquement compétentes et incuber sur de la glace pendant 20 à 30 minutes.
- Placer le mélange à 42 °C pendant 30 s (choc thermique) puis le remettre sur la glace pendant 2 min.
- Pipetter 20 μL de la bactérie directement sur des plaques de gélose contenant de l’ampicilline (AMP) et incuber à 37 °C pendant la nuit. Conservez les plaques à 4 °C jusqu’à 1 semaine.
- Inoculer 3 mL de milieu LB contenant de l’AMP avec une seule colonie de bactéries provenant des plaques de gélose. Incuber à 37 °C en agitant à 260 tr/min pendant la nuit.
- Mélanger 250 μL de la culture avec 250 μL de glycérol à 50 % (v/v) et conserver à -80 °C.
REMARQUE: Pour une préparation plus rapide à l’avenir, stockez les souches dans une plaque de 96 puits sous forme de stocks de glycérol.
- Confirmer toutes les transformations vectorielles par PCR des colonies et séquençage. Séquencez le gène, la région promotrice et le site de liaison des ribosomes.
- Transformez les déformations d’expression indiquées dans le tableau 2 avec les vecteurs d’expression correspondants à l’aide de la méthode du choc thermique.
- Test d’expression
- Inoculer 300 μL de milieux LB contenant de l’AMP avec environ 1 μL des stocks de glycérol préparés dans une plaque de puits profond de 1,3 mL. Scellez la plaque avec une membrane respirante, puis incubez à 37 °C en agitant à 260 tr/min pendant la nuit.
REMARQUE : Toutes les expressions sont effectuées séparément à ce stade. - Inoculer 300 μL de milieux LB frais contenant de l’AMP avec 1 μL des cultures de nuit. Incuber à 37 °C en agitant à 260 tr/min pendant la nuit. Après 2 h, induire les cellules avec 100 μM d’isopropyle β-D-1-thiogalactopyranoside (IPTG) et croître pendant 3 h supplémentaires.
- Mélanger 10 μL de la culture avec 10 μL de 2x tampon Laemmli et chauffer à 95 °C pendant 10 min. Faire tourner les échantillons pendant 1 min à l’aide d’une centrifugeuse de table et charger 10 μL du surnageant sur un gel PAGE. Faire fonctionner le gel dans un tampon Tris/Glycine/SDS à 200 V pendant 30 min. Rincez-le bien à l’eau désionisée. Couvrir le gel avec une tache de protéine Coomassie et incuber pendant 1 h. Déstabiliser le gel dans l’eau si nécessaire (résultats représentatifs pour l’essai d’expression de la figure 1).
REMARQUE: Utilisez des gels PAGE dégradés (4%-15% ou 4%-20%) pour obtenir une bonne séparation.
- Inoculer 300 μL de milieux LB contenant de l’AMP avec environ 1 μL des stocks de glycérol préparés dans une plaque de puits profond de 1,3 mL. Scellez la plaque avec une membrane respirante, puis incubez à 37 °C en agitant à 260 tr/min pendant la nuit.
- Restauration et nettoyage de la résine IMAC Sepharose
- Préparation des colonnes.
- Mélangez bien la résine Sepharose par vortex.
- Pipette la quantité requise de résine dans une colonne d’écoulement par gravité vide.
REMARQUE: La quantité de résine requise varie entre la purification du his-ribosome et la purification des protéines et est spécifiée dans les sections respectives. - Laver la résine avec 30 mL d’eau désionisée.
- Procéder à la recharge de la colonne comme spécifié à la section 1.4.4.
REMARQUE: Laissez toujours tout le liquide passer à travers la colonne avant de passer à l’étape suivante. Cependant, assurez-vous que la colonne ne s’assèche jamais. Lorsque vous faites passer un liquide à travers la colonne, assurez-vous d’arrêter l’écoulement ou de passer à l’étape suivante dès que le liquide atteint la résine.
- Restauration.
- Laver la colonne avec 30 mL d’eau désionisée.
- Appliquer 10 mL d’une solution d’EDTA de 0,2 M et de NaCl de 0,5 M.
- Ajouter 30 mL d’une solution de NaCl de 0,5 M.
- Laver la colonne avec 50 mL d’eau désionisée.
- Conserver dans de l’éthanol à 20 % (v/v) à 4 °C ou passer à l’étape suivante.
- Nettoyage.
ATTENTION : Portez un équipement de protection.- Laver la colonne avec 30 mL de NaOH de 0,5 M.
- Laver la colonne avec 30 mL d’eau désionisée.
- Laver la colonne avec 30 mL d’acide acétique 0,1 M.
- Laver la colonne avec 30 mL d’eau désionisée.
- Laver la colonne avec 30 mL d’éthanol à 70 % (v/v).
- Laver la colonne avec 50 mL d’eau désionisée.
- Conserver dans de l’éthanol à 20 % (v/v) à 4 °C ou passer à l’étape suivante.
- Recharge.
- Ajouter 10 mL de solution de sulfate de nickel 0,1 M à la colonne.
ATTENTION : Le sulfate de nickel est toxique. Les déchets de sulfate de nickel doivent être jetés avec les précautions indiquées par le fournisseur. - Laver la colonne avec 50 mL d’eau désionisée.
- Conserver dans de l’éthanol à 20 % (v/v)) à 4 °C ou poursuivre l’équilibrage de la colonne.
REMARQUE: Si la colonne est stockée dans de l’éthanol entre les étapes, assurez-vous d’éliminer toute trace d’éthanol en lavant la colonne avec de l’eau.
- Ajouter 10 mL de solution de sulfate de nickel 0,1 M à la colonne.
- Préparation des colonnes.
2. Expression et purification de la solution protéique OnePot
REMARQUE: Le protocole se compose de trois parties divisées en jours (Figure 2). Une procédure de préparation idéale produit 1,5 mL de solution protéique OnePot de 13,5 mg/mL, ce qui correspond à plus de mille réactions PURE de 10 μL. Cependant, la quantité et la concentration idéale de la solution varieront d’un lot à l’autre. Les utilisateurs expérimentés peuvent effectuer plusieurs préparations OnePot PURE à la fois.
Jour 1 :
- Préparer des milieux de culture bactériens et des suppléments de milieux comme décrit dans le tableau supplémentaire 1.
- Préparez et stérilisez les matériaux requis, y compris les embouts de pipette, deux plaques de puits profond de 96 et une fiole d’Erlenmeyer déconcertée de 1 L.
- Préparer des tampons et des suppléments comme décrit dans le tableau supplémentaire 2. Filtrer stériliser tous les tampons à l’aide de filtres à bouchon de bouteille (0,45 μm) et les conserver à 4 °C. Complétez tous les tampons avec 1 mM de TCEP juste avant utilisation, sauf indication contraire.
- Utilisez 2 mL de résine de sépharose pour la purification de la protéine OnePot. Préparez la colonne comme décrit à la section 1.4.
- Pour préparer les cultures de départ, combiner 20 mL de milieu LB avec 20 μL d’AMP. Dans une plaque stérile de 96 puits profonds de 1,3 mL, ajouter 300 μL du milieu dans 35 puits. Inoculez chacun d’eux avec sa souche respective, à l’exception du facteur d’allongement thermo instable (EF-Tu), et scellez la plaque avec une membrane respirante.
REMARQUE: Inoculez la plaque à l’aide d’un réplicateur à 96 puits (voir tableau des matériaux). Le volume du puits de la plaque de puits profond et le volume de la culture de démarrage sont essentiels. Des volumes de milieux plus importants ou des volumes de puits plus petits entraîneront une densité bactérienne différente en raison d’incohérences d’aération. - Pour la culture EF-Tu, inoculer 3 mL de milieu LB dans un tube de culture de 14 mL avec un capuchon encliquetable. Un seul 3 mL de culture pour EF-Tu est suffisant pour une culture d’expression OnePot.
- Incuber à 37 °C en agitant à 260 tr/min pendant la nuit.
Jour 2:
REMARQUE: Effectuez toutes les étapes à température ambiante, sauf indication contraire.
- Transférer 500 mL de milieu LB et 500 μL d’AMP dans la fiole stérile déconcertée.
- Inoculer la culture OnePot PURE avec 1675 μL de la culture EF-Tu et 55 μL de chacune des cultures de la plaque de puits profond (Tableau 2).
REMARQUE: Au cours de cette étape, la composition globale des protéines peut être ajustée en ajustant les rapports d’inoculation. Assurez-vous que le volume global d’inoculation reste constant à 3,6 mL.
FACULTATIF : Pour confirmer que toutes les souches ont poussé pendant la nuit, mesurez la densité optique des cultures de nuit à 600 nM (OD600) dans une plaque de 96 puits à l’aide d’un lecteur de plaques. Utilisez une dilution de 10x pour la mesure de la densité optique. - Incuber la culture pendant 2 h à 37 °C en agitant 260 tr/min, ou jusqu’à ce que l’OD600 de la culture atteigne 0,2-0,3.
- Induire la culture avec 500 μL de 0,1 mM IPTG et croître pendant 3 h supplémentaires.
- Récolter les cellules par centrifugation à 4 °C et 3220 x g pendant 10 min et stocker la pastille cellulaire à -80 °C jusqu’à une utilisation ultérieure.
REMARQUE: Pour optimiser le timing, préparez la solution énergétique décrite à la section 4 pendant les temps d’incubation du jour 2(tableau 1).
Jour 3:
- Mesurer les quantités de tampons nécessaires à la purification décrite dans les étapes ci-dessous et ajouter le TCEP à chacun d’eux comme indiqué dans le tableau supplémentaire 2. Stocker les tampons restants sans TCEP à 4 °C pour les purifications futures.
- Équilibrer la colonne chargée (section 2.4) avec 30 mL de tampon A. Après le passage de 25 mL de tampon A, fermez la colonne par le bas. En parallèle, passez aux étapes 2.15 à 2.17.
- Décongelez les cellules et utilisez une pipette sérologique pour ressuspender la pastille de cellule dans 7,5 mL de tampon A.
- Lyser les cellules à l’aide d’un sonicateur de sonde de 130 watts (voir Tableau des matériaux,diamètre de la pointe de la sonde: 6 mm) avec les paramètres suivants: 4 x 20 s d’impulsion allumée, 20 s d’impulsion éteinte, amplitude de 70%. Si la sonication réussit, la solution deviendra plus sombre (Figure 2).
REMARQUE: Assurez-vous de garder les cellules sur la glace pendant la sonication. Placez la sonde assez profondément dans la solution sans toucher le tube. Si une grande quantité de mousse est générée, le transfert d’énergie sera amorti. Dans ce cas, laissez la mousse se déposer, abaissez la sonde plus profondément dans la solution et prolongez le temps de sonication. - Enlever les débris cellulaires par centrifugation à 21130 x g pendant 20 min à 4 °C immédiatement après la sonication. Gardez le lysate sur la glace.
- Ajoutez le surnageant à la colonne équilibrée. Fermez la colonne par le haut et assurez-vous qu’il n’y a pas de fuite. Incuber la colonne pendant 3 h à 4 °C en rotation à l’aide d’un rotateur à tube.
- Elutez les composants non liés de la colonne et lavez avec 25 mL de tampon A.
- Laver la colonne avec 25 mL de tampon imidazole de 25 mM (23,95 mL de tampon A et 1,25 mL de tampon B).
- Éluez les protéines avec 5 mL de tampon imidazole de 450 mM (0,5 mL de tampon A et 4,5 mL de tampon B). Gardez les protéines éluées sur la glace en tout temps.
- Diluer l’éluat avec 25 mL de tampon HT, garder le mélange sur de la glace. Ajouter 15 mL à un filtre centrifuge de 15 mL et concentrer à un volume de 1,5 mL. Ajouter les 15 mL restants au filtre avec la solution concentrée et concentrer à nouveau à 1,5 mL.
- Ajouter 10 mL de tampon HT à l’échantillon concentré et concentrer à 1 mL. Ajouter une quantité égale de tampon de stock B et conserver à -80 °C jusqu’à une utilisation ultérieure.
REMARQUE: Un cycle d’échange / concentration prend environ 60 minutes de rotation à 3220 x g à 4 ° C. - Pendant l’échange de la mémoire tampon, restaurez la colonne comme indiqué à la section 1.4.
Jour 4:
- Mesurez la concentration en protéines à l’aide du test de Bradford tel que décrit par le fournisseur. Concentrer l’échantillon avec un filtre centrifuge à coupure de 0,5 mL de 3 kDa à 20 mg/mL.
REMARQUE: Diluer la solution protéique 25 fois ou 50 fois avant les mesures de concentration pour éviter de sursaturer le test de Bradford. - Pour établir la concentration idéale en protéines, effectuez un test d’expression à ce stade (section 5.2) avec différentes concentrations de la solution protéique. Pour effectuer le titrage, maintenir le volume total de la solution constant et pipeter la solution protéique OnePot, y compris le tampon de base B, à cinq rapports différents (Tableau supplémentaire 7).
- Vérifiez la composition de la protéine OnePot PURE à l’aide de SDS-PAGE (Figure 3A). Diluer 2,5 μL de l’échantillon avec 7,5 μL d’eau, mélanger avec 10 μL de tampon Laemmli 2x, puis charger 5 μL et 2,5 μL des échantillons dans le gel. Exécutez la SDS-PAGE comme spécifié dans la section 1.3.3.
- Aliquoter la solution protéique en aliquotes de 50 μL après avoir vérifié l’expression et ajusté la concentration. Conserver la solution protéique OnePot PURE à -80 °C jusqu’à nouvel usage.
REMARQUE: Si un composant protéique est soupçonné de ne pas être présent, ou est présent dans une concentration inférieure à celle prévue dans le OnePot PURE, effectuez les étapes suivantes. - Vérifiez si la culture nocturne de la souche respective a augmenté à un rythme comparable à celui des autres cultures en effectuant des mesures de densité optique (OD600)de toutes les cultures.
- Effectuer un test d’expression supplémentaire de la souche spécifique pour vérifier l’expression de la protéine suspecte.
3. Solution de ribosome
REMARQUE: Deux stratégies différentes de purification des ribosomes sont introduites, l’une pour les ribosomes marqués à l’hexaméthidine et l’autre pour les ribosomes non marqués. Le principal avantage de la méthode de purification utilisant la purification His sur une colonne d’écoulement gravitaire Ni-NTA d’affinité standard est que la purification est facile, rapide et ne nécessite pas d’équipement de laboratoire supplémentaire, tel qu’un système FPLC et une ultracentrifugeuse. Cependant, la capacité de production de protéines dans les réactions OnePot PURE est d’environ un tiers par rapport aux ribosomes sans étiquette. Par conséquent, choisissez la méthode de production de ribosomes en fonction de l’importance d’un rendement élevé pour l’application donnée.
-
Purification des ribosomes marquée par His
REMARQUE: Ce protocole utilise la souche E. coli RB1, un cadeau du professeur Wang (Université Columbia, États-Unis)18. Cette souche a une insertion génomique d’une étiquette hexa-témidine sur la terminaison C de la protéine ribosomique 50S (L7/L12), permettant la purification à l’aide d’une colonne de flux de gravité Ni-NTA. Le rendement habituel est d’environ 0,5 mL de ribosomes de 3,45 μM, ce qui est suffisant pour plus de cinq cent réactions PUREs de 10 μL.
Jour 1 :
- Préparer des milieux de culture bactériens et des suppléments de milieux comme décrit dans le tableau supplémentaire 1.
- Préparer et stériliser les matériaux requis, y compris les embouts de pipette, une fiole d’Erlenmeyer de 5 L et une fiole d’Erlenmeyer de 100 mL.
- Préparer des tampons et des suppléments comme décrit dans le tableau supplémentaire 2. Filtrer stériliser tous les tampons à l’aide de filtres de bouchon de bouteille (0,45 μm) et les stocker à 4 °C.
Jour 2:
- Pipeter 5 mL de résine sur une colonne et préparer la colonne comme spécifié au point 1.4.
REMARQUE: En raison du volume plus élevé de la résine, la restauration et la purification prennent beaucoup plus de temps. Utilisez une colonne différente pour la purification des ribosomes afin d’éviter la contamination croisée et nettoyez-la soigneusement avant la purification. - Préparer une culture nocturne de la souche E. coli RB1 en inoculant 35 mL de milieu LB dans une fiole d’Erlenmeyer de 100 mL. Incuber à 37 °C en agitant à 260 tr/min.
Jour 3:
REMARQUE: Effectuez toutes les étapes à température ambiante, sauf indication contraire.
- Ajouter 2 L de milieu LB dans une fiole stérile de 5 L, inoculer avec 12 mL de la culture pendant la nuit, puis incuber pendant 3-4 h à 37 °C tout en agitant à 260 tr/min.
REMARQUE: Alternativement, effectuer une culture bactérienne dans 4 x 500 mL de cultures dans des flacons déconcertés de 1 L. - Abreuler les cellules par centrifugation pendant 10 min à 3220 x g et 4 °C. Conserver à -80 °C jusqu’à nouvel usage.
Jour 4:
- Équilibrez la colonne préparée à l’étape 3.1.4. avec 30 mL de tampon de lyse.
- Resuspendez la pastille dans 20 mL de tampon de lyse à l’aide d’une pipette sérologique.
- Lyser les cellules avec un sonicateur de sonde de 130 watts (voir Tableau des matériaux,diamètre de la pointe de la sonde: 6 mm) sur la glace avec les paramètres suivants: 11 x 20 s d’impulsion allumée; 20 s d’impulsion désactivée, amplitude de 70 % (voir l’étape 2.16 pour plus de détails sur la procédure).
- Immédiatement après la sonication, retirer les débris cellulaires par centrifugation pendant 20 min à 21130 x g à 4 °C. Gardez le lysate sur la glace.
- Chargez le surnageant dans les colonnes et laissez-le passer.
- Lavez la colonne avec les mélanges suivants de tampons de lyse et d’élution.
- Lavage 0 : utiliser 30 mL de tampon de lyse.
- Lavage 1 : utiliser 30 mL d’imidazole de 5 mM (29 mL de tampon de lyse, 1 mL de tampon d’élution).
- Lavage 2 : utiliser 60 mL d’imidazole de 25 mM (50 mL de tampon de lyse, 10 mL de tampon d’élution).
- Lavage 3 : utiliser 30 mL d’imidazole de 40 mM (22 mL de tampon de lyse, 8 mL de tampon d’élution).
- Lavage 4 : utiliser 30 mL d’imidazole de 60 mM (18 mL de tampon de lyse, 12 mL de tampon d’élution).
- Éluez les ribosomes avec 7,5 mL de tampon d’élution. Gardez les protéines éluées sur la glace en tout temps.
- Ajouter 22 μL de β-mercaptoéthanol pur à 45 mL de tampon ribosome.
ATTENTION : β-mercaptoéthanol est toxique. Prenez des précautions de sécurité et travaillez dans une hotte aspirante. - Ajouter l’éluat dans un filtre centrifuge de 15 mL et concentrer à 1 mL.
- Ajouter 15 mL de tampon ribosome à l’échantillon concentré et concentrer à nouveau à 1 mL.
REMARQUE : Répétez deux fois l’étape précédente. - Conserver à -80 °C jusqu’à nouvel usage.
NOTE: Un cycle d’échange/concentration prend environ 60 min de centrifugation à 3220 x g à 4 °C. - Pendant l’échange de la mémoire tampon, restaurez la colonne comme indiqué à la section 1.4.
Jour 5:
- Déterminer la concentration de ribosomes en mesurant l’absorbance à 260 nM d’un échantillon dilué 1:100 dans un tampon ribosome. Une valeur d’absorbance de 10 de la solution diluée correspond à 23 μM de solution non diluée comme décrit précédemment16.
- Mettre en œuvre une concentration finale du stock de 3,45 μM. Pour ajuster la concentration, diluer les ribosomes avec un tampon ribosome ou les concentrer davantage par centrifugation à 14000 x g dans un filtre centrifuge de 3 kDa 0,5 mL à 4 °C.
REMARQUE: Pour obtenir une expression optimale du système, effectuer un titrage de concentration de ribosomes (rubrique 5.2, tableau supplémentaire 7). - Vérifier la composition des ribosomes à l’aide de la FDS-PAGE (Figure 3A) comme spécifié à la section 1.3.3. Diluer 2,5 μL de l’échantillon avec 7,5 μL d’eau, mélanger avec 10 μL de tampon Laemmli 2x, puis charger 5 μL et 2,5 μL des échantillons sur le gel.
-
Purification des ribosomes sans étiquette
REMARQUE: La purification des ribosomes sans étiquette est effectuée à l’aide d’un système FPLC (Table des matériaux) et est basée sur la chromatographie d’interaction hydrophobe utilisant 2 colonnes de butyle de 5 mL (Table des matériaux). Bien que les ribosomes puissent être purifiés de n’importe quelle souche, l’utilisation de la souche E. coli A19 (E. coli Genetic Resources at Yale CGSC) est avantageuse en raison de sa délétion RNase I22. Effectuez la purification à 4 °C dans une chambre froide ou une armoire de refroidissement. Le rendement habituel est d’environ 0,5 mL de ribosomes de 10 μM, ce qui correspond à plus de cinq cents réactions PURE de 10 μL.
Jour 1 :
- Préparer des milieux de culture bactériens et des suppléments de milieux comme décrit dans le tableau supplémentaire 1.
- Préparer et stériliser les matériaux requis, y compris les embouts de pipette, la fiole d’Erlenmeyer de 5 L et la fiole d’Erlenmeyer de 100 mL.
- Préparer des tampons et des suppléments comme décrit dans le tableau supplémentaire 2. Filtrer stériliser tous les tampons à l’aide de filtres de bouchon de bouteille (0,45 μm) et les stocker à 4 °C.
Jour 2:
- Pour préparer une culture de nuit de la souche E. coli A19, inoculer 35 mL de milieu LB dans une fiole d’Erlenmeyer de 100 mL. Incuber à 37 °C en agitant à 260 tr/min.
Jour 3:
- Transférer 2 L de milieu LB dans la fiole déconcertée stérile de 5 L, inoculer avec 30 mL de la culture pendant la nuit, puis incuber pendant 3-4 h à 37 °C tout en agitant à 200 tr/min.
- Granulés les cellules par centrifugation à 4000 x g pendant 15 min à 4 °C. Ressuspendez la pastille dans 25 mL de tampon de suspension et conservez-la à -80 °C jusqu’à une utilisation ultérieure.
Jour 4:
- Effectuez les étapes 3.2.8 à 3.2.12 en parallèle avec les étapes 3.2.13 à 3.2.19.
- Décongeler et lyser les cellules à l’aide d’un sonicateur à sonde de 130 watts (voir Tableau des matériaux et diamètre de la pointe de la sonde: 6 mm) sur la glace avec les paramètres suivants: impulsion de 12 x 20 s; 20 s d’impulsion désactivée, amplitude de 70 % (voir les détails de la procédure à l’étape 2.16).
- Retirer immédiatement les débris cellulaires par centrifugation à 20000 x g pendant 20 min à 4 °C.
- Aspirez le surnageant et mesurez le volume. Ajouter un volume égal de tampon de suspension (sel élevé) pour ajuster la concentration finale de sulfate d’ammonium à 1,5 M et bien mélanger.
- Retirer le précipité par centrifugation à 20000 x g pendant 20 min à 4 °C.
- Filtrer le surnageant à l’aide d’un filtre à seringue à membrane de polyéthersulfone de 0,45 μm avant la purification FPLC et recueillir le filtrat dans une bouteille en verre de 100 mL. Maintenir le surnageant à 4 °C en tout temps.
- Configurez le système FPLC pour la purification par chromatographie d’interaction hydrophobe à l’aide d’une double colonne de butyle (2 x 5 mL) comme suit. Pour cette configuration, un volume de colonne (CV) fait référence à un volume de 10 mL.
- Trois entrées seront nécessaires : deux comme lignes tampons et une comme ligne d’échantillonnage. En raison des paramètres par défaut du purificateur, il est pratique de choisir les lignes A1 et B1 pour le tampon C et le tampon D, respectivement, et la ligne A2 comme ligne d’échantillonnage. Appliquer un débit par défaut de 4 mL/min, sauf pour les lavages par pompe (10 mL/min) ou sauf indication contraire.
REMARQUE: Comme TCEP est un réactif coûteux, ajoutez la quantité correspondante aux tampons C et D uniquement après l’étape d’équilibrage. - Effectuer un lavage de la pompe du système dans de l’éthanol à 20 % (v/v)) pour nettoyer le système et éliminer la contamination potentielle des purifications précédentes. Réglez manuellement un débit de 0,2 mL/min et montez la colonne. Arrêtez le flux.
- Exécutez un système de lavage à la pompe avec de l’eau. Lavez la colonne avec 3 CV d’eau.
- Equilibration : placer les entrées A1 et A2 dans le tampon C et l’entrée B1 dans le tampon D sans TCEP. Exécutez un lavage à la pompe et équilibrez la colonne avec 4 CV de tampon C.
- Ajoutez TCEP aux tampons C et D.
- Préparer des tubes de 15 mL ou des tubes collecteurs de fractions ronds transparents au collecteur de fractions pour recueillir des fractions d’élution de 4 à 5 mL.
- Chargement: Placez l’entrée A2 dans la bouteille avec l’échantillon filtré. Chargez environ 90 % du volume de l’échantillon sur la colonne. Diluer l’échantillon avec 20 mL de tampon C contenant du TCEP et charger 10 mL de l’échantillon sur la colonne. Répétez l’étape de dilution au moins deux fois et chargez autant d’échantillon que possible sur la colonne. Il est essentiel de s’assurer qu’aucun air n’est aspiré dans la machine.
- Étape de lavage 1 : laver avec 3 CV de tampon C pour retirer les composants non liés.
- Étape de lavage 2 : lavage avec 5 CV de 80% de tampon C et 20% de tampon D.
- Élution: éluez le produit en appliquant 50% de tampon C et 50% de tampon D, avec un volume d’élution total de 5 CV. Collectez cette fraction dans les tubes collecteurs.
- Étape de lavage 3 : Éliminez tous les contaminants à forte interaction à l’aide du tampon D à 100 % avec un volume total de 5 CV.
- Analyser le spectre d’absorption de la fraction de l’échantillon à 260 ou 280 nM(Figure 4). Le premier pic montre les protéines non absorbées éluées pendant le chargement et la première étape de lavage; le deuxième pic montre les contaminants qui ont été élués lors de la deuxième étape de lavage. Le troisième pic surveille le produit final, et le dernier pic montre les contaminants en forte interaction. Regrouper toutes les fractions d’échantillon correspondant au troisième pic pour un traitement ultérieur. Gardez les protéines éluées sur la glace en tout temps.
- Superposez doucement la fraction récupérée sur 15 mL du tampon de coussin dans quatre tubes d’ultracentrifugation en polycarbonate. Ajouter un maximum de 15 mL de l’échantillon à 15 mL du tampon coussin. Assurez-vous de bien équilibrer le poids du tube. Abreuvoir les ribosomes par ultracentrifugation à 100000 x g à 4 °C pendant 16 h.
REMARQUE: Assurez-vous qu’aucune fissure n’est présente dans les tubes d’ultracentrifugation. - Nettoyez et réinitialisez la colonne comme suit. Un débit de 5 mL/min fonctionne bien. Placez toutes les entrées dans l’eau et exécutez un lavage à la pompe. Lavez la colonne avec 2 CV d’eau.
- Placez l’entrée dans une solution de NaOH de 0,5 M, effectuez un lavage à la pompe, puis lavez la colonne avec 3 CV de NaOH.
- Placez l’entrée dans l’eau, effectuez un lavage à la pompe, puis lavez la colonne dans 2 CV d’eau.
- Placez l’entrée dans une solution d’acide acétique de 0,1 M, effectuez un lavage à la pompe, puis lavez la colonne avec 3 CV de solution d’acide acétique.
- Laver à la pompe et laver la colonne avec 2 CV d’eau.
- Placez toutes les entrées dans de l’éthanol à 20 % (v/v), exécutez une étape de lavage par pompe et stockez la colonne dans de l’éthanol à 20 % (v/v)) en la lavant avec 3 CV d’une solution d’éthanol à 20 % (v/v).).
REMARQUE: Assurez-vous que le système ne fonctionne jamais à sec ou n’aspire jamais l’air. N’appliquez jamais de tampon directement sur l’éthanol ou d’éthanol sur le tampon. Ajoutez toujours une étape de lavage à l’eau entre les deux, sinon il y a un risque de précipiter l’encrorgement de la colonne. Assurez-vous d’ajouter suffisamment de tubes de prélèvement d’échantillons.
Jour 5:
- Jetez le surnageant et soigneusement, sans déranger la pastille translucide, lavez chaque pastille avec 0,5 mL de tampon ribosome glacé. Répétez cette étape deux fois.
- Resuspendez chacune des pastilles transparentes dans 100 μL de tampon ribosome sur glace à l’aide d’une barre d’agitation magnétique (3 mM de diamètre, 10 mM de longueur) sur un agitateur magnétique en utilisant la vitesse la plus faible possible. Recueillir les ribosomes mis enssusés et laver les tubes avec 50 μL supplémentaires de tampon ribosome.
REMARQUE: La pastille translucide est difficile à voir. Par conséquent, lavez soigneusement la pastille des côtés du tube. - Déterminer la concentration de ribosomes en mesurant l’absorbance à 260 nM de l’échantillon dilué dans un rapport de 1:100 dans le tampon ribosome. Une absorbance de 10 de la solution diluée correspond à 23 μM de solution non diluée comme décrit précédemment16.
- Mettre en œuvre une concentration finale du stock de 10 μM. Pour ajuster la concentration, diluer les ribosomes avec un tampon ribosome ou les concentrer davantage par centrifugation à 14000 x g dans un filtre centrifuge de 3 kDa à 4 °C.
REMARQUE: Pour obtenir une expression optimale du système, effectuer un titrage des ribosomes (rubrique 5.2, tableau supplémentaire 7). - Vérifier la composition du ribosome à l’avec SDS-PAGE (Figure 3A) comme spécifié à la section 1.3.3. Diluer 2,5 μL de l’échantillon avec 7,5 μL d’eau, mélanger avec 10 μL de tampon Laemmli 2x, puis charger 5 μL et 2,5 μL des échantillons dans le gel.
4. Solution énergétique
REMARQUE: La composition de la solution d’énergie 2,5x présentée ici est un exemple de solution qui a bien fonctionné pour une réaction TX-TL standard. Pour optimiser le timing, préparez la solution énergétique au cours du jour 2. La préparation de la solution d’acides aminés est expliquée en détail, suivie de la procédure de préparation finale.
- Solution d’acides aminés
REMARQUE: Préparez la solution d’acides aminés en vrac. La préparation de la quantité de solutions d’acides aminés nécessaires pour un volume final d’au moins 2000 μL réduira l’erreur de pesée pour les quantités par ailleurs très faibles. La concentration globale de la solution d’acides aminés est limitée par la solubilité des acides aminés et les concentrations respectives de la solution stockée. Pour le système PURE standard, préparer une solution avec une concentration finale de 3,25 mM. Utilisez le tableau de calcul de la solution d’acides aminés (Tableau supplémentaire 3) comme modèle. Utilisez la cystéine sous forme de sel pour assurer une solubilité suffisante. Évitez d’utiliser des méthodes de préparation d’acides aminés à base de KOH. Il est possible de peser directement les quantités exactes d’acides aminés dans la solution finale d’acides aminés sans préparer de solution stock pour tous les acides aminés. Cependant, c’est plus difficile et moins précis.- Préparer des solutions sous forme de base pour chaque acide aminé tel que décrit dans le tableau supplémentaire 3, à l’exception de la tyrosine.
REMARQUE: En raison des différentes solubilités des acides aminés dans l’eau, les concentrations suggérées respectives de la solution stock diffèrent. - La masse minimale [mg] fournit la masse minimale approximative requise pour obtenir une quantité suffisante de solution stock pour le volume global cible, à titre de référence.
NOTE: La masse minimale est calculée avec un excédent de 10%. - Pour une préparation plus facile des solutions, ne pesez pas la quantité exacte d’acide aminé, mais plutôt, pour la masse à portée de main, ajustez la quantité d’eau pour atteindre la concentration souhaitée. Calculer la quantité d’eau désionisée (eau à ajouter [μL]) nécessaire, en fonction de la masse réelle remplie (cellules jaune clair) et de la concentration souhaitée à l’aide de la feuille de calcul du tableau supplémentaire 3.
- Solubiliser les solutions de stock d’acides aminés en vortexant jusqu’à ce que tout précipité se soit dissous. Les solutions individuelles d’acides aminés peuvent être conservées à -20 °C pendant plusieurs semaines.
REMARQUE: Certains acides aminés sont difficiles à dissoudre dans l’eau; le processus peut prendre un certain temps. - Peser la quantité exacte de tyrosine nécessaire pour obtenir une concentration finale de 3,25 mM directement dans le tube pour la solution d’acide aminé.
REMARQUE: La tyrosine est très difficile à dissoudre dans l’eau. Ajoutez-le directement au lieu de préparer une solution de stock. - Ajouter les quantités correspondantes de solutions d’acides aminés et d’eau comme indiqué dans le volume final pour ajouter la colonne [μL] (cellules bleu clair) et bien vortexer la solution. Conserver la solution d’acides aminés complète à -80 °C jusqu’à nouvel emploi.
- Préparer des solutions sous forme de base pour chaque acide aminé tel que décrit dans le tableau supplémentaire 3, à l’exception de la tyrosine.
- Préparation de la solution énergétique
NOTE: Au total, la solution énergétique 2,5x contient 0,75 mM de chaque acide aminé, 29,5 mM d’acétate de magnésium, 250 mM de glutamate de potassium, 5 mM d’ATP et de GTP chacun, 2,5 mM de CTP, UTP et TCEP, respectivement, 8,75 mg / mL d’ARNt d’E. coli MRE 600, 50 mM de phosphate de créatine, 0,05 mM d’acide folinique, 5 mM de spermidine et 125 mM d’HEPES. Les nouveaux utilisateurs préparent la solution énergétique en petits lots de 200 μL. Conservez les solutions individuelles préparées conformément au tableau supplémentaire 4 à -20 °C ou -80 °C pour une utilisation ultérieure.- Décongeler toutes les solutions aqueuses mentionnées dans le tableau supplémentaire 5 sur de la glace.
- Entre-temps, préparez les solutions de stock pour les composants restants énumérés dans le tableau supplémentaire 4. Gardez toutes les solutions sur la glace après la préparation.
REMARQUE: Ajouter 500 μL de RNase et d’eau sans DNase directement dans le flacon pour dissoudre les ARNt lyophilisés. Bien mélanger par vortex doux; limiter le pipetage pour éviter d’introduire des RNases. - Ajouter les volumes calculés(tableau supplémentaire 5)de solutions d’origine et d’eau et bien mélanger à l’aide d’un vortex. Gardez la solution sur la glace en tout temps.
- Mesurer le pH de la solution en pipetant 1 μL sur une bande de pH, pour s’assurer que le pH de la solution est neutre.
- Aliquoter la solution énergétique à 50-100 μL par tube sur de la glace et la conserver à -80 °C jusqu’à une utilisation ultérieure. Tout en aliquote, vortex le stock principal fréquemment pour empêcher les composants de précipiter.
REMARQUE: En option, effectuez un test d’activité de la solution énergétique nouvellement fabriquée par rapport à des solutions énergétiques commerciales, par exemple la solution A dans PURExpress. Si une performance significativement inférieure du système avec la solution énergétique est observée, l’optimisation des concentrations d’ions, en particulier les ions magnésium, par titrage (5-20 mM) peut être avantageuse.
5. Réaction OnePot PURE
- Modèle d’ADN
REMARQUE: Les protéines codées en aval du promoteur T7 peuvent être exprimées en PURE à partir d’ADN linéaire ou circulaire. En générant un modèle d’ADN linéaire à l’aide de l’extension PCR, les étapes de clonage fastidieuses peuvent être omises. Les modèles linéaires de cette étude ont été générés par PCR comme décrit ci-dessous, en utilisant une ADN polymérase haute fidélité(Table des matériaux). Les séquences d’amorce, les températures de fusion et les réglages du thermocycleur utilisés dans cette étude sont précisés dans le tableau supplémentaire 6. La préparation du modèle d’ADN n’est pas incluse dans l’horaire quotidien.- Mettre en place une réaction PCR comme recommandé par le fournisseur de polymérase.
REMARQUE: Les paramètres optimisés pour une ADN polymérase haute fidélité(Table des matériaux)sont donnés dans le tableau supplémentaire 6. - Amplifier le gène cible (p. ex., eGFP) en tant que modèle linéaire à partir d’un plasmide ou d’un génome à l’aide d’amorces spécifiques au gène (500 nM) (pour les paramètres, voir le tableau supplémentaire 6).
- L’amplification génère de courtes extensions pour fournir des séquences de recuit pour les étapes d’extension PCR suivantes.
- Vérifiez l’amplicon sur un gel d’agarose pour la taille et la pureté correctes.
- Utilisez l’ADN amplifié comme modèle pour les étapes d’extension suivantes. Mettre en place une réaction d’au moins 50 μL.
- Exécutez 10 cycles d’amplification PCR avec les amorces d’extension (2,5 nM). Après avoir terminé les cycles d’amplification, ajoutez immédiatement les amorces finales (500 nM) à la même réaction et exécutez 30 cycles pour amplifier le produit PCR étendu. Retrouvez les températures de fusion et les séquences d’amorce dans le tableau supplémentaire 6.
- Purifiez les fragments d’ADN à l’aide d’un kit de purification de l’ADN et éluez l’ADN dans de l’eau sans nucléase au lieu d’un tampon d’élution contenant de l’EDTA.
- Vérifiez le gabarit linéaire sur un gel d’agarose pour la taille et la pureté correctes.
- Mesurez la concentration d’ADN en ng/μL à l’aide d’un spectrophotomètre UV-Vis.
- Mettre en place une réaction PCR comme recommandé par le fournisseur de polymérase.
- Mise en place de la réaction PURE
REMARQUE: La composition de réaction finale est 1x solution d’énergie, ribosomes sans étiquette ou ribosomes His-tag, protéines OnePot PURE et modèle d’ADN. Le rapport de volume de réaction comprend 40% de solution énergétique, 30% de solution de protéines et de ribosomes, et 30% d’ADN et d’eau. Les volumes de réaction typiques varient entre 5 μL et 25 μL. Quantifier l’expression d’une protéine fluorescente en continu sur un lecteur de plaques. Utilisez un Green Lys in vitro Système de marquage de traduction, qui incorpore des résidus de lysine marqués par fluorescence dans des protéines nouvellement synthétisées, pour vérifier l’expression de protéines non fluorescentes sur un gel SDS-PAGE. Un exemple de modèle de réaction est donné dans Tableau supplémentaire 7 pour aider à établir une réaction d’expression pure sans cellule. Les cellules en jaune indiquent les valeurs d’entrée de l’utilisateur, et les cellules en orange indiquent des réactifs supplémentaires à ajouter éventuellement à la réaction. Gardez les rapports de volume des composants précis pour assurer l’équilibre ionique correct. Par exemple, pour obtenir une concentration plus élevée en protéines, augmentez la concentration de solution protéique OnePot; cependant, n’augmentez pas le volume de solution protéique ajoutée à la réaction.- Remplissez la concentration [ng/μL] et la longueur [paires de bases] de l’ADN dans les cellules jaunes correspondantes dans la feuille de calcul. Utilisez 2 à 10 nM d’ADN pour la réaction.
- Remplissez le volume total de réaction souhaité en μL.
- Retirez les réactifs requis du congélateur et décongelez-les sur de la glace.
REMARQUE: La recongélation des composants est possible sans diminution des fonctionnalités. Cependant, minimisez autant que possible le nombre de cycles de gel-dégel et le temps pendant lequel les échantillons sont stockés sur la glace. - Pipettez les quantités calculées d’eau, d’ADN et de solution d’énergie d’un côté du tube PCR ou d’un coin d’un puits sur la plaque de 384 puits. Ajoutez la quantité requise de tout réactif supplémentaire du même côté. Minimiser le nombre d’échantillons par expérience pour éviter l’évaporation des échantillons et le biais du temps de début expérimental.
REMARQUE: Il est crucial de garder le composant énergétique physiquement séparé des composants protéiques pour éviter la consommation prématurée des sources d’énergie et des rendements plus faibles. - Pipettez les quantités calculées de protéines et de solution de ribosomes de l’autre côté d’un tube PCR ou du coin opposé de la plaque de 384 puits.
REMARQUE: Utilisez des mélanges maîtres dans la mesure du possible pour réduire l’impact des erreurs de pipetage. Après les tests initiaux, les solutions de ribosomes et de protéines peuvent être mélangées et stockées en une seule solution. - Faites tourner pendant une courte période (30 s) pour fusionner les composants de la réaction. Pour éviter l’évaporation lors des expériences de lecture de plaques, ajoutez 35 μL de cire liquide et scellez la plaque avec un scellant transparent (voir Tableau des matériaux).
- Incuber pendant au moins 3 h à 37 °C.
- Pour la lecture sur un lecteur de plaques, mesurez l’intensité de fluorescence à la longueur d’onde requise toutes les 2 minutes (les résultats représentatifs sont montrés à la figure 3B).
- Effectuez les étapes suivantes pour les échantillons étiquetés Green Lys.
- Après l’expression sans cellule, incuber l’échantillon avec 0,16 μg/μL de RNase A pendant 30 min à 37 °C pour éliminer le fond fluorescent du kit d’étiquetage Green Lys.
REMARQUE: Utilisez RNase A, car d’autres types de RNases ne suppriment pas suffisamment bien l’arrière-plan. - Visualisez l’expression de la protéine en exécutant SDS-PAGE comme spécifié à la section 1.3.3. Lavez doucement le gel non coloré dans de l’eau désionisée et imagez-le sur un imageur fluorescent en utilisant une longueur d’onde d’excitation de 488 nm.
- Par la suite, colorez le gel en utilisant les méthodes de coloration conventionnelles de Coomassie. Pour les paramètres appropriés, voir la section 1.3.3.
REMARQUE: Effectuer un titrage de la solution protéique avec la concentration recommandée de ribosomes et, si nécessaire, titrer les ribosomes avec la concentration optimale de protéines OnePot par la suite. Utilisez le kit commercial PURExpress ΔRibosome comme témoin positif. La solution A, le mélange factoriel et la solution de ribosome correspondent respectivement à l’énergie préparée, à la solution protéique OnePot et aux ribosomes purifiés.
Résultats
Le protocole ci-dessus est conçu pour faciliter l’établissement du système TX-TL sans cellule PURE dans n’importe quel laboratoire. Le protocole comprend une description détaillée de la préparation des trois parties distinctes du système PURE: la protéine OnePot, le ribosome et la solution énergétique. Un calendrier quotidien détaillé, qui optimise le flux de travail, est présenté dans le tableau 1. Le flux de travail est optimisé pour la purification des ribosomes marqués his, et les délais peuvent différer légèrement si la purification des ribosomes sans étiquette est effectuée. Une préparation fournit une quantité suffisante de PURE pour un minimum de cinq cent réactions de 10 μL. De plus, les solutions préparées sont stables pendant plus d’un an à -80 °C et peuvent résister à plusieurs cycles de gel-dégel.
Des niveaux de surexpression adéquats pour toutes les souches sont cruciaux pour la fonctionnalité de la solution protéique finale. La figure 1 montre la surexpression réussie dans les 36 souches individuelles utilisées par la suite pour la préparation de protéines OnePot. La variation de l’intensité de la bande des protéines sur-exprimées s’est probablement produite en raison d’un biais dans les volumes de chargement sur le gel SDS-PAGE. Les tailles de protéines attendues sont résumées dans le tableau 2. GlyRS et PheRS sont constitués de deux sous-unités de poids moléculaires différents; les 34 protéines restantes sont constituées d’une seule sous-unité. La clé de la simplicité et de l’efficacité temporelle de ce protocole est l’étape de coculturation et de co-purification(Figure 2). La solution protéique OnePot a été préparée en augmentant le rapport de la souche EF-Tu par rapport à toutes les autres souches d’expression. La composition globale des protéines finales a été analysée par SDS-PAGE (Figure 3A). À partir des gels (voies 2, 3), on remarque que l’EF-Tu (43,3 kDa) est présent à une concentration plus élevée que les autres protéines, comme prévu. Bien que le gel fournisse une bonne première indication des rapports d’expression des protéines, il est difficile de déterminer si et à quel niveau chaque protéine individuelle a été exprimée. Par conséquent, il est fortement recommandé de confirmer la surexpression dans chaque souche avant la coculture, comme indiqué ci-dessus.
Le ribosome e. coli est une machine moléculaire complexe composée de plus de 50 sous-unités protéiques individuelles23. Un spectre d’absorption représentatif à 260 nm pour la purification des ribosomes sans étiquette est représenté à la figure 4; le troisième pic est caractéristique d’une élution réussie des ribosomes. Pour les deux méthodes de purification des ribosomes, le schéma de fonctionnement attendu sur le gel SDS-PAGE (Figure 3A)18 a été observé. Nous avons observé des contaminations pour les deux purifications, bien qu’en petites quantités (<10%). Notamment, différents contaminants étaient présents dans les ribosomes sans étiquette (voies 5, 6) et étiquetés His (voies 11, 12) en raison de la variation de la méthode. Pour référence utilisateur, les gels SDS-PAGE pour les systèmes combinés sont également inclus (voies 8, 9 et 14, 15).
Enfin, les performances des systèmes préparés(Figure 3)utilisant les différentes variantes de ribosomes sont comparées. Les cycles temporels de l’expression in vitro de l’eGFP montrent que les deux systèmes PURE sont fonctionnels et produisent de l’eGFP fluorescente. Cependant, la solution protéique OnePot combinée aux ribosomes marqués His, en utilisant la concentration de ribosomes optimisée par titrage, n’a donné qu’un tiers du niveau d’expression de la version de ribosome non marquée(Figure 3B). Des résultats similaires ont été observés lorsque trois protéines de tailles différentes ont été exprimées et étiquetées à l’aide du système de tRNA in vitro Green Lys (Figure 3C). Comme on l’a vu sur le gel fluorescent, les produits pleine longueur ont été exprimés avec succès dans les deux systèmes; cependant, seulement environ la moitié du niveau d’expression a été atteint avec le système de ribosomes His-tag. En plus du marquage par fluorescence, les bandes attendues pour les trois protéines sont distinguables sur un gel coloré par Coomassie(Figure 3D). Les résultats montrent que le système d’expression introduit, qui peut être préparé en une semaine dans un laboratoire équipé d’un équipement standard, peut être utilisé pour l’expression in vitro de protéines codées en aval du promoteur T7 à partir de modèles linéaires.

Figure 1: Résultats représentatifs du test de surexpression pour toutes les souches d’expression du système PURE. Le nombre et la taille des protéines PURE sont résumés dans le tableau 2. Les nombres de protéines 21, 24 et 27 sont marqués d’une étoile pour une meilleure visualisation. Veuillez cliquer ici pour voir une version agrandie de cette figure.
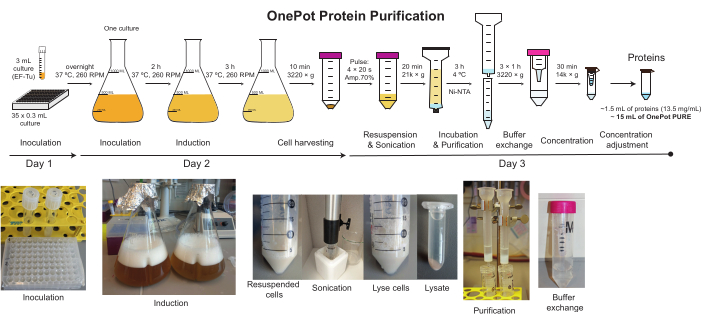
Figure 2: Purification des protéines OnePot. La représentation schématique et les photographies correspondantes de toutes les étapes impliquées dans la production de la solution protéique OnePot. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3: Performances des systèmes préparés utilisant les différentes variantes de ribosomes. (A) Gels SDS-PAGE colorés en bleu de Coomassie de la solution protéique OnePot (voies 2, 3), ribosomes sans étiquette sans solution protéique (voies 5, 6) et avec solution protéique (voies 8, 9), ribosomes marqués His sans solution protéique (voies 11, 12) et avec solution protéique (voies 14, 15). Deux concentrations différentes ont été chargées par échantillon. (B) Comparaison de l’expression eGFP des ribosomes marqués his et des ribosomes sans étiquette. L’intensité de fluorescence de l’expression in vitro de l’eGFP est surveillée au fil du temps pour une réaction PURE à l’aide de ribosomes sans étiquette (1,8 μM, bleu) et de ribosomes marqués His (0,62 μM, rouge). Les concentrations du gabarit linéaire et de la solution protéique OnePot étaient respectivement de 4 nM et 2 mg/mL. Les panneaux (C) et (D) montrent le gel SDS-PAGE de protéines synthétisées dans OnePot avec des ribosomes sans étiquette (1,8 μM, bleu, voies 3, 4, 5) et His-tag (0,62 μM, rouge, voies 6, 7, 8) étiquetés avec un kit de marquage in vitro GreenLys (C) et colorés avec du bleu Coomassie (D), respectivement. Les flèches noires indiquent les bandes attendues de protéines synthétisées : eGFP (26,9 kDa), ArgRS (64,7 kDa), T7 RNAP (98,9 kDa). Les concentrations du gabarit linéaire et de la solution protéique OnePot étaient de 4 nM et 1,6 mg/mL, respectivement. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4: Spectres d’absorbance à 260 nm. Résultats représentatifs des spectres d’absorbance à 260 nm lors de la purification de l’interaction hydrophobe des ribosomes sans étiquette. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau 1 : Un calendrier quotidien optimisé dans le temps pour la préparation de toutes les solutions OnePot PURE. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 2 : Liste des protéines PURES Veuillez cliquer ici pour télécharger ce tableau.
Tableau supplémentaire 1 : Réactifs. Le tableau énumère les concentrations, les volumes et d’autres détails spécifiques des réactifs et des composants utilisés au cours de cette étude. Veuillez cliquer ici pour télécharger ce tableau.
Tableau supplémentaire 2 : Tampons. La feuille de calcul répertorie les compositions tampons exactes pour les purifications de protéines, de ribosomes sans étiquette et de ribosomes His-tag, ainsi que les concentrations des solutions mères utilisées pour leur préparation. En outre, il calcule les quantités requises de composants en fonction du volume de tampon. Veuillez cliquer ici pour télécharger ce tableau.
Tableau supplémentaire 3 : Calculs des acides aminés. La feuille de calcul répertorie les acides aminés et leurs concentrations recommandées en solution stock requises pour la solution énergétique. Il calcule la quantité d’eau à ajouter à chaque acide aminé en fonction de la masse pesée réelle, et calcule également le volume de la solution d’acides aminés à ajouter au mélange final d’acides aminés. Veuillez cliquer ici pour télécharger ce tableau.
Tableau supplémentaire 4 : Solutions de stock pour la solution énergétique. Le tableau répertorie les concentrations et les volumes de solutions de stock nécessaires pour la solution énergétique et indique plus de détails, y compris les conditions de stockage. Veuillez cliquer ici pour télécharger ce tableau.
Tableau supplémentaire 5: Solution énergétique. Le tableau énumère les composants de la solution énergétique et leurs concentrations recommandées. En outre, il calcule les volumes requis à ajouter à la solution finale en fonction de leurs concentrations en solution stock et du volume de la solution énergétique. Veuillez cliquer ici pour télécharger ce tableau.
Tableau supplémentaire 6 : PCR. Le tableau répertorie les séquences et les concentrations des amorces utilisées pour l’extension PCR et indique les températures de fusion et les étapes du thermocycleur optimisées pour une ADN polymérase haute fidélité. Veuillez cliquer ici pour télécharger ce tableau.
Tableau supplémentaire 7: Réaction PURE. La feuille de calcul montre un exemple de configuration d’une réaction PURE. Il énumère les concentrations et les volumes utilisés des composants pour une réaction PURE utilisant des ribosomes sans étiquette ou des ribosomes sans étiquette. De plus, il calcule les rapports de volume pour les titrages de protéines et de ribosomes. Veuillez cliquer ici pour télécharger ce tableau.
Discussion
Le protocole présenté ici décrit une méthode simple, efficace en temps et en argent pour préparer un système d’expression PURE20 polyvalent basé sur la composition standard15. En utilisant le protocole avec les calendriers quotidiens fournis(tableau 1),tous les composants peuvent être préparés en 1 semaine et produire des quantités suffisantes pour des réactions PURE allant jusqu’à cinq cents 10 μL. Étant donné que les protéines utilisées dans ce protocole sont surexprimées à partir de plasmides à copie élevée et ont une faible toxicité pour E. coli, de bons niveaux d’expression sont observés pour toutes les protéines requises (Figure 1). Cela permet d’ajuster facilement les souches, et donc aussi la composition des protéines dans les cocultures, simplement en modifiant les rapports des souches d’inoculation20. Outre les protéines ribosomiques, la concentration d’EF-Tu s’est avérée d’une importance fondamentale pour les rendements d’expression6. En revanche, les changements dans la concentration des autres composants protéiques ont eu un impact relativement faible sur la robustesse du système PURE7,24. Par conséquent, en ajustant le rapport d’inoculation d’EF-Tu par rapport à tous les autres composants, une composition comparable à la composition PURE standard peut être obtenue et un système PURE avec un rendement similaireà 20 peut être atteint. Lors de la préparation de la solution protéique, il est essentiel de s’assurer que toutes les souches se développent bien et surexpriment la protéine codée après induction (Figure 1).
La fonction ribosome est essentielle pour la performance globale du système PURE24. Dans ce protocole, deux méthodes différentes de préparation de la solution de ribosome sont démontrées, à c’est-à-dire la purification du ribosome sans étiquette et la purification du ribosome marquée his. La purification des ribosomes sans étiquette est basée sur la chromatographie d’interaction hydrophobe suivie d’une centrifugation avec un coussin de saccharose, ce qui nécessite l’accès à un système de purification FPLC et à une ultracentrifugeuse15. En revanche, la méthode utilisant les ribosomes18 marqués his et la purification par chromatographie d’affinité par écoulement gravitaire ne nécessite pas d’équipement spécialisé et peut être effectuée dans la plupart des laboratoires. Cette dernière méthode apporte donc des avantages tels que la simplicité et l’accessibilité. Cependant, nous avons observé un rendement de synthèse significativement plus faible lors de l’utilisation des ribosomes marqués His dans le OnePot PURE par rapport à la variante sans étiquette(Figure 3). Selon le type d’application, ce rendement inférieur peut être acceptable.
La solution énergétique fournit les composants de faible poids moléculaire et les ARNt nécessaires pour alimenter les réactions TX-TL in vitro. Ce protocole fournit une recette pour une solution énergétique typique, qui peut être facilement ajustée en fonction des besoins de l’utilisateur. Avec l’ARNt, le NTP et le phosphate de créatine, l’abondance et la concentration des ions Mg2+ ont été cruciales pour la performance globale du système PURE8,car ils sont des cofacteurs critiques pour la transcription et la traduction. Dans certains cas, le titrage des ions peut donc grandement améliorer les performances globales de PURE. L’intégrité de l’ADN est cruciale pour la performance PURE. Ainsi, la séquence vérifie la région promotrice, le site de liaison des ribosomes et le gène cible et s’assure qu’une concentration adéquate d’ADN (<2 nM) aidera à résoudre les problèmes pouvant survenir lors de la mise en place d’une réaction PURE.
Le système PURE est un système TX-TL minimal, et des applications spécifiques peuvent donc nécessiter des ajustements supplémentaires25. Ceux-ci peuvent inclure l’incorporation de différents ARN polymérases9,26,chaperons13et facteurs protéiques tels que EF-P ou ArfA8. Bien que les souches d’expression de ces protéines puissent être incluses dans les cocultures, leur ajout séparé au système préparé peut fournir un meilleur contrôle des niveaux de protéines requis. De plus, l’inclusion de vésicules est essentielle à la production de protéinesmembranaires 10,11. L’oxydation plutôt que la réduction des environnements et une isomérase de liaison disulfure facilitent la formation correcte de liaisons disulfures, qui sont, par exemple, nécessaires pour les protéines sécrétoires12.
Il est essentiel de s’assurer que tout composant supplémentaire n’interfère pas avec la réaction. Les facteurs les plus importants à prendre en compte lors de la mise en place d’une réaction ou de l’ajout d’autres composants sont énumérés ci-dessous. S’assurer que ni des tampons incompatibles ne sont utilisés ni que les concentrations d’ions ne sont perturbées. Évitez autant que possible les solutions contenant du glycérol, des concentrations élevées de potassium, de magnésium, d’ions calcium, d’osmolytes, de pyrophosphate, d’antibiotiques ou d’EDTA. Par exemple, le remplacement d’un tampon d’élution par de l’eau pendant la purification de l’ADN peut être bénéfique car l’EDTA est un additif courant dans ce tampon. Fournir aux solutions des molécules supplémentaires chargées négativement telles que le NTP ou le dNTP nécessite d’ajuster la concentration en magnésium8, car les molécules chargées négativement se comportent comme des agents chélatants et se lient aux molécules chargées positivement. Un pH neutre est idéal pour la réaction. En conséquence, tous les composants doivent être tamponnés au pH correspondant; Ceci est particulièrement important pour les molécules très acides ou basiques telles que les NTP. Enfin, la température et le volume sont des paramètres clés de la réaction. Pour obtenir un bon rendement, il faut mettre en œuvre une température autour de 37 ° C, car des températures inférieures à 34 ° C rédiront considérablement le rendement27.
Il est pertinent de noter qu’avant de préparer le OnePot PURE, il convient de prendre en compte l’application cible et les exigences associées, telles que le volume, la pureté, la facilité de modification et l’inclusion ou l’omission de composants. Pour de nombreuses applications, le système sera un excellent choix, mais d’autres peuvent nécessiter des rendements, une ajustabilité et d’autres facteurs, que le système OnePot ne peut pas fournir. Quoi qu’il en soit, le protocole introduit sera bénéfique pour la préparation de tout système fait maison, car toutes les étapes critiques pour une telle préparation sont résumées ici.
L’un des principaux avantages du système OnePot est sa compatibilité avec le système PURExpress disponible dans le commerce, qui offre la possibilité de tester la fonctionnalité et l’intégrité de tous les composants séparément en remplaçant séquentiellement chaque composant PURExpress par son équivalent OnePot. Les avantages du système OnePot PURE, tels que l’accordabilité et la préparation facile, rapide et rentable, rendront le TX-TL sans cellules accessible à un plus grand nombre de laboratoires dans le monde entier et contribueront à étendre la mise en œuvre de cette puissante plate-forme en biologie synthétique sans cellules.
Déclarations de divulgation
Les auteurs déclarent qu’ils n’ont pas d’intérêts financiers concurrents.
Remerciements
Ces travaux ont été soutenus par le Conseil européen de la recherche dans le cadre de la subvention du programme de recherche et d’innovation Horizon 2020 de l’Union européenne 723106, d’une subvention du Fonds national suisse (182019) et de l’EPFL.
matériels
| Name | Company | Catalog Number | Comments |
| 10x Tris/Glycine/SDS buffer | Bio-Rad Laboratories | 1610732 | |
| 15 mL centrifuge tubes | VWR International | 525-0309 | |
| 384-well Black Assay Plates | Corning | 3544 | |
| 4-20% Mini-PROTEANRTM TGXTM Precast Protein Gels | Bio-Rad Laboratories | 4561096 | |
| 50 mL centrifuge tubes | VWR International | 525-0304 | |
| 96-Well Polypropylene DeepWell plate | Nunc | 260252 | |
| Acetic acid, 99.8 % | Acros | 222140010 | |
| Äkta purifier | GE Healthcare | purification of tag free ribosomes | |
| AMICON ULTRA 0.5 mL - 3 KDa | Merck Millipore | UFC500324 | |
| AMICON ULTRA 15 mL - 3 KDa | Merck Millipore | UFC900324 | |
| Amino acids | Sigma-Aldrich | LAA21-1KT | |
| Ammonium chloride | Sigma-Aldrich | 09718-250G | |
| Ammonium sulfate | Sigma-Aldrich | A4418 | |
| Ampicillin | Condalab | 6801 | |
| BenchMark Fluorescent Protein Standard | ThermoFisher | LC5928 | |
| Breathe-Easy sealing membrane | Diversified Biotech | Z380059-1PAK | |
| Centrifuge tubes polycarbonate | Beckman | 355631 | purification of tag free ribosomes |
| Chill-out Liquid Wax | Bio-Rad Laboratories | CHO1411 | |
| Creatine phosphate | Sigma-Aldrich | 27920 | |
| DNA Clean & Concentrator-25 (Capped) | Zymo | ZYM-D4034-200TS | |
| DTT | SantaCruz Biotech | sc-29089B | |
| Econo-Pac Chromatography Columns | Bio-Rad Laboratories | 7321010 | |
| EDTA (Ethylenediaminetetraacetic acid) | Sigma-Aldrich | 03609-250G | |
| Eppendorf Protein LoBind microcentrifuge tubes | VWR International / Eppendorf | 525-0133 | |
| Falcon 14 mL Round Bottom Polystyrene Test Tube, with Snap Cap | Falcon | 352051 | |
| Flasks, baffled 1000 mL 4 baffles, borosilicate glass | Scilabware | 9141173 | |
| FluoroTect Green Lys in vitro Translation Labeling System | Promega | L5001 | optional |
| Folinic acid | Sigma-Aldrich | PHR1541 | |
| Glycerol | Sigma-Aldrich | G7757-1L | |
| HEPES | Gibco | 15630-056 | |
| HiTrap Butyl HP Column | GE Healthcare | 28411005 | purification of tag free ribosomes |
| IMAC Sepharose 6 Fast Flow | GE Healthcare | 17-0921-07 | |
| Imidazole | Sigma-Aldrich | I2399 | |
| InstantBlue | Expedeon | ISB1L-1L | |
| IPTG (Isopropyl-beta-D-thiogalactoside) | Alfa Aesar | B21149.03 | |
| Laemmli buffer (2x), sample buffer | Sigma-Aldrich | S3401-1VL | |
| Lysogeny broth (LB) media | AppliChem | A0954 | |
| Magnesium acetate | Sigma-Aldrich | M0631 | |
| Magnesium chloride | Honeywell Fluka | 63020-1L | |
| Nickel Sulfate | Alfa Aesar | 15414469 | |
| NTP | ThermoFisher | R0481 | |
| Phusion High-Fidelity DNA Polymerase (2 U/µL) | ThermoFisher | F530S | |
| Potassium chloride | Sigma-Aldrich | P5405-1KG | |
| Potassium glutamate | Sigma-Aldrich | 49601 | |
| PURExpress In Vitro Protein Synthesis Kit | NEB | E6800S | |
| PURExpress Δ Ribosome Kit | NEB | E3313S | |
| Quick Start Bradford 1x Dye Reagent | Bio-Rad Laboratories | 5000205 | |
| Rapid-Flow Sterile Single Use Vacuum Filter Units | ThermoFisher | 564-0020 | |
| RNaseA solution | Promega | A7973 | |
| SealPlate film | Excel Scientific | Z369659-100EA | |
| Sodium hydroxide | Sigma-Aldrich | 6203 | |
| Spermidine | Sigma-Aldrich | S2626 | |
| Sucrose | Sigma-Aldrich | 84097 | |
| TCEP (Tris(2-carboxyethyl)phosphin -hydrochlorid) | Sigma-Aldrich | 646547-10X1mL | |
| Thickwall Polycarbonate Tube | Beckman | 355631 | |
| Trichloroacetic acid | Sigma-Aldrich | T0699 | |
| Tris base | ThermoFisher | BP152-500 | |
| tRNA | Roche | 10109541001 | |
| Ultracentrifuge Optima L-80 | Beckman | purification of tag free ribosomes | |
| Whatman GD/X syringe filters | GE Whatman | WHA68722504 | |
| β-mercaptoethanol | Sigma-Aldrich | M6250-100mL |
Références
- Laohakunakorn, N., et al. Bottom-up construction of complex biomolecular systems with cell-free synthetic biology. Frontiers in Bioengineering and Biotechnology. 8, 213(2020).
- Laohakunakorn, N. Cell-free systems: a proving ground for rational biodesign. Frontiers in Bioengineering and Biotechnology. 8, 788(2020).
- Cho, E., Lu, Y. Compartmentalizing cell-free systems: toward creating life-like artificial cells and beyond. ACS Synthetic Biology. 9 (11), 2881-2901 (2020).
- Gaut, N. J., Adamala, K. P. Reconstituting natural cell elements in synthetic cells. Advanced Biology (Weinh). 5 (3), 2000188(2021).
- Shimizu, Y., et al. Cell-free translation reconstituted with purified components. Nature Biotechnology. 19 (8), 751-755 (2001).
- Li, J., Gu, L., Aach, J., Church, G. M. Improved cell-free RNA and protein synthesis system. PLoS One. 9 (9), 106232(2014).
- Kazuta, Y., Matsuura, T., Ichihashi, N., Yomo, T. Synthesis of milligram quantities of proteins using a reconstituted in vitro protein synthesis system. Journal of Bioscience and Bioengineering. 118 (5), 554-557 (2014).
- Li, J., et al. Dissecting limiting factors of the protein synthesis using recombinant elements (PURE) system. Translation. 5 (1), Austin, Tex. 1327006(2017).
- de Maddalena, L. L., et al. GreA and GreB enhance expression of Escherichia coli RNA polymerase promoters in a reconstituted transcription-translation system. ACS Synthethic Biology. 5 (9), 929-935 (2016).
- Kuruma, Y., Ueda, T. The PURE system for the cell-free synthesis of membrane proteins. Nature Protocols. 10 (9), 1328-1344 (2015).
- Jacobs, M. L., Boyd, M. A., Kamat, N. P. Diblock copolymers enhance folding of a mechanosensitive membrane protein during cell-free expression. Proceedings of the National Academy of Sciences of the Unites States of America. 116 (10), 4031-4036 (2019).
- Shimizu, Y., Kanamori, T., Ueda, T. Protein synthesis by pure translation systems. Methods. 36 (3), San Diego, Calif. 299-304 (2005).
- Niwa, T., Kanamori, T., Ueda, T., Taguchi, H. Global analysis of chaperone effects using a reconstituted cell-free translation system. Proceedings of the National Academy of Sciences of the Unites States of America. 109 (23), 8937-8942 (2012).
- Niwa, T., et al. Large-scale analysis of macromolecular crowding effects on protein aggregation using a reconstituted cell-free translation system. Frontiers in Microbiology. 6, 1113(2015).
- Shimizu, Y., Ueda, T. PURE technology. Methods in molecular biology. 607, Clifton, N.J. 11-21 (2010).
- Horiya, S., Bailey, J. K., Krauss, I. J. Directed evolution of glycopeptides using mRNA display. Methods in Enzymology. 597, 83-141 (2017).
- Villarreal, F., et al. Synthetic microbial consortia enable rapid assembly of pure translation machinery. Nature Chemical Biology. 14 (1), 29-35 (2018).
- Wang, H. H., et al. Multiplexed in vivo His-tagging of enzyme pathways for in vitro single-pot multienzyme catalysis. ACS Synthetic Biology. 1 (2), 43-52 (2012).
- Shepherd, T., et al. De Novo Design and synthesis of a 30-cistron translation-factor module. Nucleic Acids Research. 45 (18), 10895-10905 (2017).
- Lavickova, B., Maerkl, S. J. A Simple, robust, and low-cost method to produce the PURE cell-free system. ACS Synthetic Biology. 8 (2), 455-462 (2019).
- Ederth, J., Mandava, C. S., Dasgupta, S., Sanyal, S. A single-step method for purification of active his-tagged ribosomes from a genetically engineered Escherichia coli. Nucleic Acids Research. 37 (2), 15(2009).
- Gesteland, R. F. Isolation and characterization of ribonuclease I mutants of Escherichia coli. Journal of Molecular Biology. 16 (1), 67-84 (1996).
- Li, J., et al. Cogenerating synthetic parts toward a self-replicating system. ACS Synthetic Biology. 6 (7), 1327-1336 (2017).
- Matsuura, T., Kazuta, Y., Aita, T., Adachi, J., Yomo, T. Quantifying epistatic interactions among the components constituting the protein translation system. Molecular Systems Biology. 5, 297(2009).
- Libicher, K., Hornberger, R., Heymann, M., Mutschler, H. In vitro self-replication and multicistronic expression of large synthetic genomes. Nature Communications. 11 (1), 904(2020).
- Niederholtmeyer, H., Stepanova, V., Maerkl, S. J. Implementation of cell-free biological networks at steady state. Proceedings of the National Academy of Sciences of the United States of America. 110 (40), 15985-15990 (2013).
- Lavickova, B., Laohakunakorn, N., Maerkl, S. J. A partially self-regenerating synthetic cell. Nature Communications. 11 (1), 6340(2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon