Method Article
Sistema sem células OnePot PURE
Neste Artigo
Resumo
Apresentamos um método rápido e econômico para produzir o sistema TX-TL sem células PURE recombinante usando equipamentos de laboratório padrão.
Resumo
O sistema de transcrição pure (síntese proteica definida usando elementos recombinantes) fornece um chassi atraente para biologia sintética livre de células. Infelizmente, os sistemas disponíveis comercialmente são caros, e sua sintonia é limitada. Em comparação, uma abordagem caseira pode ser personalizada com base nas necessidades do usuário. No entanto, a preparação de sistemas caseiros é demorada e árdua devido à necessidade de ribossomos, bem como 36 purificações proteicas de média escala. Simplificar a purificação de proteínas por coculturação e co-purificação permite minimizar os requisitos de tempo e trabalho. Aqui, apresentamos um método fácil, ajustável, de tempo e econômico para produzir todos os componentes do sistema PURE dentro de 1 semana, utilizando equipamentos de laboratório padrão. Além disso, o desempenho do OnePot PURE é comparável aos sistemas disponíveis comercialmente. O método de preparação OnePot PURE expande a acessibilidade do sistema PURE para mais laboratórios devido à sua simplicidade e custo-benefício.
Introdução
Os sistemas de tradução de transcrição sem células (TX-TL) constituem uma plataforma promissora para investigar e engenharia de sistemas biológicos. Eles fornecem condições de reação simplificadas e incapazes, pois não dependem mais de processos de sustentação da vida, incluindo crescimento, homeostase ou mecanismos regulatórios1. Assim, prevê-se que sistemas livres de células contribuam para a investigação de sistemas biomoleculares, ofereçam uma estrutura para testar estratégias racionais de biodesign2, e forneçam um chassi para uma futura célula sintética3,4. O sistema PURE totalmente recombinante oferece um chassi especialmente atraente devido à sua composição definida e mínima, bem como sua ajustabilidade e capacidade de sintonia5.
Desde que o primeiro sistema PURE funcional e totalmente recombinante foi criado em 20015, esforços foram feitos para expandir os limites do sistema e otimizar a composição do sistema para melhorar a produção do sistema6,7,8, permitir a regulação transcricional9,membrana10,11 e síntese de proteínas secretas12, e facilitar a dobra de proteína13,14 . Atualmente, existem três sistemas disponíveis comercialmente: PUREfrex (GeneFrontier), PURExpress (NEB) e Magic PURE (Creative Biolabs). No entanto, esses sistemas são caros, sua composição exata é proprietária e, portanto, desconhecida, e a adaptabilidade é limitada.
Os sistemas PURE preparados internamente provaram ser a opção mais econômica e inuspecível15,16. No entanto, os 37 passos necessários de purificação para proteínas e frações ribossósmos são demorados e tediosos. Várias tentativas foram feitas para melhorar a eficiência da preparação do sistema PURE17,18,19. Recentemente demonstramos que é possível cocultura e co-purificar todas as proteínas não ribossômicas necessárias presentes no sistema PURE. Este método OnePot provou ser econômico e com eficiência de tempo, reduzindo o tempo de preparação de várias semanas para 3 dias úteis. A abordagem gera um sistema PURE com capacidade de produção de proteínas comparável ao sistema PURExpress20disponível comercialmente . Ao contrário das abordagens anteriores para simplificar a preparação pura17,18,19, na abordagem OnePot todas as proteínas ainda são expressas em cepas separadas. Isso permite que o usuário sintonize a composição do sistema OnePot PURE, apenas omitindo ou adicionando cepas específicas ou ajustando os volumes de inoculação, gerando assim sistemas PURE de abandono ou alterando as relações de proteína final, respectivamente.
O protocolo aqui apresentado fornece um método detalhado para a criação do sistema OnePot PURE, como descrito anteriormente20, embora β-mercaptoetanol tenha sido substituído por tris(2-carboxyethyl)fospfina (TCEP). Além disso, dois métodos de purificação ribossosome são descritos: purificação ribossosome tradicional sem tag usando interação hidrofóbica e almofada de sacarose, adaptada de Shimizu et al.15, e purificação ribossoma Ni-NTA baseada em Wang et al.18 e Ederth et al.21, mas significativamente modificada. Este último método facilita ainda a elaboração do sistema PURE e o torna acessível a mais laboratórios, pois apenas equipamentos de laboratório padrão são necessários.
O protocolo experimental resume a preparação de um versátil sistema TX-TL sem células PURA para fornecer uma plataforma simples, tgêneo e econômica sem células, que pode ser preparada usando equipamentos de laboratório padrão dentro de uma semana. Além de introduzir a composição padrão PURE, indicamos como e onde ela pode ser ajustada, com foco principal em etapas críticas no protocolo para garantir a funcionalidade do sistema.
Protocolo
NOTA: Este protocolo descreve a preparação do sistema TX-TL livre de células a partir de componentes recombinantes. Por conveniência, o trabalho é separado em cinco partes. A primeira parte descreve as etapas de preparação, que devem ser feitas antes de iniciar o protocolo. A segunda parte descreve a preparação da solução proteica OnePot. A terceira parte descreve purificações ribossas, a quarta parte detalha a preparação da solução energética, e a última parte fornece um manual para configurar uma reação PURE. Para conveniência, os protocolos são divididos em dias e resumidos em horários diários na Tabela 1. Seguindo o cronograma, todo o sistema pode ser preparado em 1 semana por uma pessoa.
1. Trabalho preliminar
- Prepare os meios de comunicação e os suplementos de mídia da cultura bacteriana, conforme descrito na Tabela Suplementar 1. Prepare e esterilize os materiais necessários, incluindo pontas de pipeta, 96 placas de poço profundo.
- Preparação de tensão
- Transforme as cepas de expressão indicadas na Tabela 2 com os vetores de expressão correspondentes usando o método de choque térmico.
- Adicione plasmídeo purificado às bactérias quimicamente competentes e incubar no gelo por 20-30 minutos.
- Coloque a mistura a 42 °C por 30 s (choque térmico) e coloque-a de volta no gelo por 2 minutos.
- Pipeta 20 μL da bactéria diretamente em placas de ágar contendo ampicilina (AMP) e incubar a 37 °C durante a noite. Armazene as placas a 4 °C por até 1 semana.
- Inocular 3 mL de mídia LB contendo AMP com uma única colônia de bactérias das placas de ágar. Incubar a 37 °C enquanto treme a 260 rpm durante a noite.
- Misture 250 μL da cultura com 250 μL de 50% (v/v) glicerol e armazene a -80 °C.
NOTA: Para uma preparação mais rápida no futuro, armazene as cepas em uma placa de 96 poços e estoques de glicerol.
- Confirme todas as transformações vetoriais por COLÔNIA PCR e sequenciamento. Sequencie o gene, região do promotor e o local de ligação ribossosome.
- Transforme as cepas de expressão indicadas na Tabela 2 com os vetores de expressão correspondentes usando o método de choque térmico.
- Teste de expressão
- Inocula 300 μL de mídia LB contendo AMP com cerca de 1 μL dos estoques de glicerol preparados em uma placa de poço profundo de 1,3 mL. Sele a placa com uma membrana respirável e, em seguida, incubar a 37 °C enquanto treme a 260 rpm durante a noite.
NOTA: Todas as expressões são feitas separadamente neste momento. - Inocular 300 μL de mídia LB fresca contendo AMP com 1 μL das culturas noturnas. Incubar a 37 °C enquanto treme a 260 rpm durante a noite. Após 2h, induza as células com 100 μM de Isopropílico β-D-1-thiogalactopyranoside (IPTG) e cresça por mais 3 h.
- Misture 10 μL da cultura com 10 μL de tampão laemmli 2x e aqueça a 95 °C por 10 min. Gire as amostras por 1 min usando uma centrífuga de mesa e carregue 10 μL do supernatante em um gel PAGE. Execute o gel no buffer Tris/Glycine/SDS a 200 V por 30 min. Enxágüe bem com água deionizada. Cubra o gel com uma mancha de proteína Coomassie e incubar por 1h. Destan o gel na água, se necessário (resultados representativos para o teste de expressão na Figura 1).
NOTA: Use géis PAGE gradientes (4%-15% ou 4%-20%) para obter uma boa separação.
- Inocula 300 μL de mídia LB contendo AMP com cerca de 1 μL dos estoques de glicerol preparados em uma placa de poço profundo de 1,3 mL. Sele a placa com uma membrana respirável e, em seguida, incubar a 37 °C enquanto treme a 260 rpm durante a noite.
- Restauração e limpeza de resina iMAC Sepharose
- Preparação da coluna.
- Misture bem a resina sepharose com vórtice.
- Pipeta a quantidade necessária de resina em uma coluna de fluxo de gravidade vazia.
NOTA: A quantidade de resina necessária varia entre a purificação de seu ribossomo e a purificação de proteínas e é especificada nas respectivas seções. - Lave a resina com 30 mL de água deionizada.
- Proceda com a recarga da coluna conforme especificado na seção 1.4.4.
NOTA: Deixe sempre que todo o líquido passe pela coluna antes de continuar com o próximo passo. No entanto, certifique-se de que a coluna nunca seque. Sempre que passar qualquer líquido pela coluna, certifique-se de parar o fluxo ou continue para o próximo passo assim que o líquido atingir a resina.
- Restauração.
- Lave a coluna com 30 mL de água desionizada.
- Aplique 10 mL de uma solução 0,2 M EDTA e 0,5 M NaCl.
- Adicione 30 mL de uma solução NaCl de 0,5 M.
- Lave a coluna com 50 mL de água deionizada.
- Armazene em 20% (v/v) etanol a 4 °C ou continue com o próximo passo.
- Limpeza.
ATENÇÃO: Use equipamento de proteção.- Lave a coluna com 30 mL de 0,5 M NaOH.
- Lave a coluna com 30 mL de água desionizada.
- Lave a coluna com 30 mL de ácido acético de 0,1 M.
- Lave a coluna com 30 mL de água desionizada.
- Lave a coluna com 30 mL de 70% (v/v) de etanol.
- Lave a coluna com 50 mL de água deionizada.
- Armazene em 20% (v/v) etanol a 4 °C ou continue com o próximo passo.
- Re-carregando.
- Adicione 10 mL de solução de sulfato de níquel de 0,1 M à coluna.
ATENÇÃO: O sulfato de níquel é tóxico. Os resíduos de sulfato de níquel precisam ser descartados com as precauções indicadas pelo fornecedor. - Lave a coluna com 50 mL de água deionizada.
- Armazene em 20% (v/v)) etanol a 4 °C ou continue com o equilíbrio da coluna.
NOTA: Se a coluna estiver armazenada no etanol entre as etapas, certifique-se de remover todos os traços de etanol lavando a coluna com água.
- Adicione 10 mL de solução de sulfato de níquel de 0,1 M à coluna.
- Preparação da coluna.
2. Expressão e purificação da solução de proteína OnePot
NOTA: O protocolo consiste em três partes divididas em dias(Figura 2). Um procedimento de preparação ideal produz 1,5 mL de 13,5 mg/mL solução proteica OnePot, que corresponde a mais de mil reações 10 μL PURE. No entanto, o valor e a concentração ideal da solução variam de lote para lote. Usuários experientes podem realizar várias preparações do OnePot PURE por vez.
Dia 1º:
- Prepare a cultura bacteriana mídia e suplementos de mídia como descrito na Tabela Suplementar 1.
- Prepare e esterilize os materiais necessários, incluindo pontas de pipeta, duas placas de poço profundo de 96 e um frasco erlenmeyer desomado de 1 L.
- Prepare buffers e suplementos conforme descrito na Tabela Suplementar 2. O filtro esteriliza todos os buffers usando filtros de tampão de garrafa (0,45 μm) e armazená-los a 4 °C. Suplemente todos os buffers com 1 mM TCEP logo antes do uso, salvo indicação em contrário.
- Use 2 mL de resina de sepharose para a purificação de proteína OnePot. Prepare a coluna conforme descrito na seção 1.4.
- Para preparar as culturas iniciais, combine 20 mL de mídia LB com 20 μL de AMP. Em uma placa estéril de 96, 1,3 mL de poço profundo, adicione 300 μL da mídia em 35 poços. Inocular cada um deles com sua respectiva cepa, exceto fator de alongamento termo instável (EF-Tu), e selar a placa com uma membrana respirável.
NOTA: Inocular a placa usando um replicador de 96 poços (ver Tabela de Materiais). O volume do poço da placa de poço profundo e o volume da cultura inicial são essenciais. Volumes de mídia maiores ou volumes de poços menores levarão a uma densidade bacteriana diferente devido a inconsistências de aeração. - Para a cultura EF-Tu, inocular 3 mL de mídia LB em um tubo de cultura de 14 mL com uma tampa de snap. Um único 3 mL de cultura para EF-Tu é suficiente para uma cultura de expressão OnePot.
- Incubar a 37 °C enquanto treme a 260 rpm durante a noite.
Dia 2:
NOTA: Realize todas as etapas à temperatura ambiente, a menos que seja indicado o contrário.
- Transfira 500 mL de mídia LB e 500 μL de AMP para o frasco estéril e perplexo.
- Inocular a cultura OnePot PURE com 1675 μL da cultura EF-Tu e 55 μL de cada uma das culturas da placa de poço profundo(Tabela 2).
NOTA: Durante esta etapa, a composição geral da proteína pode ser ajustada afinando as relações de inoculação. Certifique-se de que o volume global de inoculação permanece constante em 3,6 mL.
OPCIONAL: Para confirmar que todas as cepas cresceram durante a noite, meça a densidade óptica das culturas durante a noite em 600 nM (OD600) em uma placa de 96 poços usando um leitor de placas. Use uma diluição de 10x para a medição da densidade óptica. - Incubar a cultura por 2h a 37 °C com um tremor de 260 rpm, ou até que o OD600 da cultura atinja 0,2-0,3.
- Induzir a cultura com 500 μL de 0,1 mM IPTG e crescer por mais 3 h.
- Colher as células por centrifugação a 4 °C e 3220 x g por 10 min e armazenar a pelota de célula a -80 °C até usar mais.
NOTA: Para otimizar o tempo, prepare a solução de energia descrita na seção 4 durante os horários de incubação no dia 2 (Tabela 1).
Dia 3:
- Meça os valores dos buffers necessários para a purificação descritos nas etapas abaixo e adicione o TCEP a todos eles conforme indicado na Tabela Suplementar 2. Armazene os buffers restantes sem TCEP a 4 °C para futuras purificações.
- Equilibre a coluna carregada (seção 2.4) com 30 mL de tampão A. Após 25 mL de tampão A ter passado, feche a coluna a partir da parte inferior. Paralelamente, continue com as etapas 2.15-2.17.
- Descongele as células e use uma pipeta sorológica para resuspensar a pelota celular em 7,5 mL de tampão A.
- Lise as células usando um sonicator de sonda de 130 watts (ver Tabela de Materiais,diâmetro da ponta da sonda: 6 mm) com os seguintes parâmetros: pulso de 4 x 20 s ligado, pulso de 20 s, 70% de amplitude. Se a sônica for bem sucedida, a solução ficará mais escura(Figura 2).
NOTA: Certifique-se de manter as células no gelo durante a sônicação. Coloque a sonda profunda o suficiente na solução sem tocar no tubo. Se uma grande quantidade de espuma for gerada, a transferência de energia será amortecido. Nesse caso, deixe a espuma se acomodar, abaixe a sonda mais profundamente na solução e amplie o tempo de sonicação. - Remova os detritos celulares por centrifugação a 21130 x g por 20 min a 4 °C imediatamente após a sônica. Mantenha o lysate no gelo.
- Adicione o supernatante à coluna equilibrada. Feche a coluna do topo e certifique-se de que não há vazamento. Incubar a coluna por 3 h a 4 °C sob rotação usando um rotador de tubo.
- Componentes elute desvinculados da coluna e lavagem com 25 mL de tampão A.
- Lave a coluna com 25 mL de tampão imidazol de 25 mM (23,95 mL de tampão A e 1,25 mL de tampão B).
- Elute as proteínas com 5 mL de 450 mM tampão imidazol (0,5 mL de tampão A e 4,5 mL de tampão B). Mantenha as proteínas elucidas no gelo o tempo todo.
- Diluir o elunato com 25 mL de tampão HT, mantenha a mistura no gelo. Adicione 15 mL a um filtro centrífugo de 15 mL e concentre-se em um volume de 1,5 mL. Adicione os 15 mL restantes ao filtro com a solução concentrada e concentre-se em 1,5 mL mais uma vez.
- Adicione 10 mL de tampão HT à amostra concentrada e concentre-se a 1 mL. Adicione uma quantidade igual de tampão de estoque B e armazene a -80 °C até que use mais.
NOTA: Uma rodada de troca/concentração leva cerca de 60 min girando a 3220 x g a 4 °C. - Durante a troca de buffer, restaure a coluna conforme especificado na seção 1.4.
Dia 4:
- Meça a concentração de proteínas usando o ensaio de Bradford, conforme descrito pelo fornecedor. Concentre a amostra com um filtro centrífugo de corte de 0,5 mL de 3 kDa a 20 mg/mL.
NOTA: Diluir a solução proteica 25 vezes ou 50 vezes antes das medidas de concentração para evitar a supersaturação do ensaio de Bradford. - Para estabelecer a concentração proteica ideal, realize um teste de expressão nesta fase (seção 5.2) com diferentes concentrações da solução proteica. Para realizar a titulação, mantenha o volume total da solução constante e pipeta a solução de proteína OnePot, incluindo o tampão de estoque B, em cinco proporções diferentes(Tabela Suplementar 7).
- Verifique a composição proteica OnePot PURE usando SDS-PAGE (Figura 3A). Diluir 2,5 μL da amostra com 7,5 μL de água, misture com 10 μL de tampão laemmli 2x e depois carregue 5 μL e 2,5 μL das amostras para o gel. Execute o SDS-PAGE conforme especificado na seção 1.3.3.
- Alíquotar a solução proteica em alíquotas de 50 μL após verificar a expressão e ajustar a concentração. Armazene a solução de proteína OnePot PURE a -80 °C até usar mais.
NOTA: Se suspeita-se que um componente proteico não esteja presente ou esteja presente em uma concentração abaixo do esperado no OnePot PURE, execute as seguintes etapas. - Verifique se a cultura da noite para o dia da respectiva cepa cresceu a uma taxa comparável às outras culturas, realizando medições de densidade óptica (OD600) de todas as culturas.
- Realize um teste de expressão adicional da cepa específica para verificar a expressão da proteína suspeita.
3. Solução ribossoma
NOTA: São introduzidas duas estratégias diferentes de purificação ribossoma, uma para hexahistidina marcada e outra para ribossomos não marcados. A principal vantagem do método de purificação usando sua purificação em uma coluna de fluxo de gravidade Ni-NTA de afinidade padrão é que a purificação é fácil, rápida e não requer equipamentos de laboratório adicionais, como um sistema FPLC e um ultracentrifuge. No entanto, a capacidade de produção de proteínas em reações OnePot PURE é em torno de um terço em comparação com ribossomos sem tag. Portanto, escolha o método de produção de ribossomo com base em se um alto rendimento é importante para a aplicação dada.
-
Sua purificação ribossoma marcada
NOTA: Este protocolo utiliza a cepa E. coli RB1, um presente do Professor Wang (Columbia University, EUA)18. Esta cepa tem uma inserção genômica de uma tag de hexahistidina no termo C da proteína ribossômica 50S (L7/L12), permitindo a purificação usando uma coluna de fluxo de gravidade Ni-NTA. O rendimento usual é de cerca de 0,5 mL de 3,45 μM ribossomos, o que é suficiente para mais de quinhentos 10 μL de reações PURAs.
Dia 1º:
- Prepare a cultura bacteriana mídia e suplementos de mídia como descrito na Tabela Suplementar 1.
- Prepare e esterilize os materiais necessários, incluindo pontas de pipeta, um frasco de 5 L Erlenmeyer e um frasco de 100 mL Erlenmeyer.
- Prepare buffers e suplementos conforme descrito na Tabela Suplementar 2. O filtro esteriliza todos os buffers usando filtros de tampão de garrafa (0,45 μm) e armazená-los a 4 °C.
Dia 2:
- Pipeta 5 mL de resina em uma coluna e prepare a coluna conforme especificado na seção 1.4.
NOTA: Devido ao maior volume da resina, a restauração e purificação demoram significativamente mais. Use uma coluna diferente para purificação ribossa para evitar contaminação cruzada e limpe-a completamente antes da purificação. - Prepare uma cultura noturna de cepa E. coli RB1 inoculando 35 mL de mídia LB em um frasco de 100 mL Erlenmeyer. Incubar a 37 °C enquanto treme a 260 rpm.
Dia 3:
NOTA: Realize todas as etapas à temperatura ambiente, a menos que seja indicado o contrário.
- Adicione 2 L de mídia LB em um frasco estéril de 5 L, inocula com 12 mL da cultura da noite para o dia e, em seguida, incubar por 3-4 h a 37 °C enquanto treme a 260 rpm.
NOTA: Alternativamente, realize a cultura bacteriana em 4 x 500 mL de culturas em frascos perplexos de 1 L. - Pelota as células por centrifugação por 10 min a 3220 x g e 4 °C. Armazene a -80 °C até que use mais.
Dia 4:
- Equilibre a coluna preparada na etapa 3.1.4. com 30 mL de tampão de lise.
- Resuspense a pelota em 20 mL de tampão de lise usando uma pipeta sorológica.
- Lise as células com um sônico de sonda de 130 watts (ver Tabela de Materiais,diâmetro da ponta da sonda: 6 mm) no gelo com os seguintes parâmetros: pulso de 11 x 20 s; 20 s pulso desligado, 70% de amplitude (ver passo 2.16 para detalhes do procedimento).
- Imediatamente após a sônicação, remova os detritos celulares por centrifugação por 20 min a 21130 x g a 4 °C. Mantenha o lysate no gelo.
- Carregue o supernante para as colunas e deixe passar.
- Lave a coluna com as seguintes misturas de lise e tampões de eluição.
- Lave 0: use 30 mL de tampão de lise.
- Lave 1: use 30 mL de imidazol de 5 mM (29 mL de tampão de lise, 1 mL de tampão de eluição).
- Lavagem 2: use 60 mL de imidazol de 25 mM (50 mL de tampão de lise, 10 mL de tampão de eluição).
- Lave 3: use 30 mL de imidazol de 40 mM (22 mL de tampão de lise, 8 mL de tampão de eluição).
- Lave 4: use 30 mL de imidazol de 60 mM (18 mL de tampão de lise, 12 mL de tampão de eluição).
- Elute os ribossomos com 7,5 mL do tampão de eluição. Mantenha as proteínas elucidas no gelo o tempo todo.
- Adicione 22 μL de puro β-mercaptoetanol a 45 mL de tampão ribossomo.
ATENÇÃO: β-mercaptoetanol é tóxico. Tome precauções de segurança e trabalhe em um capô de fumaça. - Adicione o elunato a um filtro centrífugo de 15 mL e concentre-se a 1 mL.
- Adicione 15 mL de tampão ribossomo à amostra concentrada e concentre-se novamente a 1 mL.
NOTA: Repita a etapa anterior duas vezes. - Armazene a -80 °C até que use mais.
NOTA: Uma rodada de troca/concentração leva cerca de 60 min de centrifugação a 3220 x g a 4 °C. - Durante a troca de buffer, restaure a coluna conforme especificado na seção 1.4.
Dia 5:
- Determine a concentração ribossosome medindo a absorvância a 260 nM de uma amostra diluída 1:100 em tampão ribossomo. Um valor de absorção de 10 da solução diluída corresponde a 23 μM de solução não diluída como descrito anteriormente16.
- Implementar uma concentração final de estoque de 3,45 μM. Para ajustar a concentração, diluir os ribossomos com tampão ribossomo ou co concentrar-os ainda mais por centrifugação a 14000 x g em um filtro centrífugo de 3 kDa 0,5 mL a 4 °C.
NOTA: Para alcançar a expressão ideal do sistema, realize uma titulação de concentração ribossosome (seção 5.2, Tabela Suplementar 7). - Verifique a composição ribossa usando SDS-PAGE(Figura 3A)conforme especificado na seção 1.3.3. Diluir 2,5 μL da amostra com 7,5 μL de água, misturar com 10 μL de tampão laemmli 2x e, em seguida, carregar 5 μL e 2,5 μL das amostras no gel.
-
Purificação ribossoma sem tag
NOTA: A purificação ribossosome livre de tags é realizada utilizando um sistema FPLC(Tabela de Materiais) e é baseada na cromatografia de interação hidrofóbica usando colunas butil de 2 x 5 mL(Tabela de Materiais). Embora os ribossomos possam ser purificados de qualquer cepa, o uso da cepa E. coli A19 (E. coli Genetic Resources at Yale CGSC) é vantajoso devido à sua exclusão RNase I22. Realize a purificação a 4 °C em uma sala fria ou em um armário de resfriamento. O rendimento usual é de cerca de 0,5 mL de 10 μM ribossomos, o que corresponde a mais de quinhentos 10 μL de reações PURAs.
Dia 1º:
- Prepare a cultura bacteriana mídia e suplementos de mídia como descrito na Tabela Suplementar 1.
- Prepare e esterilize os materiais necessários, incluindo pontas de pipeta, frasco de 5 L Erlenmeyer e frasco de 100 mL Erlenmeyer.
- Prepare buffers e suplementos conforme descrito na Tabela Suplementar 2. O filtro esteriliza todos os buffers usando filtros de tampão de garrafa (0,45 μm) e armazená-los a 4 °C.
Dia 2:
- Para preparar uma cultura noturna da cepa E. coli A19, inocular 35 mL de mídia LB em um frasco de 100 mL Erlenmeyer. Incubar a 37 °C enquanto treme a 260 rpm.
Dia 3:
- Transfira 2 L de mídia LB para o frasco estéril de 5 L, inocula com 30 mL da cultura da noite para o dia e, em seguida, incubar por 3-4 h a 37 °C enquanto treme a 200 rpm.
- Pelota as células por centrifugação a 4000 x g por 15 min a 4 °C. Resuspense a pelota em 25 mL de tampão de suspensão e armazene a -80 °C até usar mais.
Dia 4:
- Realizar as etapas 3.2.8-3.2.12 em paralelo com as etapas 3.2.13-3.2.19.
- Descongele elise as células usando um sônico de sonda de 130 watts (ver Tabela de Materiais e diâmetro da ponta da sonda: 6 mm) no gelo com os seguintes parâmetros: pulso de 12 x 20 s; Pulso de 20 s desligado, 70% de amplitude (veja detalhes do procedimento passo 2.16).
- Remova imediatamente os detritos celulares por centrifugação a 20000 x g por 20 min a 4 °C.
- Aspire o supernatante e meça o volume. Adicione um volume igual de tampão de suspensão (sal alto) para ajustar a concentração final de sulfato de amônio a 1,5 M e misture bem.
- Remova o precipitado por centrifugação a 20000 x g por 20 min a 4 °C.
- Filtre o supernatante usando um filtro de seringa de membrana polietroésulfone de 0,45 μm antes da purificação do FPLC e colete o filtrado em uma garrafa de vidro de 100 mL. Mantenha o supernatante a 4 °C o tempo todo.
- Configure o sistema FPLC para purificação de cromatografia de interação hidrofóbica usando uma coluna butil dupla (2 x 5 mL) da seguinte forma. Para esta configuração, um volume de coluna (CV) refere-se a um volume de 10 mL.
- Serão necessárias três entradas: duas como linhas tampão e uma como a linha de amostra. Devido às configurações padrão do purificador, é conveniente escolher as linhas A1 e B1 para buffer C e buffer D, respectivamente, e a linha A2 como a linha de amostra. Aplique uma taxa de fluxo padrão de 4 mL/min, exceto para lavagens de bomba (10 mL/min) ou, salvo indicação em contrário.
NOTA: Como o TCEP é um reagente caro, adicione o valor correspondente aos buffers C e D somente após a etapa de equilíbrio. - Realize uma lavagem da bomba do sistema em 20% ((v/v)) etanol para limpar o sistema e remover a contaminação potencial das purificações anteriores. Defina manualmente uma taxa de fluxo de 0,2 mL/min e monte a coluna. Pare o fluxo.
- Execute uma lavagem da bomba do sistema com água. Lave a coluna com 3 CV de água.
- Equilíbrio: coloque as entradas A1 e A2 no buffer C e entrada B1 no buffer D sem TCEP. Execute uma lavagem da bomba e equilibre a coluna com 4 CV de tampão C.
- Adicione TCEP aos buffers C e D.
- Prepare tubos de 15 mL ou tubos de coletor de frações redondos claros ao coletor de frações para coletar frações de eluição de 4-5 mL.
- Carregamento: Coloque a entrada A2 no frasco com a amostra filtrada. Carregue aproximadamente 90% do volume amostral na coluna. Diluir a amostra com 20 mL de tampão C contendo TCEP e carregar 10 mL da amostra na coluna. Repita a etapa de diluição pelo menos duas vezes e carregue o máximo possível de amostra na coluna. É fundamental garantir que nenhum ar seja sugado para dentro da máquina.
- Passo 1 da lavagem: lave com 3 CV de tampão C para remover os componentes não ligados.
- Lave a etapa 2: lave com 5 CV de 80% de tampão C e 20% tampão D.
- Elução: elute o produto aplicando 50% do buffer C e 50% do buffer D, com um volume total de eluição de 5 CV. Colecione esta fração nos tubos coletores.
- La lavagem passo 3: Elute todos os contaminantes fortemente interagindo usando 100% tampão D com um volume total de 5 CV.
- Analise o espectro de absorção da fração amostral em 260 ou 280 nM(Figura 4). O primeiro pico mostra as proteínas não absorvidas elucidadas durante o carregamento e a primeira etapa de lavagem; o segundo pico mostra contaminantes que foram elucidos durante a segunda etapa de lavagem. O terceiro pico monitora o produto final, e o último pico mostra os contaminantes fortemente interagindo. Acumule todas as frações amostrais correspondentes ao terceiro pico para posterior processamento. Mantenha as proteínas elucidas no gelo o tempo todo.
- Sobreponha suavemente a fração recuperada em 15 mL do tampão da almofada em quatro tubos de ultracentrifugação de policarbonato. Adicione um máximo de 15 mL da amostra a 15 mL do tampão da almofada. Certifique-se de equilibrar bem o peso do tubo. Pelle os ribossomos por ultracentrifugação a 100000 x g a 4 °C por 16 h.
NOTA: Certifique-se de que não há rachaduras nos tubos de ultracentrifugação. - Limpe e reinicie a coluna da seguinte forma. Uma vazão de 5 mL/min funciona bem. Coloque todas as entradas na água e execute uma lavagem da bomba. Lave a coluna com 2 CV de água.
- Coloque a entrada em uma solução NaOH de 0,5 M, realize uma lavagem da bomba e, posteriormente, lave a coluna com 3 CV de NaOH.
- Coloque a entrada na água, faça uma lavagem da bomba e depois lave a coluna em 2 CV de água.
- Coloque a entrada em uma solução de ácido acético de 0,1 M, realize uma lavagem da bomba e, posteriormente, lave a coluna com 3 CV de solução de ácido acético.
- Lave a bomba e lave a coluna com 2 CV de água.
- Coloque todas as entradas em 20% (v/v)) etanol, execute uma etapa de lavagem da bomba e armazene a coluna em 20% (v/v)) etanol lavando-a com 3 CV de uma solução de etanol de 20% ((v/v)).
NOTA: Certifique-se de que o sistema nunca seque ou suga o ar. Nunca aplique tampão diretamente ao etanol, ou etanol para tampão. Adicione sempre um passo de lavagem de água no meio, pois caso contrário, há o risco de precipitar entupimento da coluna. Certifique-se de adicionar tubos de coleta de amostras suficientes.
Dia 5:
- Descarte o supernatante e com cuidado, sem perturbar a pelota translúcida, lave cada pelota com 0,5 mL de tampão ribossomo gelado. Repita este passo duas vezes.
- Resuspenque cada uma das pelotas claras em 100 μL de tampão ribossomo no gelo usando uma barra de agitação magnética (3 mM de diâmetro, 10 mM de comprimento) em um agitador magnético usando a menor velocidade possível. Colete os ribossomos resuspendados e lave os tubos com um adicional de 50 μL de tampão ribossomo.
NOTA: A pelota translúcida é difícil de ver. Portanto, lave cuidadosamente a pelota das laterais do tubo. - Determine a concentração ribossosome medindo a absorvância a 260 nM da amostra diluída a uma razão de 1:100 no tampão ribossomo. Uma absorção de 10 da solução diluída corresponde a 23 μM de solução não diluída como descrito anteriormente16.
- Implementar uma concentração final de estoque de 10 μM. Para ajustar a concentração, diluir os ribossomos com tampão ribossomo ou co concentrar-nos ainda mais por centrifugação a 14000 x g em um filtro centrífuga de 3 kDa a 4 °C.
NOTA: Para obter a expressão ideal do sistema, realize a titulação ribossosome (seção 5.2, Tabela Suplementar 7). - Verifique a composição ribossa com SDS-PAGE(Figura 3A)conforme especificado na seção 1.3.3. Diluir 2,5 μL da amostra com 7,5 μL de água, misturar com 10 μL de tampão laemmli 2x e, em seguida, carregar 5 μL e 2,5 μL das amostras para o gel.
4. Solução energética
NOTA: A composição da solução de energia 2,5x introduzida aqui é um exemplo de uma solução que funcionou bem para uma reação TX-TL padrão. Para otimizar o tempo, prepare a solução de energia durante o dia 2. A preparação da solução de aminoácidos é explicada em detalhes, seguida do procedimento final de preparação.
- Solução de aminoácido
NOTA: Prepare a solução de aminoácidos a granel. Preparar a quantidade de soluções de estoque de aminoácidos necessárias para um volume final de pelo menos 2000 μL reduzirá o erro de pesagem para as quantidades muito pequenas. A concentração global da solução de aminoácidos é limitada pela solubilidade dos aminoácidos e pelas respectivas concentrações de solução de estoque. Para o sistema PURE padrão, prepare uma solução com uma concentração final de 3,25 mM. Use a tabela de cálculo da solução de aminoácidos(Tabela Complementar 3) como modelo. Use cisteína na forma de sal para garantir solubilidade suficiente. Evite usar métodos de preparação de aminoácidos à base de KOH. É possível pesar diretamente as quantidades exatas de aminoácidos na solução final de aminoácidos sem preparar solução de estoque para todos os aminoácidos. No entanto, isso é mais desafiador e menos preciso.- Prepare soluções de estoque para cada aminoácido, conforme descrito na Tabela Suplementar 3,com exceção da tyrosina.
NOTA: Devido às diferentes solubilidades dos aminoácidos na água, as respectivas concentrações sugeridas da solução de estoque diferem. - A massa mínima [mg] fornece a massa mínima aproximada necessária para obter uma quantidade suficiente de solução de estoque para o volume geral do alvo, como referência.
NOTA: A massa mínima é calculada com um superávit de 10%. - Para uma preparação mais fácil das soluções, não pese a quantidade exata de aminoácido, mas, em vez disso, para a massa em mãos, ajuste a quantidade de água para alcançar a concentração desejada. Calcule a quantidade de água deionizada (Água para adicionar [μL]) necessária, com base na massa real preenchida (células amarelas claras) e na concentração desejada utilizando a planilha na Tabela Suplementar 3.
- Solubilize as soluções de estoque de aminoácidos por vórtice até que todos os precipitados tenham se dissolvido. As soluções individuais de estoque de aminoácidos podem ser armazenadas a -20 °C por várias semanas.
NOTA: Alguns aminoácidos são difíceis de dissolver na água; o processo pode levar algum tempo. - Pesar a quantidade exata de tyrosina necessária para obter uma concentração final de 3,25 mM diretamente no tubo para a solução de aminoácidos.
NOTA: A tyrosina é muito difícil de dissolver na água. Adicione-o diretamente em vez de preparar uma solução de estoque. - Adicione as quantidades correspondentes de soluções de estoque de aminoácidos e água conforme indicado no volume final para adicionar a coluna [μL] (células azuis claras) e o vórtice da solução bem. Armazene a solução de aminoácidos completa a -80 °C até que use mais.
- Prepare soluções de estoque para cada aminoácido, conforme descrito na Tabela Suplementar 3,com exceção da tyrosina.
- Preparação da solução energética
NOTA: No total, a solução de energia de 2,5x contém 0,75 mM de cada aminoácido, 29,5 mM de acetato de magnésio, 250 mM de glutamato de potássio, 5 mM de ATP e GTP cada, 2,5 mM de CTP, UTP e TCEP, respectivamente, 8,75 mg/mL de tRNA de E. coli MRE 600, 50 mM de fosfato de creatina, 0,05 mM de ácido foliônico, 5 mM de espermidina e 125 mM de HEPES. Os usuários iniciantes preparam a solução de energia em pequenos lotes de 200 μL. Armazene as soluções individuais preparadas de acordo com a Tabela Suplementar 4 a -20 °C ou -80 °C para uso posterior.- Descongele todas as soluções aquosas mencionadas na Tabela Suplementar 5 no gelo.
- Enquanto isso, prepare as soluções de estoque para os demais componentes listados na Tabela Suplementar 4. Mantenha todas as soluções no gelo após a preparação.
NOTA: Adicione 500 μL de água RNase e DNase diretamente ao frasco para dissolver as tRNAs liofilizadas. Misture bem com vórtice suave; limitar a pipetação para evitar a introdução de RNases. - Adicione os volumes calculados(Tabela Suplementar 5) de soluções de estoque e água e misture bem usando um vórtice. Mantenha a solução no gelo o tempo todo.
- Meça o pH da solução pipetando 1 μL em uma tira de pH, para garantir que o pH da solução seja neutro.
- Aliquot a solução de energia a 50-100 μL por tubo no gelo e armazene a -80 °C até que seja mais utilizado. Durante a aliquotação, o vórtice é o estoque principal com frequência para evitar que os componentes precipitassem.
NOTA: Opcionalmente, realize um teste de atividade da solução energética recém-feita contra soluções comerciais de energia, por exemplo, solução A no PURExpress. Se observar um desempenho significativamente menor do sistema com a solução energética, a otimização das concentrações de íons, especialmente íons de magnésio, por titulação (5-20 mM) pode ser vantajosa.
5. Reação onepot PURE
- Modelo de DNA
NOTA: Proteínas codificadas a jusante do promotor T7 podem ser expressas em PURE a partir de DNA linear ou circular. Ao gerar um modelo de DNA linear usando pcr de extensão, passos de clonagem tediosos podem ser omitidos. Os modelos lineares deste estudo foram gerados por PCR conforme descrito abaixo, utilizando-se uma polimerase de DNA de alta fidelidade(Tabela de Materiais). Sequências de primer, temperaturas de fusão e as configurações do termociclamento utilizadas neste estudo são especificadas na Tabela Suplementar 6. A elaboração do modelo de DNA não está incluída na programação diária.- Configure uma reação PCR conforme recomendado pelo fornecedor de polimerase.
NOTA: Parâmetros otimizados para uma polimerase de DNA de alta fidelidade (Tabela de Materiais) são fornecidos na Tabela Suplementar 6. - Amplie o gene alvo (por exemplo, eGFP) como um modelo linear de um plasmídeo ou genoma usando primers específicos de genes (500 nM) (para os parâmetros, ver Tabela Suplementar 6).
- A amplificação gera extensões curtas para fornecer sequências de ressarcial para as seguintes etapas de extensão pcr.
- Verifique o amplicon em um gel de agarose para obter o tamanho e a pureza corretos.
- Use o DNA amplificado como modelo para as etapas subsequentes de extensão. Configure uma reação de pelo menos 50 μL.
- Execute 10 ciclos de amplificação de PCR com os primers de extensão (2,5 nM). Após completar os ciclos de amplificação, adicione imediatamente os primers finais (500 nM) à mesma reação e execute 30 ciclos para amplificar o produto PCR estendido. Encontre as temperaturas de fusão e as sequências de primer na Tabela Suplementar 6.
- Purifique os fragmentos de DNA usando um kit de purificação de DNA e elute o DNA em água livre de nuclease em vez de EDTA contendo tampão de eluição.
- Verifique o modelo linear em um gel de agarose para obter o tamanho e a pureza corretos.
- Meça a concentração de DNA em ng/μL usando um espectrômetro UV-Vis.
- Configure uma reação PCR conforme recomendado pelo fornecedor de polimerase.
- Configurando a reação PURE
NOTA: A composição final da reação é a solução de energia 1x, ribossomos sem tag ou ribossomos his-tag, proteínas OnePot PURE e modelo de DNA. A razão de volume de reação compreende 40% de solução de energia, 30% de proteína e solução ribossoma, e 30% de DNA e água. Os volumes típicos de reação variam entre 5 μL e 25 μL. Quantifique a expressão de uma proteína fluorescente continuamente em um leitor de placas. Use um Green Lys in vitro O Sistema de Rotulagem de Tradução, que incorpora resíduos de lysina fluorescente rotulados em proteínas recém-sintetizadas, para verificar a expressão de proteínas não fluorescentes em um gel SDS-PAGE. Um modelo de reação de exemplo é dado em Tabela Suplementar 7 para ajudar a estabelecer uma reação de expressão pura de livre célula. As células em amarelo indicam valores de entrada do usuário, e as células em laranja indicam reagentes adicionais a serem adicionados opcionalmente à reação. Mantenha as relações de volume dos componentes precisas para garantir o equilíbrio correto do íon. Por exemplo, para obter uma maior concentração proteica, aumente a concentração de solução de proteína OnePot; no entanto, não aumente o volume de solução proteica adicionado à reação.- Preencha a concentração [ng/μL] e o comprimento [pares de base] do DNA nas células amarelas correspondentes na planilha. Use 2-10 nM de DNA para a reação.
- Preencha o volume total de reação desejado em μL.
- Retire os reagentes necessários do congelador e descongele-os no gelo.
NOTA: O reemcongelamento dos componentes é possível sem uma diminuição na funcionalidade. No entanto, minimize o número de ciclos de congelamento e o tempo que as amostras são armazenadas no gelo tanto quanto possível. - Pipeta as quantidades calculadas de água, DNA e solução de energia para um lado do tubo PCR ou um canto de um poço na placa de 384 poços. Adicione a quantidade necessária de qualquer reagente adicional do mesmo lado. Minimize o número de amostras por experimento para evitar a evaporação da amostra e o viés experimental de início.
NOTA: É crucial manter o componente energético fisicamente separado dos componentes proteicos para evitar o consumo prematuro das fontes de energia e rendimentos mais baixos. - Pipeta as quantidades calculadas de proteína e solução ribossosome para o outro lado de um tubo PCR ou o canto oposto da placa de 384 poços.
NOTA: Use misturas mestras sempre que possível para reduzir o impacto dos erros de pipetação. Após os testes iniciais, as soluções de ribossomo e proteína podem ser misturadas e armazenadas como uma solução. - Gire por um curto período de tempo (30 s) para mesclar os componentes de reação. Para evitar a evaporação durante experimentos com leitor de placas, adicione 35 μL de cera líquida e sele a placa com um selante transparente (ver Tabela de Materiais).
- Incubar pela mínima de 3h a 37 °C.
- Para leitura em uma placa-leitor, meça a intensidade de fluorescência no comprimento de onda necessário a cada 2 minutos (os resultados representativos são mostrados na Figura 3B).
- Execute as seguintes etapas para amostras rotuladas green lys.
- Após a expressão livre de células, incubar a amostra com 0,16 μg/μL de RNase A por 30 min a 37 °C para remover o fundo fluorescente do kit de rotulagem Green Lys.
NOTA: Use RNase A, pois outros tipos de RNases não removem o fundo suficientemente bem. - Visualize a expressão proteica executando ODS-PAGE conforme especificado na seção 1.3.3. Lave o gel não manchado suavemente em água deionizada e imagem-a em um imager fluorescente usando um comprimento de onda de excitação de 488 nm.
- Posteriormente, manche o gel usando métodos convencionais de coloração coomassie. Para os parâmetros adequados, consulte a seção 1.3.3.
NOTA: Realize uma titulação da solução proteica com a concentração de ribossomo recomendada e, se necessário, titule ribossomos com a concentração de proteína OnePot ideal depois. Use o kit comercial PURExpress ΔRibosome como um controle positivo. Solução A, Factor Mix e a solução ribossosome correspondem à energia preparada, à solução proteica OnePot e aos ribossomos purificados, respectivamente.
Resultados
O protocolo acima foi projetado para facilitar o estabelecimento do sistema TX-TL sem células PURA em qualquer laboratório. O protocolo inclui uma descrição detalhada da preparação das três partes distintas do sistema PURE: a proteína OnePot, ribossomo e solução energética. Um cronograma diário detalhado, que otimiza o fluxo de trabalho, é mostrado na Tabela 1. O fluxo de trabalho é otimizado para a purificação de ribossomos marcados por Sua, e os períodos de tempo podem diferir ligeiramente se a purificação ribossomo sem tag for realizada. Uma preparação fornece uma quantidade suficiente de PURO para um mínimo de 500 10 μL reações. Além disso, as soluções preparadas são estáveis por mais de um ano a -80 °C e podem suportar múltiplos ciclos de congelamento.
Níveis adequados de superexpressão para todas as cepas são cruciais para a funcionalidade da solução final de proteína. A Figura 1 mostra uma superexpressão bem sucedida em todas as 36 cepas individuais usadas posteriormente para a preparação da proteína OnePot. A variação das intensidades da banda de proteínas super expressas ocorreu provavelmente devido a um viés no volume de carregamento do gel SDS-PAGE. Os tamanhos de proteína esperados são resumidos na Tabela 2. GlyRS e PheRS consistem em duas subunidades de vários pesos moleculares; as 34 proteínas restantes consistem em uma única subunidade. A chave para a simplicidade e eficácia do tempo deste protocolo é a etapa de cocultura e co-purificação(Figura 2). A solução proteica OnePot foi preparada aumentando a proporção de cepa EF-Tu em relação a todas as outras cepas de expressão. A composição geral das proteínas finais foi analisada pela SDS-PAGE (Figura 3A). Dos géis (faixas 2, 3), é perceptível que o EF-Tu (43,3 kDa) está presente em maior concentração em relação às outras proteínas, como esperado. Embora o gel forneça uma boa primeira indicação das relações de expressão proteica, é difícil determinar se e em que nível cada proteína individual foi expressa. Portanto, é altamente recomendável confirmar a superexpressão em cada cepa antes de coculpe, como mostrado acima.
O E. coli ribosome é uma máquina molecular complexa composta por mais de 50 subunidades proteicas individuais23. Um espectro de absorção representativo a 260 nm para purificação ribossópica sem tag é mostrado na Figura 4; o terceiro pico é característico de eluição ribossópica bem sucedida. Para ambos os métodos de purificação ribossomos, observou-se o padrão de execução esperado no gel SDS-PAGE(Figura 3A)18. Observamos contaminações para ambas as purificações, embora em pequenas quantidades (<10%). Notavelmente, diferentes contaminantes estavam presentes nos ribossomos livres de etiquetas (faixas 5, 6) e sua marcada (faixas 11, 12) ribossomos devido à variação do método. Para referência ao usuário, os géis SDS-PAGE para os sistemas combinados também estão incluídos (faixas 8, 9 e 14, 15).
Por fim, o desempenho dos sistemas preparados(Figura 3)utilizando as diferentes variantes ribossomos são comparados. Os cursos de tempo de expressão in vitro eGFP mostram que ambos os sistemas PURE são funcionais e produzem eGFP fluorescentes. No entanto, a solução proteica OnePot combinada com os ribossomos sua marcada, usando a concentração ribossosome otimizada pela titulação, rendeu apenas um terço do nível de expressão da versão ribossomo não marcada(Figura 3B). Resultados semelhantes foram observados quando três proteínas de diferentes tamanhos foram expressas e rotuladas utilizando-se o sistema de rotulagem in vitro Green Lys tRNA(Figura 3C). Como visto no gel fluorescente, os produtos de comprimento completo foram expressos com sucesso em ambos os sistemas; no entanto, apenas cerca de metade do nível de expressão foi alcançado com o sistema ribossomo His-tag. Além da rotulagem de fluorescência, as bandas esperadas para as três proteínas são distinguíveis em um gel coomassie -manchado(Figura 3D). Os resultados mostram que o sistema de expressão introduzido, que pode ser preparado dentro de uma semana em um laboratório com equipamento padrão, pode ser usado para a expressão in vitro de proteínas codificadas a jusante do promotor T7 a partir de modelos lineares.

Figura 1: Resultados representativos para o teste de superexpressão para todas as cepas de expressão do sistema PURE. Os números e tamanhos de proteínas puras são resumidos na Tabela 2. Os números de proteínas 21, 24 e 27 são marcados com uma estrela para melhor visualização. Clique aqui para ver uma versão maior desta figura.
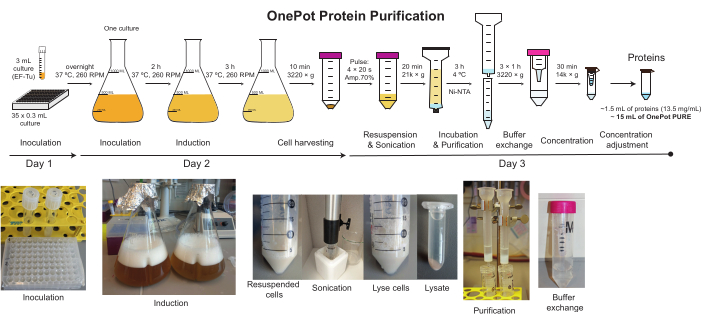
Figura 2: Purificação de proteínas OnePot. A representação esquemática e fotografias correspondentes de todas as etapas envolvidas na produção da solução proteica OnePot. Clique aqui para ver uma versão maior desta figura.

Figura 3: Desempenho dos sistemas preparados utilizando as diferentes variantes ribossomos. (A) Géis SDS-PAGE manchados de azul Coomassie da solução de proteína OnePot (faixas 2, 3), ribossomos sem etiqueta sem solução proteica (pistas 5, 6) e com solução proteica (pistas 8, 9), ribossomos sua marcado sem solução proteica (pistas 11, 12) e com solução proteica (pistas 14, 15). Duas concentrações diferentes foram carregadas por amostra. (B) Comparação da expressão eGFP de ribossomos marcados e ribossomos sem etiqueta. A intensidade de fluorescência da expressão eGFP in vitro é monitorada ao longo do tempo para uma reação PURA usando ribossomos sem tag (1,8 μM, azul) e ribossomos marcados (0,62 μM, vermelho). As concentrações do modelo linear e da solução proteica OnePot foram de 4 nM e 2 mg/mL, respectivamente. Os painéis (C) e(D) mostram o gel SDS-PAGE de proteínas sintetizadas no OnePot com tag-free (1,8 μM, azul, faixas 3, 4, 5) e ribossomos (0,62 μM, vermelho, faixas 6, 7, 8) rotulados com um kit de rotulagem in vitro GreenLys(C) e manchado com azul Coomassie(D),respectivamente. As setas pretas indicam as faixas esperadas de proteínas sintetizadas: eGFP (26,9 kDa), ArgRS (64,7 kDa), T7 RNAP (98,9 kDa). O modelo linear e as concentrações de solução proteica OnePot foram de 4 nM e 1,6 mg/mL, respectivamente. Clique aqui para ver uma versão maior desta figura.

Figura 4: Espectro de absorvância a 260 nm. Resultados representativos de espectros de absorção a 260 nm durante a purificação da interação hidrofóbica de ribossomos livres de etiquetas. Clique aqui para ver uma versão maior desta figura.
Tabela 1: Um cronograma otimizado de tempo diário para a preparação de todas as soluções OnePot PURE. Clique aqui para baixar esta Tabela.
Tabela 2: Lista de proteínas PURA Por favor clique aqui para baixar esta Tabela.
Tabela suplementar 1: Reagentes. A tabela lista concentrações, volumes e outros detalhes específicos dos reagentes e componentes utilizados durante este estudo. Clique aqui para baixar esta Tabela.
Tabela suplementar 2: Tampões. A planilha lista as composições exatas de tampão para proteínas, ribossomos sem etiqueta e purificações ribossomos his-tag, bem como as concentrações das soluções de estoque usadas para sua preparação. Além disso, calcula as quantidades necessárias de componentes com base no volume do buffer. Clique aqui para baixar esta Tabela.
Tabela complementar 3: Cálculos de aminoácidos. A planilha lista os aminoácidos e suas concentrações recomendadas de solução de estoque necessárias para a solução energética. Ele calcula a quantidade de água a ser adicionada a cada aminoácido com base na massa real ponderada, e também calcula o volume da solução de aminoácido a ser adicionada à mistura final de aminoácidos. Clique aqui para baixar esta Tabela.
Tabela Suplementar 4: Soluções de estoque para a solução energética. A tabela lista as concentrações e volumes de soluções de estoque necessárias para a solução de energia e indica mais detalhes, incluindo condições de armazenamento. Clique aqui para baixar esta Tabela.
Tabela Suplementar 5: Solução energética. A tabela lista os componentes da solução energética e suas concentrações recomendadas. Além disso, calcula seus volumes necessários a serem adicionados à solução final com base em suas concentrações de solução de estoque e no volume da solução energética. Clique aqui para baixar esta Tabela.
Tabela Suplementar 6: PCR. A tabela lista sequências e concentrações dos primers usados para a extensão PCR e indica temperaturas de fusão e passos termocicros otimizados para uma polimerase de DNA de alta fidelidade. Clique aqui para baixar esta Tabela.
Tabela suplementar 7: reação PURA. A planilha mostra uma configuração de exemplo de uma reação PURE. Ele lista as concentrações e volumes usados dos componentes para uma reação PURE usando ribossomos sem marca ou ribossomos his-tag. Além disso, calcula as relações de volume para proteínas e titulações ribossóis. Clique aqui para baixar esta Tabela.
Discussão
O protocolo aqui apresentado descreve um método simples, de tempo e econômico para preparar um versátil sistema de expressão PURA20 com base na composição padrão15. Utilizando o protocolo juntamente com os horários diários fornecidos(Tabela 1),todos os componentes podem ser preparados em 1 semana e valores de rendimento suficientes para até quinhentos 10 μL DE reações PURAs. Uma vez que as proteínas utilizadas neste protocolo são superexpressas de plasmídeos de cópia alta e têm baixa toxicidade para E. coli,bons níveis de expressão são observados para todas as proteínas necessárias(Figura 1). Isso permite o fácil ajuste das cepas e, portanto, também a composição proteica em coculturas, simplesmente modificando as proporções das cepas de inoculação20. Além das proteínas ribossômicas, a concentração de EF-Tu mostrou-se de fundamental importância para a expressão6. Em contrapartida, as mudanças na concentração dos outros componentes proteicos tiveram um impacto relativamente baixo na robustez do sistema PURE7,24. Portanto, ajustando a razão de inoculação do EF-Tu em relação a todos os outros componentes, uma composição comparável à composição PURE padrão pode ser alcançada, e um sistema PURE com rendimento semelhante20 pode ser alcançado. No preparo da solução proteica, é fundamental garantir que todas as cepas cresçam bem e expressem demais a proteína codificada após a indução(Figura 1).
A função ribosso é fundamental para o desempenho geral do sistema PURE24. Neste protocolo, dois métodos diferentes para preparar a solução ribossosome são demonstrados, ou seja, purificação ribossoma sem etiqueta e sua marca. A purificação ribossoma sem tag é baseada na cromatografia de interação hidrofóbica seguida de centrifugação com uma almofada de sacarose, que requer acesso a um sistema de purificação FPLC e um ultracentrifuge15. Em contrapartida, o método utilizando ribossomos18 e purificação de cromatografia de afinidade de fluxo gravitacional não requer equipamento especializado e pode ser realizado na maioria dos laboratórios. Este último método, portanto, traz vantagens como simplicidade e acessibilidade. No entanto, observamos um rendimento de síntese significativamente menor ao usar os ribossomos marcados por Sua no OnePot PURE em comparação com a variante sem tag(Figura 3). Com base no tipo de aplicação, esse rendimento mais baixo pode ser aceitável.
A solução de energia fornece os componentes de baixo peso molecular e tRNAs necessários para alimentar reações in vitro TX-TL. Este protocolo fornece uma receita para uma solução energética típica, que pode ser facilmente ajustada com base nas necessidades do usuário. Juntamente com o tRNA, NTP e fosfato de creatina, a abundância e concentração de íons Mg2+ têm sido cruciais para o desempenho geral do sistema PURE8,pois são cofatores críticos para transcrição e tradução. Em alguns casos, a titulação de íons pode, portanto, melhorar muito o desempenho geral do PURE. A integridade do DNA é crucial para o desempenho puro. Assim, a sequência verificando a região do promotor, o local de ligação ribossomo e o gene alvo e garantindo que uma concentração de DNA adequada (<2 nM) ajudará a solucionar problemas que podem surgir ao configurar uma reação PURA.
O sistema PURE é um sistema TX-TL mínimo, e aplicações específicas podem, portanto, exigir ajustes adicionais25. Estes podem incluir a incorporação de diferentes polímerases de RNA9,26, acompanhantes13e fatores proteicos como EF-P ou ArfA8. Embora as cepas de expressão para essas proteínas possam ser incluídas nas coculturas, adicioná-las separadamente ao sistema preparado pode fornecer um melhor controle dos níveis de proteína necessários. Além disso, a inclusão de vesículas é essencial para a produção de proteínas de membrana10,11. Oxidar ao invés de reduzir ambientes e uma isomerase de ligação dissulfeto facilitam a formação adequada de vínculos de dissulfeto, que são, por exemplo, necessárias para proteínas secretas12.
É essencial garantir que quaisquer componentes adicionais não interfiram na reação. Os fatores mais importantes a serem atentos ao configurar uma reação ou adicionar outros componentes estão listados abaixo. Certifique-se de que nem buffers incompatíveis sejam usados nem as concentrações de íons sejam perturbadas. Evite soluções que contenham glicerol, altas concentrações de potássio, magnésio, íons de cálcio, osmólises, pirofosfato, antibióticos ou EDTA, tanto quanto possível. Por exemplo, substituir um tampão de elução por água durante a purificação do DNA pode ser benéfico, pois o EDTA é um aditivo comum neste buffer. Fornecer as soluções com moléculas adicionais com pressão negativa, como NTP ou dNTP, requer o ajuste da concentração de magnésio8,pois as moléculas carregadas negativamente se comportam como agentes queradores e ligam moléculas positivamente carregadas. Um pH neutro é ideal para a reação. Assim, todos os componentes devem ser tamponados para o pH correspondente; isso é especialmente importante para moléculas altamente ácidas ou básicas, como NTPs. Por fim, temperatura e volume são parâmetros-chave para a reação. Para obter um bom rendimento, deve-se implementar uma temperatura em torno de 37 °C, pois temperaturas abaixo de 34 °C reduzirão significativamente o rendimentoem 27.
É relevante notar que antes de preparar o OnePot PURE, deve-se considerar o aplicativo-alvo e os requisitos associados, como volume, pureza, facilidade de modificação e inclusão ou omissão de componentes. Para muitos aplicativos, o sistema será uma excelente escolha, mas outros podem exigir rendimentos, ajustabilidade e outros fatores, que o sistema OnePot não pode fornecer. Independentemente disso, o protocolo introduzido será benéfico para a elaboração de qualquer sistema caseiro, uma vez que todas as etapas críticas para tal preparação são resumidas aqui.
Uma das principais vantagens do sistema OnePot é sua compatibilidade com o sistema PURExpress comercialmente disponível, que oferece a possibilidade de testar a funcionalidade e a integridade de todos os componentes separadamente, substituindo sequencialmente cada componente PURExpress pelo seu equivalente OnePot. As vantagens do sistema OnePot PURE, como a sintonia e preparação fácil, rápida e econômica, tornarão o TX-TL livre de células acessível a mais laboratórios em todo o mundo e contribuirão para a expansão da implementação desta poderosa plataforma em biologia sintética livre de células.
Divulgações
Os autores declaram que não têm interesses financeiros concorrentes.
Agradecimentos
Este trabalho foi apoiado pelo Conselho Europeu de Pesquisa sob o Programa de Pesquisa e Inovação Horizon 2020 da União Europeia, Grant 723106, uma Bolsa da Fundação Nacional de Ciência suíça (182019) e a EPFL.
Materiais
| Name | Company | Catalog Number | Comments |
| 10x Tris/Glycine/SDS buffer | Bio-Rad Laboratories | 1610732 | |
| 15 mL centrifuge tubes | VWR International | 525-0309 | |
| 384-well Black Assay Plates | Corning | 3544 | |
| 4-20% Mini-PROTEANRTM TGXTM Precast Protein Gels | Bio-Rad Laboratories | 4561096 | |
| 50 mL centrifuge tubes | VWR International | 525-0304 | |
| 96-Well Polypropylene DeepWell plate | Nunc | 260252 | |
| Acetic acid, 99.8 % | Acros | 222140010 | |
| Äkta purifier | GE Healthcare | purification of tag free ribosomes | |
| AMICON ULTRA 0.5 mL - 3 KDa | Merck Millipore | UFC500324 | |
| AMICON ULTRA 15 mL - 3 KDa | Merck Millipore | UFC900324 | |
| Amino acids | Sigma-Aldrich | LAA21-1KT | |
| Ammonium chloride | Sigma-Aldrich | 09718-250G | |
| Ammonium sulfate | Sigma-Aldrich | A4418 | |
| Ampicillin | Condalab | 6801 | |
| BenchMark Fluorescent Protein Standard | ThermoFisher | LC5928 | |
| Breathe-Easy sealing membrane | Diversified Biotech | Z380059-1PAK | |
| Centrifuge tubes polycarbonate | Beckman | 355631 | purification of tag free ribosomes |
| Chill-out Liquid Wax | Bio-Rad Laboratories | CHO1411 | |
| Creatine phosphate | Sigma-Aldrich | 27920 | |
| DNA Clean & Concentrator-25 (Capped) | Zymo | ZYM-D4034-200TS | |
| DTT | SantaCruz Biotech | sc-29089B | |
| Econo-Pac Chromatography Columns | Bio-Rad Laboratories | 7321010 | |
| EDTA (Ethylenediaminetetraacetic acid) | Sigma-Aldrich | 03609-250G | |
| Eppendorf Protein LoBind microcentrifuge tubes | VWR International / Eppendorf | 525-0133 | |
| Falcon 14 mL Round Bottom Polystyrene Test Tube, with Snap Cap | Falcon | 352051 | |
| Flasks, baffled 1000 mL 4 baffles, borosilicate glass | Scilabware | 9141173 | |
| FluoroTect Green Lys in vitro Translation Labeling System | Promega | L5001 | optional |
| Folinic acid | Sigma-Aldrich | PHR1541 | |
| Glycerol | Sigma-Aldrich | G7757-1L | |
| HEPES | Gibco | 15630-056 | |
| HiTrap Butyl HP Column | GE Healthcare | 28411005 | purification of tag free ribosomes |
| IMAC Sepharose 6 Fast Flow | GE Healthcare | 17-0921-07 | |
| Imidazole | Sigma-Aldrich | I2399 | |
| InstantBlue | Expedeon | ISB1L-1L | |
| IPTG (Isopropyl-beta-D-thiogalactoside) | Alfa Aesar | B21149.03 | |
| Laemmli buffer (2x), sample buffer | Sigma-Aldrich | S3401-1VL | |
| Lysogeny broth (LB) media | AppliChem | A0954 | |
| Magnesium acetate | Sigma-Aldrich | M0631 | |
| Magnesium chloride | Honeywell Fluka | 63020-1L | |
| Nickel Sulfate | Alfa Aesar | 15414469 | |
| NTP | ThermoFisher | R0481 | |
| Phusion High-Fidelity DNA Polymerase (2 U/µL) | ThermoFisher | F530S | |
| Potassium chloride | Sigma-Aldrich | P5405-1KG | |
| Potassium glutamate | Sigma-Aldrich | 49601 | |
| PURExpress In Vitro Protein Synthesis Kit | NEB | E6800S | |
| PURExpress Δ Ribosome Kit | NEB | E3313S | |
| Quick Start Bradford 1x Dye Reagent | Bio-Rad Laboratories | 5000205 | |
| Rapid-Flow Sterile Single Use Vacuum Filter Units | ThermoFisher | 564-0020 | |
| RNaseA solution | Promega | A7973 | |
| SealPlate film | Excel Scientific | Z369659-100EA | |
| Sodium hydroxide | Sigma-Aldrich | 6203 | |
| Spermidine | Sigma-Aldrich | S2626 | |
| Sucrose | Sigma-Aldrich | 84097 | |
| TCEP (Tris(2-carboxyethyl)phosphin -hydrochlorid) | Sigma-Aldrich | 646547-10X1mL | |
| Thickwall Polycarbonate Tube | Beckman | 355631 | |
| Trichloroacetic acid | Sigma-Aldrich | T0699 | |
| Tris base | ThermoFisher | BP152-500 | |
| tRNA | Roche | 10109541001 | |
| Ultracentrifuge Optima L-80 | Beckman | purification of tag free ribosomes | |
| Whatman GD/X syringe filters | GE Whatman | WHA68722504 | |
| β-mercaptoethanol | Sigma-Aldrich | M6250-100mL |
Referências
- Laohakunakorn, N., et al. Bottom-up construction of complex biomolecular systems with cell-free synthetic biology. Frontiers in Bioengineering and Biotechnology. 8, 213 (2020).
- Laohakunakorn, N. Cell-free systems: a proving ground for rational biodesign. Frontiers in Bioengineering and Biotechnology. 8, 788 (2020).
- Cho, E., Lu, Y. Compartmentalizing cell-free systems: toward creating life-like artificial cells and beyond. ACS Synthetic Biology. 9 (11), 2881-2901 (2020).
- Gaut, N. J., Adamala, K. P. Reconstituting natural cell elements in synthetic cells. Advanced Biology (Weinh). 5 (3), 2000188 (2021).
- Shimizu, Y., et al. Cell-free translation reconstituted with purified components. Nature Biotechnology. 19 (8), 751-755 (2001).
- Li, J., Gu, L., Aach, J., Church, G. M. Improved cell-free RNA and protein synthesis system. PLoS One. 9 (9), 106232 (2014).
- Kazuta, Y., Matsuura, T., Ichihashi, N., Yomo, T. Synthesis of milligram quantities of proteins using a reconstituted in vitro protein synthesis system. Journal of Bioscience and Bioengineering. 118 (5), 554-557 (2014).
- Li, J., et al. Dissecting limiting factors of the protein synthesis using recombinant elements (PURE) system. Translation. 5 (1), 1327006 (2017).
- de Maddalena, L. L., et al. GreA and GreB enhance expression of Escherichia coli RNA polymerase promoters in a reconstituted transcription-translation system. ACS Synthethic Biology. 5 (9), 929-935 (2016).
- Kuruma, Y., Ueda, T. The PURE system for the cell-free synthesis of membrane proteins. Nature Protocols. 10 (9), 1328-1344 (2015).
- Jacobs, M. L., Boyd, M. A., Kamat, N. P. Diblock copolymers enhance folding of a mechanosensitive membrane protein during cell-free expression. Proceedings of the National Academy of Sciences of the Unites States of America. 116 (10), 4031-4036 (2019).
- Shimizu, Y., Kanamori, T., Ueda, T. Protein synthesis by pure translation systems. Methods. 36 (3), 299-304 (2005).
- Niwa, T., Kanamori, T., Ueda, T., Taguchi, H. Global analysis of chaperone effects using a reconstituted cell-free translation system. Proceedings of the National Academy of Sciences of the Unites States of America. 109 (23), 8937-8942 (2012).
- Niwa, T., et al. Large-scale analysis of macromolecular crowding effects on protein aggregation using a reconstituted cell-free translation system. Frontiers in Microbiology. 6, 1113 (2015).
- Shimizu, Y., Ueda, T. PURE technology. Methods in molecular biology. 607, 11-21 (2010).
- Horiya, S., Bailey, J. K., Krauss, I. J. Directed evolution of glycopeptides using mRNA display. Methods in Enzymology. 597, 83-141 (2017).
- Villarreal, F., et al. Synthetic microbial consortia enable rapid assembly of pure translation machinery. Nature Chemical Biology. 14 (1), 29-35 (2018).
- Wang, H. H., et al. Multiplexed in vivo His-tagging of enzyme pathways for in vitro single-pot multienzyme catalysis. ACS Synthetic Biology. 1 (2), 43-52 (2012).
- Shepherd, T., et al. De Novo Design and synthesis of a 30-cistron translation-factor module. Nucleic Acids Research. 45 (18), 10895-10905 (2017).
- Lavickova, B., Maerkl, S. J. A Simple, robust, and low-cost method to produce the PURE cell-free system. ACS Synthetic Biology. 8 (2), 455-462 (2019).
- Ederth, J., Mandava, C. S., Dasgupta, S., Sanyal, S. A single-step method for purification of active his-tagged ribosomes from a genetically engineered Escherichia coli. Nucleic Acids Research. 37 (2), 15 (2009).
- Gesteland, R. F. Isolation and characterization of ribonuclease I mutants of Escherichia coli. Journal of Molecular Biology. 16 (1), 67-84 (1996).
- Li, J., et al. Cogenerating synthetic parts toward a self-replicating system. ACS Synthetic Biology. 6 (7), 1327-1336 (2017).
- Matsuura, T., Kazuta, Y., Aita, T., Adachi, J., Yomo, T. Quantifying epistatic interactions among the components constituting the protein translation system. Molecular Systems Biology. 5, 297 (2009).
- Libicher, K., Hornberger, R., Heymann, M., Mutschler, H. In vitro self-replication and multicistronic expression of large synthetic genomes. Nature Communications. 11 (1), 904 (2020).
- Niederholtmeyer, H., Stepanova, V., Maerkl, S. J. Implementation of cell-free biological networks at steady state. Proceedings of the National Academy of Sciences of the United States of America. 110 (40), 15985-15990 (2013).
- Lavickova, B., Laohakunakorn, N., Maerkl, S. J. A partially self-regenerating synthetic cell. Nature Communications. 11 (1), 6340 (2020).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados