Method Article
OnePot PURE Zellfreies System
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Wir präsentieren eine schnelle und kostengünstige Methode zur Herstellung des rekombinanten PURE zellfreien TX-TL-Systems mit Standard-Laborgeräten.
Zusammenfassung
Das definierte Transkriptions-Translationssystem PURE (Protein synthesis using recombinant elements) bietet ein attraktives Chassis für zellfreie synthetische Biologie. Leider sind kommerziell erhältliche Systeme teuer und ihre Abstimmbarkeit ist begrenzt. Im Vergleich dazu kann ein hausgemachter Ansatz basierend auf den Bedürfnissen der Benutzer angepasst werden. Die Herstellung von hausgemachten Systemen ist jedoch zeitaufwendig und mühsam, da Ribosomen sowie 36 Proteinreinigungen im mittleren Maßstab benötigt werden. Die Rationalisierung der Proteinreinigung durch Kokulturation und Co-Reinigung ermöglicht die Minimierung von Zeit- und Arbeitsaufwand. Hier stellen wir Ihnen eine einfache, einstellbare, zeit- und kostengünstige Methode vor, um alle PURE Systemkomponenten innerhalb von 1 Woche mit Standard-Laborgeräten herzustellen. Darüber hinaus ist die Leistung des OnePot PURE vergleichbar mit handelsüblichen Systemen. Die OnePot PURE-Präparationsmethode erweitert die Zugänglichkeit des PURE-Systems aufgrund seiner Einfachheit und Kosteneffizienz auf mehr Labore.
Einleitung
Zellfreie Transkriptionstranslationssysteme (TX-TL) stellen eine vielversprechende Plattform für die Untersuchung und Das Engineering biologischer Systeme dar. Sie bieten vereinfachte und abstimmbare Reaktionsbedingungen, da sie nicht mehr auf lebenserhaltende Prozesse angewiesen sind, einschließlich Wachstum, Homöostase oder Regulationsmechanismen1. Daher wird erwartet, dass zellfreie Systeme zur Untersuchung biomolekularer Systeme beitragen, einen Rahmen für die Erprobung rationaler Biodesignstrategien2bieten und ein Chassis für eine zukünftige synthetische Zellebereitstellen 3,4. Das voll rekombinante PURE-System bietet aufgrund seiner definierten und minimalen Zusammensetzung sowie seiner Einstellbarkeit und Abstimmbarkeit ein besonders ansprechendes Chassis5.
Seit der Etablierung des ersten funktionellen, vollständig rekombinanten PURE-Systems im Jahr 20015wurden Anstrengungen unternommen, die Systemgrenzen zu erweitern und die Zusammensetzung des Systems zu optimieren, um dieSystemausbeuten 6,7,8zuverbessern , die Transkriptionsregulation 9 ,Membran 10,11 und sekretorische Proteinsynthese12zu ermöglichen und die Proteinfaltung zu erleichtern13,14 . Heutzutage gibt es drei kommerziell erhältliche Systeme: PUREfrex (GeneFrontier), PURExpress (NEB) und Magic PURE (Creative Biolabs). Diese Systeme sind jedoch teuer, ihre genaue Zusammensetzung ist proprietär und daher unbekannt, und die Anpassungsfähigkeit ist begrenzt.
Die im eigenen Haus hergestellten PURE-Systeme erwiesen sich als die kostengünstigste und abstimmbarste Option15,16. Die erforderlichen 37 Reinigungsschritte für Protein- und Ribosomfraktionen sind jedoch zeitaufwendig und mühsam. Es wurden mehrere Versuche unternommen, die Effizienz der PURE-Systemvorbereitung17,18,19zu verbessern. Wir haben kürzlich gezeigt, dass es möglich ist, alle erforderlichen nicht-ribosomalen Proteine, die im PURE-System vorhanden sind, zu kokulturieren und zu reinigen. Diese OnePot-Methode hat sich als kostengünstig und zeiteffizient erwiesen und die Vorbereitungszeit von mehreren Wochen auf 3 Arbeitstage verkürzt. Der Ansatz erzeugt ein PURE-System mit einer Proteinproduktionskapazität, die mit dem kommerziell erhältlichen PURExpress-System20vergleichbar ist. Im Gegensatz zu den bisherigen Ansätzen zur Vereinfachung derPURE-Zubereitung 17,18,19werden beim OnePot-Ansatz alle Proteine noch in getrennten Stämmen exprimiert. Dies ermöglicht es dem Benutzer, die Zusammensetzung des OnePot PURE-Systems zu optimieren, indem er lediglich bestimmte Stämme weglässt oder hinzufügt oder die Impfvolumina anpasst, wodurch Dropout-PURE-Systeme erzeugt werden oder die endgültigen Proteinverhältnisse verändert werden.
Das hier vorgestellte Protokoll bietet eine detaillierte Methode zur Erstellung des OnePot PURE-Systems wie zuvor beschrieben20,obwohl β-Mercaptoethanol durch Tris(2-carboxyethyl)phosphin (TCEP) ersetzt wurde. Darüber hinaus werden zwei Methoden zur Ribosomenreinigung beschrieben: die traditionelle tagfreie Ribosomenreinigung mittels hydrophober Wechselwirkung und Saccharosekissen, angepasst an Shimizu et al.15,und die Ni-NTA-Ribosomenreinigung basierend auf Wang et al.18 und Ederth et al.21, aber signifikant modifiziert. Die letztgenannte Methode erleichtert die Vorbereitung des PURE-Systems weiter und macht es mehr Laboren zugänglich, da nur Standard-Laborgeräte benötigt werden.
Das experimentelle Protokoll fasst die Vorbereitung eines vielseitigen PURE-zellfreien TX-TL-Systems zusammen, um eine einfache, abstimmbare, kostengünstige zellfreie Plattform bereitzustellen, die mit Standard-Laborgeräten innerhalb einer Woche hergestellt werden kann. Neben der Einführung der STANDARDMÄßIGEN PURE-Zusammensetzung geben wir an, wie und wo sie angepasst werden kann, wobei der Schwerpunkt auf kritischen Schritten im Protokoll liegt, um die Funktionalität des Systems sicherzustellen.
Protokoll
HINWEIS: Dieses Protokoll beschreibt die Herstellung eines zellfreien TX-TL-Systems aus rekombinanten Komponenten. Der Einfachheit halber ist die Arbeit in fünf Teile unterteilt. Der erste Teil beschreibt Vorbereitungsschritte, die vor Beginn des Protokolls durchgeführt werden sollten. Der zweite Teil beschreibt die Herstellung der OnePot Proteinlösung. Der dritte Teil beschreibt Ribosomenreinigungen, der vierte Teil beschreibt die Vorbereitung der Energielösung und der letzte Teil enthält eine Anleitung zum Aufbau einer PURE-Reaktion. Der Einfachheit halber sind die Protokolle in Tage unterteilt und in Tabelle 1in Tagesplänen zusammengefasst. Nach dem Zeitplan kann das gesamte System in 1 Woche von einer Person vorbereitet werden.
1. Vorarbeiten
- Bereiten Sie die Bakterienkulturmedien und Medienergänzungen wie in der Ergänzenden Tabelle 1beschrieben vor. Bereiten Sie die erforderlichen Materialien vor und sterilisieren Sie sie, einschließlich Pipettenspitzen, 96 Deep-Well-Platten.
- Dehnungsvorbereitung
- Transformieren Sie die in Tabelle 2 angegebenen Expressionsdehnungen mit den entsprechenden Expressionsvektoren unter Verwendung der Hitzeschockmethode.
- Den chemisch kompetenten Bakterien gereinigtes Plasmid hinzufügen und 20-30 min auf Eis inkubieren.
- Die Mischung für 30 s bei 42 °C (Hitzeschock) aufstellen und dann für 2 min wieder auf Eis legen.
- 20 μL der Bakterien direkt auf Ampicillin (AMP) enthaltende Agarplatten pipettieren und über Nacht bei 37 °C inkubieren. Lagern Sie die Platten bei 4 °C für bis zu 1 Woche.
- Impfen Sie 3 ml LB-Medien, die AMP enthalten, mit einer einzigen Bakterienkolonie aus den Agarplatten. Bei 37 °C inkubieren und dabei über Nacht bei 260 U/min schütteln.
- 250 μL der Kultur mit 250 μL 50% (v/v) Glycerin mischen und bei -80 °C lagern.
HINWEIS: Für eine schnellere Vorbereitung in der Zukunft lagern Sie die Stämme in einer 96-Well-Platte als Glycerinvorräte.
- Bestätigen Sie alle Vektortransformationen durch Kolonie-PCR und Sequenzierung. Sequenzieren Sie das Gen, die Promotorregion und die Ribosomenbindungsstelle.
- Transformieren Sie die in Tabelle 2 angegebenen Expressionsdehnungen mit den entsprechenden Expressionsvektoren unter Verwendung der Hitzeschockmethode.
- Ausdruckstest
- Impfen Sie 300 μL LB-Medien, die AMP enthalten, mit etwa 1 μL der hergestellten Glycerinvorräte in einer 1,3 mL Tiefenbrunnenplatte. Verschließen Sie die Platte mit einer atmungsaktiven Membran und inkubieren Sie dann bei 37 °C, während Sie über Nacht bei 260 U / min schütteln.
HINWEIS: Alle Ausdrücke werden an dieser Stelle separat ausgeführt. - Impfen Sie 300 μL frisches LB-Medium, das AMP enthält, mit 1 μL der Übernachtkulturen. Bei 37 °C inkubieren und dabei über Nacht bei 260 U/min schütteln. Nach 2 h induzieren die Zellen mit 100 μM Isopropyl β-D-1-thiogalactopyranosid (IPTG) und wachsen für weitere 3 h.
- 10 μL der Kultur mit 10 μL 2x Laemmli-Puffer vermischen und 10 min auf 95 °C erhitzen. Drehen Sie die Proben für 1 min mit einer Tischzentrifuge und laden Sie 10 μL des Überstandes auf ein PAGE-Gel. Führen Sie das Gel in Tris / Glycin / SDS-Puffer bei 200 V für 30 min. Spülen Sie es gut mit entionisiertem Wasser ab. Das Gel mit einem Coomassie-Proteinfleck bedecken und 1 h inkubieren. Entbeinen Sie das Gel bei Bedarf in Wasser (repräsentative Ergebnisse für den Expressionstest in Abbildung 1).
HINWEIS: Verwenden Sie Gradientengele (4%-15% oder 4%-20%) PAGE Gele, um eine gute Trennung zu erreichen.
- Impfen Sie 300 μL LB-Medien, die AMP enthalten, mit etwa 1 μL der hergestellten Glycerinvorräte in einer 1,3 mL Tiefenbrunnenplatte. Verschließen Sie die Platte mit einer atmungsaktiven Membran und inkubieren Sie dann bei 37 °C, während Sie über Nacht bei 260 U / min schütteln.
- IMAC Sepharose Harzrestaurierung und -reinigung
- Säulenvorbereitung.
- Mischen Sie das Sepharose-Harz gut durch Wirbeln.
- Pipettieren Sie die erforderliche Menge Harz in eine leere Schwerkraft-Strömungssäule.
HINWEIS: Die benötigte Harzmenge variiert zwischen His-Ribosomen-Reinigung und Proteinreinigung und ist in den jeweiligen Abschnitten angegeben. - Waschen Sie das Harz mit 30 ml entionisiertem Wasser.
- Fahren Sie mit der Säulenaufladung gemäß Abschnitt 1.4.4 fort.
HINWEIS: Lassen Sie immer die gesamte Flüssigkeit durch die Säule passieren, bevor Sie mit dem nächsten Schritt fortfahren. Achten Sie jedoch darauf, dass die Säule niemals trocken läuft. Wenn Sie eine Flüssigkeit durch die Säule laufen lassen, stellen Sie sicher, dass Sie den Fluss stoppen oder mit dem nächsten Schritt fortfahren, sobald die Flüssigkeit das Harz erreicht.
- Restaurierung.
- Waschen Sie die Säule mit 30 ml entionisiertem Wasser.
- Tragen Sie 10 ml einer 0,2 M EDTA- und 0,5 M NaCl-Lösung auf.
- Fügen Sie 30 ml einer 0,5 M NaCl-Lösung hinzu.
- Waschen Sie die Säule mit 50 ml entionisiertem Wasser.
- In 20% (v/v) Ethanol bei 4 °C lagern oder mit dem nächsten Schritt fortfahren.
- Reinigung.
ACHTUNG: Tragen Sie Schutzausrüstung.- Waschen Sie die Säule mit 30 ml 0,5 M NaOH.
- Waschen Sie die Säule mit 30 ml entionisiertem Wasser.
- Waschen Sie die Säule mit 30 ml 0,1 M Essigsäure.
- Waschen Sie die Säule mit 30 ml entionisiertem Wasser.
- Waschen Sie die Säule mit 30 ml 70% (v / v) Ethanol.
- Waschen Sie die Säule mit 50 ml entionisiertem Wasser.
- In 20% (v/v) Ethanol bei 4 °C lagern oder mit dem nächsten Schritt fortfahren.
- Aufladen.
- 10 mL 0,1 M Nickelsulfatlösung in die Säule geben.
VORSICHT: Nickelsulfat ist giftig. Nickelsulfatabfälle müssen mit den vom Lieferanten angegebenen Vorsichtsmaßnahmen entsorgt werden. - Waschen Sie die Säule mit 50 ml entionisiertem Wasser.
- In 20% ((v/v)) Ethanol bei 4 °C lagern oder mit dem Säulengleichgewicht fortfahren.
HINWEIS: Wenn die Säule zwischen den Schritten in Ethanol gelagert wird, stellen Sie sicher, dass Sie alle Spuren von Ethanol entfernen, indem Sie die Säule mit Wasser waschen.
- 10 mL 0,1 M Nickelsulfatlösung in die Säule geben.
- Säulenvorbereitung.
2. Expression und Aufreinigung von OnePot Proteinlösungen
HINWEIS: Das Protokoll besteht aus drei Teilen, die in Tage unterteilt sind (Abbildung 2). Ein ideales Präparationsverfahren ergibt 1,5 ml 13,5 mg/ml OnePot Proteinlösung, was mehr als tausend 10 μL PURE Reaktionen entspricht. Die Menge und die ideale Konzentration der Lösung variieren jedoch von Charge zu Charge. Erfahrene Benutzer können mehrere OnePot PURE-Vorbereitungen gleichzeitig durchführen.
Tag 1:
- Herstellen von Bakterienkulturmedien und Medienergänzungen gemäß der Beschreibung in der ergänzenden Tabelle 1.
- Bereiten Sie die erforderlichen Materialien vor und sterilisieren Sie sie, einschließlich Pipettenspitzen, zwei 96-Deep-Well-Platten und eines 1-l-Erlenmeyerkolbens.
- Bereiten Sie Puffer und Ergänzungen vor, wie in der Ergänzenden Tabelle 2beschrieben. Filter sterilisieren Sie alle Puffer mit Flaschenverschlussfiltern (0,45 μm) und lagern Sie sie bei 4 °C. Ergänzen Sie alle Puffer direkt vor Gebrauch mit 1 mM TCEP, sofern nicht anders angegeben.
- Verwenden Sie 2 ml Sepharoseharz für die OnePot-Proteinreinigung. Bereiten Sie die Spalte wie in Abschnitt 1.4 beschrieben vor.
- Zur Herstellung der Starterkulturen werden 20 ml LB-Medien mit 20 μL AMP kombiniert. In einer sterilen 96, 1,3 ml Deep-Well-Platte werden 300 μL des Mediums in 35 Vertiefungen gegeben. Impfen Sie jeden von ihnen mit seiner jeweiligen Dehnung, mit Ausnahme des Dehnungsfaktors thermo instabil (EF-Tu), und versiegeln Sie die Platte mit einer atmungsaktiven Membran.
HINWEIS: Impfen Sie die Platte mit einem 96-Well-Replikator (siehe Materialtabelle). Das Well-Volumen der Deep-Well-Platte und das Volumen der Starterkultur sind wesentlich. Größere Medienvolumina oder kleinere Vertiefungsvolumina führen aufgrund von Belüftungsinkonsistenzen zu einer unterschiedlichen Bakteriendichte. - Für die EF-Tu-Kultur werden 3 ml LB-Medien in einem 14-ml-Kulturröhrchen mit Schnappverschluss geimpft. Eine einzige 3 ml Kultur für EF-Tu reicht für eine OnePot-Ausdruckskultur aus.
- Bei 37 °C inkubieren und dabei über Nacht bei 260 U/min schütteln.
Tag 2:
HINWEIS: Führen Sie alle Schritte bei Raumtemperatur aus, sofern nicht anders angegeben.
- 500 ml LB-Medien und 500 μL AMP in den sterilen Schalldämpferkolben geben.
- Impfen der OnePot PURE Kultur mit 1675 μL der EF-Tu Kultur und 55 μL jeder der Kulturen aus der Tiefbrunnenplatte (Tabelle 2).
HINWEIS: Während dieses Schritts kann die Gesamtproteinzusammensetzung durch Abstimmung der Impfverhältnisse angepasst werden. Achten Sie darauf, dass das Gesamtimpfvolumen konstant bei 3,6 ml bleibt.
OPTIONAL: Um zu bestätigen, dass alle Stämme über Nacht gewachsen sind, messen Sie die optische Dichte der Übernachtkulturen bei 600 nM (OD600)in einer 96-Well-Platte mit einem Plattenleser. Verwenden Sie für die optische Dichtemessung eine Verdünnung von 10x. - Inkubieren Sie die Kultur für 2 h bei 37 °C mit einem Schütteln von 260 U / min oder bis der OD600 der Kultur 0,2-0,3 erreicht.
- Induzieren Sie die Kultur mit 500 μL 0,1 mM IPTG und wachsen Sie für weitere 3 h.
- Ernten Sie die Zellen durch Zentrifugation bei 4 °C und 3220 x g für 10 min und lagern Sie das Zellpellet bei -80 °C bis zur weiteren Verwendung.
HINWEIS: Um den Zeitpunkt zu optimieren, bereiten Sie die in Abschnitt 4 beschriebene Energielösung während der Inkubationszeiten am Tag 2 vor (Tabelle 1).
Tag 3:
- Messen Sie die Mengen an Puffern, die für die in den folgenden Schritten beschriebene Reinigung benötigt werden, und fügen Sie TCEP zu allen puffern hinzu, wie in der ergänzenden Tabelle 2angegeben. Lagern Sie die restlichen Puffer ohne TCEP bei 4 °C für zukünftige Reinigungen.
- Die geladene Säule (Abschnitt 2.4) wird mit 30 ml Puffer A ausgeglichen. Nachdem 25 ml Puffer A durchgelassen sind, schließen Sie die Säule von unten. Fahren Sie parallel mit den Schritten 2.15-2.17 fort.
- Tauen Sie die Zellen auf und verwenden Sie eine serologische Pipette, um das Zellpellet in 7,5 ml Puffer A zu resuspendieren.
- Lysieren Sie die Zellen mit einem 130-Watt-Sondenschallgerät (siehe Materialtabelle,Sondenspitzendurchmesser: 6 mm) mit folgenden Parametern: 4 x 20 s Puls an, 20 s Puls aus, 70% Amplitude. Wenn die Beschallung erfolgreich ist, wird die Lösung dunkler (Abbildung 2).
HINWEIS: Achten Sie darauf, die Zellen während der Beschallung auf Eis zu halten. Legen Sie die Sonde tief genug in die Lösung, ohne das Röhrchen zu berühren. Wenn eine große Menge Schaum erzeugt wird, wird die Energieübertragung gedämpft. Lassen Sie in diesem Fall den Schaum absetzen, senken Sie die Sonde tiefer in die Lösung und verlängern Sie die Beschallungszeit. - Entfernen Sie die Zelltrümmer durch Zentrifugation bei 21130 x g für 20 min bei 4 °C unmittelbar nach der Beschallung. Halten Sie das Lysat auf Eis.
- Fügen Sie den Überstand der äquilibrierten Spalte hinzu. Schließen Sie die Säule von oben und stellen Sie sicher, dass keine Leckage vorhanden ist. Inkubieren Sie die Säule für 3 h bei 4 °C unter Rotation mit einem Rohrrotator.
- Lösen Sie ungebundene Bauteile aus der Säule und waschen Sie sie mit 25 ml Puffer A.
- Waschen Sie die Säule mit 25 ml 25 mM Imidazolpuffer (23,95 ml Puffer A und 1,25 ml Puffer B).
- Eluieren Sie die Proteine mit 5 ml 450 mM Imidazolpuffer (0,5 ml Puffer A und 4,5 ml Puffer B). Bewahren Sie die eluierten Proteine immer auf Eis auf.
- Verdünnen Sie das Eluat mit 25 ml HT-Puffer, halten Sie die Mischung auf Eis. 15 mL in einen 15 mL Zentrifugalfilter geben und auf ein Volumen von 1,5 mL konzentrieren. Die restlichen 15 ml werden mit der konzentrierten Lösung in den Filter gegeben und erneut auf 1,5 ml konzentriert.
- 10 ml HT-Puffer in die konzentrierte Probe geben und auf 1 ml konzentrieren. Fügen Sie eine gleiche Menge Lagerpuffer B hinzu und lagern Sie bei -80 °C bis zur weiteren Verwendung.
HINWEIS: Eine Runde Austausch/Konzentration dauert ca. 60 min Spinnen bei 3220 x g bei 4 °C. - Stellen Sie während des Pufferaustauschs die Spalte wie in Abschnitt 1.4 beschrieben wieder her.
Tag 4:
- Messen Sie die Proteinkonzentration mit dem Bradford-Assay, wie vom Lieferanten beschrieben. Konzentrieren Sie die Probe mit einem 0,5 ml 3 kDa Cutoff-Zentrifugalfilter auf 20 mg/ml.
HINWEIS: Verdünnen Sie die Proteinlösung vor den Konzentrationsmessungen 25- oder 50-fach, um eine Übersättigung des Bradford-Assays zu vermeiden. - Um die ideale Proteinkonzentration zu ermitteln, wird in diesem Stadium (Abschnitt 5.2) ein Expressionstest mit unterschiedlichen Konzentrationen der Proteinlösung durchgeführt. Um die Titration durchzuführen, halten Sie das Gesamtvolumen der Lösung konstant und pipettieren Sie die OnePot-Proteinlösung, einschließlich Stammpuffer B, in fünf verschiedenen Verhältnissen (Ergänzende Tabelle 7).
- Überprüfen Sie die OnePot PURE Proteinzusammensetzung mit SDS-PAGE (Abbildung 3A). Verdünnen Sie 2,5 μL der Probe mit 7,5 μL Wasser, mischen Sie mit 10 μL 2x Laemmli-Puffer und laden Sie dann 5 μL und 2,5 μL der Proben in das Gel. Führen Sie die SDS-PAGE wie in Abschnitt 1.3.3 beschrieben aus.
- Aliquotieren Sie die Proteinlösung in 50 μL Aliquots, nachdem die Expression überprüft und die Konzentration angepasst wurde. Die OnePot PURE Proteinlösung bis zur weiteren Anwendung bei -80 °C lagern.
HINWEIS: Wenn der Verdacht besteht, dass eine Proteinkomponente nicht oder in einer niedriger als erwarteten Konzentration im OnePot PURE vorhanden ist, führen Sie die folgenden Schritte aus. - Prüfen Sie, ob die Übernachtungskultur des jeweiligen Stammes mit einer vergleichbaren Rate wie die anderen Kulturen gewachsen ist, indem Sie optische Dichtemessungen (OD600) aller Kulturen durchführen.
- Führen Sie einen zusätzlichen Expressionstest des spezifischen Stammes durch, um die Expression des verdächtigen Proteins zu überprüfen.
3. Ribosomenlösung
HINWEIS: Es werden zwei verschiedene Ribosomenreinigungsstrategien eingeführt, eine für Hexahistidin-markierte und eine für nicht markierte Ribosomen. Der Hauptvorteil der Reinigungsmethode mit His-Reinigung auf einer Ni-NTA-Schwerkraftstromsäule mit Standardaffinität besteht darin, dass die Reinigung einfach und schnell ist und keine zusätzliche Laborausrüstung wie ein FPLC-System und eine Ultrazentrifuge erfordert. Die Proteinproduktionskapazität in OnePot PURE-Reaktionen beträgt jedoch etwa ein Drittel im Vergleich zu tagfreien Ribosomen. Wählen Sie daher die Methode zur Ribosomenproduktion basierend darauf, ob eine hohe Ausbeute für die jeweilige Anwendung wichtig ist.
-
Ribosomenreinigung mit seinem Schlagwort
HINWEIS: Dieses Protokoll verwendet den Stamm E. coli RB1, ein Geschenk von Professor Wang (Columbia University, USA)18. Dieser Stamm hat eine genomische Insertion eines Hexahistidin-Tags auf dem C-Terminus des ribosomalen 50S-Proteins (L7 / L12), was eine Reinigung mit einer Ni-NTA-Schwerkraftflusssäule ermöglicht. Die übliche Ausbeute liegt bei etwa 0,5 ml 3,45 μM Ribosomen, was für mehr als fünfhundert 10 μL PURE-Reaktionen ausreicht.
Tag 1:
- Herstellen von Bakterienkulturmedien und Medienergänzungen gemäß der Beschreibung in der ergänzenden Tabelle 1.
- Bereiten Sie die erforderlichen Materialien vor und sterilisieren Sie sie, einschließlich Pipettenspitzen, eines 5-Liter-Erlenmeyerkolbens und eines 100-ml-Erlenmeyerkolbens.
- Bereiten Sie Puffer und Ergänzungen vor, wie in der Ergänzenden Tabelle 2beschrieben. Filter sterilisieren Sie alle Puffer mit Flaschenverschlussfiltern (0,45 μm) und lagern Sie sie bei 4 °C.
Tag 2:
- 5 ml Harz werden in eine Säule pipettiert und die Säule gemäß Abschnitt 1.4 vorbereitet.
HINWEIS: Aufgrund des höheren Harzvolumens dauern die Restauration und Reinigung deutlich länger. Verwenden Sie eine andere Säule für die Ribosomenreinigung, um Kreuzkontaminationen zu vermeiden, und reinigen Sie sie vor der Reinigung gründlich. - Bereiten Sie eine Übernachtkultur von E. coli RB1-Stamm vor, indem Sie 35 ml LB-Medien in einem 100-ml-Erlenmeyerkolben impfen. Bei 37 °C inkubieren und dabei bei 260 U/min schütteln.
Tag 3:
HINWEIS: Führen Sie alle Schritte bei Raumtemperatur aus, sofern nicht anders angegeben.
- 2 l LB-Medien in einen 5-Liter-Reaktor geben, mit 12 ml der Übernachtkultur impfen und dann 3-4 h bei 37 °C unter Schütteln mit 260 U/min inkubieren.
HINWEIS: Alternativ können Sie die Bakterienkultivierung in 4 x 500 ml Kulturen in 1 L Prallenkolben durchführen. - Pelletieren Sie die Zellen durch Zentrifugation für 10 min bei 3220 x g und 4 °C. Bis zur weiteren Verwendung bei -80 °C lagern.
Tag 4:
- Die in Schritt 3.1.4 vorbereitete Spalte wird ausgeglichen. mit 30 ml Lysepuffer.
- Resuspendieren Sie das Pellet in 20 ml Lysepuffer mit einer serologischen Pipette.
- Lyse der Zellen mit einem 130-Watt-Sondenschallgerät (siehe Materialtabelle,Sondenspitzendurchmesser: 6 mm) auf Eis mit folgenden Parametern: 11 x 20 s Impuls an; 20 s Puls aus, 70% Amplitude (siehe Schritt 2.16 für Verfahrensdetails).
- Unmittelbar nach der Beschallung entfernen Sie die Zelltrümmer durch Zentrifugation für 20 min bei 21130 x g bei 4 °C. Halten Sie das Lysat auf Eis.
- Laden Sie den Überstand in die Säulen und lassen Sie ihn passieren.
- Waschen Sie die Säule mit den folgenden Mischungen von Lyse- und Elutionspuffern.
- Waschen Sie 0: Verwenden Sie 30 ml Lysepuffer.
- Waschen Sie 1: Verwenden Sie 30 ml 5 mM Imidazol (29 ml Lysepuffer, 1 ml Elutionspuffer).
- Waschen Sie 2: Verwenden Sie 60 ml 25 mM Imidazol (50 ml Lysepuffer, 10 ml Elutionspuffer).
- Waschen Sie 3: Verwenden Sie 30 ml 40 mM Imidazol (22 ml Lysepuffer, 8 ml Elutionspuffer).
- Waschen Sie 4: Verwenden Sie 30 ml 60 mM Imidazol (18 ml Lysepuffer, 12 ml Elutionspuffer).
- Eluieren Sie die Ribosomen mit 7,5 ml des Elutionspuffers. Bewahren Sie die eluierten Proteine immer auf Eis auf.
- Fügen Sie 22 μL reines β-Mercaptoethanol zu 45 ml Ribosomenpuffer hinzu.
ACHTUNG: β-Mercaptoethanol ist giftig. Treffen Sie Sicherheitsvorkehrungen und arbeiten Sie in einem Abzug. - Das Eluat in einen 15-ml-Zentrifugalfilter geben und auf 1 ml konzentrieren.
- 15 ml Ribosomenpuffer in die konzentrierte Probe geben und erneut auf 1 ml konzentrieren.
HINWEIS: Wiederholen Sie den vorherigen Schritt zweimal. - Bis zur weiteren Verwendung bei -80 °C lagern.
HINWEIS: Eine Runde Austausch/Konzentration dauert etwa 60 min Zentrifugation bei 3220 x g bei 4 °C. - Stellen Sie während des Pufferaustauschs die Spalte wie in Abschnitt 1.4 beschrieben wieder her.
Tag 5:
- Bestimmen Sie die Ribosomenkonzentration durch Messung der Absorption bei 260 nM einer Probe, die 1:100 im Ribosomenpuffer verdünnt ist. Ein Absorptionswert von 10 der verdünnten Lösung entspricht 23 μM unverdünnter Lösung wie zuvor beschrieben16.
- Implementieren Sie eine endgültige Stammkonzentration von 3,45 μM. Zur Einstellung der Konzentration verdünnen Sie die Ribosomen mit Ribosomenpuffer oder konzentrieren Sie sie weiter durch Zentrifugation bei 14000 x g in einem 3 kDa 0,5 ml Zentrifugalfilter bei 4 °C.
ANMERKUNG: Um eine optimale Systemexpression zu erreichen, führen Sie eine Ribosomenkonzentrationstitration durch (Abschnitt 5.2, Ergänzende Tabelle 7). - Überprüfen Sie die Ribosomenzusammensetzung mit SDS-PAGE (Abbildung 3A) wie in Abschnitt 1.3.3 angegeben. Verdünnen Sie 2,5 μL der Probe mit 7,5 μL Wasser, mischen Sie sie mit 10 μL 2x Laemmli-Puffer und laden Sie dann 5 μL und 2,5 μL der Proben auf das Gel.
-
Tag-freie Ribosomenreinigung
HINWEIS: Die Tag-freie Ribosomenreinigung erfolgt mit einem FPLC-System (Table of Materials) und basiert auf hydrophober Wechselwirkungschromatographie unter Verwendung von 2 x 5 ml Butylsäulen ( Tableof Materials). Obwohl Ribosomen von jedem Stamm gereinigt werden können, ist die Verwendung des E. coli A19 (E. coli Genetic Resources at Yale CGSC) Stammes aufgrund seiner RNase I Deletion22vorteilhaft. Führen Sie die Reinigung bei 4 °C entweder in einem Kühlraum oder in einem Kühlschrank durch. Die übliche Ausbeute liegt bei etwa 0,5 ml 10 μM Ribosomen, was mehr als fünfhundert 10 μL PURE Reaktionen entspricht.
Tag 1:
- Herstellen von Bakterienkulturmedien und Medienergänzungen gemäß der Beschreibung in der ergänzenden Tabelle 1.
- Bereiten Sie die erforderlichen Materialien vor und sterilisieren Sie sie, einschließlich Pipettenspitzen, 5 L Erlenmeyerkolben und 100 ml Erlenmeyerkolben.
- Bereiten Sie Puffer und Ergänzungen vor, wie in der Ergänzenden Tabelle 2beschrieben. Filter sterilisieren Sie alle Puffer mit Flaschenverschlussfiltern (0,45 μm) und lagern Sie sie bei 4 °C.
Tag 2:
- Um eine Nachtkultur des E. coli A19-Stammes vorzubereiten, werden 35 ml LB-Medien in einem 100-ml-Erlenmeyerkolben geimpft. Bei 37 °C inkubieren und dabei bei 260 U/min schütteln.
Tag 3:
- 2 l LB-Medien werden in den 5-Liter-Sterilkolben überführt, mit 30 ml der Übernachtkultur geimpft und dann 3-4 h bei 37 °C unter Schütteln bei 200 U/min inkubiert.
- Pelletieren Sie die Zellen durch Zentrifugation bei 4000 x g für 15 min bei 4 °C. Das Pellet in 25 mL Suspensionspuffer resuspenieren und bei -80 °C bis zur weiteren Verwendung lagern.
Tag 4:
- Führen Sie die Schritte 3.2.8-3.2.12 parallel zu den Schritten 3.2.13-3.2.19 aus.
- Auftauen und Lysieren der Zellen mit einem 130-Watt-Sondenschallgerät (siehe Materialtabelle und Sondenspitzendurchmesser: 6 mm) auf Eis mit folgenden Parametern: 12 x 20 s Impuls an; 20 s Impuls aus, 70% Amplitude (siehe Schritt 2.16 Verfahrensdetails).
- Entfernen Sie die Zelltrümmer sofort durch Zentrifugation bei 20000 x g für 20 min bei 4 °C.
- Aspirieren Sie den Überstand und messen Sie das Volumen. Fügen Sie ein gleiches Volumen Suspensionspuffer (hohes Salz) hinzu, um die Endkonzentration von Ammoniumsulfat auf 1,5 M einzustellen und gut zu mischen.
- Entfernen Sie den Niederschlag durch Zentrifugation bei 20000 x g für 20 min bei 4 °C.
- Filtern Sie den Überstand vor der FPLC-Reinigung mit einem 0,45 μm Polyethersulfon-Membranspritzenfilter und sammeln Sie das Filtrat in einer 100 ml Glasflasche. Halten Sie den Überstand immer bei 4 °C.
- Richten Sie das FPLC-System für die hydrophobe Interaktionschromatographie-Reinigung mit einer doppelten Butylsäule (2 x 5 ml) wie folgt ein. Für diesen Aufbau bezieht sich ein Spaltenvolumen (CV) auf ein Volumen von 10 ml.
- Es werden drei Einlässe benötigt: zwei als Pufferlinien und eine als Probenlinie. Aufgrund der Standardeinstellungen des Luftreinigers ist es praktisch, die Zeilen A1 und B1 für Puffer C bzw. Puffer D und die Zeile A2 als Probenlinie zu wählen. Wenden Sie eine Standarddurchflussrate von 4 ml/min an, außer bei Pumpenwäschen (10 ml/min) oder wenn nicht anders angegeben.
HINWEIS: Da TCEP ein kostspieliges Reagenz ist, fügen Sie die entsprechende Menge erst nach dem Gleichgewichtsschritt zu den Puffern C und D hinzu. - Führen Sie eine Systempumpenwäsche in 20% ((v/v)) Ethanol durch, um das System zu reinigen und mögliche Verunreinigungen aus früheren Reinigungen zu entfernen. Stellen Sie manuell eine Durchflussrate von 0,2 ml/min ein und montieren Sie die Säule. Stoppen Sie den Fluss.
- Führen Sie eine Systempumpenwäsche mit Wasser durch. Waschen Sie die Säule mit 3 CV Wasser.
- Equilibrierung: Platzieren Sie die Eingänge A1 und A2 in Puffer C und den Einlass B1 in Puffer D ohne TCEP. Führen Sie eine Pumpenwäsche durch und gleichen Sie die Säule mit 4 CV Puffer C aus.
- Fügen Sie TCEP zu den Puffern C und D hinzu.
- Bereiten Sie 15-ml-Rohre oder klare runde Fraktionskollektorrohre zum Fraktionssammler vor, um 4-5 ml Elutionsfraktionen zu sammeln.
- Beladung: Legen Sie den Einlass A2 mit der gefilterten Probe in die Flasche. Laden Sie ca. 90% des Probenvolumens auf die Säule. Verdünnen Sie die Probe mit 20 ml TCEP-haltigem Puffer C und laden Sie 10 ml der Probe auf die Säule. Wiederholen Sie den Verdünnungsschritt mindestens zweimal und laden Sie so viel Probe wie möglich auf die Säule. Es ist wichtig sicherzustellen, dass keine Luft in die Maschine gesaugt wird.
- Waschschritt 1: Waschen sie mit 3 CV Puffer C, um die ungebundenen Komponenten zu entfernen.
- Waschschritt 2: Waschen mit 5 CV von 80% Puffer C und 20% Puffer D.
- Elution: Eluieren Sie das Produkt durch Auftragen von 50% von Puffer C und 50% von Puffer D, mit einem Gesamtelutionsvolumen von 5 CV. Sammeln Sie diese Fraktion in den Kollektorrohren.
- Waschschritt 3: Elkulieren Sie alle stark wechselwirkenden Verunreinigungen mit 100% Puffer D mit einem Gesamtvolumen von 5 CV.
- Analysieren Sie das Absorptionsspektrum der Probenfraktion bei 260 oder 280 nM (Abbildung 4). Der erste Peak zeigt die nicht absorbierten Proteine, die während der Beladung und des ersten Waschschritts eluiert werden; der zweite Peak zeigt Verunreinigungen, die beim zweiten Waschschritt eluiert wurden. Der dritte Peak überwacht das Endprodukt, und der letzte Peak zeigt die stark wechselwirkenden Verunreinigungen. Alle Probenfraktionen, die dem dritten Peak entsprechen, zur Weiterverarbeitung bündeln. Bewahren Sie die eluierten Proteine immer auf Eis auf.
- Legen Sie die zurückgewonnene Fraktion vorsichtig auf 15 ml des Kissenpuffers in vier Polycarbonat-Ultrazentrifugationsrohren auf. Fügen Sie maximal 15 ml der Probe zu 15 ml des Kissenpuffers hinzu. Achten Sie darauf, das Gewicht des Rohres gut auszugleichen. Pelletieren Sie die Ribosomen durch Ultrazentrifugation bei 100000 x g bei 4 °C für 16 h.
HINWEIS: Stellen Sie sicher, dass in den Ultrazentrifugationsrohren keine Risse vorhanden sind. - Reinigen und setzen Sie die Spalte wie folgt zurück. Ein Durchfluss von 5 ml/min funktioniert gut. Legen Sie alle Einlässe ins Wasser und führen Sie eine Pumpenwäsche durch. Waschen Sie die Säule mit 2 CV Wasser.
- Legen Sie den Einlass in eine 0,5 M NaOH-Lösung, führen Sie eine Pumpenwäsche durch und waschen Sie anschließend die Säule mit 3 CV NaOH.
- Legen Sie den Einlass in Wasser, führen Sie eine Pumpenwäsche durch und waschen Sie dann die Säule in 2 CV Wasser.
- Legen Sie den Einlass in eine 0,1 M Essigsäurelösung, führen Sie eine Pumpwäsche durch und waschen Sie anschließend die Säule mit 3 CV Essigsäurelösung.
- Pumpen waschen und waschen Sie die Säule mit 2 CV Wasser.
- Alle Einlässe in 20% ((v/v)) Ethanol geben, einen Pumpenwaschschritt ausführen und die Säule in 20% ((v/v)) Ethanol lagern, indem sie mit 3 CV einer 20% igen ((v/v)) Ethanollösung gewaschen wird.
HINWEIS: Stellen Sie sicher, dass das System niemals trocken läuft oder Luft ansaugt. Tragen Sie niemals Puffer direkt auf Ethanol oder Ethanol auf Puffer auf. Fügen Sie immer einen Wasserwaschschritt dazwischen hinzu, da sonst die Gefahr besteht, dass Ausscheidungen die Säule verstopfen. Stellen Sie sicher, dass Sie genügend Probenentnahmeröhrchen hinzufügen.
Tag 5:
- Entsorgen Sie den Überstand und waschen Sie vorsichtig, ohne das durchscheinende Pellet zu stören, jedes Pellet mit 0,5 ml eiskaltem Ribosomenpuffer. Wiederholen Sie diesen Schritt zweimal.
- Resuspendieren Sie jedes der klaren Pellets in 100 μL Ribosomenpuffer auf Eis mit einem Magnetrührstab (3 mM Durchmesser, 10 mM Länge) auf einem Magnetrührer mit der geringstmöglichen Geschwindigkeit. Sammeln Sie die resuspendierten Ribosomen und waschen Sie die Röhrchen mit zusätzlichen 50 μL Ribosomenpuffer.
HINWEIS: Das lichtdurchlässige Pellet ist schwer zu sehen. Waschen Sie daher das Pellet vorsichtig von den Seiten des Röhrchens. - Bestimmen Sie die Ribosomenkonzentration durch Messung der Absorption bei 260 nM der Probe, die im Verhältnis 1:100 im Ribosomenpuffer verdünnt ist. Eine Absorption von 10 der verdünnten Lösung entspricht 23 μM unverdünnter Lösung wie zuvor beschrieben16.
- Implementieren Sie eine endgültige Stammkonzentration von 10 μM. Zur Einstellung der Konzentration verdünnen Sie die Ribosomen mit Ribosomenpuffer oder konzentrieren Sie sie weiter durch Zentrifugation bei 14000 x g in einem 3 kDa Zentrifugalfilter bei 4 °C.
HINWEIS: Um eine optimale Systemexpression zu erreichen, führen Sie eine Ribosomentitration durch (Abschnitt 5.2, Ergänzende Tabelle 7). - Überprüfen Sie die Ribosomenzusammensetzung mit SDS-PAGE (Abbildung 3A) wie in Abschnitt 1.3.3 angegeben. Verdünnen Sie 2,5 μL der Probe mit 7,5 μL Wasser, mischen Sie mit 10 μL 2x Laemmli-Puffer und laden Sie dann 5 μL und 2,5 μL der Proben in das Gel.
4. Energielösung
HINWEIS: Die Zusammensetzung für die hier vorgestellte 2,5-fache Energielösung ist ein Beispiel für eine Lösung, die für eine Standard-TX-TL-Reaktion gut funktionierte. Um das Timing zu optimieren, bereiten Sie die Energielösung an Tag 2 vor. Die Herstellung der Aminosäurelösung wird ausführlich erläutert, gefolgt vom abschließenden Zubereitungsverfahren.
- Aminosäurelösung
HINWEIS: Bereiten Sie die Aminosäurelösung in großen Mengen vor. Die Herstellung der Menge an Aminosäurenstammlösungen, die für ein Endvolumen von mindestens 2000 μL erforderlich ist, reduziert den Wägefehler für die ansonsten sehr kleinen Mengen. Die Gesamtkonzentration der Aminosäurelösung ist durch die Löslichkeit der Aminosäuren und die jeweiligen Stammlösungskonzentrationen begrenzt. Bereiten Sie für das Standard-PURE-System eine Lösung mit einer Endkonzentration von 3,25 mM vor. Verwenden Sie die Berechnungstabelle für Aminosäurelösungen (Ergänzende Tabelle 3) als Vorlage. Verwenden Sie Cystein in Salzform, um eine ausreichende Löslichkeit zu gewährleisten. Vermeiden Sie die Verwendung von KOH-basierten Aminosäure-Präparationsmethoden. Es ist möglich, die genauen Mengen an Aminosäuren direkt in die endgültige Aminosäurelösung zu wiegen, ohne die Stammlösung für alle Aminosäuren vorzubereiten. Dies ist jedoch anspruchsvoller und weniger präzise.- Bereiten Sie Stammlösungen für jede Aminosäure vor, wie in der ergänzenden Tabelle 3beschrieben, mit Ausnahme von Tyrosin.
HINWEIS: Aufgrund der unterschiedlichen Löslichkeiten der Aminosäuren in Wasser unterscheiden sich die jeweils vorgeschlagenen Konzentrationen der Stammlösung. - Die minimale Masse [mg] liefert die ungefähre Mindestmasse, die erforderlich ist, um eine ausreichende Menge an Stammlösung für das Zielgesamtvolumen als Referenz zu erhalten.
HINWEIS: Die minimale Masse wird mit einem Überschuss von 10% berechnet. - Für eine einfachere Herstellung der Lösungen nicht die genaue Menge an Aminosäure wiegen, sondern stattdessen für die vorliegende Masse die Wassermenge anpassen, um die gewünschte Konzentration zu erreichen. Berechnen Sie die benötigte Menge an deionisiertem Wasser (Wasser zum Hinzufügen [μL]), basierend auf der tatsächlich eingefüllten Masse (hellgelbe Zellen) und der gewünschten Konzentration unter Verwendung der Tabelle in Der Ergänzenden Tabelle 3.
- Lösen Sie die Aminosäure-Stammlösungen durch Wirbeln auf, bis sich der gesamte Niederschlag aufgelöst hat. Die einzelnen Aminosäure-Stammlösungen können bei -20 °C für mehrere Wochen gelagert werden.
HINWEIS: Einige Aminosäuren sind schwer in Wasser aufzulösen; Der Vorgang kann einige Zeit in Anspruch nehmen. - Die genaue Menge an Tyrosin, die benötigt wird, um eine Endkonzentration von 3,25 mM zu erhalten, wird direkt in das Röhrchen für die Aminosäurelösung eingewogen.
HINWEIS: Tyrosin ist sehr schwer in Wasser aufzulösen. Fügen Sie es direkt hinzu, anstatt eine Standardlösung vorzubereiten. - Fügen Sie die entsprechenden Mengen an Aminosäurestocklösungen und Wasser hinzu, wie im Endvolumen angegeben, um die [μL] -Säule (hellblaue Zellen) hinzuzufügen und die Lösung gut zu wirbeln. Die fertige Aminosäurelösung wird bis zur weiteren Anwendung bei -80 °C gelagert.
- Bereiten Sie Stammlösungen für jede Aminosäure vor, wie in der ergänzenden Tabelle 3beschrieben, mit Ausnahme von Tyrosin.
- Vorbereitung der Energielösung
ANMERKUNG: Insgesamt enthält die 2,5-fache Energielösung jeweils 0,75 mM jeder Aminosäure, 29,5 mM Magnesiumacetat, 250 mM Kaliumglutamat, je 5 mM ATP und GTP, 2,5 mM CTP, UTP und TCEP, 8,75 mg/ml tRNA aus E. coli MRE 600, 50 mM Kreatinphosphat, 0,05 mM Folinsäure, 5 mM Spermidin und 125 mM HEPES. Erstanwender bereiten die Energielösung in Kleinserien von 200 μL vor. Lagern Sie die nach Ergänzungstabelle 4 hergestellten Einzellösungen bei -20 °C oder -80 °C für die spätere Verwendung.- Alle in der Ergänzenden Tabelle 5 genannten wässrigen Lösungen auf Eis auftauen.
- Bereiten Sie in der Zwischenzeit die Lagerlösungen für die übrigen komponenten vor, die in der ergänzenden Tabelle 4aufgeführt sind. Halten Sie alle Lösungen nach der Zubereitung auf Eis.
HINWEIS: Geben Sie 500 μL RNase und DNase-freies Wasser direkt in die Durchstechflasche, um die lyophilisierten tRNAs aufzulösen. Durch sanftes Wirbeln gut mischen; Begrenzen Sie das Pipettieren, um die Einführung von RNases zu vermeiden. - Addieren Sie die berechneten Volumina (Ergänzende Tabelle 5) von Stammlösungen und Wasser und mischen Sie gut mit einem Wirbel. Bewahren Sie die Lösung immer auf Eis auf.
- Messen Sie den pH-Wert der Lösung, indem Sie 1 μL auf einen pH-Streifen pipettieren, um sicherzustellen, dass der pH-Wert der Lösung neutral ist.
- Aliquotieren Sie die Energielösung bei 50-100 μL pro Röhrchen auf Eis und lagern Sie sie bei -80 °C bis zur weiteren Verwendung. Beim Aliquotieren den Hauptbestand häufig verwirbeln, um zu verhindern, dass die Komponenten ausfallen.
HINWEIS: Führen Sie optional einen Aktivitätsassay der neu hergestellten Energielösung gegen kommerzielle Energielösungen durch, z. B. Lösung A in PURExpress. Wird eine deutlich geringere Leistung des Systems mit der Energielösung beobachtet, kann eine Optimierung der Ionenkonzentrationen, insbesondere Magnesiumionen, durch Titration (5-20 mM) vorteilhaft sein.
5. OnePot PURE Reaktion
- DNA-Vorlage
HINWEIS: Proteine, die nach dem T7-Promotor kodiert werden, können in PURE entweder aus linearer oder zirkulärer DNA exprimiert werden. Durch die Generierung einer linearen DNA-Schablone mittels Erweiterung PCR können langwierige Klonschritte entfallen. Die linearen Schablonen für diese Studie wurden durch PCR wie unten beschrieben unter Verwendung einer High-Fidelity-DNA-Polymerase (Table of Materials) generiert. Primersequenzen, Schmelztemperaturen und die in dieser Studie verwendeten Thermocycler-Einstellungen sind in der ergänzenden Tabelle 6 angegeben. Die Erstellung der DNA-Vorlage ist nicht im Tagesablauf enthalten.- Richten Sie eine PCR-Reaktion ein, wie vom Polymerase-Lieferanten empfohlen.
HINWEIS: Optimierte Parameter für eine High-Fidelity-DNA-Polymerase (Tabelle der Materialien) sind in der Ergänzenden Tabelle 6 angegeben. - Amplifizieren Sie das Zielgen (z. B. eGFP) als lineare Vorlage aus einem Plasmid oder Genom unter Verwendung genspezifischer Primer (500 nM) (für die Parameter siehe Ergänzende Tabelle 6).
- Die Amplifikation erzeugt kurze Erweiterungen, um Glühsequenzen für die folgenden Erweiterungs-PCR-Schritte bereitzustellen.
- Überprüfen Sie das Amplikon auf einem Agarosegel auf korrekte Größe und Reinheit.
- Verwenden Sie die amplifizierte DNA als Vorlage für die nachfolgenden Erweiterungsschritte. Richten Sie eine Reaktion von mindestens 50 μL ein.
- Führen Sie 10 PCR-Amplifikationszyklen mit den Erweiterungsprimern (2,5 nM) durch. Nach Abschluss der Amplifikationszyklen fügen Sie sofort die endgültigen Primer (500 nM) zu derselben Reaktion hinzu und führen Sie 30 Zyklen durch, um das erweiterte PCR-Produkt zu verstärken. Die Schmelztemperaturen und Primersequenzen finden Sie in der Ergänzenden Tabelle 6.
- Reinigen Sie die DNA-Fragmente mit einem DNA-Reinigungskit und eluieren Sie die DNA in nukleasefreiem Wasser anstelle von EDTA-haltigem Elutionspuffer.
- Überprüfen Sie die lineare Schablone eines Agarosegels auf korrekte Größe und Reinheit.
- Messen Sie die DNA-Konzentration in ng/μL mit einem UV-Vis-Spektralphotometer.
- Richten Sie eine PCR-Reaktion ein, wie vom Polymerase-Lieferanten empfohlen.
- Einrichten der PURE-Reaktion
HINWEIS: Die endgültige Reaktionszusammensetzung besteht aus 1x Energielösung, Tag-freien Ribosomen oder His-Tag-Ribosomen, OnePot PURE-Proteinen und DNA-Template. Das Reaktionsvolumenverhältnis umfasst 40% Energielösung, 30% Protein- und Ribosomenlösung sowie 30% DNA und Wasser. Typische Reaktionsvolumina variieren zwischen 5 μL und 25 μL. Quantifizieren Sie die Expression eines fluoreszierenden Proteins kontinuierlich auf einem Plate-Reader. Verwenden Sie eine grüne Lys in vitro Translation Labeling System, das fluoreszierend markierte Lysinreste in neu synthetisierte Proteine einbaut, um die Expression von nicht-fluoreszierenden Proteinen auf einem SDS-PAGE-Gel zu überprüfen. Ein Beispiel für eine Reaktionsvorlage ist in Ergänzende Tabelle 7 um eine PURE zellfreie Expressionsreaktion zu etablieren. Zellen in Gelb zeigen Benutzereingabewerte an, und Zellen in Orange zeigen zusätzliche Reagenzien an, die optional zur Reaktion hinzugefügt werden sollen. Halten Sie die Volumenverhältnisse der Komponenten präzise, um die richtige Ionenbilanz zu gewährleisten. Um beispielsweise eine höhere Proteinkonzentration zu erreichen, erhöhen Sie die OnePot-Proteinlösungskonzentration. Erhöhen Sie jedoch nicht das Volumen der Proteinlösung, die der Reaktion zugesetzt wird.- Geben Sie die Konzentration [ng/μL] und länge [Basenpaare] der DNA in den entsprechenden gelben Zellen in der Tabelle ein. Verwenden Sie 2-10 nM DNA für die Reaktion.
- Füllen Sie das gewünschte Gesamtreaktionsvolumen in μL aus.
- Nehmen Sie die benötigten Reagenzien aus dem Gefrierschrank und tauen Sie sie auf Eis auf.
HINWEIS: Ein erneutes Einfrieren der Komponenten ist ohne Einschränkung der Funktionalität möglich. Minimieren Sie jedoch die Anzahl der Gefrier-Tau-Zyklen und die Zeit, in der Proben auf Eis gespeichert werden, so weit wie möglich. - Pipettieren Sie die berechneten Mengen an Wasser, DNA und Energielösung auf eine Seite des PCR-Röhrchens oder eine Ecke eines Brunnens auf der 384-Well-Platte. Fügen Sie die erforderliche Menge eines zusätzlichen Reagenzes auf derselben Seite hinzu. Minimieren Sie die Anzahl der Proben pro Experiment, um probenverdampfung und experimentelle Startzeitverzerrungen zu vermeiden.
HINWEIS: Es ist wichtig, die Energiekomponente physikalisch von den Proteinkomponenten getrennt zu halten, um einen vorzeitigen Verbrauch der Energiequellen und geringere Erträge zu vermeiden. - Die berechneten Mengen an Protein- und Ribosomenlösung werden auf die andere Seite eines PCR-Röhrchens oder die gegenüberliegende Ecke der 384-Well-Platte pipettiert.
HINWEIS: Verwenden Sie nach Möglichkeit Master-Mischungen, um die Auswirkungen von Pipettierfehlern zu reduzieren. Nach ersten Tests können die Ribosomen- und Proteinlösungen gemischt und als eine Lösung gelagert werden. - Drehen Sie für eine kurze Zeit (30 s), um die Reaktionskomponenten zusammenzuführen. Um die Verdampfung während der Plate-Reader-Experimente zu verhindern, fügen Sie 35 μL flüssiges Wachs hinzu und versiegeln Sie die Platte mit einem transparenten Dichtmittel (siehe Materialtabelle).
- Mindestens 3 h bei 37 °C inkubieren.
- Für die Anzeige auf einem Plattenleser messen Sie alle 2 Minuten die Fluoreszenzintensität bei der erforderlichen Wellenlänge (repräsentative Ergebnisse sind in Abbildung 3B dargestellt).
- Führen Sie die folgenden Schritte für Green Lys-beschriftete Proben aus.
- Nach der zellfreien Expression wird die Probe mit 0,16 μg/μL RNase A für 30 min bei 37 °C inkubiert, um den fluoreszierenden Hintergrund des Green Lys-Markierungskits zu entfernen.
HINWEIS: Verwenden Sie RNase A, da andere Arten von RNases den Hintergrund nicht ausreichend gut entfernen. - Visualisieren Sie die Proteinexpression, indem Sie SDS-PAGE wie in Abschnitt 1.3.3 beschrieben ausführen. Waschen Sie das ungefärbte Gel vorsichtig in deionisiertem Wasser und bilden Sie es auf einem Fluoreszenzbildner mit einer Anregungswellenlänge von 488 nm ab.
- Anschließend färben Sie das Gel mit herkömmlichen Coomassie-Färbemethoden. Für die geeigneten Parameter siehe Abschnitt 1.3.3.
HINWEIS: Führen Sie eine Titration der Proteinlösung mit der empfohlenen Ribosomenkonzentration durch und titrieren Sie anschließend die Ribosomen mit der optimalen OnePot-Proteinkonzentration. Verwenden Sie das handelsübliche PURExpress ΔRibosome Kit als Positivkontrolle. Lösung A, Factor Mix und die Ribosomenlösung entsprechen der hergestellten Energie, der OnePot-Proteinlösung bzw. den gereinigten Ribosomen.
Ergebnisse
Das obige Protokoll wurde entwickelt, um die Etablierung des zellfreien TX-TL-Systems PURE in jedem Labor zu erleichtern. Das Protokoll enthält eine detaillierte Beschreibung der Vorbereitung der drei verschiedenen Teile des PURE-Systems: das OnePot-Protein, das Ribosom und die Energielösung. Ein detaillierter Tagesablauf, der den Workflow optimiert, ist in Tabelle 1 dargestellt. Der Workflow ist für die Reinigung von His-getaggten Ribosomen optimiert, und die Zeitrahmen können sich geringfügig unterscheiden, wenn eine tagfreie Ribosomenreinigung durchgeführt wird. Ein Präparat liefert eine ausreichende Menge an PURE für mindestens fünfhundert 10 μL-Reaktionen. Darüber hinaus sind die vorbereiteten Lösungen mehr als ein Jahr lang bei -80 °C stabil und können mehreren Frost-Tau-Zyklen standhalten.
Ausreichende Überexpressionswerte für alle Stämme sind entscheidend für die Funktionalität der endgültigen Proteinlösung. Abbildung 1 zeigt die erfolgreiche Überexpression in allen 36 Einzelstämmen, die anschließend für die OnePot-Proteinzubereitung verwendet wurden. Variationen in den Bandenintensitäten der überexprimierten Proteine traten höchstwahrscheinlich aufgrund einer Verzerrung der Ladevolumina auf das SDS-PAGE-Gel auf. Die erwarteten Proteingrößen sind in Tabelle 2zusammengefasst. GlyRS und PheRS bestehen aus zwei Untereinheiten unterschiedlicher Molekulargewichte; die restlichen 34 Proteine bestehen aus einer einzigen Untereinheit. Der Schlüssel zur Einfachheit und Zeitwirksamkeit dieses Protokolls ist der Kokulturations- und Co-Reinigungsschritt (Abbildung 2). Die OnePot-Proteinlösung wurde hergestellt, indem das Verhältnis des EF-Tu-Stammes zu allen anderen Expressionsstämmen erhöht wurde. Die Gesamtzusammensetzung der endgültigen Proteine wurde mittels SDS-PAGE analysiert (Abbildung 3A). Von den Gelen (Bahnen 2, 3) fällt auf, dass EF-Tu (43,3 kDa) erwartungsgemäß in einer höheren Konzentration im Vergleich zu den anderen Proteinen vorliegt. Während das Gel einen guten ersten Hinweis auf Proteinexpressionsverhältnisse liefert, ist es schwierig zu bestimmen, ob und auf welcher Ebene jedes einzelne Protein exprimiert wurde. Daher wird dringend empfohlen, die Überexpression in jedem Stamm vor der Kokulturation zu bestätigen, wie oben gezeigt.
Das E. coli-Ribosom ist eine komplexe molekulare Maschine, die aus über 50 einzelnen Proteinuntereinheiten besteht23. Ein repräsentatives Absorptionsspektrum bei 260 nm für die tagfreie Ribosomenreinigung ist in Abbildung 4dargestellt; der dritte Peak ist charakteristisch für eine erfolgreiche Ribosomenelution. Für beide Ribosomenreinigungsmethoden wurde das erwartete Ablaufmuster auf dem SDS-PAGE-Gel (Abbildung 3A)18 beobachtet. Wir beobachteten Kontaminationen für beide Reinigungen, wenn auch in kleinen Mengen (<10%). Insbesondere waren in den Tag-freien (Bahnen 5, 6) und His-markierten (Bahnen 11, 12) Ribosomen aufgrund der Variation der Methode unterschiedliche Verunreinigungen vorhanden. Zur Benutzerreferenz sind auch die SDS-PAGE-Gele für die kombinierten Systeme enthalten (Bahnen 8, 9 und 14, 15).
Abschließend wird die Leistungsfähigkeit der präparierten Systeme (Abbildung 3) unter Verwendung der verschiedenen Ribosomenvarianten verglichen. Die Zeitverläufe der in vitro eGFP-Expression zeigen, dass beide PURE-Systeme funktionsfähig sind und fluoreszierendes eGFP produzieren. Die OnePot-Proteinlösung in Kombination mit den His-getaggten Ribosomen ergab jedoch unter Verwendung der durch Titration optimierten Ribosomenkonzentration nur ein Drittel des Expressionsniveaus der nicht markierten Ribosomenversion (Abbildung 3B). Ähnliche Ergebnisse wurden beobachtet, als drei Proteine unterschiedlicher Größe mit dem Green Lys tRNA in vitro Markierungssystem exprimiert und markiert wurden (Abbildung 3C). Wie auf dem fluoreszierenden Gel zu sehen ist, wurden Produkte in voller Länge in beiden Systemen erfolgreich exprimiert; allerdings wurde nur etwa die Hälfte des Expressionsniveaus mit dem His-Tag-Ribosomensystem erreicht. Neben der Fluoreszenzmarkierung sind die erwarteten Banden für alle drei Proteine auf einem Coomassie-gefärbten Gel unterscheidbar (Abbildung 3D). Die Ergebnisse zeigen, dass das eingeführte Expressionssystem, das innerhalb einer Woche in einem Labor mit Standardausrüstung hergestellt werden kann, für die In-vitro-Expression von Proteinen verwendet werden kann, die nach dem T7-Promotor aus linearen Schablonen kodiert sind.

Abbildung 1: Repräsentative Ergebnisse für den Überexpressionstest für alle Expressionsstämme des PURE-Systems. Pure Proteinzahlen und -größen sind in Tabelle 2zusammengefasst. Die Proteinnummern 21, 24 und 27 sind zur besseren Visualisierung mit einem Stern markiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
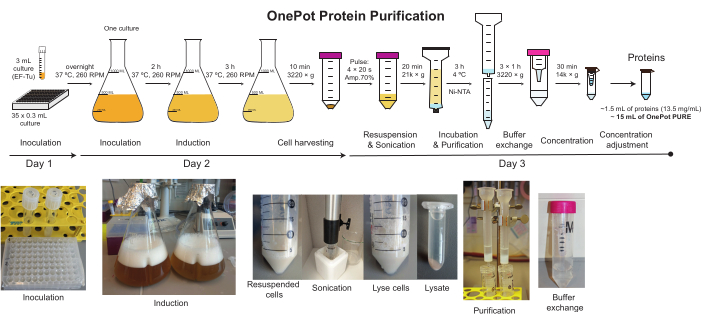
Abbildung 2: OnePot Proteinreinigung. Die schematische Darstellung und die entsprechenden Fotos aller Schritte, die an der Herstellung der OnePot-Proteinlösung beteiligt sind. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Leistungsfähigkeit der hergestellten Systeme unter Verwendung der verschiedenen Ribosomenvarianten. (A) Coomassie blau gefärbte SDS-PAGE Gele der OnePot Proteinlösung (Bahnen 2, 3), tagfreie Ribosomen ohne Proteinlösung (Bahnen 5, 6) und mit Proteinlösung (Bahnen 8, 9), His-markierte Ribosomen ohne Proteinlösung (Bahnen 11, 12) und mit Proteinlösung (Bahnen 14, 15). Pro Probe wurden zwei verschiedene Konzentrationen geladen. (B) Vergleich der eGFP-Expression von His-markierten Ribosomen und tagfreien Ribosomen. Die Fluoreszenzintensität der in vitro eGFP-Expression wird im Laufe der Zeit für eine PURE-Reaktion mit tagfreien Ribosomen (1,8 μM, blau) und His-getaggten Ribosomen (0,62 μM, rot) überwacht. Die Konzentrationen der linearen Schablone und der OnePot-Proteinlösung betrugen 4 nM bzw. 2 mg/ml. Die Panels (C) und (D) zeigen das SDS-PAGE-Gel von Proteinen, die in OnePot mit Tag-freien (1,8 μM, Blau, Bahnen 3, 4, 5) und His-Tag-Ribosomen (0,62 μM, Rot, Bahnen 6, 7, 8) synthetisiert und mit einem GreenLys-In-vitro-Markierungskit (C) markiert und mit Coomassie-Blau (D) gefärbt sind. Die schwarzen Pfeile zeigen die erwarteten Banden der synthetisierten Proteine an: eGFP (26,9 kDa), ArgRS (64,7 kDa), T7 RNAP (98,9 kDa). Die Konzentrationen der linearen Template- und OnePot-Proteinlösung betrugen 4 nM bzw. 1,6 mg/ml. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Absorptionsspektren bei 260 nm. Repräsentative Ergebnisse von Absorptionsspektren bei 260 nm während der hydrophoben Wechselwirkungsreinigung von tagfreien Ribosomen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Tabelle 1: Ein täglicher zeitoptimierter Zeitplan für die Vorbereitung aller OnePot PURE Lösungen. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Tabelle 2: PURE Proteinliste Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle 1: Reagenzien. Die Tabelle listet Konzentrationen, Volumina und andere spezifische Details der Reagenzien und Komponenten auf, die während dieser Studie verwendet wurden. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle 2: Puffer. Die Tabelle listet die genauen Pufferzusammensetzungen für Protein-, Tag-freie Ribosomen- und His-Tag-Ribosomenreinigungen sowie die Konzentrationen der für ihre Herstellung verwendeten Stammlösungen auf. Darüber hinaus berechnet es die erforderlichen Mengen an Komponenten basierend auf dem Puffervolumen. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle 3: Aminosäureberechnungen. Die Tabelle listet die Aminosäuren und ihre empfohlenen Stammlösungskonzentrationen auf, die für die Energielösung erforderlich sind. Es berechnet die Menge an Wasser, die jeder Aminosäure zugesetzt werden soll, basierend auf der tatsächlich gewogenen Masse, und berechnet auch das Volumen der Aminosäurelösung, die der endgültigen Aminosäuremischung zugesetzt werden soll. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle 4: Bestandslösungen für die Energielösung. Die Tabelle listet die Konzentrationen und Volumina der für die Energielösung benötigten Stammlösungen auf und gibt weitere Details, einschließlich der Lagerbedingungen, an. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle 5: Energielösung. Die Tabelle listet die Komponenten der Energielösung und ihre empfohlenen Konzentrationen auf. Darüber hinaus berechnet es ihre erforderlichen Volumina, die der endgültigen Lösung hinzugefügt werden sollen, basierend auf ihren Stammlösungskonzentrationen und dem Volumen der Energielösung. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle 6: PCR. Die Tabelle listet Sequenzen und Konzentrationen der für die Erweiterungs-PCR verwendeten Primer auf und zeigt Schmelztemperaturen und Thermocyclerschritte an, die für eine High-Fidelity-DNA-Polymerase optimiert sind. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Ergänzende Tabelle 7: REINE Reaktion. Die Tabelle zeigt ein Beispiel für die Einrichtung einer PURE-Reaktion. Es listet die verwendeten Konzentrationen und Volumina der Komponenten für eine PURE-Reaktion unter Verwendung von Tag-freien Ribosomen oder His-Tag-Ribosomen auf. Darüber hinaus berechnet es die Volumenverhältnisse für Protein- und Ribosomtitrationen. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Diskussion
Das hier vorgestellte Protokoll beschreibt ein einfaches, zeit- und kostengünstiges Verfahren zur Herstellung eines vielseitigen PURE-Expressionssystems20 auf Basis der Standardzusammensetzung15. Durch die Verwendung des Protokolls zusammen mit den mitgelieferten Tagesplänen (Tabelle 1) können alle Komponenten in 1 Woche vorbereitet werden und mengenmäßige Mengen ergeben, die für bis zu fünfhundert 10 μL PURE-Reaktionen ausreichen. Da die in diesem Protokoll verwendeten Proteine aus Plasmiden mit hoher Kopie überexprimiert werden und eine geringe Toxizität für E. coliaufweisen, werden gute Expressionsniveaus für alle erforderlichen Proteine beobachtet (Abbildung 1). Dies ermöglicht die einfache Anpassung der Stämme und damit auch der Proteinzusammensetzung in Kokulturen, indem einfach die Verhältnisse der Impfstämme modifiziert werden20. Neben den ribosomalen Proteinen erwies sich die Konzentration von EF-Tu als von grundlegender Bedeutung für die Expressionergibt 6. Im Gegensatz dazu hatten Änderungen in der Konzentration der anderen Proteinbestandteile einen relativ geringen Einfluss auf die Robustheit des PURE-Systems7,24. Daher kann durch Anpassung des Impfverhältnisses von EF-Tu in Bezug auf alle anderen Komponenten eine vergleichbare Zusammensetzung zur Standard-PURE-Zusammensetzung erreicht und ein PURE-System mit einer ähnlichenAusbeute von 20 erreicht werden. Bei der Herstellung der Proteinlösung ist es entscheidend sicherzustellen, dass alle Stämme gut wachsen und das kodierte Protein nach der Induktion überexprimieren (Abbildung 1).
Die Ribosomenfunktion ist der Schlüssel für die Gesamtleistung des PURE-Systems24. In diesem Protokoll werden zwei verschiedene Methoden zur Herstellung der Ribosomenlösung demonstriert, nämlich die Tag-freie und die His-getaggte Ribosomenreinigung. Die tagfreie Ribosomenreinigung basiert auf einer hydrophoben Interaktionschromatographie, gefolgt von einer Zentrifugation mit einem Saccharosekissen, die den Zugang zu einem FPLC-Reinigungssystem und einer Ultrazentrifuge15erfordert. Im Gegensatz dazu erfordert die Methode, die His-getaggte Ribosomen18 und Schwerkraftflussaffinitätschromatographie-Reinigung verwendet, keine spezielle Ausrüstung und kann in den meisten Labors durchgeführt werden. Die letztere Methode bringt daher Vorteile wie Einfachheit und Zugänglichkeit mit sich. Allerdings beobachteten wir eine signifikant geringere Syntheseausbeute bei Verwendung der His-getaggten Ribosomen im OnePot PURE im Vergleich zur Tag-freien Variante (Abbildung 3). Je nach Art der Anwendung kann diese geringere Ausbeute akzeptabel sein.
Die Energielösung liefert die niedermolekularen Komponenten und tRNAs, die erforderlich sind, um in vitro TX-TL-Reaktionen anzutreiben. Dieses Protokoll bietet ein Rezept für eine typische Energielösung, die leicht an die Benutzerbedürfnisse angepasst werden kann. Zusammen mit tRNA, NTP und Kreatinphosphat waren die Häufigkeit und Konzentration vonMg2+-Ionen entscheidend für die Gesamtleistung des PURE-Systems8,da sie kritische Cofaktoren für Transkription und Translation sind. In einigen Fällen kann die Titration von Ionen daher die gesamte PURE-Leistung erheblich verbessern. DNA-Integrität ist entscheidend für die PURE-Leistung. Daher hilft die Sequenzverifizierung der Promotorregion, der Ribosomenbindungsstelle und des Zielgens und die Sicherstellung, dass eine ausreichende DNA-Konzentration (<2 nM) bei der Behebung von Problemen, die beim Einrichten einer PURE-Reaktion auftreten können.
Das PURE-System ist ein minimales TX-TL-System, und spezifische Anwendungen können daher zusätzliche Anpassungen erfordern25. Diese können den Einbau verschiedener RNA-Polymerasen9,26, Chaperone13und Proteinfaktoren wie EF-P oder ArfA8umfassen . Obwohl die Expressionsstämme für diese Proteine in die Kokulturen aufgenommen werden können, kann die separate Zugabe zu dem vorbereiteten System eine bessere Kontrolle der erforderlichen Proteinspiegel ermöglichen. Weiterhin ist der Einschluss von Vesikeln essentiell für die Produktion von Membranproteinen10,11. Oxidierende statt reduzierende Umgebungen und eine Disulfidbindungsisomerase erleichtern eine ordnungsgemäße Disulfidbindungsbildung, die beispielsweise für sekretorische Proteine erforderlich sind12.
Es muss unbedingt sichergestellt werden, dass zusätzliche Komponenten die Reaktion nicht stören. Die wichtigsten Faktoren, auf die Beim Einrichten einer Reaktion oder beim Hinzufügen anderer Komponenten zu achten ist, sind unten aufgeführt. Stellen Sie sicher, dass weder inkompatible Puffer verwendet werden noch die Ionenkonzentrationen gestört werden. Vermeiden Sie Lösungen, die Glycerin, hohe Konzentrationen von Kalium, Magnesium, Calciumionen, Osmolyten, Pyrophosphat, Antibiotika oder EDTA enthalten, so weit wie möglich. Zum Beispiel kann das Ersetzen eines Elutionspuffers durch Wasser während der DNA-Reinigung von Vorteil sein, da EDTA ein üblicher Zusatzstoff in diesem Puffer ist. Die Versorgung der Lösungen mit zusätzlichen negativ geladenen Molekülen wie NTP oder dNTP erfordert eine Anpassung der Magnesiumkonzentration8, da sich die negativ geladenen Moleküle als Chelatbildner verhalten und positiv geladene Moleküle binden. Ein neutraler pH-Wert ist ideal für die Reaktion. Dementsprechend sollten alle Komponenten auf den entsprechenden pH-Wert gepuffert werden; Dies ist besonders wichtig für stark saure oder basische Moleküle wie NTPs. Schließlich sind Temperatur und Volumen Schlüsselparameter für die Reaktion. Um eine gute Ausbeute zu erzielen, sollte man eine Temperatur um 37 °C implementieren, da Temperaturen unter 34 °C die Ausbeute deutlich reduzieren27.
Es ist wichtig zu beachten, dass vor der Vorbereitung des OnePot PURE die Zielanwendung und die damit verbundenen Anforderungen wie Volumen, Reinheit, Einfache Änderung und Aufnahme oder Wegfall von Komponenten berücksichtigt werden sollten. Für viele Anwendungen ist das System eine ausgezeichnete Wahl, aber andere erfordern möglicherweise Erträge, Anpassungsfähigkeit und andere Faktoren, die das OnePot-System nicht bieten kann. Unabhängig davon wird das eingeführte Protokoll für die Vorbereitung eines hausgemachten Systems von Vorteil sein, da alle kritischen Schritte für eine solche Vorbereitung hier zusammengefasst sind.
Einer der Hauptvorteile des OnePot-Systems ist seine Kompatibilität mit dem handelsüblichen PURExpress-System, das die Möglichkeit bietet, die Funktionalität und Integrität aller Komponenten separat zu testen, indem jede PURExpress-Komponente nacheinander durch ihr OnePot-Äquivalent ersetzt wird. Die Vorteile des OnePot PURE-Systems, wie Abstimmbarkeit und einfache, schnelle und kostengünstige Zubereitung, werden zellfreies TX-TL für mehr Labore weltweit zugänglich machen und dazu beitragen, die Implementierung dieser leistungsstarken Plattform in der zellfreien synthetischen Biologie zu erweitern.
Offenlegungen
Die Autoren erklären, dass sie keine konkurrierenden finanziellen Interessen haben.
Danksagungen
Diese Arbeit wurde vom Europäischen Forschungsrat im Rahmen des Horizon 2020 Research and Innovation Program Grant der Europäischen Union 723106, einem Stipendium des Schweizerischen Nationalfonds (182019) und der EPFL unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 10x Tris/Glycine/SDS buffer | Bio-Rad Laboratories | 1610732 | |
| 15 mL centrifuge tubes | VWR International | 525-0309 | |
| 384-well Black Assay Plates | Corning | 3544 | |
| 4-20% Mini-PROTEANRTM TGXTM Precast Protein Gels | Bio-Rad Laboratories | 4561096 | |
| 50 mL centrifuge tubes | VWR International | 525-0304 | |
| 96-Well Polypropylene DeepWell plate | Nunc | 260252 | |
| Acetic acid, 99.8 % | Acros | 222140010 | |
| Äkta purifier | GE Healthcare | purification of tag free ribosomes | |
| AMICON ULTRA 0.5 mL - 3 KDa | Merck Millipore | UFC500324 | |
| AMICON ULTRA 15 mL - 3 KDa | Merck Millipore | UFC900324 | |
| Amino acids | Sigma-Aldrich | LAA21-1KT | |
| Ammonium chloride | Sigma-Aldrich | 09718-250G | |
| Ammonium sulfate | Sigma-Aldrich | A4418 | |
| Ampicillin | Condalab | 6801 | |
| BenchMark Fluorescent Protein Standard | ThermoFisher | LC5928 | |
| Breathe-Easy sealing membrane | Diversified Biotech | Z380059-1PAK | |
| Centrifuge tubes polycarbonate | Beckman | 355631 | purification of tag free ribosomes |
| Chill-out Liquid Wax | Bio-Rad Laboratories | CHO1411 | |
| Creatine phosphate | Sigma-Aldrich | 27920 | |
| DNA Clean & Concentrator-25 (Capped) | Zymo | ZYM-D4034-200TS | |
| DTT | SantaCruz Biotech | sc-29089B | |
| Econo-Pac Chromatography Columns | Bio-Rad Laboratories | 7321010 | |
| EDTA (Ethylenediaminetetraacetic acid) | Sigma-Aldrich | 03609-250G | |
| Eppendorf Protein LoBind microcentrifuge tubes | VWR International / Eppendorf | 525-0133 | |
| Falcon 14 mL Round Bottom Polystyrene Test Tube, with Snap Cap | Falcon | 352051 | |
| Flasks, baffled 1000 mL 4 baffles, borosilicate glass | Scilabware | 9141173 | |
| FluoroTect Green Lys in vitro Translation Labeling System | Promega | L5001 | optional |
| Folinic acid | Sigma-Aldrich | PHR1541 | |
| Glycerol | Sigma-Aldrich | G7757-1L | |
| HEPES | Gibco | 15630-056 | |
| HiTrap Butyl HP Column | GE Healthcare | 28411005 | purification of tag free ribosomes |
| IMAC Sepharose 6 Fast Flow | GE Healthcare | 17-0921-07 | |
| Imidazole | Sigma-Aldrich | I2399 | |
| InstantBlue | Expedeon | ISB1L-1L | |
| IPTG (Isopropyl-beta-D-thiogalactoside) | Alfa Aesar | B21149.03 | |
| Laemmli buffer (2x), sample buffer | Sigma-Aldrich | S3401-1VL | |
| Lysogeny broth (LB) media | AppliChem | A0954 | |
| Magnesium acetate | Sigma-Aldrich | M0631 | |
| Magnesium chloride | Honeywell Fluka | 63020-1L | |
| Nickel Sulfate | Alfa Aesar | 15414469 | |
| NTP | ThermoFisher | R0481 | |
| Phusion High-Fidelity DNA Polymerase (2 U/µL) | ThermoFisher | F530S | |
| Potassium chloride | Sigma-Aldrich | P5405-1KG | |
| Potassium glutamate | Sigma-Aldrich | 49601 | |
| PURExpress In Vitro Protein Synthesis Kit | NEB | E6800S | |
| PURExpress Δ Ribosome Kit | NEB | E3313S | |
| Quick Start Bradford 1x Dye Reagent | Bio-Rad Laboratories | 5000205 | |
| Rapid-Flow Sterile Single Use Vacuum Filter Units | ThermoFisher | 564-0020 | |
| RNaseA solution | Promega | A7973 | |
| SealPlate film | Excel Scientific | Z369659-100EA | |
| Sodium hydroxide | Sigma-Aldrich | 6203 | |
| Spermidine | Sigma-Aldrich | S2626 | |
| Sucrose | Sigma-Aldrich | 84097 | |
| TCEP (Tris(2-carboxyethyl)phosphin -hydrochlorid) | Sigma-Aldrich | 646547-10X1mL | |
| Thickwall Polycarbonate Tube | Beckman | 355631 | |
| Trichloroacetic acid | Sigma-Aldrich | T0699 | |
| Tris base | ThermoFisher | BP152-500 | |
| tRNA | Roche | 10109541001 | |
| Ultracentrifuge Optima L-80 | Beckman | purification of tag free ribosomes | |
| Whatman GD/X syringe filters | GE Whatman | WHA68722504 | |
| β-mercaptoethanol | Sigma-Aldrich | M6250-100mL |
Referenzen
- Laohakunakorn, N., et al. Bottom-up construction of complex biomolecular systems with cell-free synthetic biology. Frontiers in Bioengineering and Biotechnology. 8, 213 (2020).
- Laohakunakorn, N. Cell-free systems: a proving ground for rational biodesign. Frontiers in Bioengineering and Biotechnology. 8, 788 (2020).
- Cho, E., Lu, Y. Compartmentalizing cell-free systems: toward creating life-like artificial cells and beyond. ACS Synthetic Biology. 9 (11), 2881-2901 (2020).
- Gaut, N. J., Adamala, K. P. Reconstituting natural cell elements in synthetic cells. Advanced Biology (Weinh). 5 (3), 2000188 (2021).
- Shimizu, Y., et al. Cell-free translation reconstituted with purified components. Nature Biotechnology. 19 (8), 751-755 (2001).
- Li, J., Gu, L., Aach, J., Church, G. M. Improved cell-free RNA and protein synthesis system. PLoS One. 9 (9), 106232 (2014).
- Kazuta, Y., Matsuura, T., Ichihashi, N., Yomo, T. Synthesis of milligram quantities of proteins using a reconstituted in vitro protein synthesis system. Journal of Bioscience and Bioengineering. 118 (5), 554-557 (2014).
- Li, J., et al. Dissecting limiting factors of the protein synthesis using recombinant elements (PURE) system. Translation. 5 (1), 1327006 (2017).
- de Maddalena, L. L., et al. GreA and GreB enhance expression of Escherichia coli RNA polymerase promoters in a reconstituted transcription-translation system. ACS Synthethic Biology. 5 (9), 929-935 (2016).
- Kuruma, Y., Ueda, T. The PURE system for the cell-free synthesis of membrane proteins. Nature Protocols. 10 (9), 1328-1344 (2015).
- Jacobs, M. L., Boyd, M. A., Kamat, N. P. Diblock copolymers enhance folding of a mechanosensitive membrane protein during cell-free expression. Proceedings of the National Academy of Sciences of the Unites States of America. 116 (10), 4031-4036 (2019).
- Shimizu, Y., Kanamori, T., Ueda, T. Protein synthesis by pure translation systems. Methods. 36 (3), 299-304 (2005).
- Niwa, T., Kanamori, T., Ueda, T., Taguchi, H. Global analysis of chaperone effects using a reconstituted cell-free translation system. Proceedings of the National Academy of Sciences of the Unites States of America. 109 (23), 8937-8942 (2012).
- Niwa, T., et al. Large-scale analysis of macromolecular crowding effects on protein aggregation using a reconstituted cell-free translation system. Frontiers in Microbiology. 6, 1113 (2015).
- Shimizu, Y., Ueda, T. PURE technology. Methods in molecular biology. 607, 11-21 (2010).
- Horiya, S., Bailey, J. K., Krauss, I. J. Directed evolution of glycopeptides using mRNA display. Methods in Enzymology. 597, 83-141 (2017).
- Villarreal, F., et al. Synthetic microbial consortia enable rapid assembly of pure translation machinery. Nature Chemical Biology. 14 (1), 29-35 (2018).
- Wang, H. H., et al. Multiplexed in vivo His-tagging of enzyme pathways for in vitro single-pot multienzyme catalysis. ACS Synthetic Biology. 1 (2), 43-52 (2012).
- Shepherd, T., et al. De Novo Design and synthesis of a 30-cistron translation-factor module. Nucleic Acids Research. 45 (18), 10895-10905 (2017).
- Lavickova, B., Maerkl, S. J. A Simple, robust, and low-cost method to produce the PURE cell-free system. ACS Synthetic Biology. 8 (2), 455-462 (2019).
- Ederth, J., Mandava, C. S., Dasgupta, S., Sanyal, S. A single-step method for purification of active his-tagged ribosomes from a genetically engineered Escherichia coli. Nucleic Acids Research. 37 (2), 15 (2009).
- Gesteland, R. F. Isolation and characterization of ribonuclease I mutants of Escherichia coli. Journal of Molecular Biology. 16 (1), 67-84 (1996).
- Li, J., et al. Cogenerating synthetic parts toward a self-replicating system. ACS Synthetic Biology. 6 (7), 1327-1336 (2017).
- Matsuura, T., Kazuta, Y., Aita, T., Adachi, J., Yomo, T. Quantifying epistatic interactions among the components constituting the protein translation system. Molecular Systems Biology. 5, 297 (2009).
- Libicher, K., Hornberger, R., Heymann, M., Mutschler, H. In vitro self-replication and multicistronic expression of large synthetic genomes. Nature Communications. 11 (1), 904 (2020).
- Niederholtmeyer, H., Stepanova, V., Maerkl, S. J. Implementation of cell-free biological networks at steady state. Proceedings of the National Academy of Sciences of the United States of America. 110 (40), 15985-15990 (2013).
- Lavickova, B., Laohakunakorn, N., Maerkl, S. J. A partially self-regenerating synthetic cell. Nature Communications. 11 (1), 6340 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten