Method Article
Sondeo de las propiedades estructurales y dinámicas del tráfico de nanoestructuras subcelulares mediante espectroscopia de fluctuación espaciotemporal
En este artículo
Resumen
El análisis de desplazamiento cuadrático medio (iMSD) derivado de imágenes se aplica a los macropinosomas para resaltar su naturaleza intrínseca de evolución en el tiempo en términos de propiedades estructurales y dinámicas. Los macropinosomas se comparan con los gránulos secretores de insulina (ISG) como referencia para las estructuras subcelulares con propiedades estructurales / dinámicas promedio invariantes en el tiempo.
Resumen
El desplazamiento cuadrático medio derivado de imágenes (iMSD) se utiliza para abordar las propiedades estructurales y dinámicas de las nanoestructuras subcelulares, como las vesículas involucradas en el tráfico endo/exocitótico de solutos y biomoléculas. iMSD se basa en imágenes de lapso de tiempo estándar, es compatible con cualquier configuración óptica y no necesita detenerse en objetos individuales para extraer trayectorias. A partir de cada traza de iMSD, se calcula y combina un triplete único de parámetros estructurales y dinámicos promedio (es decir, tamaño, difusividad local, coeficiente anómalo) para construir la "firma iMSD" de la nanoestructura en estudio.
La potencia de este enfoque se demuestra aquí con el caso ejemplar de los macropinosomas. Estas vesículas evolucionan en el tiempo, cambiando su tamaño promedio, número y propiedades dinámicas que pasan de las primeras a las últimas etapas del tráfico intracelular. Como control, los gránulos secretores de insulina (ISG) se utilizan como referencia para las estructuras subcelulares que viven en un estado estacionario en el que las propiedades estructurales y dinámicas promedio de toda la población de objetos son invariantes en el tiempo. El análisis de iMSD destaca cuantitativamente estas características peculiares y allana el camino a aplicaciones similares a nivel subcelular, tanto en los estados fisiológicos como patológicos.
Introducción
Las nanoestructuras subcelulares (por ejemplo, vesículas endocíticas/secretoras, orgánulos) desempeñan un papel fundamental en la regulación de la señalización celular1. El ajuste adecuado de sus características estructurales (por ejemplo, tamaño) y/o dinámicas (por ejemplo, difusividad) determina cómo responde la célula a los estímulos internos o externos 2,3,4. Con base en esta evidencia, no es sorprendente que las alteraciones de estas características se encuentren en muchas condiciones patológicas. Los ejemplos abarcan el papel de la endocitosis mal regulada en el cáncer 2,3, las alteraciones estructurales y dinámicas encontradas a nivel de ISG en células β expuestas a condiciones de diabetes tipo 25, la mala regulación de las propiedades estructurales y de transporte lisosomales en la leucodistrofia de células globoides o la lipidosisgalactosilceramida 6, y las disfunciones en la vía endo-lisosomal en trastornos neurodegenerativos (por ejemplo, enfermedad de Alzheimer)7.
En este contexto, los investigadores han demostrado recientemente que el rendimiento de los métodos de microscopía óptica estándar puede mejorarse ajustando adecuadamente la resolución de muestreo espacial y temporal8. Esto, a su vez, puede proporcionar una mayor comprensión de los procesos biológicos de relevancia. En la práctica, esto es posible gracias a un algoritmo de análisis de fluctuación espaciotemporal, que extrae simultáneamente las propiedades estructurales y dinámicas promedio de la difusión de objetos directamente de la pila estándar de imágenes de microscopía óptica sin necesidad de conocimiento preliminar sobre el objeto biológico de interés y extracción de trayectorias de un solo objeto. Toda esta información se incluye en una única salida del método: un seguimiento iMSD9 (los detalles sobre la derivación y el análisis del seguimiento iMSD se dan en el Archivo Complementario 1).
El protocolo experimental resultante consta de unos pocos pasos. En primer lugar, la obtención de imágenes de la región de interés se realiza a alta resolución temporal. Luego, las funciones de correlación espacio-temporal promedio se calculan a partir de la pila de imágenes. Finalmente, mediante el ajuste gaussiano de la serie de funciones de correlación, la 'ley de difusión' promedio se obtiene directamente de las imágenes y se analiza para reconocer el modo de difusión del objeto. El potencial del método ya se demostró para una variedad de objetos biológicos, que van desde moléculas hasta nanopartículas e incluso orgánulos / estructuras subcelulares completas 9,10,11,12,13,14,15.
Este artículo informa sobre la aplicación de iMSD a macropinosomas para resaltar su naturaleza intrínseca e irreversible de evolución en el tiempo en términos de sus propiedades estructurales y dinámicas promedio (es decir, a nivel de toda la población). Además, estas vesículas endocíticas se comparan con las ISG como referencia para estructuras subcelulares en un "estado estacionario", es decir, un estado en el que las propiedades estructurales / dinámicas promedio de toda la población de gránulos permanecen constantes en cualquier punto de tiempo. La macropinocitosis define una serie de eventos iniciados por la reorganización extensa (o agitación) de la membrana plasmática para formar una estructura macropinocítica externa que luego se internaliza16. Los macropinosomas en etapa temprana formados son muy similares a los fagosomas. Al mismo tiempo, se pueden distinguir de otras formas de vesículas endocíticas debido a su gran tamaño característico, heterogeneidad morfológica y falta de estructuras de capa de proteínas.
Los ensayos bioquímicos revelaron que, tras la internalización, los macropinosomas se enriquecen progresivamente con marcadores proteicos de otras vías endocíticas, lo que a su vez sugiere que sus identidades cambian continuamente durante el tráfico17. Utilizando anticuerpos contra marcadores conocidos de la vía endosomal, se demostró que los macropinosomas adoptan progresivamente características endosomas clásicas: disminuyen de tamaño, se convierten en estructuras endocíticas tardías (por ejemplo, lisosomas) o eventualmente pierden su identidad a través de la recuperación mediada por membrana de marcadores moleculares específicos (por ejemplo, clasificación de nexinas)18,19 . El escenario general es que cada macropinosoma dentro de la célula cambia irreversiblemente su identidad estructural y dinámica (así como molecular) durante el tráfico desde la membrana plasmática hasta su destino intracelular final. Como resultado, las propiedades estructurales/dinámicas/moleculares de toda la población de macropinosomas también están cambiando a lo largo del mismo camino temporal. Siendo intrínsecamente sensible a las propiedades promedio de toda la población de objetos observados, el método iMSD representa cuantitativamente la "naturaleza evolutiva" mediante la cuantificación de parámetros promedio clave, es decir, la difusividad local y el coeficiente anómalo (propiedades dinámicas) y el tamaño promedio de los macropinosomas (propiedad estructural) en cualquier etapa de su tráfico intracelular.
A modo de comparación, se realizaron mediciones similares en una estructura bien conocida de membrana intracelular, el ISG, en un modelo de células β. Al igual que los macropinosomas, la regulación de las propiedades estructurales y dinámicas de los ISG, desde su génesis en la Red Trans Golgi (TGN) hasta su exocitosis en la membrana plasmática, es fundamental para la correcta ejecución de la función ISG20. Sin embargo, a diferencia de los macropinosomas, los ISG viven en un "estado estacionario" en el que, en cualquier momento, todas las etapas funcionales / estructurales / moleculares de la vida útil de ISG están simultáneamente presentes dentro de la célula, y cada una está representada por una subpoblación específica de ISG. Esto significa que aunque cada gránulo evoluciona irreversiblemente de la biogénesis a la secreción, se espera que las propiedades estructurales / dinámicas promedio de toda la población de gránulos permanezcan constantes en cualquier punto de tiempo (a menos que las condiciones de estado estacionario cambien, por ejemplo, por estímulos externos como la glucosa, el colesterol y las citoquinas13). Esto se confirma mediante el análisis de iMSD.
Protocolo
1. Preparación de la muestra
- Antes del experimento de microscopía, células de subcultivo en platos adecuados para aplicaciones de microscopía.
- Lave dos veces una placa tratada con cultivo de tejidos de 10 cm de células confluentes HeLa o INS 1E (insulinoma similar a células β) con 0,01 M 1x PBS, agregue 1 ml de tripsina-EDTA al 0,05 % (1x) y colóquela en una incubadora de 37 °C, humidificada, al 5% de CO2 durante 5 min.
- Resuspender las células separadas agregando 9 mL de medio DMEM completo (para células HeLa) o RPMI 1640 (para células INS-1E), y recolectar los 10 mL finales en tubos de centrífuga.
- Sembrar aproximadamente 2 × 105 celdas en cada plato de 35 mm x 10 mm en un volumen final de 1 mL del medio. Incubar las células durante 24 h a 37 °C y 5% CO2.
- Para etiquetar fluorescentemente los lisosomas, use LysoTracker Red DND-99.
- Diluya la solución madre en 1 ml de medio precalentado hasta una concentración final de colorante de 70 nM.
- Reemplace el medio del plato con un medio fresco que contenga LysoTracker. Incubar las células en un medio que contenga LysoTracker durante 20 min a 37 °C en una atmósfera de 5% de CO2 , y lavarlas dos veces con medio fresco antes del experimento.
- Para etiquetar fluorescentemente los macropinosomas, use fluoresceína isotiocianato-dextrano de 70 kDa. Lavar las células subcultivadas tres veces con solución salina tamponada con fosfato (PBS) 0,01 M 1x, sustituir por medio que contenga dextrano (1 mg/ml) e incubar a 37 °C durante 30 min. Antes de continuar con el experimento de microscopía, lave las células tres veces con medio fresco.
- Para etiquetar fluorescentemente los ISG en las células INS-1E, transfectar las células utilizando el reactivo de transfección (consulte la Tabla de materiales) y el plásmido13 de proteína fluorescente verde mejorada con péptido C (EGFP) de acuerdo con el protocolo del fabricante, e incube a 37 ° C durante 24 h en una atmósfera de CO2 al 5% antes del experimento.
2. Adquisición de datos
- Para dejar que el microscopio se equilibre a la temperatura y la atmósfera deseadas, encienda el sistema de control de la incubadora del microscopio al menos 2 h antes del experimento.
NOTA: Cada adquisición es una serie de lapso de tiempo. - Adquiera las imágenes utilizando un microscopio confocal invertido equipado con un objetivo de inmersión en agua de apertura numérica (NA) de 60x y 1.2.
- Utilice un láser de argón de 488 nm para la excitación de EGFP (células transfectadas) y macropinosomas marcados con fluoresceína. Recoja la emisión de fluorescencia entre 500 y 600 nm utilizando un detector de tubo fotomultiplicador estándar.
- Utilice un láser HeNe de 543 nm para excitar Lysotracker y recoger su emisión de fluorescencia entre 555 y 655 nm.
- Establezca el diámetro del agujero de detección en el tamaño de 1 Airy. Para cada adquisición, recopile una serie de 1000 fotogramas secuenciales. Establezca el tiempo de permanencia de píxeles en 2 μs/píxel para un tiempo de fotograma de 129 ms.
NOTA: Cada fotograma constaba de 256 x 256 píxeles (16 bits/píxel) con una dimensión física de 69 nm/píxel, correspondiente aproximadamente a un área de 17 μm x 17 μm.
3. i Cálculode MSD
NOTA: Para ejecutar correctamente el cálculo, utilice un software capaz de cálculo numérico y programación de scripts. El script específico (es decir, para el archivo de script 'iMSD.m', consulte El archivo de soporte 1) debe estar presente en el mismo directorio que contiene la serie de imágenes que se va a procesar. Cada imagen de la serie debe guardarse como un archivo '.tif' distinto.
- Para inicializar correctamente los parámetros instrumentales utilizados para las adquisiciones, abra iMSD.m con el editor de texto del software y modifique su primera sección de la siguiente manera.
- Establezca N como el número de fotogramas en la serie temporal (por ejemplo, 1000 en este protocolo).
- Establezca px_size: tamaño de píxel, expresado en μm (por ejemplo, 0,069 en este protocolo).
- Conjunto f: resolución temporal de cada fotograma, expresada en segundos (por ejemplo, 0,129 en este protocolo).
- Establecer filtro: entrada binaria para la corrección de fondo, establezca el valor en '0' para procesar imágenes sin procesar o establezca el valor en '1' para realizar una resta de fondo basada en umbrales.
- Establezca av_toll: umbral para la corrección de fondo; cualquier píxel con intensidad inferior a este valor se establecerá como 0 si Filter=1.
- Establezca el bit como el número entero que determina la intensidad del muestreo (por ejemplo, 8 bits, 16 bits).
- Guarde y ejecute el archivo de script iMSD.m editado.
- Compruebe la ejecución del script.
NOTA: El estado de procesamiento del cálculo se puede comprobar en la ventana de comandos; si se produce algún problema grave, el proceso se interrumpirá y se mostrará un mensaje de advertencia para mostrar el tipo de error y el código de línea relacionado. De lo contrario, siga los pasos 3.3.1-3.3.3.- Importe la pila de imágenes y reste el fondo (si es necesario).
- Calcular la función de correlación espaciotemporal G(ξ,η,τ) utilizando el método de Fourier.
- Ajuste la función de correlación espaciotemporal con una función gaussiana 2D.
NOTA: Al final del proceso, los valores de salida de los procedimientos de ajuste σ2(τ) se mostrarán en la ventana de comandos: se informan los promedios, el error y la bondad correspondiente (R2) de la conexión.
- Compruebe la salida gráfica.
NOTA: La curva iMSD y las curvas de ajuste correspondientes se muestran en tres paneles separados, cada uno para un tipo diferente de ecuación de ajuste utilizada: difusión browniana, difusión anómala o difusión confinada. Para cada curva, el valor R2 se informa en la leyenda del gráfico. - Compruebe la salida de texto.
NOTA: Al final del proceso, se crea un archivo '.xls' con el mismo nombre del archivo '.tif' original de lapso de tiempo. La primera hoja contiene los valores de ξ, η, τ y σ2 calculados para cada retardo de tiempo. Los parámetros de entrada y los valores de salida calculados principales se informan en la segunda hoja, es decir, el coeficiente de difusión, el coeficiente anómalo y la intersección del eje Y σ20 .
Resultados
El flujo de trabajo general del método se presenta en la Figura 1. Recapitula los principales pasos presentados en la sección de protocolo, desde la preparación de la muestra (Figura 1A) hasta la imagen de lapso de tiempo de nanoestructuras intracelulares (Figura 1B), el análisis de fluctuación para el cálculo de la serie de funciones de correlación espaciotemporal (Figura 1C) y el ajuste para la derivación de las propiedades estructurales / dinámicas promedio del objeto en estudio (Figura 1D).
Un parámetro crítico es la resolución de tiempo adoptada para obtener imágenes del objeto subcelular de interés. Este valor experimental establecerá el umbral de tiempo en el que se medirá el desplazamiento medio mínimo de los objetos de interés. Sin embargo, la condición preferida es establecer una resolución de tiempo de la imagen en la que el objeto de interés aparece "inmóvil" dentro del marco capturado, es decir, muestra un tamaño característico que, en promedio, no se deforma debido a la velocidad de la imagen. Esto es técnicamente posible si el objeto de interés es una estructura subcelular u orgánulo encerrado en una membrana (como en este caso). Típicamente, las estructuras subcelulares exhiben coeficientes de difusión local (D, μm2/s, ver Tabla 1) que son varios órdenes de magnitud más bajos que los de moléculas individuales aisladas en el citoplasma (por ejemplo, GFP21). La validación se puede realizar inmovilizando artificialmente el orgánulo de interés (por ejemplo, mediante fijación química). De hecho, esta condición puede servir como referencia para determinar el tamaño real del orgánulo en las condiciones experimentales utilizadas (por ejemplo, longitud de onda de excitación, tamaño de píxel, objetivo).
Aquí, aunque los lisosomas se utilizaron como orgánulos de prueba para este procedimiento, los resultados son válidos independientemente de la estructura objetivo. La Figura 2A muestra una imagen de un lisosoma fijo (es decir, inmóvil) junto con una adquisición realizada en células vivas a la resolución temporal adecuada (es decir, típicamente por debajo de 100 ms / marco; por ejemplo, 65 ms / marco en el ejemplo de la Figura 2B) y una adquisición realizada intencionalmente a una resolución temporal muy baja (por ejemplo, 10 s / marco en el ejemplo de la Figura 2C ). Para cada condición, el tamaño del objeto difusor se extrae de la siguiente manera: i) la herramienta de línea en el software ImageJ deriva un perfil de intensidad del punto; ii) el perfil de intensidad es trazado e interpolado por una función gaussiana para calcular el ancho total a la mitad del valor máximo (FWHM) que, a su vez, se utiliza como una estimación del diámetro del punto (Figura 2D). Como se esperaba y se muestra en la gráfica de la Figura 2E, la adquisición a muy alta resolución temporal (es decir, 65 ms/fotograma) arroja un tamaño medio de la estructura cercano al obtenido en la muestra fija, ya sea utilizando la herramienta estándar descrita anteriormente o extrayendo la intersección del eje y iMSD. En cambio, la adquisición a velocidad lenta produce un aumento en el tamaño aparente de la estructura, debido a la dinámica natural de la estructura durante la obtención de imágenes. En condiciones experimentales subóptimas, la información estructural/dinámica extraída no refleja fielmente las propiedades intrínsecas del objeto objeto de estudio.
Una vez que se seleccionan los principales parámetros experimentales, se pueden producir conjuntos de datos para las estructuras intracelulares objetivo. Para los macropinosomas, después de 20 min de incubación de las células con 70 kDa dextrans, las series temporales de las estructuras intracelulares marcadas se adquirieron en diferentes puntos de tiempo después del tratamiento, desde 30 min hasta aproximadamente 180 min. Curiosamente, se detecta un cambio gradual en las propiedades estructurales y dinámicas de los macropinosomas durante el tráfico (Figura 3; las distribuciones de σ02, Dm, α y N para macropinosomas se informan en las gráficas de la izquierda). Si bien no se detectan cambios evidentes en la difusividad local (Dm) de los macropinosomas durante el tráfico, tanto el tamaño característico (σ02) como el modo general de movimiento (α) evolucionan en el tiempo.
Cabe destacar que durante el tráfico se observa una disminución en el tamaño promedio de los macropinosomas (Figura 3A, izquierda), junto con un aumento concomitante en la naturaleza subdiffusiva de su movimiento (es decir, denotado como una disminución en los valores de α, Figura 3C, panel izquierdo). Además, se extrajo el número de macropinosomas de cada adquisición: los resultados, reportados en la Figura 3D, panel izquierdo, revelan claramente un aumento en el número de macropinosomas en el tiempo. Todos estos resultados están de acuerdo con las expectativas porque se supone que los macropinosomas marcados con dextrano se originan como vesículas aisladas y grandes encerradas en la membrana plasmática (que también son competentes para el movimiento a lo largo de los componentes citoesqueléticos), pero se supone que se comunican gradualmente con la vía endo-lisosomal formada por una gran población de estructuras más pequeñas y difusoras al azar.
Como se anticipó anteriormente, los resultados para los macropinosomas contrastan con mediciones similares realizadas en ISG (Figura 3, columna derecha). Los gránulos de insulina no muestran una tendencia de evolución temporal de los parámetros estructurales/dinámicos derivados de iMSD (y su número promedio dentro de la célula) en la misma ventana de tiempo observada para los macropinosomas. Además, los valores característicos de σ02, Dm y α son bastante diferentes de los de los lisosomas, utilizados nuevamente como referencia. Este resultado confirma la idea, como se anticipó anteriormente, de que los gránulos se sondean en un "estado estacionario" en el que, en cualquier momento, las propiedades estructurales / dinámicas promedio de toda la población de ISG no cambian (es decir, permanecen constantes, a menos que las condiciones del estado estacionario cambien, por ejemplo, debido a estímulos externos).
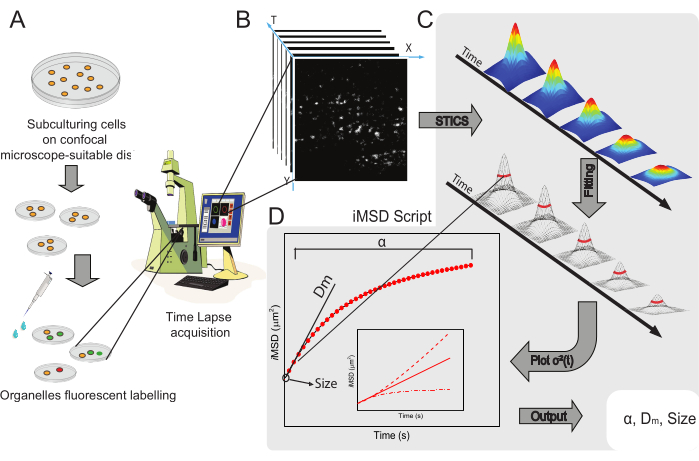
Figura 1: Flujo de trabajo experimental. (A) Las células se enchaparon 24 h (48 h para experimentos de transfección) antes de los experimentos confocales en placas celulares adecuadas para aplicaciones microscópicas. Las células se trataron adecuadamente de acuerdo con el método de etiquetado (ver protocolo) para teñir el orgánulo citoplasmático de interés. (B) Una adquisición confocal típica consiste en una pila de imágenes (time-lapse) de una porción citoplasmática de una célula viva, que describe la evolución temporal de la dinámica de orgánulos marcados. (C) La película de lapso de tiempo se analiza con un script matlab hecho a medida, primero calculando la función de correlación de imágenes espaciotemporales y el ajuste gaussiano para trazar curvas iMSD (D) y parámetros de ajuste extraídos relacionados que describen los parámetros de dinámica estructural de los orgánulos fotografiados. Abreviaturas: iMSD = desplazamiento cuadrático medio derivado de imágenes; STICS = espectroscopia de correlación de imágenes espaciotemporales; α = coeficiente de difusión anómalo; Dm = difusividad local; σ2(τ) = varianza. Haga clic aquí para ver una versión más grande de esta figura.
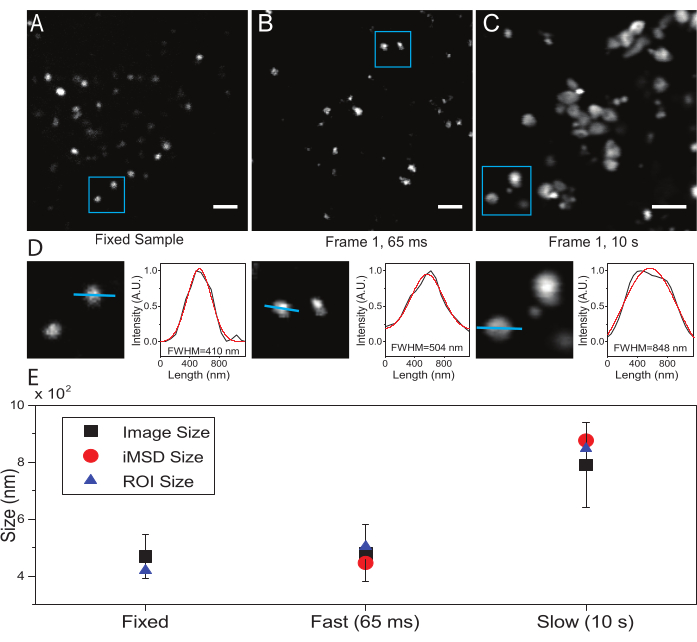
Figura 2: Parámetros experimentales adecuados. (A) Imagen ejemplar de lisosomas teñidos en una muestra fija. Barra de escala = 2 μm. (B) Primer fotograma de una pila de imágenes de lisosomas teñidos en una célula viva, adquirido con los parámetros apropiados. Resolución temporal: 65 ms/frame. Barra de escala = 2 μm. (C) El primer fotograma de una pila de imágenes de lisosomas teñidos en una célula viva, adquiridas a baja velocidad: es visible la deformación artificiosa del tamaño aparente del lisosoma debido al movimiento de orgánulos durante la imagen. Resolución temporal: 10 s/frame. Barra de escala = 2 μm. (D) Ejemplo de cálculo de tamaño para lisosomas fotografiados en un ROI azul de (A), (B) y (C). El perfil de intensidad a lo largo de la línea azul fue equipado con una función gaussiana para recuperar el FWHM, es decir, una estimación del tamaño del punto. Los valores de FWHM se informan para cada ajuste. (E) Representación gráfica de los valores de tamaño obtenidos mediante el análisis de imágenes descritos en el panel (D) para todos los lisosomas fotografiados (cuadrado negro, valor medio y desviación estándar), para los lisosomas encerrados dentro del ROI azul (triángulo azul), y recuperados por el análisis iMSD (círculo rojo). Esta cifra es de 22. Abreviaturas: ROI = región de interés; FWHM = ancho completo a la mitad como máximo; iMSD = desplazamiento cuadrático medio derivado de imágenes. Haga clic aquí para ver una versión más grande de esta figura.
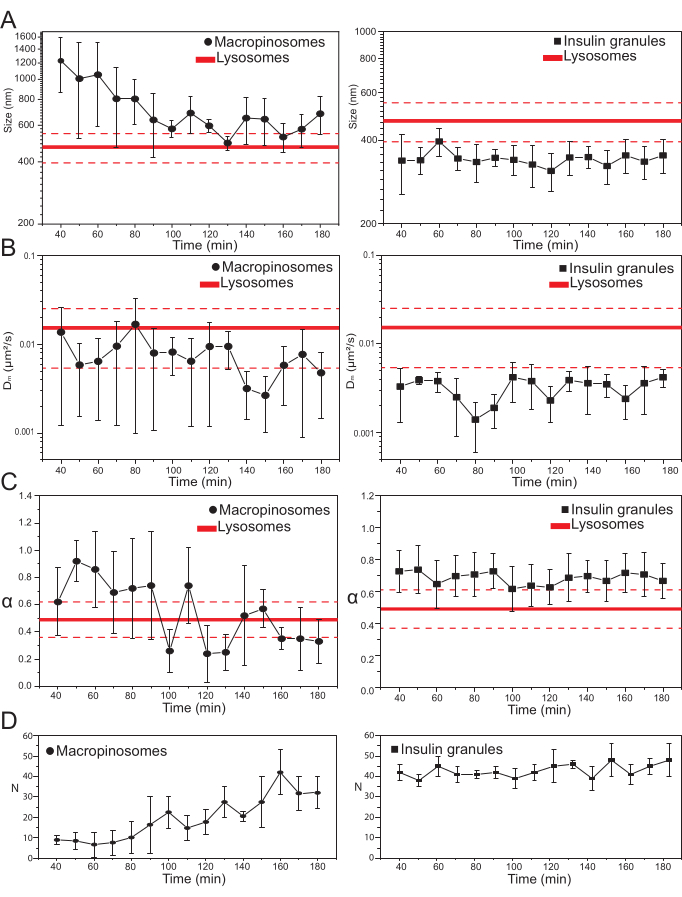
Figura 3: Evolución temporal de orgánulos marcados. (A) Gráficos de valores de tamaño extraídos de iMSD vs. progresión temporal de adquisiciones posteriores representadas como una media de valores medidos en adquisiciones realizadas en una ventana de tiempo de 10 minutos. A la izquierda, reducción progresiva en el tamaño promedio de los macropinosomas (círculos negros) y a la derecha, el tamaño invariante en el tiempo de los gránulos secretores de insulina (cuadrados negros) en comparación con los lisosomas, representado como valor de tamaño promedio (línea roja más gruesa) ± desviación estándar (líneas rojas discontinuas). (B) y (C) Progresión temporal de Dm y coeficientes α para macropinosomas (izquierda) y gránulos de insulina (derecha) extraídos mediante análisis iMSD. (D) Evolución temporal de los números de macropinosomas marcados y gránulos de insulina medidos en el primer fotograma de cada película de lapso de tiempo adquirida. Abreviaturas: iMSD = desplazamiento cuadrático medio derivado de imágenes; α = coeficiente de difusión anómalo; Dm = difusividad local, N = número. Haga clic aquí para ver una versión más grande de esta figura.
| Orgánulo | Etiquetado | Línea celular | Tamaño (nm) | Dm ( × 10-3 μm2/s) | α | N | Ref. | ||
| Endosoma temprano (EE) | CellLight Early Endosome GFP | Hela | 395±74 | 3.0±2.4 | 1.02±0.20 | 40 | 10 | ||
| Endosoma tardío (LE) | CellLight Late Endosome GFP | Hela | 693±102 | 15.4±10.6 | 0,57±0,16 | 58 | 10 | ||
| Lisosoma (LY) | LysoTracker DND-99 | Hela | 471±76 | 15,3±9,0 | 0,49±0,13 | 143 | 10, 14 | ||
| Caveola (CAV) | Caveolina-EGFP | Hela | 405±49 | 3.1±1.8 | 1.00±0.22 | 15 | 10 | ||
| Vescicle recubierto de clatrina (CCV) | Transferrin-Alexa 488 | Hela | 513±62 | 16.2±9.9 | 0,48±0,17 | 33 | 10 | ||
| Gránulo de insulina (IG) | Péptido C-EGFP | INS-1E | 335±56 | 3.0±1.7 | 0,70±0,14 | 107 | 11 | ||
| Macropinosoma temprano (EMCR) | Fluoresceína-Dextrano 70 kDa | Hela | 979±423 | 8.3±9 | 0,79±0,27 | 36 | 10 | ||
| Macropinosoma intermedio (IMCR) | Fluoresceína-Dextrano 70 kDa | Hela | 702±180 | 13.7±19.9 | 0,60±0,38 | 29 | 10 | ||
| Macropinosoma tardío (LMCR) | Fluoresceína-Dextrano 70 kDa | Hela | 592±127 | 5.8±4.7 | 0,39±0,21 | 21 | 10 | ||
Tabla 1: Parámetros estructurales y dinámicos extraídos de MSD. La tabla muestra los valores de tamaño, Dm y coeficiente de α medido para diferentes orgánulos, especificando las estrategias de etiquetado, la línea celular utilizada y el número de adquisiciones analizadas. Los valores se notifican como media ± desviación estándar. Abreviaturas: iMSD = desplazamiento cuadrático medio derivado de imágenes; α = coeficiente de difusión anómalo; Dm = difusividad local, N = número; GFP = proteína fluorescente verde; EGFP = proteína fluorescente verde mejorada.
Archivo complementario 1: Detalles sobre la derivación y el análisis del seguimiento de iMSD. Haga clic aquí para descargar este archivo.
Archivo de soporte 1: Haga clic aquí para descargar este archivo.
Discusión
Las propiedades y ventajas de iMSD son evidentes en comparación con las técnicas disponibles para recuperar información análoga. Para la información estructural, la opción preferida es el análisis de microscopía electrónica de transmisión (TEM). Mediante este método, se pueden recuperar detalles ultraestructurales a resolución molecular e incluso más allá, incluso para nanoestructuras subcelulares. Sin embargo, la peculiar resolución espacial de TEM se logra a expensas de la información en la dimensión temporal, lo cual es de interés aquí. Para compensar esto, los recientes avances en las tecnologías de imágenes de células vivas son de particular interés. Estos incluyen nuevos marcadores fluorescentes con mayores rendimientos (por ejemplo, brillo y fotoestabilidad), procedimientos de etiquetado optimizados y detectores más sensibles. Además, se dispone de herramientas analíticas para abordar tanto la estructura (por ejemplo, el "tamaño" mediante el análisis basado en fasores de la espectroscopia de correlación de imágenes locales, PLICS23, la agregación/oligomerización mediante el análisis de número y brillo24) como la dinámica (por ejemplo, la ley de difusión mediante el seguimiento de una sola partícula, es decir, (SPT)25,26,27,28 ) parámetros en la escala subcelular. El método SPT permite el acceso directo a la trayectoria del objeto y su MSD. Sin embargo, la desventaja es la necesidad de una baja densidad de la sonda y etiquetas muy brillantes y muchas trayectorias de un solo objeto que se midan para satisfacer los criterios estadísticos. Con respecto a la resolución temporal de la medición, las sondas fotoestables inorgánicas (por ejemplo, puntos cuánticos o nanopartículas metálicas) pueden aumentar el rendimiento de SPT, pero a expensas de procedimientos complejos de producción y etiquetado.
En comparación con estos estándares, el método iMSD descrito aquí muestra algunas ventajas clave. En primer lugar, este enfoque se puede utilizar junto con etiquetas fluorescentes relativamente tenues, como las proteínas fluorescentes codificadas genéticamente (por ejemplo, la aplicación a isG). Por lo tanto, en comparación con SPT, se logra una mayor resolución temporal (utilizando la misma etiqueta) debido a la menor cantidad de fotones requeridos8. En segundo lugar, el método iMSD está limitado solo por la resolución temporal, pero no por la difracción. De hecho, a pesar de la configuración óptica limitada por difracción utilizada, se pueden medir los desplazamientos moleculares promedio incluso por debajo del límite de difracción, como ya se ha demostrado para los flujos moleculares mediante el uso de STICS29. La resolución real en la medición de desplazamientos depende de la precisión (en términos de señal a ruido) con la que se puede medir la función de correlación, lo que explica por qué no está limitada por la difracción. Por lo tanto, parece claro que el desplazamiento mínimo que se puede medir depende de la difusividad del objeto de interés y la resolución temporal de la configuración de la imagen.
En este sentido, es importante considerar que la aplicación a nanoestructuras subcelulares, como macropinosomas o gránulos de insulina, con un microscopio de barrido láser es óptima: la velocidad de escaneo disponible es significativamente mayor que la dinámica del objeto de interés. En tal caso, el movimiento de los objetos durante la adquisición es insignificante, y la función de correlación puede ser aproximada por una función gaussiana. Finalmente, el enfoque iMSD se puede aplicar fácilmente a una amplia gama de configuraciones de microscopía óptica comercial basadas en escaneo ráster o imágenes basadas en cámaras de campo amplio, sin necesidad de calibración del sistema (requerida solo si se necesita lograr una estimación precisa del tamaño de partícula). Un parámetro importante para que el método funcione es el muestreo espacial adecuado. Como regla general, para alcanzar una convergencia satisfactoria del algoritmo de ajuste, el tamaño mínimo de la región de interés para la obtención de imágenes debe ser al menos 3 veces mayor que el desplazamiento máximo de interés.
En conclusión, el método iMSD solo requiere un microscopio equipado para una adquisición rápida. La estructura de interés se puede etiquetar a cualquier fluoróforo orgánico o codificado genéticamente, lo que permite imágenes multicanal. Se prevé que el análisis cruzado de iMSD se utilizará en un futuro próximo para seleccionar subpoblaciones de nanoestructuras subcelulares y revelar sus interacciones y codifusión dentro de la célula, siendo este último un tema candente en la biofísica celular. Si se pierde algún detalle por el análisis de iMSD, esto ciertamente está relacionado con la gran cantidad de información molecular dentro de las nanoestructuras subcelulares dinámicas. Dicha información se promedia inevitablemente durante la medición debido a la mala resolución temporal. Teóricamente, sin embargo, no existe un límite técnico debido a la posibilidad de recuperar información molecular, siempre que se puedan alcanzar velocidades de adquisición suficientes8. Debido a las continuas mejoras en la velocidad/sensibilidad del detector y las tecnologías de imágenes, se prevé que la información sobre todo el compartimento subcelular y sus componentes moleculares se extraerá de un único conjunto de datos.
Divulgaciones
Los autores no tienen ningún conflicto de intereses que declarar.
Agradecimientos
Este trabajo ha recibido financiación del Consejo Europeo de Investigación (CEI) en el marco del Programa de Investigación e Innovación Horizonte 2020 de la Unión Europea (acuerdo de subvención n.º 866127, proyecto CAPTUR3D).
Materiales
| Name | Company | Catalog Number | Comments |
| 100x Penicillin-Streptomycin-Glutamine | Gibco | 10378-016 | Cell medium supplement |
| C-peptide-EGFP | Plasmid | ||
| DMEM High Glucose | Gibco | 31053028 | Cell medium (HeLa) |
| FBS | Gibco | 10082147 | Cell medium supplement |
| Fluorescein isothiocyanate-dextran 70 kDa | Sigma Aldrich | 46945-100MG-F | Reagent |
| HeLa | ATCC | CCL-61 | Cell Line |
| Lipofectamine 2000 | TermoFisher | 11668019 | Trasfection reagent |
| Lysotracker Red DND-99 | Gibco | L7528 | Reagent |
| Matlab | MathWork | Software | |
| Microscope-suitable cell dishes | Willco | GWSt-3522 | Petri dishes |
| Olympus FV1000 | Olympus Japan | Confocal microscope | |
| RPMI 1640 | Gibco | 11835063 | Cell medium (INS-1E) |
Referencias
- Murphy, J. E., Padilla, B. E., Hasdemir, B., Cottrell, G. S., Bunnett, N. W. Endosomes: a legitimate platform for the signaling train. Proceedings of the National Academy of Sciences of the United States of America. 106 (42), 17615-17622 (2009).
- Mosesson, Y., Mills, G. B., Yarden, Y. Derailed endocytosis: an emerging feature of cancer. Nature Reviews Cancer. 8 (11), 835-850 (2008).
- Mellman, I., Yarden, Y. Endocytosis and cancer. Cold Spring Harbor Perspectives in Biology. 5 (12), 016949 (2013).
- Di Fiore, P. P. Endocytosis, signaling and cancer, much more than meets the eye. Preface. Molecular Oncology. 3 (4), 273-279 (2009).
- Bogan, J. S., Xu, Y., Hao, M. Cholesterol accumulation increases insulin granule size and impairs membrane trafficking. Traffic. 13 (11), 1466-1480 (2012).
- Ballabio, A., Gieselmann, V. Lysosomal disorders: From storage to cellular damage. Biochimica et Biophysica Acta. 1793 (4), 684-696 (2009).
- Hu, Y. -. B., Dammer, E. B., Ren, R. -. J., Wang, G. The endosomal-lysosomal system: from acidification and cargo sorting to neurodegeneration. Translational Neurodegeneration. 4, 18 (2015).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. Spatiotemporal fluctuation analysis: a powerful tool for the future nanoscopy of molecular processes. Biophysical Journal. 111 (4), 679-685 (2016).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. Fast spatiotemporal correlation spectroscopy to determine protein lateral diffusion laws in live cell membranes. Proceedings of the National Academy of Sciences of the United States of America. 110 (30), 12307-12312 (2013).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. From fast fluorescence imaging to molecular diffusion law on live cell membranes in a commercial microscope. Journal of Visualized Experiments: JoVE. (92), e51994 (2014).
- Digiacomo, L., et al. Dynamic fingerprinting of sub-cellular nanostructures by image mean square displacement analysis. Scientific Reports. 7 (1), 14836 (2017).
- Durso, W., et al. Lysosome dynamic properties during neuronal stem cell differentiation studied by spatiotemporal fluctuation spectroscopy and organelle tracking. International Journal of Molecular Sciences. 21 (9), 3397 (2020).
- Ferri, G., et al. Insulin secretory granules labelled with phogrin-fluorescent proteins show alterations in size, mobility and responsiveness to glucose stimulation in living β-cells. Scientific Reports. 9 (1), 2890 (2019).
- Durso, W., D'Autilia, F., Amodeo, R., Marchetti, L., Cardarelli, F. Probing labeling-induced lysosome alterations in living cells by imaging-derived mean squared displacement analysis. Biochemical and Biophysical Research Communications. 503 (4), 2704-2709 (2018).
- Digiacomo, L., Digman, M. A., Gratton, E., Caracciolo, G. Development of an image Mean Square Displacement (iMSD)-based method as a novel approach to study the intracellular trafficking of nanoparticles. Acta Biomaterialia. 42, 189-198 (2016).
- Swanson, J. A., Watts, C. Macropinocytosis. Trends in Cell Biology. 5 (11), 424-428 (1995).
- Jones, A. T. Macropinocytosis: searching for an endocytic identity and role in the uptake of cell penetrating peptides. Journal of Cellular and Molecular Medicine. 11 (4), 670-684 (2007).
- Falcone, S., et al. Macropinocytosis: regulated coordination of endocytic and exocytic membrane traffic events. Journal of Cell Science. 119, 4758-4769 (2006).
- Kerr, M. C., et al. Visualisation of macropinosome maturation by the recruitment of sorting nexins. Journal of Cell Science. 119, 3967-3980 (2006).
- Rorsman, P., Renstrom, E. Insulin granule dynamics in pancreatic beta cells. Diabetologia. 46 (8), 1029-1045 (2003).
- Di Rienzo, C., Piazza, V., Gratton, E., Beltram, F., Cardarelli, F. Probing short-range protein Brownian motion in the cytoplasm of living cells. Nature Communications. 5 (1), 5891 (2014).
- Ferri, G., et al. Time-lapse confocal imaging datasets to assess structural and dynamic properties of subcellular nanostructures. Scientific Data. 5 (1), 180191 (2018).
- Scipioni, L., Gratton, E., Diaspro, A., Lanzanò, L. Phasor analysis of local ICS detects heterogeneity in size and number of intracellular vesicles. Biophysical Journal. 111 (3), 619-629 (2016).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical Journal. 94 (6), 2320-2332 (2008).
- Li, C. H., Bai, L., Li, D. D., Xia, S., Xu, T. Dynamic tracking and mobility analysis of single GLUT4 storage vesicle in live 3T3-L1 cells. Cell Research. 14 (6), 480-486 (2004).
- Donovan, K. W., Bretscher, A. Tracking individual secretory vesicles during exocytosis reveals an ordered and regulated process. Journal of Cell Biology. 210 (2), 181-189 (2015).
- Westphal, V., et al. Video-rate far-field optical nanoscopy dissects synaptic vesicle movement. Science. 320 (5873), 246-249 (2008).
- Tabei, S. M. A., et al. Intracellular transport of insulin granules is a subordinated random walk. Proceedings of the National Academy of Sciences of the United States of America. 110 (13), 4911-4916 (2013).
- Hebert, B., Costantino, S., Wiseman, P. W. Spatiotemporal image correlation spectroscopy (STICS) theory, verification, and application to protein velocity mapping in living CHO cells. Biophysical Journal. 88 (5), 3601-3614 (2005).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados