Method Article
Untersuchung struktureller und dynamischer Eigenschaften des Handels subzellulärer Nanostrukturen durch raumzeitliche Fluktuationsspektroskopie
In diesem Artikel
Zusammenfassung
Die iMSD-Analyse (Imaging-derived mean square displacement) wird auf Makropinosomen angewendet, um ihre intrinsische zeitentwickelnde Natur in Bezug auf strukturelle und dynamische Eigenschaften hervorzuheben. Macropinosomen werden dann mit insulinsekretorischen Granulaten (ISGs) als Referenz für subzelluläre Strukturen mit zeitinvarianten durchschnittlichen strukturellen/dynamischen Eigenschaften verglichen.
Zusammenfassung
Imaging-derived mean square displacement (iMSD) wird verwendet, um die strukturellen und dynamischen Eigenschaften von subzellulären Nanostrukturen zu untersuchen, wie z.B. Vesikel, die am endo/exozytotischen Transport von gelösten Stoffen und Biomolekülen beteiligt sind. iMSD basiert auf Standard-Zeitraffer-Bildgebung, ist mit jedem optischen Setup kompatibel und muss nicht auf einzelnen Objekten verweilen, um Trajektorien zu extrahieren. Aus jeder iMSD-Spur wird ein eindeutiges Triplett durchschnittlicher struktureller und dynamischer Parameter (d.h. Größe, lokale Diffusivität, anomaler Koeffizient) berechnet und kombiniert, um die "iMSD-Signatur" der untersuchten Nanostruktur zu bilden.
Die Wirksamkeit dieses Ansatzes wird hier mit dem beispielhaften Fall von Makropinosomen bewiesen. Diese Vesikel entwickeln sich im Laufe der Zeit und verändern ihre durchschnittliche Größe, Anzahl und dynamischen Eigenschaften, die von den frühen bis zu den späten Stadien des intrazellulären Verkehrs reichen. Als Kontrolle werden Insulinsekretorgranula (ISGs) als Referenz für subzelluläre Strukturen verwendet, die in einem stationären Zustand leben, in dem die durchschnittlichen strukturellen und dynamischen Eigenschaften der gesamten Objektpopulation in der Zeit invariant sind. Die iMSD-Analyse hebt diese besonderen Merkmale quantitativ hervor und ebnet den Weg zu ähnlichen Anwendungen auf subzellulärer Ebene, sowohl im physiologischen als auch im pathologischen Zustand.
Einleitung
Subzelluläre Nanostrukturen (z.B. endozytische/sekretorische Vesikel, Organellen) spielen eine zentrale Rolle bei der zellsignalisierendenRegulation 1. Die richtige Abstimmung ihrer strukturellen (z. B. Größe) und/oder dynamischen (z. B. Diffusivität) Eigenschaften bestimmt, wie die Zelle auf interne oder externe Reize reagiert 2,3,4. Basierend auf diesen Beweisen ist es nicht verwunderlich, dass Veränderungen dieser Eigenschaften in vielen pathologischen Zuständen gefunden werden. Beispiele umfassen die Rolle der falsch regulierten Endozytose bei Krebs 2,3, die strukturellen und dynamischen Veränderungen, die auf der Ebene der ISGs in β-Zellen gefunden wurden, die Typ-2-Diabetes-Erkrankungenausgesetzt waren 5, die Fehlregulation lysosomaler Struktur- und Transporteigenschaften bei Globoidzellleukodystrophie oder Galactosylceramidlipidose6 und Dysfunktionalitäten im endo-lysosomalen Signalweg bei neurodegenerativen Erkrankungen (z. B. Alzheimer-Krankheit)7.
In diesem Zusammenhang haben Forscher kürzlich bewiesen, dass die Leistung von Standardmethoden der optischen Mikroskopie durch die richtige Abstimmung der räumlichen und zeitlichen Abtastauflösung8 verbessert werden kann. Dies wiederum kann weitere Einblicke in relevante biologische Prozesse liefern. In der Praxis wird dies durch einen Algorithmus der räumlich-zeitlichen Fluktuationsanalyse ermöglicht, der gleichzeitig die durchschnittlichen strukturellen und dynamischen Eigenschaften von diffusen Objekten direkt aus dem Standardstapel optischer Mikroskopiebilder extrahiert, ohne dass Vorkenntnisse über das biologische Objekt von Interesse und die Extraktion von Einzelobjekttrajektorien erforderlich sind. Alle diese Informationen sind in einer einzigen Ausgabe der Methode enthalten: einem iMSD-Trace9 (Details zur iMSD-Trace-Ableitung und -Analyse finden Sie in der Ergänzungsdatei 1).
Das resultierende experimentelle Protokoll besteht aus wenigen Schritten. Zunächst wird die Bildgebung der interessierenden Region mit hoher zeitlicher Auflösung durchgeführt. Anschließend werden aus dem Bildstapel durchschnittliche räumlich-zeitliche Korrelationsfunktionen berechnet. Schließlich wird durch Gaußsche Anpassung der Reihe von Korrelationsfunktionen das durchschnittliche "Diffusionsgesetz" direkt aus der Bildgebung erhalten und analysiert, um den Objektdiffusionsmodus zu erkennen. Das Potenzial der Methode wurde bereits für eine Vielzahl von biologischen Objekten nachgewiesen, die von Molekülen über Nanopartikel bis hin zu ganzen subzellulären Organellen/Strukturen reichen 9,10,11,12,13,14,15.
Dieses Papier berichtet über die Anwendung von iMSD auf Makropinosomen, um ihre intrinsische, irreversible zeitentwickelnde Natur in Bezug auf ihre durchschnittlichen (d.h. auf der gesamten Bevölkerungsebene) strukturellen und dynamischen Eigenschaften hervorzuheben. Darüber hinaus werden diese endozytären Vesikel mit ISGs als Referenz für subzelluläre Strukturen in einem "stationären Zustand" verglichen, d.h. einem Zustand, in dem die durchschnittlichen strukturellen/dynamischen Eigenschaften der gesamten Granulatpopulation zu jedem Zeitpunkt konstant bleiben. Macropinozytose definiert eine Reihe von Ereignissen, die durch die umfangreiche Reorganisation (oder Rüschen) der Plasmamembran ausgelöst werden, um eine externe makropinozytäre Struktur zu bilden, die dann internalisiertwird 16. Die gebildeten Makropinosomen im Frühstadium sind den Phagosomen sehr ähnlich. Gleichzeitig unterscheiden sie sich von anderen Formen endozytischer Vesikel durch ihre charakteristische Größe, morphologische Heterogenität und fehlende Proteinhüllenstrukturen.
Biochemische Assays zeigten, dass Makropinosomen nach der Internalisierung allmählich mit Proteinmarkern anderer endozytischer Signalwege angereichert werden, was wiederum darauf hindeutet, dass sich ihre Identität während des Handels ständig ändert17. Unter Verwendung von Antikörpern gegen bekannte Marker des Endosomenweges wurde gezeigt, dass Makropinosomen schrittweise klassische endosomale Merkmale annehmen: Sie nehmen an Größe ab, entwickeln sich zu späten endozytären Strukturen (z. B. Lysosomen) oder verlieren schließlich ihre Identität durch membranvermittelten Abruf spezifischer molekularer Marker (z. B. Sortierung von Nexinen)18,19 . Das Gesamtszenario ist, dass jedes einzelne Makropinosom innerhalb der Zelle seine strukturelle und dynamische (sowie molekulare) Identität während des Transports von der Plasmamembran zu seinem endgültigen intrazellulären Schicksal irreversibel ändert. Infolgedessen verändern sich auch die strukturellen/dynamischen/molekularen Eigenschaften der gesamten Population von Makropinosomen entlang des gleichen zeitlichen Pfades. Da die iMSD-Methode intrinsisch empfindlich auf die durchschnittlichen Eigenschaften der gesamten Population der beobachteten Objekte reagiert, bildet sie die "sich entwickelnde Natur" quantitativ durch die Quantifizierung der wichtigsten Durchschnittsparameter ab, d.h. der lokalen Diffusivität und des anomalen Koeffizienten (dynamische Eigenschaften) und der durchschnittlichen Größe der Makropinosomen (strukturelle Eigenschaft) in jedem Stadium ihres intrazellulären Verkehrs.
Zum Vergleich wurden ähnliche Messungen an einer bekannten intrazellulären membranumschlossenen Struktur, der ISG, in einem Modell von β-Zellen durchgeführt. Wie bei Makropinosomen ist die Regulation der strukturellen und dynamischen Eigenschaften von ISGs, von ihrer Entstehung im Trans-Golgi-Netzwerk (TGN) bis zu ihrer Exozytose an der Plasmamembran, entscheidend für die ordnungsgemäße Ausführung der ISG-Funktion20. Im Gegensatz zu Makropinosomen leben ISGs jedoch in einem "stationären Zustand", in dem zu jeder Zeit alle funktionellen/strukturellen/molekularen Stadien der ISG-Lebensdauer gleichzeitig in der Zelle vorhanden sind und jedes durch eine spezifische Subpopulation von ISGs repräsentiert wird. Dies bedeutet, dass, obwohl sich jedes einzelne Granulat irreversibel von der Biogenese zur Sekretion entwickelt, erwartet wird, dass die durchschnittlichen strukturellen / dynamischen Eigenschaften der gesamten Granulatpopulation zu jedem Zeitpunkt konstant bleiben (es sei denn, die stationären Zustandsbedingungen werden beispielsweise durch äußere Reize wie Glukose, Cholesterin und Zytokineverändert 13). Dies wird durch die iMSD-Analyse bestätigt.
Protokoll
1. Probenvorbereitung
- Vor dem Mikroskopie-Experiment Subkulturzellen in Schalen, die für Mikroskopie-Anwendungen geeignet sind.
- Waschen Sie eine 10 cm mit Gewebekultur behandelte Schale aus konfluenten HeLa- oder INS 1E-Zellen (Insulinom β-Zell-ähnlich) zweimal mit 0,01 M 1x PBS, fügen Sie 1 ml 0,05% Trypsin-EDTA (1x) hinzu und legen Sie es für 5 min in einen 37 °C starken, befeuchteten 5% CO 2-Inkubator.
- Resuspendieren Sie die abgelösten Zellen durch Zugabe von 9 ml vollständigem DMEM- (für HeLa-Zellen) oder RPMI 1640 (für INS-1E-Zellen) Medium und sammeln Sie die letzten 10 ml in Zentrifugenröhrchen.
- Samen Sie ca. 2 × 105 Zellen in jeder 35 mm x 10 mm Schale in einem Endvolumen von 1 ml des Mediums. Inkubieren Sie die Zellen für 24 h bei 37 °C und 5% CO2.
- Um Lysosomen fluoreszierend zu markieren, verwenden Sie LysoTracker Red DND-99.
- Verdünnen Sie die Stammlösung in 1 ml vorgewärmtem Medium auf eine endgültige Farbstoffkonzentration von 70 nM.
- Ersetzen Sie das Medium aus der Schale durch frisches LysoTracker-haltiges Medium. Inkubieren Sie die Zellen in LysoTracker-haltigem Medium für 20 min bei 37 °C in einer 5% CO2-Atmosphäre und waschen Sie sie vor dem Experiment zweimal mit frischem Medium.
- Um Makropinosomen fluoreszierend zu markieren, verwenden Sie 70-kDa Fluorescein-Isothiocyanat-Dextran. Waschen Sie die subkultivierten Zellen dreimal mit 0,01 M 1x phosphatgepufferter Kochsalzlösung (PBS), ersetzen Sie sie durch Dextran-haltiges Medium (1 mg / ml) und inkubieren Sie bei 37 ° C für 30 min. Bevor Sie mit dem Mikroskopie-Experiment fortfahren, waschen Sie die Zellen dreimal mit frischem Medium.
- Um ISGs in INS-1E-Zellen fluoreszierend zu markieren, transfizieren Sie die Zellen mit Transfektionsreagenz (siehe Materialtabelle) und C-peptide-enhanced green fluorescent protein (EGFP) Plasmid13 gemäß dem Protokoll des Herstellers und inkubieren Sie vor dem Experiment bei 37 ° C für 24 h in einer 5% CO2 -Atmosphäre.
2. Datenerfassung
- Damit sich das Mikroskop bei der gewünschten Temperatur und Atmosphäre ausgleicht, schalten Sie das Inkubator-Steuerungssystem des Mikroskops mindestens 2 Stunden vor dem Experiment ein.
HINWEIS: Jede Akquisition ist eine Zeitrafferreihe. - Erfassen Sie die Bilder mit einem invertierten konfokalen Mikroskop, das mit einem 60-fachen Wassertauchobjektiv (NA) mit 1,2-facher numerischer Apertur ausgestattet ist.
- Verwenden Sie einen 488-nm-Argon-Laser zur Anregung von EGFP- (transfizierten Zellen) und Fluorescein-markierten Makropinosomen. Sammeln Sie die Fluoreszenzemission zwischen 500 und 600 nm mit einem Standard-Photomultiplier-Röhrendetektor.
- Verwenden Sie einen 543 nm HeNe-Laser, um den Lysotracker anzuregen und seine Fluoreszenzemission zwischen 555 und 655 nm zu sammeln.
- Stellen Sie den Durchmesser des Detektionslochs auf die Größe 1 Airy ein. Sammeln Sie für jede Akquisition eine Reihe von 1000 aufeinanderfolgenden Frames. Stellen Sie die Pixelverweilzeit auf 2 μs/Pixel für eine Framezeit von 129 ms ein.
HINWEIS: Jeder Frame bestand aus 256 x 256 Pixeln (16 Bit/Pixel) mit einer physikalischen Größe von 69 nm/Pixel, was in etwa einer Fläche von 17 μm x 17 μm entspricht.
3. iMSD-Berechnung
HINWEIS: Um die Berechnung ordnungsgemäß auszuführen, verwenden Sie eine Software, die numerische Berechnungen und Skriptprogrammierung ermöglicht. Das spezifische Skript (d. h. für die Skriptdatei 'iMSD.m' siehe Unterstützende Datei 1) muss im selben Verzeichnis vorhanden sein, das die zu verarbeitende Bildserie enthält. Jedes Bild der Serie muss als eigenständige ".tif" -Datei gespeichert werden.
- Um die für die Akquisitionen verwendeten instrumentellen Parameter ordnungsgemäß zu initialisieren, öffnen Sie iMSD.m mit dem Software-Texteditor und ändern Sie den ersten Abschnitt wie folgt.
- Legen Sie N als Anzahl der Frames in der Zeitreihe fest (z. B. 1000 in diesem Protokoll).
- Set px_size: Pixelgröße, ausgedrückt in μm (z. B. 0,069 in diesem Protokoll).
- Set f: zeitliche Auflösung jedes Frames, ausgedrückt in Sekunden (z. B. 0,129 in diesem Protokoll).
- Legen Sie Filter fest: binäre Eingabe für die Hintergrundkorrektur, setzen Sie den Wert auf '0', um Rohbilder zu verarbeiten, oder setzen Sie den Wert auf '1', um eine schwellenwertbasierte Hintergrundsubtraktion durchzuführen.
- Legen Sie av_toll fest: Schwellenwert für die Hintergrundkorrektur; Jedes Pixel mit einer Intensität, die niedriger als dieser Wert ist, wird als 0 festgelegt, wenn Filter=1 ist.
- Legen Sie bit als ganzzahlige Zahl fest, die die Intensitätsabtastung bestimmt (z. B. 8 Bit, 16 Bit).
- Speichern Sie die bearbeitete Skriptdatei iMSD.m, und führen Sie sie aus.
- Überprüfen Sie die Skriptausführung.
HINWEIS: Der Berechnungsverarbeitungsstatus kann im Befehlsfenster überprüft werden. Wenn ein schwerwiegendes Problem auftritt, wird der Prozess unterbrochen, und es wird eine Warnmeldung angezeigt, um den Fehlertyp und den zugehörigen Zeilencode anzuzeigen. Führen Sie andernfalls die Schritte 3.3.1-3.3.3 aus.- Importieren Sie den Bildstapel und subtrahieren Sie den Hintergrund (falls erforderlich).
- Berechnen Sie die raumzeitliche Korrelationsfunktion G(ξ,η,τ) mit der Fourier-Methode.
- Passen Sie die räumlich-zeitliche Korrelationsfunktion an eine Gaußsche 2D-Funktion an.
HINWEIS: Am Ende des Prozesses werden die Ausgabewerte der σ 2(τ)-Formgebungsverfahren im Befehlsfenster angezeigt: Mittelwerte, Fehler und entsprechende Güte (R2) der Formstückung werden gemeldet.
- Überprüfen Sie die grafische Ausgabe.
HINWEIS: Die iMSD-Kurve und die entsprechenden Anpassungskurven werden in drei separaten Feldern dargestellt, die jeweils für eine andere Art von Anpassungsgleichung verwendet werden: Brownsche Diffusion, anomale Diffusion oder begrenzte Diffusion. Für jede Kurve wird derR2-Wert in der Diagrammlegende gemeldet. - Überprüfen Sie die Textausgabe.
HINWEIS: Am Ende des Prozesses wird eine .xls Datei mit dem gleichen Namen wie die ursprüngliche Zeitraffer-.tif Datei erstellt. Das erste Blatt enthält die Werte ξ, η, τ und σ2, die für jede Zeitverzögerung berechnet werden. Die Eingabeparameter und die hauptsächlich berechneten Ausgangswerte werden im zweiten Blatt angegeben, d. h. Diffusionskoeffizient, anomaler Koeffizient und y-Achse σ20-Schnittpunkt .
Ergebnisse
Der allgemeine Workflow der Methode ist in Abbildung 1 dargestellt. Es rekapituliert die im Protokollabschnitt vorgestellten Hauptschritte, von der Probenvorbereitung (Abbildung 1A) über die Zeitrafferbildgebung intrazellulärer Nanostrukturen (Abbildung 1B), die Fluktuationsanalyse für die Berechnung der Reihe der räumlich-zeitlichen Korrelationsfunktionen (Abbildung 1C) bis hin zur Anpassung an die Ableitung der durchschnittlichen strukturellen/dynamischen Eigenschaften des untersuchten Objekts (Abbildung 1D).
Ein kritischer Parameter ist die Zeitauflösung, die für die Abbildung des subzellulären Objekts von Interesse verwendet wird. Dieser experimentelle Wert legt die Zeitschwelle fest, bei der die minimale durchschnittliche Verschiebung der Objekte von Interesse gemessen wird. Die bevorzugte Bedingung ist jedoch die Einstellung einer Zeitauflösung der Bildgebung, bei der das Objekt von Interesse innerhalb des aufgenommenen Rahmens "unbeweglich" erscheint, d.h. es zeigt eine charakteristische Größe an, die im Durchschnitt aufgrund der Bildgebungsgeschwindigkeit nicht verformt wird. Dies ist technisch möglich, wenn der Gegenstand von Interesse eine membranumschlossene subzelluläre Struktur oder Organelle ist (wie in diesem Fall). Typischerweise weisen subzelluläre Strukturen lokale Diffusionskoeffizienten (D, μm2/s, siehe Tabelle 1) auf, die um mehrere Größenordnungen niedriger sind als die isolierter Einzelmoleküle im Zytoplasma (z. B. GFP21). Die Validierung kann durch künstliche Immobilisierung der interessierenden Organelle (z. B. durch chemische Fixierung) erfolgen. Tatsächlich kann diese Bedingung als Referenz dienen, um die tatsächliche Organellengröße unter den verwendeten experimentellen Bedingungen (z. B. Anregungswellenlänge, Pixelgröße, Ziel) zu bestimmen.
Obwohl hier Lysosomen als Testorganellen für dieses Verfahren verwendet wurden, sind die Ergebnisse unabhängig von der Zielstruktur gültig. Abbildung 2A zeigt ein Bild eines festen (d. h. unbeweglichen) Lysosoms zusammen mit einer Aufnahme, die an lebenden Zellen mit der entsprechenden zeitlichen Auflösung (d. h. typischerweise unter 100 ms/Frame; z. B. 65 ms/Frame im Beispiel in Abbildung 2B) durchgeführt wird, und eine Abnahme, die absichtlich mit sehr niedriger zeitlicher Auflösung durchgeführt wurde (z. B. 10 s/frame im Beispiel in Abbildung 2C ). Für jede Bedingung wird die Größe des diffundierenden Objekts wie folgt extrahiert: i) ein Intensitätsprofil des Spots wird durch das Linienwerkzeug in der ImageJ-Software abgeleitet; ii) Das Intensitätsprofil wird durch eine Gaußsche Funktion dargestellt und interpoliert, um den FWHM-Wert (Full Width at Half Maximum) zu berechnen, der wiederum als Schätzung des Spotdurchmessers verwendet wird (Abbildung 2D). Wie erwartet und im Diagramm von Abbildung 2E gezeigt, ergibt die Erfassung mit sehr hoher zeitlicher Auflösung (d. h. 65 ms/Frame) eine durchschnittliche Größe der Struktur, die der in der festen Stichprobe erhaltenen nahe kommt, entweder unter Verwendung des oben beschriebenen Standardwerkzeugs oder durch Extrahieren des i-MSD-y-Achsenabschnitts. Stattdessen führt die Erfassung mit langsamer Geschwindigkeit zu einer Zunahme der scheinbaren Größe der Struktur, was auf die natürliche Strukturdynamik während der Bildgebung zurückzuführen ist. Unter suboptimalen experimentellen Bedingungen spiegeln die extrahierten strukturellen/dynamischen Informationen die intrinsischen Eigenschaften des untersuchten Objekts nicht getreu wider.
Sobald die wichtigsten experimentellen Parameter ausgewählt sind, können Datensätze für die intrazellulären Zielstrukturen erstellt werden. Für Makropinosomen wurden nach 20 min Inkubation der Zellen mit 70 kDa Dextrans Zeitreihen der markierten intrazellulären Strukturen zu verschiedenen Zeitpunkten nach der Behandlung von 30 min bis ca. 180 min aufgenommen. Interessanterweise wird während des Handels eine allmähliche Veränderung der strukturellen und dynamischen Eigenschaften von Makropinosomen festgestellt (Abbildung 3; Verteilungen von σ02, Dm, α und N für Makropinosomen werden in den Diagrammen auf der linken Seite angegeben). Während während des Verkehrs keine offensichtlichen Veränderungen der lokalen Diffusivität (Dm) von Makropinosomen festgestellt werden, entwickeln sich sowohl die charakteristische Größe (σ02) als auch die allgemeine Bewegungsweise (α) im Laufe der Zeit.
Besonders hervorzuheben ist, dass während des Verkehrs eine Abnahme der durchschnittlichen Größe der Makropinosomen beobachtet wird (Abbildung 3A, links), zusammen mit einer gleichzeitigen Zunahme der subdiffusiven Natur ihrer Bewegung (d. h. bezeichnet als Abnahme der α-Werte, Abbildung 3C, linkes Feld). Darüber hinaus wurde die Anzahl der Makropinosomen aus jeder Akquisition extrahiert: Die Ergebnisse, die in Abbildung 3D, linkes Panel, dargestellt sind, zeigen deutlich eine Zunahme der Anzahl der Makropinosomen im Laufe der Zeit. All diese Ergebnisse stimmen gut mit den Erwartungen überein, da Dextran-markierte Makropinosomen als isolierte, große, membranumschlossene Vesikel an der Plasmamembran entstehen sollen (die auch für die Bewegung entlang zytoskelettaler Komponenten kompetent sind), aber allmählich mit dem endo-lysosomalen Weg kommunizieren sollen, der aus einer großen Population kleinerer und zufällig diffuser Strukturen besteht.
Wie oben erwartet, stehen die Ergebnisse für Makropinosomen im Gegensatz zu ähnlichen Messungen, die an ISGs durchgeführt wurden (Abbildung 3, rechte Spalte). Insulingranulate zeigen keinen sich zeitlich entwickelnden Trend der von iMSD abgeleiteten strukturellen / dynamischen Parameter (und ihrer durchschnittlichen Anzahl innerhalb der Zelle) im selben Zeitfenster, das für Makropinosomen beobachtet wurde. Darüber hinaus unterscheiden sich die charakteristischen Werte vonσ 02, Dm und α erheblich von denen von Lysosomen, die wiederum als Referenz verwendet werden. Dieses Ergebnis bestätigt die Idee, wie oben erwartet, dass Granulate in einem "stationären Zustand" untersucht werden, in dem die durchschnittlichen strukturellen/dynamischen Eigenschaften der gesamten Population von ISGs jederzeit unverändert bleiben (d.h. sie bleiben konstant, es sei denn, die stationären Zustandsbedingungen ändern sich beispielsweise aufgrund äußerer Reize).
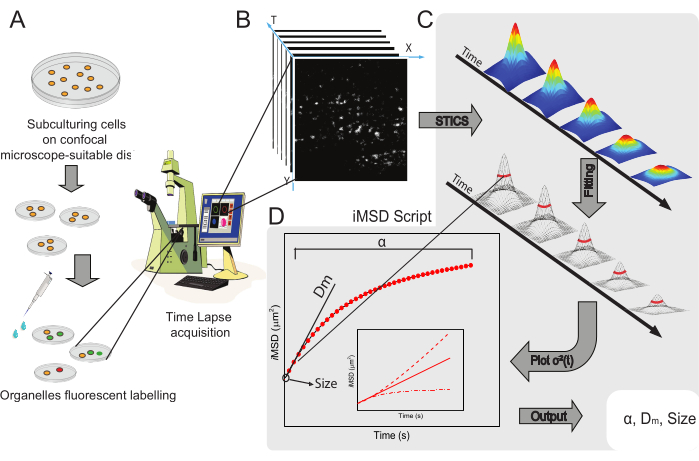
Abbildung 1: Experimenteller Arbeitsablauf . (A) Die Zellen wurden 24 h (48 h für Transfektionsexperimente) vor konfokalen Experimenten auf Zellschalen plattiert, die für mikroskopische Anwendungen geeignet sind. Die Zellen wurden dann nach der Markierungsmethode (siehe Protokoll) entsprechend behandelt, um die interessierende zytoplasmatische Organelle zu färben. (B) Eine typische konfokale Aufnahme besteht aus einem Stapel von Bildern (Zeitraffer) eines zytoplasmatischen Teils einer lebenden Zelle, der die zeitliche Entwicklung der markierten Organellendynamik beschreibt. (C) Der Zeitrafferfilm wird mit einem maßgeschneiderten Matlab-Skript analysiert, wobei zunächst die räumlich-zeitliche Bildkorrelationsfunktion und die Gaußsche Anpassung berechnet werden, um iMSD-Kurven (D) und verwandte extrahierte Anpassungsparameter zu zeichnen, die strukturelle Dynamikparameter von abgebildeten Organellen beschreiben. Abkürzungen: iMSD = Imaging-derived mean square displacement; STICS = räumlich-zeitliche Bildkorrelationsspektroskopie; α = anomaler Diffusionskoeffizient; Dm = lokale Diffusivität; σ2(τ) = Varianz. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
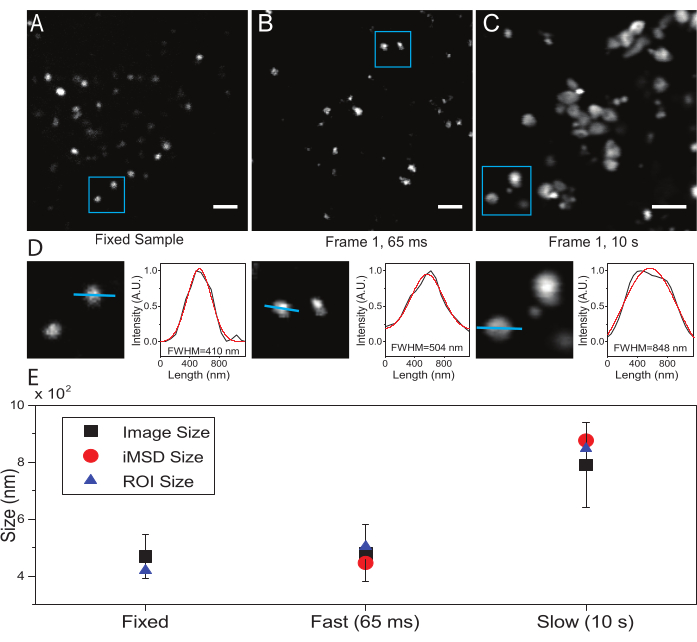
Abbildung 2: Richtige experimentelle Parameter. (A) Beispielhafte Darstellung von gefärbten Lysosomen in einer festen Probe. Maßstabsleiste = 2 μm. (B) Der erste Rahmen eines Stapels von Bildern von gefärbten Lysosomen in einer lebenden Zelle, aufgenommen mit den entsprechenden Parametern. Zeitliche Auflösung: 65 ms/Bild. Maßstabsleiste = 2 μm. (C) Der erste Rahmen eines Stapels von Bildern von gefärbten Lysosomen in einer lebenden Zelle, aufgenommen mit niedriger Geschwindigkeit: Artefaktische Verformung der scheinbaren Lysosomengröße aufgrund von Organellenbewegungen während der Bildgebung ist sichtbar. Zeitliche Auflösung: 10 s/Bild. Maßstabsleiste = 2 μm. (D) Beispiel für die Größenberechnung für abgebildete Lysosomen in einem blauen ROI von (A), (B) und (C). Das Intensitätsprofil entlang der blauen Linie wurde mit einer Gaußschen Funktion ausgestattet, um die FWHM abzurufen, d.h. eine Schätzung der Spotgröße. FWHM-Werte werden für jedes Fitting gemeldet. (E) Grafische Darstellung der Größenwerte, die durch die in Panel (D) beschriebene Bildanalyse für alle abgebildeten Lysosomen (schwarzes Quadrat, Mittelwert und Standardabweichung), für Lysosomen, die innerhalb des blauen ROI (blaues Dreieck) eingeschlossen sind, erhalten und durch iMSD-Analyse (roter Kreis) abgerufen wurden. Diese Zahl stammt aus dem Jahr 22. Abkürzungen: ROI = Region of interest; FWHM = volle Breite bei halbem Maximum; iMSD = Imaging-derived mean square displacement. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
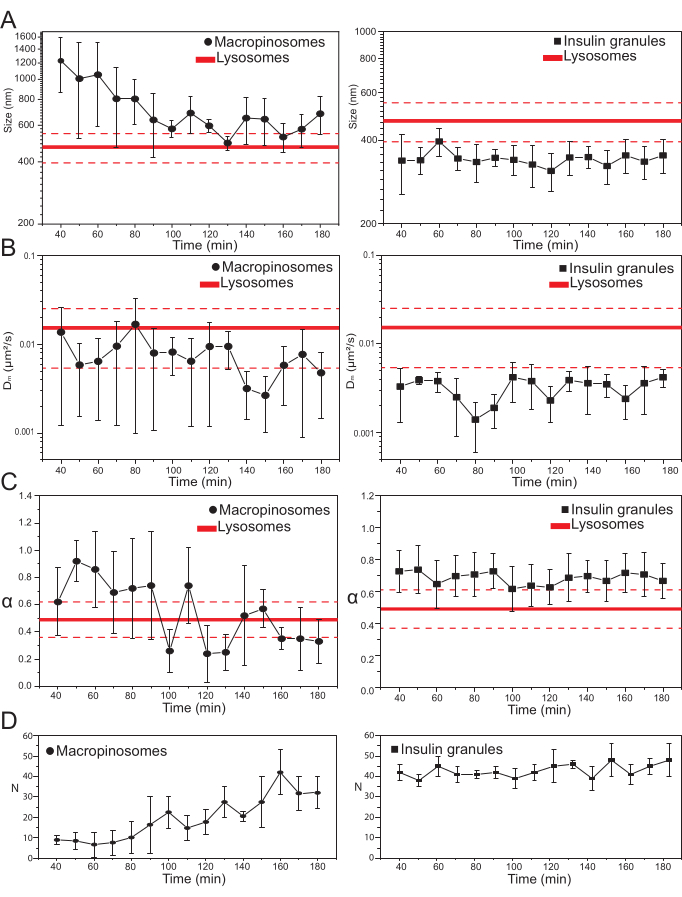
Abbildung 3: Zeitliche Entwicklung der markierten Organellen . (A) Plots von iMSD-extrahierten Größenwerten im Vergleich zum zeitlichen Verlauf nachfolgender Akquisitionen, die als Mittelwert der Messwerte in Akquisitionen dargestellt werden, die in einem 10-minütigen Zeitfenster durchgeführt werden. Links fortschreitende Reduktion der durchschnittlichen Größe von Makropinosomen (schwarze Kreise) und rechts die zeitinvariante Größe von insulinsekretorischen Granula (schwarze Quadrate) im Vergleich zu Lysosomen, dargestellt als Durchschnittsgrößenwert (dickere rote Linie) ± Standardabweichung (gestrichelte rote Linien). (B) und (C) Zeitprogression von Dm und α Koeffizienten für Makropinosomen (links) und Insulingranula (rechts), die durch iMSD-Analyse extrahiert wurden. (D) Zeitliche Entwicklung der Anzahl markierter Makropinosomen und Insulingranulate, gemessen im ersten Bild jedes erfassten Zeitrafferfilms. Abkürzungen: iMSD = Imaging-derived mean square displacement; α = anomaler Diffusionskoeffizient; Dm = lokale Diffusivität, N = Zahl. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
| Organell | Kennzeichnung | Zelllinie | Größe (nm) | Dm ( × 10-3 μm2/s) | α | N | Schiri. | ||
| Frühes Endosom (EE) | CellLight Early Endosom GFP | Hela | Artikel 395±74 | 3,0±2,4 | 1,02±0,20 | 40 | 10 | ||
| Spätes Endosom (LE) | CellLight Spätes Endosom GFP | Hela | Artikel 693±102 | 15,4±10,6 | 0,57±0,16 | 58 | 10 | ||
| Lysosom (LY) | LysoTracker DND-99 | Hela | 471±76 | 15,3±9,0 | 0,49±0,13 | 143 | 10, 14 | ||
| Caveola (CAV) | Caveolin-EGFP | Hela | 405±49 | 3,1±1,8 | 1,00±0,22 | 15 | 10 | ||
| Clathrin beschichtete Veskikel (CCV) | Transferrin-Alexa 488 | Hela | 513±62 | 16,2±9,9 | 0,48±0,17 | 33 | 10 | ||
| Insulingranulat (IG) | C-Peptid-EGFP | INS-1E | 335±56 | 3,0±1,7 | 0,70±0,14 | 107 | 11 | ||
| Frühes Macropinosom (EMCR) | Fluorescein-Dextran 70 kDa | Hela | 979±423 | 8,3±9 | 0,79±0,27 | 36 | 10 | ||
| Intermediäres Macropinosom (IMCR) | Fluorescein-Dextran 70 kDa | Hela | 702±180 | 13,7±19,9 | 0,60±0,38 | 29 | 10 | ||
| Spätes Macropinosom (LMCR) | Fluorescein-Dextran 70 kDa | Hela | 592±127 | 5,8±4,7 | 0,39±0,21 | 21 | 10 | ||
Tabelle 1: Strukturelle und dynamische iMSD-extrahierte Parameter. Die Tabelle zeigt die Werte Größe, Dm und α Koeffizienten, die für verschiedene Organellen gemessen wurden, und gibt die Markierungsstrategien, die verwendete Zelllinie und die Anzahl der analysierten Akquisitionen an. Werte werden als mittlere ± Standardabweichung angegeben. Abkürzungen: iMSD = Imaging-derived mean square displacement; α = anomaler Diffusionskoeffizient; Dm = lokale Diffusivität, N = Zahl; GFP = grün fluoreszierendes Protein; EGFP = Enhanced Green Fluorescent Protein.
Ergänzende Datei 1: Details zur iMSD-Trace-Ableitung und -Analyse. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Unterstützende Datei 1: Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Die Eigenschaften und Vorteile von iMSD sind im Vergleich zu den verfügbaren Techniken zum Abrufen analoger Informationen offensichtlich. Für strukturelle Informationen ist die Transmissionselektronenmikroskopie (TEM) -Analyse die bevorzugte Wahl. Mit dieser Methode können ultrastrukturelle Details in molekularer Auflösung und sogar darüber hinaus abgerufen werden, auch für subzelluläre Nanostrukturen. Dennoch wird die eigentümliche räumliche Auflösung von TEM auf Kosten der Informationen in der zeitlichen Dimension erreicht, was hier von Interesse ist. Um dies zu kompensieren, sind die jüngsten Fortschritte bei der Live-Cell-Bildgebungstechnologie von besonderem Interesse. Dazu gehören neue fluoreszierende Marker mit erhöhter Leistung (z. B. Helligkeit und Photostabilität), optimierte Markierungsverfahren und empfindlichere Detektoren. Darüber hinaus stehen Analysewerkzeuge zur Verfügung, um sowohl strukturelle (z. B. "Größe" durch phasorbasierte Analyse der lokalen Bildkorrelationsspektroskopie, PLICS 23, Aggregation/Oligomerisierung durch Number&Brightness-Analyse 24) als auch dynamische (z. B. Diffusionsgesetz durch Einzelpartikelverfolgung, d. H. (SPT) 25,26,27,28 ) Parameter auf der subzellulären Skala. Die SPT-Methode ermöglicht den direkten Zugriff auf die Objekttrajektorie und ihre MSD. Der Nachteil ist jedoch, dass eine geringe Dichte der Sonde und sehr helle Markierungen und viele Einzelobjekttrajektorien gemessen werden müssen, um statistische Kriterien zu erfüllen. In Bezug auf die zeitliche Auflösung der Messung können anorganische, fototaugliche Sonden (z. B. Quantenpunkte oder Metallnanopartikel) die SPT-Leistung erhöhen, jedoch auf Kosten komplexer Produktions- und Markierungsverfahren.
Im Vergleich zu diesen Standards zeigt die hier beschriebene iMSD-Methode einige entscheidende Vorteile. Erstens kann dieser Ansatz in Verbindung mit relativ schwachen fluoreszierenden Tags verwendet werden, wie z. B. genetisch kodierten fluoreszierenden Proteinen (z. B. der Anwendung auf ISGs). Somit wird im Vergleich zu SPT eine höhere zeitliche Auflösung (bei gleicher Beschriftung) aufgrund der geringeren Menge an benötigten Photonenerreicht 8. Zweitens ist die iMSD-Methode nur durch die zeitliche Auflösung, nicht aber durch die Beugung begrenzt. Tatsächlich können trotz des verwendeten beugungsbegrenzten optischen Aufbaus durchschnittliche molekulare Verschiebungen sogar unterhalb der Beugungsgrenze gemessen werden, wie bereits für molekulare Strömungen unter Verwendung von STICS29 gezeigt wurde. Die tatsächliche Auflösung bei der Messung von Verschiebungen hängt davon ab, wie genau (in Bezug auf Signal zu Rauschen) die Korrelationsfunktion gemessen werden kann, was erklärt, warum sie nicht durch Beugung begrenzt ist. Somit scheint klar zu sein, dass die minimale Verschiebung, die gemessen werden kann, von der Diffusivität des interessierenden Objekts und der zeitlichen Auflösung des Bildaufbaus abhängt.
In diesem Zusammenhang ist es wichtig zu berücksichtigen, dass die Anwendung auf subzelluläre Nanostrukturen wie Makropinosomen oder Insulingranulate mit einem Laser-Scanning-Mikroskop optimal ist: Die verfügbare Scangeschwindigkeit ist deutlich höher als die Dynamik des interessierenden Objekts. In einem solchen Fall ist die Bewegung der Objekte während der Erfassung vernachlässigbar, und die Korrelationsfunktion kann durch eine Gaußsche Funktion approximiert werden. Schließlich kann der iMSD-Ansatz leicht auf eine Vielzahl von kommerziellen optischen Mikroskopie-Setups angewendet werden, die auf Raster-Scan oder Weitfeld-Kamera-basierter Bildgebung basieren, ohne dass eine Systemkalibrierung erforderlich ist (nur erforderlich, wenn eine genaue Schätzung der Partikelgröße erreicht werden muss). Ein wichtiger Parameter für das Funktionieren der Methode ist die richtige räumliche Probenahme. Um eine zufriedenstellende Konvergenz des Anpassungsalgorithmus zu erreichen, sollte die Mindestgröße des interessierenden Bereichs für die Bildgebung in der Regel mindestens 3-mal größer sein als die maximale Verschiebung des Interesses.
Zusammenfassend lässt sich sagen, dass die iMSD-Methode nur ein Mikroskop erfordert, das für eine schnelle Erfassung ausgestattet ist. Die interessierende Struktur kann mit jedem genetisch kodierten oder organischen Fluorophor markiert werden, wodurch eine Mehrkanal-Bildgebung ermöglicht wird. Es ist vorgesehen, dass die Cross-iMSD-Analyse in naher Zukunft verwendet wird, um Subpopulationen subzellulärer Nanostrukturen auszuwählen und ihre Wechselwirkungen und Kodiffusion innerhalb der Zelle aufzudecken, wobei letzteres ein heißes Thema in der zellulären Biophysik ist. Wenn durch die iMSD-Analyse ein Detail verloren geht, hängt dies sicherlich mit der großen Menge an molekularer Information in dynamischen subzellulären Nanostrukturen zusammen. Solche Informationen werden während der Messung aufgrund der schlechten zeitlichen Auflösung unweigerlich gemittelt. Theoretisch gibt es jedoch keine technische Grenze aufgrund der Möglichkeit, molekulare Informationen abzurufen, vorausgesetzt, dass ausreichende Erfassungsgeschwindigkeiten erreicht werdenkönnen 8. Aufgrund der kontinuierlichen Verbesserungen der Detektorgeschwindigkeit / -empfindlichkeit und der Bildgebungstechnologien ist vorgesehen, dass Informationen über das gesamte subzelluläre Kompartiment und seine molekularen Bestandteile aus einem einzigen Datensatz extrahiert werden.
Offenlegungen
Die Autoren haben keinen Interessenkonflikt zu erklären.
Danksagungen
Diese Arbeit wurde vom Europäischen Forschungsrat (ERC) im Rahmen des Forschungs- und Innovationsprogramms Horizon 2020 der Europäischen Union (Finanzhilfevereinbarung Nr. 866127, Projekt CAPTUR3D) gefördert.
Materialien
| Name | Company | Catalog Number | Comments |
| 100x Penicillin-Streptomycin-Glutamine | Gibco | 10378-016 | Cell medium supplement |
| C-peptide-EGFP | Plasmid | ||
| DMEM High Glucose | Gibco | 31053028 | Cell medium (HeLa) |
| FBS | Gibco | 10082147 | Cell medium supplement |
| Fluorescein isothiocyanate-dextran 70 kDa | Sigma Aldrich | 46945-100MG-F | Reagent |
| HeLa | ATCC | CCL-61 | Cell Line |
| Lipofectamine 2000 | TermoFisher | 11668019 | Trasfection reagent |
| Lysotracker Red DND-99 | Gibco | L7528 | Reagent |
| Matlab | MathWork | Software | |
| Microscope-suitable cell dishes | Willco | GWSt-3522 | Petri dishes |
| Olympus FV1000 | Olympus Japan | Confocal microscope | |
| RPMI 1640 | Gibco | 11835063 | Cell medium (INS-1E) |
Referenzen
- Murphy, J. E., Padilla, B. E., Hasdemir, B., Cottrell, G. S., Bunnett, N. W. Endosomes: a legitimate platform for the signaling train. Proceedings of the National Academy of Sciences of the United States of America. 106 (42), 17615-17622 (2009).
- Mosesson, Y., Mills, G. B., Yarden, Y. Derailed endocytosis: an emerging feature of cancer. Nature Reviews Cancer. 8 (11), 835-850 (2008).
- Mellman, I., Yarden, Y. Endocytosis and cancer. Cold Spring Harbor Perspectives in Biology. 5 (12), 016949(2013).
- Di Fiore, P. P. Endocytosis, signaling and cancer, much more than meets the eye. Preface. Molecular Oncology. 3 (4), 273-279 (2009).
- Bogan, J. S., Xu, Y., Hao, M. Cholesterol accumulation increases insulin granule size and impairs membrane trafficking. Traffic. 13 (11), 1466-1480 (2012).
- Ballabio, A., Gieselmann, V. Lysosomal disorders: From storage to cellular damage. Biochimica et Biophysica Acta. 1793 (4), 684-696 (2009).
- Hu, Y. -B., Dammer, E. B., Ren, R. -J., Wang, G. The endosomal-lysosomal system: from acidification and cargo sorting to neurodegeneration. Translational Neurodegeneration. 4, 18(2015).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. Spatiotemporal fluctuation analysis: a powerful tool for the future nanoscopy of molecular processes. Biophysical Journal. 111 (4), 679-685 (2016).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. Fast spatiotemporal correlation spectroscopy to determine protein lateral diffusion laws in live cell membranes. Proceedings of the National Academy of Sciences of the United States of America. 110 (30), 12307-12312 (2013).
- Di Rienzo, C., Gratton, E., Beltram, F., Cardarelli, F. From fast fluorescence imaging to molecular diffusion law on live cell membranes in a commercial microscope. Journal of Visualized Experiments: JoVE. (92), e51994(2014).
- Digiacomo, L., et al. Dynamic fingerprinting of sub-cellular nanostructures by image mean square displacement analysis. Scientific Reports. 7 (1), 14836(2017).
- Durso, W., et al. Lysosome dynamic properties during neuronal stem cell differentiation studied by spatiotemporal fluctuation spectroscopy and organelle tracking. International Journal of Molecular Sciences. 21 (9), 3397(2020).
- Ferri, G., et al. Insulin secretory granules labelled with phogrin-fluorescent proteins show alterations in size, mobility and responsiveness to glucose stimulation in living β-cells. Scientific Reports. 9 (1), 2890(2019).
- Durso, W., D'Autilia, F., Amodeo, R., Marchetti, L., Cardarelli, F. Probing labeling-induced lysosome alterations in living cells by imaging-derived mean squared displacement analysis. Biochemical and Biophysical Research Communications. 503 (4), 2704-2709 (2018).
- Digiacomo, L., Digman, M. A., Gratton, E., Caracciolo, G. Development of an image Mean Square Displacement (iMSD)-based method as a novel approach to study the intracellular trafficking of nanoparticles. Acta Biomaterialia. 42, 189-198 (2016).
- Swanson, J. A., Watts, C. Macropinocytosis. Trends in Cell Biology. 5 (11), 424-428 (1995).
- Jones, A. T. Macropinocytosis: searching for an endocytic identity and role in the uptake of cell penetrating peptides. Journal of Cellular and Molecular Medicine. 11 (4), 670-684 (2007).
- Falcone, S., et al. Macropinocytosis: regulated coordination of endocytic and exocytic membrane traffic events. Journal of Cell Science. 119, Pt 22 4758-4769 (2006).
- Kerr, M. C., et al. Visualisation of macropinosome maturation by the recruitment of sorting nexins. Journal of Cell Science. 119, Pt 19 3967-3980 (2006).
- Rorsman, P., Renstrom, E. Insulin granule dynamics in pancreatic beta cells. Diabetologia. 46 (8), 1029-1045 (2003).
- Di Rienzo, C., Piazza, V., Gratton, E., Beltram, F., Cardarelli, F. Probing short-range protein Brownian motion in the cytoplasm of living cells. Nature Communications. 5 (1), 5891(2014).
- Ferri, G., et al. Time-lapse confocal imaging datasets to assess structural and dynamic properties of subcellular nanostructures. Scientific Data. 5 (1), 180191(2018).
- Scipioni, L., Gratton, E., Diaspro, A., Lanzanò, L. Phasor analysis of local ICS detects heterogeneity in size and number of intracellular vesicles. Biophysical Journal. 111 (3), 619-629 (2016).
- Digman, M. A., Dalal, R., Horwitz, A. F., Gratton, E. Mapping the number of molecules and brightness in the laser scanning microscope. Biophysical Journal. 94 (6), 2320-2332 (2008).
- Li, C. H., Bai, L., Li, D. D., Xia, S., Xu, T. Dynamic tracking and mobility analysis of single GLUT4 storage vesicle in live 3T3-L1 cells. Cell Research. 14 (6), 480-486 (2004).
- Donovan, K. W., Bretscher, A. Tracking individual secretory vesicles during exocytosis reveals an ordered and regulated process. Journal of Cell Biology. 210 (2), 181-189 (2015).
- Westphal, V., et al. Video-rate far-field optical nanoscopy dissects synaptic vesicle movement. Science. 320 (5873), 246-249 (2008).
- Tabei, S. M. A., et al. Intracellular transport of insulin granules is a subordinated random walk. Proceedings of the National Academy of Sciences of the United States of America. 110 (13), 4911-4916 (2013).
- Hebert, B., Costantino, S., Wiseman, P. W. Spatiotemporal image correlation spectroscopy (STICS) theory, verification, and application to protein velocity mapping in living CHO cells. Biophysical Journal. 88 (5), 3601-3614 (2005).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten