Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Determinación de la respiración mitocondrial y la glucólisis en muestras de tejido retiniano ex vivo
En este artículo
Resumen
Aquí se describe un protocolo detallado para realizar el ensayo de estrés mitocondrial y el ensayo de velocidad glucolítica en muestras de tejido retiniano ex vivo utilizando un bioanalizador comercial.
Resumen
La respiración mitocondrial es una vía crítica de generación de energía en todas las células, especialmente en los fotorreceptores de la retina que poseen un metabolismo altamente activo. Además, los fotorreceptores también exhiben una alta glucólisis aeróbica como las células cancerosas. Las mediciones precisas de estas actividades metabólicas pueden proporcionar información valiosa sobre la homeostasis celular en condiciones fisiológicas y en estados de enfermedad. Se han desarrollado ensayos basados en microplacas de alto rendimiento para medir la respiración mitocondrial y diversas actividades metabólicas en células vivas. Sin embargo, una gran mayoría de estos están desarrollados para células cultivadas y no han sido optimizados para muestras de tejido intacto y para su aplicación ex vivo. Aquí se describe un protocolo detallado paso a paso, utilizando tecnología de fluorescencia basada en microplacas, para medir directamente la tasa de consumo de oxígeno (OCR) como indicador de la respiración mitocondrial, así como la tasa de acidificación extracelular (ECAR) como indicador de glucólisis, en tejido retiniano ex vivo intacto. Este método se ha utilizado para evaluar con éxito las actividades metabólicas en la retina del ratón adulto y demostrar su aplicación en la investigación de los mecanismos celulares del envejecimiento y la enfermedad.
Introducción
Las mitocondrias son orgánulos esenciales que regulan el metabolismo celular, la señalización, la homeostasis y la apoptosis mediante la coordinación de múltiples procesos fisiológicos cruciales1. Las mitocondrias sirven como la fuente de energía en la célula para generar trifosfato de adenosina (ATP) a través de la fosforilación oxidativa (OXPHOS) y proporcionar energía que soporta casi todos los eventos celulares. La mayoría del oxígeno celular se metaboliza en las mitocondrias, donde sirve como el aceptor final de electrones en la cadena de transporte de electrones (ETC) durante la respiración aeróbica. También se pueden producir bajas cantidades de ATP a partir de la glucólisis en el citosol, donde la glucosa se convierte en piruvato, que puede convertirse en lactato o transportarse a las mitocondrias y oxidarse a acetil-CoA, un sustrato en el ciclo del ácido tricarboxílico (ciclo TCA).
La retina es uno de los tejidos metabólicamente más activos en mamíferos2, mostrando altos niveles de respiración mitocondrial y un consumo de oxígeno extremadamente alto3. Los fotorreceptores de bastón y cono contienen una alta densidad de mitocondrias4, y OXPHOS genera la mayor cantidad de ATP en la retina5. Además, la retina también depende en gran medida de la glucólisis aeróbica6,7 al convertir la glucosa en lactato5. Los defectos mitocondriales se asocian a diversas enfermedades neurodegenerativas8,9; y con sus altas demandas energéticas únicas, la retina es especialmente vulnerable a los defectos metabólicos, incluidos los que afectan a OXPHOS4 mitocondrial y la glucólisis10. La disfunción mitocondrial y los defectos de la glucólisis están implicados en enfermedades degenerativas de retinal11,12 y macular13, degeneración macular asociada a la edad10,14,15,16 y retinopatía diabética17,18. Por lo tanto, las mediciones precisas de la respiración mitocondrial y la glucólisis pueden proporcionar parámetros importantes para evaluar la integridad y la salud de la retina.
La respiración mitocondrial se puede medir a través de la determinación de la tasa de consumo de oxígeno (OCR). Dado que la conversión de glucosa en piruvato y posteriormente en lactato da como resultado la extrusión de protones y la acidificación del entorno extracelular, las mediciones de la tasa de acidificación extracelular (ECAR) proporcionan una indicación del flujo de glucólisis. Como la retina está compuesta por múltiples tipos de células con relaciones íntimas y sinergia activa, incluido el intercambio de sustratos6, es imperativo analizar la función mitocondrial y el metabolismo en el contexto de todo el tejido retiniano con laminación y circuitos intactos. Durante las últimas décadas, los electrodos de O2 tipo Clark y otros microelectrodos de oxígeno se han utilizado para medir el consumo de oxígeno en la retina19,20,21. Estos electrodos de oxígeno tienen limitaciones importantes en la sensibilidad, el requisito de un gran volumen de muestra y la necesidad de agitación continua de la muestra en suspensión, lo que generalmente conduce a la interrupción del contexto celular y tisular. El protocolo descrito aquí se desarrolló utilizando una técnica de fluorescencia basada en microplacas para medir el metabolismo energético mitocondrial en tejido de retina de ratón ex vivo recién diseccionado. Permite mediciones en tiempo real de rendimiento medio de OCR y ECAR simultáneamente utilizando una pequeña muestra (punzón de 1 mm) de tejido retiniano ex vivo, evitando la necesidad de suspensión y agitación continua.
Aquí se demuestra el procedimiento experimental para el ensayo de estrés mitocondrial y el ensayo de tasa glucolítica en discos de punción retiniana recién diseccionados. Este protocolo permite la medición de las actividades metabólicas relacionadas con las mitocondrias en un contexto tisular ex vivo. A diferencia de los ensayos realizados con células cultivadas, las lecturas obtenidas aquí reflejan el metabolismo energético combinado a nivel tisular y están influenciadas por las interacciones entre los diferentes tipos de células dentro del tejido. El protocolo se modifica a partir de una versión publicada anteriormente22,23 para adaptarse a la nueva generación del analizador de flujo extracelular Agilent Seahorse de 24 pocillos (XFe24) con placa de captura de islotes. El medio de ensayo, las concentraciones de compuestos de inyección y el número/duración de los ciclos de ensayo también se han optimizado para el tejido retiniano. Se proporciona un protocolo detallado paso a paso para la preparación de discos perforados de retina. Se puede obtener más información sobre la configuración del programa y el análisis de datos en la guía del usuario del fabricante24,25,26.
Protocolo
Todos los protocolos de ratón fueron aprobados por el Comité de Cuidado y Uso de Animales del Instituto Nacional del Ojo (NEI ASP # 650). Los ratones fueron alojados en condiciones de luz-oscuridad de 12 h y cuidados siguiendo las recomendaciones de la Guía para el Cuidado y Uso de Animales de Laboratorio, el Instituto de Recursos Animales de Laboratorio y la Política del Servicio de Salud Pública sobre Cuidado Humanitario y Uso de Animales de Laboratorio.
1. Cartucho del sensor hidratante y preparación del medio de ensayo
- El día antes del experimento, agregue 1 ml del medio de calibración a cada pozo de la placa de utilidad. Coloque la cubierta Hydro-Booster en la parte superior y baje el cartucho del sensor a través de la abertura de la cubierta. Compruebe que el sensor está sumergido en el medio de calibración. Incubar el cartucho del sensor durante la noche en una incubadora libre de CO2 a 37 °C para activar los fluoróforos.
NOTA: Para evitar la evaporación, la incubadora se humidifica manteniendo una bandeja de agua en el interior, y el cassette del cartucho del sensor se envuelve con una envoltura de plástico transparente. - Prepare el medio de ensayo reconstituyendo el medio Seahorse DMEM con la adición de glucosa, piruvato y glutamina a las concentraciones deseadas. En los ensayos reportados en este artículo, la concentración final de sustratos en el medio de ensayo son: 6 mM de glucosa, 0.12 mM de piruvato y 0.5 mM de glutamina. Para cada placa de ensayo, 40 ml del medio de ensayo se preparan frescos el día del experimento.
- Configure el programa de ensayo en el analizador siguiendo las instrucciones del fabricante26. En el ensayo demostrado aquí, el protocolo se establece de la siguiente manera: 5 ciclos de mediciones para la línea de base, luego inyectar el puerto A, seguido de 4 ciclos de mediciones, luego inyectar el puerto B y seguido de 4 ciclos de mediciones. Cada ciclo se compone de mezcla (3 min), espera (2 min) y medida (3 min).
2. Revestimiento de inserciones de malla de microplaca de captura de islotes
- Prepare la mezcla de recubrimiento combinando 20 μL del medio de fijación celular (por ejemplo, Cell-Tak) con 171 μL de bicarbonato de sodio de 0,1 M y 9 μL de 1 M de NaOH.
- Abra la tapa del cassette que contiene las inserciones de malla. Pipete 8 μL de la mezcla de recubrimiento a cada inserción de malla. Use una punta de pipeta para untar / extender suavemente la gota para distribuir la mezcla de recubrimiento por igual en todo el inserto de malla.
- Cierre el cassette y permita que los insertos de malla se incuben a temperatura ambiente durante al menos 25 minutos para la adsorción.
- Lave el inserto de malla pipeteando 4 ml del medio de ensayo directamente sobre los insertos de malla. Agite suavemente el cassette para asegurarse de que todos los insertos de malla se laven con el medio de ensayo.
- Mantenga el inserto de malla a un lado. Está listo para usar.
3. Preparación de compuestos de inyección
- Sacar las alícuotas de Bam15 (10 mM), Rotenona (10 mM), Antimicina A (10 mM) y 2-DG (500 mM) desde -80 °C congelador y descongelar a temperatura ambiente.
NOTA: El stock 2-DG está listo para usar. Los otros medicamentos deben diluirse en stock de trabajo. - Calentar 10 ml del medio de ensayo en un baño de agua a 37 °C.
- Diluir 10 mM bam15 stock a 50 μM de material de trabajo utilizando un procedimiento de dilución de dos pasos: mezclar 20 μL de stock de 10 mM con 20 μL de DMSO para obtener 5 mM de stock intermedio. A continuación, mezcle 10 μL del material intermedio anterior a 5 mM con 990 μL de medio de ensayo precalentado para obtener el material de trabajo final de 50 μM.
- Diluir y combinar 10 mM de rotenona y 10 mM de antimicina A en 10 μM de rotenona/antimicina A (Podredumbre/AA) en dos pasos de diluciones: mezclar 10 μL cada uno de 10 mM de rotenona y 10 mM de antimicina A con 80 μL de DMSO para obtener 1 mM de podredumbre/AA de material intermedio. A continuación, mezcle 10 μL del material intermedio anterior de 1 mM con 990 μL de medio de ensayo precalentado para obtener el material de trabajo final de 10 μM Rot/AA.
- Prepare recién preparados los materiales de trabajo de compuestos de inyección mencionados anteriormente el día del experimento y déjelos a un lado a temperatura ambiente hasta que se carguen en los puertos de inyección del cartucho del sensor.
4. Disección retiniana y preparación del punzón retiniano
- Eutanasia de un ratón por asfixia por CO2 siguiendo las Directrices AVMA sobre Eutanasia27.
NOTA: No deje al animal en una cámara de CO2 más tiempo del necesario para la eutanasia. - Enucle los ojos y colóquelos en un tampón de PBS 1x viejo de hielo en una placa de Petri y luego colóquelo bajo un microscopio de disección.
- Retire con cuidado, cortando con microscisores, los músculos rectos adicionales unidos fuera del globo ocular y corte el nervio óptico.
- Use una aguja de 30 G para perforar un orificio en el borde de la córnea (limbo); esto sirve como el sitio de inserción para los microscisores. Luego, use una microescisora de disección fina para hacer un corte circular a lo largo del borde de la córnea, separándola de la copa del ojo posterior.
- Use fórceps de disección afilados para eliminar la córnea, el cristalino y el humor vítreo de la copa del ojo.
- Use microescisores de disección fina para hacer varios cortes pequeños en la capa escleral en el borde de la copa del ojo. Evite cortar la capa de retina. Use dos pinzas de disección afiladas para sujetar el tejido escleral a cada lado del corte y tire con mucho cuidado de la capa escleral para extraerla de la retina neural. Repita esto alrededor de la copa del ojo hasta que se extirpe toda la esclerótica y se obtenga una copa de retina intacta.
- Use microescisores de disección y haga cortes radiales en la copa de la retina para aplanarla y generar varias secciones distintas.
NOTA: Dependiendo de las habilidades de disección de la persona y la experiencia en el manejo de tejido retiniano fresco, la copa de la retina se puede cortar para generar de 3 a 5 secciones distintas. - Use un punzón de biopsia de 1 mm de diámetro para cortar un disco de retina de cada sección de la copa retiniana aplanada.
NOTA: Se debe tener cuidado de perforar los discos de retina a la misma distancia de la cabeza del nervio óptico. - Use fórceps para transferir los insertos de malla pre-recubiertos en la placa de Petri de disección. Con la ayuda de dos cepillos de pestañas superfinos, coloque el disco de punzonado de retina en el inserto de malla. El disco perforador de retina se coloca en el centro del inserto de malla con la capa de células ganglionares hacia abajo tocando la malla y la capa fotorreceptora hacia arriba.
NOTA: Con frecuencia, algunas células RPE permanecen unidas a los fotorreceptores, y la pigmentación de estas células se puede utilizar como un indicador de la orientación del disco punzante retiniano.
5. Carga de los puertos de inyección del cartucho del sensor y calibración
- Saque el cassette de la placa del cartucho del sensor hidratado de la incubadora de 37 °C. Retire la cubierta hydro-Booster y vuelva a colocar el cartucho del sensor en la placa de servicio.
- Cargue el volumen deseado de soluciones de material de trabajo compuesto de inyección en los puertos apropiados. Sostenga la punta de la pipeta en un ángulo de 45 °. Inserte la punta de la pipeta hasta la mitad en un puerto de inyección con el bisel de la punta contra la pared opuesta del puerto de inyección y cargue suavemente el compuesto en cada puerto. Evite introducir burbujas de aire.
- Consulte la guía del usuario del instrumento para conocer el volumen del compuesto cargado en cada puerto de inyección para un ensayo específico. En los experimentos presentados en este artículo, 68 μL de material de trabajo bam15 de 50 μM (para el ensayo de estrés mitocondrial) o 68 μL de material de trabajo rot/AA de 10 μM (para el ensayo de velocidad glucolítica) se cargan en el puerto A; 75 μL de material de trabajo 10 μM Rot/AA (para ensayo de estrés mitocondrial) o 75 μL de material de trabajo 2-DG de 500 mM (para ensayo de velocidad glucolítica) se cargan en el puerto B.
- Puertos de inyección de carga de todos los pozos de la placa, incluidos los pozos de corrección de fondo y los pozos en blanco para garantizar una inyección adecuada. Cargue la solución compuesta respectiva en cada puerto para los pozos de corrección de fondo. El medio de ensayo puede ser sustituido, en lugar de la solución compuesta, en cada uno de los puertos de los pozos del banco.
- Coloque la placa del cartucho del sensor cargada, con la tapa apagada, en la máquina analizadora para iniciar la calibración antes de la ejecución del ensayo. Una vez finalizada la calibración, el programa se detendrá automáticamente, esperando el reemplazo de la placa de utilidad con la placa de captura del islote que contiene punzones de retina.
6. Carga de la placa de captura del islote e inicio de la ejecución del ensayo
- Añadir 607 μL del medio de ensayo a cada pocillo de la placa de captura del islote
- Use fórceps para agarrar el borde del inserto de malla que contiene discos perforadores de retina en la parte superior y sáquelo de la placa de Petri. Golpee ligeramente la parte inferior del inserto de malla en un pañuelo absorbente para eliminar el líquido adicional y colóquelo en el pozo de la placa de captura del islote. Repita este paso hasta que todos los insertos de malla con punzones de retina se coloquen en la placa de captura del islote. Llene los pozos de corrección de fondo y los pozos en blanco con inserciones de malla vacías.
- Use dos pinzas Graefe para presionar con cuidado y suavidad el borde de cada inserto de malla y asegúrese de que estén insertadas de manera segura en la parte inferior de la placa de captura del islote.
- Coloque la placa de captura del islote cargada en una incubadora de 37 °C durante 5 minutos para calentarse.
- Expulse la placa de utilidad después de que se complete la calibración y reemplácela por una y reemplácela con la placa de captura de islotes, con la tapa apagada, que contenga punzones de retina.
- Reanude la ejecución del ensayo.
7. Ejecute la terminación y el almacenamiento de datos
- Una vez completada la ejecución, expulse el cartucho del sensor y la placa de captura del islote que contiene punzones retinianos. Los datos se guardan automáticamente como archivo .asyr.
- Utilice el software de análisis de datos asociado para ver y analizar los datos siguiendo la guía del usuario del fabricante26.
- Utilice la función Exportar para exportar el archivo .xslx de los datos, que se puede ver y analizar utilizando un software de hoja de cálculo.
8. Guardar la muestra de punzón retiniano
- Después del ensayo, saque la placa de la máquina, retire el cartucho del sensor y retire suavemente el medio de ensayo de cada pozo con una pipeta.
- Vuelva a aplicar la cubierta y selle los lados de la placa con la tira de parafilm.
- Conservar a -80 °C.
- Para la normalización, cuantifique el contenido total de ADN o proteína del punzón en cada pozo.
9. Análisis de datos
- Ensayo de estrés mitocondrial
NOTA: El valor de OCR medido (totalOCR) representa el consumo total de oxígeno por parte del tejido. Después de la inyección de Bam15 (desacoplador), el OCR aumenta desde el nivel basal (totalOCRbasal) hasta el nivel máximo (totalOCRmax) y disminuye después de la inyección de Rot/AA. El valor de OCR residual después de la inyección de Rot/AA (totalOCRRot/AA) representa el consumo de oxígeno no mitocondrial.- Calcule el consumo de oxígeno relacionado con las mitocondrias como:
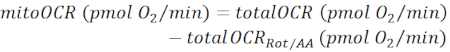 (Eq. 1) 28
(Eq. 1) 28
- Calcule la capacidad de reserva mitocondrial (MRC) como:
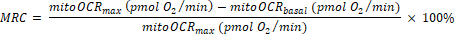 (Eq. 2) 29
(Eq. 2) 29
NOTA: La última lectura entre las 5 mediciones antes de la inyección de Bam15 se toma como el valor "basal" (para totalOCRbasal y mitoOCRbasal). La lectura más alta entre las 4 mediciones después de la inyección de Bam15 se utiliza como valor "máximo" (para totalOCRmax y mitoOCRmax). La lectura más baja entre las 4 mediciones después de la inyección de Rot/AA se utiliza como totalOCRRot/AA.
- Calcule el consumo de oxígeno relacionado con las mitocondrias como:
- Ensayo de velocidad glucolítica
NOTA: El valor ECAR medido (totalECAR) representa la acidificación total del medio por la actividad metabólica del tejido. En general, la acidificación del microambiente extracelular resulta principalmente por extrusión del producto glicolítico, el lactato. El catabolismo de sustratos en el ciclo mitocondrial del TCA da como resultado la producción de CO2, que también acidifica el medio extracelular a través de la hidratación al bicarbonato.- Substract mitocondrial contribuyó a la acidificación media (mitoECAR) de totalECAR para obtener el glycoECAR.
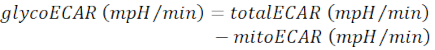 (Eq. 3) 28
(Eq. 3) 28
NOTA: La respiración mitocondrial y el ciclo de TCA son procesos fuertemente acoplados. La producción de CO2 a partir de las mitocondrias es una función de la tasa de OXPHOS, que es medible por mitoOCR. - Calcule el mitoECAR como:
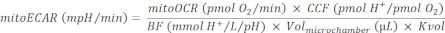 (Eq. 4) 28
(Eq. 4) 28
donde, el CCF (Factor de Contribución de CO2) es un valor de relación calculado empíricamente, que representa la cantidad de contribución de H + de la acidificación mediada por CO2 frente a cada consumo de O2 de OXPHOS. El CCF para este sistema está predeterminado a ser 0.6028. La medición precisa de la acidificación del medio está determinada por la capacidad tampón del medio, la sensibilidad del sensor de pH del instrumento y la capacidad efectiva de la cámara de medición. Aquí, el BF (Buffer Factor) es un parámetro de la capacidad de buffer experimental in situ, que representa la cantidad de H+ u OH- añadida a la cámara de medición efectiva para cambiar el nivel de pH en 1 unidad. Cuando se utiliza un medio de ensayo personalizado, el BF se puede determinar titulando cantidades conocidas de ácido en el medio de ensayo siguiendo el protocolo del factor tampón30. El Seahorse DMEM medium pH 7.4 utilizado en este protocolo tiene un BF predeterminado de 2.60 mmol H +/ L / pH. La placa de captura de islotes utilizada en este protocolo tiene una Volmicrocámara = 16,6 μL31. El factor de escalado de volumen, Kvol, es una constante determinada empíricamente. El valor de Kvol no está disponible para la placa de captura de islotes, pero se puede calcular a partir del valor de la microplaca28, teniendo en cuenta la diferencia de volumen en sus microcámaras, que es de 0,41.
NOTA: La inyección de rot /AA apaga la respiración mitocondrial y obliga al tejido a cambiar a la glucólisis para la producción de ATP, lo que lleva a una mayor extrusión de lactato y un aumento en la medición de ECAR. La glucólisis se interrumpe con la inyección de 2-DG, y la medición residual de ECAR revela acidificación no glicolítica y no mitocondrial del medio. - Calcule la capacidad de reserva glucolítica (GRC) como:
 (Eq. 5) 32
(Eq. 5) 32
donde, la última lectura entre las 5 mediciones antes de la inyección de Rot/AA se toma como el valor "basal" (glicoECARbasal). La lectura más alta entre las 4 mediciones después de la inyección de Rot/AA se utiliza como valor "máximo" (glycoECARmax). La lectura más baja entre las 4 mediciones después de la inyección de 2-DG se utiliza como glycoECAR2-DG.
- Substract mitocondrial contribuyó a la acidificación media (mitoECAR) de totalECAR para obtener el glycoECAR.
- Normalización
NOTA: La normalización es esencial cuando se comparan las lecturas de tejidos retinianos de diferentes grupos de edad o entre muestras de tipo salvaje y patológicas/degenerativas, que pueden diferir en el número de células.- Utilice kits disponibles comercialmente para evaluar el contenido de ADN en cada disco punzante retiniano33,34.
- Alternativamente, use un tampón de ensayo de radioinmunoprecipitación (tampón RIPA) para extraer la proteína total del punzón retiniano y use el contenido de proteína para la normalización.
NOTA: El área de superficie de la retina de un ratón adulto se ha determinado previamente que es de alrededor de 20 mm2, y cada retina contiene ~ 6.5 millones de células35. Por lo tanto, cada punzón retiniano de 1 mm de diámetro es ~ 1/25 de una sola retina y contiene ~ 260K células. Uno puede referirse a estos números al comparar los datos de un punzón retiniano con los de otras muestras de tejido o células cultivadas,
Resultados
Los datos reportados aquí son ensayos representativos de estrés mitocondrial que muestran trazas de OCR (Figura 1) y ensayos de tasa glucolítica que muestran trazas de OCR y trazas de ECAR (Figura 2), que se realizaron utilizando discos perforados de retina de 1 mm recién diseccionados de ratones transgénicos Nrl-L-EGFP de 4 meses de edad36 (fondo C57B / L6). Estos ratones expresan GFP específicamente en fotorreceptores de ...
Discusión
Aquí se proporcionan instrucciones detalladas para realizar ensayos basados en microplacas de la respiración mitocondrial y la actividad de la glucólisis utilizando discos perforadores de retina recién diseccionados ex vivo. El protocolo se ha optimizado para: 1) garantizar el uso de un medio de ensayo adecuado para el tejido retiniano ex vivo; 2) emplear el tamaño adecuado de los discos perforadores de retina para obtener lecturas de OCR y ECAR que se encuentren dentro del rango de detección ópt...
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo es apoyado por el Programa de Investigación Intramuros del Instituto Nacional del Ojo (ZIAEY000450 y ZIAEY000546).
Materiales
| Name | Company | Catalog Number | Comments |
| 1X PBS | Thermo Fisher | 14190-144 | |
| 2-Deoxy glucose (2-DG), 500 mM stock solution | Sigma | D6134 | Dissolve in Seahorse XF DMEM medium, prepare ahead of time |
| 30-gauge needle | BD Precision Glide | 305106 | |
| Antimycin A, 10 mM stock solution | Sigma | A8674 | Dissolve in DMSO, prepare ahead of time |
| Bam15, 10 mM stock solution | TimTec | ST056388 | Dissolve in DMSO, prepare ahead of time |
| Biopsy puncher, 1 mm | Integra Miltex | 33-31AA | |
| Cell-Tak | Corning Life Sciences | CB40240 | |
| CO2 asphyxiation chamber | |||
| Dissection forceps-Dumont #5 | Fine Science Tools | 11251-10 | Stright tip |
| Dissection forceps-Dumont #7 | Fine Science Tools | 11274-20 | Curved tip |
| Dissection microscope | |||
| DMSO | Sigma | D2438 | |
| Graefe forceps | Fine Science Tools | 11051-10 | Curved, Serrated tip |
| Microscissors | Fine Science Tools | 15004-08 | Curved tip |
| NaOH solution, 1 M | Sigma-Aldrich | S8263 | Aqueous solution, prepare ahead of time |
| Rotenone, 10 mM stock solution | Sigma | R8875 | Dissolve in DMSO, prepare ahead of time |
| Seahorse calibration medium | Agilent | 100840-000 | |
| Seahorse XF 1.0 M glucose | Agilent | 103577-100 | |
| Seahorse XF 100 mM pyruvate | Agilent | 103578-100 | |
| Seahorse XF 200 mM glutamine | Agilent | 103579-100 | |
| Seahorse XF DMEM medium | Agilent | 103575-100 | pH 7.4, with 5 mM HEPES |
| Seahorse XFe24 Islet Capture FluxPak | Agilent | 103518-100 | Containing Sensor Cartridge and Islet Capture microplate |
| Seahorse XFe24, Extra Cellular Flux Analyzer | Agilent | ||
| Sodium bicarbonate solution, 0.1 M | Sigma-Aldrich | S5761 | Aqueous solution, prepare ahead of time |
| Superfine eyelash brush | Ted Pella | 113 |
Referencias
- Nunnari, J., Suomalainen, A. Mitochondria: In sickness and in health. Cell. 148 (6), 1145-1159 (2012).
- Wong-Riley, M. T. Energy metabolism of the visual system. Eye Brain. 2, 99-116 (2010).
- Yu, D. Y., Cringle, S. J. Oxygen distribution and consumption within the retina in vascularised and avascular retinas and in animal models of retinal disease. Progress in Retina and Eye Research. 20, 175-208 (2001).
- Barot, M., Gokulgandhi, M. R., Mitra, A. K. Mitochondrial dysfunction in retinal diseases. Current Eye Research. 36 (12), 1069-1077 (2011).
- Joyal, J. S., Gantner, M. L., Smith, L. E. H. Retinal energy demands control vascular supply of the retina in development and disease: The role of neuronal lipid and glucose metabolism. Progress in Retina and Eye Research. 64, 131-156 (2018).
- Hurley, J. B., Lindsay, K. J., Du, J. Glucose, lactate, and shuttling of metabolites in vertebrate retinas. Journal of Neuroscience Research. 93 (7), 1079-1092 (2015).
- Haydinger, C. D., Kittipassorn, T., Peet, D. J. Power to see-Drivers of aerobic glycolysis in the mammalian retina: A review. Clinical and Experimental Ophthalmology. 48 (8), 1057-1071 (2020).
- Wright, A. F., et al. Lifespan and mitochondrial control of neurodegeneration. Nature Genetics. 36, 1153-1158 (2004).
- Bossy-Wetzel, E., Schwarzenbacher, R., Lipton, S. A. Molecular pathways to neurodegeneration. Nature Medicine. 10, 2-9 (2004).
- Leveillard, T., Philp, N. J., Sennlaub, F. Is retinal metabolic dysfunction at the center of the pathogenesis of age-related macular degeneration. International Journal of Molecular Sciences. 20 (3), (2019).
- Vlachantoni, D., et al. Evidence of severe mitochondrial oxidative stress and a protective effect of low oxygen in mouse models of inherited photoreceptor degeneration. Human Molecular Genetics. 20 (2), 322-335 (2011).
- Grenell, A., et al. Loss of MPC1 reprograms retinal metabolism to impair visual function. Proceedings of the National Academy of Science U. S. A. 116 (9), 3530-3535 (2019).
- Wright, A. F., Chakarova, C. F., Abd El-Aziz, M. M., Bhattacharya, S. S. Photoreceptor degeneration: genetic and mechanistic dissection of a complex trait. Nature Reviews in Genetics. 11 (4), 273-284 (2010).
- Jarrett, S. G., Boulton, M. E. Consequences of oxidative stress in age-related macular degeneration. Molecular Aspects of Medicine. 33 (4), 399-417 (2012).
- Rozing, M., et al. Age-related macular degeneration: A two-level model hypothesis. Progress in Retina Eye Research. 76, 100825 (2020).
- Yokosako, K., et al. Glycolysis in patients with age-related macular degeneration. Open Ophthalmology Journal. 8, 39-47 (2014).
- Bek, T. Mitochondrial dysfunction and diabetic retinopathy. Mitochondrion. 36, 4-6 (2017).
- Yumnamcha, T., Guerra, M., Singh, L. P., Ibrahim, A. S. Metabolic dysregulation and neurovascular dysfunction in diabetic retinopathy. Antioxidants. 9 (12), (2020).
- Futterman, S., Kinoshita, J. H. Metabolism of the retina. I. Respiration of cattle retina. Journal of Biological Chemistry. 234 (4), 723-726 (1959).
- Linsenmeier, R. A. Effects of light and darkness on oxygen distribution and consumption in the cat retina. Journal of General Physiology. 88 (4), 521-542 (1986).
- Medrano, C. J., Fox, D. A. Oxygen consumption in the rat outer and inner retina: light- and pharmacologically-induced inhibition. Experiments in Eye Research. 61 (3), 273-284 (1995).
- Kooragayala, K. Quantification of oxygen consumption in retina ex vivo demonstrates limited reserve capacity of photoreceptor mitochondria. Investigative Ophthalmology and Visual Science. 56 (13), 8428-8436 (2015).
- Adlakha, Y. K., Swaroop, A. Determination of mitochondrial oxygen consumption in the retina ex vivo: applications for retinal disease. Methods in Molecular Biology. 1753, 167-177 (2018).
- . Agilent Mitocondrial stress test user guide Available from: https://www.agilent.com/cs/library/usermanuals/public/XF_Cell_Mito_Stress_Test_Kit_User_Guide.pdf (2021)
- . Agilent Glycolytic rate assay user guide Available from: https://www.agilent.com/cs/library/usermanuals/public/103344-400.pdf (2021)
- . Agilent wave 2.6 user guide Available from: https://www.agilent.com/cs/library/usermanuals/public/103344-400.pdf (2021)
- . AVMA Guidelines for the Euthanasia of Animals Available from: https://www.avma.org/sites/default/files/2020-01/2020-Euthanasia-Final-1-17-20.pdf (2021)
- . Improving Quantification of Cellular Glycolytic Rate Using Agilent Seahorse XF Technology Available from: https://www.agilent.com/cs/library/whitepaper/public/whitepaper-improve-quantification-of-cellular-glycolytic-rate-cell-analysis-5991-7894en-agilent.pdf (2021)
- . Report Generator User Guide Agilent Seahorse XF Cell Mito Stress Test Available from: https://www.agilent.com/cs/library/usermanuals/public/Report_Generator_User_Guide_Seahorse_XF_Cell_Mito_Stress_Test_Single_File.pdf (2021)
- . Agilent Seahorse XF Buffer Factor Protocol Available from: https://www.agilent.com/cs/library/usermanuals/public/usermanual-xf-buffer-factor-protocol-cell-analysis-S7888-10010en-agilent.pdf (2021)
- . Agilent sensor cartridges and cell culture microplates Available from: https://www.agilent.com/cs/library/brochures/5991-8657EN_seahorse_plastics_brochure.pdf (2021)
- . Agilent Seahorse XF Glycolysis Stress Test Kit User Guide Available from: https://www.agilent.com/cs/library/usermanuals/public/XF_Glycolysis_Stress_Test_Kit_User_Guide.pdf (2021)
- Fan, Y. Y. A bioassay to measure energy metabolism in mouse colonic crypts, organoids, and sorted stem cells. American Journal of Physiology-Gastrointestinal and Liver Physiology. 309, 1-9 (2015).
- Huang, L., et al. Ductal pancreatic cancer modeling and drug screening using human pluripotent stem cell- and patient-derived tumor organoids. Nature Medicine. 21 (11), 1364-1371 (2015).
- Jeon, C. J., Strettoi, E., Masland, R. H. The major cell populations of the mouse retina. Journal of Neuroscience. 18 (21), 8936-8946 (1998).
- Akimoto, M., et al. Targeting of GFP to newborn rods by Nrl promoter and temporal expression profiling of flow-sorted photoreceptors. Proceedings of the National Academy of Science U. S. A. 103 (10), 3890-3895 (2006).
- Kenwood, B. M., et al. Identification of a novel mitochondrial uncoupler that does not depolarize the plasma membrane. Molecular Metabolism. 3 (2), 114-123 (2014).
- Corso-Diaz, X., et al. Genome-wide profiling identifies DNA methylation signatures of aging in rod photoreceptors associated with alterations in energy metabolism. Cell Reports. 31 (3), 107525 (2020).
- Berkowitz, B. A., et al. Mitochondrial respiration in outer retina contributes to light-evoked increase in hydration in vivo. Investigative Ophthalmology and Visual Science. 59 (15), 5957-5964 (2018).
- Joyal, J. S., et al. Retinal lipid and glucose metabolism dictates angiogenesis through the lipid sensor Ffar1. Nature Medicine. 22 (4), 439-445 (2016).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados