Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Détermination de la respiration mitochondriale et de la glycolyse dans des échantillons de tissu rétinien ex vivo
Dans cet article
Résumé
Décrit ici est un protocole détaillé pour effectuer un test de stress mitochondrial et un test de taux glycolytique dans des échantillons de tissu rétinien ex vivo à l’aide d’un bioanalyseur commercial.
Résumé
La respiration mitochondriale est une voie essentielle génératrice d’énergie dans toutes les cellules, en particulier les photorécepteurs rétiniens qui possèdent un métabolisme très actif. En outre, les photorécepteurs présentent également une glycolyse aérobie élevée comme les cellules cancéreuses. Des mesures précises de ces activités métaboliques peuvent fournir des informations précieuses sur l’homéostasie cellulaire dans des conditions physiologiques et dans des états pathologiques. Des tests à base de microplaques à haut débit ont été développés pour mesurer la respiration mitochondriale et diverses activités métaboliques dans les cellules vivantes. Cependant, une grande majorité d’entre eux sont développés pour des cellules cultivées et n’ont pas été optimisés pour des échantillons de tissus intacts et pour une application ex vivo. Décrit ici est un protocole détaillé étape par étape, utilisant la technologie de fluorescence à base de microplaques, pour mesurer directement le taux de consommation d’oxygène (OCR) comme indicateur de la respiration mitochondriale, ainsi que le taux d’acidification extracellulaire (ECAR) comme indicateur de glycolyse, dans le tissu rétinien ex vivo intact. Cette méthode a été utilisée pour évaluer avec succès les activités métaboliques dans la rétine de souris adulte et démontrer son application dans l’étude des mécanismes cellulaires du vieillissement et de la maladie.
Introduction
Les mitochondries sont des organites essentiels qui régulent le métabolisme cellulaire, la signalisation, l’homéostasie et l’apoptose en coordonnant plusieurs processus physiologiques cruciaux1. Les mitochondries servent de centrale électrique dans la cellule pour générer de l’adénosine triphosphate (ATP) par phosphorylation oxydative (OXPHOS) et fournir de l’énergie qui soutient presque tous les événements cellulaires. La majorité de l’oxygène cellulaire est métabolisée dans les mitochondries, où il sert d’accepteur final d’électrons dans la chaîne de transport d’électrons (ETC) pendant la respiration aérobie. De faibles quantités d’ATP peuvent également être produites à partir de la glycolyse dans le cytosol, où le glucose est converti en pyruvate, qui peut être converti en lactate ou être transporté dans les mitochondries et oxydé en acétyl-CoA, un substrat du cycle de l’acide tricarboxylique (cycle TCA).
La rétine est l’un des tissus les plus métaboliquement actifs chez les mammifères2, présentant des niveaux élevés de respiration mitochondriale et une consommation d’oxygène extrêmement élevée3. Les photorécepteurs des bâtonnets et des cônes contiennent une densité élevée de mitochondries4, et OXPHOS génère la plupart de l’ATP dans la rétine5. De plus, la rétine repose aussi fortement sur la glycolyse aérobie6,7 en convertissant le glucose en lactate5. Les défauts mitochondriaux sont associés à diverses maladies neurodégénératives8,9; et avec ses demandes énergétiques élevées uniques, la rétine est particulièrement vulnérable aux défauts métaboliques, y compris ceux affectant OXPHOS4 mitochondrial et la glycolyse10. Le dysfonctionnement mitochondrial et les défauts de glycolyse sont impliqués dans les maladies dégénératives rétiniennes11,12 et maculaires13, la dégénérescence maculaire liée à l’âge10,14,15,16 et la rétinopathie diabétique17,18. Par conséquent, des mesures précises de la respiration mitochondriale et de la glycolyse peuvent fournir des paramètres importants pour évaluer l’intégrité et la santé de la rétine.
La respiration mitochondriale peut être mesurée par la détermination du taux de consommation d’oxygène (OCR). Étant donné que la conversion du glucose en pyruvate et par la suite en lactate entraîne l’extrusion de protons et l’acidification de l’environnement extracellulaire, les mesures du taux d’acidification extracellulaire (ECAR) fournissent une indication du flux de glycolyse. Comme la rétine est composée de plusieurs types de cellules avec des relations intimes et une synergie active, y compris l’échange de substrats6, il est impératif d’analyser la fonction mitochondriale et le métabolisme dans le contexte de tissu rétinien entier avec une lamination et des circuits intacts. Au cours des dernières décennies, les électrodes O2 de type Clark et d’autres microélectrodes d’oxygène ont été utilisées pour mesurer la consommation d’oxygène dans la rétine19,20,21. Ces électrodes d’oxygène ont des limites majeures en termes de sensibilité, l’exigence d’un grand volume d’échantillon et la nécessité d’une agitation continue de l’échantillon en suspension, ce qui entraîne généralement la perturbation du contexte cellulaire et tissulaire. Le protocole décrit ici a été développé à l’aide d’une technique de fluorescence à base de microplaques pour mesurer le métabolisme énergétique mitochondrial dans le tissu rétinien ex vivo de souris fraîchement disséqué. Il permet des mesures en temps réel à mi-débit de l’OCR et de l’ECAR simultanément à l’aide d’un petit échantillon (poinçon de 1 mm) de tissu rétinien ex vivo tout en évitant le besoin de suspension et d’agitation continue.
Démontrée ici est la procédure expérimentale pour le test de stress mitochondrial et le test de taux glycolytique sur des disques de perforation rétiniens fraîchement disséqués. Ce protocole permet de mesurer les activités métaboliques liées aux mitochondries dans un contexte tissulaire ex vivo. Contrairement aux essais effectués à l’aide de cellules cultivées, les lectures obtenues ici reflètent le métabolisme énergétique combiné au niveau tissulaire et sont influencées par les interactions entre les différents types de cellules dans le tissu. Le protocole est modifié par rapport à une version précédemment publiée22,23 pour s’adapter à la nouvelle génération de l’analyseur de flux extracellulaire Agilent Seahorse 24-wells (XFe24) avec plaque Islet Capture. Le milieu d’essai, les concentrations de composés d’injection et le nombre/la durée des cycles d’essai ont également été optimisés pour le tissu rétinien. Un protocole détaillé étape par étape est donné pour la préparation des disques de poinçonnage rétiniens. Plus d’informations sur la configuration du programme et l’analyse des données peuvent être obtenues à partir du guide de l’utilisateur du fabricant24,25,26.
Protocole
Tous les protocoles de souris ont été approuvés par le Comité de soins et d’utilisation des animaux du National Eye Institute (NEI ASP# 650). Les souris ont été logées dans des conditions claires-sombres de 12 heures et soignées en suivant les recommandations du Guide pour les soins et l’utilisation des animaux de laboratoire, de l’Institut des ressources des animaux de laboratoire et de la Politique du service de santé publique sur les soins et l’utilisation sans cruauté des animaux de laboratoire.
1. Cartouche de capteur hydratante et préparation du milieu d’essai
- La veille de l’expérience, ajouter 1 mL du milieu d’étalonnage à chaque puits de la plaque utilitaire. Placez le couvercle Hydro-Booster sur le dessus et abaissez la cartouche du capteur par l’ouverture du couvercle. Vérifiez que le capteur est immergé dans le milieu d’étalonnage. Incuber la cartouche du capteur pendant la nuit dans un incubateur sans CO2 à 37 °C pour activer les fluorophores.
REMARQUE: Pour éviter l’évaporation, l’incubateur est humidifié en gardant un plateau d’eau à l’intérieur et la cassette de la cartouche du capteur est enveloppée d’une pellicule de plastique transparent. - Préparer le milieu d’essai en reconstituant le milieu DMEM seahorse avec l’ajout de glucose, de pyruvate et de glutamine aux concentrations souhaitées. Dans les essais rapportés dans cet article, la concentration finale des substrats dans le milieu d’essai est: 6 mM de glucose, 0,12 mM de pyruvate et 0,5 mM de glutamine. Pour chaque plaque d’essai, 40 mL du milieu d’essai sont préparés frais le jour de l’expérience.
- Configurez le programme de dosage dans l’analyseur en suivant les instructions du fabricant26. Dans le test démontré ici, le protocole est défini comme suit: 5 cycles de mesures pour la ligne de base, puis injecter le port A, suivi de 4 cycles de mesures, puis injecter le port B et suivi de 4 cycles de mesures. Chaque cycle est composé de mélanger (3 min), d’attendre (2 min) et de mesurer (3 min).
2. Inserts de maille de revêtement de microplaque de capture d’îlots
- Préparer le mélange d’enrobage en combinant 20 μL du milieu de fixation cellulaire (p. ex., Cell-Tak) avec 171 μL de bicarbonate de sodium de 0,1 M et 9 μL de 1 M de NaOH.
- Ouvrez le couvercle de la cassette contenant des inserts en maille. Pipette 8 μL du mélange de revêtement à chaque insert de maille. Utilisez une pointe de pipette pour étaler doucement la gouttelette afin de répartir le mélange de revêtement de manière égale dans tout l’insert en maille.
- Fermez la cassette et laissez les inserts en maille incuber à température ambiante pendant au moins 25 minutes pour l’adsorption.
- Lavez l’insert en maille en pipetant 4 mL du milieu d’essai directement sur les inserts en maille. Secouez doucement la cassette pour vous assurer que tous les inserts en maille sont lavés avec le support d’essai.
- Gardez l’insert de maille de côté. Il est prêt à l’emploi.
3. Préparation des composés d’injection
- Sortir les aliquotes de Bam15 (10 mM), roténone (10 mM), antimycine A (10 mM) et 2-DG (500 mM) du congélateur à -80 °C et décongeler à température ambiante.
REMARQUE: Le stock 2-DG est prêt à l’emploi. Les autres médicaments doivent être dilués dans le stock de travail. - Réchauffer 10 mL du milieu d’essai dans un bain-marie à 37 °C.
- Diluer 10 mM de stock Bam15 à 50 μM de stock de travail à l’aide d’une procédure de dilution en deux étapes : mélanger 20 μL de stock de 10 mM avec 20 μL de DMSO pour obtenir un stock intermédiaire de 5 mM. Mélanger ensuite 10 μL du stock intermédiaire supérieur à 5 mM avec 990 μL de milieu d’essai préchauffé pour obtenir le stock de travail final de 50 μM.
- Diluer et combiner 10 mM de roténone et 10 mM d’antimycine A sur un stock de 10 μM de roténone/antimycine A (Rot/AA) par deux étapes de dilutions : mélanger 10 μL chacun de 10 mM de roténone et 10 mM de stock d’antimycine A avec 80 μL de DMSO pour obtenir 1 mM de stock intermédiaire rot/aA. Mélangez ensuite 10 μL du stock intermédiaire supérieur à 1 mM avec 990 μL de milieu d’essai préchauffé pour obtenir le stock final de 10 μM de rotule/AA.
- Préparez fraîchement les stocks de travail de composés d’injection mentionnés ci-dessus le jour de l’expérience et mettez-les de côté à température ambiante jusqu’à ce qu’ils soient chargés dans les orifices d’injection de la cartouche du capteur.
4. Dissection rétinienne et préparation du poinçon rétinien
- Euthanasier une souris par asphyxie au CO2 en suivant les lignes directrices de l’AVMA sur l’euthanasie27.
REMARQUE: Ne laissez pas l’animal dans une chambre à CO2 plus longtemps que le temps nécessaire pour l’euthanasie. - Énucléez les yeux et placez-les dans un tampon PBS 1x vieux de glace dans une boîte de Pétri, puis placez-le sous un microscope à dissection.
- Retirez soigneusement, en coupant avec des microcisseurs, les muscles droits supplémentaires attachés à l’extérieur du globe oculaire et coupez le nerf optique.
- Utilisez une aiguille de 30 G pour percer un trou au bord de la cornée (limbe); cela sert de site d’insertion pour les microscisseurs. Ensuite, utilisez une fine microscissive de dissection pour faire une coupe circulaire le long du bord de la cornée, en la séparant de la coupe oculaire postérieure.
- Utilisez des pinces à dissection pointues pour retirer la cornée, le cristallin et l’humeur vitrée de la coupe oculaire.
- Utilisez des microcissives à dissection fine pour faire plusieurs petites coupures sur la couche sclérale au bord de la coupe oculaire. Évitez de couper la couche de rétine. Utilisez deux pinces à dissection pointues pour maintenir le tissu scléral de chaque côté de la coupe et tirez très soigneusement sur la couche sclérale pour l’enlever de la rétine neurale. Répétez ceci autour de la coupe oculaire jusqu’à ce que toute la sclérotique soit enlevée et qu’une coupe rétinienne intacte soit obtenue.
- Utilisez des microcisseurs de dissection et faites des coupes radiales sur la coupe rétinienne pour l’aplatir et générer plusieurs sections distinctes.
REMARQUE: Selon les compétences de dissection de la personne et son expérience dans la manipulation de tissu rétinien frais, la coupe rétinienne peut être coupée pour générer 3 à 5 sections distinctes. - Utilisez un poinçonneur de biopsie de 1 mm de diamètre pour couper un disque rétinien de chaque section de la coupe rétinienne aplatie.
REMARQUE: Il faut prendre soin d’obtenir les disques rétiniens perforés à égale distance de la tête du nerf optique. - Utilisez des pinces pour transférer les inserts en maille pré-enduits dans la boîte de Pétri disséquante. À l’aide de deux brosses à cils superfines, placez le disque de poinçon rétinien sur l’insert en maille. Le disque de poinçonnage rétinien est placé au centre de l’insert de maille avec la couche de cellules ganglionnaires vers le bas touchant la maille et la couche de photorécepteurs vers le haut.
REMARQUE: Fréquemment, certaines cellules RPE restent attachées aux photorécepteurs, et la pigmentation de ces cellules peut être utilisée comme indicateur de l’orientation du disque de poinçon rétinien.
5. Chargement des orifices d’injection de la cartouche du capteur et étalonnage
- Sortez la cassette de la plaque de cartouche de capteur hydratée de l’incubateur à 37 °C. Retirez le couvercle de l’Hydro-Booster et replacez la cartouche du capteur sur la plaque utilitaire.
- Chargez le volume souhaité de solutions de base de travail de composés d’injection dans des orifices appropriés. Maintenez l’embout de la pipette à un angle de 45 °. Insérez l’embout de la pipette à mi-chemin dans un orifice d’injection avec le biseau de l’embout contre la paroi opposée du port d’injection et chargez doucement le composé dans chaque orifice. Évitez d’introduire des bulles d’air.
- Reportez-vous au guide de l’utilisateur de l’instrument pour connaître le volume du composé chargé dans chaque orifice d’injection pour un dosage spécifique. Dans les expériences présentées dans cet article, 68 μL de 50 μM de stock de travail Bam15 (pour le test de stress mitochondrial) ou 68 μL de stock de travail Rot/AA de 10 μM (pour le dosage du taux glycolytique) sont chargés dans le port A; 75 μL de 10 μM de stock de travail Rot/AA (pour le dosage du stress mitochondrial) ou 75 μL de 500 mM de stock de travail 2-DG (pour le dosage du taux glycolytique) sont chargés dans le port B.
- Ports d’injection de charge de tous les puits de la plaque, y compris les puits de correction de fond et les puits à blanc pour assurer une injection correcte. Chargez la solution composée respective dans chaque port pour les puits de correction de fond. Le milieu d’essai peut être substitué, au lieu de la solution composée, dans chacun des orifices des puits de la rive.
- Placez la plaque de cartouche de capteur chargée, avec le couvercle fermé, dans l’analyseur pour lancer l’étalonnage avant l’exécution du test. Une fois l’étalonnage terminé, le programme s’interrompt automatiquement, attendant le remplacement de la plaque utilitaire par la plaque de capture d’îlot contenant des poinçons rétiniens.
6. Chargement de la plaque de capture des îlots et démarrage de l’essai
- Ajouter 607 μL du milieu d’essai à chaque puits de la plaque de capture des îlots
- Utilisez des pinces pour saisir le bord de l’insert en maille contenant des disques de poinçonnage rétiniens sur le dessus et le sortir de la boîte de Pétri. Tapotez légèrement le fond de l’insert en maille sur un tissu absorbant pour éliminer le liquide supplémentaire et le mettre dans le puits de la plaque de capture des îlots. Répétez cette étape jusqu’à ce que tous les inserts de maille avec des poinçons rétiniens soient placés dans la plaque de capture des îlots. Remplissez les puits de correction d’arrière-plan et les puits vierges avec des inserts de maille vides.
- Utilisez deux pinces Graefe pour appuyer soigneusement et doucement sur le bord de chaque insert en maille et assurez-vous qu’elles sont bien insérées au bas de la plaque de capture des îlots.
- Placez la plaque de capture des îlots chargés dans un incubateur à 37 °C pendant 5 minutes pour la réchauffer.
- Éjectez la plaque utilitaire une fois l’étalonnage terminé et remplacez-la par une et remplacez-la par la plaque de capture des îlots, avec couvercle fermé, contenant des poinçons rétiniens.
- Reprenez l’exécution du test.
7. Exécuter la résiliation et le stockage des données
- Une fois l’exécution terminée, éjectez la cartouche du capteur et la plaque de capture de l’îlot contenant des poinçons rétiniens. Les données sont automatiquement enregistrées en tant que fichier .asyr.
- Utilisez le logiciel d’analyse de données associé pour afficher et analyser les données en suivant le guide de l’utilisateur du fabricant26.
- Utilisez la fonction Exporter pour exporter le fichier .xslx des données, qui peut être visualisé et analysé à l’aide d’un tableur.
8. Enregistrement de l’échantillon de poinçon rétinien
- Après le test, retirez la plaque de la machine, retirez la cartouche du capteur et retirez doucement le milieu d’essai de chaque puits à l’aide d’une pipette.
- Remettez le couvercle et scellez les côtés de la plaque avec la bande de parafilm.
- Conserver à -80 °C.
- Pour la normalisation, quantifiez la teneur totale en ADN ou en protéines du poinçon dans chaque puits.
9. Analyse des données
- Test de stress mitochondrial
REMARQUE: La valeur OCR mesurée (totalOCR) représente la consommation totale d’oxygène par le tissu. Après l’injection de Bam15 (découpleur), l’OCR augmente du niveau basal (totalOCRbasal) au niveau maximal (totalOCRmax) et diminue après l’injection de Rot/AA. La valeur résiduelle de l’OCR après injection de Rot/AA (totalOCRRot/AA) représente la consommation d’oxygène non mitochondriale.- Calculer la consommation d’oxygène liée aux mitochondries comme suit:
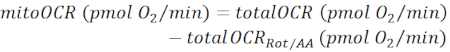 (Eq. 1) 28 ans
(Eq. 1) 28 ans
- Calculer la capacité de réserve mitochondriale (MRC) comme suit :
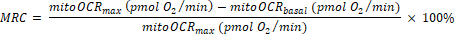 (Eq. 2) 29
(Eq. 2) 29
REMARQUE: La dernière lecture parmi les 5 mesures avant l’injection de Bam15 est prise comme valeur « basale » (pour totalOCRbasal et mitoOCRbasal). La lecture la plus élevée parmi les 4 mesures suivant l’injection de Bam15 est utilisée comme valeur « max » (pour totalOCRmax et mitoOCRmax). La lecture la plus basse parmi les 4 mesures suivant l’injection de Rot/AA est utilisée comme totalOCRRot/AA.
- Calculer la consommation d’oxygène liée aux mitochondries comme suit:
- Dosage du taux glycolytique
NOTE: La valeur ECAR mesurée (totalECAR) représente l’acidification totale du milieu par l’activité métabolique du tissu. En général, l’acidification du micro-environnement extracellulaire résulte principalement de l’extrusion du produit glycolytique, le lactate. Le catabolisme des substrats dans le cycle mitochondrial du TCA entraîne la production de CO2, qui acidifie également le milieu extracellulaire par l’hydratation en bicarbonate.- La sous-race mitochondriale a contribué à l’acidification moyenne (mitoECAR) de totalECAR pour obtenir le glycoECAR.
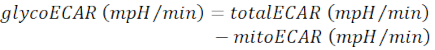 (Eq. 3) 28 ans
(Eq. 3) 28 ans
REMARQUE: La respiration mitochondriale et le cycle TCA sont des processus fortement couplés. La production de CO2 à partir des mitochondries est fonction du taux d’OXPHOS, qui est mesurable par mitoOCR. - Calculez le mitoECAR comme suit :
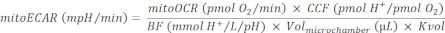 (Eq. 4) 28 ans
(Eq. 4) 28 ans
où, le CCF (CO2 Contribution Factor) est une valeur de ratio calculée empiriquement, représentant la quantité de contribution H+ de l’acidification médiée par le CO2 par rapport à chaque consommation d’O2 d’OXPHOS. Le CCF pour ce système est prédéterminé à 0,6028. La mesure précise de l’acidification du milieu est déterminée par la capacité tampon du milieu, la sensibilité du capteur de pH de l’instrument et la capacité effective de la chambre de mesure. Ici, le BF (Buffer Factor) est un paramètre de la capacité tampon expérimentale in situ, représentant la quantité de H+ ou OH- ajoutée à la chambre de mesure efficace pour modifier le niveau de pH de 1 unité. Lorsque le milieu d’essai personnalisé est utilisé, le BF peut être déterminé en titrant les quantités connues d’acide dans le milieu d’essai en suivant le protocole Buffer Factor30. Le milieu DMEM hippocampe pH 7,4 utilisé dans ce protocole a un BF prédéterminé de 2,60 mmol H+/L/pH. La plaque de capture d’îlots utilisée dans ce protocole a une Volmicrochambre = 16,6 μL31. Le facteur d’échelle de volume, Kvol, est une constante déterminée empiriquement. La valeur de Kvol n’est pas disponible pour la plaque de capture des îlots, mais peut être calculée à partir de la valeur de la microplaque28, en tenant compte de la différence de volume dans leurs microchambres, à 0,41.
REMARQUE: L’injection de la rotule / AA arrête la respiration mitochondriale et force le tissu à passer à la glycolyse pour la production d’ATP, ce qui entraîne une extrusion de lactate plus élevée et une augmentation de la mesure ECAR. La glycolyse est arrêtée par injection de 2-DG et la mesure ECAR résiduelle révèle une acidification non glycolytique et non mitochondriale du milieu. - Calculer la capacité de réserve glycolytique (GRC) comme suit :
 (Eq. 5) 32
(Eq. 5) 32
où, la dernière lecture parmi les 5 mesures avant l’injection de Rot/AA est prise comme valeur « basale » (glycoECARbasal). La lecture la plus élevée parmi les 4 mesures après l’injection de Rot/AA est utilisée comme valeur « max » (glycoECARmax). La lecture la plus basse parmi les 4 mesures après l’injection de 2-DG est utilisée comme glycoECAR2-DG.
- La sous-race mitochondriale a contribué à l’acidification moyenne (mitoECAR) de totalECAR pour obtenir le glycoECAR.
- Normalisation
REMARQUE: La normalisation est essentielle lors de la comparaison des lectures des tissus rétiniens de différents groupes d’âge ou entre des échantillons de type sauvage et pathologiques / dégénératifs, qui peuvent différer dans le nombre de cellules.- Utilisez des kits disponibles en commerce pour évaluer la teneur en ADN de chaque disque de poinçonnage rétinien33,34.
- Alternativement, utilisez un tampon de dosage de radioimmunoprécipitation (tampon RIPA) pour extraire la protéine totale du poinçon rétinien et utilisez la teneur en protéines pour la normalisation.
REMARQUE: La surface d’une rétine de souris adulte a été préalablement déterminée à environ 20 mm2, et chaque rétine contient environ 6,5 millions de cellules35. Par conséquent, chaque poinçon rétinien de 1 mm de diamètre est ~ 1/25 d’une seule rétine et contient ~ 260K cellules. On peut se référer à ces chiffres en comparant les données d’un poinçon rétinien à celles d’autres échantillons de tissus ou de cellules cultivées,
Résultats
Les données rapportées ici sont un test de stress mitochondrial représentatif montrant une trace OCR (Figure 1) et un test de taux glycolytique montrant une trace OCR et une trace ECAR (Figure 2), qui ont été effectués à l’aide de disques de perforation rétiniens de 1 mm fraîchement disséqués de souris Nrl-L-EGFP transgéniques de 4 mois36 (fond C57B / L6). Ces souris expriment la GFP spécifiquement dans les photor?...
Discussion
Vous trouverez ici des instructions détaillées pour effectuer des tests sur microplaques de la respiration mitochondriale et de l’activité de glycolyse à l’aide de disques de perforation rétiniens ex vivo fraîchement disséqués. Le protocole a été optimisé pour : 1) assurer l’utilisation d’un milieu d’essai approprié pour le tissu rétinien ex vivo; 2) utiliser la taille appropriée des disques de poinçonnage rétiniens pour obtenir des lectures OCR et ECAR qui se situent dans la p...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail est soutenu par le programme de recherche intra-muros de l’Institut national de l’œil (ZIAEY000450 et ZIAEY000546).
matériels
| Name | Company | Catalog Number | Comments |
| 1X PBS | Thermo Fisher | 14190-144 | |
| 2-Deoxy glucose (2-DG), 500 mM stock solution | Sigma | D6134 | Dissolve in Seahorse XF DMEM medium, prepare ahead of time |
| 30-gauge needle | BD Precision Glide | 305106 | |
| Antimycin A, 10 mM stock solution | Sigma | A8674 | Dissolve in DMSO, prepare ahead of time |
| Bam15, 10 mM stock solution | TimTec | ST056388 | Dissolve in DMSO, prepare ahead of time |
| Biopsy puncher, 1 mm | Integra Miltex | 33-31AA | |
| Cell-Tak | Corning Life Sciences | CB40240 | |
| CO2 asphyxiation chamber | |||
| Dissection forceps-Dumont #5 | Fine Science Tools | 11251-10 | Stright tip |
| Dissection forceps-Dumont #7 | Fine Science Tools | 11274-20 | Curved tip |
| Dissection microscope | |||
| DMSO | Sigma | D2438 | |
| Graefe forceps | Fine Science Tools | 11051-10 | Curved, Serrated tip |
| Microscissors | Fine Science Tools | 15004-08 | Curved tip |
| NaOH solution, 1 M | Sigma-Aldrich | S8263 | Aqueous solution, prepare ahead of time |
| Rotenone, 10 mM stock solution | Sigma | R8875 | Dissolve in DMSO, prepare ahead of time |
| Seahorse calibration medium | Agilent | 100840-000 | |
| Seahorse XF 1.0 M glucose | Agilent | 103577-100 | |
| Seahorse XF 100 mM pyruvate | Agilent | 103578-100 | |
| Seahorse XF 200 mM glutamine | Agilent | 103579-100 | |
| Seahorse XF DMEM medium | Agilent | 103575-100 | pH 7.4, with 5 mM HEPES |
| Seahorse XFe24 Islet Capture FluxPak | Agilent | 103518-100 | Containing Sensor Cartridge and Islet Capture microplate |
| Seahorse XFe24, Extra Cellular Flux Analyzer | Agilent | ||
| Sodium bicarbonate solution, 0.1 M | Sigma-Aldrich | S5761 | Aqueous solution, prepare ahead of time |
| Superfine eyelash brush | Ted Pella | 113 |
Références
- Nunnari, J., Suomalainen, A. Mitochondria: In sickness and in health. Cell. 148 (6), 1145-1159 (2012).
- Wong-Riley, M. T. Energy metabolism of the visual system. Eye Brain. 2, 99-116 (2010).
- Yu, D. Y., Cringle, S. J. Oxygen distribution and consumption within the retina in vascularised and avascular retinas and in animal models of retinal disease. Progress in Retina and Eye Research. 20, 175-208 (2001).
- Barot, M., Gokulgandhi, M. R., Mitra, A. K. Mitochondrial dysfunction in retinal diseases. Current Eye Research. 36 (12), 1069-1077 (2011).
- Joyal, J. S., Gantner, M. L., Smith, L. E. H. Retinal energy demands control vascular supply of the retina in development and disease: The role of neuronal lipid and glucose metabolism. Progress in Retina and Eye Research. 64, 131-156 (2018).
- Hurley, J. B., Lindsay, K. J., Du, J. Glucose, lactate, and shuttling of metabolites in vertebrate retinas. Journal of Neuroscience Research. 93 (7), 1079-1092 (2015).
- Haydinger, C. D., Kittipassorn, T., Peet, D. J. Power to see-Drivers of aerobic glycolysis in the mammalian retina: A review. Clinical and Experimental Ophthalmology. 48 (8), 1057-1071 (2020).
- Wright, A. F., et al. Lifespan and mitochondrial control of neurodegeneration. Nature Genetics. 36, 1153-1158 (2004).
- Bossy-Wetzel, E., Schwarzenbacher, R., Lipton, S. A. Molecular pathways to neurodegeneration. Nature Medicine. 10, 2-9 (2004).
- Leveillard, T., Philp, N. J., Sennlaub, F. Is retinal metabolic dysfunction at the center of the pathogenesis of age-related macular degeneration. International Journal of Molecular Sciences. 20 (3), (2019).
- Vlachantoni, D., et al. Evidence of severe mitochondrial oxidative stress and a protective effect of low oxygen in mouse models of inherited photoreceptor degeneration. Human Molecular Genetics. 20 (2), 322-335 (2011).
- Grenell, A., et al. Loss of MPC1 reprograms retinal metabolism to impair visual function. Proceedings of the National Academy of Science U. S. A. 116 (9), 3530-3535 (2019).
- Wright, A. F., Chakarova, C. F., Abd El-Aziz, M. M., Bhattacharya, S. S. Photoreceptor degeneration: genetic and mechanistic dissection of a complex trait. Nature Reviews in Genetics. 11 (4), 273-284 (2010).
- Jarrett, S. G., Boulton, M. E. Consequences of oxidative stress in age-related macular degeneration. Molecular Aspects of Medicine. 33 (4), 399-417 (2012).
- Rozing, M., et al. Age-related macular degeneration: A two-level model hypothesis. Progress in Retina Eye Research. 76, 100825 (2020).
- Yokosako, K., et al. Glycolysis in patients with age-related macular degeneration. Open Ophthalmology Journal. 8, 39-47 (2014).
- Bek, T. Mitochondrial dysfunction and diabetic retinopathy. Mitochondrion. 36, 4-6 (2017).
- Yumnamcha, T., Guerra, M., Singh, L. P., Ibrahim, A. S. Metabolic dysregulation and neurovascular dysfunction in diabetic retinopathy. Antioxidants. 9 (12), (2020).
- Futterman, S., Kinoshita, J. H. Metabolism of the retina. I. Respiration of cattle retina. Journal of Biological Chemistry. 234 (4), 723-726 (1959).
- Linsenmeier, R. A. Effects of light and darkness on oxygen distribution and consumption in the cat retina. Journal of General Physiology. 88 (4), 521-542 (1986).
- Medrano, C. J., Fox, D. A. Oxygen consumption in the rat outer and inner retina: light- and pharmacologically-induced inhibition. Experiments in Eye Research. 61 (3), 273-284 (1995).
- Kooragayala, K. Quantification of oxygen consumption in retina ex vivo demonstrates limited reserve capacity of photoreceptor mitochondria. Investigative Ophthalmology and Visual Science. 56 (13), 8428-8436 (2015).
- Adlakha, Y. K., Swaroop, A. Determination of mitochondrial oxygen consumption in the retina ex vivo: applications for retinal disease. Methods in Molecular Biology. 1753, 167-177 (2018).
- . Agilent Mitocondrial stress test user guide Available from: https://www.agilent.com/cs/library/usermanuals/public/XF_Cell_Mito_Stress_Test_Kit_User_Guide.pdf (2021)
- . Agilent Glycolytic rate assay user guide Available from: https://www.agilent.com/cs/library/usermanuals/public/103344-400.pdf (2021)
- . Agilent wave 2.6 user guide Available from: https://www.agilent.com/cs/library/usermanuals/public/103344-400.pdf (2021)
- . AVMA Guidelines for the Euthanasia of Animals Available from: https://www.avma.org/sites/default/files/2020-01/2020-Euthanasia-Final-1-17-20.pdf (2021)
- . Improving Quantification of Cellular Glycolytic Rate Using Agilent Seahorse XF Technology Available from: https://www.agilent.com/cs/library/whitepaper/public/whitepaper-improve-quantification-of-cellular-glycolytic-rate-cell-analysis-5991-7894en-agilent.pdf (2021)
- . Report Generator User Guide Agilent Seahorse XF Cell Mito Stress Test Available from: https://www.agilent.com/cs/library/usermanuals/public/Report_Generator_User_Guide_Seahorse_XF_Cell_Mito_Stress_Test_Single_File.pdf (2021)
- . Agilent Seahorse XF Buffer Factor Protocol Available from: https://www.agilent.com/cs/library/usermanuals/public/usermanual-xf-buffer-factor-protocol-cell-analysis-S7888-10010en-agilent.pdf (2021)
- . Agilent sensor cartridges and cell culture microplates Available from: https://www.agilent.com/cs/library/brochures/5991-8657EN_seahorse_plastics_brochure.pdf (2021)
- . Agilent Seahorse XF Glycolysis Stress Test Kit User Guide Available from: https://www.agilent.com/cs/library/usermanuals/public/XF_Glycolysis_Stress_Test_Kit_User_Guide.pdf (2021)
- Fan, Y. Y. A bioassay to measure energy metabolism in mouse colonic crypts, organoids, and sorted stem cells. American Journal of Physiology-Gastrointestinal and Liver Physiology. 309, 1-9 (2015).
- Huang, L., et al. Ductal pancreatic cancer modeling and drug screening using human pluripotent stem cell- and patient-derived tumor organoids. Nature Medicine. 21 (11), 1364-1371 (2015).
- Jeon, C. J., Strettoi, E., Masland, R. H. The major cell populations of the mouse retina. Journal of Neuroscience. 18 (21), 8936-8946 (1998).
- Akimoto, M., et al. Targeting of GFP to newborn rods by Nrl promoter and temporal expression profiling of flow-sorted photoreceptors. Proceedings of the National Academy of Science U. S. A. 103 (10), 3890-3895 (2006).
- Kenwood, B. M., et al. Identification of a novel mitochondrial uncoupler that does not depolarize the plasma membrane. Molecular Metabolism. 3 (2), 114-123 (2014).
- Corso-Diaz, X., et al. Genome-wide profiling identifies DNA methylation signatures of aging in rod photoreceptors associated with alterations in energy metabolism. Cell Reports. 31 (3), 107525 (2020).
- Berkowitz, B. A., et al. Mitochondrial respiration in outer retina contributes to light-evoked increase in hydration in vivo. Investigative Ophthalmology and Visual Science. 59 (15), 5957-5964 (2018).
- Joyal, J. S., et al. Retinal lipid and glucose metabolism dictates angiogenesis through the lipid sensor Ffar1. Nature Medicine. 22 (4), 439-445 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon