É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Determinação da respiração mitocondrial e glicolise em amostras de tecido retinal ex vivo
Neste Artigo
Resumo
Descrito aqui é um protocolo detalhado para a realização de ensaios de estresse mitocondrial e ensaio de taxa glicólítica em amostras de tecido de retina ex vivo usando um bioanalzer comercial.
Resumo
A respiração mitocondrial é um caminho crítico gerador de energia em todas as células, especialmente fotorreceptores de retina que possuem um metabolismo altamente ativo. Além disso, fotorreceptores também exibem alta glicólise aeróbica como células cancerosas. Medições precisas dessas atividades metabólicas podem fornecer informações valiosas sobre a homeostase celular em condições fisiológicas e em estados de doenças. Ensaios baseados em microplacos de alto rendimento foram desenvolvidos para medir a respiração mitocondrial e várias atividades metabólicas em células vivas. No entanto, a grande maioria delas são desenvolvidas para células cultivadas e não foram otimizadas para amostras de tecido intacto e para aplicação ex vivo. Descrito aqui é um protocolo passo-a-passo detalhado, utilizando a tecnologia de fluorescência baseada em microplaca, para medir diretamente a taxa de consumo de oxigênio (OCR) como um indicador de respiração mitocondrial, bem como taxa de acidificação extracelular (ECAR) como um indicador de glicolise, em tecido de retina ex vivo intacto. Este método tem sido usado para avaliar com sucesso as atividades metabólicas na retina do camundongo adulto e demonstrar sua aplicação na investigação de mecanismos celulares de envelhecimento e doença.
Introdução
Mitocôndrias são organelas essenciais que regulam o metabolismo celular, sinalização, homeostase e apoptose coordenando múltiplos processos fisiológicos cruciais1. Mitocôndrias servem como a potência na célula para gerar triphosfato de adenosina (ATP) através da fosforilação oxidativa (OXPHOS) e fornecem energia que suporta quase todos os eventos celulares. A maioria do oxigênio celular é metabolizada em mitocôndrias, onde serve como o aceitador final de elétrons na cadeia de transporte de elétrons (ETC) durante a respiração aeróbica. Baixas quantidades de ATP também podem ser produzidas a partir da glicolise no citosol, onde a glicose é convertida em piruvato, que pode ser ainda convertida em lactato ou ser transportada para mitocôndrias e oxidada para acetil-CoA, um substrato no ciclo de ácido tricarboxílico (ciclo TCA).
A retina é um dos tecidos mais metabolicamente ativos em mamíferos2, apresentando altos níveis de respiração mitocondrial e consumo de oxigênio extremamente alto3. Os fotorreceptores da haste e do cone contêm uma alta densidade de mitocôndrias4, e oxphos gera a maioria de ATP na retina5. Além disso, a retina também depende fortemente da glicólise aeróbica6,7, convertendo glicose em lactato5. Os defeitos mitocondriais estão associados a diversas doenças neurodegenerativas8,9; e com suas demandas únicas de alta energia, a retina é especialmente vulnerável a defeitos metabólicos, incluindo aqueles que afetam oxphos4 mitocondrial4 e glicolise10. A disfunção mitocondrial e os defeitos na glicólise estão implicados em doenças degenerativas da retina 11,12 e macular13, degeneração macular relacionada à idade10,14,15,16 e retinopatia diabética17,18. Portanto, medições precisas da respiração mitocondrial e da glicolise podem fornecer parâmetros importantes para avaliar a integridade e a saúde da retina.
A respiração mitocondrial pode ser medida através da determinação da taxa de consumo de oxigênio (OCR). Dado que a conversão da glicose em piruvato e, posteriormente, para lactato resulta em extrusão de prótons em e acidificação do ambiente extracelular, as medidas da taxa de acidificação extracelular (ECAR) fornecem uma indicação de fluxo de glicolise. Como a retina é composta de múltiplos tipos de células com relações íntimas e sinergia ativa, incluindo a troca de substratos6, é imperativo analisar a função mitocondrial e o metabolismo no contexto de todo o tecido retinente com laminação intacta e circuitos. Nas últimas décadas, os eletrodos O2 tipo Clark e outros microeletrodos de oxigênio têm sido usados para medir o consumo de oxigênio na retina19,20,21. Esses eletrodos de oxigênio têm grandes limitações na sensibilidade, exigência de um grande volume amostral e a necessidade de agitação contínua da amostra suspensa, o que geralmente leva à interrupção do contexto celular e tecidual. O protocolo descrito aqui foi desenvolvido usando uma técnica de fluorescência à base de microplaca para medir o metabolismo de energia mitocondrial em tecido de retina de camundongos ex vivo recém-dissecado. Permite medições em tempo real de média entrada tanto do OCR quanto do ECAR simultaneamente usando uma pequena amostra (soco de 1 mm) de tecido ex vivo retina, evitando a necessidade de suspensão e agitação contínua.
Demonstrado aqui é o procedimento experimental para ensaio de estresse mitocondrial e ensaio de taxa glicolítico em discos de perfuração de retina recém-dissecados. Este protocolo permite a medição de atividades metabólicas relacionadas a mitocôndrias em um contexto de tecido ex vivo. Diferente dos ensaios realizados com células cultivadas, as leituras obtidas aqui refletem o metabolismo energético combinado no nível do tecido e são influenciadas por interações entre os diferentes tipos celulares dentro do tecido. O protocolo é modificado a partir de uma versão anteriormente publicada22,23 para se adaptar à nova geração do analisador Agilent Seahorse extracelular fluxo 24-wells (XFe24) com placa de Captura de Ilhéus. O meio de ensaio, as concentrações de compostos de injeção e o número/duração dos ciclos de ensaio também foram otimizados para o tecido retiniano. Um protocolo passo a passo detalhado é dado para a preparação de discos de perfuração de retina. Mais informações sobre a configuração do programa e análise de dados podem ser obtidas no guia de usuário do fabricante24,25,26.
Protocolo
Todos os protocolos de camundongos foram aprovados pelo Comitê de Cuidado e Uso de Animais do Instituto Nacional de Olhos (NEI ASP nº 650). Os camundongos foram alojados em condições claras e escuras 12h e atendidos seguindo as recomendações do Guia de Cuidado e Uso de Animais de Laboratório, do Instituto de Recursos Animais Laboratoriais e da Política de Serviços Públicos de Saúde sobre Cuidados Humanos e Uso de Animais de Laboratório.
1. Cartucho de sensor hidratante e preparação do meio de ensaio
- No dia anterior ao experimento, adicione 1 mL do meio de calibração a cada poço da placa de utilidade. Coloque a tampa Hydro-Booster na parte superior e baixe o cartucho do sensor através da abertura na tampa. Verifique se o sensor está submerso no meio de calibração. Incubar o cartucho do sensor durante a noite em uma incubadora livre de CO2 a 37 °C para ativar os fluoroforos.
NOTA: Para evitar a evaporação, a incubadora é umidificada mantendo uma bandeja de água dentro, e o do cartucho do sensor é embrulhado com plástico transparente. - Prepare o meio de ensaio reconstituindo o meio DMEM do cavalo marinho com a adição de glicose, piruvato e glutamina às concentrações desejadas. Nos ensaios relatados neste artigo, a concentração final de substratos no meio de ensaio são: 6 mM de glicose, 0,12 mM de piruvato e 0,5 mM de glutamina. Para cada placa de ensaio, 40 mL do meio de ensaio é preparado fresco no dia do experimento.
- Configure o programa de ensaio no analisador seguindo as instruções do fabricante26. No ensaio aqui demonstrado, o protocolo é definido da seguinte forma: 5 ciclos de medições para a linha de base, depois injetar a porta A, seguido por 4 ciclos de medições, depois injetar a porta B e seguido por 4 ciclos de medições. Cada ciclo é composto de mistura (3 min), espera (2 min) e medida (3 min).
2. Revestimento de pastilhas de malha de microplatos de captura de ilhotas
- Prepare a mistura de revestimento combinando 20 μL do meio de fixação celular (por exemplo, Cell-Tak) com 171 μL de 0,1 M de bicarbonato de sódio e 9 μL de 1 M NaOH.
- Abra a tampa do contendo pastilhas de malha. Pipeta 8 μL da mistura de revestimento para cada inserção de malha. Use uma ponta de pipeta para esfregar suavemente/espalhar a gota ao redor para distribuir a mistura de revestimento igualmente ao longo da inserção da malha.
- Feche o e deixe as pastilhas de malha incubarem à temperatura ambiente por pelo menos 25 minutos para adsorção.
- Lave a inserção da malha ao tubo de 4 mL do meio de ensaio diretamente nas pastilhas de malha. Agite suavemente o para garantir que todas as pastilhas de malha sejam lavadas com o meio de ensaio.
- Mantenha a pastilha de malha de lado. Está pronto para usar.
3. Preparação de compostos de injeção
- Retire as alíquotas de estoque de Bam15 (10 mM), Rotenone (10 mM), Antimicina A (10 mM) e 2-DG (500 mM) a partir de -80 °C congelador e descongele à temperatura ambiente.
NOTA: O estoque de 2 DG está pronto para uso. As outras drogas precisam ser diluídas para o estoque de trabalho. - Aqueça 10 mL do meio de ensaio em um banho de água de 37 °C.
- Diluir 10 mM Bam15 estoque para 50 μM de estoque de trabalho usando um procedimento de diluição de duas etapas: misturar 20 μL de estoque de 10 mM com 20 μL de DMSO para obter 5 mM de estoque intermediário. Em seguida, misture 10 μL do estoque intermediário acima de 5 mM com 990 μL de meio de ensaio pré-aquecido para obter o estoque final de 50 μM de trabalho.
- Diluir e combinar 10 mM rotenona e 10 mM de antimicina A a 10 μM Rotenone/Antimicina A (Rot/AA) estoque de trabalho por duas etapas de diluições: misturar 10 μL cada um de 10 mM Rotenone e 10 mM antimicina A estoque com 80 μL de DMSO para obter 1 mM Rot/AA estoque intermediário. Em seguida, misture 10 μL do estoque intermediário acima de 1 mM com 990 μL de meio de ensaio pré-aquecido para obter o estoque final de 10 μM Rot/AA de trabalho.
- Prepare-os recentemente os estoques de trabalho acima mencionados de compostos de injeção no dia do experimento e reserve-os à temperatura ambiente até carregar em portas de injeção do cartucho do sensor.
4. Dissecção de retina e preparação de soco de retina
- Eutanásia um rato por asfixia de CO2 seguindo as Diretrizes avma sobre eutanásia27.
NOTA: Não deixe o animal em uma câmara de CO2 por mais tempo do que o tempo necessário para a eutanásia. - Enuclear os olhos e colocar em gelo velho 1x PBS tampão em uma placa de Petri e, em seguida, colocá-lo sob um microscópio de dissecção.
- Remova cuidadosamente, cortando com microscisores, os músculos de reto extra ligados fora do globo ocular e corte o nervo óptico.
- Use uma agulha de 30 G para fazer um buraco na borda da córnea (limbus); este serve como o local de inserção para os microscissores. Em seguida, use um microscissores de dissecção fina para fazer um corte circular ao longo da borda da córnea, separando-o do copo posterior dos olhos.
- Use fórceps de dissecção afiados para remover a córnea, a lente e o humor vítreo longe do copo ocular.
- Use microscisores de dissecção fina para fazer vários pequenos cortes na camada escleral na borda do copo ocular. Evite cortar a camada de retina. Use dois fórceps de dissecção afiados para segurar o tecido escleral em cada lado do corte e puxe cuidadosamente a camada escleral para removê-lo da retina neural. Repita isso ao redor do copo ocular até que toda a esclera seja removida e um copo de retina intacto seja obtido.
- Use microscisores de dissecção e faça cortes radiais no copo de retina para achatá-lo e gerar várias seções distintas.
NOTA: Dependendo das habilidades de dissecção da pessoa e da experiência no manuseio de tecido retiniano fresco, o copo de retina pode ser cortado para gerar de 3 a 5 seções distintas. - Use um perfurador de biópsia de 1 mm de diâmetro para cortar um disco de retina de cada seção do copo de retina achatado.
NOTA: Deve-se tomar cuidado para que os discos de retina sejam perfurados a uma distância igual da cabeça do nervo óptico. - Use fórceps para transferir as pastilhas de malha pré-revestidas para a placa de petri dissecando. Com a ajuda de dois pincéis de cílios superfinos, coloque o disco de perfuração da retina sobre a inserção da malha. O disco de perfuração da retina é colocado no centro da inserção de malha com o lado da camada de célula de gânglio para baixo tocando a camada de malha e fotorreceptor voltada para cima.
NOTA: Frequentemente, algumas células RPE permanecem presas aos fotorreceptores, e a pigmentação dessas células pode ser usada como um indicador da orientação do disco de perfuração da retina.
5. Carregando as portas de injeção e calibração do cartucho do sensor
- Tire o da placa do cartucho do sensor hidratado da incubadora de 37 °C. Remova a tampa Hydro-Booster e coloque o cartucho do sensor de volta na placa de utilidade.
- Carregue o volume desejado de soluções de estoque de composto de injeção em portas apropriadas. Segure a ponta da pipeta no ângulo de 45°. Insira a ponta da pipeta no meio do caminho em uma porta de injeção com o bisel da ponta contra a parede oposta da porta de injeção e carregue suavemente o composto em cada porta. Evite introduzir bolhas de ar.
- Consulte o guia do usuário do instrumento para o volume do composto carregado em cada porta de injeção para um ensaio específico. Nos experimentos apresentados neste artigo, 68 μL de 50 μM Bam15 (para ensaio de estresse mitocondrial) ou 68 μL de 10 μM Rot/AA (para ensaio de taxa glicolítico) é carregado na porta A; 75 μL de 10 μM Rot/AA working stock (para ensaio de estresse mitocondrial) ou 75 μL de 500 mM 2-DG de estoque de trabalho (para ensaio de taxa glicolítico) é carregado na porta B.
- Carregar portas de injeção de todos os poços da placa, incluindo poços de correção de fundo e poços em branco para garantir a injeção adequada. Carregue a respectiva solução composta em cada porta para os poços de correção de fundo. O meio de ensaio pode ser substituído, em vez da solução composta, em cada um dos portos dos poços bancários.
- Coloque a placa do cartucho do sensor carregado, com tampa desligada, na máquina do analisador para iniciar a calibração antes da execução do ensaio. Após o fim da calibração, o programa fará uma pausa automática, esperando a substituição da placa de utilidade pela placa de captura de ilhota contendo socos de retina.
6. Carregar a placa de captura de ilhotas e iniciar a execução do ensaio
- Adicione 607 μL do meio de ensaio a cada poço da placa de captura de ilhotas
- Use fórceps para pegar a borda da inserção de malha contendo discos de perfuração de retina em cima e tirá-lo da placa de Petri. Bata levemente a parte inferior da inserção da malha em um tecido absorvente para remover líquido extra e coloque-o no poço da placa de captura de ilhotas. Repita esta etapa até que todas as pastilhas de malha com socos de retina sejam colocadas na placa de captura de ilhotas. Encha poços de correção de fundo e poços em branco com pastilhas de malha vazias.
- Use duas fórceps Graefe para pressionar cuidadosamente e suavemente a borda de cada inserção de malha e certificar-se de que estas estão bem inseridas na parte inferior da placa de captura de ilhotas.
- Coloque a placa de captura de ilhota carregada em uma incubadora de 37 °C por 5 minutos para aquecer.
- Ejete a placa de utilidade após a calibração ser concluída e substitua-a por uma e substitua-a pela placa de captura de ilhota, com tampa desligada, contendo socos na retina.
- Retome a execução do ensaio.
7. Executar a rescisão e o armazenamento de dados
- Após a execução ser concluída, ejete o cartucho do sensor e a placa de captura de ilhotas contendo socos na retina. Os dados são salvos automaticamente como arquivo .asyr.
- Use o software de análise de dados associado para visualizar e analisar os dados seguindo o guia de usuário do fabricante26.
- Use a função Exportar para exportar o arquivo .xslx dos dados, que podem ser visualizados e analisados usando software de planilha.
8. Salvando a amostra de soco de retina
- Após o ensaio, retire a placa da máquina, remova o cartucho do sensor e remova suavemente o meio de ensaio de cada poço usando uma pipeta.
- Aplique a tampa de volta e sele os lados da placa com a tira do parafilm.
- Armazém a -80 °C.
- Para normalização, quantifique o dna total ou o teor proteico do soco em cada poço.
9. Análise de dados
- Ensaio de estresse mitocondrial
NOTA: O valor OCR medido (totalOCR) representa o consumo total de oxigênio pelo tecido. Após a injeção de Bam15 (uncoupler), o OCR aumenta do nível basal (totalOCRbasal) para o nível máximo (totalOCRmax) e desce após a injeção de Rot/AA. O valor residual de OCR após a injeção de Rot/AA (totalOCRRot/AA) representa o consumo de oxigênio não mitocondrial.- Calcule o consumo de oxigênio relacionado a mitocôndrias como:
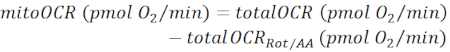 (Eq. 1) 28
(Eq. 1) 28
- Calcule a capacidade de reserva mitocondrial (MRC) como:
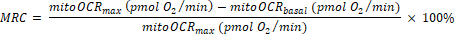 (Eq. 2) 29
(Eq. 2) 29
NOTA: A última leitura entre as 5 medidas antes da injeção de Bam15 é tomada como o valor "basal" (para totalOCRbasal e mitoOCRbasal). A leitura mais alta entre as 4 medidas após a injeção de Bam15 é usada como valor "máximo" (para totalOCRmax e mitoOCRmax). A leitura mais baixa entre as 4 medidas após a injeção de Rot/AA é usada como totalOCRRot/AA.
- Calcule o consumo de oxigênio relacionado a mitocôndrias como:
- Ensaio de taxa glicolítico
NOTA: O valor ecar medido (totalECAR) representa a acidificação total do meio pela atividade metabólica do tecido. Em geral, a acidificação do microambiente extracelular resulta principalmente pela extrusão do produto glicolítico, o lactato. O catabolismo dos substratos no ciclo TCA mitocondrial resulta na produção de CO2, que também acidifica o meio extracelular através da hidratação ao bicarbonato.- Mitocondrial substrato contribuiu com acidificação média (mitoECAR) da totalECAR para obter o glicoECAR.
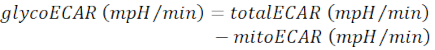 (Eq. 3) 28
(Eq. 3) 28
NOTA: A respiração mitocondrial e o ciclo TCA são processos fortemente acoplados. A produção de CO2 a partir de mitocôndrias é uma função da taxa de OXPHOS, que é mensurável pelo mitoOCR. - Calcule o mitoECAR como:
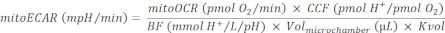 (Eq. 4) 28
(Eq. 4) 28
onde, o CCF (Fator de Contribuição de CO2) é um valor de razão empiricamente calculado, representando a quantidade de contribuição H+ da acidificação mediada por CO2 versus cada consumo de O2 do OXPHOS. CCF para este sistema é pré-determinado para ser 0,6028. A medição precisa da acidificação média é determinada pela capacidade tampão do meio, pela sensibilidade do sensor de pH do instrumento e pela capacidade efetiva da câmara de medição. Aqui, o BF (Buffer Factor) é um parâmetro da capacidade de buffer experimental in situ, representando a quantidade de H+ ou OH- adicionado à câmara de medição eficaz para alterar o nível de pH por 1 unidade. Quando o meio de ensaio personalizado é usado, o BF pode ser determinado titulando quantidades conhecidas de ácido no meio de ensaio seguindo o protocolo Fator Tampão30. O MH médio seahorse 7.4 usado neste protocolo tem um BF pré-determinado de 2,60 mmol H+/L/pH. A placa de captura de ilhotas usada neste protocolo tem um Volmicrochamber = 16,6 μL31. O fator de dimensionamento de volume, Kvol, é uma constante empiricamente determinada. O valor kvol não está disponível para a placa de captura de ilhotas, mas pode ser calculado a partir do valor da microplaca28, contabilizando a diferença de volume em seus microchambers, para 0,41.
NOTA: A injeção do Rot/AA desliga a respiração mitocondrial e força o tecido a mudar para glicólise para produção de ATP, levando a uma maior extrusão de lactato e um aumento na medição do ECAR. A glicólise é cessada com injeção de 2-DG, e a medição residual do ECAR revela acidificação não glicolítica e não mitocondrial do meio. - Calcule a capacidade de reserva glicolícica (GRC) como:
 (Eq. 5) 32
(Eq. 5) 32
onde, a última leitura entre as 5 medidas antes da injeção de Rot/AA é tomada como o valor "basal" (glycoECARbasal). A leitura mais alta entre as 4 medidas após a injeção de Rot/AA é usada como valor "máximo" (glycoECARmax). A leitura mais baixa entre as 4 medidas após a injeção de 2DG é usada como glicoECAR2-DG.
- Mitocondrial substrato contribuiu com acidificação média (mitoECAR) da totalECAR para obter o glicoECAR.
- Normalização
NOTA: A normalização é essencial quando se compara as leituras de tecidos da retina de diferentes faixas etárias ou entre amostras do tipo selvagem e patológica/degenerativa, que podem diferir em números celulares.- Use kits commericamente disponíveis para avaliar o conteúdo de DNA em cada disco de perfuração de retina33,34.
- Alternativamente, use o tampão de ensaio radioimunoprecipitação (tampão RIPA) para extrair proteína total do soco da retina e use o teor de proteína para normalização.
NOTA: A área de superfície de uma retina de camundongo adulto foi previamente determinada em torno de 20 mm2, e cada retina contém ~6,5 milhões de células35. Assim, cada soco de retina de 1 mm de diâmetro é ~1/25 de uma única retina e contém ~260K células. Pode-se referir a esses números ao comparar os dados de um soco de retina com os de outras amostras de tecido ou células cultivadas,
Resultados
Os dados aqui relatados são ensaios representativos de estresse mitocondrial mostrando traços OCR (Figura 1) e ensaio de taxa glicolítico mostrando traços OCR e traço ECAR (Figura 2), que foram realizados usando discos de perfuração de retina de 1 mm recém-dissecados a partir de 4 meses de camundongos nrl-L-EGFP transgênicos de 4 meses de idade( fundo C57B/L6). Esses camundongos expressam GFP especificamente em fotorrece...
Discussão
Fornecido aqui estão instruções detalhadas para a realização de ensaios baseados em microplaca de atividade de respiração mitocondrial e glicolise usando discos de perfuração de retina ex vivo, recém-dissecados. O protocolo foi otimizado para: 1) garantir o uso de um meio de ensaio adequado para tecido de retina ex vivo; 2) empregar o tamanho adequado dos discos de perfuração de retina para obter leituras de OCR e ECAR que se enquadram no alcance ideal de detecção da máquina; 3) revestime...
Divulgações
Os autores não têm nada a revelar.
Agradecimentos
Este trabalho é apoiado pelo Programa de Pesquisa Intramural do National Eye Institute (ZIAEY000450 e ZIAEY000546).
Materiais
| Name | Company | Catalog Number | Comments |
| 1X PBS | Thermo Fisher | 14190-144 | |
| 2-Deoxy glucose (2-DG), 500 mM stock solution | Sigma | D6134 | Dissolve in Seahorse XF DMEM medium, prepare ahead of time |
| 30-gauge needle | BD Precision Glide | 305106 | |
| Antimycin A, 10 mM stock solution | Sigma | A8674 | Dissolve in DMSO, prepare ahead of time |
| Bam15, 10 mM stock solution | TimTec | ST056388 | Dissolve in DMSO, prepare ahead of time |
| Biopsy puncher, 1 mm | Integra Miltex | 33-31AA | |
| Cell-Tak | Corning Life Sciences | CB40240 | |
| CO2 asphyxiation chamber | |||
| Dissection forceps-Dumont #5 | Fine Science Tools | 11251-10 | Stright tip |
| Dissection forceps-Dumont #7 | Fine Science Tools | 11274-20 | Curved tip |
| Dissection microscope | |||
| DMSO | Sigma | D2438 | |
| Graefe forceps | Fine Science Tools | 11051-10 | Curved, Serrated tip |
| Microscissors | Fine Science Tools | 15004-08 | Curved tip |
| NaOH solution, 1 M | Sigma-Aldrich | S8263 | Aqueous solution, prepare ahead of time |
| Rotenone, 10 mM stock solution | Sigma | R8875 | Dissolve in DMSO, prepare ahead of time |
| Seahorse calibration medium | Agilent | 100840-000 | |
| Seahorse XF 1.0 M glucose | Agilent | 103577-100 | |
| Seahorse XF 100 mM pyruvate | Agilent | 103578-100 | |
| Seahorse XF 200 mM glutamine | Agilent | 103579-100 | |
| Seahorse XF DMEM medium | Agilent | 103575-100 | pH 7.4, with 5 mM HEPES |
| Seahorse XFe24 Islet Capture FluxPak | Agilent | 103518-100 | Containing Sensor Cartridge and Islet Capture microplate |
| Seahorse XFe24, Extra Cellular Flux Analyzer | Agilent | ||
| Sodium bicarbonate solution, 0.1 M | Sigma-Aldrich | S5761 | Aqueous solution, prepare ahead of time |
| Superfine eyelash brush | Ted Pella | 113 |
Referências
- Nunnari, J., Suomalainen, A. Mitochondria: In sickness and in health. Cell. 148 (6), 1145-1159 (2012).
- Wong-Riley, M. T. Energy metabolism of the visual system. Eye Brain. 2, 99-116 (2010).
- Yu, D. Y., Cringle, S. J. Oxygen distribution and consumption within the retina in vascularised and avascular retinas and in animal models of retinal disease. Progress in Retina and Eye Research. 20, 175-208 (2001).
- Barot, M., Gokulgandhi, M. R., Mitra, A. K. Mitochondrial dysfunction in retinal diseases. Current Eye Research. 36 (12), 1069-1077 (2011).
- Joyal, J. S., Gantner, M. L., Smith, L. E. H. Retinal energy demands control vascular supply of the retina in development and disease: The role of neuronal lipid and glucose metabolism. Progress in Retina and Eye Research. 64, 131-156 (2018).
- Hurley, J. B., Lindsay, K. J., Du, J. Glucose, lactate, and shuttling of metabolites in vertebrate retinas. Journal of Neuroscience Research. 93 (7), 1079-1092 (2015).
- Haydinger, C. D., Kittipassorn, T., Peet, D. J. Power to see-Drivers of aerobic glycolysis in the mammalian retina: A review. Clinical and Experimental Ophthalmology. 48 (8), 1057-1071 (2020).
- Wright, A. F., et al. Lifespan and mitochondrial control of neurodegeneration. Nature Genetics. 36, 1153-1158 (2004).
- Bossy-Wetzel, E., Schwarzenbacher, R., Lipton, S. A. Molecular pathways to neurodegeneration. Nature Medicine. 10, 2-9 (2004).
- Leveillard, T., Philp, N. J., Sennlaub, F. Is retinal metabolic dysfunction at the center of the pathogenesis of age-related macular degeneration. International Journal of Molecular Sciences. 20 (3), (2019).
- Vlachantoni, D., et al. Evidence of severe mitochondrial oxidative stress and a protective effect of low oxygen in mouse models of inherited photoreceptor degeneration. Human Molecular Genetics. 20 (2), 322-335 (2011).
- Grenell, A., et al. Loss of MPC1 reprograms retinal metabolism to impair visual function. Proceedings of the National Academy of Science U. S. A. 116 (9), 3530-3535 (2019).
- Wright, A. F., Chakarova, C. F., Abd El-Aziz, M. M., Bhattacharya, S. S. Photoreceptor degeneration: genetic and mechanistic dissection of a complex trait. Nature Reviews in Genetics. 11 (4), 273-284 (2010).
- Jarrett, S. G., Boulton, M. E. Consequences of oxidative stress in age-related macular degeneration. Molecular Aspects of Medicine. 33 (4), 399-417 (2012).
- Rozing, M., et al. Age-related macular degeneration: A two-level model hypothesis. Progress in Retina Eye Research. 76, 100825 (2020).
- Yokosako, K., et al. Glycolysis in patients with age-related macular degeneration. Open Ophthalmology Journal. 8, 39-47 (2014).
- Bek, T. Mitochondrial dysfunction and diabetic retinopathy. Mitochondrion. 36, 4-6 (2017).
- Yumnamcha, T., Guerra, M., Singh, L. P., Ibrahim, A. S. Metabolic dysregulation and neurovascular dysfunction in diabetic retinopathy. Antioxidants. 9 (12), (2020).
- Futterman, S., Kinoshita, J. H. Metabolism of the retina. I. Respiration of cattle retina. Journal of Biological Chemistry. 234 (4), 723-726 (1959).
- Linsenmeier, R. A. Effects of light and darkness on oxygen distribution and consumption in the cat retina. Journal of General Physiology. 88 (4), 521-542 (1986).
- Medrano, C. J., Fox, D. A. Oxygen consumption in the rat outer and inner retina: light- and pharmacologically-induced inhibition. Experiments in Eye Research. 61 (3), 273-284 (1995).
- Kooragayala, K. Quantification of oxygen consumption in retina ex vivo demonstrates limited reserve capacity of photoreceptor mitochondria. Investigative Ophthalmology and Visual Science. 56 (13), 8428-8436 (2015).
- Adlakha, Y. K., Swaroop, A. Determination of mitochondrial oxygen consumption in the retina ex vivo: applications for retinal disease. Methods in Molecular Biology. 1753, 167-177 (2018).
- . Agilent Mitocondrial stress test user guide Available from: https://www.agilent.com/cs/library/usermanuals/public/XF_Cell_Mito_Stress_Test_Kit_User_Guide.pdf (2021)
- . Agilent Glycolytic rate assay user guide Available from: https://www.agilent.com/cs/library/usermanuals/public/103344-400.pdf (2021)
- . Agilent wave 2.6 user guide Available from: https://www.agilent.com/cs/library/usermanuals/public/103344-400.pdf (2021)
- . AVMA Guidelines for the Euthanasia of Animals Available from: https://www.avma.org/sites/default/files/2020-01/2020-Euthanasia-Final-1-17-20.pdf (2021)
- . Improving Quantification of Cellular Glycolytic Rate Using Agilent Seahorse XF Technology Available from: https://www.agilent.com/cs/library/whitepaper/public/whitepaper-improve-quantification-of-cellular-glycolytic-rate-cell-analysis-5991-7894en-agilent.pdf (2021)
- . Report Generator User Guide Agilent Seahorse XF Cell Mito Stress Test Available from: https://www.agilent.com/cs/library/usermanuals/public/Report_Generator_User_Guide_Seahorse_XF_Cell_Mito_Stress_Test_Single_File.pdf (2021)
- . Agilent Seahorse XF Buffer Factor Protocol Available from: https://www.agilent.com/cs/library/usermanuals/public/usermanual-xf-buffer-factor-protocol-cell-analysis-S7888-10010en-agilent.pdf (2021)
- . Agilent sensor cartridges and cell culture microplates Available from: https://www.agilent.com/cs/library/brochures/5991-8657EN_seahorse_plastics_brochure.pdf (2021)
- . Agilent Seahorse XF Glycolysis Stress Test Kit User Guide Available from: https://www.agilent.com/cs/library/usermanuals/public/XF_Glycolysis_Stress_Test_Kit_User_Guide.pdf (2021)
- Fan, Y. Y. A bioassay to measure energy metabolism in mouse colonic crypts, organoids, and sorted stem cells. American Journal of Physiology-Gastrointestinal and Liver Physiology. 309, 1-9 (2015).
- Huang, L., et al. Ductal pancreatic cancer modeling and drug screening using human pluripotent stem cell- and patient-derived tumor organoids. Nature Medicine. 21 (11), 1364-1371 (2015).
- Jeon, C. J., Strettoi, E., Masland, R. H. The major cell populations of the mouse retina. Journal of Neuroscience. 18 (21), 8936-8946 (1998).
- Akimoto, M., et al. Targeting of GFP to newborn rods by Nrl promoter and temporal expression profiling of flow-sorted photoreceptors. Proceedings of the National Academy of Science U. S. A. 103 (10), 3890-3895 (2006).
- Kenwood, B. M., et al. Identification of a novel mitochondrial uncoupler that does not depolarize the plasma membrane. Molecular Metabolism. 3 (2), 114-123 (2014).
- Corso-Diaz, X., et al. Genome-wide profiling identifies DNA methylation signatures of aging in rod photoreceptors associated with alterations in energy metabolism. Cell Reports. 31 (3), 107525 (2020).
- Berkowitz, B. A., et al. Mitochondrial respiration in outer retina contributes to light-evoked increase in hydration in vivo. Investigative Ophthalmology and Visual Science. 59 (15), 5957-5964 (2018).
- Joyal, J. S., et al. Retinal lipid and glucose metabolism dictates angiogenesis through the lipid sensor Ffar1. Nature Medicine. 22 (4), 439-445 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoExplore Mais Artigos
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados