Method Article
Interrogatorio de mecanobiología totalmente óptica de proteína asociada a sí en cáncer humano y células normales utilizando un sistema multifuncional
* Estos autores han contribuido por igual
En este artículo
Resumen
Este documento presenta un protocolo detallado sobre cómo utilizar un sistema integrado multifuncional y programable por el usuario que permite la obtención automática de imágenes multicanal y el análisis mecanobiológico para dilucidar la mecanosensibilidad de la proteína asociada al Sí (YAP).
Resumen
Las imágenes multifuncionales a largo plazo y el análisis de células vivas requieren una coordinación funcional y optimizada de varias plataformas de hardware y software. Sin embargo, el control manual de varios equipos producidos por diferentes fabricantes requiere mucha mano de obra y requiere mucho tiempo, lo que puede disminuir la precisión, la reproducibilidad y la calidad de los datos adquiridos. Por lo tanto, un sistema todo en uno y programable por el usuario que permita la adquisición automática, multifuncional y a largo plazo de imágenes y sea compatible con la mayoría de las plataformas de microscopía fluorescente puede beneficiar a la comunidad científica. Este documento presenta los protocolos operativos completos de la utilización de un novedoso sistema de software integrado que consiste en (1) un programa de software construido en el hogar, titulado "Programa de integración multifuncional automática (AMFIP)", que permite la adquisición automática de imágenes multicanal, y (2) un conjunto de paquetes de análisis de imágenes cuantitativas y computación de tracción celular.
Este sistema integrado se aplica para revelar la relación previamente desconocida entre la distribución espacio-temporal de la proteína asociada al Sí (YAP) mecanosensible y la mecánica celular, incluida la propagación y tracción celular, en células normales humanas (B2B) y células de cáncer de pulmón (PC9) diseñadas por CRISPR / Cas9. Aprovechando la capacidad de control y lectura multicanal de este sistema, el resultado muestra: (1) las células normales B2B y las células cancerosas PC9 muestran una relación distinta entre la expresión de YAP, la tracción y la dinámica celular durante los procesos de propagación y migración celular; y (2) las células cancerosas PC9 aplican fuerzas perinucleares notables sobre los sustratos. En resumen, este documento presenta un protocolo paso a paso detallado sobre cómo utilizar un sistema integrado programable por el usuario que permite obtener imágenes y análisis multifuncionales automáticos para dilucidar la mecanosensibilidad de YAP. Estas herramientas abren la posibilidad de exploraciones detalladas de dinámicas de señalización multifacéticas en el contexto de la fisiología y patología celular.
Introducción
El objetivo general de este método es permitir imágenes multifuncionales totalmente ópticas y análisis de células vivas. Un programa de imágenes todo en uno que permite la coordinación automática de dispositivos optoelectrónicos multifuncionales reducirá las operaciones manuales intensivas en mano de obra y propensas a errores y es esencial para que los investigadores realicen imágenes de células vivas a largo plazo1,2,3,4. Sin embargo, la mayoría de los programas públicos existentes en la comunidad de investigación biomédica solo se aplican a dispositivos optoelectrónicos limitados o requieren hardware adicional para la coordinación de diferentes equipos5,6,7,8,9. Recientemente, se ha desarrollado un programa de código abierto y basado en software, titulado "Programa de integración multifuncional automática (AMFIP)", que permite imágenes multicanal y de lapso de tiempo. Basado en el lenguaje Java y la interfaz de programación de aplicaciones (API) de μManager11,12, AMFIP se desarrolló como un complemento en μManager que ejecuta scripts Java personalizados para lograr comunicaciones basadas en software de múltiples plataformas de hardware y software optoelectrónicas, incluidas, entre otras, las de Nikon. El establecimiento de AMFIP abre la posibilidad de interrogatorio programable y multifuncional de comportamientos celulares. En este documento se desarrolla un sistema experimental y computacional integrado que combina AMFIP con análisis de imágenes digitales y microscopía de fuerza de tracción celular. El sistema permite la elucidación de la mecanobiología YAP distinta en líneas celulares B2B normales humanas (Figura 1) y cáncer de pulmón PC9 (Figura 2) diseñadas por CRISPR / Cas9. El sistema proporciona a la comunidad científica una solución integral que evita la demanda de comprar dispositivos de control adicionales que pueden no estar disponibles y / o compatibles con todos los sistemas de imágenes.
Los protocolos presentados en este documento presentan cómo (1) aplicar AMFIP para realizar imágenes automáticas a largo plazo para ambas líneas celulares de ingeniería CRISPR / Cas9 que expresan YAP etiquetado con mNEonGreen2; y (2) combinar Fiji ImageJ, MATLAB y Origin para el análisis cuantitativo de la relación nuclear/citoplasma (N/C) de YAP basada en su intensidad fluorescente (Figura 3 y Figura 4), campo de desplazamiento celular (Figura 1C y Figura 2C) y campo de tracción celular (Figura 1D y Figura 2D ). Los resultados sugieren que (1) durante las primeras 10 h de propagación celular sobre los sustratos que tienen rigidez mecánica fisiológicamente relevante13,14,15,16,17,18, la relación YAP N/C de células B2B individuales muestra una variación y fluctuación dependiente del tiempo más notable en comparación con la de las células PC9 individuales (Figura 5 y Figura 6 ); y (2) las células cancerosas PC9 generan una tracción notable en sus regiones perinucleares (Figura 7). El sistema integrado y las metodologías descritas en este protocolo trascienden los tipos específicos de células y moléculas optogenéticas. Los investigadores pueden aplicar los protocolos para personalizar sus experimentos específicos de interrogación de células vivas y dilucidar la dinámica de señalización multifacética en el contexto de la fisiología y la patología celular.
Protocolo
1. Generación de una línea celular estable de cáncer de pulmón humano (PC9) editada por CRISPR / Cas9 y una línea celular epitelial bronquial humana (Beas2B) que expresan endógenamente la proteína YAP etiquetada con mNeonGreen21-10/11
- Realizar la reacción en cadena de la polimerasa (PCR) para amplificar la secuencia de ADN que codifica la 11ª hebra de la proteína de fluorescencia, mNeonGreen2, utilizando la ADN polimerasa de alta fidelidad (ver la Tabla de Materiales).
- Incorpore la secuencia de ADN amplificada en el locus genómico YAP de las líneas celulares PC9 y B2B utilizando el sistema de edición de genes CRISPR-Cas9.
NOTA: Esta secuencia de ADN complementa las hebras 1-10 de mNeonGreen2 para emitir fluorescencia. El mapa de secuencia genómica de YAP-mNeonGreen21-10/11 se muestra en la Figura Suplementaria S1. El mapa contiene las secuencias genómicas, donantes y mNeonGreen2 etiquetadas. - Compruebe la expresión de mNeonGreen2 diseñada por CRISPR/Cas9 con un microscopio de epifluorescencia (consulte la Tabla de materiales). Debido a que el mNeonGreen2 se marca en YAP cada vez que las células expresan YAP en el contexto de su red reguladora de genes nativos, verifique la presencia de la intensidad de fluorescencia en ambas células diseñadas por CRISPR / Cas9 y compárela con la de las células parentales (control).
NOTA: Para seguir este protocolo, utilice (1) un láser de 488 nm (47,5 mW/mm2) para la excitación, (2) un objetivo 40x (apertura numérica (NA) = 0,95) y un filtro de emisión de paso de banda (ET525/50 nm) para la medición de fluorescencia, y (3) software ImageJ para medir, cuantificar y comparar las intensidades de fluorescencia. - Confirmar la correcta integración de mNeonGreen211 extrayendo ADN genómico de las líneas celulares editadas por CRISPR/Cas9; realizar PCR utilizando cebadores que flanquean el inserto genómico y secuenciación para confirmar la inserción en el loci genómico correcto19,20.
- Derribe el mNeonGreen211 utilizando el sistema de edición de genes CRISPR/Cas9 y compruebe la reducción de la intensidad de fluorescencia en las células utilizando los mismos sistemas de microscopio y parámetros de imagen descritos en el paso 1.3.
NOTA: Este paso confirma la integración correcta de mNeonGreen211 mediante la comparación de intensidades de fluorescencia. Las células diseñadas por CRISPR/Cas9 sin derribo y las células parentales se utilizan como control. - Recolecte las células con la proteína marcada de interés a través de la clasificación de células activadas por fluorescencia (FACS).
- Para preparar las células para la clasificación de FACS, tripsinícelas y resuspóndelas en solución salina tamponada con fosfato (PBS).
- Recolecte células con fluorescencia mNeonGreen2 por encima del nivel de fondo de las líneas celulares parentales en dos rondas enriquecedoras de clasificación FACS.
NOTA: La línea de tiempo para generar las líneas celulares editadas por CRISPR/Cas9 que se describen aquí es del orden de 1-2 meses. Todas las líneas celulares se ponen a disposición del público previa solicitud para que otros laboratorios de investigación puedan reproducir los resultados.
2. Mantenimiento de células PC9 y B2B
- Mantener ambas líneas celulares en incubadoras de cultivo de tejidos humidificados con 5% de CO2 a 37 °C.
- Cultivo 106 células PC9 y Beas2B marcadas endógenamente en matraces de 75 cm2 con 12 ml de medio RPMI-1640 suplementado con suero bovino fetal al 10% y 100 μg/ml de penicilina-estreptomicina. Subcultivo de ambas líneas celulares cuando la confluencia celular alcanza ~80%.
- Pruebe ambas líneas celulares para detectar micoplasma cada 3 meses utilizando un kit de detección de micoplasma, siguiendo estrictamente todos los protocolos recomendados por el fabricante.
- Guarde las líneas celulares en un congelador de -80 °C.
- Use las líneas celulares que están <20 pasajes del deshielo para todos los experimentos.
3. Configuración del entorno de hardware y software
- Configuración del entorno de hardware del experimento
- Conecte el controlador confocal y el microscopio invertido al ordenador (consulte la Tabla de materiales).
- Instale la plataforma de software (Tabla de Materiales).
- Encienda el controlador confocal y el microscopio invertido. A continuación, inicie Elements.
- Abra los paneles de control del microscopio confocal, láser e invertido en Elements. A continuación, verifique si los tres paneles funcionan correctamente probando el movimiento de la etapa motorizada, la conmutación de los objetivos del microscopio y el escaneo espacial de las líneas láser.
- Configuración del entorno de software de AMFIP
- Instale IntelliJ, Java Development Kit 14.0, μManager versión 2.0 gamma y Fiji ImageJ en el equipo.
- Abra el proyecto AMFIP descargado de GitHub (enlace: https://github.com/njheadshotz/AMFIP) en IntelliJ.
- Haga clic en configuración | | del compilador Procesadores de anotaciones y marque Habilitar procesamiento de anotaciones.
- Haga clic en estructura del proyecto | Artefactos y crear un archivo JAR. Establezca el directorio de salida en mmplugins en el directorio μManager .
- Haga clic en estructura del proyecto | Bibliotecas y añadir mmplugins y plugins bajo el directorio μManager .
- Haga clic en agregar configuración en el menú desplegable Ejecutar y cree una aplicación.
- Ingrese ij. ImageJ en la clase Main.
- Escriba -Xmx3000m -Dforce.annotation.index=true en la opción VM.
- Establezca el directorio μManager en el directorio Work.
- Haga clic en Ejecutar para activar μManager con el complemento AMFIP.
- Conecte μManager con el microscopio invertido.
- Agregue el controlador adaptativo del microscopio invertido21 al directorio μManager .
- Abra μManager. Haga clic en dispositivos | Asistente de configuración de hardware y cree una nueva configuración.
- Agregue el controlador Ti2 en Dispositivos disponibles.
- Seleccione todos los dispositivos periféricos y guarde el nuevo archivo de configuración.
- Reinicie μManager y seleccione el archivo de configuración en el paso 3.2.4 en Configuración de inicio de Micro-Manager.
4. Preparación del gel
- Trate la cubierta de vidrio con 3-aminopropiltripmetoxisilano durante 7 min a temperatura ambiente (24 °C).
- Use agua desionizada (DI) para enjuagar la cubierta de vidrio y seque la cubierta durante 20 minutos a 160 °C.
- Trate la cubierta de vidrio con glutaraldehído al 0,5% durante 30 min y enjuague con agua DI.
- Mezcle la solución de acrilamida, la solución de N,N′-metilendinbisacrilamida (bis) y las perlas fluorescentes suspendidas en solución salina tamponada con HEPES de 10 mM. Utilizar solución de persulfato de amonio al 10% (p/v) y N,N,N′,N′-tetrametilendiamina (TEMED) como iniciadores de la polimerización. Cambiar el porcentaje de cada componente para conseguir la rigidez mecánica deseada de los hidrogeles de poliacrilamida (PAA) siguiendo los protocolos establecidos anteriormente descritos13,14.
NOTA: En este protocolo, gel de 2 kPa: acrilamida = 12.5% y bis-acrilamida = 6.5%; Gel de 5 kPa: acrilamida = 12,5% y bis-acrilamida = 21,5%; y gel de 40 kPa: acrilamida = 12,5% y bis-acrilamida = 31,5%. Todos los % enumerados son porcentaje de volumen. - Después de 35 min, pelar la cubierta de vidrio del hidrogel PAA solidificado y lavar el hidrogel con solución salina tamponada con HEPES de 50 mM dos veces (5 min cada vez).
- Trate la superficie del hidrogel con una solución hidratada de hidracina durante 6 h.
- Enjuague el hidrogel con ácido acético durante 30 min. Retire el ácido acético y enjuague con PBS durante 30 min.
- Oxidar la solución de fibronectina (50 μg/ml en PBS) con periodato de sodio durante 30 min.
- Cubra la superficie del hidrogel con la solución de fibronectina oxidada y espere 35 min.
- Añadir PBS para sumergir el hidrogel y almacenar a 4 °C. Cubra todas las placas de Petri que contienen los hidrogeles con papel de aluminio para evitar cualquier exposición a la luz a los hidrogeles.
5. Cultivo celular
NOTA: Realizar cultivo celular mediante técnica aséptica.
- Unir las cubiertas de vidrio con los hidrogeles PAA a la placa de Petri con fondo de vidrio de 35 mm para evitar la deriva física de los geles durante los procesos de siembra celular e imágenes.
- Usando pinzas limpias esterilizadas, levante la cubierta (con el hidrogel PAA en la parte superior) de la placa de Petri que contiene los geles preparados.
- Use una toallita seca para limpiar las gotas de agua en la superficie inferior de la cubierta de vidrio.
- Use las pinzas esterilizadas para sujetar la cubierta de vidrio.
- Coloque pequeñas gotas (1-5 μL) de pegamento de cianoacrilato en las dos esquinas diagonales de la superficie inferior.
- Use toallitas esterilizadas para eliminar el exceso de pegamento.
- Use las pinzas esterilizadas para reemplazar la cubierta en la placa de Petri con fondo de vidrio. Presione ligeramente las esquinas de la cubierta para asegurarse de que las gotas de pegamento hagan contacto completo con la superficie de la placa de Petri.
- Vuelva a colocar la tapa en la placa de Petri para minimizar la evaporación de PBS en los hidrogeles PAA. Espere 3 minutos para permitir que el pegamento se solidifique y se seque en la placa de Petri.
- Llene la placa de Petri con 4 ml de PBS.
- Repita los pasos anteriores 5.1.1-5.1.8 para las muestras de hidrogel PAA restantes en las placas de Petri utilizadas para la obtención de imágenes.
- Utilice etanol al 75% para esterilizar la superficie exterior de todas las placas de Petri y transferirlas al gabinete de bioseguridad del cultivo de tejidos. Encienda la luz ultravioleta durante 5 minutos y esterilice las muestras.
- Sembra las células en la superficie superior del gel.
- Apague la luz ultravioleta. Saque el matraz (que contiene células B2B/PC9) de la incubadora de 37 °C en el gabinete de bioseguridad. Utilice una pipeta conectada a una bomba de vacío para aspirar todo el medio de cultivo y agregue 5 ml de PBS para lavar el matraz.
- Agregue 2 ml de tripsina al 0.05% para separar las células de la parte inferior del matraz.
- Coloque el matraz en la incubadora. Espere 5 min.
- Transfiera el matraz al gabinete de bioseguridad. Añadir 8 ml de medio de cultivo fresco al matraz y pipetear hacia arriba y hacia abajo varias veces para suspender las células de forma homogénea.
- Transfiera todos los 10 ml de la suspensión celular a un tubo de 15 ml y centrífuga a 300 × g durante 5 min.
- Revise el pellet celular en la parte inferior del tubo. Incline lentamente el tubo horizontalmente y use la pipeta aspiradora para extraer todo el medio de cultivo del tubo sin tocar el gránulo celular. A continuación, agregue 8 ml de medio de cultivo fresco y pipetee hacia arriba y hacia abajo varias veces hasta que todas las células se mezclen homogéneamente con el medio.
- Deposite 100 μL de la suspensión celular (150 células/μL) sobre la superficie del gel y espere 5 min. A continuación, agregue lentamente 4 ml de medio de cultivo fresco a las placas de Petri; evite agregar el medio fresco directamente sobre el gel.
- Coloque la placa de Petri en la incubadora de 37 °C. Espere para permitir que las células se adhieran a la superficie del gel (B2B: 0.5-1 h; PC9: 4-5 h).
6. Imágenes celulares
NOTA: AMFIP permite la obtención de imágenes automáticas, multicanal y a largo plazo mediante la coordinación con diferentes sistemas de hardware y software: (1) AMFIP manipula μManager para mover automáticamente la etapa motorizada del microscopio Ti2-E a múltiples campos de visión (FOV) y adquirir imágenes de campo brillante a través de una cámara monocromática (Tabla de materiales); y (2) AMFIP activa múltiples archivos de macro dentro de Elements con un script Java personalizado para realizar operaciones automáticas para imágenes confocales de pila z y la conmutación de diferentes canales láser (405 nm y 488 nm).
- Establezca el entorno para imágenes a largo plazo.
- Coloque la cámara de ambiente en la etapa motorizada del microscopio invertido. Ajuste el caudal de CO2 a 160 ml/min y ajuste la temperatura de la cámara (parte superior: 44 °C; baño: 42 °C; etapa: 40 °C). A continuación, agregue 40 ml de agua purificada en el baño de la cámara.
- Saque la placa de Petri con fondo de vidrio con células de la incubadora y colóquela en la cámara de ambiente.
- Encienda el controlador confocal y el microscopio invertido. Cambie la trayectoria de la luz hacia la derecha y observe las celdas que se conectan usando μManager. Si se han adherido suficientes células al gel, transfiera la placa de Petri de vuelta a la incubadora. Si no se han adherido suficientes células al gel, continúe la incubación celular durante otros 30 minutos para las células B2B y 60 minutos para las células PC9.
- Corta dos pequeños trozos de cinta adhesiva y pégalos en la cámara alrededor del orificio circular. A continuación, aplique un poco de pegamento adhesivo sobre la cinta (solo en el área que cubrirá la placa de Petri).
- Saque la placa de Petri de la incubadora. A continuación, coloque lentamente la placa de Petri en la cámara y deje que el fondo de la placa haga contacto con el pegamento.
- Presione la tapa de la placa de Petri durante 1 minuto para permitir que el pegamento haga contacto completo con la placa de Petri y se solidifique. A continuación, empuje suavemente la placa de Petri horizontalmente para confirmar que la placa de Petri es inamovible en la cámara.
- Cierre la tapa de la cámara.
- Establezca los parámetros de adquisición de imágenes para imágenes de campo brillante.
- Abra IntelliJ y establezca un parámetro T1 (por ejemplo, 120 s) en la línea 93 del archivo Elements_script.java. Asegúrese de que este valor es mayor que el tiempo de ejecución de la macro en Elementos utilizados para la imagen confocal de un campo de visión (FOV). Haga clic en el botón Ejecutar para iniciar el proyecto AMFIP IntelliJ.
- Haga clic en el botón Live and Multi-D Acq. en la interfaz principal de μManager. A continuación, cambie la trayectoria de la luz del microscopio invertido a la derecha para obtener imágenes de campo brillante, cambie al objetivo 10x y abra la luz del diodo emisor de luz (LED) (la fuente de luz para las imágenes de campo brillante; intensidad: 5%).
- Haga clic en la trayectoria de la luz, el objetivo del microscopio y el botón de la lámpara LED en el panel Elements Ti2 o presione manualmente los botones correspondientes en el microscopio.
- Ajuste el joystick XY y la perilla del plano Z para encontrar la posición correcta y el plano de enfoque del gel en la placa de Petri. Utilice un objetivo 10x para encontrar los FOV apropiados de múltiples células individuales unidas al gel.
- Marque la casilla Posiciones múltiples (XY) en la ventana Adquisición multidimensional . Haga clic en el botón Editar lista de posiciones... y observe la ventana Lista de posiciones del escenario que aparece. A continuación, cambie el objetivo a 40x, aumente la intensidad de la luz LED al 15%, reajuste la etapa motorizada XY para localizar los FOV y registre las coordenadas haciendo clic en el botón Marcar en la ventana Lista de posición del escenario .
- Graba 67 FOVs deseados. Haga clic en el botón Guardar como... en la ventana Lista de posiciones del escenario para registrar las coordenadas. Introduzca T1 (el parámetro, por ejemplo, 120 s, definido en el paso 6.2.1) en el intervalo de tiempo de adquisición de imágenes a T1 en la sección Puntos de tiempo en la ventana Adquisición multidimensional .
- Establezca la adquisición de imágenes para imágenes 2D-YAP y cuentas.
- Abra Elementos, cambie la trayectoria de la luz a la derecha para obtener imágenes confocales y apague la luz LED. A continuación, haga clic en el botón Eliminar enclavamiento y encienda el canal láser FITC (para imágenes YAP) marcando la casilla FITC .
- Ajuste la velocidad de escaneo a 1 fotograma por 2 s haciendo clic en el botón 1/2 y gire la perilla del plano Z para encontrar la posición Z de las celdas adjuntas rápidamente. Registre los límites inferior y superior de la pila Z.
- Haga clic en Macro en la cinta superior, seleccione Editor de macros en el menú desplegable Macro e introduzca los valores del paso 6.3.2 en un archivo de macros.
- Encienda el canal láser de 4′,6-diamidino-2-fenilindole (DAPI) (para imágenes de cuentas) marcando la casilla DAPI para encontrar y registrar la posición Z enfocada de las perlas. Vaya al editor de macros e introduzca los valores registrados en el archivo de macros.
- Establezca la tarea de mover la etapa motorizada usando AMFIP.
- Vaya a μManager y haga clic en Plugins | Automatización para abrir la interfaz gráfica de usuario (GUI) de AMFIP. Haga clic en los botones Agregar punto o Quitar punto para adquirir el número exacto de FOV seleccionados. Introduzca las coordenadas registradas de los MOV en el panel Coordenadas.
- Defina el tiempo total del experimento en el campo de texto Tiempo total del experimento .
- Haga clic en el botón Configuración de tiempo adicional y defina el intervalo de tiempo T2 (por ejemplo, 30 min) para mover la etapa motorizada a cada FOV.
- Maximice el tamaño de la ventana de Elements y arrastre la GUI de AMFIP al lado derecho de la pantalla para evitar que la GUI perturbe las operaciones automáticas del cursor.
- Haga clic en el botón Entrar . Una vez finalizada la primera macro, haga clic en el botón Adquirir! en la ventana Adquisición multidimensional .
- Disolver las células después de la adquisición de la imagen.
- Después de finalizar la imagen a largo plazo, detenga la tarea AMFIP haciendo clic en el botón Pausa en la ventana del complemento de automatización y en el botón Detener en la ventana Adquisición multidimensional .
- Abra Elements y configure la imagen de la pila Z haciendo clic en los botones Superior e Inferior en la ventana Adquisición de ND (configure el rango Z para que sea mayor que el rango Z de las cuentas). Cambie la trayectoria de la luz hacia la derecha y abra la luz LED (intensidad: 15%).
- Retire lenta y cuidadosamente las tapas de la cámara y la placa de Petri. Mientras tanto, monitoree la vista de campo brillante para detectar cualquier deriva del FOV.
- Usando una pipeta de plástico para tomar 0,5 ml de solución de dodecil sulfato de sodio (SDS), sostenga cuidadosamente la pipeta de plástico un poco por encima del medio de cultivo en la placa de Petri y agregue 1-2 gotas de la solución SDS en el medio de cultivo.
- Una vez que las celdas en la vista de campo brillante se disuelvan, cambie la trayectoria de la luz a la izquierda, cierre la luz LED, haga clic en el botón Eliminar enclavamiento .
- Ejecute la imagen de la pila Z. Guarde la pila de imágenes y asígnele el nombre Reference_N (N es el número de secuencia de cada FOV).
- Haga clic en el botón Posiciones múltiples (XY) en la ventana Adquisición multidimensional . A continuación, seleccione el siguiente FOV y haga clic en el botón Ir a para mover la etapa motorizada al segundo FOV.
- Repita el paso 6.5.7 para cada FOV.
7. Medición de la relación YAP N/C
- Realice análisis de imágenes para medir la relación YAP N/C utilizando el software Fiji ImageJ (Figura 4).
- Abra Fiji ImageJ. Importe la pila de imágenes de campo brillante para todos los FOV adquiridos por μManager.
- Abra el menú desplegable Imagen y seleccione Pilas | Herramientas | Guardián de rebanadas. A continuación, exporte la pila de imágenes de campo brillante para cada FOV.
- Importe la imagen de fluorescencia del canal FITC y superpóngala con la imagen de campo brillante para el mismo FOV. Para ello, elija la imagen fluorescente y seleccione Superposición | Añadir imagen... (Imagen para agregar: la imagen de campo brillante; La ubicación X e Y depende del tamaño de la imagen de campo brillante adquirida por diferentes cámaras; Opacidad: 60-70).
- Abra el menú desplegable Analizar y seleccione Establecer medidas.... Seleccione Área; Densidad integrada y valor gris medio.
- Haga clic en el botón Selecciones a mano alzada en la interfaz principal de ImageJ.
- Dibuja el contorno del cuerpo celular y el núcleo deseado. A continuación, haga clic en Analizar | Mida o pulse el botón M del teclado.
- Observe la ventana Resultados que aparece. Tenga en cuenta que los valores de la columna Área representan el área de la región seleccionada (μm2) y los valores de la columna IntDen representan la intensidad de fluorescencia de la región seleccionada.
- Calcule la relación YAP N/C utilizando las siguientes fórmulas (1), (2) y (3):
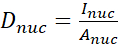 (1)
(1)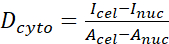 (2)
(2)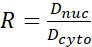 (3)
(3)
Donde Inuc e Icel representan la intensidad relativa del núcleo y el cuerpo celular, y Anuc y Acel representan el área del núcleo y el cuerpo celular. R es la relación YAP N/C. - Guarde los contornos para el cálculo futuro de la fuerza de tracción dipolar y el desplazamiento pericelular/perinuclear. Para hacer esto, haga clic en Analizar | Herramientas | Guardar coordenadas XY...
8. Medición del campo de tracción
- Aplique microscopía de fuerza de tracción a través de los complementos Fiji ImageJ22,23.
- Abra Fiji ImageJ.
- Importe la pila de imágenes de cuentas para un FOV.
- Seleccione la división que muestra la distribución más clara de cuentas y extráigala haciendo clic en Imágenes | Pilas | Herramientas | Guardián de rebanadas.
- Importe la pila de imágenes de la referencia para el mismo FOV.
- Elija el sector con el mismo brillo y contraste que el sector en el paso 8.1.3. A continuación, extráigalo como imagen de referencia.
- Seleccione imágenes | Pilas | Herramientas | Concatenar para combinar los dos sectores de los pasos 8.1.3 y 8.1.5 (seleccione la imagen de referencia como primer segmento).
- Seleccione Plugins | | de coincidencia de plantillas Alinear sectores en pila o plugins | Estabilizador de imagen para alinear las dos rebanadas.
- Seleccione | de imagen Pilas | Apilar a imágenes. A continuación, seleccione | de imagen Tablas de búsqueda | Verde para convertir el color de la primera división a verde y seleccionar Imagen | Tablas de búsqueda | Rojo para convertir el color de la segunda rebanada a rojo.
- Seleccione | de imagen | de color Combinar canales para combinar las dos imágenes.
- Superponga la imagen con la imagen de campo brillante del mismo FOV y utilice esta imagen superpuesta para observar el desplazamiento de cuentas.
- Seleccione Plugins | | PIV PIV iterativo (Básico).... Establezca el tamaño de la ventana de interrogación en 128/256; 64/128; 32/64 (al menos cuatro cuentas por ventana de interrogatorio). Establezca el umbral de correlación en 0,6.
- Haga clic en Aceptar. Una vez finalizado el cálculo, guarde el archivo de texto con los datos sin procesar del desplazamiento de cuentas en una carpeta ordinaria creada por el usuario.
- Seleccione Plugins | | FTTC FTTC y elija el archivo de texto en el paso 8.1.9.
- Ingrese el tamaño de píxel (μm), el módulo de Young del gel (Pascal) y el ancho y alto de la trama según el experimento y la imagen de las cuentas.
- Haga clic en Aceptar para guardar automáticamente el archivo de texto que contiene los datos sin procesar de la fuerza de tracción en el mismo directorio que el archivo de texto en el paso 8.1.12.
- Utilice un software de gráficos (Tabla de materiales) para trazar el campo de tracción con la misma escala para varias celdas (Figura 1B, C y Figura 2B, C).
- Inserte el archivo de texto que contiene los datos sin procesar de tracción en una hoja de cálculo.
- Cree una nueva hoja, introduzca las coordenadas Y de tracción en la primera fila (organice de valores altos a valores bajos) y las coordenadas X en la primera columna (organice de baja a alta).
- Introduzca el valor de la tracción en cada coordenada a partir de los datos sin procesar.
- Guarde la hoja en el paso 8.2.2 como un archivo *.csv.
- Origen abierto.
- Haga clic en archivo | Abra e importe el archivo *.csv en el paso 8.2.4. Seleccione todas las celdas y haga clic en Trazar | Contorno| Contorno - Relleno de color.
- En la ventana Trazado: plotvm , seleccione Y en las columnas para establecer automáticamente los valores Y en la primera fila y los valores X en la primera columna. A continuación, asigne un nombre al título y haga clic en Aceptar.
- En la ventana del gráfico que aparece, haga doble clic en el mapa de calor.
- Haga clic en Niveles en la ventana Mapa de colores/Contornos . A continuación, cambie el nivel de escala a un rango razonable (0300 en este análisis) y haga clic en Aceptar.
- Haga clic en Líneas, desmarque Mostrar solo en los niveles principales y marque Ocultar todo. A continuación, haga clic en Aceptar.
- Haga clic con el botón derecho en el gráfico y seleccione Exportar gráficos.... Guarde la imagen en la ruta especificada.
- Utilice MATLAB para calcular la tracción de la celda dipolar.
- Guarde el archivo de texto de datos sin procesar de tracción (del paso 8.1.12) y el archivo de coordenadas de la región límite de celda de interés (ROI) (del paso 7.1.9) en la misma carpeta definida en el paso 8.1.12. Transfiera todos los archivos de MATLAB que se encuentran en el paquete AMFIP a esta carpeta.
- Abra MATLAB. Abra la carpeta definida en el paso 8.1.12 y abra el archivo de función de cálculo de tracción dipolar absdipole.m transferido a esta carpeta en el paso 8.3.1.
- Lea los dos archivos de texto/csv del paso 8.3.1 en el espacio de trabajo de MATLAB y asigne una matriz a dos variables (por ejemplo, tracción y roi).
- Ejecute la función absdiple (tracción, roi)..
NOTA: La primera columna de la salida es la fuerza de tracción dipolar en nN (nano-Newton). La segunda columna de la salida es el ángulo de la fuerza de tracción dipolar con respecto al eje horizontal.
Resultados
Distribución y dinámica distintas de YAP en el cáncer PC9 diseñado por CRISPR / Cas9 y las células normales B2B durante la propagación celular
Las imágenes representativas de fluorescencia de la distribución de YAP en células B2B y PC9 individuales en geles PAA de 2, 5, 40 kPa y fundas de vidrio se muestran en la Figura 1A y la Figura 2A. La localización nuclear de YAP en células B2B aumentó con el aumento de la rigidez del sustrato (Figura 1A), mientras que las células PC9 mostraron una concentración similar de YAP en el núcleo y el citoplasma en sustratos de rigidez variable (Figura 2A). Las imágenes representativas de fluorescencia de la distribución de YAP en células B2B y PC9 individuales y PC9 en el sustrato de hidrogel de 5 kPa (desde la 0ª h hasta la 10ª h después de las células unidas a los sustratos) se muestran en la Figura 1B y la Figura 2B, respectivamente. La célula B2B aumentó monótonamente el área de propagación a lo largo del tiempo junto con una disminución en la relación YAP N / C (Figura 1B), mientras que la célula PC9 mantuvo un área de propagación celular comparativamente invariable, orientación y relación YAP N / C durante todo el proceso de propagación de 10 h (Figura 2B). Durante las 10 h de duración de la propagación temprana, la célula B2B representativa deformó constitutivamente la superficie del sustrato y aplicó tracción celular de evolución temporal en toda el área celular (Figura 1C y Figura 1D).
En contraste, la célula PC9 representativa solo desarrolló desplazamiento y tracción en los dos extremos del cuerpo celular y su tracción disminuyó después de 7.5 h (Figura 2C y Figura 2D). En la Figura Suplementaria S2 y la Figura Suplementaria S3 se proporcionan más imágenes de lapso de tiempo y mediciones de tracción de células B2B y PC9 en la etapa de propagación temprana. También se observaron otros modos de dinámica celular PC9 (Figura 6). En paralelo a estas diferentes características de propagación, las células B2B y PC9 mostraron una distribución y dinámica YAP distintas (Figura 3). En un gel de 5 kPa, YAP en las células B2B se concentró en el núcleo a la 0ª h y se distribuyó de manera más homogénea por todo el cuerpo celular a la 10ª h. Sin embargo, las células PC9 mostraron una distribución más homogénea de YAP en el núcleo y el citoplasma a lo largo de las 10 h completas del proceso de propagación. Para analizar cuantitativamente la actividad y translocación de YAP en células B2B y PC9, se calculó la relación YAP N/C utilizando el algoritmo descrito en la Figura 4.
Para investigar más a fondo la distinta dinámica de YAP, se compararon los cambios temporales en la relación YAP N/C, el área célula/núcleo y la tracción de múltiples células B2B individuales (n = 10) y células PC9 (n = 5) (Figura 5). Se encontró que la relación promedio YAP N/C de las células B2B disminuyó de 2.54 ± 0.22 a 1.79 ± 0.21 (n = 10; p = 0.0022**; Figura 5A), mientras que la relación promedio YAP N/C de las células PC9 cambió de 1.92 ± 0.26 a 1.57 ± 0.07 (n = 5; p = 0.187 (no significativo (ns)); Figura 5A). La tracción dipolar promedio de las células B2B cambió de 256,17 ± 123,69 nN a 287,44 ± 99,79 nN (p = 0,7593 (ns); Figura 5B). La tracción dipolar promedio de las células PC9 cambió de 141,19 ± 33,62 nN a 168,52 ± 73,01 nN (p = 0,7137 (ns); Figura 5B). El área de propagación celular promedio de las células B2B aumentó de 613,89 ± 102,43 μm2 a 942,51 ± 226,71 μm2 (p = 0,0512 (ns); Figura 5C).
El área de propagación celular promedio de las células PC9 cambió de 495,78 ± 97,04 μm2 a 563,95 ± 89,92 μm2 (p = 0,5804 (ns); Figura 5C). El área media de propagación del núcleo de las células B2B aumentó de 181,55 ± 36,18 μm2 a 239,38 ± 43,12 μm2 (p = 0,1217 (ns); Figura 5D) y el área media de propagación del núcleo de las células PC9 cambió de 133,31 ± 30,05 μm2 a 151,93 ± 22,49 μm2 (p = 0,5944 (ns); Figura 5D). Estos resultados sugieren que (1) las células B2B muestran una relación YAP N/C constitutivamente dependiente de la rigidez del sustrato; (2) la tracción de las células B2B es mayor que la de las células PC9; y (3) en contraste con las células B2B, las células PC9 muestran un aumento limitado en el área celular y cambios en la relación YAP N / C durante el proceso de propagación de 10 h.
Correlación de la distribución y dinámica de YAP con los estados de migración de las células B2B
Se comparó la relación YAP N/C y la tracción dipolar de todas las células B2B (n = 10) y PC9 (n = 5) en función del área de propagación celular y el área de propagación del núcleo. La relación YAP N/C y la tracción dipolar de las células PC9 no se correlacionaron claramente con sus rangos de área de propagación de células pequeñas y núcleos (Figura 6). En contraste, la relación YAP N/C y la tracción dipolar de las células B2B parecieron seguir dos tendencias distintas (Figura 6A y Figura 6C), lo que sugiere que podría haber dos grupos de células B2B que coexisten en este experimento. En el primer grupo, la relación YAP N/C y la tracción dipolar aumentan junto con la ampliación del área de propagación celular y alcanzan sus máximos a ~ 1000 μm2 (Figura 6C y Figura 6D, indicadas por la línea discontinua amarilla). En el segundo grupo, la relación YAP N/C y la tracción dipolar aumentan a un ritmo más lento con la ampliación del área de propagación celular y mantienen valores casi constantes cuando el área de propagación celular continúa aumentando (Figura 6C, D, indicada por la línea discontinua verde).
Las células cancerosas PC9 generan tracciones en las regiones perinucleares
Las células PC9 individuales y en expansión desplazan los sustratos en las regiones perinucleares, a partir de la 6ª h de cultivo (Figura 7C). Para visualizar el desplazamiento perinuclear causado por la tracción celular, superpusimos las imágenes de perlas fluorescentes tomadas antes (rojo) y después (verde) de la eliminación de las células de los sustratos (consulte la sección de protocolo para obtener más detalles). Las cuentas que no tienen ningún desplazamiento aparecerán amarillas en las imágenes superpuestas, es decir, la adición de colores rojo y verde. En contraste, las cuentas que se desplazan de sus posiciones de reposo debido a la tracción celular mostrarán colores verdes y rojos separados.
En particular, tanto en las células PC9 (Figura 7C, D) como B2B (Figura 7E), se observó desplazamiento de perlas en el citoplasma y dentro del núcleo, además de las que se encuentran en el límite celular. Para resaltar el desplazamiento perinuclear, la ecuación de Boussinesq de la teoría de la elasticidad lineal se utiliza para predecir el desplazamiento teórico 2D generado por una fuerza dipolar hipotética en el límite celular (línea discontinua negra en la Figura 7B)24. Comparando esta curva teórica con el desplazamiento real del sustrato medido a lo largo del mismo eje (línea discontinua blanca en la Figura 7D), se encontró que los desplazamientos reales dentro del núcleo eran 1.5-8 veces más grandes que el valor teórico (Figura 7B), lo que indica la existencia de fuerza de tracción en las regiones perinucleares.

Figura 1: Cambios en la expresión/distribución de YAP, campo de desplazamiento de sustrato y campo de tracción de una célula normal B2B en sustratos de rigidez variable y durante la propagación temprana. (A) La expresión de YAP de una célula B2B sembrada en geles PAA de 2, 5 y 40 kPa y una cubierta de vidrio después de 60 h desde la unión inicial de sustrato celular. (B) La célula B2B se sembró en un gel PAA de 5 kPa y se tomó una imagen durante 10 h después de la fijación inicial del sustrato celular. La expresión de YAP está representada por la intensidad de fluorescencia verde. Nota: La intensidad de YAP dentro del núcleo disminuye gradualmente, pero sigue siendo mayor que la del citoplasma con el tiempo. Las barras de color indican los niveles de expresión YAP (verde = alta expresión; negro = baja expresión) en (A) y (B). (C) La deformación del sustrato (superpuesta con la imagen de campo brillante) en la ubicación de la celda está representada por el campo de desplazamiento en cada punto de tiempo. La dirección y la magnitud del desplazamiento se muestran por la dirección de la flecha y el color, respectivamente. El desplazamiento se hace más grande en los extremos del cuerpo de las células B2B a medida que aumenta el área de propagación celular. La barra de color indica la magnitud del desplazamiento (carmesí = alta magnitud; negro = baja magnitud). (D) Campo de tracción (superpuesto con la imagen de campo brillante) calculado a partir del campo de desplazamiento. La tracción se concentra en el límite de las células B2B. Los contornos punteados blancos y amarillos delinean los límites de la célula y el núcleo, respectivamente. La barra de color indica la magnitud de la tracción (carmesí = alta magnitud; negro = baja magnitud). Barras de escala = 20 μm. Abreviaturas: YAP = proteína asociada al Sí; PAA = poliacrilamida. Haga clic aquí para ver una versión más grande de esta figura.
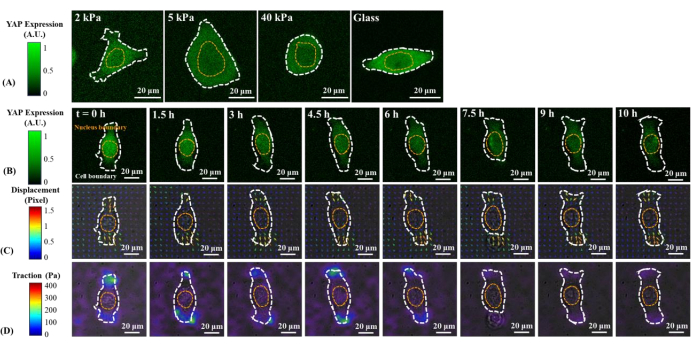
Figura 2: Cambios en la expresión/distribución de YAP, campo de desplazamiento de sustrato y campo de tracción de una célula cancerosa PC9 en sustratos de rigidez variable y durante la propagación temprana. (A) La expresión YAP de una célula PC9 sembrada en geles PAA de 2, 5 y 40 kPa y una cubierta de vidrio después de 65 h desde la fijación inicial del sustrato celular. (B) La célula PC9 se sembró en un gel PAA de 5 kPa y se tomó una imagen durante 10 h después de la fijación inicial del sustrato celular. La expresión de YAP está representada por la intensidad de fluorescencia verde. Nota: La intensidad del YAP se estabiliza a partir de 1,5 h. Las barras de color indican los niveles de expresión YAP (verde = alta expresión; negro = baja expresión) en (A) y (B). (C) La deformación del sustrato (superpuesta con la imagen de campo brillante) en la ubicación de la celda está representada por el campo de desplazamiento de perla fluorescente en cada punto de tiempo. La dirección y la magnitud del desplazamiento se muestran por la dirección de la flecha y el color, respectivamente. El campo de desplazamiento causado por las células PC9 es menor que el causado por la célula B2B. A lo largo del proceso de propagación de 10 h, el área de las células PC9 permanece casi constante. La barra de color indica la magnitud del desplazamiento (carmesí = alta magnitud; negro = baja magnitud). (D) Campo de tracción (superpuesto con la imagen de campo brillante) calculado a partir del campo de desplazamiento. La tracción generada por esta célula PC9 representativa disminuye gradualmente de la 6ª h a la 10ª h. Los contornos punteados blancos y amarillos delinean los límites de la célula y el núcleo, respectivamente. La barra de color indica la magnitud de la tracción (carmesí = alta magnitud; negro = baja magnitud). Barras de escala = 20 μm. Abreviaturas: YAP = proteína asociada al Sí; PAA = poliacrilamida. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Distribución de YAP en células B2B y PC9 en la etapa temprana de propagación. (A) La intensidad YAP de la célula B2B se mide a lo largo del eje rojo asignado en la 0ª y la 10ª h. (B) En la 0ª h, la intensidad de YAP muestra diferencias dramáticas de concentración entre el núcleo y el citoplasma. A las 10 h , la intensidad de YAP se vuelve más homogénea en todo el cuerpo celular. (C) La intensidad YAP de la célula PC9 se mide a lo largo del eje azul asignado en la 0ª y la 10ª h. (D) En la 0ª h, la intensidad de YAP en el núcleo parece mayor que la del citoplasma, aunque la diferencia no es tan notable como la de las células B2B. A las 10 h , la intensidad de YAP en el núcleo todavía aparece ligeramente superior a la del citoplasma, con una tendencia de variación similar a la de la 0ª h. Barras de escala = 20 μm (A, C). Abreviatura: YAP = Proteína asociada al Sí. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Medición de la relación YAP N/C. (1) Aplicar Fiji ImageJ para dibujar el contorno del núcleo y medir su área proyectada 2D Anuc. (2) Medir la intensidad de fluorescencia dentro del núcleo Inuc. (3) Dibuje el contorno del cuerpo celular y mida su área proyectada Acel. (4) Medir la intensidad de fluorescencia dentro de la célula Icel. (5) Calcular la densidad del núcleo YAP Dnuc, la densidad del citoplasma YAP Dcyto, y su relación R: Dnuc=Inuc/Anuc; Dcyto=(Icel-Inuc)/(Acel-Anuc); R=Dnuc/Dcyto. La barra de color indica los niveles de expresión YAP (verde = alta expresión; negro = baja expresión). Barra de escala = 20 μm. Abreviaturas: YAP = proteína asociada al Sí; N = núcleo; C = citoplasma. Haga clic aquí para ver una versión más grande de esta figura.
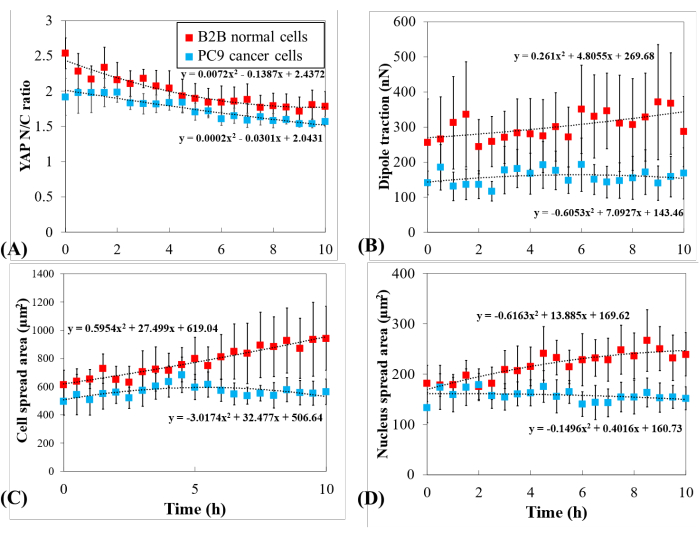
Figura 5: Expresión distinta de YAP, morfología celular/núcleo y tracción celular en el cáncer PC9 y las células normales B2B durante la propagación celular. (A) Cambio en la relación YAP N/C durante las primeras 10 h de propagación unicelular. La relación promedio YAP N/C de las células B2B (columna roja; n = 10) cambió de 2.54 ± 0.22 a 1.79 ± 0.21 (n = 10; p = 0.0022**) mientras que la relación promedio YAP N/C de las celdas PC9 (columna azul; n = 5) cambió de 1.92 ± 0.26 a 1.57 ± 0.07 (p = 0.187 (ns)). (B) La tracción dipolar promedio en función del tiempo. La tracción dipolar promedio de las células B2B cambió de 256.17 ± 123.69 nN a 287.44 ± 99.79 nN (p = 0.7593 (ns)) y la tracción dipolar promedio de las células PC9 cambió de 141.19 ± 33.62 nN a 168.52 ± 73.01 nN (p = 0.7137 (ns)). (C) El área celular promedio en función del tiempo. El área de propagación celular promedio de las células B2B aumentó de 613.89 ± 102.43 μm2 a 942.51 ± 226.71 μm2 (p = 0.0512 (ns)) y el área promedio de propagación celular de las células PC9 cambió de 495.78 ± 97.04 μm2 a 563.95 ± 89.92 μm2 (p = 0.5804 (ns)). (D) El área media del núcleo en función del tiempo. El área media de propagación del núcleo de las células B2B aumentó de 181,55 ± 36,18 μm2 a 239,38 ± 43,12 μm2 (p = 0,1217 (ns)) y el área media de propagación del núcleo de las células PC9 cambió de 133,31 ± 30,05 μm2 a 151,93 ± 22,49 μm2 (p = 0,5944 (ns)). Abreviaturas: YAP = proteína asociada al Sí; N = núcleo; C = citoplasma; ns = no significativo. Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: Relación YAP N/C y fuerza de tracción dipolar en función del área de propagación de la célula y el núcleo. La relación YAP N/C y la tracción dipolar de las células B2B (n = 10) y las células PC9 (n = 5) se calculan desde la 6ª h hasta la 10ª h después de unirse al sustrato. (A) Relación YAP N/C en función del área de propagación celular. Las proporciones YAP N/C de las células B2B varían de 1.16 a 2.53, mientras que las relaciones YAP N/C de las células PC9 varían de 1.27 a 1.88. El área de propagación celular de las células B2B varía de 391,94 μm2 a 1986,40 μm2. El área de propagación celular de las células PC9 varía de 284,46 μm2 a 830,12 μm2. (B) Relación YAP N/C en función del área de propagación del núcleo. El área de propagación del núcleo de las células B2B varía de 107,09 μm2 a 514,28 μm2. El área de propagación del núcleo de las células PC9 varía de 58,03 μm2 a 259,65 μm2. Tracción dipolar de las células B2B en función del área de propagación celular (C) y el área de propagación del núcleo (D). Las células B2B que se propagan y no migran muestran una mayor tracción (de 47,50 nN a 1051,48 nN) con un área celular y de núcleo más baja. Mientras se propagan y migran, las células B2B muestran una menor tracción (de 105.80 nN a 310.28 nN) con rangos más grandes de área celular y núcleo. Abreviaturas: YAP = proteína asociada al Sí; N = núcleo; C = citoplasma. Haga clic aquí para ver una versión más grande de esta figura.
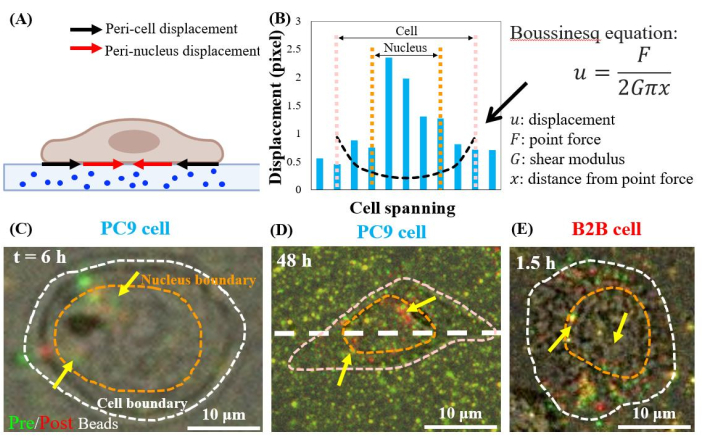
Figura 7: Desplazamiento perinuclear en células PC9 B2B normales y cancerosas. (A) Diagrama esquemático de vista lateral del desplazamiento perinuclear y pericelular medido a partir del desplazamiento de perlas en el sustrato. (B) El desplazamiento del sustrato debajo de la celda PC9 se mide a lo largo del eje de la celda (línea discontinua blanca en 7D). El desplazamiento teórico generado por la fuerza dipolar en el límite celular se muestra mediante la ecuación de Boussinesq (curva discontinua negra). (C) y (D) Imágenes de cuentas fluorescentes superpuestas con celdas (rojas) y sin (verdes) para las celdas PC9 a la 6ª h después de la conexión (vista superior). Las cuentas amarillas (superposición exacta de colores rojo y verde) indican que no hay desplazamiento. Las cuentas verdes y rojas separadas (apuntadas por flechas amarillas) representan el desplazamiento perinuclear. Las flechas amarillas indican estas manchas perinúcleo contraídas ubicadas en la periferia del núcleo. (E) Desplazamiento perinuclear generado por la célula B2B a 1,5 h después de la unión célula-sustrato. Barras de escala = 10 μm (C–E). Haga clic aquí para ver una versión más grande de esta figura.
Figura suplementaria S1: El mapa de secuencia genómica de YAP-mNeonGreen21-10/11. Haga clic aquí para descargar este archivo.
Figura suplementaria S2: Cambios en la expresión/distribución de YAP, campo de desplazamiento de sustrato y campo de tracción de células normales B2B durante la propagación temprana. (A, D, G, J, M) La célula B2B se sembró en un gel PAA de 5 kPa y se tomó una imagen durante 10 h después de la unión inicial de la célula-sustrato. La expresión de YAP está representada por la intensidad de fluorescencia verde. Nota: La intensidad de YAP dentro del núcleo disminuye gradualmente, pero permanece más alta que en el citoplasma con el tiempo. Las barras de color indican los niveles de expresión YAP (verde = alta expresión; negro = baja expresión) en (A, D, G, J, M). (B, E, H, K, N) La deformación del sustrato (superpuesta con la imagen de campo brillante) en la ubicación de la celda está representada por el campo de desplazamiento en cada punto de tiempo. La dirección y la magnitud del desplazamiento se muestran por la dirección de la flecha y el color, respectivamente. El desplazamiento se hace más grande en la periferia del cuerpo de las células B2B a medida que aumenta el área de propagación celular. Las barras de color indican la magnitud del desplazamiento (carmesí = alta magnitud; negro = baja magnitud) en (B, E, H, K, N).. (C, F, I, L, O) Campo de tracción (superpuesto con la imagen de campo brillante) calculado a partir del campo de desplazamiento mediante microscopía de fuerza de tracción. La tracción se concentra en la periferia de las células B2B. Las barras de color indican la magnitud de tracción (carmesí = alta magnitud; negro = baja magnitud) en (C, F, I, L, O).. Barras de escala = 20 μm. Abreviaturas: YAP = proteína asociada al Sí; PAA = poliacrilamida. Haga clic aquí para descargar este archivo.
Figura suplementaria S3: Cambios en la expresión/distribución de YAP, campo de desplazamiento de sustrato y campo de tracción de las células cancerosas PC9 durante la propagación temprana. (A, D, G, J) La célula PC9 se sembró en un gel PAA de 5 kPa y se tomó una imagen durante 10 h después de la unión inicial de la célula-sustrato. La expresión de YAP está representada por la intensidad de fluorescencia verde. Nota: La intensidad de YAP dentro del núcleo disminuye gradualmente, pero permanece similar o ligeramente inferior a la del citoplasma con el tiempo. Las barras de color indican los niveles de expresión YAP (verde = alta expresión; negro = baja expresión) en (A, D, G, J). (B, E, H, K) La deformación del sustrato (superpuesta con la imagen de campo brillante) en la ubicación de la celda está representada por el campo de desplazamiento en cada punto de tiempo. La dirección y la magnitud del desplazamiento se muestran por la dirección de la flecha y el color, respectivamente. El desplazamiento se hace más grande en la periferia del cuerpo celular PC9 a medida que aumenta el área de propagación celular. Las barras de color indican la magnitud del desplazamiento (carmesí = alta magnitud; negro = baja magnitud) en (B, E, H, K).. (C, F, I, L) Campo de tracción (superpuesto con la imagen de campo brillante) calculado a partir del campo de desplazamiento. La tracción se concentra en la periferia de las células PC9. Las barras de color indican la magnitud de tracción (carmesí = alta magnitud; negro = baja magnitud) en (C, F, I, L).. Barras de escala = 20 μm. Abreviaturas: YAP = proteína asociada al Sí; PAA = poliacrilamida. Haga clic aquí para descargar este archivo.
Discusión
El proceso de obtención de imágenes (paso 6.3) es fundamental para garantizar que las imágenes de fluorescencia sean de calidad suficientemente buena para obtener resultados de cuantificación válidos. Las imágenes z-stack de proteínas fluorescentes o perlas deben tener un rango z que sea lo suficientemente grande como para incluir las imágenes enfocadas para todas las posiciones Z que abarca la muestra. Otro paso crítico es recolectar las imágenes de referencia de las perlas fluorescentes después de disolver las células (paso 6.5). Debido a que las imágenes de referencia deben tomarse en las mismas posiciones en el paso 6.3, no se debe inducir ningún desplazamiento relativo entre la placa de Petri, la cámara de ambiente y el microscopio. Los investigadores que realizan el paso de disolución deben tener cuidado de quitar la tapa de la placa de Petri y asegurarse de que la perturbación mecánica aplicada no sea lo suficientemente grande como para alterar la ubicación de la placa en la cámara de ambiente.
A continuación se proporcionan soluciones para resolver algunos errores que pueden ocurrir durante los experimentos. Si no se activa ninguna macro después de hacer clic en Entrar en el paso 6.4, lo más probable es que se deba a que el área inferior izquierda de la pantalla está ocupada por una ventana que no es elemento. En tal caso, el área inferior izquierda de la ventana debe borrarse para que las macros se puedan activar en Elements. Otro error común es que las imágenes de campo brillante aparecen negras. Este problema es causado por un intervalo de tiempo insuficiente entre las adquisiciones de fluorescencia y las imágenes de campo brillante. Los ligeros retrasos en el conteo del tiempo de imágenes de fluorescencia pueden acumularse con el tiempo y causar retrasos considerables e interferir con las imágenes de campo brillante. Una solución es ajustar la duración de un ciclo de imágenes de todas las posiciones para que sea menor que (no igual a) el intervalo de tiempo entre el inicio de movimientos consecutivos. Esta operación actualiza el conteo de tiempo y elimina el error acumulativo al comienzo de cada ciclo de imágenes.
Esta tecnología de interrogación totalmente óptica admite (1) una amplia gama de hardware / software, que incluye, entre otros, Nikon, (2) diversos tipos de sistemas de hidrogel validados, incluidos geles de gelatina, PEG, Matrigel y colágeno I, y (3) la personalización programable basada en diferentes necesidades de los investigadores. Sin embargo, si alguna de las funciones de control de nivel inferior no está disponible en un microscopio comercial, la personalización de las funciones utilizando AMFIP se convierte en un desafío. Otra limitación de esta técnica es la deriva espacial de la muestra tanto en el plano XY como en el de enfoque (Z). Aunque esta limitación se puede superar durante el post-procesamiento de las imágenes, es esencial mejorar la función de autoenfoque para corregir la deriva en tiempo real de las muestras. Esta mejora aumentará el rendimiento del proceso de obtención de imágenes y reducirá el error potencial causado por la deriva durante los experimentos.
Los mecanotransductores, como el YAP, pueden servir como nuevas dianas terapéuticas para el desarrollo de prometedoras terapias contra el cáncer25,26,27. Los datos emergentes sugieren que YAP promueve la proliferación e invasión de células cancerosas. La translocación YAP inducida por la mecánica del citoplasma al núcleo activa la transcripción de genes relacionados con la migración celular, proliferación, invasión y apoptosis, dando lugar a comportamientos celulares aberrantes28,29,30,31. Este trabajo tuvo como objetivo explorar la correlación potencial de la relación YAP N / C y la mecánica celular en dos líneas celulares típicas de cáncer de pulmón humano y líneas celulares normales. Durante el período de propagación celular de 10 h, las células PC9 muestran concentraciones similares de YAP en el núcleo y el citoplasma (Figura 3D y Figura 5A). Las células B2B muestran una mayor concentración de YAP en el núcleo que en el citoplasma (Figura 3C y Figura 5A). Esta relación encontrada durante la etapa de propagación temprana es diferente de la mayoría de los hallazgos publicados que comparan la concentración de YAP en el núcleo entre las células normales y las cancerosas. Aunque no necesariamente en la etapa de propagación temprana, la mayoría de los hallazgos publicados muestran que la YAP está más concentrada en el núcleo de las células cancerosas que en el núcleo de las células normales27,28. Solo un estudio sobre el cáncer de mama reportó una excepción32 que muestra que la YAP está más concentrada en el citoplasma, lo que concuerda con nuestras observaciones actuales realizadas en células PC9 de cáncer de pulmón. Según el mejor conocimiento de los autores, este trabajo es el primero en mostrar una relación YAP N / C más baja en una línea celular de cáncer de pulmón humano. Los autores plantean la hipótesis de que la razón de una relación YAP N / C estable en las células PC9 podría deberse a la baja variación en el área de propagación de la célula / núcleo y la tracción en las células PC9 en la etapa temprana de propagación. La disección de los mecanismos moleculares subyacentes de la baja relación YAP N/C en células PC9 y B2B está en curso.
Durante las primeras 10 h de propagación, estas dos líneas celulares muestran una relación distinta entre la relación YAP N/C, la tracción celular y el área de propagación (Figura 5). Para las células B2B, una mayor relación YAP N/C se correlaciona con un área de propagación celular y núcleo más alta (Figura 6A,B), lo que es consistente con los datos reportados de otras células normales33. Curiosamente, aunque la tendencia de desarrollo de esta relación se encuentra generalmente en todas las células B2B registradas, se encuentran dos grados diferentes (alto y bajo) de esta relación. Las células B2B que se propagan y migran simultáneamente muestran una menor tracción y un área de propagación celular y núcleo más alta con una relación YAP N/C más alta (2.05 ± 0.32). Para las células B2B que se propagan y permanecen en el mismo lugar, muestran una mayor tracción y un área de propagación celular y núcleo más baja con una relación YAP N / C más baja (1.74 ± 0.21). Estos dos grados de relaciones se demuestran en los grupos de datos dispersos bifurcados (Figura 6C,D). Como se informó en la literatura, las células normales estacionarias, como las células NIH 3T3 de fibroblastos embrionarios, tienen mayor tracción que las células migratorias34. Los datos reportados en este documento sugieren que las células B2B que se propagan y no migran aplicaron una tracción más alta que las células B2B que se propagan y migran, lo que probablemente sugiere que se necesita una alta tracción para que las células no migratorias se estabilicen en el sustrato.
Además, estos datos muestran que las células B2B normales estacionarias generan una mayor fuerza perinuclear, mientras que investigaciones anteriores realizadas por otros investigadores informaron solo una mayor tracción celular generada en la periferia de las células estacionarias34,35,36,37. Los autores piensan que la diferencia en la tendencia intrínseca de la migración en los experimentos podría causar estos resultados contradictorios. En los experimentos publicados, se había utilizado el micropatronaje de forma cuadrada para confinar la propagación de células individuales e inhibir la migración; se desconoce si las células tenían la tendencia a migrar. Como las células migratorias a menudo muestran una alta fuerza de tracción en la periferia de las células38, es probable que las células con tendencia a migrar aún mantengan una alta tracción periférica a pesar de que su migración está restringida. En este estudio, las células estacionarias no están restringidas por ningún micropatrón, pero no migran, lo que indica que las células tienden a mantener su estado no migratorio. Otra posibilidad es que la forma celular definida por el micropatrones pueda afectar a la distribución de las adherencias focales y las fuerzas de tracción39. Los resultados de este estudio se generaron sin ningún micropatronaje confinante y representan la distribución de la fuerza de las células estacionarias en su forma original.
Hasta la fecha, sólo una publicación hasta la fecha informó específicamente del hallazgo de fuerzas perinucleares en células normales (fibroblastos embrionarios de ratón), potencialmente causadas por la tapa de actina que se extiende a través del núcleo40. La translocación del citoplasma al núcleo del YAP se correlaciona con el aumento de la fuerza perinuclear40. Una búsqueda exhaustiva de la literatura relevante no arrojó ninguna publicación que informe una fuerza perinuclear o la tapa de actina en las células cancerosas. Un estudio indirecto sobre células cancerosas de melanoma demostró que el borde de actina (otra organización de actina perinuclear ubicada alrededor pero no cubriendo el núcleo) reduce las tasas de migración celular41, lo que sugiere indirectamente la existencia de una fuerza perinuclear. Sin embargo, no se reportan datos experimentales directos. En este estudio, los autores encontraron que tanto las células PC9 como las B2B muestran desplazamiento y tracción perinuclear. Los mecanismos de generación de las fuerzas perinucleares y sus efectos siguen siendo controvertidos. En células normales, se ha informado que la tapa de actina desempeña un papel en la regulación de la morfología del núcleo y la organización de la cromatina42, transmitiendo señales mecánicas de adherencias focales al núcleo a través de enlazadores del complejo de nucleoesqueletos y citoesqueletos (LINC)43, y regulando la migración celular44. Lamin A/C está relacionado con la formación e interrupción de la actina cap40,41,42,43,44. Sin embargo, el informe que afirmaba que la tapa de actina genera una fuerza perinuclear no consideraba el papel potencial de la acción rim40. En las células cancerosas, la sobreexpresión de Lamin A facilita la formación de un borde de actina y restringe la migración de células cancerosas. La sobreexpresión de Lamin B reduce la formación del borde de actina y promueve la migración. La fuerza perinuclear podría estar involucrada en este proceso debido a la existencia de la organización de la actina perinuclear y el efecto de Lamin A. Sin embargo, los resultados de este estudio no mostraron ninguna evidencia de fuerzas perinucleares medidas o el comportamiento de la tapa de actina. Por lo tanto, el descubrimiento de fuerzas perinucleares en células PC9 en este estudio es el primer informe que muestra fuerzas y desplazamientos perinucleares en células de cáncer de pulmón. Los autores están investigando actualmente los mecanismos moleculares y las funciones de las fuerzas perinucleares en células PC9 y B2B diseñadas por CRISPR / Cas9.
Más allá del interrogatorio de mecanobiología totalmente óptico que se demuestra en este artículo, el sistema multifuncional integrado se puede aplicar para sondear ópticamente una miríada de otras señales fisiológicas y patobiológicas esenciales en sistemas vivos. Por ejemplo, el laboratorio de los autores ha establecido recientemente múltiples líneas celulares de cáncer humano transducidas de manera estable que coexpresan tres proteínas de membrana sensibles a la luz: indicador de voltaje de membrana QuasAr2 (excitación: 640 nm; emisión: 660 nm-740 nm), despolarizador de voltaje de membrana CheRiff (excitación: 488 nm) e hiperpolarizador de voltaje de membrana eNpHR3 (excitación: 590 nm). Estas tres proteínas funcionales pueden ser activadas por líneas láser de espectro-ortogonal de una manera libre de diafonía, lo que permite comunicaciones de señalización bidireccional totalmente ópticas (lectura y control) de electrofisiología de membrana. Utilizando un sistema optoelectrónico integrado y una pinza de parche manual, los autores han validado el control totalmente óptico y la lectura del voltaje de membrana (Vm) en células cancerosas humanas individuales y esferoides tumorales multicelulares. El interrogatorio de electrofisiología totalmente óptica abre la posibilidad de exploraciones detalladas de la bioelectricidad previamente inaccesible en las células cancerosas, lo que puede ayudar a avanzar en la biología tumoral desde un nuevo eje.
Divulgaciones
No hay conflictos de intereses que declarar.
Agradecimientos
Este proyecto cuenta con el apoyo financiero del Cancer Pilot Award de UF Health Cancer Center (X. T. y D. S.) y el Gatorade Award Start-up Package (X. T.). Los autores aprecian sinceramente las discusiones intelectuales y los apoyos técnicos del Dr. Jonathan Licht (UFHCC), el Dr. Rolf Renne (UFHCC), el Dr. Ji-Hyun Lee (Bioestadística, UF), el Dr. Hugh Fan (MAE, UF), el Dr. Warren Dixon (MAE, UF), el Dr. Ghatu Subhash (MAE, UF), el Dr. Mark Sheplak (MAE & ECE, UF), el Dr. Malisa Sarntinoranont (MAE, UF), el Dr. Scott Banks (MAE, UF), Dr. Matthew Traum (MAE, UF), Dr. David Hahn (Universidad de Arizona), Dr. Weihong Wang (Oracle Corporation), Dr. Youhua Tan (Universidad Politécnica de Hong Kong) y el Equipo de Apoyo de Nikon (Dres. José Serrano-Vélez, Larry Kordon y Jon Ekman). Los autores están profundamente agradecidos por el apoyo generoso y efectivo de todos los miembros de los laboratorios de investigación de Tang, Siemann y Guan y todos los miembros del personal de mae & ECE & Physics & Radiation Oncology Departments, UF.
Materiales
| Name | Company | Catalog Number | Comments |
| (3-Aminopropyl)triethoxysilane | Sigma-aldrich | 440140 | |
| 0.05 % Trypsin | Corning | 25-051-CI | |
| 75 cm2 flask | Corning | 430641U | |
| 8 Benchtop Centrifuge | Thermo | 75007210 | |
| A1R confocal system | Nikon | HD25 | |
| Acetic acid | Sigma-aldrich | 695092 | glacial, ACS reagent, ≥99.7% |
| BEAS-2B (B2B) cells | Sigma-aldrich | 95102433 | human epithelial cells from lung tissue |
| Carboxylate-Modified Microspheres | Invitrogen | F8797 | |
| Culture medium (RPMI-1640) | Gibco | 11875093 | |
| Desktop Computer | Dell | 2018 | with Windows 10 operating system |
| Environmental chamber TIZB | Tokai Hit | TIZB | |
| Fetal bovine serum (FBS) | Gibco | 26140 | |
| Fibronectin Human Protein, Plasma | Gibco | 33016015 | |
| Fiji ImageJ | National Institutes of Health and the Laboratory for Optical and Computational Instrumentation | 1.53k | |
| Glass-bottom petri dish | MatTek | P35G-1.5-14-C | |
| HEPES buffered saline | Sigma-aldrich | 51558 | |
| Hydrazine hydrate solution | Sigma-aldrich | 53847 | |
| IntelliJ IDEA | JetBrains | 2020 | Java development platform |
| Java Development Kit | Oracle | 14.0 | |
| Kimwipe | Kimtech Science | 3066-05 | |
| MATLAB | MathWorks | 2020b | |
| Monochrome Camera | FLIR | BFS-U3-70S7M-C | |
| MycoAlert Mycoplasma Detection Kit | Lonza | LT07-218 | |
| N,N′-Methylenebisacrylamide solution | Sigma-aldrich | M1533 | |
| NIS-Elements software platform | Nikon | 4.50 | software platform |
| Origin | OriginLab | OriginPro 2017 (Learning Edition) | data analysis and graphing software |
| Penicillin-streptomycin | Gibco | 15140122 | |
| PC9 cells | Sigma-aldrich | 90071810 | human adenocarcinoma cells from lung tissue |
| Phosphate buffered saline (PBS) | Gibco | 10010023 | |
| Phusion High-Fidelity DNA Polymerase | New England Biolabs | F-553S | high-fidelity DNA polymerase |
| Scotch tape | Scotch | adhesive tape | |
| Sodium dodecyl sulfate solution | Sigma-aldrich | 05030 | |
| Super glue | Gorilla | cyanoacrylate glue | |
| Ti2-E inverted microscope | Nikon | MEA54000 | |
| TI2-S-SE-E Motorized Stage with Encoder | Nikon | MEC56120 | |
| μManager | version 2.0 gamma | open source microscopy software (https://micro-manager.org/) |
Referencias
- Werley, C., Boccardo, S., Rigamonti, A., Hansson, E., Cohen, A. Multiplexed optical sensors in arrayed islands of cells for multimodal recordings of cellular physiology. Nature Communications. 11 (1), 3881 (2020).
- Yang, B., et al. Epi-illumination SPIM for volumetric imaging with high spatial-temporal resolution. Nature Methods. 16 (6), 501-504 (2019).
- Saraswathibhatla, A., Galles, E. E., Notbohm, J. Spatiotemporal force and motion in collective cell migration. Scientific Data. 7 (1), 197 (2020).
- Saraswathibhatla, A., Henkes, S., Galles, E. E., Sknepnek, R., Notbohm, J. Coordinated tractions control the size of a collectively moving pack in a cell monolayer. Extreme Mechanics Letters. 48, 101438 (2021).
- Wang, W., Kim, C. K., Ting, A. Y. Molecular tools for imaging and recording neuronal activity. Nature Chemical Biology. 15 (2), 101-110 (2019).
- Eliceiri, K. W., et al. Biological imaging software tools. Nature Methods. 9 (7), 697-710 (2012).
- Carpenter, A. E., Kamentsky, L., Eliceiri, K. W. A call for bioimaging software usability. Nature Methods. 9 (7), 666-670 (2012).
- Skylaki, S., Hilsenbeck, O., Schroeder, T. Challenges in long-term imaging and quantification of single-cell dynamics. Nature Biotechnology. 34 (11), 1137-1144 (2016).
- Lin, M. Z., Schnitzer, M. J. Genetically encoded indicators of neuronal activity. Nature Neuroscience. 19 (9), 1142-1153 (2016).
- Luo, Q., et al. Automatic multi-functional integration program (AMFIP) towards all-optical mechanobiology interrogation. bioRxiv. , (2021).
- Edelstein, A., Amodaj, N., Hoover, K., Vale, R., Stuurman, N. Computer control of microscopes using manager. Current Protocols in Molecular Biology. 92 (1), 14-20 (2010).
- Tulpule, A., et al. Kinase-mediated RAS signaling via membraneless cytoplasmic protein granules. Cell. 184 (10), 2649-2664 (2021).
- Tang, X., Tofangchi, A., Anand, S. V., Saif, T. A. A novel cell traction force microscopy to study multi-cellular system. PLOS Computational Biology. 10 (6), 1003631 (2014).
- Tang, X., et al. Mechanical force affects expression of an in vitro metastasis-like phenotype in HCT-8 cells. Biophysical Journal. 99 (8), 2460-2469 (2010).
- Guimarães, C. F., Gasperini, L., Marques, A. P., Reis, R. L. The stiffness of living tissues and its implications for tissue engineering. Nature Reviews Materials. 5, 351-370 (2020).
- Phelps, E. A., et al. Maleimide cross-linked bioactive PEG hydrogel exhibits improved reaction kinetics and cross-linking for cell encapsulation and in situ delivery. Advanced Materials. 24 (1), 64-70 (2012).
- Bajaj, P., Tang, X., Saif, T. A., Bashir, R. Stiffness of the substrate influences the phenotype of embryonic chicken cardiac myocytes. Journal of Biomedical Materials Research. Part A. 95 (4), 1261-1269 (2010).
- Temples, M. N., Adjei, I. M., Nimocks, P. M., Djeu, J., Sharma, B. Engineered three-dimensional tumor models to study natural killer cell suppression. ACS Biomaterials Science & Engineering. 6 (7), 4179-4199 (2020).
- Feng, S., et al. Improved split fluorescent proteins for endogenous protein labeling. Nature Communications. 8, 370 (2017).
- Guan, J., Liu, H., Shi, X., Feng, S., Huang, B. Tracking multiple genomic elements using correlative CRISPR imaging and sequential DNA FISH. Biophysical Journal. 112 (6), 1077-1084 (2017).
- . Micro-Manager Available from: https://micro-manager.org/wiki/NikonTi2 (2021)
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Martiel, J. L., et al. Measurement of cell traction forces with ImageJ. Methods in Cell Biology. 125, 269-287 (2015).
- Okumurai, I. A. On the generalization of Cerruti's problem in an elastic half-space. Doboku Gakkai Ronbunshu. 1995, 1-10 (1995).
- Piccolo, S., Dupont, S., Cordenonsi, M. The biology of YAP/TAZ: hippo signaling and beyond. Physiological Reviews. 94 (4), 1287-1312 (2014).
- Hong, W. W., Guan, K. L. The YAP and TAZ transcription co-activators: Key downstream effectors of the mammalian Hippo pathway. Seminars in Cell and Developmental Biology. 23 (7), 785-793 (2012).
- Zanconato, F., Cordenonsi, M., Piccolo, S. YAP/TAZ at the roots of cancer. Cancer Cell. 29 (6), 783-803 (2016).
- Wang, Y., et al. Overexpression of yes-associated protein contributes to progression and poor prognosis of non-small-cell lung cancer. Cancer Science. 101 (5), 1279-1285 (2010).
- Li, H., et al. Inhibition of YAP suppresses CML cell proliferation and enhances efficacy of imatinib in vitro and in vivo. Journal of Experimental & Clinical Cancer Research. 35 (1), 134 (2016).
- Tang, X., et al. A mechanically-induced colon cancer cell population shows increased metastatic potential. Molecular Cancer. 13, 131 (2014).
- Panciera, T., Azzolin, L., Cordenonsi, M., Piccolo, S. Mechanobiology of YAP and TAZ in physiology and disease. Nature Reviews Molecular Cell Biology. 18 (12), 758-770 (2017).
- Yuan, M., et al. Yes-associated protein (YAP) functions as a tumor suppressor in breast. Cell Death and Differentiation. 15 (11), 1752-1759 (2008).
- Koushki, N., et al. Lamin A redistribution mediated by nuclear deformation determines dynamic localization of YAP. bioRxiv. , (2020).
- Chang, S. S., Rape, A. D., Wong, S. A., Guo, W. H., Wang, Y. L. Migration regulates cellular mechanical states. Molecular Biology of the Cell. 30 (26), 3104-3111 (2019).
- Lee, J., Abdeen, A. A., Tang, X., Saif, T. A., Kilian, K. A. Geometric guidance of integrin mediated traction stress during stem cell differentiation. Biomaterials. 69, 174-183 (2015).
- Lee, J., Abdeen, A., Tang, X., Saif, T. A., Kilian, K. A. Matrix directed adipogenesis and neurogenesis of mesenchymal stem cells derived from adipose tissue and bone marrow. Acta Biomaterialia. 42, 46-55 (2016).
- Tang, X., Bajaj, P., Bashir, R., Saif, T. A. How far cardiac cells can see each other mechanically. Soft Matter. 7 (13), 6151-6158 (2011).
- Dembo, M., Wang, Y. L. Stresses at the cell-to-substrate interface during locomotion of fibroblasts. Biophysical Journal. 76 (4), 2307-2316 (1999).
- Rape, A., Guo, W. H., Wang, Y. L. The regulation of traction force in relation to cell shape and focal adhesions. Biomaterials. 32 (8), 2043-2051 (2011).
- Shiu, J. Y., Aires, L., Lin, Z., Vogel, V. Nanopillar force measurements reveal actin-cap-mediated YAP mechanotransduction. Nature Cell Biology. 20 (3), 262-271 (2018).
- Fracchia, A., Asraf, T., Salmon-Divon, M., Gerlitz, G. Increased lamin B1 levels promote cell migration by altering perinuclear actin organization. Cells. 9 (10), 2161 (2020).
- Ramdas, N. M., Shivashankar, G. V. Cytoskeletal control of nuclear morphology and chromatin o1rganization. Journal of Molecular Biology. 427 (3), 695-706 (2015).
- Khatau, S. B., et al. A perinuclear actin cap regulates nuclear shape. Proceedings of the National Academy of Sciences of the United States of America. 106 (45), 19017-19022 (2009).
- Kim, D. H., Cho, S., Wirtz, D. Tight coupling between nucleus and cell migration through the perinuclear actin cap. Journal of Cell Science. 127 (11), 2528-2541 (2014).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados