Method Article
Seguimiento de partículas sensibles a la masa para caracterizar la dinámica de macromoléculas asociada a la membrana
En este artículo
Resumen
Este protocolo describe un enfoque de procesamiento de imágenes basado en iSCAT y seguimiento de una sola partícula que permite la investigación simultánea de la masa molecular y el comportamiento difusivo de las macromoléculas que interactúan con las membranas lipídicas. Las instrucciones paso a paso para la preparación de muestras, la conversión de masa a contraste, la adquisición de películas y el posprocesamiento se proporcionan junto con instrucciones para prevenir posibles trampas.
Resumen
Las interacciones de corta duración o transitorias de macromoléculas en y con membranas lipídicas, una interfaz donde tienen lugar una multitud de reacciones biológicas esenciales, son inherentemente difíciles de evaluar con métodos biofísicos estándar. La introducción del seguimiento de partículas sensibles a la masa (MSPT) constituye un paso importante hacia una caracterización cuantitativa exhaustiva de dichos procesos. Técnicamente, esto fue posible gracias al advenimiento de la fotometría de masas (MP) basada en microscopía de dispersión interferométrica (iSCAT). Cuando la estrategia de eliminación de fondo se optimiza para revelar el movimiento bidimensional de las partículas asociadas a la membrana, esta técnica permite el análisis en tiempo real tanto de la difusión como de la masa molecular de macromoléculas no etiquetadas en membranas biológicas. Aquí, se describe un protocolo detallado para realizar y analizar el seguimiento de partículas sensibles a la masa de los sistemas asociados a la membrana. Las mediciones realizadas en un fotómetro de masas comercial alcanzan una resolución de tiempo en el régimen de milisegundos y, dependiendo del sistema MP, un límite de detección de masa de hasta 50 kDa. Para mostrar el potencial de MSPT para el análisis en profundidad de la dinámica de macromoléculas catalizadas por membrana en general, se presentan los resultados obtenidos para sistemas proteicos ejemplares como el interactor de membrana nativo anexina V.
Introducción
Una vez percibidas simplemente como una barrera contra la amplia gama de condiciones físicas ambientales, las membranas biológicas se consideran hoy en día entidades funcionales y plataformas catalíticas 1,2. Sobre la base de su capacidad para localizar, amplificar y dirigir señales en respuesta a reacciones de macromoléculas asociadas a la membrana, las interfaces lipídicas constituyen un elemento crucial para una amplia variedad de procesos celulares, como el tráfico de membranas y las cascadas de señalización 3,4,5. Sirviendo como un sitio de nucleación para el ensamblaje de complejos estables, la unión de membrana a menudo se basa en un equilibrio dinámico entre las formas de macromoléculas asociadas a la membrana y citosólicas y, por lo tanto, es de naturaleza transitoria 6,7.
A pesar de su gran importancia en biología, hasta ahora ha sido difícil desarrollar métodos que puedan proporcionar acceso a las heterogeneidades compositivas, espaciales y temporales de las reacciones de macromoléculas asociadas a la membrana en tiempo real 7,8. Para resolver los procesos moleculares subyacentes, dos aspectos experimentales son decisivos: la resolución de tiempo suficiente y la sensibilidad de una sola partícula. Por lo tanto, las técnicas de promedio de conjunto, como la recuperación de fluorescencia después del fotoblanqueo (FRAP), pero también la espectroscopia de correlación de fluorescencia (FCS), mucho más sensible, tienen limitaciones, ya que desacoplan en gran medida la información espacial de la información temporal9. Por lo tanto, un paso importante hacia la caracterización de la dinámica de moléculas individuales ha sido el advenimiento del seguimiento de partículas individuales (SPT) en combinación con microscopía altamente sensible. En particular, dos enfoques del Subcomité han demostrado ser eficaces a este respecto. En primer lugar, la utilización de fluoróforos como etiquetas y los correspondientes sistemas de detección de fluorescencia allanaron el camino para la precisión nanométrica y la resolución de tiempo de milisegundos 10,11,12. En segundo lugar, la detección basada en dispersión utilizando nanopartículas de oro mejoró tanto la precisión de localización como la resolución de tiempo en el rango de subnanométricos y microsegundos, respectivamente 13,14,15,16. A pesar de las muchas ventajas de ambos enfoques y sus contribuciones significativas con respecto a la comprensión mecanicista de los sistemas asociados a la membrana 17,18, ambas técnicas han sido hasta ahora limitadas: requieren el etiquetado de las moléculas de interés, lo que potencialmente perturba su comportamiento nativo y son insensibles a la composición molecular de las partículas asociadas a la membrana 19,20.
Ambas limitaciones han sido superadas recientemente por la introducción de un nuevo enfoque basado en la dispersión interferométrica (iSCAT) denominado fotometría de masas (MP)21,22,23. Esta técnica permite la determinación de distribuciones de masa en solución de biomoléculas según su contraste iSCAT al aterrizar en una interfaz de vidrio. Sin embargo, para la detección y caracterización de moléculas móviles que se difunden en membranas lipídicas, se tuvo que desarrollar un enfoque de análisis de imágenes más sofisticado. Mientras tanto, esto se ha implementado con éxito y permite detectar, rastrear y determinar la masa molecular de biomoléculas individuales no etiquetadas que se difunden en una interfaz lipídica24,25. Conocida como fotometría de masas dinámica o seguimiento de partículas sensibles a la masa (MSPT), esta técnica ahora permite la evaluación de interacciones complejas de macromoléculas al registrar directamente los cambios en la masa molecular de las entidades rastreadas y, por lo tanto, abre nuevas posibilidades para el análisis mecanicista de la dinámica molecular asociada a la membrana.
Aquí, se presenta un protocolo detallado para la preparación de muestras, imágenes y la canalización de análisis de datos requerida para MSPT. En particular, se discuten los requisitos de la muestra y los posibles problemas que pueden ocurrir durante la medición y el análisis. Además, el potencial sin precedentes para analizar sistemas de macromoléculas que interactúan con la membrana se muestra a través de varios resultados representativos.
Protocolo
1. Preparación de la muestra
- Generación de vesículas multilamelares (MLV)
- Calcule la cantidad de lípidos disueltos por cloroformo de acuerdo con la mezcla de lípidos deseada y el volumen de suspensión requerido.
NOTA: Se recomienda una concentración final de vesículas de 4 mg/ml de lípidos para el tampón de resuspensión (reacción). - Pipetear el volumen calculado de lípidos en un vial de vidrio de 1,5 ml utilizando pipetas de desplazamiento positivo equipadas con puntas de vidrio.
- Evapore el disolvente lipídico bajo una tenue corriente de nitrógeno y gire constantemente el vial para garantizar una distribución equitativa de los lípidos en las paredes de vidrio.
- Asegúrese de la evaporación completa del disolvente colocando el vial bajo un flujo constante de nitrógeno durante 15 minutos.
- Elimine los rastros residuales de cloroformo mediante secado al vacío en un desecador al vacío durante una hora adicional.
- Rehidrate la mezcla lipídica en el tampón de resuspensión (reacción) deseado y vórtice completamente la suspensión hasta que la película lipídica se haya disuelto de las paredes del vial.
NOTA: El tampón de reacción debe garantizar la actividad y la estabilidad de la proteína. El tampón de reacción utilizado en este estudio contiene 50 mM Tris-HCl (pH = 7.5), 150 mM KCl y 5 mM MgCl2. Tenga en cuenta que cualquier tampón utilizado para diluir lípidos o proteínas debe filtrarse para eliminar las impurezas de partículas interferentes (consulte el paso 5).
- Calcule la cantidad de lípidos disueltos por cloroformo de acuerdo con la mezcla de lípidos deseada y el volumen de suspensión requerido.
- Generación de pequeñas vesículas unilamelares (SUV)
- Para ciclos consecutivos de congelación-descongelación de la resuspensión lipídica (paso 1.1.6), hervir 500 ml de agua en un vaso de precipitados en una placa caliente (entre 70 °C y 99 °C) y preparar un recipiente con nitrógeno líquido.
- Congelación por choque de la resuspensión lipídica en el nitrógeno líquido. Transfiera el vial al vaso de precipitados con agua caliente hasta que la solución se descongele por completo. Repita este ciclo de congelación-descongelación 8-10 veces o hasta que la mezcla previamente turbia parezca clara.
PRECAUCIÓN: Use prendas y equipos de seguridad adecuados, como gafas, guantes y pinzas para evitar cualquier contacto directo con el nitrógeno líquido, el vial de lípidos congelados o el agua hirviendo. - Para la generación de una distribución de vesícula monodispersa, ensamble un extrusor lipídico y pruebe su integridad con tampón de reacción para asegurarse de que no tenga fugas.
NOTA: Si se observa una fuga, vuelva a ensamblar cuidadosamente el extrusor de lípidos hasta que no se eviden derrames de tampón. - Extruir la suspensión lipídica durante 37 pasa a través de una membrana nucleoporora con un tamaño de poro de 50 nm26. El número de pasadas debe ser desigual para garantizar que la mezcla final de SUV atraviese la membrana de nucleoporas y, por lo tanto, esté libre de agregados lipídicos o vesículas multilamelares. Las vesículas extruidas se utilizarán más adelante para formar bicapas lipídicas soportadas (ver pasos 6 y 7).
NOTA: Los SUV también pueden formarse por sonicación de la mezcla de lípidos rehidratados. Sin embargo, la preparación a través de la extrusión proporciona una distribución más monodispersa de los SUV, lo que facilita la ruptura de la vesícula durante la formación de la bicapa lipídica soportada. Las vesículas extruidas se pueden almacenar en el refrigerador durante un máximo de 3 días.
2. Limpieza de portaobjetos de microscopio
- Distribuya un número igual de portaobjetos de microscopio (No. #1.5; 0.17 mm de espesor) con dimensiones de 24 mm x 60 mm y 24 mm x 24 mm en soportes de microscopio de politetrafluoretileno (PTFE).
- Transfiera los soportes de PTFE a vasos de precipitados que contengan agua ultrapura y sonicarlos durante 15 minutos a temperatura ambiente.
NOTA: Dependiendo del vaso de precipitados, el volumen de agua debe ajustarse para cubrir completamente el soporte de PTFE. - Use pinzas para quitar los soportes del vaso de precipitados y reemplace el agua con isopropanol ultrapuro. Inserte el soporte en el vaso de precipitados que contiene isopropanol y sonicato nuevamente durante 15 minutos.
NOTA: Dependiendo del vaso de precipitados, el volumen del isopropanol debe ajustarse para cubrir completamente el soporte de PTFE. - Reemplace el isopropanol con agua ultrapura y sonice el vaso de precipitados que contiene los soportes durante 15 min.
- Retire los soportes de PTFE de los vasos de precipitados y seque los portaobjetos del microscopio en el soporte bajo un flujo constante de gas nitrógeno o aire comprimido.
NOTA: Asegúrese de una limpieza adecuada de los portaobjetos de la cubierta mediante el uso de guantes, vasos de precipitados limpios y película de parafina para cubrir cada vaso de precipitados. De lo contrario, el polvo residual podría causar fluctuaciones significativas en el fondo durante las mediciones de MSPT.
3. Hidrofilización de portaobjetos de microscopio
NOTA: Para obtener una bicapa lipídica homogénea y fluida, la hidrofilización de los portaobjetos es esencial y debe llevarse a cabo justo antes del montaje de la cámara de flujo.
- Coloque los soportes de PTFE que contengan solo portaobjetos de microscopio de 24 mm x 60 mm en un limpiador de plasma con oxígeno como gas de proceso y limpie los portaobjetos de microscopio con plasma (parámetros utilizados en este trabajo: 30% de potencia, presión de oxígeno de 0,3 mbar durante 30 s; consulte la Tabla de materiales para obtener detalles del limpiador de plasma utilizado).
NOTA: Para obtener membranas fluidas, los parámetros de limpieza por plasma, como la potencia, la presión de oxígeno y el tiempo de limpieza, deben ajustarse para cada instrumento. Para ello, se recomienda el uso de lípidos marcados fluorescentemente para asegurar la fluidez de la membrana, que puede cuantificarse con la recuperación de fluorescencia después de experimentos de fotoblanqueo (FRAP)27. Si los parámetros no están optimizados para la configuración respectiva, la difusión de la membrana podría verse afectada debido a la reducción de la fluidez de la membrana.
4. Montaje de cámaras de flujo
- Antes del montaje de la cámara de flujo, mantenga listos los siguientes componentes: portaobjetos de microscopio limpios (24 mm x 24 mm), portaobjetos de microscopio hidrofilizados (24 mm x 60 mm), papel de aluminio, cartón plano, bisturí y cinta de doble cara.
- Envuelva el cartón plano con papel de aluminio.
- Extienda los portaobjetos de microscopio limpios de 24 mm x 24 mm sobre el papel de aluminio con suficiente distancia entre sí.
- Coloque tiras de cinta de doble cara en los bordes superior e inferior de las diapositivas.
- Elimine cada portaobjetos de microscopio con el bisturí, de modo que se pueda quitar del papel de aluminio. Como resultado, cada diapositiva debe tener rayas de cinta adhesiva de doble cara unidas a los bordes superior e inferior de la diapositiva (consulte la Figura 1).
- Conecte la corredera de 24 mm x 24 mm con las dos tiras de cinta de doble cara a la corredera hidrofilizada de 24 mm x 60 mm para formar una trayectoria de flujo entre las diapositivas de microscopio más pequeñas y más grandes.
NOTA: Para garantizar cámaras de flujo limpias, use constantemente guantes y asegúrese de que el banco de trabajo esté libre de polvo.
5. Filtración de tampones de reacción
- Filtre estéril todos los tampones de reacción a través de membranas de acetato de celulosa de 0,45 μm para garantizar una señal de fondo mínima durante las mediciones de MSPT.
NOTA: Si la presencia de nucleótidos, como el ATP, es esencial para un experimento exitoso, tenga en cuenta un posible aumento en la señal de fondo. Se recomienda usar solo cantidades mínimas que aún aseguren la actividad de las proteínas.
6. Formación de bicapa lipídica (SLB) soportada
NOTA: Se recomienda realizar la formación de bicapas lipídicas soportadas en el fotómetro de masas para garantizar visualmente la propagación exitosa de la vesícula y la eliminación completa de las vesículas no fusionadas.
- Diluya los SUV recién extruidos (consulte el paso 1 para obtener más detalles) a una concentración final de 0,4 mg/ ml en el tampón de reacción requerido. Opcionalmente, para promover la ruptura de la vesícula, agregue 2 mM CaCl2 a la suspensión de la vesícula.
NOTA: Los cationes divalentes pueden causar la agregación de algunos lípidos como PiP2. Para las mezclas que contienen tales lípidos, absténgase de usar CaCl2 para la promoción de la ruptura de la vesícula u otros cationes divalentes en el tampón de resuspensión. Si es necesario para el experimento, se pueden agregar cationes divalentes después de la formación exitosa de la bicapa lipídica soportada. - Enjuague 50 μL de la suspensión de vesículas en la cámara de flujo (paso 4) e incube la cámara durante 2 min.
NOTA: Los tampones, vesículas o soluciones proteicas se pueden enjuagar a través de la cámara de flujo con un pequeño trozo de tejido en remojo. Sin embargo, también es posible utilizar un sistema de bomba mecánica. - Retire las vesículas no fusionadas mediante el lavado repetido (al menos tres veces) de la cámara de flujo con 200 μL del tampón de reacción cada vez.
NOTA: Las vesículas deben lavarse a fondo de la cámara de flujo para garantizar una señal de fondo estable durante las mediciones de MSPT.
7. Generación de curva de calibración
NOTA: Para convertir el contraste de las partículas detectadas en masa molecular, su señal debe calibrarse utilizando proteínas de tamaños conocidos. Se recomienda ajustar el régimen estándar de tamaño de proteína para cubrir el rango de masas moleculares esperadas para el sistema de interés.
- Biotinilación de proteínas estándar con un residuo de cisteína
- Calcule la cantidad adecuada de maleimida-biotina para la proteína estándar de acuerdo con las instrucciones del fabricante.
- Incubar la proteína estándar con el volumen determinado de maleimida-biotina durante 1 h a temperatura ambiente.
- Para eliminar la maleimida-biotina no conjugada del complejo conjugado biotina-proteína, realice una cromatografía de exclusión de tamaño en una columna adecuada para la proteína de interés.
- Determine la concentración de proteína utilizando un ensayo de Bradford.
NOTA: Para almacenar la proteína estándar para mediciones adicionales, congele la proteína en alícuotas de un solo uso en nitrógeno líquido y guárdelas a -80 ° C.
- Medición de proteínas estándar para la curva de calibración
- En una cámara de flujo, prepare una bicapa lipídica soportada con 0,4 mg/ml de SUV extruidos (consulte los pasos 1 y 6 para obtener más detalles) que contenga 0,01 mol% (v/v) biotinyl Cap PE (1,2-dioleoyl-sn-glycero-3-phosphoethanolamine-N-cap biotinyl).
- Añadir 50 μL de estreptavidina divalente de 2,5 nM a la bicapa de la cámara de flujo e incubar durante 10 min.
NOTA: La estreptavidina divalente ha sido expresada y purificada como se describe en Howarth et al.28. También se puede utilizar estreptavidina tetravalente. Sin embargo, el uso de estreptavidina divalente puede reducir las posibles estequiometrías de reacción entre los lípidos biotinilados y las proteínas estándar que se conjugan a una fracción de biotina para facilitar la asignación de especies. - Eliminar la estreptavidina divalente no unida con 100 μL de tampón de reacción.
- Añadir 50 μL de proteína estándar conjugada con biotina de 100 nM a la bicapa de la cámara de flujo e incubar durante 2 min.
NOTA: Dependiendo de la eficiencia de biotinilación y si se utiliza estreptavidina divalente o tetravalente, las concentraciones óptimas de proteína estándar conjugada con biotina y estreptavidina pueden variar. - Realice la medición de MSPT de acuerdo con los detalles descritos en el paso 8.
PRECAUCIÓN: Las condiciones de imagen deben ser idénticas tanto para la muestra como para los estándares de calibración.
8. Imágenes
- Formación de SLB y preparación de muestras
- Como se describe con más detalle en el paso 6, introduzca LOS SUV de la mezcla lipídica deseada (25 μL) en la cámara de flujo de muestra y forme una bicapa lipídica soportada. Lave bien la cámara (tres veces) con 100 μL de tampón de reacción para eliminar todas las vesículas no fusionadas.
- Añadir 50 μL de la proteína de interés a la cámara de muestra.
NOTA: Como MSPT es un método de una sola partícula, la concentración de proteínas debe mantenerse en el rango de pM a nM para permitir la detección y el seguimiento de partículas sin perturbaciones.
- Adquisición de vídeo
- Establezca las condiciones de imagen deseadas, como el tamaño del campo de visión (FOV), la velocidad de fotogramas, el tiempo de exposición y el tiempo de adquisición en el software de adquisición.
NOTA: Las siguientes configuraciones han demostrado funcionar para MSPT en un fotómetro de masas comercial (consulte la Tabla de materiales): FOV de 128 píxeles x 35 píxeles, una velocidad de fotogramas de 1 kHz que resulta en aproximadamente 200 cuadros por segundo después de un promedio posterior de fotogramas de 5 veces y un tiempo de exposición de 0,95 ms. - Ajuste el enfoque de forma automática o manual. Si es necesario, mueva el campo de visión a una posición con una membrana homogénea utilizando el control lateral.
- Cree una carpeta de proyecto y comience a grabar la película. Al finalizar la grabación, especifique un nombre de archivo en el cuadro de diálogo solicitado por el software de adquisición. La película se guarda automáticamente en la carpeta del proyecto como un archivo MP para su posterior análisis.
NOTA: Registre al menos tres réplicas en diferentes cámaras de flujo para garantizar la integridad de las membranas individuales y la reproducibilidad de los resultados. La duración de la película se puede establecer de antemano y depende del tipo de experimento. En la mayoría de los casos, se recomienda un tiempo de adquisición entre 5 min y 7 min.
PRECAUCIÓN: De forma predeterminada, las grabaciones de película en el software de adquisición de fotómetros de masas comerciales se comprimen antes de guardarse para reducir el espacio de almacenamiento. Sin embargo, la compresión de archivos debe desactivarse para habilitar el análisis de datos personalizado como se describe en este protocolo. Los detalles sobre cómo desactivar la compresión de archivos se pueden encontrar en el manual del usuario del fabricante.
- Establezca las condiciones de imagen deseadas, como el tamaño del campo de visión (FOV), la velocidad de fotogramas, el tiempo de exposición y el tiempo de adquisición en el software de adquisición.
9. Análisis de datos
NOTA: La canalización de análisis de datos va acompañada de dos blocs de notas interactivos de Jupyter (MSPT analysis.ipynb, Movie visualization.ipynb). Los blocs de notas de Jupyter y los módulos de Python personalizados asociados necesarios para realizar el análisis de MSPT que se describe a continuación están disponibles en un repositorio público: https://github.com/MSPT-toolkit/MSPT-toolkit. Para obtener instrucciones detalladas sobre el análisis a continuación, se remite a los lectores a MSPT analysis.ipynb al que se accede utilizando el enlace anterior.
- Procesamiento de vídeo
- Elimine la dispersión estática dominante de la luz con el algoritmo de estimación de fondo en píxeles utilizando la función image_processing.mp_reader .
- Para aplicar la eliminación de fondo, elija la opción continuous_median para el modo de parámetro y establezca una longitud adecuada para la ventana mediana deslizante (window_length) en la sección B.1 del bloc de notas. Opcionalmente, guarde las películas después de la eliminación del fondo para usarlas para la detección de partículas y la vinculación de trayectoria (estableciendo el parámetro save_processed_movies en True).
NOTA: Ajuste el tamaño de la ventana (window_length) a valores entre 101 y 2001 dependiendo de la densidad de partículas en la membrana, el coeficiente de difusión esperado, la velocidad de fotogramas de adquisición y la velocidad de procesamiento requerida.
PRECAUCIÓN: La estrategia de eliminación de fondo funciona bien si la membrana no está demasiado densamente empaquetada y si la difusión de las partículas es lo suficientemente rápida (es decir, cada píxel está la mayor parte del tiempo no ocupado con una partícula). De lo contrario, el contraste de las partículas se subestimará sistemáticamente, ya que no se pueden distinguir adecuadamente de la señal de fondo. Esto se puede compensar aumentando el tamaño medio de la ventana a costa de la velocidad computacional. Sin embargo, tenga en cuenta que establecer el tamaño de la ventana demasiado grande puede influir negativamente en la salida debido a la deriva de la muestra. Una inspección visual de los videos procesados es crucial.
- Para aplicar la eliminación de fondo, elija la opción continuous_median para el modo de parámetro y establezca una longitud adecuada para la ventana mediana deslizante (window_length) en la sección B.1 del bloc de notas. Opcionalmente, guarde las películas después de la eliminación del fondo para usarlas para la detección de partículas y la vinculación de trayectoria (estableciendo el parámetro save_processed_movies en True).
- Detecte partículas y su posición respectiva a lo largo de la película utilizando la función particle_fitting.particle_fitter (véase la sección B.2 del cuaderno).
- Ajuste la sensibilidad de la detección de partículas con el parámetro umbral (trilla; consulte la sección B.1 del cuaderno), que se utiliza para resaltar los puntos candidatos mediante la binarización de imágenes. El efecto de la variación de los parámetros de umbral en la sensibilidad de detección puntual se puede examinar en un bloc de notas independiente (Movie visualization.ipynb). Los resultados de la detección de partículas se guardan automáticamente en archivos CSV en un subdirectorio del archivo de película.
NOTA: No se recomienda establecer el parámetro de umbral arbitrariamente bajo (por ejemplo, para películas tomadas con el fotómetro de masa utilizado, un parámetro de umbral por debajo de 0.0005) ya que los puntos candidatos estarán dominados por ruido espurio y, por lo tanto, prolongarán el tiempo de procesamiento.
- Ajuste la sensibilidad de la detección de partículas con el parámetro umbral (trilla; consulte la sección B.1 del cuaderno), que se utiliza para resaltar los puntos candidatos mediante la binarización de imágenes. El efecto de la variación de los parámetros de umbral en la sensibilidad de detección puntual se puede examinar en un bloc de notas independiente (Movie visualization.ipynb). Los resultados de la detección de partículas se guardan automáticamente en archivos CSV en un subdirectorio del archivo de película.
- Elimine la dispersión estática dominante de la luz con el algoritmo de estimación de fondo en píxeles utilizando la función image_processing.mp_reader .
- Vincule partículas en fotogramas consecutivos en trayectorias utilizando el paquete de Python trackpy (v.0.5.0)29.
NOTA: La vinculación de la trayectoria se realiza sobre la marcha después de la detección puntual. Como resultado, un archivo CSV adicional que contiene la información de trayectoria se almacena en un subdirectorio del archivo CSV de detección de partículas.- Elimine las trayectorias con muy pocos puntos utilizando el parámetro minimum_trajectory_length (véase la sección B.1 del cuaderno) para permitir una determinación sólida de los coeficientes de difusión. Para obtener explicaciones detalladas sobre los otros parámetros de las funciones de trackpy , consulte la documentación de trackpy .
- Análisis de trayectoria
- En la sección C.1 del bloc de notas, especifique la velocidad de fotogramas (frame_rate) y el tamaño de píxel en nm (pixel_size), que se utilizaron para la adquisición de películas. Cree una lista de archivos CSV que contengan la información de trayectoria devuelta por trackpy (consulte el paso 9.2) con la función trajectory_analysis.get_csv_files (sección C.2 del bloc de notas).
- Además, especifique un nombre de archivo de salida para el contenedor HDF5, que se utiliza para almacenar los resultados de la conexión en el disco (sección C.3 del bloc de notas). Analice todas las trayectorias con la función trajectory_analysis.fit_trajectories en la sección C.4 del bloc de notas, que recorre en iteración la lista de archivos CSV. Esta función utiliza la distribución de distancia de salto (JDD)30 y el análisis de desplazamiento cuadrático medio (MSD)31 para estimar el coeficiente de difusión de cada trayectoria.
- Convertir la mediana de contraste de cada trayectoria a su masa correspondiente utilizando la relación contraste-masa obtenida de la calibración MSPT (ver sección 7). Especifique la pendiente (pendiente) y la intersección en Y (desplazamiento) de la línea de calibración, que relaciona el contraste iSCAT con la masa molecular (función trajectory_analysis.apply_calibration; consulte la sección C.5 del cuaderno). Esta función agrega una columna con la masa mediana de la trayectoria a cada trama de datos.
- Evalúe la densidad aparente de partículas en la membrana con la función trajectory_analysis.membrane_density, que devuelve el valor de densidad mediana en términos de partículas detectadas y trayectorias presentes durante cada fotograma (consulte la sección C.6 del cuaderno) como columnas adicionales en el marco de datos.
NOTA: Como una parte de las partículas se perderá durante el proceso de detección y enlace de trayectoria, las densidades reales de partículas pueden ser más altas. Para obtener resultados fiables con respecto a las densidades de partículas, así como a los histogramas de masa, inspeccione visualmente las instantáneas de películas representativas para verificar que las condiciones de medición sean plausibles para el seguimiento de una sola partícula (consulte el paso 9.1.1).
10. Visualización de datos
- Ilustrar la correlación de la masa y el coeficiente de difusión con la estimación bidimensional de la densidad del núcleo (KDE), que se basa en el paquete python fastkde (v.1.0.19; https://pypi.org/project/fastkde/).
- Para generar la gráfica, especifique el archivo HDF5 que contiene los resultados de MSPT (consulte el paso 9.3.2 y la sección D.1 del bloc de notas) y seleccione un único (sección D.2 del bloc de notas) o un marco de datos concatenado (sección D.3 del bloc de notas) como datos de entrada para la función plotting.generate_2D_KDE (sección D.4 del bloc de notas).
NOTA: Cada conjunto de datos trazado debe contener idealmente más de 1.000 trayectorias para un 2D-KDE fiable.
Resultados
Siguiendo el protocolo detallado aquí para la preparación de bicapas lipídicas soportadas (SLB) en cámaras de flujo (Figura 1), se puede reconocer claramente un patrón similar a una mota en la vista nativa de todas las condiciones mostradas (Figura 2). Este efecto es causado por la rugosidad superficial del vidrio, que generalmente domina la señal de dispersión y conduce a condiciones visualmente indistinguibles (vidrio, vidrio con SLB o vidrio con SLB y proteínas unidas). La presencia de vesículas, sin embargo, es claramente distinta debido a la gran sección transversal de dispersión de las vesículas y permite la observación de la ruptura de vesículas y la fusión en membranas homogéneas (Figura 2B y Película suplementaria 1). Al eliminar la señal de dispersión estática de la superficie de vidrio con un enfoque ratiométrico que enfatiza los elementos dinámicos dentro del campo de visión24,25, se pueden descubrir proteínas no etiquetadas que se difunden en la membrana (Figura 2D) mientras que un SLB vacío (Figura 2C) o el vidrio en sí (Figura 2A) aparece como una imagen ruidosa.
El fondo inherente de las mediciones MSPT se puede estimar localmente dividiendo cada valor de píxel a través de la mediana de n píxeles anteriores y posteriores de la película en la misma posición de imagen (Figura 3). Como resultado, las macromoléculas aparecen como funciones isotrópicas de dispersión de puntos (PSF) cuyo movimiento en la membrana se puede observar, rastrear y cuantificar. De hecho, la disponibilidad tanto del contraste como del comportamiento dinámico permite la relación directa del tamaño molecular de una partícula con su respectivo comportamiento difusivo, todo ello sin necesidad de etiquetar la partícula. Sin embargo, para interpretar el contraste iSCAT determinado durante los experimentos MSPT, es esencial realizar una calibración que traduzca la amplitud de la señal en masa molecular. Esto se puede lograr uniendo biomoléculas de masa conocida a un SLB a través de un complejo de biotina-estreptavidina-biotina (Figura 4A). Como estrategia ejemplar, se pueden utilizar variantes biotiniladas de albúmina sérica bovina (BSA), proteína A (prA), fosfatasa alcalina (AP) y fibronectina (FN), que se unen a la estreptavidina (STP) que a su vez está unida a los lípidos que contienen biotina (Biotinyl Cap PE) en la membrana. Como se muestra en la Figura 4A, el contraste cada vez más pronunciado de estas macromoléculas ejemplares refleja el creciente peso molecular de los respectivos estándares biotinilados. Al asignar cada pico de los histogramas de contraste (Figura 4B) a la masa correspondiente del estado oligómero de la proteína estándar, se revela una relación lineal entre contraste y masa21,22 y posteriormente se puede utilizar para el análisis de sistemas de macromoléculas desconocidos (Figura 4C).
Un buen ejemplo que demuestra la aplicabilidad y las capacidades de MSPT para analizar pesos moleculares y, por lo tanto, estudiar los estados de oligómeros y los eventos de oligomerización es la consideración de la aldolasa biotinilada y la IgG biotinilada (Figura 5). La aldolasa se informa comúnmente como un homotetrámero32. Sin embargo, la distribución de masa resuelta por MSPT presenta cuatro picos distintos, lo que destaca la presencia de múltiples poblaciones (Figura 5A). Si bien el primer pico menor corresponde a la estreptavidina desocupada y se puede esperar debido a la configuración en este tipo de experimento, también se pueden detectar complejos de aldolasa con solo dos subunidades (2SU) o seis subunidades (6SU) (Figura 5B). Curiosamente, los complejos tetra- y hexaméricos aldolasa-estreptavidina muestran un coeficiente de difusión reducido en comparación con la aldolasa dimérica y la estreptavidina solas, lo que indica un aumento de la resistencia viscosa, por ejemplo, a través de la unión de un segundo lípido biotinilado a la estreptavidina. Del mismo modo, la IgG biotinilada exhibe tres picos en la distribución de masa, con el primer pico nuevamente igualando la masa de una sola estreptavidina. La masa del pico más abundante corresponde a la masa de una cadena ligera y una pesada (1SU), es decir, la mitad de un anticuerpo IgG. El anticuerpo completo con dos mitades idénticas (2SU) se detecta en aproximadamente el 11% de los casos. La disminución del coeficiente de difusión con el aumento de los tamaños complejos indica interacciones de la estreptavidina con más de un lípido biotinilado o arrastre adicional causado por la IgG adjunta, o ambos.
Además del único análisis de los estados de oligómeros dependientes de la membrana, MSPT también confiere la ventaja particular de correlacionar el comportamiento difusivo de una macromolécula de interés con su estado de oligómero. Se muestran resultados representativos para este tipo de análisis para la anexina V (AnV) y la subunidad B de la toxina del cólera (CTxB), que se unen a la dioleoilfosfatidilserina (DOPS) o glicoesfingolípidos (GM1), respectivamente, incorporados en la membrana (Figura 6A). Ambas estimaciones de densidad de núcleo (KDE) presentan distribuciones unimodales de masa y difusión, lo que indica una sola especie abundante con un comportamiento difusivo similar. Se encontró que la posición máxima de la masa molecular y el coeficiente de difusión era de 49,8 ± 2,2 kDa y 1,4 ± 0,1 μm2/s, respectivamente, para AnV, así como de 62,7 ± 3,1 kDa y 0,4 ± 0,1 μm2/s, respectivamente, para CTxB. Los coeficientes de difusión medidos son comparables a los valores previamente informados obtenidos de AFM de alta velocidad y FRAP33,34. La masa ligeramente reducida en comparación con la masa de la macromolécula esperada (52 kDa para un trímero AnV, 65 kDa para un pentámero CTxB) puede indicar la presencia de complejos más pequeños con menos subunidades en el conjunto. Si bien la diferencia de masa entre las proteínas es pequeña y cercana al límite de detección especificado del microscopio (≈50 kDa), sus coeficientes de difusión difieren considerablemente. En una mezcla equimolar, por ejemplo, comparando la difusión de la mezcla con la distribución de AnV y CTxB solos, se puede concluir que AnV es más abundante en la membrana que CTxB (Figura 6B). Sin embargo, si la concentración de CTxB se duplica en comparación con la concentración de AnV, el equilibrio se desplaza hacia CTxB como la proteína predominante en la membrana. Como se ilustra para las mezclas de AnV y CTxB, MSPT no solo permite discriminar macromoléculas asociadas a membranas según su peso molecular, sino que también permite la discriminación de diferentes poblaciones de macromoléculas según su comportamiento difusivo.
Al igual que con todas las técnicas de microscopía, algunos requisitos experimentales son cruciales para lograr la calidad deseada de los datos. Un ejemplo importante en este contexto son los cubrehojas limpiados a fondo. En general, esto se considera un requisito previo para los experimentos de molécula única relacionados con la microscopía, pero MSPT es particularmente sensible a las impurezas de la muestra. El aumento de la dispersión que se origina en la superficie de vidrio de las cubiertas sin limpiar impide cualquier medición cuantitativa de iSCAT. En particular, incluso la suciedad residual o las partículas de polvo en vidrio insuficientemente limpiado pueden causar distorsiones de imagen notables, reconocibles como puntos brillantes en el modo de imagen nativo (Figura 7A). Aunque estos defectos son eliminados por la estimación de fondo debido a su naturaleza estática, la determinación precisa del contraste de una partícula podría verse afectada y, por lo tanto, influir negativamente en su análisis cuantitativo. Otro problema común encontrado en los experimentos MSPT son las vesículas restantes que flotan (rodeadas de naranja) a través del campo de visión o las vesículas no fusionadas que están pegadas (rodeadas de azul) en una posición específica en la membrana y aparecen como grandes dispersores pulsantes (Figura 7B). Para minimizar su aparición e interferencia con la adquisición de películas, se recomienda lavar a fondo el SLB antes de agregar la proteína y usar mezclas recién preparadas de pequeñas vesículas unilamelares (SUV) y cationes divalentes.
Un factor que debe tenerse en cuenta para el diseño de experimentos de seguimiento de partículas sensibles a la masa es la densidad de macromoléculas asociadas con la interfaz de membrana. De hecho, las altas densidades de partículas en la membrana pueden causar problemas por dos razones: i) La vinculación de las detecciones de partículas de fotogramas consecutivos en trayectorias se vuelve ambigua y, por lo tanto, aumenta la probabilidad de errores y coeficientes de difusión mal juzgados. ii) La masa de las partículas, que se extrae de la amplitud de su correspondiente ajuste PSF, se subestima sistemáticamente y los picos de masa se amplían porque la separación de la señal de fondo estática de la señal de partícula dinámica es cada vez más difícil (Figura 7C). Actualmente, la evaluación visual de la calidad de los datos en el proceso de adquisición de videos MSPT es difícil en los microscopios comerciales disponibles porque la vista ratiométrica implementada en el software de adquisición está utilizando la eliminación de fondo establecida para la fotometría de masas21 en lugar del algoritmo basado en la mediana descrito aquí y en las referencias24,25 (Figura 7D ). La eliminación continua de fondo basada en la media utilizada para visualizar las moléculas de aterrizaje en la fotometría de masas hace que las partículas difusoras aparezcan como frentes oscuros con colas brillantes, lo que hace que las manchas parezcan altamente anisotrópicas e interfiere con el ajuste de PSF durante el procedimiento de detección. Por lo tanto, el uso del procesamiento de imágenes basado en medias implementado en el software de adquisición no es adecuado para el análisis de biomoléculas difusoras en membranas.
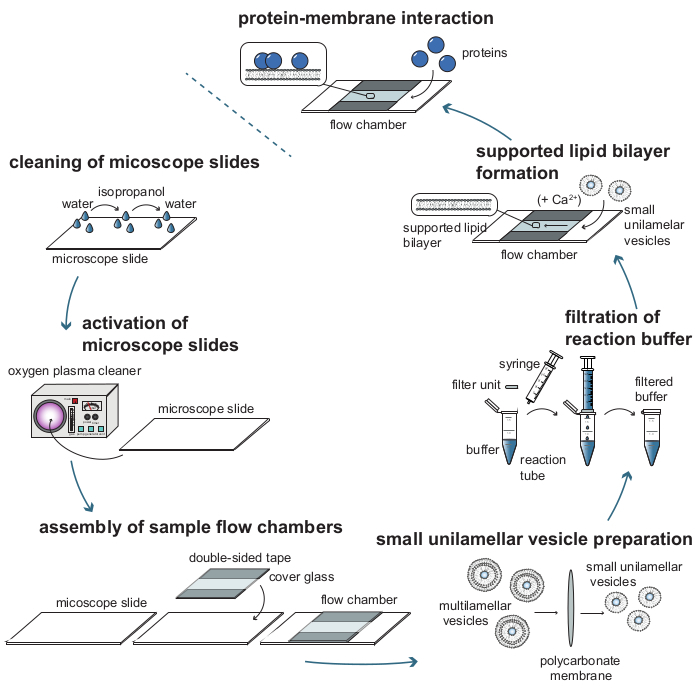
Figura 1: Diagrama de flujo de proceso de los pasos individuales necesarios para analizar las interacciones proteína-membrana con el seguimiento de partículas sensibles a la masa (MSPT). Para preparar muestras para las mediciones de MSPT, los portaobjetos de cubierta de vidrio deben limpiarse a fondo y activarse con un plasma de oxígeno. Después de su ensamblaje en cámaras de flujo de muestra, se preparan pequeñas vesículas unilamelares (SUV) para la formación de bicapa lipídica (SLB) soportada y todos los tampones de reacción se filtran para reducir la dispersión de fondo. Los SUV se agregan para formar bicapas lipídicas en las cámaras de flujo. Opcionalmente, se pueden agregar cationes divalentes como los iones Ca2+ a los SUV para promover la ruptura de la vesícula. Por fin, las bajas concentraciones de la proteína de interés se introducen en la cámara de reacción. Haga clic aquí para ver una versión más grande de esta figura.
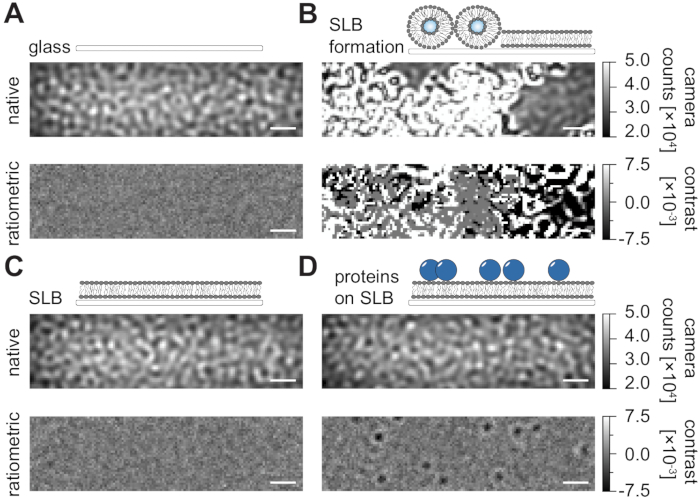
Figura 2: Vista nativa y ratiométrica de superficies ejemplares relevantes para mediciones MSPT. Imágenes representativas de la rugosidad superficial de un portaobjetos de cubierta de vidrio (A), durante la formación de una bicapa lipídica soportada (B), con una bicapa lipídica soportada intacta (C) y de proteínas ejemplares reconstituidas en un SLB (D). Los cuatro ejemplos se muestran en el modo nativo, al que se puede acceder durante la medición en sí, y como imágenes ratiométricas procesadas después de la eliminación del fondo basado en la mediana. Las barras de escala representan 1 μm. Para el análisis de los datos (ver cuaderno de Jupyter acompañado; paso 9), se utilizaron los siguientes parámetros: tamaño medio de la ventana (window_length) = 1001. Haga clic aquí para ver una versión más grande de esta figura.
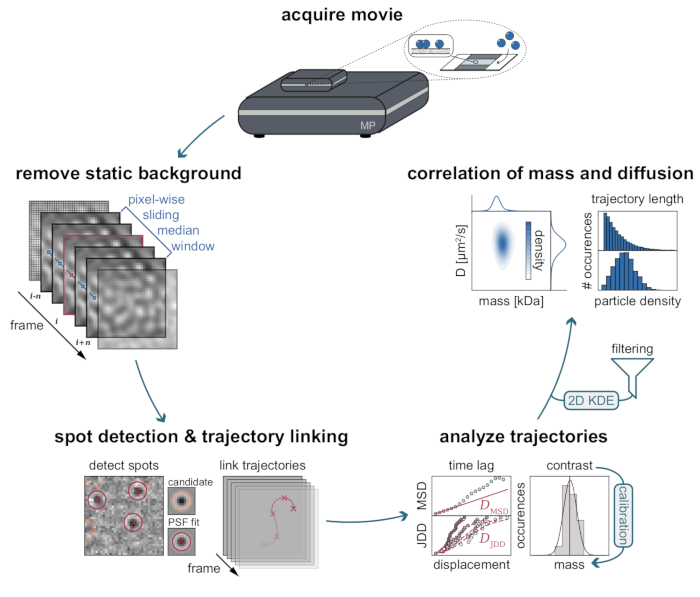
Figura 3: Diagrama paso a paso de las etapas requeridas para la recopilación y análisis de datos MSPT. Después de la adquisición de datos para la muestra de interés en el fotómetro de masas, las películas se procesan para eliminar el fondo estático a través de un enfoque de mediana deslizante en píxeles. A partir de entonces, las partículas candidatas se identifican y se ajustan mediante una función de dispersión de puntos (PSF) antes de su enlace en las trayectorias de las partículas. Para permitir la determinación del coeficiente de difusión para cada partícula, se emplea el análisis de desplazamiento cuadrático medio (MSD) o distribución de distancia de salto (JDD). En esta etapa, los valores de contraste se pueden transformar en masas moleculares de acuerdo con la relación contraste-masa determinada a través de la estrategia de calibración. Como paso final, las trayectorias se pueden filtrar en función de su longitud o densidad de partículas de membrana y visualizarse mediante la estimación bidimensional de la densidad del núcleo (2D-KDE). Haga clic aquí para ver una versión más grande de esta figura.
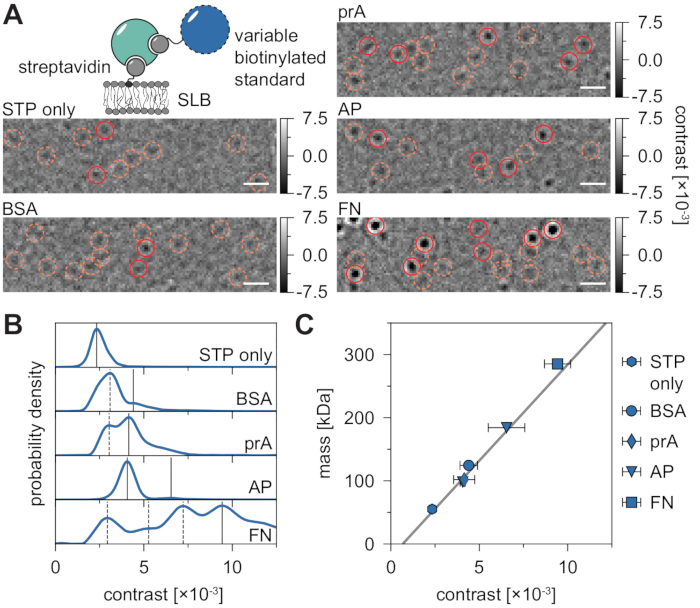
Figura 4: Calibración de la relación masa-contraste para las mediciones de MSPT. (A) Marcos ratiométricos representativos obtenidos para complejos de proteínas estándar de estreptavidina ejemplares que se difunden en una bicapa lipídica soportada que contiene un pequeño porcentaje de lípidos biotinilados (DOPC: DOPG: Relación de PE de la tapa de biotinilo de 70: 29.99: 0.01 mol%). Como estándares de peso molecular modelo, se muestran la estreptavidinamonovalente 28 (solo STP) o la estreptavidinadivalente 28 en complejo con albúmina sérica bovina biotinilada (BSA), proteína biotinilada A (prA), fosfatasa alcalina biotinilada (AP) o fibronectina biotinilada (FN). Los puntos candidatos se resaltan en naranja (círculos discontinuos) y las detecciones de partículas exitosas se resaltan en rojo (círculos sólidos). Las barras de escala representan 1 μm. (B) Distribuciones de densidad de probabilidad de los valores de contraste obtenidos para las cinco proteínas estándar del modelo. Todos los datos mostrados representan distribuciones agrupadas de tres experimentos independientes por condición: STP solo n = 82,719; BSA n = 9.034; prA n = 22.204; AP n = 69.065, y FN n = 71.759 trayectorias. En comparación con el número de partículas determinado para membranas con proteínas, el número de partículas detectadas en una bicapa vacía es insignificante a densidades moderadas de membrana (Figura suplementaria 1). Los picos de contraste considerados para la calibración de masa se marcan a través de líneas continuas, mientras que los discontinuos representan estados de oligómeros no considerados. (C) Curva de calibración de contraste a masa derivada de los contrastes máximos en el panel D y las respectivas masas de secuencia de los complejos. Las barras de error muestran el error estándar de las ubicaciones de pico estimadas por bootstrapping (100 remuestreos con 1.000 trayectorias cada uno). Para el análisis de datos (consulte Jupyter notebook; paso 9), se utilizaron los siguientes parámetros: tamaño medio de ventana (window_length) = 1.001 fotogramas, umbral de detección (thresh) = 0,00055, rango de búsqueda (dmax) = 4 píxeles, memoria (max_frames_to_vanish) = 0 fotogramas, longitud mínima de trayectoria (minimum_trajectory_length) = 7 fotogramas (solo STP), 9 fotogramas (BSA/FN), 15 fotogramas (prA), 10 fotogramas (AP). Haga clic aquí para ver una versión más grande de esta figura.
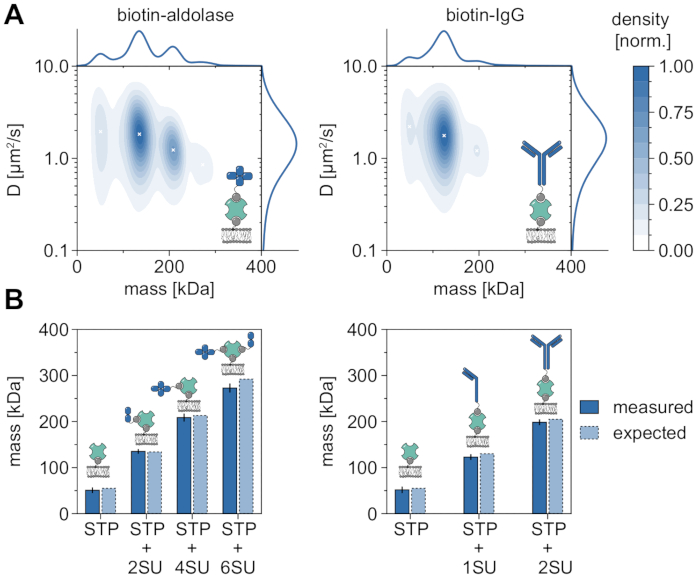
Figura 5: Desciframiento de los estados de oligómeros de proteínas asociadas a la membrana. (A) Estimaciones de la densidad del núcleo 2D tanto de la masa como del coeficiente de difusión de la estreptavidina tetravalente en complejo con aldolasa biotinilada (panel izquierdo) o con un anticuerpo IgG anti-conejo de cabra modificado con biotina (panel derecho). La reconstitución de ambos complejos se ha realizado en una bicapa lipídica soportada que contiene DOPC, DOPG y Biotinyl Cap PE en una proporción de 70:29.99:0.01 mol%, respectivamente. En total, se han incluido 116.787 trayectorias de tres réplicas independientes para el complejo estreptavidina-aldolasa (densidad de partículas de 0,1 μm-2) y 348.405 para el compuesto estreptavidina-IgG (densidad de partículas de 0,1 μm-2). Solo se incluyeron partículas con una longitud de pista de al menos cinco fotogramas. Se presentan distribuciones de probabilidad marginal tanto de masa molecular (arriba) como de coeficiente de difusión (derecha). La x negra en ambos paneles marca los máximos locales respectivos del KDE. (B) Comparación de masas de oligómeros determinadas para el complejo de estreptavidina tetravalente con aldolasa modificada con biotina (panel izquierdo) o IgG biotinilada (panel derecho) con, según las masas de secuencia, pesos moleculares esperados. La abreviatura SU se introduce en nombre de la subunidad de la proteína de intereses. Las barras de error muestran el error estándar de las ubicaciones de pico estimadas por bootstrapping (100 remuestreos con 1.000 trayectorias cada uno). Para el análisis de datos (ver cuaderno Jupyter acompañado; paso 9), se utilizaron los siguientes parámetros: tamaño medio de ventana (window_length) = 1.001 fotogramas, umbral de detección (thresh) = 0,00055, rango de búsqueda (dmax) = 4 píxeles, memoria (max_frames_to_vanish) = 0 fotogramas, longitud mínima de trayectoria (minimum_trajectory_length) = 5. Haga clic aquí para ver una versión más grande de esta figura.
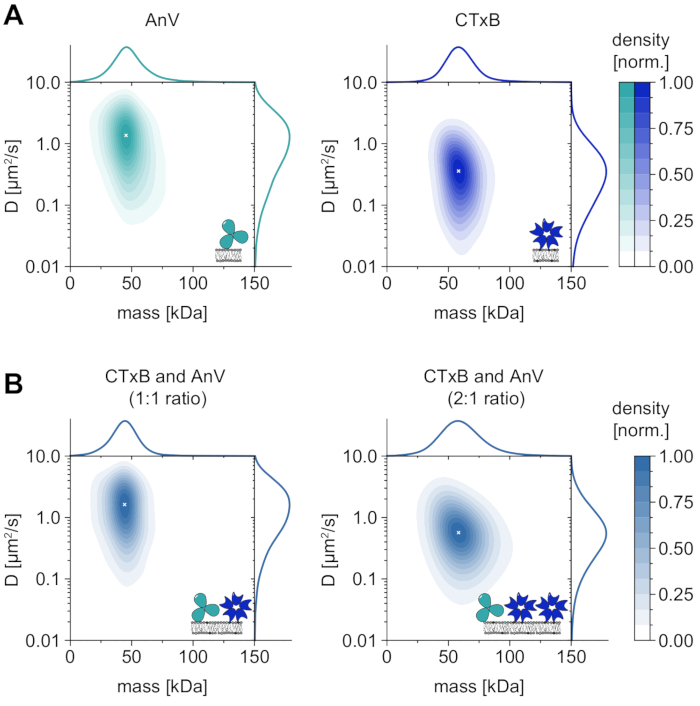
Figura 6: Disolución del comportamiento difusivo de las proteínas nativas que interactúan con la membrana anexina V (AnV) y la subunidad B de la toxina del cólera (CTxB). (A) Estimaciones de la densidad del núcleo 2D tanto de la masa como del coeficiente de difusión de la anexina V (panel izquierdo) y la subunidad B de la toxina del cólera (panel derecho). Para la reconstitución de la membrana AnV y CTxB, se han utilizado composiciones lipídicas de 80:20 mol% DOPC a DOPS y 99.99:0.01 mol% DOPC a GM1, respectivamente. En total se han incluido 206.819 trayectorias de tres réplicas independientes para AnV (densidad de partículas de 0,1 μm-2) y 142.895 trayectorias para CTxB (densidad de partículas de 0,2 μm-2). (B) Estimaciones de densidad de kernel 2D de mezclas CTxB y AnV en una proporción de 1:1 (panel izquierdo) o 2:1 (panel derecho), respectivamente. La reconstitución de mezclas de proteínas se realizó en una bicapa lipídica soportada que contenía lípidos DOPC, DOPS y GM1 en una proporción de 80: 19.99: 0.01 mol%. En total se han incluido 42.696 trayectorias de tres réplicas independientes para la mezcla 1:1 (densidad de partículas de 0,1 μm-2) y 264.561 trayectorias para la relación 2:1 (densidad de partículas de 0,3 μm-2). Tanto para (A) como para (B), solo se incluyeron partículas con una longitud de pista de al menos cinco fotogramas. Se presentan distribuciones de probabilidad marginal tanto de masa molecular (arriba) como de coeficiente de difusión (derecha). La x blanca en cada panel marca el máximo global respectivo del KDE. Para el análisis de datos (ver cuaderno Jupyter acompañado; paso 9), se utilizaron los siguientes parámetros: tamaño medio de ventana (window_length) = 1.001 fotogramas, umbral de detección (trilla) = 0,00055, rango de búsqueda (dmax) = 4 píxeles, memoria (max_frames_to_vanish) = 0 fotogramas, longitud mínima de trayectoria (minimum_trajectory_length) = 5. Haga clic aquí para ver una versión más grande de esta figura.
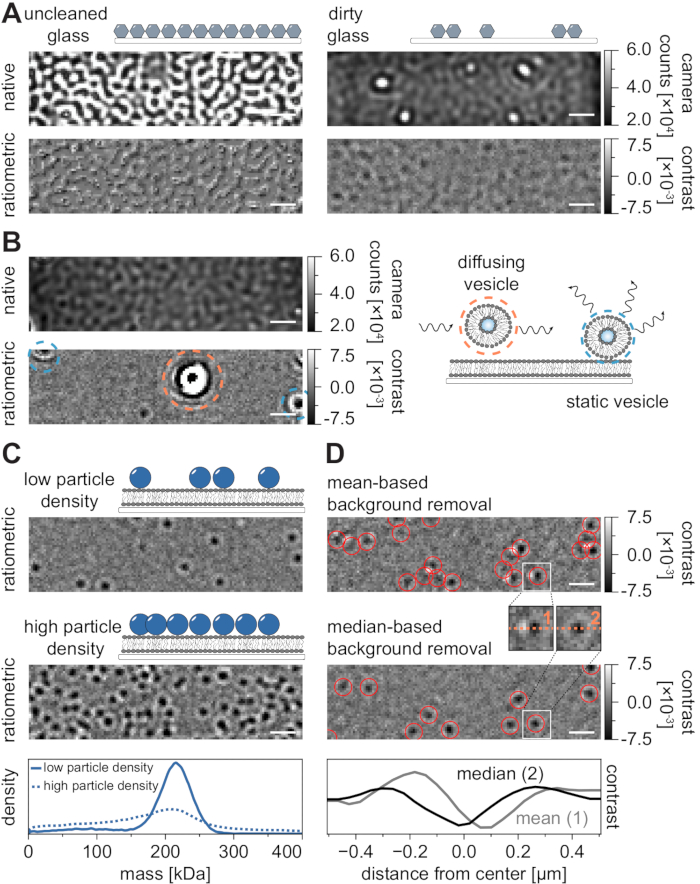
Figura 7: Posibles complicaciones en el curso de las mediciones de MSPT o durante el análisis de datos. (A) Imágenes representativas de la rugosidad de la superficie que se muestran tanto en la vista ratiométrica nativa como en la procesada (eliminación de fondo basada en la mediana) de un portaobjetos de vidrio de cubierta sin limpiar. En ambos casos, los puntos brillantes constituyen impurezas superficiales residuales que impiden las mediciones sin artefactos. (B) Imágenes ejemplares de vesículas residuales en el campo de visión después de un lavado insuficiente de la membrana. Tanto las vesículas estáticas (resaltadas en azul) como las difusoras (resaltadas en naranja) afectarán la calidad de la medición, ya sea debido a la pulsación y el movimiento o debido a su movimiento direccional, respectivamente. (C) Como técnica de una sola partícula, MSPT requiere bajas densidades de partículas (imagen representativa, panel superior) para permitir la vinculación adecuada y la determinación de masa de cada partícula. En el caso de altas densidades de partículas de membrana (panel medio), el ajuste de partículas se ve afectado, lo que afecta la determinación de masa (ver panel inferior). (D) Imágenes ratiométricas representativas de partículas que se difunden en una interfaz de membrana después de la eliminación de fondo basada en medias (panel superior) o en la mediana. Para la difusión de partículas, la estrategia de eliminación de fondo basada en la media produce imágenes distorsionadas del PSF de la partícula, como se puede ver en los pequeños recuadros entre el panel superior y medio. Por el contrario, los PSF de partículas no distorsionadas se pueden obtener a través del enfoque basado en la mediana. Panel inferior: Comparación de perfiles de línea a través del centro del PSF obtenidos después de la eliminación de fondo basada en medias o medianas. Para todas las imágenes nativas y ratiométricas que se muestran en esta figura, las barras de escala representan 1 μm. Para el análisis de datos (ver cuaderno Jupyter acompañado; paso 9), se utilizaron los siguientes parámetros: tamaño medio de ventana (window_length) = 1.001 fotogramas, umbral de detección (thresh) = 0,00055, rango de búsqueda (dmax) = 4 píxeles, memoria (max_frames_to_vanish) = 0 fotogramas, longitud mínima de trayectoria (minimum_trajectory_length) = 5. Haga clic aquí para ver una versión más grande de esta figura.
Figura complementaria 1: Comparación de membranas libres de proteínas y ocupadas. Imágenes representativas de una bicapa lipídica soportada intacta antes (A) y después (B) de la adición de estreptavidina purificada (STP). Los puntos candidatos que se ajustaron con éxito al modelo PSF están rodeados de rojo. (C) Distribuciones de probabilidad de contraste de partículas detectadas en una membrana vacía (fondo de membrana, gris) y en una bicapa con partículas de estreptavidina difusora (azul). Ambas distribuciones de probabilidad representan los datos agrupados de tres experimentos independientes con parámetros idénticos de adquisición y análisis de películas. Para el análisis de datos (ver cuaderno Jupyter acompañado; paso 9), se utilizaron los siguientes parámetros: tamaño medio de ventana (window_length) = 1.001 fotogramas, umbral de detección (trilla) = 0,00055, rango de búsqueda (dmax) = 4 píxeles, memoria (max_frames_to_vanish) = 0 fotogramas, longitud mínima de trayectoria (minimum_trajectory_length) = 7 fotogramas. Haga clic aquí para descargar este archivo.
Película complementaria 1: Película ejemplar que muestra la ruptura y fusión de vesículas en una membrana homogénea registrada con el fotómetro de masas. Tamaño medio de ventana (window_length) de procesamiento de imágenes = 1.001 fotogramas. Barra de escala: 1 μm. Rango de recuento de cámaras: negro = 16,892; blanco = 65.408. Haga clic aquí para descargar esta película.
Película complementaria 2: Películas ejemplares que muestran la difusión de complejos de anexina V (arriba) y aldolasa biotinilada (abajo) en una bicapa obtenida de mediciones MSPT. Tamaño medio de ventana (window_length) de procesamiento de imágenes = 1.001 fotogramas. Barra de escala: 1 μm. Rango de contraste de dispersión interferométrica: negro = -0.0075; blanco = 0,0075. Haga clic aquí para descargar esta película.
Discusión
El protocolo presentado extiende la fotometría de masas21, una técnica que analiza la masa de biomoléculas individuales que se adsorben en vidrio, a una herramienta aún más versátil capaz de medir simultáneamente la masa y la difusión de biomoléculas que interactúan con membranas no etiquetadas. Esta extensión de análisis se consigue mediante la implementación de una estrategia de eliminación de fondo modificada adaptada al movimiento lateral de las moléculas24,25. En general, la eliminación del fondo es de suma importancia para los enfoques basados en iSCAT, ya que la fuerte dispersión de la rugosidad de la superficie del vidrio representa el principal impedimento del análisis, y la determinación precisa del fondo local de cada píxel es esencial para la cuantificación de la masa y la ubicación de las partículas. Además del análisis de imágenes adaptado al movimiento de las partículas, la posterior detección de partículas, la vinculación de trayectorias y el análisis de datos completan la novedosa expansión de MP en el seguimiento de partículas sensibles a la masa (MSPT).
En general, los portaobjetos de cubierta de vidrio limpiados a fondo y un entorno de trabajo limpio son requisitos críticos para el desempeño exitoso de los experimentos MSPT. Debido a la ausencia de etiquetado de macromoléculas, la señal adquirida es inherentemente no selectiva. Las muestras limpias, así como el manejo adecuado de las muestras, son, por lo tanto, cruciales para garantizar que las observaciones no puedan malinterpretarse. En particular, cuando se examinan moléculas de bajo peso molecular, se aprueban las mediciones de control de membranas libres de proteínas para evaluar las contribuciones de fondo (Figura suplementaria 1). Además de la inclusión de mediciones de control, se recomienda seguir los pasos de preparación que se muestran en la Figura 2 para cada cámara de flujo. Cuando se combinan, estas medidas de seguridad garantizarán que la señal detectada se origine en la biomolécula de interés y no, por ejemplo, en una cámara de flujo, tampón o membrana contaminada.
Además de las precauciones con respecto al diseño experimental, también se debe tener cuidado durante el procesamiento de imágenes MSPT. Durante el procesamiento de vídeo, el valor de tres parámetros debe elegirse cuidadosamente para garantizar resultados correctos: i) la longitud de la ventana mediana para la eliminación del fondo, ii) el umbral para la detección de partículas y iii) el radio de búsqueda máximo durante la asignación de enlace. Una ventana mediana más grande (i) generalmente facilita la separación de las partículas difusoras del fondo cuasi-constante superpuesto. Sin embargo, para tamaños de ventana demasiado grandes, la deriva de la muestra eventualmente se notará y disminuirá la precisión de la estimación de fondo. Los ajustes óptimos dependen en gran medida de las propiedades de la muestra y las condiciones de medición. Sin embargo, un valor de 1.001 se puede utilizar como un punto de partida robusto. El parámetro umbral (ii) debe ajustarse en función de la masa molecular más baja esperada en la muestra. No se recomienda un valor por debajo de 0,0005 para las mediciones tomadas con el fotómetro de masas utilizado en este estudio. Para acelerar los tiempos de análisis, se pueden elegir valores más altos si se espera una muestra con alto peso molecular. El radio de búsqueda en el enlace de trayectoria (iii) especifica la distancia radial máxima en píxeles en la que se buscará la ubicación desplazada de la partícula en fotogramas consecutivos. Debe adaptarse a la partícula más rápida de la muestra y, si se prefiere, se podría usar un rango de búsqueda adaptativo (consulte la documentación de trackpy) para reducir el tiempo de cálculo. Especialmente durante la fase inicial de un proyecto, se recomienda volver a analizar las películas con parámetros variables para validar los resultados obtenidos.
A la luz de la naturaleza de una sola molécula de MSPT, se debe evitar medir a altas densidades de partículas de membrana, ya que pueden interferir con el contraste preciso y la determinación de masa. Se ha demostrado que las densidades por debajo de una partícula por micrómetro cuadrado son favorables para las mediciones de MSPT24. Una consideración adicional son los coeficientes de difusión esperados en la muestra. Aunque es aplicable a una amplia gama de coeficientes de difusión, MSPT tiene un límite inferior de coeficientes de difusión accesibles. El confinamiento local a una región de pocos píxeles durante una parte significativa del período de ventana mediana fusiona la partícula con el fondo estático. Para las condiciones de imagen utilizadas en este protocolo, no se recomienda la medición de coeficientes de difusión por debajo de 0,01 μm2/s. A esta velocidad de difusión, por ejemplo, el desplazamiento cuadrático medio de una partícula durante la mediana ventana de tamaño medio es de aproximadamente 4 píxeles y, por lo tanto, de tamaño similar a la extensión del PSF. Como consecuencia, es probable que la estimación de fondo estático contenga contribuciones de señal de la propia partícula, lo que resulta en un contraste aparentemente reducido de la partícula hasta que finalmente se acerca al nivel de ruido. Sin embargo, los coeficientes de difusión de macromoléculas que oscilan entre 0,05 y 10 μm2/s pueden resolverse claramente.
Para ampliar aún más el rango de aplicaciones MSPT, se puede imaginar un avance del algoritmo de fondo basado en la mediana a través de la eliminación de píxeles que están ocupados temporalmente con una partícula, o mediante la corrección de la deriva de la muestra que permite tamaños de ventana medianos más grandes. Ambos enfoques aliviarían los problemas relacionados con las mediciones a altas densidades de partículas y la difusión lenta. Las mejoras en términos de menor sensibilidad de masa están en el horizonte con una nueva generación de fotómetros de masas, que pueden proporcionar acceso a biomoléculas menores de 50 kDa. Por lo tanto, los futuros experimentos MSPT podrán estudiar la dinámica de una sola molécula y las interacciones relacionadas con la membrana para una gama aún más amplia de imitaciones de membrana, como bicapas amortiguadas y sistemas macromoleculares.
Divulgaciones
Los autores no tienen conflictos de intereses.
Agradecimientos
Agradecemos sinceramente el apoyo de Philipp Kukura, Gavin Young y el equipo de software de Refeyn y reconocemos su ayuda al compartir partes del código de análisis de imágenes. Agradecemos a cryo-EM MPIB Core Facility por proporcionar acceso al fotómetro de masas comercial Refeyn. F.S. agradece el apoyo y la financiación otorgados por Jürgen Plitzko y Wolfgang Baumeister. T.H. y P.S. recibieron financiación a través de la Deutsche Forschungsgemeinschaft (DFG, Fundación Alemana de Investigación) - Project-ID 201269156 - SFB 1032 (A09). N.H. fue apoyado por una subvención de retorno de DFG HU 2462/3-1. P.S. reconoce el apoyo a través de la red de investigación MaxSynBio a través de la iniciativa de financiación conjunta del Ministerio Federal Alemán de Educación e Investigación (BMBF) y la Sociedad Max Planck.
Materiales
| Name | Company | Catalog Number | Comments |
| annexin V | Sigma Aldrich | #SRP8026 | examplary membrane-interacting protein |
| Bio-Rad Protein Assay | Bio-Rad Laboratories Inc. | #5000006 | bradford assay kit to determine protein stock concentrations |
| biotin labeled bovine albumin | Sigma Aldrich | #A8549 | examplary protein that can be used as standard protein for MSPT |
| cholera toxin subunit B | Sigma Aldrich | #SAE0069 | examplary membrane-interacting protein |
| cover glasses, #1.5, 24 x 24 mm | Paul Marienfeld GmbH & Co. KG | #0102062 | |
| cover glasses, #1.5, 24 x 60 mm | Paul Marienfeld GmbH & Co. KG | #0102242 | |
| dioleoyl-sn-glycero-3-phosphocholine (DOPC) | Avanti Polar Lipids | #850375 | lipid - in the form of extruded small unilamellar vesicles required for supported lipid bilayer formation |
| dioleoyl-sn-glycero-3-phosphoethanolamine-N-cap biotinyl (18:1 Biotinyl Cap PE | Avanti Polar Lipids | #870273 | lipid - in the form of extruded small unilamellar vesicles required for supported lipid bilayer formation |
| dioleoyl-sn-glycero-3-phosphoglycerol (DOPG) | Avanti Polar Lipids | #840475 | lipid - in the form of extruded small unilamellar vesicles required for supported lipid bilayer formation |
| dioleoyl-sn-glycero-3-phospho-L-serine (DOPS) | Avanti Polar Lipids | #840035 | lipid - in the form of extruded small unilamellar vesicles required for supported lipid bilayer formation |
| double-sided tape | tesa | #57912-00000-02 | needed for the assembly of glass sample chambers |
| Extruder | Avanti Polar Lipids | #610023 | Lipid extruder to enable monodisperse vesicle distributions |
| EZ-Link Maleimide-PEG2-Biotin | Thermo Fisher Scientific | #A39261 | maileimide-fused biotin that can be used to biotinylate standard proteins for MSPT |
| Fibronectin (Biotinylated) | Cytoskeleton Inc. | #FNR03-A | examplary protein that can be used as standard protein for MSPT |
| Gel Filtration HMW Calibration Kit | Cytiva | #28403842 | standard proteins, e.g. aldolase that can be biotinylated and used as molecular weight standards for MSPT |
| GM1 Ganglioside (Brain, Ovine-Sodium Salt) | Avanti Polar Lipids | #860065 | lipid - in the form of extruded small unilamellar vesicles required for supported lipid bilayer formation |
| Goat anti-Rabbit IgG (H+L) Secondary Antibody, Biotin | Thermo Fisher Scientific, Waltham, USA) | #31820 | examplary protein to highlight the existence of different protein states |
| Isopropanol, 99.5%, for spectroscopy | Thermo Fisher Scientific | #10003643 | |
| Low Autofluorescence Immersion Oil | Olympus K.K. | #IMMOIL-F30CC | |
| pET21a-Streptavidin-Alive | Addgene | #20860 | required to express and purify divalent streptavidin in combination with each other |
| pET21a-Streptavidin-Dead | Addgene | #20859 | required to express and purify divalent streptavidin in combination with each other |
| Pierce Alkaline Phosphatase, biotinylated | Thermo Fisher Scientific | #29339 | examplary protein that can be used as standard protein for MSPT |
| Pierce Protein A, Biotinylated | Thermo Fisher Scientific | #29989 | examplary protein that can be used as standard protein for MSPT |
| Refeyn Acquire | Refeyn Ltd. | control software for Refeyn OneMP | |
| Refeyn One | Refeyn Ltd. | - | mass photometer |
| sterile syringe filters 0.45 µm cellulose acetate membrane | VWR International | #514-0063 | needed to filter particles from the buffer of interest |
| tetravalent streptavidin | Thermo Fisher Scientific | #SNN1001 | tetravalent streptavidin to enable the presence of several biotin binding sites |
| Whatman Nuclepore Hydrophilic Membrane, 0.05 µm Pore Size, 25 mm Circle | Cytiva | #110603 | a pore size of 50 nm is recommended for supported lipid bilayer formation in the context of MSPT |
| Zepto model 2 plasma cleaner | Diener electronic GmbH | - |
Referencias
- Robertson, J. L. The lipid bilayer membrane and its protein constituents. Journal of General Physiology. 150 (11), 1472-1483 (2018).
- Grecco, H. E., Schmick, M., Bastiaens, P. I. H. Signaling from the Living Plasma Membrane. Cell. 144 (6), 897-909 (2011).
- Cho, W., Stahelin, R. V. Membrane-protein interactions in cell signaling and membrane trafficking. Annual Review of Biophysics and Biomolecular Structure. 34, 119-151 (2005).
- Whited, A. M., Johs, A. The interactions of peripheral membrane proteins with biological membranes. Chemistry and Physics of Lipids. 192, 51-59 (2015).
- Gonzalez, L., Scheller, R. H. Regulation of membrane trafficking: Structural insights from a Rab/effector complex. Cell. 96 (6), 755-758 (1999).
- Sezgin, E., Levental, I., Mayor, S., Eggeling, C. The mystery of membrane organization: composition, regulation and roles of lipid rafts. Nature Reviews Molecular Cell Biology. 18 (6), 361-374 (2017).
- Bagheri, Y., Ali, A. A., You, M. Current methods for detecting cell membrane transient interactions. Frontiers in Chemistry. 8, 603259 (2020).
- Miller, H., Zhou, Z., Shepherd, J., Wollman, A. J. M., Leake, M. C. Single-molecule techniques in biophysics: A review of the progress in methods and applications. Reports on Progress in Physics. 81 (2), 024601 (2018).
- Manzo, C., Garcia-Parajo, M. F. A review of progress in single particle tracking: From methods to biophysical insights. Reports on Progress in Physics. 78 (12), 124601 (2015).
- Gelles, J., Schnapp, B. J., Sheetz, M. P. Tracking kinesin-driven movements with nanometre-scale precision. Nature. 331 (6155), 450-453 (1988).
- Funatsu, T., Harada, Y., Tokunaga, M., Saito, K., Yanagida, T. Imaging of single fluorescent molecules and individual ATP turnovers by single myosin molecules in aqueous solution. Nature. 374 (6522), 555-559 (1995).
- Schmidt, T., Schütz, G. J., Baumgartner, W., Gruber, H. J., Schindler, H. Imaging of single molecule diffusion. Proceedings of the National Academy of Sciences of the United States of America. 93 (7), 2926-2929 (1996).
- Taylor, R. W., et al. Interferometric scattering microscopy reveals microsecond nanoscopic protein motion on a live cell membrane. Nature Photonics. 13 (7), 480-487 (2019).
- Kukura, P., et al. High-speed nanoscopic tracking of the position and orientation of a single virus. Nature Methods. 6 (12), 923-927 (2009).
- Jacobsen, V., Stoller, P., Brunner, C., Vogel, V., Sandoghdar, V. Interferometric optical detection and tracking of very small gold nanoparticles at a water-glass interface. Optics Express. 14 (1), 405 (2006).
- Ueno, H., et al. Simple dark-field microscopy with nanometer spatial precision and microsecond temporal resolution. Biophysical Journal. 98 (9), 2014-2023 (2010).
- Loose, M., Fischer-Friedrich, E., Herold, C., Kruse, K., Schwille, P. Min protein patterns emerge from rapid rebinding and membrane interaction of MinE. Nature Structural and Molecular Biology. 18 (5), 577-583 (2011).
- Bezeljak, U., Loya, H., Kaczmarek, B., Saunders, T. E., Loose, M. Stochastic activation and bistability in a Rab GTPase regulatory network. Proceedings of the National Academy of Sciences of the United States of America. 117 (12), 6504-6549 (2020).
- Ha, T., Tinnefeld, P. Photophysics of fluorescent probes for single-molecule biophysics and super-resolution imaging. Annual Review of Physical Chemistry. 63 (1), 595-617 (2012).
- Garcia-Parajo, M. F., Segers-Nolten, G. M. J., Veerman, J. A., Greve, J., Van Hulst, N. F. Real-time light-driven dynamics of the fluorescence emission in single green fluorescent protein molecules. Proceedings of the National Academy of Sciences of the United States of America. 97 (13), 7237-7242 (2000).
- Young, G., et al. Quantitative mass imaging of single biological macromolecules. Science. 360 (6387), 423-427 (2018).
- Piliarik, M., Sandoghdar, V. Direct optical sensing of single unlabelled proteins and super-resolution imaging of their binding sites. Nature Communications. 5, 4495 (2014).
- Cole, D., Young, G., Weigel, A., Sebesta, A., Kukura, P. Label-free single-molecule imaging with numerical-aperture-shaped interferometric scattering microscopy. ACS Photonics. 4 (2), 211-216 (2017).
- Heermann, T., Steiert, F., Ramm, B., Hundt, N., Schwille, P. Mass-sensitive particle tracking to elucidate the membrane-associated MinDE reaction cycle. Nature Methods. 18 (10), 1239-1246 (2021).
- Foley, E. D. B., Kushwah, M. S., Young, G., Kukura, P. Mass photometry enables label-free tracking and mass measurement of single proteins on lipid bilayers. Nature Methods. 18 (10), 1247-1252 (2021).
- Voss, O. H., Lee, H. N., Tian, L., Krzewski, K., Coligan, J. E. Liposome preparation for the analysis of lipid-receptor interaction and efferocytosis. Current Protocols in Immunology. 120, 1-21 (2018).
- Pincet, F., et al. FRAP to characterize molecular diffusion and interaction in various membrane environments. PLoS ONE. 11 (7), 0158457 (2016).
- Howarth, M., et al. A monovalent streptavidin with a single femtomolar biotin binding site. Nature Methods. 3 (4), 267-273 (2006).
- Allan, D., et al. soft-matter/trackpy: Trackpy v.0.5.0. Zenodo. , (2021).
- Weimann, L., et al. A quantitative comparison of single-dye tracking analysis tools using Monte Carlo simulations. PLoS ONE. 8 (5), 64287 (2013).
- Michalet, X. Mean square displacement analysis of single-particle trajectories with localization error: Brownian motion in an isotropic medium. Physical Review E - Statistical, Nonlinear, and Soft Matter Physics. 82 (4), 041914 (2010).
- Sygusch, J., Beaudry, D., Allaire, M. Molecular architecture of rabbit skeletal muscle aldolase at 2.7-A resolution. Proceedings of the National Academy of Sciences of the United States of America. 84 (22), 7846-7850 (1987).
- Heath, G. R., Scheuring, S. High-speed AFM height spectroscopy reveals µs-dynamics of unlabeled biomolecules. Nature Communications. 9 (1), 4983 (2018).
- Day, C. A., Kenworthy, A. K. Mechanisms underlying the confined diffusion of cholera toxin B-subunit in intact cell membranes. PLOS ONE. 7 (4), 34923 (2012).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados