Method Article
Rastreamento de partículas sensíveis à massa para caracterizar dinâmicas de macromolécula associadas à membrana
Neste Artigo
Resumo
Este protocolo descreve uma abordagem de processamento de imagem baseada em iSCAT e rastreamento de partículas únicas que permite a investigação simultânea da massa molecular e o comportamento difuso de macromoléculas interagindo com membranas lipídicas. Instruções passo a passo para preparação da amostra, conversão em massa para contraste, aquisição de filmes e pós-processamento são fornecidas juntamente com direções para evitar possíveis armadilhas.
Resumo
Interações de curta duração ou transitórias de macromoléculas em e com membranas lipídicas, uma interface onde uma infinidade de reações biológicas essenciais ocorrem, são inerentemente difíceis de avaliar com métodos biofísicos padrão. A introdução do rastreamento de partículas sensíveis à massa (MSPT) constitui um passo importante para uma caracterização quantitativa completa de tais processos. Tecnicamente, isso foi possível através do advento da microscopia de dispersão interferométrica (iSCAT) à base de fotometria em massa (MP). Quando a estratégia de remoção de fundo é otimizada para revelar o movimento bidimensional de partículas associadas à membrana, esta técnica permite a análise em tempo real tanto da difusão quanto da massa molecular de macromoléculas não rotuladas em membranas biológicas. Aqui, é descrito um protocolo detalhado para executar e analisar o rastreamento de partículas sensíveis à massa de sistemas associados à membrana. As medições realizadas em um fotômetro de massa comercial alcançam resolução de tempo no regime de milissegundos e, dependendo do sistema MP, um limite de detecção em massa até 50 kDa. Para mostrar o potencial do MSPT para a análise aprofundada da dinâmica da macromolécula catalisada por membrana em geral, são apresentados resultados obtidos para sistemas proteicos exemplares, como o interagedor de membrana nativa anexo V.
Introdução
Uma vez meramente percebidas como uma barreira contra a ampla gama de condições físicas ambientais, as membranas biológicas são hoje consideradas entidades funcionais e plataformas catalíticas 1,2. Com base em sua capacidade de localização, amplificação e sinais diretos em resposta a reações macromoléculas associadas à membrana, as interfaces lipídicas constituem um elemento crucial para uma grande variedade de processos celulares, como o tráfico de membranas e as cascatasde sinalização 3,4,5. Servindo como um local de nucleação para a montagem de complexos estáveis, o apego da membrana muitas vezes se baseia em um equilíbrio dinâmico entre formas de macromoléculas associadas à membrana e citotócidas e, portanto, é de natureza transitória 6,7.
Apesar de sua grande importância na biologia, até agora tem sido desafiador desenvolver métodos que possam fornecer acesso às heterogêneses composicionais, espaciais e temporais das reações macromoléculas associadas à membrana emtempo real 7,8. Para resolver os processos moleculares subjacentes, dois aspectos experimentais são decisivos: resolução de tempo suficiente e sensibilidade a partículas únicas. Portanto, técnicas médias de conjunto, como a recuperação da fluorescência após o fotobleaching (FRAP), mas também a espectroscopia de correlação de fluorescência muito mais sensível (FCS) têm limitações, uma vez que em grande parte descoplam informações espaciais da informação temporal9. Um passo importante para a caracterização da dinâmica individual das moléculas tem sido, portanto, o advento do rastreamento de partículas únicas (SPT) em combinação com microscopia altamente sensível. Em particular, duas abordagens SPT provaram ser eficazes nesse sentido. Em primeiro lugar, a utilização de fluoroforos como rótulos e os sistemas correspondentes de detecção de fluorescência abriu caminho para a precisão de nanômetros e a resolução de tempo milissegundo 10,11,12. Em segundo lugar, a detecção baseada em dispersão usando nanopartículas de ouro melhorou tanto a precisão de localização quanto a resolução de tempo para a faixa subnômetro e microssegundo, respectivamente 13,14,15,16. Apesar das muitas vantagens de ambas as abordagens e de suas contribuições significativas em relação à compreensão mecanicista dos sistemas associados à membrana17,18, ambas as técnicas têm sido até agora limitadas: exigem rotulagem das moléculas de interesse, o que potencialmente perturba seu comportamento nativo e são insensíveis à composição molecular de partículas associadas à membrana19,20.
Ambas as limitações foram recentemente superadas pela introdução de uma nova abordagem baseada em dispersão interferométrica (iSCAT) denominada fotometria em massa (MP)21,22,23. Esta técnica permite a determinação de distribuições em massa em solução de biomoléculas de acordo com seu contraste iSCAT ao pousar em uma interface de vidro. No entanto, para a detecção e caracterização de moléculas móveis difundidas em membranas lipídicas, uma abordagem de análise de imagem mais sofisticada teve que ser desenvolvida. Isso foi implementado com sucesso e permite detectar, rastrear e determinar a massa molecular de biomoléculas não rotuladas únicas difundidas em uma interface lipídica24,25. Chamada de fotometria de massa dinâmica ou rastreamento de partículas sensíveis à massa (TMT), essa técnica agora permite a avaliação de interações complexas de macromoléculas registrando diretamente mudanças na massa molecular das entidades rastreadas e, portanto, abre novas possibilidades para a análise mecanicista da dinâmica molecular associada à membrana.
Aqui, é apresentado um protocolo detalhado para preparação de amostras, imagens e o pipeline de análise de dados necessário para o MSPT. Em particular, são discutidos os requisitos amostrais e possíveis problemas que podem ocorrer durante a medição e análise. Além disso, o potencial incomparável de analisar sistemas macromoléculas que interagem por membrana é apresentado através de vários resultados representativos.
Protocolo
1. Preparação da amostra
- Geração de vesículas multilamellar (MLVs)
- Calcule a quantidade de lipídios dissolvidos com clorofórmio de acordo com a mistura lipídica desejada e o volume de suspensão necessário.
NOTA: Recomenda-se uma concentração final de vesícula de lipídios de 4 mg/mL para o tampão de ressuspensão (reação). - Pipeta o volume calculado de lipídios em um frasco de vidro de 1,5 mL usando pipetas de deslocamento positivas equipadas com pontas de vidro.
- Evaporar o solvente lipídico sob um fluxo fraco de nitrogênio e girar constantemente o frasco para garantir a distribuição igual dos lipídios nas paredes de vidro.
- Certifique-se de evaporação completa do solvente colocando o frasco sob um fluxo constante de nitrogênio por 15 minutos.
- Remova traços residuais de clorofórmio por secagem a vácuo em um dessecador a vácuo por mais uma hora.
- Reidratar a mistura lipídica no tampão de ressuspensão (reação) desejado e vórtice completamente a suspensão até que o filme lipídide tenha sido dissolvido das paredes do frasco.
NOTA: O tampão de reação deve garantir atividade e estabilidade da proteína. O tampão de reação utilizado neste estudo contém 50 mM Tris-HCl (pH = 7,5), 150 mM KCl e 5 mM MgCl2. Observe que qualquer tampão usado para diluir lipídios ou proteínas precisa ser filtrado para remover impurezas de partículas interferentes (ver passo 5).
- Calcule a quantidade de lipídios dissolvidos com clorofórmio de acordo com a mistura lipídica desejada e o volume de suspensão necessário.
- Geração de pequenos vesículos unilamellar (SUVs)
- Para ciclos consecutivos de congelamento da ressursão lipídica (passo 1.1.6), ferva 500 mL de água em um béquer em uma placa quente (entre 70 °C e 99 °C) e prepare um recipiente com nitrogênio líquido.
- Congelar o lipídio de ressuspensão no nitrogênio líquido. Transfira o frasco para o béquer com água quente até que a solução esteja completamente descongelada. Repita este ciclo de congelamento 8-10 vezes ou até que a mistura anteriormente turva pareça clara.
ATENÇÃO: Utilize roupas de segurança e equipamentos adequados, como óculos, luvas e pinças para evitar qualquer contato direto com o nitrogênio líquido, o frasco lipídico congelado ou a água fervente. - Para a geração de uma distribuição de vesícula monodisperse, monte um lipídio-extrudido e teste sua integridade com tampão de reação para garantir que ele não vaze.
NOTA: Se o vazamento for observado, rearquiba cuidadosamente o lipídio-extrudido até que nenhum derramamento de tampão seja evidente. - Extrude a suspensão lipídica para 37 passa por uma membrana nucleopore com um tamanho de poros de 50 nm26. O número de passes deve ser desigual para garantir que a mistura final do SUV cruze a membrana nucleopore e, portanto, esteja livre de agregados lipídes ou vesículas multilamellar. As vesículas extrudadas serão usadas mais tarde para formar bicamadas lipídicas suportadas (ver passos 6 e 7).
NOTA: Os SUVs também podem ser formados pela sônica da mistura lipídica rehidratada. No entanto, a preparação via extrusão proporciona uma distribuição mais monodisperse de SUVs, o que facilita a ruptura vesícula durante a formação de bicamadas lipídicas suportadas. Vesículas extrudadas podem ser armazenadas na geladeira por um máximo de 3 dias.
2. Limpeza de slides de microscópio
- Distribua um número igual de slides de microscópio (Nº 1,5; 0,17 mm de espessura) com dimensões de 24 mm x 60 mm e 24 mm x 24 mm em porta-microscópios politetrafluortileno (PTFE).
- Transfira os titulares do PTFE em béquers contendo água ultrapura e sonicá-los por 15 minutos a temperatura ambiente.
NOTA: Dependendo do béquer, o volume de água precisa ser ajustado para cobrir completamente o titular do PTFE. - Use pinças para remover os suportes do béquer e substitua a água por isopropanol ultrauso. Insira o suporte no béquer contendo isopropanol e sonicato novamente por 15 minutos.
NOTA: Dependendo do béquer, o volume do isopropanol precisa ser ajustado para cobrir completamente o titular do PTFE. - Substitua o isopropanol por água ultrauso e sonicar o béquer contendo os suportes por 15 minutos.
- Remova os suportes de PTFE dos béquers e seque os slides do microscópio no suporte sob um fluxo constante de gás nitrogênio ou ar comprimido.
NOTA: Certifique-se de limpeza adequada dos slides de cobertura usando luvas, béquers limpos e filme de parafina para cobrir cada béquer. Caso contrário, a poeira residual pode causar flutuações significativas de fundo durante as medições do MSPT.
3. Hidrofilização de lâminas de microscópio
NOTA: Para obter uma bicamada lipídica homogênea e fluida, a hidrofilização dos slides é essencial e deve ser realizada pouco antes da montagem da câmara de fluxo.
- Coloque os suportes de PTFE contendo apenas 24 mm x 60 mm de microscópio desliza em um limpador de plasma com oxigênio como gás de processo e limpe os slides do microscópio com plasma (parâmetros utilizados neste trabalho: 30% de potência, pressão de oxigênio de 0,3 mbar para 30 s; consulte Tabela de Materiais para obter detalhes do limpador de plasma usado).
NOTA: Para obter membranas fluidas, parâmetros de limpeza plasmática, como energia, pressão de oxigênio e tempo de limpeza devem ser ajustados para cada instrumento. Para isso, recomenda-se o uso de lipídios fluorescentes rotulados para garantir a fluidez da membrana, que pode ser quantificada com recuperação de fluorescência após experimentos de fotobleaching (FRAP)27. Se os parâmetros não forem otimizados para a respectiva configuração, a difusão da membrana pode ser prejudicada devido à redução da fluidez da membrana.
4. Montagem de câmaras de fluxo
- Antes da montagem da câmara de fluxo, mantenha os seguintes componentes prontos: lâminas de microscópio limpos (24 mm x 24 mm), lâminas de microscópio hidrofilizado (24 mm x 60 mm), papel alumínio, papelão plano, bisturi e fita dupla face.
- Enrole o papelão plano com papel alumínio.
- Espalhe os slides de microscópio de 24 mm x 24 mm limpos na folha de alumínio com distância suficiente entre si.
- Conecte tiras de fita de dois lados nas bordas superior e inferior dos slides.
- Extirpar cada slide de microscópio com o bisturi, de talto que possa ser removido da folha de alumínio. Como resultado, cada slide deve ter listras de fita dupla face anexadas às bordas superior e inferior do slide (ver Figura 1).
- Conecte o slide de 24 mm x 24 mm com as duas tiras de fita de dupla face ao slide hidrofilizado de 24 mm x 60 mm para formar um caminho de fluxo entre os slides do microscópio menor e maior.
NOTA: Para garantir câmaras de fluxo limpas, use constantemente luvas e certifique-se de que a bancada esteja livre de poeira.
5. Filtragem de buffers de reação
- Filtro estéril todos os buffers de reação através de membranas de acetato de celulose de 0,45 μm para garantir o mínimo de sinal de fundo durante as medições de MSPT.
NOTA: Se a presença de nucleotídeos, como o ATP, é essencial para um experimento bem-sucedido, esteja ciente de um potencial aumento no sinal de fundo. Recomenda-se usar apenas quantidades mínimas que ainda garantam a atividade proteica.
6. Formação de bicamada lipídica suportada (SLB)
NOTA: Recomenda-se realizar a formação de bicamadas lipídicas suportadas no fotômetro de massa para garantir visualmente a propagação de vesículas bem sucedidas e a remoção completa de vesículas não afusadas.
- Diluir SUVs recém-extrudados (ver passo 1 para mais detalhes) a uma concentração final de 0,4 mg/mL no buffer de reação necessário. Opcionalmente, para promover a ruptura vesícula, adicione 2 mM CaCl2 à suspensão vesícula.
NOTA: Os cations divalent podem causar a agregação de alguns lipídios, como o PiP2. Para misturas contendo tais lipídios, evite usar CaCl2 para a promoção de ruptura vesícula ou outros cáings divalent no tampão de resuspensão. Se necessário para o experimento, os cations divalent podem ser adicionados após a formação bem sucedida do bicamadorídeo lipíduo suportado. - Lave 50 μL da suspensão da vesícula na câmara de fluxo (passo 4) e incubar a câmara por 2 min.
NOTA: Tampões, vesículas ou soluções proteicas podem ser lavadas através da câmara de fluxo com um pequeno pedaço de tecido de imersão. No entanto, também é possível usar um sistema de bomba mecânica. - Remova vesículas não afundadas através de lavagem repetida (pelo menos três vezes) da câmara de fluxo com 200 μL do buffer de reação cada vez.
NOTA: As vesículas precisam ser completamente lavadas para fora da câmara de fluxo para garantir um sinal de fundo estável durante as medições do MSPT.
7. Geração de curva de calibração
NOTA: Para converter o contraste das partículas detectadas em massa molecular, seu sinal precisa ser calibrado usando proteínas de tamanhos conhecidos. Recomenda-se ajustar o regime padrão de tamanho da proteína para cobrir a gama de massas moleculares esperadas para o sistema de interesse.
- Biotintilação de proteínas padrão com resíduo de cisteína
- Calcule a quantidade apropriada de maleimida-biotina para a proteína padrão de acordo com as instruções do fabricante.
- Incubar a proteína padrão com o volume determinado de maleimida-biotina por 1 h à temperatura ambiente.
- Para remover maleimida-biotina não conjugada do complexo biotin-proteína conjugado, realize cromatografia de exclusão de tamanho em uma coluna adequada para a proteína de interesse.
- Determine a concentração de proteínas usando um Ensaio de Bradford.
NOTA: Para armazenar a proteína padrão para novas medidas, congele a proteína em alíquotas de uso único em nitrogênio líquido e armazene-as a -80 °C.
- Medição de proteínas padrão para a curva de calibração
- Em uma câmara de fluxo, prepare uma bicamada lipídica suportada com SUVs extrudados de 0,4 mg/mL (ver passos 1 e 6 para mais detalhes) contendo 0,01 mol% (v/v) Biotinyl Cap PE (1,2-dioleoyl-sn-glycero-3-phosphoethanolamina-N-biocloin).
- Adicione 50 μL de 2,5 nM divalent streptavidin à bicamadas na câmara de fluxo e incubar por 10 minutos.
NOTA: Divalent streptavidin foi expressa e purificada como descrito em Howarth et al.28. Tetravalent streptavidin também pode ser usado. No entanto, o uso de streptavidina divalent pode reduzir a possível reação entre lipídios biotinilados e proteínas padrão que são conjugadas a uma moiety biotina, a fim de facilitar a atribuição de espécies. - Remova o streptavidin divalent desvinculado com 100 μL de tampão de reação.
- Adicione 50 μL de proteína padrão biotina de 100 nM conjugada à bicamada na câmara de fluxo e incubar por 2 min.
NOTA: Dependendo da eficiência da biotintilação e se a estreptavidina di ou tetravalente é usada, as concentrações ideais de proteína padrão biotina-conjugada e streptavidina podem variar. - Realize a medição do MSPT de acordo com os detalhes descritos na etapa 8.
ATENÇÃO: As condições de imagem precisam ser idênticas tanto para os padrões de amostra quanto para a calibração.
8. Imagem
- Formação de SLB e preparação de amostras
- Como descrito em mais detalhes na etapa 6, introduza os SUVs da mistura lipídica desejada (25 μL) na câmara de fluxo de amostra e forme uma bicamada lipídica suportada. Lave bem a câmara (três vezes) com 100 μL de tampão de reação para remover todas as vesículas não afusadas.
- Adicione 50 μL da proteína de interesse à câmara de amostra.
NOTA: Como o MSPT é um método de partícula única, a concentração de proteínas deve ser mantida na faixa pM a nM para permitir a detecção e rastreamento de partículas não perturbadas.
- Aquisição de vídeo
- Defina as condições de imagem desejadas, como o tamanho do campo de visão (FOV), taxa de quadros, tempo de exposição e tempo de aquisição no software de aquisição.
NOTA: As seguintes configurações provaram funcionar para o MSPT em um fotômetro de massa comercial (ver Tabela de Materiais): FOV de 128 pixels x 35 pixels, uma taxa de quadros de 1 kHz resultando em cerca de 200 quadros por segundo após a média subsequente de 5 vezes e um tempo de exposição de 0,95 ms. - Ajuste o foco automaticamente ou manualmente. Se necessário, mova o FOV para uma posição com uma membrana homogênea usando o controle lateral.
- Crie uma pasta de projeto e comece a gravar o filme. Após a conclusão da gravação, especifique um nome de arquivo na caixa de diálogo solicitada pelo software de aquisição. O filme é então automaticamente salvo na pasta do projeto como um arquivo MP para análise subsequente.
NOTA: Registo pelo menos três réplicas em diferentes câmaras de fluxo para garantir a integridade das membranas individuais e a reprodutibilidade dos resultados. A duração do filme pode ser definida com antecedência e depende do tipo de experimento. Na maioria dos casos, recomenda-se um tempo de aquisição entre 5 min e 7 min.
ATENÇÃO: Por padrão, gravações de filmes no software comercial de aquisição de fotômetros em massa são compactadas antes de serem salvas para reduzir o espaço de armazenamento. No entanto, a compactação de arquivos precisa ser desligada para permitir a análise personalizada de dados, conforme descrito neste protocolo. Detalhes sobre como desativar a compactação de arquivos podem ser encontrados no manual do usuário do fabricante.
- Defina as condições de imagem desejadas, como o tamanho do campo de visão (FOV), taxa de quadros, tempo de exposição e tempo de aquisição no software de aquisição.
9. Análise de dados
NOTA: O pipeline de análise de dados é acompanhado por dois notebooks Jupyter interativos (análise MSPT.ipynb, Visualization.ipynb). Os notebooks Jupyter e os módulos Python personalizados necessários para realizar a análise MSPT descrita abaixo estão disponíveis em um repositório público: https://github.com/MSPT-toolkit/MSPT-toolkit. Para obter instruções detalhadas sobre a análise abaixo, os leitores são encaminhados para análise do MSPT.ipynb acessado usando o link acima.
- Processamento de vídeo
- Remova a dispersão estática dominante da luz com o algoritmo de estimativa de fundo em termos de pixel usando a função image_processing.mp_reader .
- Para aplicar a remoção de fundo, escolha a opção continuous_median para o modo parâmetro e defina um comprimento apropriado para a janela mediana deslizante (window_length) na seção de notebook B.1. Opcionalmente, salve os filmes após a remoção de fundo para ser usado para detecção de partículas e vinculação de trajetória (definindo o parâmetro save_processed_movies para True).
NOTA: Ajuste o tamanho da janela (window_length) para valores entre 101 e 2001, dependendo da densidade de partículas na membrana, do coeficiente de difusão esperado, da taxa de quadro de aquisição e da velocidade de processamento necessária.
ATENÇÃO: A estratégia de remoção de fundo funciona bem se a membrana não estiver muito densamente embalada e se a difusão das partículas for suficientemente rápida (ou seja, cada pixel é na maior parte do tempo não ocupado com uma partícula). Caso contrário, o contraste das partículas será sistematicamente subestimado, pois elas não podem ser adequadamente distinguidas do sinal de fundo. Isso pode ser compensado aumentando o tamanho médio da janela ao custo da velocidade computacional. No entanto, esteja ciente de que a configuração do tamanho da janela muito grande pode influenciar negativamente a saída devido à deriva da amostra. Uma inspeção visual dos vídeos processados é crucial.
- Para aplicar a remoção de fundo, escolha a opção continuous_median para o modo parâmetro e defina um comprimento apropriado para a janela mediana deslizante (window_length) na seção de notebook B.1. Opcionalmente, salve os filmes após a remoção de fundo para ser usado para detecção de partículas e vinculação de trajetória (definindo o parâmetro save_processed_movies para True).
- Detecte partículas e sua respectiva posição ao longo do filme usando a função particle_fitting.particle_fitter (ver seção de caderno B.2).
- Sintonize a sensibilidade da detecção de partículas com o parâmetro limiar (thresh; consulte a seção de caderno B.1), que é usada para destacar pontos de candidatos por binarização de imagem. O efeito de parâmetros de limiar variados na sensibilidade à detecção de manchas pode ser examinado em um notebook separado (Visualização de filme.ipynb). Os resultados da detecção de partículas são automaticamente salvos em arquivos CSV em um subdiretório do arquivo de filme.
NOTA: A definição do parâmetro de limiar arbitrariamente baixa (por exemplo, para filmes feitos com o fotômetro de massa usado, um parâmetro limiar abaixo de 0,0005) não é recomendada, pois as manchas de candidato serão dominadas por ruídos espúrios e, portanto, prolongar o tempo de processamento.
- Sintonize a sensibilidade da detecção de partículas com o parâmetro limiar (thresh; consulte a seção de caderno B.1), que é usada para destacar pontos de candidatos por binarização de imagem. O efeito de parâmetros de limiar variados na sensibilidade à detecção de manchas pode ser examinado em um notebook separado (Visualização de filme.ipynb). Os resultados da detecção de partículas são automaticamente salvos em arquivos CSV em um subdiretório do arquivo de filme.
- Remova a dispersão estática dominante da luz com o algoritmo de estimativa de fundo em termos de pixel usando a função image_processing.mp_reader .
- Vincule partículas em quadros consecutivos em trajetórias usando o trackpy do pacote Python (v.0.5.0)29.
NOTA: A ligação de trajetória é realizada na mosca após a detecção do local. Como resultado, um arquivo CSV adicional contendo as informações de trajetória é armazenado em um subdiretório do arquivo CSV de detecção de partículas.- Remova as trajetórias com poucos pontos usando o parâmetro minimum_trajectory_length (ver seção de caderno B.1) para permitir uma determinação robusta dos coeficientes de difusão. Para obter explicações detalhadas sobre os outros parâmetros das funções trackpy , consulte a documentação do trackpy .
- Análise de trajetória
- Na seção de notebook C.1, especifique a taxa de quadros (frame_rate) e o tamanho do pixel em nm (pixel_size), que foram usados para aquisição de filmes. Crie uma lista de arquivos CSV contendo as informações de trajetória devolvidas pelo trackpy (ver etapa 9.2) com a função trajectory_analysis.get_csv_files (seção de notebook C.2).
- Além disso, especifique um nome de arquivo de saída para o recipiente HDF5, que é usado para armazenar os resultados de montagem em disco (seção notebook C.3). Analise todas as trajetórias com a função trajectory_analysis.fit_trajetórias na seção de notebook C.4, que itera através da lista de arquivos CSV. Esta função utiliza a distribuição de distância de salto (JDD)30 e a análise de deslocamento quadrado médio (MSD)31 para estimar o coeficiente de difusão de cada trajetória.
- Converta o contraste mediano de cada trajetória em sua massa correspondente utilizando a relação de massa de contraste obtida a partir da calibração MSPT (ver seção 7). Especifique a inclinação (inclinação) e a interceptação y (offset) da linha de calibração, que relaciona o contraste iSCAT com a massa molecular (função trajectory_analysis.apply_calibration; ver seção de notebook C.5). Esta função adiciona uma coluna com a massa mediana da trajetória a cada quadro de dados.
- Avalie a densidade de partículas aparente na membrana com a função trajectory_analysis.membrana_densidade, que retorna o valor médio de densidade em termos de partículas detectadas e trajetórias presentes durante cada quadro (ver seção notebook C.6) como colunas adicionais no quadro de dados.
NOTA: Como uma porção de partículas será perdida durante o processo de detecção e conexão de trajetória, as densidades reais de partículas podem ser maiores. Para obter resultados confiáveis em relação às densidades de partículas, bem como histogramas em massa, inspecione visualmente instantâneos de filmes representativos para verificar se as condições de medição são plausíveis para o rastreamento de partículas únicas (ver passo 9.1.1).
10. Visualização de dados
- Ilustre a correlação do coeficiente de massa e difusão com a estimativa de densidade do kernel bidimensional (KDE), que é baseada no fastkde do pacote Python (v.1.0.19; https://pypi.org/project/fastkde/).
- Para gerar o plot, especifique o arquivo HDF5 contendo os resultados do MSPT (ver etapa 9.3.2 e seção notebook D.1) e selecione um único (seção de notebook D.2) ou um quadro de dados concatenado (seção notebook D.3) como dados de entrada para a função plotting.generate_2D_KDE (seção de notebook D.4).
NOTA: Cada conjunto de dados plotado deve conter idealmente mais de 1.000 trajetórias para um 2D-KDE confiável.
Resultados
Seguindo o protocolo detalhado aqui para a preparação de bicamadas lipídicas suportadas (SLBs) em câmaras de fluxo (Figura 1), pode-se reconhecer claramente um padrão semelhante a manchas na visão nativa de todas as condições exibidas (Figura 2). Este efeito é causado pela rugosidade superficial do vidro, que geralmente domina o sinal de dispersão e leva a condições visualmente indistinguíveis (vidro, vidro com SLB, ou vidro com SLB e proteínas anexadas). A presença de vesículas, no entanto, é claramente distinta devido à grande seção transversal de dispersão das vesículas e permite a observação da ruptura e fusão de vesículas em membranas homogêneas (Figura 2B e Filme Suplementar 1). Ao remover o sinal de dispersão estática da superfície do vidro com uma abordagem ratiométrica que enfatiza os elementos dinâmicos dentro do campo de visão24,25, pode-se descobrir proteínas sem rótulo difundindo na membrana (Figura 2D) enquanto um SLB vazio (Figura 2C) ou o próprio vidro (Figura 2A) aparece como uma imagem barica.
O fundo inerente das medições de MSPT pode ser estimado localmente dividindo cada valor de pixel através da mediana de n precedentes e pixels sucessores do filme na mesma posição de imagem (Figura 3). Como resultado, macromoléculas aparecem como funções isotropicais de propagação de pontos (PSFs) cujo movimento na membrana pode ser observado, rastreado e quantificado. De fato, a disponibilidade tanto de contraste quanto de comportamento dinâmico permite a relação direta do tamanho molecular de uma partícula com seu respectivo comportamento difusivo, tudo sem a necessidade de rotular a partícula. No entanto, para interpretar o contraste iSCAT determinado durante os experimentos do MSPT, é essencial realizar uma calibração que traduz a amplitude do sinal em massa molecular. Isso pode ser conseguido anexando biomoléculas de massa conhecida a um SLB através de um complexo biotina-streptavidin-biotina (Figura 4A). Como estratégia exemplar, pode-se usar variantes biotinilatas de gróbios bovinos albumina (BSA), proteína A (prA), fosfatase alcalina (AP) e fibronectina (FN), que se ligam ao streptavidina (STP) que em si está ligado a lipídios contendo biotina (Biotinyl Cap PE) na membrana. Como mostrado na Figura 4A, o contraste cada vez mais pronunciado dessas macromoléculas exemplares reflete o aumento do peso molecular dos respectivos padrões biotinilados. Ao atribuir cada pico dos histogramas de contraste (Figura 4B) à massa correspondente do estado oligômero da proteína padrão, uma relação linear entre contraste e massa é revelada21,22 e pode ser posteriormente usada para a análise de sistemas macromolecule desconhecidos (Figura 4C).
Um bom exemplo demonstrando a aplicabilidade e as capacidades do MSPT para analisar pesos moleculares e, portanto, estudar estados oligômeros e eventos de oligomerização é a consideração da aldolase biotinilada e do IgG biotinilado (Figura 5). Aldolase é comumente relatado como um homotetramer32. No entanto, a distribuição em massa resolvida pelo MSPT apresenta quatro picos distintos, o que destaca a presença de múltiplas populações (Figura 5A). Enquanto o primeiro pico menor corresponde a streptavidina desocupado e pode ser esperado devido à configuração neste tipo de experimento, complexos de aldolase com apenas duas subunidades (2SU) ou seis subunidades (6SU) também podem ser detectados (Figura 5B). Curiosamente, os complexos tetra e hexameric aldolase-streptavidin apresentam um coeficiente de difusão reduzido quando comparados apenas à adolase dimérica e streptavidin, indicando um aumento do arrasto viscoso, por exemplo, através da fixação de um segundo lipídio biotinilado ao streptavidin. Da mesma forma, o IgG biotinylated exibe três picos na distribuição em massa, com o primeiro pico novamente correspondendo à massa de um único streptavidin. A massa do pico mais abundante corresponde à massa de uma cadeia leve e uma pesada (1SU), ou seja, metade de um anticorpo IgG. O anticorpo completo com duas metades idênticas (2SU) é detectado em cerca de 11% dos casos. A diminuição do coeficiente de difusão com tamanhos complexos crescentes indica interações do streptavidina com mais de um lipídio biotinilado ou arrasto adicional causado pelo IgG anexado, ou ambos.
Além da análise única dos estados oligômeros dependentes da membrana, o MSPT também confere a vantagem particular de correlacionar o comportamento difuso de uma macromolécula de interesse com seu estado oligômero. Os resultados representativos para este tipo de análise são mostrados para a subunidade de toxina de cólerina V (AnV) e cólera toxina B (CTxB), que se ligam à dioleoylphosphatidylserine (DOPS) ou glicosfingolipids (GM1), respectivamente, incorporadas na membrana (Figura 6A). Ambas as estimativas de densidade do kernel (KDEs) apresentam distribuições unimodais de massa e difusão, indicando uma única espécie abundante com comportamento difusivo semelhante. A posição máxima do coeficiente de massa molecular e difusão foi de 49,8 ± 2,2 kDa e 1,4 ± 0,1 μm2/s, respectivamente, para AV, bem como 62,7 ± 3,1 kDa e 0,4 ± 0,1 μm2/s, respectivamente, para CTxB. Os coeficientes de difusão medidos são comparáveis aos valores relatados anteriormente obtidos a partir de AFM de alta velocidade e FRAP33,34. A massa ligeiramente reduzida em comparação com a massa da macromolécula esperada (52 kDa para um aparador de AnV, 65 kDa para um pentamer CTxB) pode indicar a presença de complexos menores com menos subunidades no conjunto. Embora a diferença de massa entre as proteínas seja pequena e próxima do limite de detecção especificado do microscópio (≈50 kDa), seus coeficientes de difusão diferem consideravelmente. Em uma mistura equimolar, por exemplo, comparando a difusão da mistura apenas à distribuição de AnV e CTxB, pode-se concluir que o AnV é mais abundante na membrana do que o CTxB (Figura 6B). No entanto, se a concentração de CTxB for dobrada em comparação com a concentração de GNV, o equilíbrio é deslocado para CTxB como a proteína predominante na membrana. Como ilustrado para misturas de AnV e CTxB, o MSPT não só permite discriminar macromoléculas associadas à membrana de acordo com seu peso molecular, mas também permite a discriminação de diferentes populações macromoléculas de acordo com seu comportamento difusivo.
Como em todas as técnicas de microscopia, alguns requisitos experimentais são cruciais para alcançar a qualidade desejada dos dados. Um exemplo importante neste contexto são as coberturas completamente limpas. Em geral, este é considerado um pré-requisito para experimentos de molécula única relacionadas à microscopia, mas o MSPT é particularmente sensível às impurezas amostrais. O aumento da dispersão originária da superfície de vidro de tampas impuras impede qualquer medição quantitativa do iSCAT. Notavelmente, mesmo partículas residuais de sujeira ou poeira em vidro insuficientemente limpo podem causar notáveis distorções de imagem, reconhecíveis como pontos brilhantes no modo de imagem nativo (Figura 7A). Embora esses defeitos sejam removidos pela estimativa de fundo devido à sua natureza estática, a determinação precisa do contraste de uma partícula pode ser prejudicada e, portanto, influencia negativamente sua análise quantitativa. Outro problema comum encontrado nos experimentos do MSPT são as vesículas remanescentes que flutuam (cercadas em laranja) pelo campo de visão ou vesículas não fundidas que estão presas (cercadas em azul) em uma posição específica na membrana e aparecem como grandes dispersores pulsantes (Figura 7B). Para minimizar sua ocorrência e interferência na aquisição de filmes, recomenda-se lavar bem o SLB antes de adicionar a proteína e usar misturas recém-preparadas de pequenas vesículas unilamellar (SUVs) e cáções divalentes.
Um fator que deve ser levado em conta para o projeto de experimentos de rastreamento de partículas sensíveis à massa é a densidade de macromoléculas associadas à interface da membrana. Altas densidades de partículas na membrana podem de fato causar problemas por duas razões: i) A ligação de detecções de partículas de quadros consecutivos em trajetórias torna-se ambígua e, portanto, aumenta a probabilidade de erros e coeficientes de difusão mal julgados. ii) A massa de partículas, que é extraída da amplitude de seu ajuste PSF correspondente, torna-se sistematicamente subestimada e os picos de massa se ampliam porque a separação do sinal de fundo estático do sinal de partícula dinâmica é cada vez mais difícil (Figura 7C). Atualmente, a avaliação visual da qualidade dos dados no processo de aquisição de vídeos MSPT é difícil nos microscópios comerciais disponíveis porque a visão racionada implementada no software de aquisição está usando a remoção de fundo estabelecida para fotometria em massa21 em vez do algoritmo baseado em mediana descrito aqui e nas referências24,25 (Figura 7D ). A remoção contínua de fundo baseada em média usada para visualizar moléculas de pouso na fotometria em massa faz com que partículas de difusão apareçam como frentes escuras com caudas brilhantes, o que faz com que as manchas pareçam altamente anisotrópicos e interfere com o encaixe do PSF durante o procedimento de detecção. Assim, o uso do processamento de imagem baseado em média implementado no software de aquisição é inadequado para a análise de biomoléculas difusas em membranas.
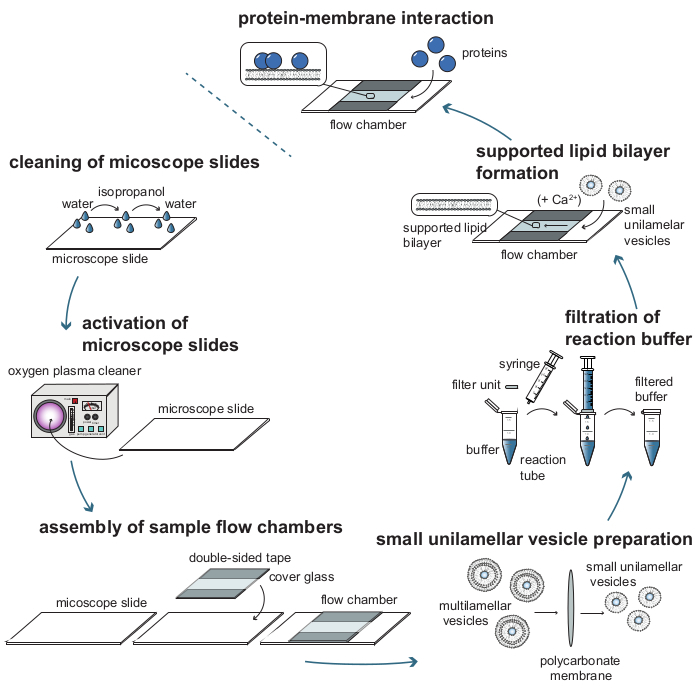
Figura 1: Diagrama de fluxo de processo das etapas individuais necessárias para analisar as interações proteína-membrana com rastreamento de partículas sensíveis à massa (MSPT). Para preparar amostras para as medições do MSPT, as lâminas de cobertura de vidro devem ser completamente limpas e ativadas com um plasma de oxigênio. Após sua montagem em câmaras de fluxo de amostra, pequenas vesículas unilamellar (SUVs) são preparadas para a formação de bicamadas lipídicas suportadas (SLB) e todos os buffers de reação são filtrados para reduzir a dispersão de fundo. Os SUVs são adicionados para formar bicamadas lipídicas nas câmaras de fluxo. Opcionalmente, cations divalent, como íons Ca2+ podem ser adicionados aos SUVs para promover a ruptura vesícula. Por fim, baixas concentrações da proteína de interesse são lavadas na câmara de reação. Clique aqui para ver uma versão maior desta figura.
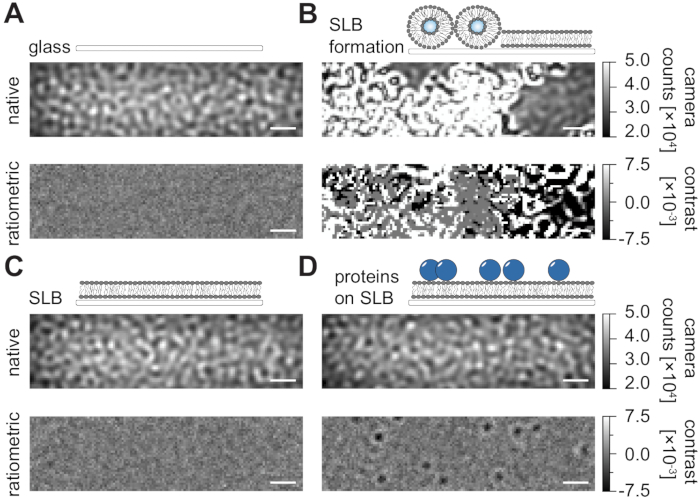
Figura 2: Visão nativa e racionmétrica de superfícies exemplares relevantes para as medições de TMPT. Imagens representativas da rugosidade superficial de um slide de tampa de vidro (A), durante a formação de uma bicamada lipídica suportada (B), com uma bicamada lipídica intacta (C) e de proteínas exemplares reconstituídas em um SLB (D). Todos os quatro exemplos são exibidos no modo nativo, que pode ser acessado durante a medição em si, e como imagens racionadas processadas após a remoção de fundo baseada na mediana. As barras de escala representam 1 μm. Para análise de dados (ver notebook Jupyter acompanhado; etapa 9), foram utilizados os seguintes parâmetros: tamanho médio da janela (window_length) = 1001. Clique aqui para ver uma versão maior desta figura.
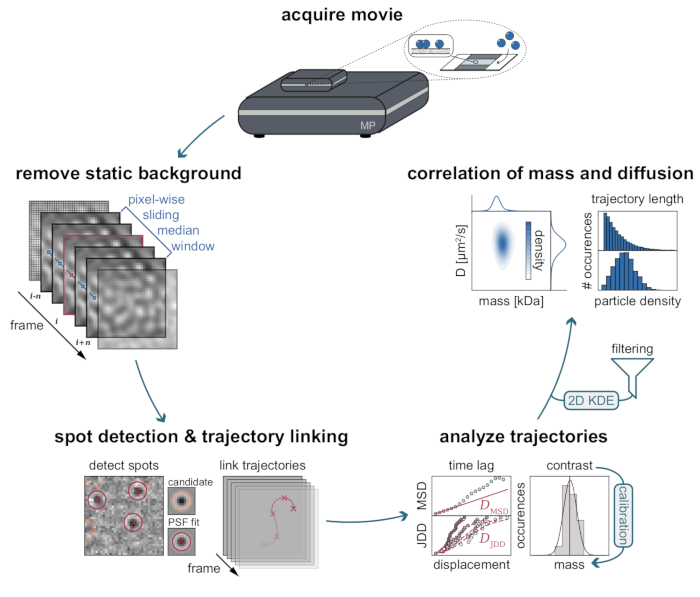
Figura 3: Diagrama passo a passo das etapas necessárias para coleta e análise de dados do MSPT. Após a aquisição de dados para a amostra de interesse no fotômetro de massa, os filmes são processados para remover o fundo estático através de uma abordagem mediana deslizante em termos de pixels. Posteriormente, as partículas candidatas são identificadas e montadas por uma função de propagação de ponto (PSF) antes de sua ligação em trajetórias de partículas. Para permitir a determinação do coeficiente de difusão para cada partícula, é utilizada a análise de deslocamento quadrado médio (MSD) ou distribuição de distância de salto (JDD). Nesta fase, os valores de contraste podem ser transformados em massas moleculares de acordo com a relação contraste-massa determinada através da estratégia de calibração. Como etapa final, as trajetórias podem ser filtradas com base em seu comprimento ou densidade de partículas de membrana e visualizadas pela estimativa de densidade bidimensional do kernel (2D-KDE). Clique aqui para ver uma versão maior desta figura.
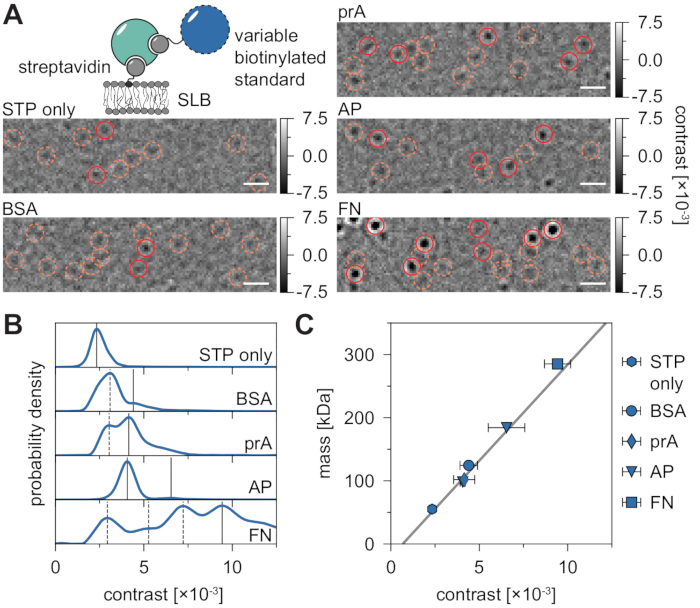
Figura 4: Calibração da relação massa-contraste para medições de MSPT. (A) Quadros métricos de razão representativa obtidos para complexos proteicos padrão-proptavidin exemplares difundidos em uma bicamada lipídica suportada contendo uma pequena porcentagem de lipídios biotinilados (DOPC:DOPG:Biotinyl Cap PE razão de 70:29.99:0.01). Como modelo de padrões de peso molecular, a monovalente streptavidin28 (somente STP) ou a estreptavidinadivalenta 28 em complexo com albumina de soro bovino biotinína (BSA), proteína biotinilada A (prA), fosfatase alcalina biotinilada (AP) ou fibronectina biotinilada (FN) são mostradas. As manchas de candidatos são destacadas em laranja (círculos tracejados) e detecções de partículas bem sucedidas são destacadas em vermelho (círculos sólidos). As barras de escala representam distribuições de densidade de probabilidade de 1 μm. (B) de valores de contraste obtidos para as cinco proteínas padrão modelo. Todos os dados apresentados representam distribuições agrupadas de três experimentos independentes por condição: STP apenas n = 82.719; BSA n = 9.034; prA n = 22.204; AP n = 69.065, e FN n = 71.759 trajetórias. Em comparação com os números de partículas determinados para membranas com proteínas, o número de partículas detectadas em uma bicamada vazia é insignificante em densidades de membrana moderada (Figura Suplementar 1). Os picos de contraste considerados para a calibração de massa são marcados através de linhas contínuas, enquanto os tracejados representam estados oligômeros não considerados. (C) Curva de calibração contraste-a-massa derivada de contrastes de pico no painel D e nas respectivas massas sequenciais dos complexos. As barras de erro exibem o erro padrão dos locais de pico estimados pelo bootstrapping (100 resamples com 1.000 trajetórias cada). Para análise de dados (ver notebook Jupyter; etapa 9), foram utilizados os seguintes parâmetros: tamanho médio da janela (window_length) = 1.001 quadros, limiar de detecção (thresh) = 0,00055, alcance de pesquisa (dmax) = 4 pixels, memória (max_frames_to_vanish) = 0 quadros, comprimento mínimo da trajetória (minimum_trajectory_length) = 7 quadros (somente STP), 9 quadros (BSA/FN), 15 quadros (prA), 10 quadros (AP). Clique aqui para ver uma versão maior desta figura.
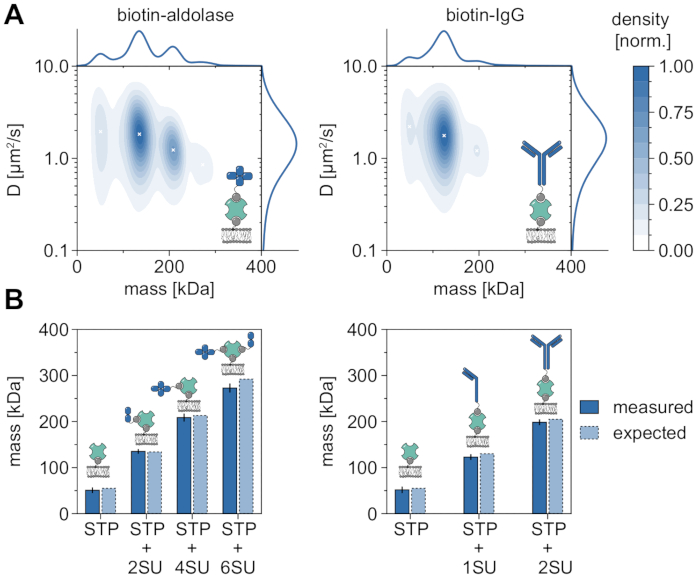
Figura 5: Decifrando estados oligômeros de proteínas associadas à membrana. (A) Estimativas de densidade de kernel 2D de massa e coeficiente de difusão de estreptavidina tetravalente em complexo com aldolase biotiningada (painel esquerdo) ou com um anticorpo IgG de cabra modificado por biotina (painel direito). A reconstituição de ambos os complexos foi realizada em uma bicamada lipídica suportada contendo DOPC, DOPG e Biotinyl Cap PE em uma razão de 70:29.99:0.01 mol%, respectivamente. No total, foram incluídas 116.787 trajetórias de três réplicas independentes para o complexo streptavidin-aldolase (densidade de partículas de 0,1 μm-2) e 348.405 para o composto streptavidin-IgG (densidade de partículas de 0,1 μm-2). Apenas partículas com comprimento de pista de pelo menos cinco quadros foram incluídas. São apresentadas distribuições marginais de probabilidades tanto de massa molecular (superior) quanto de coeficiente de difusão (à direita). O x preto em ambos os painéis marcam as respectivas máximas locais do KDE. (B) Comparação de massas oligômeras determinadas para o complexo de estreptavidina tetravalente com aldolase modificada por biotina (painel esquerdo) ou IgG biotinilado (painel direito) com, de acordo com as massas sequenciais, pesos moleculares esperados. A abreviação SU é introduzida em nome da subunidade da proteína dos interesses. As barras de erro exibem o erro padrão dos locais de pico estimados pelo bootstrapping (100 resamples com 1.000 trajetórias cada). Para análise de dados (ver notebook Jupyter acompanhado; etapa 9), foram utilizados os seguintes parâmetros: tamanho médio da janela (window_length) = 1.001 quadros, limiar de detecção (thresh) = 0,00055, alcance de pesquisa (dmax) = 4 pixels, memória (max_frames_to_vanish) = 0 quadros, comprimento mínimo da trajetória (minimum_trajectory_length) = 5. Clique aqui para ver uma versão maior desta figura.
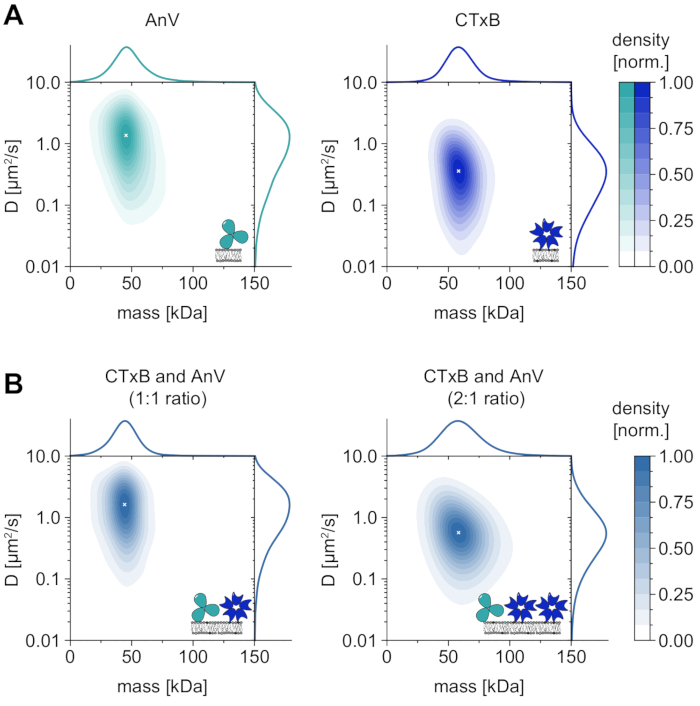
Figura 6: Dissolvendo o comportamento difusivo das proteínas nativas que interagem membrana anexam V (AnV) e subunidade de toxina de cólera B (CTxB). (A) estimativas de densidade de kernel 2D tanto de massa quanto de coeficiente de difusão do anexo V (painel esquerdo) e subunidade de toxina de cólera B (painel direito). Para a reconstituição da membrana AnV e CTxB, foram utilizadas composições lipídicas de 80:20 mol% DOPC para DOPS e 99,99:0,01 mol% DOPC para GM1, respectivamente. No total, foram incluídas 206.819 trajetórias de três réplicas independentes para AV (densidade de partículas de 0,1 μm-2) e 142.895 trajetórias para CTxB (densidade de partículas de 0,2 μm-2). (B) Estimativas de densidade do kernel 2D das misturas CTxB e AnV em uma razão de 1:1 (painel esquerdo) ou 2:1 (painel direito), respectivamente. A reconstituição de misturas proteicas foi realizada em uma bicamada lipídica suportada contendo lipídios DOPC, DOPS e GM1 em uma razão de 80:19.99:0.01 mol%. No total foram incluídas 42.696 trajetórias de três réplicas independentes para a mistura 1:1 (densidade de partículas de 0,1 μm-2) e 264.561 trajetórias para a razão 2:1 (densidade de partículas de 0,3 μm-2). Para ambos (A) e (B), foram incluídas apenas partículas com comprimento de faixa de pelo menos cinco quadros. São apresentadas distribuições marginais de probabilidades tanto de massa molecular (superior) quanto de coeficiente de difusão (à direita). O x branco em cada painel marca o respectivo máximo global do KDE. Para análise de dados (ver notebook Jupyter acompanhado; etapa 9), foram utilizados os seguintes parâmetros: tamanho médio da janela (window_length) = 1.001 quadros, limiar de detecção (thresh) = 0,00055, alcance de pesquisa (dmax) = 4 pixels, memória (max_frames_to_vanish) = 0 quadros, comprimento mínimo da trajetória (minimum_trajectory_length) = 5. Clique aqui para ver uma versão maior desta figura.
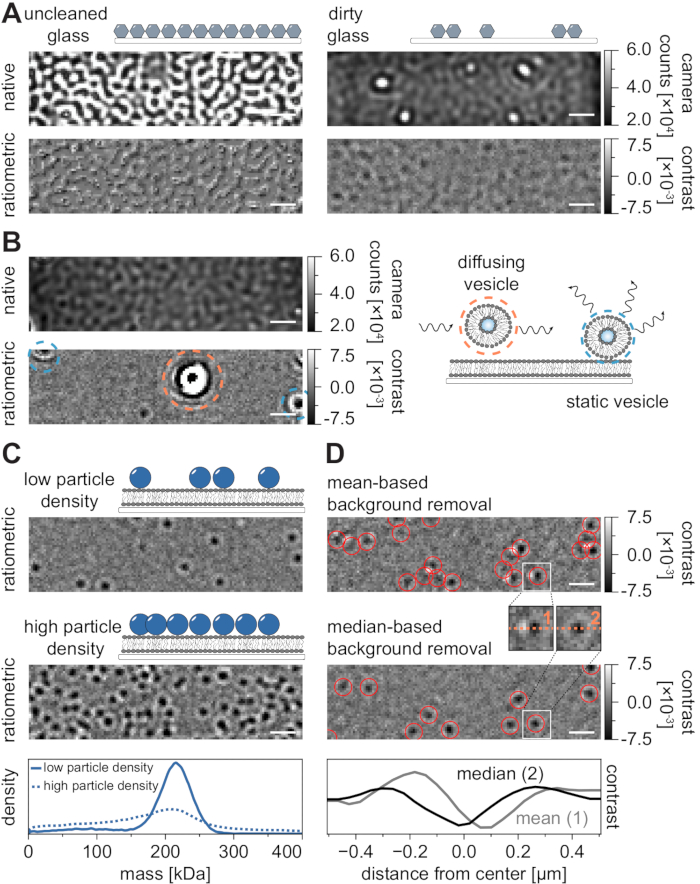
Figura 7: Complicações potenciais no curso das medições do MSPT ou durante a análise de dados. (A) Imagens representativas da rugosidade superficial exibidas tanto na visão nativa quanto na razão de fundo processada (remoção de fundo mediana) de um deslizamento de vidro de cobertura impura. Em ambos os casos, os pontos brilhantes constituem impurezas residuais da superfície que impedem as medições livres de artefatos. (B) Imagens exemplares de vesículas residuais no campo de visão após lavagem insuficiente da membrana. Tanto as vesículas estáticas (destacadas em azul) quanto a difusão (destacada em laranja) prejudicarão a qualidade da medição, seja devido ao pulsante e à agitação ou devido ao seu movimento direcional, respectivamente. (C) Como técnica de partícula única, o MSPT requer baixas densidades de partículas (imagem representativa, painel superior) para permitir a ligação adequada e a determinação em massa de cada partícula. No caso de altas densidades de partículas de membrana (painel médio), o encaixe de partículas é prejudicado, o que afeta a determinação em massa (ver painel inferior). (D) Imagens de proporção representativa de partículas difundindo em uma interface de membrana após a base média (painel superior) ou a remoção de fundo baseada em mediana. Para difundimento de partículas, a estratégia de remoção de fundo baseada em média produz imagens distorcidas do PSF da partícula, como pode ser visto nos pequenos insets entre o painel superior e médio. Em contraste, PSFs de partículas não retorcidos podem ser obtidos através da abordagem baseada em mediana. Painel inferior: Comparação dos perfis de linha através do centro do PSF obtido após a remoção de fundo média ou mediana. Para todas as imagens nativas e racionadas exibidas nesta figura, as barras de escala representam 1 μm. Para análise de dados (ver notebook Jupyter acompanhado; etapa 9), foram utilizados os seguintes parâmetros: tamanho médio da janela (window_length) = 1.001 quadros, limiar de detecção (thresh) = 0,00055, alcance de pesquisa (dmax) = 4 pixels, memória (max_frames_to_vanish) = 0 quadros, comprimento mínimo da trajetória (minimum_trajectory_length) = 5. Clique aqui para ver uma versão maior desta figura.
Figura suplementar 1: Comparação de membranas livres de proteínas e ocupadas. Imagens representativas de uma bicamada lipídica intacta suportada antes (A) e depois (B) a adição de streptavidina purificado (STP). As vagas de candidatos que foram bem encaixadas no modelo PSF estão cercadas em vermelho. (C) Distribuições de probabilidade de contraste de partículas detectadas em uma membrana vazia (fundo de membrana, cinza) e em uma bicamadas com partículas estreptavidinas difusas (azul). Ambas as distribuições de probabilidade representam os dados agrupados de três experimentos independentes com parâmetros idênticos de aquisição e análise de filmes. Para análise de dados (ver notebook Jupyter acompanhado; etapa 9), foram utilizados os seguintes parâmetros: tamanho médio da janela (window_length) = 1.001 quadros, limiar de detecção (thresh) = 0,00055, alcance de pesquisa (dmax) = 4 pixels, memória (max_frames_to_vanish) = 0 quadros, comprimento mínimo da trajetória (minimum_trajectory_length) = 7 quadros. Clique aqui para baixar este Arquivo.
Filme suplementar 1: Filme exemplar mostrando a ruptura e fusão de vesículas em uma membrana homogênea registrada com o fotômetro de massa. Tamanho médio da janela de processamento de imagem (window_length) = 1.001 quadros. Barra de escala: 1 μm. Faixa de contagem de câmeras: preto = 16.892; branco = 65.408. Clique aqui para baixar este Filme.
Filme suplementar 2: Filmes exemplares que mostram a difusão dos complexos de anexo V (superior) e aldolase biotinína (inferior) em uma bicamada, obtidos a partir de medições do MSPT. Tamanho médio da janela de processamento de imagem (window_length) = 1.001 quadros. Barra de escala: 1 μm. Faixa de contraste de dispersão interferométrica: preto = -0,0075; branco = 0,0075. Clique aqui para baixar este Filme.
Discussão
O protocolo apresentado estende a fotometria em massa21, técnica que analisa a massa de adsorçamentos biomoléculas únicos no vidro, a uma ferramenta ainda mais versátil capaz de medir simultaneamente a massa e a difusão de biomoléculas não rotuladas que interagem com membranas. Esta extensão de análise é alcançada através da implementação de uma estratégia modificada de remoção de fundo adaptada ao movimento lateral das moléculas24,25. Em geral, a remoção de fundo é de extrema importância para abordagens baseadas em iSCAT, uma vez que a forte dispersão da rugosidade da superfície de vidro representa o principal impedimento de análise, e a determinação precisa do fundo local de cada pixel é essencial para a quantificação da massa e localização de partículas. Além da análise de imagem adaptada ao movimento de partículas, a detecção subsequente de partículas, a vinculação de trajetórias e a análise de dados completam a nova expansão da MP no rastreamento de partículas sensíveis à massa (MSPT).
Em geral, lâminas de tampa de vidro completamente limpas e um ambiente de trabalho limpo são requisitos críticos para o desempenho bem-sucedido dos experimentos do MSPT. Devido à ausência de rotulagem macromolécula, o sinal adquirido é inerentemente não seletivo. Amostras limpas, bem como o manuseio adequado da amostra, são, portanto, cruciais para garantir que as observações não possam ser mal interpretadas. Em particular, quando moléculas de baixo peso molecular são examinadas, as medidas de controle de membranas livres de proteínas são endossadas para avaliar as contribuições de fundo (Figura Suplementar 1). Além da inclusão das medições de controle, recomenda-se, assim, acompanhar as etapas de preparação exibidas na Figura 2 para cada câmara de fluxo. Quando combinados, essas medidas de segurança garantirão que o sinal detectado se origine da biomolécula de interesse e não, por exemplo, de uma câmara de fluxo, tampão ou membrana contaminada.
Além das precauções relativas ao desenho experimental, é preciso tomar cuidado durante o processamento de imagens do MSPT. Durante o processamento de vídeo, o valor para três parâmetros deve ser escolhido cuidadosamente para garantir os resultados corretos: i) o comprimento da janela mediana para remoção de fundo, ii) o limiar para detecção de partículas e iii) o raio máximo de pesquisa durante a atribuição de vinculação. Uma janela mediana maior (i) geralmente facilita a separação de partículas difusas do fundo quase constante sobreposto. No entanto, para tamanhos de janelas muito grandes a deriva amostra eventualmente se tornará perceptível e diminuirá a precisão da estimativa de fundo. As configurações ideais dependem fortemente das propriedades da amostra e das condições de medição. No entanto, um valor de 1.001 pode ser usado como um ponto de partida robusto. O parâmetro limiar (ii) deve ser ajustado dependendo da menor massa molecular esperada na amostra. Um valor abaixo de 0,0005 não é recomendado para medições feitas com o fotômetro de massa utilizado neste estudo. Para acelerar os tempos de análise, valores mais elevados podem ser escolhidos se uma amostra com alto peso molecular for esperada. O raio de pesquisa na ligação de trajetória (iii) especifica a distância radial máxima em pixels nos quais o local deslocado da partícula será procurado em quadros consecutivos. Ele deve ser adaptado à partícula mais rápida da amostra e, se favorecido, um intervalo de pesquisa adaptativo (ver documentação do trackpy) poderia ser usado em vez disso para reduzir o tempo de computação. Especialmente durante a fase inicial de um projeto, recomenda-se a re-análise dos filmes com parâmetros variados para validar os resultados obtidos.
À luz da natureza de molécula única do MSPT, deve-se evitar medir em altas densidades de partículas de membrana, pois essas podem interferir com contraste preciso e determinação de massa. Foi demonstrado que densidades abaixo de uma partícula per micrômetro quadrado são favoráveis para as medições de MSPT24. Uma consideração adicional são os coeficientes de difusão esperados na amostra. Embora aplicável a uma ampla gama de coeficientes de difusão, o MSPT tem um limite menor de coeficientes de difusão acessíveis. O confinamento local a uma região de poucos pixels durante uma parte significativa do período médio da janela mescla a partícula com o fundo estático. Para as condições de imagem utilizadas neste protocolo, não é recomendada a medição de coeficientes de difusão abaixo de 0,01 μm2/s. Nesta velocidade de difusão, por exemplo, o deslocamento médio ao quadrado de uma partícula durante a janela mediana de tamanho médio é de cerca de 4 pixels e, portanto, de tamanho semelhante à extensão do PSF. Como consequência, a estimativa de fundo estático provavelmente conterá contribuições de sinal da própria partícula, o que resulta em um contraste aparentemente reduzido da partícula até que ela eventualmente se aproxime do nível de ruído. No entanto, os coeficientes de difusão de macromolécula que variam entre 0,05 e 10 μm2/s podem ser claramente resolvidos.
Para ampliar ainda mais o alcance de aplicações MSPT, pode-se prever um avanço do algoritmo de fundo baseado em mediana através da eliminação de pixels que estão temporariamente ocupados com uma partícula, ou por correção de deriva de amostra permitindo tamanhos de janelas medianas maiores. Ambas as abordagens aliviariam os problemas relativos às medições em altas densidades de partículas e difusão lenta. Melhorias em termos de menor sensibilidade à massa estão no horizonte com uma nova geração de fotômetros de massa, que podem fornecer acesso a biomoléculas menores que 50 kDa. Portanto, futuros experimentos de MSPT serão capazes de estudar dinâmicas de molécula única e interações relacionadas à membrana para uma gama ainda maior de imitações de membrana, como bicamadas amortecidas e sistemas macromoleculares.
Divulgações
Os autores não têm conflitos de interesse.
Agradecimentos
Agradecemos sinceramente o apoio de Philipp Kukura, Gavin Young e da equipe de software refeyn e reconhecemos sua ajuda compartilhando partes do código de análise de imagens. Agradecemos ao Cryo-EM MPIB Core Facility por fornecer acesso ao fotômetro de massa comercial Refeyn. A F.S. agradece o apoio e o financiamento concedidos por Jürgen Plitzko e Wolfgang Baumeister. T.H. e P.S. receberam financiamento através da Deutsche Forschungsgemeinschaft (DFG, German Research Foundation) - Project-ID 201269156 - SFB 1032 (A09). N.H. foi apoiado por uma concessão de retorno do DFG HU 2462/3-1. A P.S. reconhece o apoio por meio da rede de pesquisa MaxSynBio por meio da iniciativa de financiamento conjunto do Ministério da Educação e Pesquisa da Alemanha (BMBF) e da Sociedade Max Planck.
Materiais
| Name | Company | Catalog Number | Comments |
| annexin V | Sigma Aldrich | #SRP8026 | examplary membrane-interacting protein |
| Bio-Rad Protein Assay | Bio-Rad Laboratories Inc. | #5000006 | bradford assay kit to determine protein stock concentrations |
| biotin labeled bovine albumin | Sigma Aldrich | #A8549 | examplary protein that can be used as standard protein for MSPT |
| cholera toxin subunit B | Sigma Aldrich | #SAE0069 | examplary membrane-interacting protein |
| cover glasses, #1.5, 24 x 24 mm | Paul Marienfeld GmbH & Co. KG | #0102062 | |
| cover glasses, #1.5, 24 x 60 mm | Paul Marienfeld GmbH & Co. KG | #0102242 | |
| dioleoyl-sn-glycero-3-phosphocholine (DOPC) | Avanti Polar Lipids | #850375 | lipid - in the form of extruded small unilamellar vesicles required for supported lipid bilayer formation |
| dioleoyl-sn-glycero-3-phosphoethanolamine-N-cap biotinyl (18:1 Biotinyl Cap PE | Avanti Polar Lipids | #870273 | lipid - in the form of extruded small unilamellar vesicles required for supported lipid bilayer formation |
| dioleoyl-sn-glycero-3-phosphoglycerol (DOPG) | Avanti Polar Lipids | #840475 | lipid - in the form of extruded small unilamellar vesicles required for supported lipid bilayer formation |
| dioleoyl-sn-glycero-3-phospho-L-serine (DOPS) | Avanti Polar Lipids | #840035 | lipid - in the form of extruded small unilamellar vesicles required for supported lipid bilayer formation |
| double-sided tape | tesa | #57912-00000-02 | needed for the assembly of glass sample chambers |
| Extruder | Avanti Polar Lipids | #610023 | Lipid extruder to enable monodisperse vesicle distributions |
| EZ-Link Maleimide-PEG2-Biotin | Thermo Fisher Scientific | #A39261 | maileimide-fused biotin that can be used to biotinylate standard proteins for MSPT |
| Fibronectin (Biotinylated) | Cytoskeleton Inc. | #FNR03-A | examplary protein that can be used as standard protein for MSPT |
| Gel Filtration HMW Calibration Kit | Cytiva | #28403842 | standard proteins, e.g. aldolase that can be biotinylated and used as molecular weight standards for MSPT |
| GM1 Ganglioside (Brain, Ovine-Sodium Salt) | Avanti Polar Lipids | #860065 | lipid - in the form of extruded small unilamellar vesicles required for supported lipid bilayer formation |
| Goat anti-Rabbit IgG (H+L) Secondary Antibody, Biotin | Thermo Fisher Scientific, Waltham, USA) | #31820 | examplary protein to highlight the existence of different protein states |
| Isopropanol, 99.5%, for spectroscopy | Thermo Fisher Scientific | #10003643 | |
| Low Autofluorescence Immersion Oil | Olympus K.K. | #IMMOIL-F30CC | |
| pET21a-Streptavidin-Alive | Addgene | #20860 | required to express and purify divalent streptavidin in combination with each other |
| pET21a-Streptavidin-Dead | Addgene | #20859 | required to express and purify divalent streptavidin in combination with each other |
| Pierce Alkaline Phosphatase, biotinylated | Thermo Fisher Scientific | #29339 | examplary protein that can be used as standard protein for MSPT |
| Pierce Protein A, Biotinylated | Thermo Fisher Scientific | #29989 | examplary protein that can be used as standard protein for MSPT |
| Refeyn Acquire | Refeyn Ltd. | control software for Refeyn OneMP | |
| Refeyn One | Refeyn Ltd. | - | mass photometer |
| sterile syringe filters 0.45 µm cellulose acetate membrane | VWR International | #514-0063 | needed to filter particles from the buffer of interest |
| tetravalent streptavidin | Thermo Fisher Scientific | #SNN1001 | tetravalent streptavidin to enable the presence of several biotin binding sites |
| Whatman Nuclepore Hydrophilic Membrane, 0.05 µm Pore Size, 25 mm Circle | Cytiva | #110603 | a pore size of 50 nm is recommended for supported lipid bilayer formation in the context of MSPT |
| Zepto model 2 plasma cleaner | Diener electronic GmbH | - |
Referências
- Robertson, J. L. The lipid bilayer membrane and its protein constituents. Journal of General Physiology. 150 (11), 1472-1483 (2018).
- Grecco, H. E., Schmick, M., Bastiaens, P. I. H. Signaling from the Living Plasma Membrane. Cell. 144 (6), 897-909 (2011).
- Cho, W., Stahelin, R. V. Membrane-protein interactions in cell signaling and membrane trafficking. Annual Review of Biophysics and Biomolecular Structure. 34, 119-151 (2005).
- Whited, A. M., Johs, A. The interactions of peripheral membrane proteins with biological membranes. Chemistry and Physics of Lipids. 192, 51-59 (2015).
- Gonzalez, L., Scheller, R. H. Regulation of membrane trafficking: Structural insights from a Rab/effector complex. Cell. 96 (6), 755-758 (1999).
- Sezgin, E., Levental, I., Mayor, S., Eggeling, C. The mystery of membrane organization: composition, regulation and roles of lipid rafts. Nature Reviews Molecular Cell Biology. 18 (6), 361-374 (2017).
- Bagheri, Y., Ali, A. A., You, M. Current methods for detecting cell membrane transient interactions. Frontiers in Chemistry. 8, 603259(2020).
- Miller, H., Zhou, Z., Shepherd, J., Wollman, A. J. M., Leake, M. C. Single-molecule techniques in biophysics: A review of the progress in methods and applications. Reports on Progress in Physics. 81 (2), 024601(2018).
- Manzo, C., Garcia-Parajo, M. F. A review of progress in single particle tracking: From methods to biophysical insights. Reports on Progress in Physics. 78 (12), 124601(2015).
- Gelles, J., Schnapp, B. J., Sheetz, M. P. Tracking kinesin-driven movements with nanometre-scale precision. Nature. 331 (6155), 450-453 (1988).
- Funatsu, T., Harada, Y., Tokunaga, M., Saito, K., Yanagida, T. Imaging of single fluorescent molecules and individual ATP turnovers by single myosin molecules in aqueous solution. Nature. 374 (6522), 555-559 (1995).
- Schmidt, T., Schütz, G. J., Baumgartner, W., Gruber, H. J., Schindler, H. Imaging of single molecule diffusion. Proceedings of the National Academy of Sciences of the United States of America. 93 (7), 2926-2929 (1996).
- Taylor, R. W., et al. Interferometric scattering microscopy reveals microsecond nanoscopic protein motion on a live cell membrane. Nature Photonics. 13 (7), 480-487 (2019).
- Kukura, P., et al. High-speed nanoscopic tracking of the position and orientation of a single virus. Nature Methods. 6 (12), 923-927 (2009).
- Jacobsen, V., Stoller, P., Brunner, C., Vogel, V., Sandoghdar, V. Interferometric optical detection and tracking of very small gold nanoparticles at a water-glass interface. Optics Express. 14 (1), 405(2006).
- Ueno, H., et al. Simple dark-field microscopy with nanometer spatial precision and microsecond temporal resolution. Biophysical Journal. 98 (9), 2014-2023 (2010).
- Loose, M., Fischer-Friedrich, E., Herold, C., Kruse, K., Schwille, P. Min protein patterns emerge from rapid rebinding and membrane interaction of MinE. Nature Structural and Molecular Biology. 18 (5), 577-583 (2011).
- Bezeljak, U., Loya, H., Kaczmarek, B., Saunders, T. E., Loose, M. Stochastic activation and bistability in a Rab GTPase regulatory network. Proceedings of the National Academy of Sciences of the United States of America. 117 (12), 6504-6549 (2020).
- Ha, T., Tinnefeld, P. Photophysics of fluorescent probes for single-molecule biophysics and super-resolution imaging. Annual Review of Physical Chemistry. 63 (1), 595-617 (2012).
- Garcia-Parajo, M. F., Segers-Nolten, G. M. J., Veerman, J. A., Greve, J., Van Hulst, N. F. Real-time light-driven dynamics of the fluorescence emission in single green fluorescent protein molecules. Proceedings of the National Academy of Sciences of the United States of America. 97 (13), 7237-7242 (2000).
- Young, G., et al. Quantitative mass imaging of single biological macromolecules. Science. 360 (6387), 423-427 (2018).
- Piliarik, M., Sandoghdar, V. Direct optical sensing of single unlabelled proteins and super-resolution imaging of their binding sites. Nature Communications. 5, 4495(2014).
- Cole, D., Young, G., Weigel, A., Sebesta, A., Kukura, P. Label-free single-molecule imaging with numerical-aperture-shaped interferometric scattering microscopy. ACS Photonics. 4 (2), 211-216 (2017).
- Heermann, T., Steiert, F., Ramm, B., Hundt, N., Schwille, P. Mass-sensitive particle tracking to elucidate the membrane-associated MinDE reaction cycle. Nature Methods. 18 (10), 1239-1246 (2021).
- Foley, E. D. B., Kushwah, M. S., Young, G., Kukura, P. Mass photometry enables label-free tracking and mass measurement of single proteins on lipid bilayers. Nature Methods. 18 (10), 1247-1252 (2021).
- Voss, O. H., Lee, H. N., Tian, L., Krzewski, K., Coligan, J. E. Liposome preparation for the analysis of lipid-receptor interaction and efferocytosis. Current Protocols in Immunology. 120, 1-21 (2018).
- Pincet, F., et al. FRAP to characterize molecular diffusion and interaction in various membrane environments. PLoS ONE. 11 (7), 0158457(2016).
- Howarth, M., et al. A monovalent streptavidin with a single femtomolar biotin binding site. Nature Methods. 3 (4), 267-273 (2006).
- Allan, D., et al. soft-matter/trackpy: Trackpy v.0.5.0. Zenodo. , (2021).
- Weimann, L., et al. A quantitative comparison of single-dye tracking analysis tools using Monte Carlo simulations. PLoS ONE. 8 (5), 64287(2013).
- Michalet, X. Mean square displacement analysis of single-particle trajectories with localization error: Brownian motion in an isotropic medium. Physical Review E - Statistical, Nonlinear, and Soft Matter Physics. 82 (4), 041914(2010).
- Sygusch, J., Beaudry, D., Allaire, M. Molecular architecture of rabbit skeletal muscle aldolase at 2.7-A resolution. Proceedings of the National Academy of Sciences of the United States of America. 84 (22), 7846-7850 (1987).
- Heath, G. R., Scheuring, S. High-speed AFM height spectroscopy reveals µs-dynamics of unlabeled biomolecules. Nature Communications. 9 (1), 4983(2018).
- Day, C. A., Kenworthy, A. K. Mechanisms underlying the confined diffusion of cholera toxin B-subunit in intact cell membranes. PLOS ONE. 7 (4), 34923(2012).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados