Method Article
Cocultivo y transducción de timocitos murinos en células estromales que expresan 4 similares a Delta para estudiar oncogenes en leucemia de células T
En este artículo
Resumen
Este protocolo describe el aislamiento de timocitos doble negativos del timo de ratón seguido de transducción retroviral y cocultivo en el sistema de cocultivo de líneas celulares del estroma del estroma de médula ósea que expresa 4 en forma delta (OP9-DL4) para un análisis funcional adicional.
Resumen
Los timocitos inmaduros de ratón transducidos se pueden diferenciar en células T in vitro utilizando el sistema de cocultivo de líneas celulares estromales de médula ósea que expresan 4 en forma delta (OP9-DL4). Como la transducción retroviral requiere células en división para la integración transgénica, OP9-DL4 proporciona un entorno in vitro adecuado para cultivar células progenitoras hematopoyéticas. Esto es particularmente ventajoso cuando se estudian los efectos de la expresión de un gen específico durante el desarrollo normal de las células T y la leucemogénesis, ya que permite a los investigadores eludir el proceso lento de generación de ratones transgénicos. Para lograr resultados exitosos, se deben realizar cuidadosamente una serie de pasos coordinados que implican la manipulación simultánea de diferentes tipos de células. Aunque estos son procedimientos muy bien establecidos, la falta de una fuente común en la literatura a menudo significa que se requieren una serie de optimizaciones, que pueden llevar mucho tiempo. Este protocolo ha demostrado ser eficiente en la transducción de timocitos primarios seguidos de diferenciación en células OP9-DL4. Aquí se detalla un protocolo que puede servir como una guía rápida y optimizada para el cocultivo de timocitos transducidos retroviralmente en células estromales OP9-DL4.
Introducción
La línea celular estromal de médula ósea OP9 proporciona un sistema in vitro útil para la inducción de linfopoyesis a partir de varias fuentes de progenitores1. Los primeros estudios que utilizaron células OP9 demostraron que la falta de expresión del factor estimulante de colonias de macrófagos (MCSF) contribuyó a la capacidad de la línea celular OP9 para apoyar la hematopoyesis y la diferenciación de células B de las células madre hematopoyéticas derivadas de la médula ósea (HSC), como también se demostró más tarde para las células madre embrionarias (ESC)2,3,4,5 . En estudios previos, la generación de células OP9 que expresan 1/4 en forma delta (OP9-DL1/OP9-DL4) permitió la inducción del compromiso del linaje de células T6 y demostró la capacidad de recapitular con éxito la maduración tímica 7,8. Brevemente, el desarrollo de células T se ha descrito mediante la expresión secuencial de moléculas CD4 y CD8. Los timocitos inmaduros son "dobles negativos" (DN, CD4− CD8−) y pueden subdividirse según la expresión superficial de CD44 y CD25. Los timocitos DN se diferencian a través de la etapa inmadura positiva simple (ISP), caracterizada por la expresión de CD8 en ratones y CD4 en humanos, seguida de la etapa doble positiva (DP), caracterizada por la coexpresión de CD4 y CD8, y, finalmente, la etapa madura positiva única, caracterizada por la expresión de CD4 o CD89. Las HSC expresan el receptor Notch1, que normalmente interactúa con el delta-like 4 (DL4) expresado en las células epiteliales tímicas para inducir la diferenciación del linaje T10. Por lo tanto, el interés en el modelo OP9-DL1/4 ha aumentado progresivamente, lo que ha llevado al uso extensivo de este enfoque en una amplia variedad de aplicaciones en las últimas dos décadas 8,11,12,13. Aunque DL1 y DL4 son capaces de soportar la diferenciación de células T in vitro, muestran requerimientos diferenciales in vivo, y algunos estudios han sugerido que OP9_DL4 es más eficiente que OP9_DL1 en la recapitulación del ambiente tímico del ratón 7,14.
Entre las aplicaciones potenciales del sistema OP9-DL1/4, existe un interés particular en la combinación de este sistema con la transducción de células DN o HSCs con vectores retrovirales. Esta combinación es una forma efectiva de manipular la expresión génica durante el desarrollo normal de células T y la leucemogénesis y ha demostrado ser un método eficiente para inducir o inhibir la función de un gen de interés15,16,17. Este modelo se ha utilizado con especial éxito para estudiar la colaboración entre los oncogenes que impulsan la leucemia15, ya que es flexible y permite examinar los efectos de múltiples combinaciones de genes en un tiempo razonable, en contraste con la generación de ratones transgénicos. Además, modelos similares han sido utilizados previamente para evaluar los efectos de la introducción de oncogenes en células normales15,16,17. Además, la transducción retroviral requiere células en división para la integración transgénica18, y aunque la transducción lentiviral superaría esta limitación al eliminar la necesidad de células en división para la integración transgénica, no hemos podido lograr la transducción exitosa de timocitos DN utilizando vectores lentivirales. Por lo tanto, OP9-DL1/DL4 es una herramienta adecuada para el crecimiento de células progenitoras hematopoyéticas.
El protocolo estándar para la linfopoyesis de timocitos transducidos en OP9-DL4 implica una serie de pasos coordinados que deben realizarse cuidadosamente para lograr un resultado exitoso. Aunque estas son técnicas que han estado sirviendo bien a la comunidad durante muchos años, a menudo los protocolos disponibles en la literatura están fragmentados. Como resultado, cada laboratorio se ve obligado a adaptar y optimizar diferentes etapas del procedimiento, lo que puede llevar mucho tiempo. Aquí, este protocolo describe el aislamiento de timocitos DN del timo de ratón, seguido de transducción retroviral y cocultivo en células estromales OP9-DL4 para un análisis funcional adicional. Este protocolo establecido ha demostrado ser eficiente y reproducible en la transducción de timocitos primarios, seguido de la diferenciación en células OP9-DL4 o la inducción de leucemia linfoblástica aguda de células T15.
Protocolo
Todos los experimentos con animales descritos fueron aprobados por el Comité Institucional de Bioseguridad (IBC) y el Comité de Cuidado y Uso de Animales (ACUC) de los NIH. Consulte la Tabla de materiales para obtener detalles relacionados con todos los reactivos y materiales utilizados en este protocolo. Consulte las directrices publicadas19 para obtener más detalles sobre el cultivo de células productoras de retrovirus y los procedimientos de mantenimiento. Consulte la figura 1 para obtener información general sobre el protocolo.
1. Inicio del cultivo de células OP9-DL4 (Día 1)
- Descongele rápidamente las células OP9-DL4 sosteniendo el vial y agitando suavemente en un baño maría a 37 °C. Transfiera inmediatamente las células a un tubo de centrífuga que contenga 5 ml del medio OP9 (medio MEM-alfa, 10% FBS, 50 U/ml penicilina/estreptomicina, 55 μM 2-mercaptoetanol, 2 mM L-glutamina) para eliminar los agentes crioprotectores.
- Centrifugar a 300 × g durante 5 min a temperatura ambiente. Desechar el sobrenadante y resuspender las células en 1 ml de medio OP9.
- Introducir 5 ml de medio OP9 en un matraz T25 y añadir las células resuspendidas a este matraz. Cultivo a 37 °C y 5%CO2.
- Después de 2 a 3 días, subcultive las células usando una división 1:3 (pase las celdas todos los lunes, miércoles y viernes).
NOTA: Divida las celdas OP9-DL4 antes de que alcancen la confluencia. El matraz OP9-DL4 debe tener un 80%-90% de confluente en los días 6-7 y en los días 8-9+ para el cocultivo con timocitos. Por lo tanto, es importante planificar cuántas células OP9-DL4 se requerirán en esos días mientras se dividen las células OP9-DL4.- Deseche el medio del matraz y lave la monocapa con 1x PBS. Desechar el PBS, añadir 1 ml de tripsina al 0,25% e incubar durante 1-5 min a 37 °C, o hasta que las células se hayan desprendido del matraz. Golpee el matraz para desalojar las celdas.
NOTA: (Opcional) El progreso de la disociación celular se puede comprobar mediante microscopía. - Añadir 5 ml de medio OP9 para inactivar la tripsina y resuspender las células enjuagando la superficie cubierta de células del matraz durante la resuspensión.
- Centrifugar la suspensión celular a 300 × g durante 5 min a temperatura ambiente y volver a suspender en 3 ml de medio OP9 (para una división 1:3).
- Introducir 1 ml de la suspensión celular en un matraz T25 que ya contenga 5 ml de medio OP9 fresco. Congelar/desechar las celdas restantes.
- Para congelar las células OP9-DL4, resuspenda las células en 90% FBS/10% DMSO en una proporción de 1 ml de medio de congelación a un vial de células.
- Congelar en crioviales de 1 ml a -80 °C o en nitrógeno líquido, dependiendo de los requisitos futuros. Almacenar en nitrógeno líquido para un almacenamiento prolongado.
- Deseche el medio del matraz y lave la monocapa con 1x PBS. Desechar el PBS, añadir 1 ml de tripsina al 0,25% e incubar durante 1-5 min a 37 °C, o hasta que las células se hayan desprendido del matraz. Golpee el matraz para desalojar las celdas.
2. Inicio del cultivo de la línea celular productora de retrovirus (Día 1)
- Descongele rápidamente la línea celular productora de retrovirus sosteniendo el vial y agitando suavemente en un baño maría a 37 °C. Transfiera inmediatamente las células a un tubo de centrífuga que contenga 5 ml de medio celular productor de retrovirus (RPC: DMEM, 10% FBS) para eliminar el agente crioprotector.
- Centrifugar a 300 × g durante 5 min a temperatura ambiente. Deseche el sobrenadante y resuspenda las células en 1 ml de medio RPC.
- Introducir 5 ml de medio RPC en un matraz T25 y añadir las células resuspendidas a este matraz. Cultivo a 37 °C y 5%CO2.
- Pasar las células cada 2-3 días en una división de 1:5 a 1:8.
- Golpee el matraz para desalojar las células y resuspenda las células pipeteando agresivamente.
NOTA: Estas células se pueden eliminar de la superficie del matraz mediante pipeteo agresivo sin tripsina. Alternativamente, tripsinizar (0,05% tripsina/0,53 mM EDTA) hasta que las células se desprendan fácilmente y puedan pipetearse fácilmente en una suspensión de una sola célula.
- Golpee el matraz para desalojar las células y resuspenda las células pipeteando agresivamente.
- Centrifugar la suspensión celular a 300 × g durante 5 min, y resuspender en 5 ml de medio RPC (para una división 1:5).
- Introducir 1 ml de la suspensión celular en un matraz T25 que ya contenga 5 ml de medio RPC fresco. Congelar/desechar las celdas restantes. Congelar las células productoras de retrovirus de la misma manera que se describe para las células OP9-DL4 (pasos 1.4.4.1–1.4.4.2).
3. Inicio del cultivo de las células productoras de retrovirus en placas de 6 pocillos (días 4-5)
- Sembrar las células productoras de retrovirus 18-24 h antes de la transfección a una confluencia del 70%-90% en cada pocillo de una placa de cultivo de tejido de 6 pocillos en 2 ml de medio RPC, e incubar a 37 °C y 5% deCO2.
NOTA: Transfecte dos pocillos de las células productoras de retrovirus por cada 2-5 × 105 a 1 × 106 timocitos que se transducirán.
4. Transfectar las células productoras de retrovirus para generar el retrovirus que contiene los genes de interés (Días 5-6)
- Reemplace el medio con medio RPC fresco 1 h antes de la transfección.
- Prepare mezclas de lipofección (para cada pocillo de una placa de 6 pocillos; escale según sea necesario): Diluya 4 μg de ADN (2 μg de plásmido auxiliar (pCL-Eco) y 2 μg de plásmido de transferencia (pMIG) en 250 μL de medio sérico reducido. Mezclar suavemente.
- Mezclar 10 μL de reactivo de transfección con 250 uL del medio sérico reducido a partir del paso 4.2. Incubar durante 5 min a temperatura ambiente.
- Después de 5 minutos de incubación, combinar el ADN diluido con el reactivo de transfección diluido (volumen total = 500 μL). Mezclar suavemente e incubar durante 20-25 min a temperatura ambiente.
- Añadir suavemente los 500 μL de mezclas de ADN/reactivos de transfección al pocillo que contiene las células productoras de retrovirus dejando caer sobre las células con un movimiento circular. Mezclar suavemente balanceando la placa hacia adelante y hacia atrás, e incubar la placa en una incubadora a 37 °C durante 16-24 h.
NOTA: Las células subconfluentes (80–90%) son las más adecuadas para la transfección y potencialmente generan el título viral más alto. Como las células productoras de retrovirus se desprenden muy fácilmente del matraz, evite movimientos bruscos al manipular estas células.
5. Cambio del medio celular productor de retrovirus (días 6–7)
- Reemplace el medio viejo aproximadamente 16 h después de la transfección con 2 ml de medio RPC fresco. Continuar incubando las células a 37 °C durante 20-24 h.
- Evaluar la eficiencia de la transfección bajo un microscopio de fluorescencia (opcional).
NOTA: En este protocolo, las células positivas para GFP son las celdas de interés (consulte la figura 2). Este paso depende del vector de retrovirus seleccionado para usar en este protocolo (si un gen reportero está presente en la columna vertebral).
6. Preparación de timocitos y agotamiento de células CD4+ y CD8+ (días 6-7)
- Cosechar el timo de un ratón C57BL/6 de 4-6 semanas de edad. Para obtener más detalles sobre la recolección del timo, consulte Xing y Hogquist20.
NOTA: Los ratones fueron sacrificados por inhalación deCO2 seguida de dislocación cervical. - Prepare una suspensión unicelular de timocitos colocando el timo en 5 ml de PBS en una placa de Petri. Usando portaobjetos de vidrio estéril, coloque el timo entre las superficies esmeriladas de los portaobjetos y frote suavemente los portaobjetos juntos, enrollando el timo entre los dos portaobjetos. Enjuague los portaobjetos de vidrio para recoger las células y deseche el tejido estromal tímico remanente.
NOTA: El rendimiento estimado de un timo es de 90 × 10 6-100 × 106 células, y aproximadamente el 1% de las células permanecerán después del agotamiento de CD4 y CD8. Utilice tejido de timo de ratones más jóvenes para un mejor rendimiento de las células, ya que la celularidad del timo del ratón en las primeras semanas después del nacimiento aumenta rápidamente, alcanza una meseta a las 4-6 semanas de edad e involuciona progresivamenteposteriormente 21. Cuente las células usando un contador automático de células sanguíneas o cualquier método alternativo. - Filtrar la suspensión tímica a través de un filtro de 30 μm o 40 μm pipeteando 5 ml de la suspensión tímica a través de un filtro celular. Centrifugar las células a 300 × g durante 10 min.
- Retire el sobrenadante y lisar los glóbulos rojos con tampón de lisis ACK agregando (dependiendo del tamaño de la muestra) 1 ml de tampón por tubo durante 1 minuto. Agregue 5 ml de tampón de agotamiento celular (500 ml de PBS [pH 7.2], 0.5% BSA, 2 mM 0.5M EDTA, pH 8.0) para desactivar el tampón de lisis ACK, centrifugar a 300 × g durante 10 min y resuspender en 1-5 ml de tampón de agotamiento celular para contar.
NOTA: Reservar lo siguiente en hielo antes del agotamiento (pre-agotamiento): 70 μL de suspensión celular para contar (varíe esto dependiendo del método de celda de conteo elegido) y 200 μL de suspensión celular para determinar la eficiencia del agotamiento mediante tinción de CD4 y CD8 y análisis por citometría de flujo22,23. - Contar las células y centrifugar a 300 × g durante 10 min. Resuspender las células a 1 × 107/80 μL de tampón de agotamiento celular. Añadir 10 μL de microperlas CD4 y CD8 por 1 × 107 células. Mezclar bien e incubar durante 15 minutos en la oscuridad en la nevera (2–8 °C).
- Prepare una columna de agotamiento enjuagándola con 2 ml de tampón de agotamiento y desechando el flujo continuo.
- Lavar las células del paso 6.6 añadiendo 1-2 ml de tampón de agotamiento por 1 × 107 células, y centrifugar a 300 × g durante 10 min. Retire y deseche el sobrenadante.
- Resuspender hasta 1,25 × 108 células en 500 μL de tampón de agotamiento. Aplique la suspensión de celdas a la columna y recoja el flujo continuo (celdas sin etiquetar). Lave la columna 2x con 1 ml de tampón y recoja el flujo continuo.
NOTA: Agregue un nuevo búfer sólo cuando el depósito de columna esté vacío. Reservar lo siguiente en hielo después del agotamiento (control posterior al agotamiento): 70 μL de suspensión celular para contar (varíe esto según el método de conteo de células elegido) y 1,000 μL de suspensión celular para determinar la eficiencia del agotamiento mediante tinción de CD4 y CD8 y análisis por citometría de flujo22,23. - Tinción de los controles de eficiencia de agotamiento dividiendo los 200 μL de células recolectadas antes del agotamiento en cuatro tubos FACS: sin teñir, CD4 con tinción simple, CD8 con tinción simple y CD4 / CD8 con doble tinción (use las muestras no teñidas y las de una sola tinción para configurar los parámetros de citometría de flujo). Utilice los 1.000 μL de células recogidas después del agotamiento para teñir CD4 y CD8 y comparar con la muestra de doble tinción recogida antes del agotamiento (ver resultados típicos del agotamiento en la Figura 3).
NOTA: Ajuste los volúmenes para la tinción por citometría de flujo de acuerdo con la recomendación del fabricante del anticuerpo (p. ej., 1 μL de anti-CD4-FITC + 0,5 μL de anti-CD8-PE por 1 x 106 células en 50 μL de tampón).
7. Cultivo de timocitos en células OP9-DL4 (días 6-7)
- Colocar 2–5 × 105 a 1 × 106 timocitos posdepleción en un matraz T25 de 80%–90% de células OP9-DL4 confluentes en medio OP9 que contenga citocinas (IL-7 recombinante de 10 ng/ml y hFLT-3 recombinante). Cultivo a 37 °C y 5%CO2. Crecer durante aproximadamente 24 h en células OP9-DL4.
NOTA: Esta duración es necesaria para que las células T sean transducibles (ver un resultado típico en la Figura 4). Reservar un matraz T25 del cocultivo de timocitos y células OP9-DL4 para utilizarlo como control (no transducido). Los timocitos no transducidos se teñirán junto con los timocitos transducidos (paso 9.1) como control negativo para evaluar la eficiencia de la transducción. Los timocitos no transducidos también se pueden usar para medir el efecto de la expresión transgénica en la diferenciación celular. Deseche el medio que contiene citoquinas después de 1 mes.
8. Recolección del retrovirus del sobrenadante y uso para transducir los timocitos (Días 8-9)
- Recoja el sobrenadante que contiene los retrovirus de las células transfectadas inclinando la placa de 6 pocillos y colocando una jeringa de 3-5 ml en la parte inferior de la placa mientras tira del émbolo para aspirar el sobrenadante. Reemplace el medio con 2 ml de medio RPC nuevo. Continuar incubando las células a 37 °C durante 20-24 h para la segunda transducción en el paso 8.9.
- Filtrar el sobrenadante de retrovirus a través de un filtro de jeringa de 0,45 μm y recoger el filtrado en un tubo de 50 ml.
NOTA: No congele el sobrenadante retroviral. Use una preparación de virus recién hecha para la transducción. - Recolectar timocitos del cultivo OP9-DL4 mediante pipeteo agresivo para eliminar los timocitos y las células OP9-DL4 de la superficie del matraz. Filtre la suspensión celular a través de un filtro celular de 40 μm para eliminar la mayoría de las células OP9-DL4 y recoja el filtrado en un tubo de 50 ml.
NOTA: Las células OP9 son muy adherentes. Aunque el pipeteo agresivo puede eliminar algunas de las células OP9-DL4 del matraz, la interrupción de la monocapa OP9-DL4 durante este proceso es mínima, y las células OP9-DL4 que se desprenden se filtrarán con el filtro celular de 40 μm, ya que las células OP9-DL4 son mucho más grandes que los timocitos primarios. Si las células OP9-DL4 todavía son 80%-90% confluentes, retire los timocitos y vuelva a colocarlos en el mismo matraz OP9-DL4. Alternativamente, se debe preparar un nuevo matraz OP9-DL4. - Centrifugar los timocitos en el filtrado a partir del paso 8.3 a 300 × g durante 5 min. Deseche el sobrenadante.
- En el tubo de 50 ml, resuspender los timocitos en 0,5–1 ml de medio OP9 + citocinas (ver paso 7.1). Agregue 1-2 ml de medio RPC que contenga el virus (el doble de medio RPC que medio de timocito). Añadir bromuro de hexametrina (concentración madre de 10 μg/μL) para obtener 8 μg de bromuro de hexametrina/ml de suspensión celular total.
- Espinocular centrifugando las células a 850 × g durante 1 h a temperatura ambiente.
- Resuspender las células en 6 ml de medio OP9 + citocinas por matraz (ver paso 7.1) y añadir la suspensión de nuevo a la monocapa de células OP9-DL4.
NOTA: Alternativamente, tenga nuevos matraces OP9-DL4 listos para recibir los timocitos transducidos. Si los timocitos se van a volver a colocar en un matraz OP9-DL4 usado, asegúrese de agregar medio OP9 fresco a las células OP9-DL4 durante la 1 h de espinoculación de timocitos para mantener las células sanas y garantizar una confluencia del 80% al 90%. - Incubar a 37 °C durante la noche.
- Repita los pasos 8.2 a 8.7 utilizando un nuevo pocillo del sobrenadante celular que contiene retrovirus.
9. Mantenimiento de timocitos transducidos en cultivo OP9-DL4 durante 2-5 días o congelación según sea necesario (Día 9+)
- Evaluar la diferenciación de timocitos por citometría de flujo mediante la tinción de timocitos para marcadores de desarrollo de células T como CD4, CD8, CD25 y CD4423. Vea los resultados típicos de la diferenciación de timocitos en la Figura 5.
Resultados
La eficiencia del agotamiento se puede evaluar citométricamente marcando la fracción celular magnéticamente no marcada para CD4 y CD8 después de la separación celular inmunomagnética (MACS) y analizando esto en un diagrama de puntos bivariado bidimensional (Figura 3). Un buen rendimiento de células doble negativas (CD4−, CD8−) es del 95% o superior, como se representa en la Figura 3. Dos de las causas más comunes de menor rendimiento son el cálculo erróneo de las microperlas basado en el número de células y el número de células marcadas que exceden la capacidad de la columna. Se recomienda elegir el número correcto de columnas MACS según el número de celdas etiquetadas. Cuando se trabaja con timocitos, el número de células marcadas (células DP y SP) es casi igual al número total de células (más del 96%). El recuento de células se puede hacer en un contador automático de células, una cámara Neubauer o con cualquier método alternativo. Como resultado, los volúmenes asignados para el recuento de celdas pueden necesitar ser ajustados dependiendo de la máquina específica y el método de conteo elegido.
Es importante determinar el número de células presentes antes y después del agotamiento. Estos recuentos celulares son necesarios para calcular el número de columnas de depleción de LD necesarias y para distribuir uniformemente los timocitos DN en el número apropiado de matraces OP9-DL4. Los controles para la citometría de flujo (células no marcadas, células marcadas para CD4 y células marcadas para CD8) se pueden realizar con la muestra reservada previa al agotamiento, ya que contiene más células. Sin embargo, como se espera que la mayoría de las células se retengan dentro de la columna, la muestra posterior al agotamiento que se etiquetará requerirá un volumen mayor. En consecuencia, es posible que sea necesario realizar ajustes de acuerdo con el protocolo de etiquetado seleccionado.
Cuando se utilizan vectores que expresan marcadores cribables, como un gen de fluorescencia, la transfección y la transducción se pueden evaluar de manera aproximada y empírica mediante microscopía de fluorescencia (Figura 2). La eficiencia de la transducción se puede analizar recolectando el timocito de la monocapa OP9-DL4, como se describe en el paso 8.3, y observando la expresión de un gen de fluorescencia por citometría de flujo. La eficiencia de la transducción utilizando un vector retroviral vacío con GFP como gen informador fue del 84,2% (Figura 4).
La diferenciación de células T en células OP9-DL4 se puede observar 4 días después de la transducción. La citometría de flujo generalmente se realiza para evaluar la diferenciación celular inducida por el cocultivo en células OP9-DL4 y / o la expresión transgénica, como se representa en la Figura 5, donde las células se marcaron para CD4, CD8, CD44 y CD25. Hay varias combinaciones posibles de etiquetado de moléculas de superficie celular que han demostrado ser útiles para investigar los mecanismos moleculares y celulares del desarrollo de células T en ratones24,25,26,27. Por lo tanto, los paneles de anticuerpos fluorescentes pueden variar según la cuestión de interés que se aborde. La transducción de timocitos DN con el vector retroviral vacío pMIG, mostrada en el panel B, presentó aproximadamente las mismas proporciones de positivos simples (CD4+ o CD8+), dobles positivos (CD4+/CD8+), dobles negativos (CD4−/CD8−), y sus subetapas dobles negativas 1-4 (DN1-DN4) que los timocitos no transducidos, mostrados en el panel A, lo que indica que el desarrollo de células T no se vio afectado por el proceso de transducción.
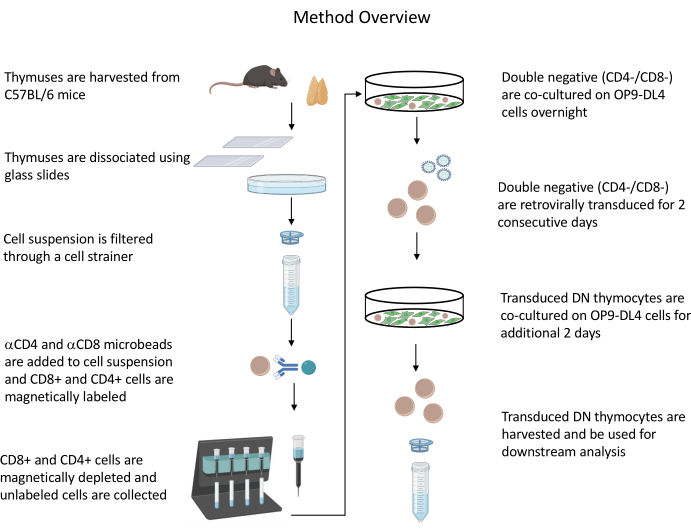
Figura 1: Diagrama de los pasos del aislamiento, transducción y cocultivo de timocitos. Abreviatura: DN = doble negativo. Haga clic aquí para ver una versión más grande de esta figura.
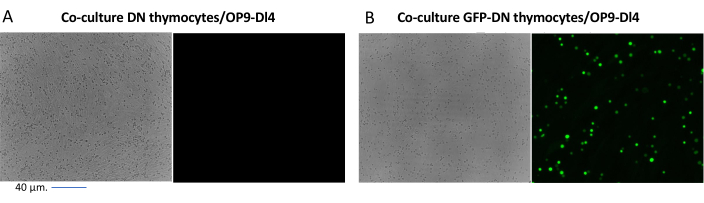
Figura 2: Microscopía de fluorescencia de cocultivo de timocitos transducidos por GFP o timocitos no transducidos y células OP9-DL4. (A) timocitos no transducidos y (B) timocitos murinos estables que expresan GFP en OP9-DL4 al día 3 después de la segunda transducción. Se utilizó un microscopio de fluorescencia Olympus-IX71 con una lente 40x y un filtro 480/30 adecuado para la detección de GFP. Barra de escala = 40 μm. Abreviaturas: GFP = proteína verde fluorescente; DN = doble negativo. Haga clic aquí para ver una versión más grande de esta figura.
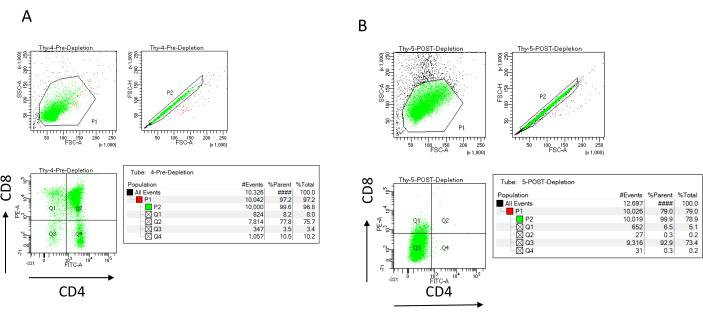
Figura 3: Citometría de flujo de la depleción de timocitos. Gráficos de citometría de flujo representativos que analizan la expresión de CD4 y CD8 en timocitos obtenidos de ratones hembra C57BL/6J de 7-8 semanas de edad (A) antes del agotamiento y (B) después del agotamiento de CD4+ y CD8+ utilizando microperlas y columnas LD de acuerdo con las instrucciones del fabricante. Los diagramas de puntos de la izquierda muestran puertas basadas en el tamaño y la complejidad del evento (FCS-A y SSC-A, respectivamente). El panel central muestra FSC-H frente a FSC-A para bloquear celdas individuales y excluir dobletes. En los gráficos de la derecha, las células se definieron como CD4 y CD8 desde la puerta de una sola célula. Abreviaturas: FSC-A = área de pico de dispersión hacia adelante; SSC-A = área de pico de dispersión lateral; FSC-H = altura del pico de dispersión hacia adelante; FITC = isotiocianato de fluoresceína; PE = ficoeritrina. Haga clic aquí para ver una versión más grande de esta figura.
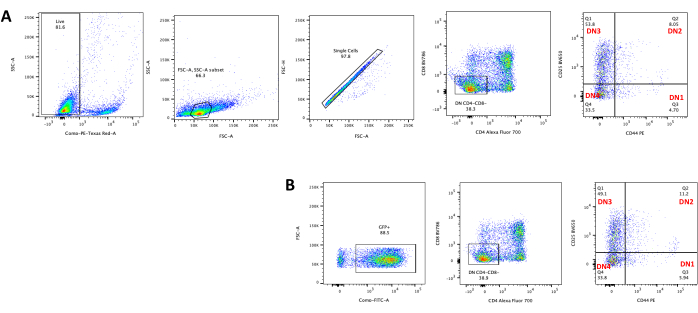
Figura 4: Citometría de flujo de la eficiencia de transducción retroviral del timocito. (A) timocitos no transducidos cocultivados en OP9-DL4; (B) timocitos transducidos retroviralmente en OP9-DL4 en el día 3 después de la transducción. Abreviaturas: FSC-A = área de pico de dispersión hacia adelante; SSC-A = área de pico de dispersión lateral; FSC-H = altura del pico de dispersión hacia adelante; FITC = isotiocianato de fluoresceína; PE = ficoeritrina; DN = doble negativo. Haga clic aquí para ver una versión más grande de esta figura.
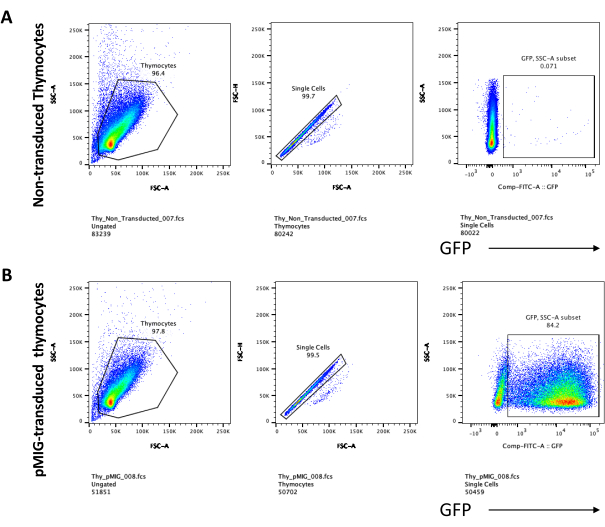
Figura 5: Citometría de flujo de timocitos transducidos después de 4 días de cocultivo OP9-DL4. (A) Sin transducir y (B) transducido con retrovirus pMIG. Las células se cerraron primero en células vivas y luego se cerraron en función del tamaño y la complejidad (FCS-A y SSC-A, respectivamente), seguidas de trazar FSC-H versus FSC-A para bloquear celdas individuales y excluir dobletes. Para las células no transducidas, utilizamos la siguiente estrategia de puerta. Desde la puerta unicelular, las células se definieron como positivas simples (CD4+ o CD8+), dobles positivas (CD4+/CD8+) y dobles negativas (CD4−/CD8−). Luego, a partir de la puerta de doble negativo (CD4−/CD8−), las células se definieron como CD44+, CD25+, CD44+/CD25+ y CD44−/CD25−, como se muestra en el panel A. Para las células transducidas pMIG, que se muestran en el panel B, desde la puerta de una sola célula, las células se definieron primero como GFP + / GFP −, y luego a partir de células GFP +, la distribución de la población celular en las principales etapas de desarrollo de células T, según lo definido por la expresión de CD4, CD8, CD44 y CD25, se determinó utilizando la misma estrategia de puerta que para las células no traducidas. Abreviaturas: FSC-A = área de pico de dispersión hacia adelante; SSC-A = área de pico de dispersión lateral; FSC-H = altura del pico de dispersión hacia adelante; FITC = isotiocianato de fluoresceína; GFP = proteína verde fluorescente. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
El protocolo descrito aquí se desarrolló específicamente para estudios de células T DN (CD4−/CD8−) derivadas del timo con transfección retroviral seguida de un modelo de diferenciación OP9-DL4. Sin embargo, es probable que las células diana que serán sometidas a este protocolo de transducción seguido de diferenciación celular tengan una utilidad interdisciplinaria más amplia. Por lo tanto, además de los timocitos inmaduros, las células madre hematopoyéticas, como las células derivadas del hígado fetal o la médula ósea, podrían usarse potencialmente en este protocolo.
El sistema OP9-DL4 ha demostrado ser un modelo eficaz para estudiar la función génica en una variedad de aspectos, incluyendo la diferenciación celular17 y la oncogénesis15. Si bien la modificación retroviral de progenitores hematopoyéticos es una técnica bien establecida que permite una modificación genética estable, combinar la inducción de la diferenciación celular en el sistema OP9-DL4 y la transducción retroviral requiere cuidado y habilidad. El aspecto crítico para lograr el éxito con este protocolo es garantizar que todos los pasos estén bien coordinados, ya que el protocolo implica el uso de tres tipos de células diferentes que deben mantenerse saludables y en la confluencia ideal requerida para cada etapa específica. Con esto en mente, es importante realizar todos los análisis de puntos de control de calidad después de la ejecución de cada paso antes de pasar al siguiente paso. Esto asegurará que todos los pasos estén funcionando. Por lo tanto, la comprobación de las eficiencias de agotamiento, transfección y transducción es importante para la ejecución correcta de este protocolo (consulte un resultado típico para la eficiencia de la transducción en la Figura 4). La buena eficiencia de la transducción de células primarias está relacionada con un título viral alto. Por lo general, los insertos más grandes dan como resultado títulos de virus más bajos18. Para fines de entrenamiento, utilizamos un vector vacío para representar los resultados que se pueden obtener con este protocolo. En nuestra experiencia, las eficiencias de transducción y transfección varían según el tamaño del inserto, especialmente considerando los genes reporteros que expresan la columna vertebral viral, como GFP. Una estrategia que se puede utilizar cuando se estudia la interacción de más de un gen es clonar cada gen en un vector de transferencia diferente, seguido de la producción de virus individuales y, finalmente, la cotransducción de la célula diana. En ese caso, se puede aplicar un paso de selección para eliminar las células transducidas individualmente y retener solo las células cotransducidas.
Vale la pena señalar que la mayoría de las líneas celulares de la capa de alimentación estromal OP9-DL1/DL4 han sido modificadas genéticamente para expresar GFP como parte de las construcciones DL1 o DL47. En este protocolo, utilizamos un vector retroviral que también expresa GFP; sin embargo, es más brillante que la proteína GFP expresada por las células OP9-DL4 y no interfiere con la inspección de transducción cuando se visualiza el cocultivo bajo el microscopio de fluorescencia.
Las células OP9 se diferencian en adipocitos después de muchos pasajes, largos períodos en cultivo o en condiciones de sobreconfluencia19. Esto se evidencia por el desarrollo de grandes vacuolas. Por lo tanto, las células OP9 que presentan estas características no deben utilizarse en este protocolo. La transfección de células productoras de retrovirus demasiado confluentes dará como resultado un título de virus bajo. De hecho, la etapa subconfluente es cuando las células son más transfectables. Además, la transfección de células productoras de retrovirus de baja confluencia disminuirá el estrés celular en el proceso de transfección y dará el título de virus más alto.
Si bien, en este protocolo, no titulamos el sobrenadante del virus, la titulación del sobrenadante del virus debe considerarse en algunos casos, como en ausencia de un gen informador en el vector retroviral, lo que impediría la determinación indirecta de la producción viral, o en los casos en que el diseño experimental requiere que se integre un número más preciso de copias del transgén en el genoma de la célula diana. Sin embargo, es importante tener en cuenta que el título de sobrenadante del vector retroviral disminuye significativamente cuando se almacena a −80 °C o 4 °C hasta que se obtienen los resultados de la titulación. Por lo tanto, el uso de sobrenadante de virus recién preparado para la transducción producirá una mejor eficiencia de transducción.
El timo contiene un gran número de timocitos dobles positivos (DP) (más del 85%) y aproximadamente un 10% de15 células simples positivas (CD4 o CD8), que son los timocitos en etapa post-DN. Las células DP no pueden sobrevivir in vitro para la manipulación retroviral, mientras que las células SP son intransducibles. Por lo tanto, este protocolo se puede aplicar para generar timocitos DN transducibles por vectores retrovirales.
Divulgaciones
Los autores no tienen conflictos de intereses que revelar
Agradecimientos
Este trabajo fue apoyado por el programa intramuros del Instituto Nacional del Cáncer, proyecto ZIABC009287. OP9-DL4 se obtuvo del Dr. Juan Carlos Zúñiga-Pflücker (Sunnybrook Health Sciences Centre, Toronto, ON, Canadá). Los autores agradecen al Programa de Ciencias de Animales de Laboratorio NCI-Frederick por su continua asistencia técnica y asesoramiento y aportes experimentales, así como a Jeff Carrel, Megan Karwan y Kimberly Klarmann por su asistencia en citometría de flujo. Agradecemos a Howard Young por sus consejos y aportes críticos.
Materiales
| Name | Company | Catalog Number | Comments |
| 2-mercaptoethanol | Sigma | M3148 | |
| ACK Lysis buffer | Lonza | 10-548E | |
| BSA | Cell Signaling Technology | 9998S | |
| CD4 Microbeads | Miltenyi | 130-049-201 | |
| CD8 Microbeads | Miltenyi | 130-049-401 | |
| Centrifuge 5910R | eppendorf | 5942IP802353 | |
| DMEM | Corning | 10-013-CV | |
| EDTA | Invitrogen | 15575-038 | |
| Fetal calf serum HyClone FBS | ThermoScientific | SH30910.03 | |
| LD columns | Miltenyi | 130-042-901 | |
| L-glutamine | Sigma | G7513 | Freeze glutamine in aliquots and use freshly-thawed glutamine |
| Lipofectamine 2000 | Invitrogen | P/N 52887 | |
| MEM-alpha Medium | Gibco | 12561-072 | |
| OPTI-MEM I Reducing Serum Medium | Invitrogen | 31985-062 | |
| PBS pH 7.2 | Corning | 21-040-CV | |
| pcL-Eco Plasmid | Addgene | 12371 | |
| penicillin/streptomycin | Gibco | 15140-122 | |
| pMIG Plasmid | Addgene | 6492 | |
| Polybrene | Chemicon | TR-1003-G | |
| Pre-Separation Filters | Miltenyi | 130-041-407 | |
| recombinant hFLT-3L | PeproTech | 300-19 | |
| recombinant IL-7 | Peprotech | 217-17 | |
| Retrovial packaging cell line Phoenix-Eco | Orbigen | RVC-10002 | |
| Syringe filter (0.45 µm) | Millipore | SLHV033RS |
Referencias
- Zuniga-Pflucker, J. C. T-cell development made simple. Nature Reviews Immunology. 4 (1), 67(2004).
- Kodama, H., Nose, M., Niida, S., Nishikawa, S., Nishikawa, S. Involvement of the c-kit receptor in the adhesion of hematopoietic stem cells to stromal cells. Experimental Hematology. 22 (10), 979-984 (1994).
- Nakano, T., Kodama, H., Honjo, T. Generation of lymphohematopoietic cells from embryonic stem cells in culture. Science. 265 (5175), 1098-1101 (1994).
- Nakano, T., Kodama, H., Honjo, T. In vitro development of primitive and definitive erythrocytes from different precursors. Science. 272 (5262), 722-724 (1996).
- Ueno, H., et al. A stromal cell-derived membrane protein that supports hematopoietic stem cells. Nature Immunology. 4 (5), 457-463 (2003).
- Jaleco, A. C., et al. Differential effects of Notch ligands Delta-1 and Jagged-1 in human lymphoid differentiation. Journal of Experimental Medicine. 194 (7), 991-1002 (2001).
- Mohtashami, M., et al. Direct comparison of Dll1- and Dll4-mediated Notch activation levels shows differential lymphomyeloid lineage commitment outcomes. Journal of Immunology. 185 (2), 867-876 (2010).
- Schmitt, T. M., Zuniga-Pflucker, J. C. Induction of T cell development from hematopoietic progenitor cells by delta-like-1 in vitro. Immunity. 17 (6), 749-756 (2002).
- Mazzucchelli, R., Durum, S. K. Interleukin-7 receptor expression: Intelligent design. Nature Reviews Immunology. 7 (2), 144-154 (2007).
- Robey, E. A., Bluestone, J. A. Notch signaling in lymphocyte development and function. Current Opinion in Immunology. 16 (3), 360-366 (2004).
- Lavaert, M., et al. Integrated scRNA-Seq identifies human postnatal thymus seeding progenitors and regulatory dynamics of differentiating immature thymocytes. Immunity. 52 (6), 1088-1104 (2020).
- Roh, K. H., Roy, K. Engineering approaches for regeneration of T lymphopoiesis. Biomaterials Research. 20 (20), (2016).
- Zlotoff, D. A., et al. CCR7 and CCR9 together recruit hematopoietic progenitors to the adult thymus. Blood. 115 (10), 1897-1905 (2010).
- Holmes, R., Zuniga-Pflucker, J. C. The OP9-DL1 system: Generation of T-lymphocytes from embryonic or hematopoietic stem cells in vitro. Cold Spring Harbor Protocols. 2009 (2), (2009).
- Cramer, S. D., et al. Mutant IL-7Ralpha and mutant NRas are sufficient to induce murine T cell acute lymphoblastic leukemia. Leukemia. 32 (8), 1795-1882 (2018).
- Treanor, L. M., et al. Interleukin-7 receptor mutants initiate early T cell precursor leukemia in murine thymocyte progenitors with multipotent potential. Journal of Experimental Medicine. 211 (4), 701-713 (2014).
- Yokoyama, K., et al. In vivo leukemogenic potential of an interleukin 7 receptor alpha chain mutant in hematopoietic stem and progenitor cells. Blood. 122 (26), 4259-4263 (2013).
- Simmons, A., Alberola-Ila, J. Retroviral transduction of T cells and T cell precursors. Methods in Molecular Biology. 1323, 99-108 (2016).
- Retroviral systems. , Nolan Lab. Available from: https://web.stanford.edu/group/nolan/_OldWebsite/retroviral_systems/retsys.html (2022).
- Xing, Y., Hogquist, K. A. Isolation, identification, and purification of murine thymic epithelial cells. Journal of Visualized Experiments. (90), e51780(2014).
- Gray, D. H., et al. Developmental kinetics, turnover, and stimulatory capacity of thymic epithelial cells. Blood. 108 (12), 3777-3785 (2006).
- Godfrey, D. I., Kennedy, J., Suda, T., Zlotnik, A. A developmental pathway involving four phenotypically and functionally distinct subsets of CD3-CD4-CD8- triple-negative adult mouse thymocytes defined by CD44 and CD25 expression. Journal of Immunology. 150 (10), 4244-4252 (1993).
- Wang, Y. B., Edinger, M., Mittar, D., McIntyre, C. Studying mouse thymocyte development using multiparametric flow cytometry: An efficient method to improve an 8-color panel on the BD FACSVerse™ system. BD Biosciences–Application Note. , (2012).
- Sakaguchi, N., et al. Altered thymic T-cell selection due to a mutation of the ZAP-70 gene causes autoimmune arthritis in mice. Nature. 426 (6965), 454-460 (2003).
- He, X., Park, K., Kappes, D. J. The role of ThPOK in control of CD4/CD8 lineage commitment. Annual Review of Immunology. 28, 295-320 (2010).
- Wang, Y., et al. A conserved CXXC motif in CD3epsilon is critical for T cell development and TCR signaling. PLoS Biology. 7 (12), e1000253(2009).
- Aliahmad, P., Kadavallore, A., de la Torre, B., Kappes, D., Kaye, J. TOX is required for development of the CD4 T cell lineage gene program. Journal of Immunology. 187 (11), 5931-5940 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados