Method Article
Co-culture et transduction de thymocytes murins sur des cellules stromales exprimant 4-Delta-like pour étudier les oncogènes dans la leucémie à cellules T
Dans cet article
Résumé
Ce protocole décrit l’isolement de thymocytes doublement négatifs du thymus de souris, suivi d’une transduction rétrovirale et d’une co-culture sur le système de co-culture de lignée cellulaire stromale de moelle osseuse de type delta 4-exprimant la moelle osseuse (OP9-DL4) pour une analyse fonctionnelle plus approfondie.
Résumé
Les thymocytes immatures de souris transduits peuvent être différenciés en lymphocytes T in vitro à l’aide du système de co-culture de lignées cellulaires stromales de moelle osseuse de type delta à 4 exprimant la moelle osseuse (OP9-DL4). Comme la transduction rétrovirale nécessite des cellules en division pour l’intégration des transgènes, OP9-DL4 fournit un environnement in vitro approprié pour la culture de cellules progénitrices hématopoïétiques. Ceci est particulièrement avantageux lors de l’étude des effets de l’expression d’un gène spécifique au cours du développement normal des lymphocytes T et de la leucémogenèse, car cela permet aux chercheurs de contourner le processus fastidieux de génération de souris transgéniques. Pour obtenir des résultats positifs, une série d’étapes coordonnées impliquant la manipulation simultanée de différents types de cellules doit être soigneusement effectuée. Bien qu’il s’agisse de procédures très bien établies, l’absence d’une source commune dans la littérature signifie souvent qu’une série d’optimisations sont nécessaires, ce qui peut prendre beaucoup de temps. Ce protocole s’est avéré efficace pour transduire les thymocytes primaires suivis d’une différenciation sur les cellules OP9-DL4. Détaillé ici est un protocole qui peut servir de guide rapide et optimisé pour la co-culture de thymocytes transduits rétroviralement sur des cellules stromales OP9-DL4.
Introduction
La lignée cellulaire stromale de moelle osseuse OP9 fournit un système in vitro utile pour l’induction de la lymphopoïèse à partir de plusieurs sources de progéniteurs1. Les premières études utilisant des cellules OP9 ont démontré que l’absence d’expression du facteur de stimulation des colonies de macrophages (MCSF) contribuait à la capacité de la lignée cellulaire OP9 à soutenir l’hématopoïèse et la différenciation des cellules B à partir de cellules souches hématopoïétiques (CSH) dérivées de la moelle osseuse, comme cela a également été démontré plus tard pour les cellules souches embryonnaires (CSE)2,3,4,5 . Dans des études antérieures, la génération de cellules OP9 exprimant 1/4 de type delta (OP9-DL1/OP9-DL4) a permis l’induction de l’engagement de la lignée des lymphocytes T6 et a démontré la capacité de récapituler avec succès la maturation thymique 7,8. Brièvement, le développement des lymphocytes T a été décrit par l’expression séquentielle des molécules CD4 et CD8. Les thymocytes immatures sont « doublement négatifs » (DN, CD4− CD8−) et peuvent être subdivisés en fonction de l’expression de surface de CD44 et CD25. Les thymocytes DN se différencient par le stade monopositif immature (ISP), caractérisé par l’expression de CD8 chez la souris et de CD4 chez l’homme, suivi du stade double positif (DP), caractérisé par la co-expression de CD4 et CD8, et, enfin, du stade monopositif mature, caractérisé par l’expression de CD4 ou CD89. Les CSH expriment le récepteur Notch1, qui interagit normalement avec le delta-like 4 (DL4) exprimé sur les cellules épithéliales thymiques pour induire la différenciation de la lignée T10. Par conséquent, l’intérêt pour le modèle OP9-DL1/4 a progressivement augmenté, conduisant à l’utilisation extensive de cette approche dans une grande variété d’applications au cours des deux dernières décennies 8,11,12,13. Bien que DL1 et DL4 soient tous deux capables de soutenir la différenciation des lymphocytes T in vitro, ils présentent des exigences différentielles in vivo, et quelques études ont suggéré que OP9_DL4 est plus efficace que OP9_DL1 pour récapituler l’environnement thymique de la souris 7,14.
Parmi les applications potentielles du système OP9-DL1/4, il y a un intérêt particulier pour la combinaison de ce système avec la transduction de cellules DN ou CSH avec des vecteurs rétroviraux. Cette combinaison est un moyen efficace de manipuler l’expression génique au cours du développement normal des lymphocytes T et de la leucémogenèse et s’est avérée être une méthode efficace pour induire ou inhiber la fonction d’un gène d’intérêt15,16,17. Ce modèle a été particulièrement utilisé avec succès pour étudier la collaboration entre les oncogènes à l’origine de la leucémie15 car il est flexible et permet d’examiner les effets de multiples combinaisons de gènes dans un délai raisonnable, contrairement à la génération de souris transgéniques. De plus, des modèles similaires ont été utilisés précédemment pour évaluer les effets de l’introduction d’oncogènes dans les cellules normales15,16,17. De plus, la transduction rétrovirale nécessite des cellules en division pour l’intégration transgénique18, et bien que la transduction lentivirale permettrait de surmonter cette limitation en éliminant le besoin de cellules en division pour l’intégration transgénique, nous n’avons pas été en mesure de réussir la transduction des thymocytes DN en utilisant des vecteurs lentiviraux. Ainsi, OP9-DL1/DL4 est un outil approprié pour la croissance de cellules progénitrices hématopoïétiques.
Le protocole standard pour la lymphopoïèse des thymocytes transduits sur OP9-DL4 implique une série d’étapes coordonnées qui doivent être soigneusement effectuées pour obtenir un résultat positif. Bien qu’il s’agisse de techniques qui servent bien la communauté depuis de nombreuses années, les protocoles disponibles dans la littérature sont souvent fragmentés. En conséquence, chaque laboratoire est obligé d’adapter et d’optimiser différentes étapes de la procédure, ce qui peut prendre beaucoup de temps. Ici, ce protocole décrit l’isolement des thymocytes DN du thymus de souris, suivi d’une transduction rétrovirale et d’une co-culture sur des cellules stromales OP9-DL4 pour une analyse fonctionnelle plus approfondie. Ce protocole établi s’est avéré efficace et reproductible dans la transduction des thymocytes primaires, suivie d’une différenciation sur les cellules OP9-DL4 ou de l’induction de la leucémie lymphoblastique aiguë à cellules T15.
Protocole
Toutes les expériences sur les animaux décrites ont été approuvées par le Comité institutionnel de biosécurité (IBC) et le Comité de soin et d’utilisation des animaux (ACUC) des NIH. Voir le tableau des matériaux pour plus de détails sur tous les réactifs et matériaux utilisés dans ce protocole. Reportez-vous aux lignes directrices publiées19 pour plus de détails sur la culture cellulaire et les procédures d’entretien des producteurs de rétrovirus. Voir la figure 1 pour une vue d’ensemble du protocole.
1. Démarrage de la culture de cellules OP9-DL4 (Jour 1)
- Décongeler rapidement les cellules OP9-DL4 en tenant le flacon et en agitant doucement au bain-marie à 37 °C. Transférer immédiatement les cellules dans un tube centrifuge contenant 5 mL du milieu OP9 (milieu MEM-alpha, FBS à 10 %, 50 U/mL de pénicilline/streptomycine, 55 μM de 2-mercaptoéthanol, 2 mM de L-glutamine) pour éliminer les agents cryoprotecteurs.
- Centrifuger à 300 × g pendant 5 min à température ambiante. Jeter le surnageant et remettre les cellules en suspension dans 1 mL de milieu OP9.
- Placer 5 mL de milieu OP9 dans une fiole T25 et ajouter les cellules en suspension dans cette fiole. Culture à 37 °C et 5 % de CO2.
- Après 2 à 3 jours, sous-culture des cellules en utilisant une division 1:3 (passage des cellules tous les lundis, mercredis et vendredis).
REMARQUE: Divisez les cellules OP9-DL4 avant qu’elles n’atteignent la confluence. Le flacon OP9-DL4 doit être confluent à 80 % à 90 % les jours 6 et 7 et les jours 8 à 9+ pour la coculture avec des thymocytes. Par conséquent, il est important de planifier le nombre de cellules OP9-DL4 nécessaires ces jours-là lors de la division des cellules OP9-DL4.- Jeter le milieu de la fiole et laver la monocouche avec 1x PBS. Jeter le PBS, ajouter 1 mL de trypsine à 0,25 % et incuber pendant 1 à 5 minutes à 37 °C, ou jusqu’à ce que les cellules se soient délogées de la fiole. Frappez la fiole pour déloger les cellules.
NOTE: (Facultatif) La progression de la dissociation cellulaire peut être vérifiée par microscopie. - Ajouter 5 mL de milieu OP9 pour inactiver la trypsine et remettre les cellules en suspension en rinçant la surface recouverte de cellules du ballon pendant la remise en suspension.
- Centrifuger la suspension cellulaire à 300 × g pendant 5 min à température ambiante et la remettre en suspension dans 3 mL de milieu OP9 (pour une division 1:3).
- Placer 1 mL de la suspension cellulaire dans une fiole T25 contenant déjà 5 mL de milieu OP9 frais. Congeler/jeter les cellules restantes.
- Pour congeler les cellules OP9-DL4, remettre les cellules en suspension dans du FBS à 90 % / 10 % de DMSO dans un rapport de 1 mL de milieu de congélation pour un flacon de cellules.
- Congeler dans des flacons de 1 mL à −80 °C ou dans de l’azote liquide, selon les besoins futurs. Conserver dans de l’azote liquide pour un stockage prolongé.
- Jeter le milieu de la fiole et laver la monocouche avec 1x PBS. Jeter le PBS, ajouter 1 mL de trypsine à 0,25 % et incuber pendant 1 à 5 minutes à 37 °C, ou jusqu’à ce que les cellules se soient délogées de la fiole. Frappez la fiole pour déloger les cellules.
2. Démarrage de la culture de la lignée cellulaire productrice de rétrovirus (Jour 1)
- Décongeler rapidement la lignée cellulaire productrice de rétrovirus en tenant le flacon et en agitant doucement au bain-marie à 37 °C. Transférer immédiatement les cellules dans un tube centrifuge contenant 5 mL de milieu cellulaire producteur de rétrovirus (RPC : DMEM, 10 % FBS) pour éliminer l’agent cryoprotecteur.
- Centrifuger à 300 × g pendant 5 min à température ambiante. Jeter le surnageant et remettre les cellules en suspension dans 1 mL de milieu RPC.
- Placer 5 mL de milieu RPC dans une fiole T25 et ajouter les cellules en suspension dans cette fiole. Culture à 37 °C et 5 % de CO2.
- Passez les cellules tous les 2 à 3 jours à une répartition de 1:5 à 1:8.
- Frappez la fiole pour déloger les cellules et remettez les cellules en suspens en les pipetant agressivement.
NOTE: Ces cellules peuvent être retirées de la surface du flacon par pipetage agressif sans trypsine. Alternativement, trypsiniser (0,05% trypsine / 0,53 mM EDTA) jusqu’à ce que les cellules se détachent facilement et peuvent être facilement pipetées dans une suspension unicellulaire.
- Frappez la fiole pour déloger les cellules et remettez les cellules en suspens en les pipetant agressivement.
- Centrifuger la suspension cellulaire à 300 × g pendant 5 min, puis remettre en suspension dans 5 mL de milieu RPC (pour une division 1:5).
- Placer 1 mL de la suspension cellulaire dans une fiole T25 contenant déjà 5 mL de milieu RPC frais. Congeler/jeter les cellules restantes. Congeler les cellules productrices de rétrovirus de la même manière que celle décrite pour les cellules OP9-DL4 (étapes 1.4.4.1 à 1.4.4.2).
3. Début de la culture des cellules productrices de rétrovirus dans des plaques à 6 puits (jours 4 et 5)
- Ensemencer les cellules productrices de rétrovirus 18–24 h avant la transfection à une confluence de 70 à 90 % dans chaque puits d’une plaque de culture tissulaire à 6 puits dans 2 mL de milieu RPC, et incuber à 37 °C et 5 % de CO2.
REMARQUE : Transfecter deux puits de cellules productrices de rétrovirus pour chaque 2 à 5 × 105 à 1 × 106 thymocytes qui seront transduits.
4. Transfecter les cellules productrices de rétrovirus pour générer le rétrovirus contenant les gènes d’intérêt (jours 5 et 6)
- Remplacer le milieu par un milieu RPC frais 1 h avant la transfection.
- Préparer des mélanges de lipofection (pour chaque puits d’une plaque à 6 puits) – augmenter au besoin) : Diluer 4 μg d’ADN (2 μg de plasmide auxiliaire (pCL-Eco) et 2 μg de plasmide de transfert (pMIG) dans 250 μL de milieu sérique réduit. Mélanger délicatement.
- Mélanger 10 μL de réactif de transfection avec 250 uL du milieu sérique réduit de l’étape 4.2. Incuber pendant 5 min à température ambiante.
- Après 5 min d’incubation, combiner l’ADN dilué avec le réactif de transfection dilué (volume total = 500 μL). Mélanger doucement et incuber pendant 20–25 min à température ambiante.
- Ajouter doucement les 500 μL de mélanges de réactifs ADN/transfection dans le puits contenant les cellules productrices de rétrovirus en les déposant sur les cellules avec un mouvement circulaire. Mélanger doucement en balançant la plaque d’avant en arrière et incuber la plaque dans un incubateur à 37 °C pendant 16–24 h.
REMARQUE : Les cellules sous-confluentes (80 % à 90 %) conviennent mieux à la transfection et génèrent potentiellement le titre viral le plus élevé. Comme les cellules productrices de rétrovirus se délogent très facilement du flacon, évitez les mouvements brusques lors de la manipulation de ces cellules.
5. Changement du milieu cellulaire producteur de rétrovirus (jours 6 et 7)
- Remplacer l’ancien milieu environ 16 h après la transfection par 2 mL de milieu RPC frais. Continuer à incuber les cellules à 37 °C pendant 20–24 h.
- Évaluer l’efficacité de la transfection au microscope à fluorescence (facultatif).
REMARQUE : Dans ce protocole, les cellules GFP positives sont les cellules d’intérêt (voir Figure 2). Cette étape dépend du vecteur rétroviral choisi pour être utilisé dans ce protocole (si un gène rapporteur est présent dans le squelette).
6. Préparation des thymocytes et déplétion des cellules CD4+ et CD8+ (jours 6 et 7)
- Récoltez le thymus d’une souris C57BL/6 âgée de 4 à 6 semaines. Pour plus de détails sur la récolte du thymus, reportez-vous à Xing et Hogquist20.
REMARQUE : Les souris ont été euthanasiées par inhalation de CO2 suivie d’une luxation cervicale. - Préparer une suspension unicellulaire de thymocytes en plaçant le thymus dans 5 ml de PBS dans une boîte de Pétri. À l’aide de lames de verre stériles, placez le thymus entre les surfaces givrées des lames et frottez doucement les lames ensemble, en roulant le thymus entre les deux lames. Rincez les lames de verre pour recueillir les cellules et jetez le reste du tissu stromal thymique.
REMARQUE : Le rendement estimé d’un thymus est de 90 × 10 6–100 × 106 cellules, et environ 1 % des cellules resteront après l’épuisement de CD4 et CD8. Utilisez le tissu du thymus de souris plus jeunes pour un meilleur rendement en cellules, car la cellularité du thymus de souris dans les premières semaines après la naissance augmente rapidement, atteint un plateau à l’âge de 4 à 6 semaines et s’involue progressivement par lasuite21. Comptez les cellules à l’aide d’un compteur automatique de cellules sanguines ou de toute autre méthode. - Filtrer la suspension thymique à travers un filtre de 30 μm ou 40 μm en pipetant 5 mL de la suspension thymique à travers une passoire cellulaire. Centrifuger les cellules à 300 × g pendant 10 min.
- Retirer le surnageant et lyser les globules rouges avec un tampon de lyse ACK en ajoutant (selon la taille de l’échantillon) 1 mL de tampon par tube pendant 1 min. Ajouter 5 mL de tampon d’épuisement cellulaire (500 mL de PBS [pH 7,2], 0,5 % BSA, 2 mM 0,5M EDTA, pH 8,0) pour désactiver le tampon de lyse ACK, centrifuger à 300 × g pendant 10 minutes et remettre en suspension dans 1 à 5 mL de tampon de déplétion cellulaire pour compter.
REMARQUE : Réserver sur la glace avant l’épuisement (avant l’épuisement) : 70 μL de suspension cellulaire à compter (varient selon la méthode de cellule de comptage choisie) et 200 μL de suspension cellulaire pour déterminer l’efficacité de la déplétion par coloration pour CD4 et CD8 et analyse par cytométrie en flux22,23. - Compter les cellules et centrifuger à 300 × g pendant 10 min. Remettez les cellules en suspension à 1 × 107/80 μL de tampon d’épuisement cellulaire. Ajouter 10 μL de microbilles CD4 et CD8 par 1 × 107 cellules. Bien mélanger et incuber pendant 15 min dans l’obscurité au réfrigérateur (2–8 °C).
- Préparez une colonne d’épuisement en la rinçant avec 2 ml de tampon d’épuisement et en jetant le tampon d’écoulement.
- Laver les cellules de l’étape 6.6 en ajoutant 1 à 2 mL de tampon de déplétion par 1 × 107 cellules, et centrifuger à 300 × g pendant 10 min. Retirez et jetez le surnageant.
- Remettez en suspension jusqu’à 1,25 × 108 cellules dans 500 μL de tampon d’épuisement. Appliquez la suspension cellulaire sur la colonne et collectez le flux continu (cellules non étiquetées). Lavez la colonne 2x avec 1 mL de tampon et recueillez le flux.
REMARQUE : N’ajoutez un nouveau tampon que lorsque le réservoir de colonne est vide. Réserver sur la glace après l’épuisement (contrôle après déplétion) : 70 μL de suspension cellulaire à compter (varient selon la méthode de cellule de comptage choisie) et 1 000 μL de suspension cellulaire pour déterminer l’efficacité de la déplétion par coloration pour CD4 et CD8 et analyse par cytométrie de flux22,23. - Colorer les contrôles d’efficacité de déplétion en divisant les 200 μL de cellules recueillies avant l’épuisement en quatre tubes FACS : non colorés, CD4 mono-colorés, CD8 mono-colorés et CD4/CD8 double-colorés (utilisez les échantillons non colorés et monocolorés pour établir les paramètres de cytométrie en flux). Utiliser les 1 000 μL de cellules prélevées après l’épuisement pour colorer les CD4 et CD8 et comparer avec l’échantillon à double coloration prélevé avant l’épuisement (voir les résultats typiques de la déplétion à la figure 3).
REMARQUE : Ajustez les volumes pour la coloration par cytométrie en flux selon la recommandation du fabricant de l’anticorps (p. ex., 1 μL d’anti-CD4-FITC + 0,5 μL d’anti-CD8-PE par 1 x 106 cellules dans 50 μL de tampon).
7. Culture de thymocytes sur des cellules OP9-DL4 (jours 6 et 7)
- Placer 2–5 × 105 à 1 × 106 thymocytes post-déplétion dans une fiole T25 de cellules OP9-DL4 confluentes à 80 %–90 % dans un milieu OP9 contenant des cytokines (IL-7 recombinante 10 ng/mL et hFLT-3 recombinante). Culture à 37 °C et 5 % de CO2. Cultiver pendant environ 24 h sur des cellules OP9-DL4.
REMARQUE : Cette durée est nécessaire pour rendre les lymphocytes T transductibles (voir un résultat typique à la figure 4). Réserver une fiole T25 de co-culture de thymocytes et de cellules OP9-DL4 à utiliser comme témoin (non transduit). Les thymocytes non transduits seront colorés avec les thymocytes transducs (étape 9.1) comme contrôle négatif pour évaluer l’efficacité de la transduction. Les thymocytes non transduits peuvent également être utilisés pour mesurer l’effet de l’expression du transgène sur la différenciation cellulaire. Jetez le milieu contenant des cytokines après 1 mois.
8. Récolter le rétrovirus du surnageant et l’utiliser pour transduire les thymocytes (jours 8 et 9)
- Recueillir le surnageant contenant les rétrovirus des cellules transfectées en inclinant la plaque à 6 puits et en plaçant une seringue de 3 à 5 ml au fond de la plaque tout en tirant sur le piston pour aspirer le surnageant. Remplacer le milieu par 2 ml de milieu RPC frais. Continuer à incuber les cellules à 37 °C pendant 20–24 h pour la deuxième transduction à l’étape 8.9.
- Filtrer le surnageant rétroviral à travers un filtre à seringue de 0,45 μm et recueillir le filtrat dans un tube de 50 mL.
REMARQUE : Ne congelez pas le surnageant rétroviral. Utilisez une préparation virale fraîchement préparée pour la transduction. - Recueillir les thymocytes de la culture OP9-DL4 par pipetage agressif pour éliminer les thymocytes et les cellules OP9-DL4 de la surface du flacon. Filtrer la suspension cellulaire à travers une crépine cellulaire de 40 μm pour éliminer la plupart des cellules OP9-DL4 et recueillir le filtrat dans un tube de 50 mL.
REMARQUE: Les cellules OP9 sont très adhérentes. Bien qu’un pipetage agressif puisse éliminer certaines des cellules OP9-DL4 du flacon, la perturbation de la monocouche OP9-DL4 au cours de ce processus est minime, et les cellules OP9-DL4 qui se détachent seront filtrées avec la crépine cellulaire de 40 μm, car les cellules OP9-DL4 sont beaucoup plus grandes que les thymocytes primaires. Si les cellules OP9-DL4 sont encore confluentes à 80 % à 90 %, retirez les thymocytes et replaquez-les dans le même flacon OP9-DL4. Une nouvelle fiole OP9-DL4 doit également être préparée. - Centrifuger les thymocytes dans le filtrat à partir de l’étape 8.3 à 300 × g pendant 5 min. Jetez le surnageant.
- Dans le tube de 50 mL, remettre les thymocytes en suspension dans 0,5 à 1 mL de milieu OP9 + cytokines (voir étape 7.1). Ajouter 1 à 2 mL de milieu RPC contenant le virus (deux fois plus de milieu RPC que de milieu thymocytaire). Ajouter le bromure d’hexaméthrine (concentration stockaire de 10 μg/μL) pour obtenir 8 μg de bromure d’hexaméthrine/mL de suspension cellulaire totale.
- Spinoculate par centrifugation des cellules à 850 × g pendant 1 h à température ambiante.
- Remettez les cellules en suspension dans 6 mL de milieu OP9 + cytokines par fiole (voir étape 7.1) et ajoutez la suspension sur la monocouche cellulaire OP9-DL4.
REMARQUE: Vous pouvez également avoir de nouvelles fioles OP9-DL4 prêtes à recevoir les thymocytes transduits. Si les thymocytes doivent être remis dans une fiole OP9-DL4 usagée, assurez-vous d’ajouter un milieu OP9 frais aux cellules OP9-DL4 pendant les 1 h de spinoculation thymocytaire pour garder les cellules en bonne santé et assurer une confluence de 80% à 90%. - Incuber à 37 °C pendant une nuit.
- Répétez les étapes 8.2 à 8.7 à l’aide d’un nouveau puits du surnageant cellulaire contenant le rétrovirus.
9. Maintenir les thymocytes transduits sur culture OP9-DL4 pendant 2 à 5 jours ou congeler au besoin (Jour 9+)
- Évaluer la différenciation des thymocytes par cytométrie de flux en colorant les thymocytes pour les marqueurs de développement des lymphocytes T tels que CD4, CD8, CD25 et CD4423. Voir les résultats typiques de la différenciation des thymocytes à la figure 5.
Résultats
L’efficacité de la déplétion peut être évaluée cytométriquement en marquant la fraction cellulaire magnétiquement non marquée pour CD4 et CD8 après séparation cellulaire immunomagnétique (MACS) et en l’analysant sur un diagramme à points bivarié bidimensionnel (Figure 3). Un bon rendement de cellules doublement négatives (CD4−, CD8−) est de 95 % ou plus, comme le montre la figure 3. Deux des causes les plus courantes de rendement inférieur sont l’erreur de calcul des microbilles basée sur le nombre de cellules et le nombre de cellules marquées dépassant la capacité de la colonne. Il est recommandé de choisir le nombre correct de colonnes MACS en fonction du nombre de cellules étiquetées. Lorsque vous travaillez avec des thymocytes, le nombre de cellules marquées (cellules DP et SP) est presque égal au nombre total de cellules (plus de 96%). Le comptage des cellules peut être effectué dans un compteur de cellules automatique, une chambre de Neubauer ou avec toute autre méthode. Par conséquent, les volumes alloués au comptage des cellules peuvent devoir être ajustés en fonction de la machine spécifique et de la méthode de comptage choisie.
Il est important de déterminer le nombre de cellules présentes avant et après l’épuisement. Ces numérations cellulaires sont nécessaires pour calculer le nombre de colonnes de déplétion LD nécessaires et pour répartir uniformément les thymocytes DN dans le nombre approprié de flacons OP9-DL4. Les contrôles de la cytométrie en flux (cellules non marquées, cellules marquées pour CD4 et cellules marquées pour CD8) peuvent être effectués avec l’échantillon de pré-déplétion réservé, car il contient plus de cellules. Cependant, comme la plupart des cellules devraient être conservées dans la colonne, l’échantillon post-déplétion à étiqueter nécessitera un volume plus élevé. Par conséquent, des ajustements peuvent être nécessaires en fonction du protocole d’étiquetage sélectionné.
Lors de l’utilisation de vecteurs qui expriment des marqueurs contrôlables, tels qu’un gène de fluorescence, la transfection et la transduction peuvent être évaluées grossièrement et empiriquement par microscopie à fluorescence (Figure 2). L’efficacité de la transduction peut être analysée en récoltant le thymocyte de la monocouche OP9-DL4, comme décrit à l’étape 8.3, et en examinant l’expression d’un gène de fluorescence par cytométrie en flux. L’efficacité de la transduction à l’aide d’un vecteur rétroviral vide avec GFP comme gène rapporteur était de 84,2% (Figure 4).
La différenciation des lymphocytes T sur les cellules OP9-DL4 peut être observée 4 jours après la transduction. La cytométrie en flux est généralement réalisée pour évaluer la différenciation cellulaire induite par la co-culture sur les cellules OP9-DL4 et / ou l’expression transgénique, comme représenté sur la figure 5, où les cellules ont été marquées pour CD4, CD8, CD44 et CD25. Il existe plusieurs combinaisons possibles de marquage de molécules de surface cellulaire qui se sont révélées utiles pour étudier les mécanismes moléculaires et cellulaires du développement des lymphocytes T chez la souris24,25,26,27. Par conséquent, les panels d’anticorps fluorescents peuvent varier en fonction de la question d’intérêt traitée. La transduction des thymocytes DN avec le vecteur rétroviral vide pMIG, illustrée dans le panel B, présentait approximativement les mêmes proportions de positifs simples (CD4+ ou CD8+), de doubles positifs (CD4+/CD8+), de doubles négatifs (CD4−/CD8−) et de ses sous-stades doublement négatifs 1–4 (DN1–DN4) que les thymocytes non transduits, montrés dans le panel A, ce qui indique que le développement des lymphocytes T n’a pas été affecté par le processus de transduction.
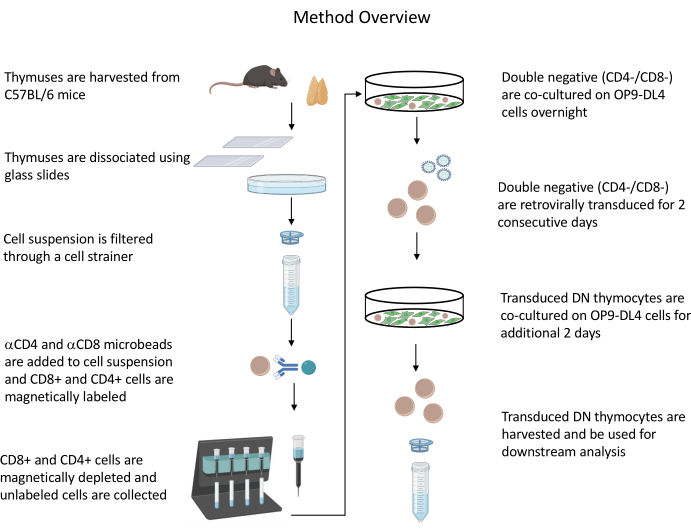
Figure 1 : Schéma des étapes de l’isolement, de la transduction et de la co-culture des thymocytes. Abréviation : DN = double négatif. Veuillez cliquer ici pour voir une version agrandie de cette figure.
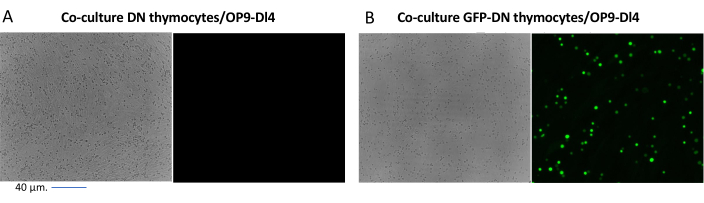
Figure 2 : Microscopie à fluorescence en coculture de thymocytes transduits par GFP ou de thymocytes non transduits et de cellules OP9-DL4. (A) Thymocytes non transduits et (B) thymocytes murins stables exprimant la GFP sur OP9-DL4 au jour 3 après la deuxième transduction. Un microscope à fluorescence Olympus-IX71 avec une lentille 40x et un filtre 480/30 adapté à la détection GFP a été utilisé. Barre d’échelle = 40 μm. Abréviations : GFP = protéine fluorescente verte; DN = double négatif. Veuillez cliquer ici pour voir une version agrandie de cette figure.
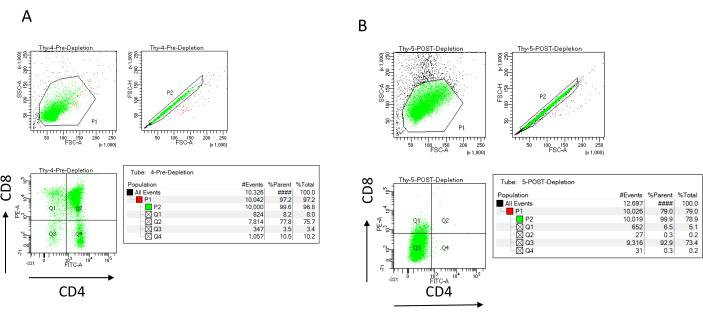
Figure 3 : Cytométrie en flux de la déplétion des thymocytes. Tracés représentatifs de cytométrie en flux analysant l’expression de CD4 et CD8 sur des thymocytes obtenus à partir de souris femelles C57BL/6J âgées de 7 à 8 semaines (A) avant la déplétion et (B) après la déplétion de CD4+ et CD8+ à l’aide de microbilles et de colonnes LD conformément aux instructions du fabricant. Les diagrammes à points à gauche montrent les portes en fonction de la taille et de la complexité de l’événement (FCS-A et SSC-A, respectivement). Le panneau du milieu montre FSC-H par rapport à FSC-A pour ouvrir des cellules individuelles et exclure les doublets. Dans les graphiques de droite, les cellules ont été définies comme CD4 et CD8 à partir de la porte unicellulaire. Abréviations : FSC-A = zone de pic de diffusion vers l’avant; SSC-A = aire de pic de diffusion latérale; FSC-H = hauteur du pic de diffusion vers l’avant; FITC = isothiocyanate de fluorescéine; PE = phycoérythrine. Veuillez cliquer ici pour voir une version agrandie de cette figure.
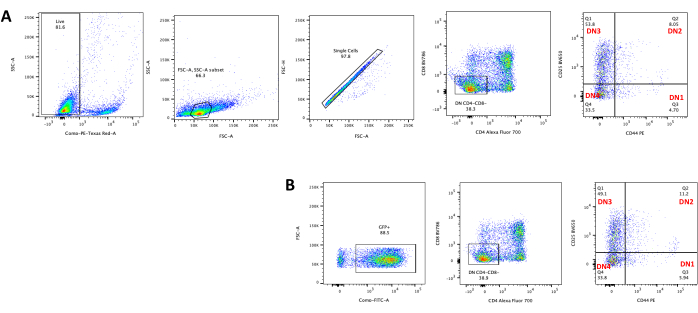
Figure 4 : Cytométrie en flux de l’efficacité de la transduction rétrovirale des thymocytes. (A) Thymocytes non transduits co-cultivés sur OP9-DL4; (B) thymocytes transduits rétroviralement sur OP9-DL4 au jour 3 après la transduction. Abréviations : FSC-A = zone de pic de diffusion vers l’avant; SSC-A = aire de pic de diffusion latérale; FSC-H = hauteur du pic de diffusion vers l’avant; FITC = isothiocyanate de fluorescéine; PE = phycoérythrine; DN = double négatif. Veuillez cliquer ici pour voir une version agrandie de cette figure.
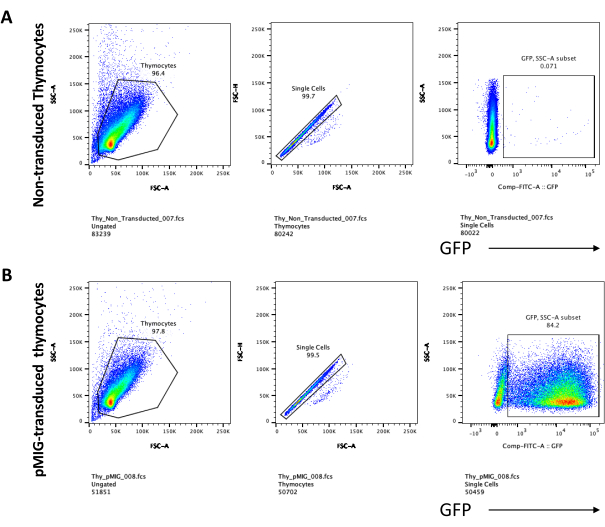
Figure 5 : Cytométrie en flux de thymocytes transduits après 4 jours de co-culture OP9-DL4. (A) non transduit et (B) transduit avec le rétrovirus pMIG. Les cellules ont d’abord été fermées sur des cellules vivantes, puis fermées en fonction de la taille et de la complexité (FCS-A et SSC-A, respectivement), puis ont été repérées par le tracé FSC-H par rapport au FSC-A pour la porte sur des cellules individuelles et exclure les doublets. Pour les cellules non transdudues, nous avons utilisé la stratégie de porte suivante. À partir de la porte unicellulaire, les cellules ont été définies comme étant des positifs simples (CD4+ ou CD8+), des doubles positifs (CD4+/CD8+) et des doubles négatifs (CD4−/CD8−). Ensuite, à partir de la porte des doubles négatifs (CD4−/CD8−), les cellules ont été définies comme CD44+, CD25+, CD44+/CD25+ et CD44−/CD25−, comme le montre le panneau A. Pour les cellules transduites pMIG, montrées dans le panneau B, à partir de la porte unicellulaire, les cellules ont d’abord été définies comme GFP+/GFP−, puis à partir de cellules GFP+, la distribution de la population cellulaire dans les principaux stades de développement des lymphocytes T, tels que définis par l’expression de CD4, CD8, CD44 et CD25, a été déterminée en utilisant la même stratégie de porte que pour les cellules non tradues. Abréviations : FSC-A = zone de pic de diffusion vers l’avant; SSC-A = aire de pic de diffusion latérale; FSC-H = hauteur du pic de diffusion vers l’avant; FITC = isothiocyanate de fluorescéine; GFP = protéine fluorescente verte. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Le protocole décrit ici a été développé spécifiquement pour les études sur les lymphocytes T DN dérivés du thymus (CD4−/CD8−) avec transfection rétrovirale suivie d’un modèle de différenciation OP9-DL4. Cependant, il est probable que les cellules cibles qui seront soumises à ce protocole de transduction suivi de différenciation cellulaire auront une utilité interdisciplinaire plus large. Ainsi, en plus des thymocytes immatures, les cellules souches hématopoïétiques, telles que les cellules dérivées du foie fœtal ou de la moelle osseuse, pourraient potentiellement être utilisées dans ce protocole.
Le système OP9-DL4 s’est avéré être un modèle efficace pour étudier la fonction des gènes dans une variété d’aspects, y compris la différenciation cellulaire17 et l’oncogenèse15. Bien que la modification rétrovirale des progéniteurs hématopoïétiques soit une technique bien établie qui permet une modification génétique stable, la combinaison de l’induction de la différenciation cellulaire sur le système OP9-DL4 et de la transduction rétrovirale nécessite des soins et des compétences. L’aspect critique pour réussir avec ce protocole est de s’assurer que toutes les étapes sont bien coordonnées, car le protocole implique l’utilisation de trois types de cellules différentes qui doivent être maintenues en bonne santé et à la confluence idéale requise pour chaque étape spécifique. Dans cet esprit, il est important d’effectuer toutes les analyses de points de contrôle de la qualité après l’exécution de chaque étape avant de passer à l’étape suivante. Cela garantira que toutes les étapes fonctionnent. Par conséquent, la vérification de l’efficacité de la déplétion, de la transfection et de la transduction est importante pour la bonne exécution de ce protocole (voir un résultat typique pour l’efficacité de la transduction à la figure 4). Une bonne efficacité de transduction des cellules primaires est liée à un titre viral élevé. En règle générale, les encarts plus grands entraînent des titres de virus plus faibles18. À des fins d’entraînement, nous utilisons un vecteur vide pour représenter les résultats qui peuvent être obtenus avec ce protocole. D’après notre expérience, les efficacités de transduction et de transfection varient en fonction de la taille de l’insert, en particulier compte tenu des gènes rapporteurs exprimant le squelette viral, tels que la GFP. Une stratégie qui peut être utilisée lors de l’étude de l’interaction de plus d’un gène consiste à cloner chaque gène dans un vecteur de transfert différent, suivi de la production de virus individuels et, enfin, de la co-transduction de la cellule cible. Dans ce cas, une étape de sélection peut être appliquée pour éliminer les cellules transduites individuellement et ne conserver que les cellules co-transduites.
Il convient de noter que la majorité des lignées cellulaires de couche d’alimentation stromale OP9-DL1/DL4 ont été génétiquement modifiées pour exprimer la GFP dans le cadre des constructions DL1 ou DL47. Dans ce protocole, nous avons utilisé un vecteur rétroviral qui exprime également la GFP ; cependant, elle est plus brillante que la protéine GFP exprimée par les cellules OP9-DL4 et n’interfère pas avec l’inspection de transduction lors de la visualisation de la co-culture au microscope à fluorescence.
Les cellules OP9 se différencient en adipocytes après de nombreux passages, de longues périodes en culture, ou dans des conditions de surconfluence19. Ceci est démontré par le développement de grandes vacuoles. Par conséquent, les cellules OP9 présentant ces caractéristiques ne doivent pas être utilisées dans ce protocole. Le transfectage de cellules productrices de rétrovirus trop confluentes entraînera un faible titre de virus. En effet, le stade sous-confluent est celui où les cellules sont les plus transfectables. De plus, le transfectage de cellules productrices de rétrovirus à faible confluence diminuera le stress cellulaire dans le processus de transfection et donnera le titre de virus le plus élevé.
Bien que, dans ce protocole, nous ne titreons pas le surnageant du virus, le titrage du surnageant viral doit être envisagé dans certains cas, comme en l’absence d’un gène rapporteur dans le vecteur rétroviral, ce qui empêcherait la détermination indirecte de la production virale, ou dans les cas où la conception expérimentale nécessite un nombre plus précis de copies du transgène à intégrer dans le génome de la cellule cible. Cependant, il est important de noter que le titre du surnageant vecteur rétroviral diminue considérablement lorsqu’il est stocké à -80 °C ou 4 °C jusqu’à ce que les résultats du titrage soient obtenus. Par conséquent, l’utilisation d’un surnageant de virus fraîchement préparé pour la transduction donnera une meilleure efficacité de transduction.
Le thymus contient un grand nombre de thymocytes doublement positifs (DP) (plus de 85%) et environ 10% decellules 15 simples positives (CD4 ou CD8), qui sont les thymocytes post-DN. Les cellules DP ne peuvent pas survivre in vitro pour la manipulation rétrovirale, tandis que les cellules SP sont intransductibles. Par conséquent, ce protocole peut être appliqué pour générer des thymocytes DN transductibles à vecteur rétroviral.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer
Remerciements
Ce travail a été soutenu par le programme intra-muros de l’Institut national du cancer, projet ZIABC009287. OP9-DL4 a été obtenu du Dr Juan Carlos Zúñiga-Pflücker (Sunnybrook Health Sciences Centre, Toronto, ON, Canada). Les auteurs remercient le NCI-Frederick Laboratory Animal Sciences Program pour son assistance technique continue et ses conseils et contributions expérimentaux, ainsi que Jeff Carrel, Megan Karwan et Kimberly Klarmann pour son assistance en cytométrie en flux. Nous sommes reconnaissants à Howard Young pour ses conseils et ses commentaires essentiels.
matériels
| Name | Company | Catalog Number | Comments |
| 2-mercaptoethanol | Sigma | M3148 | |
| ACK Lysis buffer | Lonza | 10-548E | |
| BSA | Cell Signaling Technology | 9998S | |
| CD4 Microbeads | Miltenyi | 130-049-201 | |
| CD8 Microbeads | Miltenyi | 130-049-401 | |
| Centrifuge 5910R | eppendorf | 5942IP802353 | |
| DMEM | Corning | 10-013-CV | |
| EDTA | Invitrogen | 15575-038 | |
| Fetal calf serum HyClone FBS | ThermoScientific | SH30910.03 | |
| LD columns | Miltenyi | 130-042-901 | |
| L-glutamine | Sigma | G7513 | Freeze glutamine in aliquots and use freshly-thawed glutamine |
| Lipofectamine 2000 | Invitrogen | P/N 52887 | |
| MEM-alpha Medium | Gibco | 12561-072 | |
| OPTI-MEM I Reducing Serum Medium | Invitrogen | 31985-062 | |
| PBS pH 7.2 | Corning | 21-040-CV | |
| pcL-Eco Plasmid | Addgene | 12371 | |
| penicillin/streptomycin | Gibco | 15140-122 | |
| pMIG Plasmid | Addgene | 6492 | |
| Polybrene | Chemicon | TR-1003-G | |
| Pre-Separation Filters | Miltenyi | 130-041-407 | |
| recombinant hFLT-3L | PeproTech | 300-19 | |
| recombinant IL-7 | Peprotech | 217-17 | |
| Retrovial packaging cell line Phoenix-Eco | Orbigen | RVC-10002 | |
| Syringe filter (0.45 µm) | Millipore | SLHV033RS |
Références
- Zuniga-Pflucker, J. C. T-cell development made simple. Nature Reviews Immunology. 4 (1), 67 (2004).
- Kodama, H., Nose, M., Niida, S., Nishikawa, S., Nishikawa, S. Involvement of the c-kit receptor in the adhesion of hematopoietic stem cells to stromal cells. Experimental Hematology. 22 (10), 979-984 (1994).
- Nakano, T., Kodama, H., Honjo, T. Generation of lymphohematopoietic cells from embryonic stem cells in culture. Science. 265 (5175), 1098-1101 (1994).
- Nakano, T., Kodama, H., Honjo, T. In vitro development of primitive and definitive erythrocytes from different precursors. Science. 272 (5262), 722-724 (1996).
- Ueno, H., et al. A stromal cell-derived membrane protein that supports hematopoietic stem cells. Nature Immunology. 4 (5), 457-463 (2003).
- Jaleco, A. C., et al. Differential effects of Notch ligands Delta-1 and Jagged-1 in human lymphoid differentiation. Journal of Experimental Medicine. 194 (7), 991-1002 (2001).
- Mohtashami, M., et al. Direct comparison of Dll1- and Dll4-mediated Notch activation levels shows differential lymphomyeloid lineage commitment outcomes. Journal of Immunology. 185 (2), 867-876 (2010).
- Schmitt, T. M., Zuniga-Pflucker, J. C. Induction of T cell development from hematopoietic progenitor cells by delta-like-1 in vitro. Immunity. 17 (6), 749-756 (2002).
- Mazzucchelli, R., Durum, S. K. Interleukin-7 receptor expression: Intelligent design. Nature Reviews Immunology. 7 (2), 144-154 (2007).
- Robey, E. A., Bluestone, J. A. Notch signaling in lymphocyte development and function. Current Opinion in Immunology. 16 (3), 360-366 (2004).
- Lavaert, M., et al. Integrated scRNA-Seq identifies human postnatal thymus seeding progenitors and regulatory dynamics of differentiating immature thymocytes. Immunity. 52 (6), 1088-1104 (2020).
- Roh, K. H., Roy, K. Engineering approaches for regeneration of T lymphopoiesis. Biomaterials Research. 20 (20), (2016).
- Zlotoff, D. A., et al. CCR7 and CCR9 together recruit hematopoietic progenitors to the adult thymus. Blood. 115 (10), 1897-1905 (2010).
- Holmes, R., Zuniga-Pflucker, J. C. The OP9-DL1 system: Generation of T-lymphocytes from embryonic or hematopoietic stem cells in vitro. Cold Spring Harbor Protocols. 2009 (2), (2009).
- Cramer, S. D., et al. Mutant IL-7Ralpha and mutant NRas are sufficient to induce murine T cell acute lymphoblastic leukemia. Leukemia. 32 (8), 1795-1882 (2018).
- Treanor, L. M., et al. Interleukin-7 receptor mutants initiate early T cell precursor leukemia in murine thymocyte progenitors with multipotent potential. Journal of Experimental Medicine. 211 (4), 701-713 (2014).
- Yokoyama, K., et al. In vivo leukemogenic potential of an interleukin 7 receptor alpha chain mutant in hematopoietic stem and progenitor cells. Blood. 122 (26), 4259-4263 (2013).
- Simmons, A., Alberola-Ila, J. Retroviral transduction of T cells and T cell precursors. Methods in Molecular Biology. 1323, 99-108 (2016).
- . Retroviral systems Available from: https://web.stanford.edu/group/nolan/_OldWebsite/retroviral_systems/retsys.html (2022)
- Xing, Y., Hogquist, K. A. Isolation, identification, and purification of murine thymic epithelial cells. Journal of Visualized Experiments. (90), e51780 (2014).
- Gray, D. H., et al. Developmental kinetics, turnover, and stimulatory capacity of thymic epithelial cells. Blood. 108 (12), 3777-3785 (2006).
- Godfrey, D. I., Kennedy, J., Suda, T., Zlotnik, A. A developmental pathway involving four phenotypically and functionally distinct subsets of CD3-CD4-CD8- triple-negative adult mouse thymocytes defined by CD44 and CD25 expression. Journal of Immunology. 150 (10), 4244-4252 (1993).
- Wang, Y. B., Edinger, M., Mittar, D., McIntyre, C. Studying mouse thymocyte development using multiparametric flow cytometry: An efficient method to improve an 8-color panel on the BD FACSVerse™ system. BD Biosciences–Application Note. , (2012).
- Sakaguchi, N., et al. Altered thymic T-cell selection due to a mutation of the ZAP-70 gene causes autoimmune arthritis in mice. Nature. 426 (6965), 454-460 (2003).
- He, X., Park, K., Kappes, D. J. The role of ThPOK in control of CD4/CD8 lineage commitment. Annual Review of Immunology. 28, 295-320 (2010).
- Wang, Y., et al. A conserved CXXC motif in CD3epsilon is critical for T cell development and TCR signaling. PLoS Biology. 7 (12), e1000253 (2009).
- Aliahmad, P., Kadavallore, A., de la Torre, B., Kappes, D., Kaye, J. TOX is required for development of the CD4 T cell lineage gene program. Journal of Immunology. 187 (11), 5931-5940 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon