Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Ensayo de sedimentación basado en concanavalina A para medir la unión al sustrato de las fosfatasas de glucano
* Estos autores han contribuido por igual
En este artículo
Resumen
Este método describe un ensayo de sedimentación in vitro basado en lectinas para cuantificar la afinidad de unión de la glucano fosfatasa y la amilopectina. Este ensayo de cosedimentación es confiable para medir la unión del sustrato de glucano fosfatasa y se puede aplicar a varios sustratos de glucano solubilizados.
Resumen
Las fosfatasas de glucano pertenecen a la familia más grande de fosfatasas de especificidad dual (DSP) que desfosforilan sustratos de glucano, como el glucógeno en animales y el almidón en plantas. Las estructuras cristalinas de la glucano fosfatasa con sustratos de glucano modelo revelan distintas interfaces de unión a glucanos hechas de DSP y dominios de unión a carbohidratos. Sin embargo, las mediciones cuantitativas de las interacciones glucano-glucano fosfatasa con sustratos fisiológicamente relevantes son fundamentales para la comprensión biológica de la familia de enzimas glucano fosfatasa y la regulación del metabolismo energético. Este manuscrito informa de un ensayo de sedimentación in vitro basado en Concanavalina A (ConA) diseñado para detectar la afinidad de unión al sustrato de las fosfatasas de glucano contra diferentes sustratos de glucano. Como prueba de concepto, se determinó la constante de disociación (KD) de la glucano fosfatasa Arabidopsis thaliana Almidón Exceso4 (SEX4) y amilopectina. La caracterización de los mutantes SEX4 y otros miembros de la familia de enzimas glucano fosfatasa demuestra aún más la utilidad de este ensayo para evaluar la unión diferencial de las interacciones proteína-carbohidrato. Estos datos demuestran la idoneidad de este ensayo para caracterizar una amplia gama de proteínas que interactúan con el almidón y el glucógeno.
Introducción
Las fosfatasas de glucano son miembros de una subfamilia funcionalmente diversa de fosfatasas de especificidad dual (DSP) dentro de la superfamilia1 de la proteína tirosina fosfatasa (PTP). Se han encontrado en la mayoría de las formas de vida, incluyendo organismos fotosintéticos ampliamente divergentes, humanos, vertebrados y algunos invertebrados y protistas 2,3,4. Las plantas contienen tres fosfatasas de glucano conocidas: Almidón Exceso4 (SEX4), Como Sexo Cuatro1 (LSF1) y Como Sexo Cuatro2 (LSF2)5,6,7. Las plantas que carecen de glucano fosfatasas muestran tasas disminuidas de degradación transitoria del almidón y acumulación de almidón en las hojas 8,9. Laforin es el miembro fundador de la familia glucano fosfatasa que desfosforila glucógeno en vertebrados y humanos 3,10. Las mutaciones de laforina resultan en la enfermedad neurodegenerativa de Lafora, una forma autosómica recesiva fatal de epilepsia11. Las glucano fosfatasas son necesarias para el metabolismo del glucógeno y del almidón y han surgido como enzimas importantes para modular el contenido de almidón en las plantas y tratar la enfermedad neurodegenerativa de Lafora12,13. Estudios recientes de cristalografía de rayos X sobre fosfatasas de glucano con sustratos de glucano modelo han arrojado luz sobre la unión al sustrato y el mecanismo catalítico de la desfosforilación de glucanos14,15,16,17. Sin embargo, la comprensión actual de cómo las fosfatasas de glucano se unen a sus sustratos fisiológicos es incompleta.
El almidón es un polímero insoluble de glucosa hecho de 80% -90% de amilopectina y 10% -20% de amilosa18. Los sustratos para las fosfatasas de glucano vegetal son moléculas de carbohidratos fosforilados, como el glucógeno y los gránulos de almidón. Los residuos de glucosilo fosforilados están presentes en una proporción de 1:600 fosfato:residuo de glucosil. Curiosamente, los fosfatos están presentes sólo en las moléculas de amilopectina19. La principal glucano fosfatasa de la planta SEX4 actúa sobre el gránulo de almidón para desfosforilar las moléculas de amilopectina. La estructura cristalina de rayos X de SEX4 combinada con estudios de mutagénesis guiada por estructura ha demostrado las especificidades únicas del sustrato de SEX4 para diferentes posiciones dentro de una estructura de glucano15. Recientemente hemos demostrado que la actividad biológicamente relevante de SEX4 sólo puede observarse cuando actúa sobre sus sustratos de amilopectina solubilizados20. Sin embargo, comprender las interacciones glucano-SEX4 ha demostrado ser difícil debido a la complejidad estructural del sustrato, las especificidades de unión más amplias y las bajas afinidades de unión entre la proteína y sus sustratos. Estos problemas han obstaculizado la capacidad de utilizar métodos comúnmente utilizados en las interacciones proteína-ligando, como la calorimetría de titulación isotérmica (ITC), la espectroscopia de resonancia magnética nuclear (RMN) y los ensayos basados en ensayos de inmunoadsorción ligados a enzimas (ELISA).
Curiosamente, gran parte de nuestra comprensión de las interacciones carbohidratos-proteínas proviene del estudio de las lectinas. La concanavalina A (ConA) es una familia de proteínas lectinas de leguminosas originalmente extraídas del frijol jack. ConA se une a los carbohidratos con alta especificidad, lo que es ventajoso para su uso en aplicaciones de administración y orientación de fármacos. La unión de ConA a una variedad de sustratos que contienen α-D-manosil y α-D-glucosilo no reductores ha sido ampliamente estudiada19,20. Las perlas de sefarosa unidas a ConA disponibles comercialmente se usan comúnmente para purificar glicoproteínas y glicolípidos21. ConA se une a estos glucanos a través de los grupos hidroxilo C3, C4 y C6 de los residuos de glucosa. Las perlas de ConA-Sepharose también se han utilizado con éxito para medir la unión de las interacciones glucógeno-proteína y almidón-proteína22,23. En este estudio, utilizamos perlas de ConA-Sepharose para desarrollar un ensayo de unión para medir las especificidades de unión de las interacciones glucano fosfatasa-amilopectina.
Anteriormente, se empleó un ensayo de sedimentación basado en ConA para evaluar la capacidad de unión al sustrato de glucano fosfatasa14,20,24. En este estudio, se utilizó la misma estrategia para desarrollar un método novedoso para determinar la afinidad de unión de las interacciones glucano-glucano fosfatasa y carbohidratos. Este método también tiene una ventaja para investigar varias interacciones carbohidratos-proteínas solubilizadas.
Protocolo
1. Preparación de perlas de ConA-Sepharose
- Hacer 250 ml de un tampón de unión que contenga 67 mM HEPES (pH 7.5), 10 mM MgCl 2 y 0.2 mM CaCl2. Ajustar el pH con una solución de NaOH 1 M.
- Pipetear 250 μL de suspensión de perlas de ConA-Sepharose en un tubo de microcentrífuga de 1,5 ml. Centrifugar el contenido a 10.000 x g durante 30 s a 4 °C. Deseche el sobrenadante.
NOTA: Se necesitan 250 μL de perlas de ConA-Sepharose en un tubo de microcentrífuga de 1,5 ml para cada concentración de amilopectina utilizada para el ensayo. - Añadir 750 μL del tampón de unión a cada tubo que contenga 250 μL de perlas de ConA-Sepharosa. Centrifugar los tubos a 10.000 x g durante 1 min a 4 °C. Retire el sobrenadante. Repita este paso 2 veces para asegurarse de que las perlas se lavan adecuadamente y se equilibran con el tampón de unión.
2. Preparación de soluciones de amilopectina
- Haga una solución madre de 10 mg/ml de amilopectina de patata. La amilopectina es insoluble en agua y puede ser solubilizada por calor. Para solubilizar, agregue 0.1 g de amilopectina de papa a 10 ml de agua destilada. Calentar la suspensión en un baño maría a 80 °C durante 1 h o hasta que la solución ya no esté turbia.
- Permita que la solución vuelva a la temperatura ambiente (RT), con vórtices repetidos para evitar la formación de grumos.
- El tratamiento alcalino con alcohol es un método alternativo para solubilizar sustratos de amilopectina. Para solubilizar usando este método, siga los pasos a continuación.
- Suspender 0,5 g de sustrato de amilopectina en 5 mL de etanol al 20% y 5 mL de NaOH 2 M. Revuelva el contenido vigorosamente durante 15-20 minutos en RT.
- A continuación, agregue 10 ml de agua y ajuste el pH de la solución a 6.5 agregando 2 M HCl. Aumente el volumen de la solución resultante a 50 ml con agua destilada para hacer una solución de amilopectina de 10 mg / ml.
- Diluir la solución de amilopectina solubilizada de 10 mg/ml para hacer una serie de 2 ml de soluciones diluidas de amilopectina. Por ejemplo, realice medias diluciones de 10 mg/ml para preparar una serie de concentraciones de amilopectina (5 mg/ml, 2,5 mg/ml, 1,25 mg/ml, 0,625 mg/ml, 0,3125 mg/ml, 0,156 mg/ml, 0,078 mg/ml, 0,039 mg/ml, 0,019 mg/ml y 0 mg/ml).
3. Preparación de ConA-Sefarosa: perlas de amilopectina
- Añadir 250 μL de cada solución diluida de amilopectina a 1,5 ml de tubos de microcentrífuga que contengan 250 μL de perlas de ConA-Sefarosa preequilibradas en tampón de unión. Mezcla bien el contenido. Etiquete los tubos con la concentración de amilopectina correspondiente.
- Incubar el contenido en una rueda giratoria a 4 °C durante 30 min.
NOTA: No hay cambios en el complejo ConA-Sepharose:amilopectina unida a lo largo del tiempo después de 20 min. El tiempo de incubación de 30 minutos se eligió variando los tiempos de incubación de 10 minutos a 1 h para garantizar que se alcanzara el equilibrio. - Centrifugar los tubos a 10.000 x g durante 1 min. Recoja el sobrenadante en un tubo de microcentrífuga de 1,5 ml recién etiquetado. Guarde estas fracciones sobrenadantes para realizar el ensayo de D-glucosa12 (hidrólisis ácida de amilopectina seguida de determinación UV de glucosa mediante ensayo enzimático). Este paso es necesario para garantizar que toda la amilopectina esté unida a las perlas.
- Añadir 750 μL de tampón de unión a las perlas de ConA-Sepharosa:amilopectina. Centrifugar los tubos a 10.000 x g durante 1 min. Deseche el sobrenadante para eliminar cualquier molécula de amilopectina no unida.
- Repita el paso 3.4 para garantizar un lavado suficiente. Cada tubo ahora contiene perlas de ConA-Sepharose unidas a cantidades variables de sustratos de amilopectina.
4. Incubación de SEX4 con ConA-Sefarosa: perlas de amilopectina
- Mezcle 250 μL de Perlas de ConA-Sefarosa:amilopectina con 100 μL del tampón de unión que incluye 10 μg de proteína SEX4, 10 mM de ditiothreitol (DTT) y 10 μM de cóctel inhibidor de proteasa (PIC). Tenga en cuenta que el volumen total en cada tubo es de 350 μL.
NOTA: Se agrega un cóctel de inhibidores de proteasa como medida de precaución para evitar cualquier degradación innecesaria de SEX4. Este es un paso opcional. En este ensayo, se utiliza la proteína recombinante Arabidopsis thaliana SEX4 (AtSEX4). La proteína purificada contiene una etiqueta de histidina N-terminal necesaria para detectar la proteína a través de la quimioluminiscencia. La información detallada sobre las purificaciones de glucano fosfatasa se describe en publicaciones anteriores14,20,24. - Incubar la proteína y la suspensión de perlas de ConA-Sefaro: amilopectina a 4 °C durante 45 min con una rotación suave.
NOTA: El tiempo de incubación de 45 minutos se elige para garantizar que se alcance el equilibrio para el complejo. - Centrifugar los tubos a 10.000 x g durante 1 min. Pipetear 50 μL del sobrenadante con cuidado utilizando una punta de carga de gel en un nuevo tubo de microcentrífuga de 1,5 ml. Añadir 20 μL de 4x colorante SDS-PAGE y 10 μL de agua a cada tubo que contenga 50 μL de las fracciones sobrenadantes recogidas. Calentar las muestras a 95 °C durante 10 min. Guarde estos ejemplos para ejecutar los geles SDS-PAGE. Asegúrese de que 10 tubos nuevos etiquetados como "sobrenadante (S)" tengan las concentraciones de sustrato correspondientes.
- Agregue 750 μL del tampón de unión a las perlas ConA-Sepharose:amilopectina: SEX4 para eliminar cualquier proteína no unida de las perlas. Centrifugar los tubos a 10.000 x g durante 1 min. Repita este paso una vez más para garantizar un lavado adecuado. Deseche el sobrenadante.
- Añadir 20 μL de 4x colorante SDS-PAGE y 80 μL de agua destilada en los tubos que contienen perlas ConA-Sepharose:amilopectina:SEX4 lavadas. Calentar las muestras a 95 °C durante 10 min y centrifugar a 10.000 x g durante 1 min.
- Deseche el pellet y guarde el sobrenadante para ejecutar los geles SDS-PAGE. Pipetear 80 μL del sobrenadante en tubos nuevos y etiquetarlos como "pellet (P)".
5. Ejecución de geles SDS-PAGE
- Cargue 40 μL de las muestras de proteína no unidas (realizadas en el paso 2.3, etiquetado como S) en pocillos de gel de poliacrilamida prefabricada al 4% -12% desde la concentración de sustrato más baja hasta la más alta, pero mantenga el primer carril libre para cargar el marcador de peso molecular de la proteína. Use un segundo gel para cargar 10 muestras de proteína unidas hechas en el paso 2.5 (etiquetado como P).
- Agregue el búfer de ejecución SDS-PAGE 1x recién preparado a ambas cámaras del aparato. Pase el gel a 150 V durante 35 minutos o hasta que el frente del tinte llegue al fondo del gel.
- Retire el gel de rodaje del aparato y retire los espaciadores y las placas de vidrio. Use el gel separado para ejecutar un análisis de Western blot.
6. Western blotting para la detección de quimioluminiscencia14,15
NOTA: Este método se puede modificar/adaptar fácilmente dependiendo del equipo de Western Blotting que los usuarios tengan en sus laboratorios.
- Haga 1 L de tampón de transferencia que contenga 5.8 g de Tris base, 2.9 g de glicina, 0.37 g de SDS y 200 ml de metanol.
- Transfiera las proteínas separadas por tamaño del gel de poliacrilamida a una membrana de nitrocelulosa. Ensamble brevemente las esponjas, los papeles de filtro, el gel y la membrana de nitrocelulosa de acuerdo con el protocolo de transferencia occidental14,15. Funcionar a 70 V durante 1 h.
- Para evitar la unión de proteínas inespecíficas, incubar la membrana de nitrocelulosa que contiene la solución proteica de albúmina sérica bovina (BSA) al 1% -5% o proteína láctea en 50 ml de tampón TBST (20 mM Tris [pH 7.5], 150 mM NaCl, 0.1% Tween 20) durante 1 h. Lave la membrana 3 veces usando tampón TBST para eliminar cualquier solución de bloqueo no unida.
- Incubar la membrana con un anticuerpo ligado a la peroxidasa de rábano picante (HRP) específico para la proteína marcada con His durante 1 h. Lave la membrana 3 veces en tampón TBST para eliminar cualquier anticuerpo no unido. Utilice una dilución 1:2.000 de anticuerpos contra TBST para una reproducibilidad y sensibilidad óptimas.
- El anticuerpo HRP unido a la enzima se une específicamente a la etiqueta de histidina de la proteína SEX4, que produce una banda en presencia de reactivos de quimioluminiscencia. Para imágenes digitales, haga una solución de partes iguales de soluciones de sustrato quimioluminiscente (750 μL cada una) en un tubo de 1,5 ml. Incubar la membrana durante al menos 5 minutos en la solución.
- Coloque la proteína de membrana hacia abajo en el escáner de borrones y ejecute el software de adquisición para cuantificar la proteína en las fracciones de pellet y sobrenadante.
7. Análisis de datos
- Realice las mediciones cuantitativas de la señal utilizando el software de adquisición con el escáner de borrones. Normalizar todas las mediciones cuantitativas en las fracciones sobrenadante y pellet a la proteína total cargada.
NOTA: El software permite cuantificar la intensidad de cada banda proteica en las fracciones sobrenadante y pellet. - En el experimento de unión a la saturación, trace el porcentaje de concentración de amilopectina unida a proteínas versus amilopectina. Ajuste los datos a Y = Bmax x X / (K D + X), utilizando software de análisis de datos para calcular KD.
NOTA: Bmax es la unión específica máxima, el eje Y es el porcentaje de unión a proteínas, el eje X es la concentración de amilopectina.
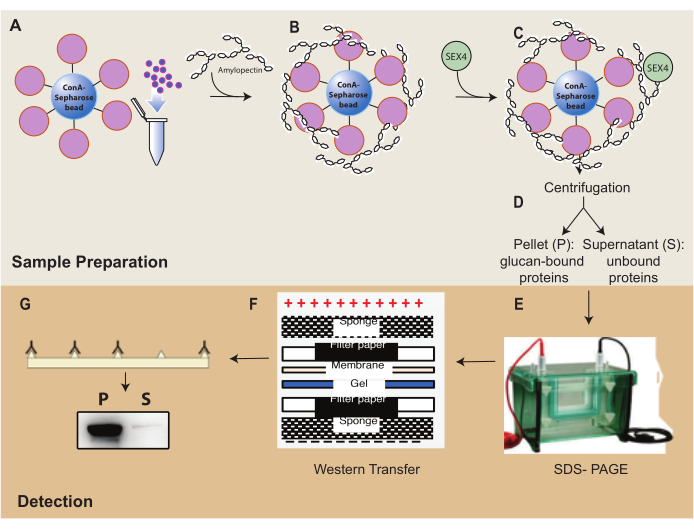
Figura 1: Descripción general del flujo de trabajo del ensayo de sedimentación de ConA-Sepharose. (A) Preparación de perlas de ConA-Sepharose. (B) Incubación con sustrato de amilopectina. (C) Incubación con proteína SEX4. (D) Separación de fracciones de proteínas unidas y no unidas mediante centrifugación. (E) Separación de proteínas a través de SDS-PAGE. (F) Análisis de Western blot. (G) Detección de quimioluminiscencia de la proteína SEX4 marcada con His. Haga clic aquí para ver una versión más grande de esta figura.
Resultados
Una de las características clave de la familia de proteínas glucano fosfatasa es su capacidad para unirse a sustratos de glucano. En primer lugar, se analizó la capacidad de unión de SEX4 a las perlas de ConA-Sepharose:amilopectina mediante SDS-PAGE (Figura 2A). La albúmina sérica bovina (BSA) sirvió como control negativo para detectar cualquier unión inespecífica de proteínas a las perlas de ConA-Sefarosa:amilopectina. El análisis SDS-PAGE de proteínas mostró la presencia de pr...
Discusión
Este estudio demuestra el desarrollo exitoso de un nuevo ensayo de sedimentación in vitro que permite determinar la afinidad de unión de las interacciones glucano-glucano fosfatasa. El diseño del ensayo aprovecha la unión específica de la lectina ConA a los glucanos a través de los residuos hidroxilo de glucosa para capturar indirectamente sustratos de carbohidratos solubilizados en perlas de Sepharose. Esto permite la separación de fracciones de proteínas unidas y no unidas mediante ce...
Divulgaciones
Los autores declaran no tener conflictos de intereses.
Agradecimientos
Este estudio fue apoyado por el premio MCB-2012074 de la National Science Foundation. Los autores agradecen al Dr. Craig W. Vander Kooi del Departamento de Bioquímica y Biología Molecular de la Universidad de Florida por sus valiosas discusiones y apoyo. Los autores también agradecen al Dr. Matthew S. Gentry del Departamento de Bioquímica y Biología Molecular de la Universidad de Florida por su apoyo. Sara Lagalwar, presidenta del programa de Neurociencia de Skidmore College, por permitirnos usar el escáner de borrones de dígitos C LICOR para imágenes de Western Blot.
Materiales
| Name | Company | Catalog Number | Comments |
| 6x-His Tag monoclonal antibody (HIS.H8), HRP | Therm Fisher Scientific | MA1-21315-HRP | |
| Biorad gel electrophoresis and Western blot kit | Biorad | 1703930 | |
| Calcium chloride | Sigma-Aldrich | 208291 | |
| C-Digit blot scanner | LICOR | 3600-00 | Blot scanner |
| Complete protease inhibitor cocktail | Sigma-Aldrich | 11836170001 | |
| Concanavalin A-sepharose beads | Sigma-Aldrich | C9017 | This product contains in 0.1 M acetate buffer, pH 6, containing 1 M NaCl, 1 mM CaCl2, 1 mM MnCl2, and 1 mM MgCl2 in 20% ethanol |
| Centrifuge | Eppendorf | 5425R | |
| Glycine | Fisher Scientific | BP381-5 | |
| GraphPad Prism 8.0 software | GraphPad | Version 8.0 | Data analysis software |
| HEPES | Sigma-Aldrich | H8651 | |
| Image Studio | LICOR | 3600-501 | Acquisition Software |
| Magnesium chloride | Sigma-Aldrich | M2670 | |
| Methanol | Fisher Scientific | A452SK-4 | |
| Sodium dodecyl sulfate | Fisher Scientific | PI28312 | |
| Potato amylopectin | Sigma-Aldrich | A8515 | |
| Precast SDSPAGE Gels | Genscript | M00653S | |
| Tris base | Fisher Scientific | BP154-1 | |
| Tween 20 | Fisher Scientific | MP1TWEEN201 | |
| Westernsure premium chemiluminescence substrate | LI-COR | 926-95000 |
Referencias
- Meekins, D. A., Vander Kooi, C. W., Gentry, M. S. Structural mechanisms of plant glucan phosphatases in starch metabolism. The FEBS Journal. 283 (13), 2427-2447 (2016).
- Gentry, M. S., et al. The phosphatase laforin crosses evolutionary boundaries and links carbohydrate metabolism to neuronal disease. The Journal of Cell Biology. 178 (3), 477-488 (2007).
- Worby, C. A., Gentry, M. S., Dixon, J. E. Laforin, a dual specificity phosphatase that dephosphorylates complex carbohydrates. The Journal of Biological Chemistry. 281 (41), 30412-30418 (2006).
- Gentry, M. S., Pace, R. M. Conservation of the glucan phosphatase laforin is linked to rates of molecular evolution and the glucan metabolism of the organism. BMC Evolutionary Biology. 9, 138 (2009).
- Niittyla, T., et al. Similar protein phosphatases control starch metabolism in plants and glycogen metabolism in mammals. The Journal of Biological Chemistry. 281 (17), 11815-11818 (2006).
- Kotting, O., et al. STARCH-EXCESS4 is a laforin-like Phosphoglucan phosphatase required for starch degradation in Arabidopsis thaliana. The Plant Cell. 21 (1), 334-346 (2009).
- Comparot-Moss, S., et al. A putative phosphatase, LSF1, is required for normal starch turnover in Arabidopsis leaves. Plant Physiology. 152 (2), 685-697 (2010).
- Zeeman, S. C., Northrop, F., Smith, A. M., Rees, T. A starch-accumulating mutant of Arabidopsis thaliana deficient in a chloroplastic starch-hydrolysing enzyme. The Plant Journal: For Cell and Molecular Biology. 15 (3), 357-365 (1998).
- Kotting, O., et al. Identification of a novel enzyme required for starch metabolism in Arabidopsis leaves. The phosphoglucan, water dikinase. Plant Physiology. 137 (1), 242-252 (2005).
- Tagliabracci, V. S., et al. Laforin is a glycogen phosphatase, deficiency of which leads to elevated phosphorylation of glycogen in vivo. Proceedings of the National Academy of Sciences. 104 (49), 19262-19266 (2007).
- Gentry, M. S., Guinovart, J. J., Minassian, B. A., Roach, P. J., Serratosa, J. M. Lafora disease offers a unique window into neuronal glycogen metabolism. The Journal of Biological Chemistry. 293 (19), 7117-7125 (2018).
- Brewer, M. K., et al. Targeting pathogenic lafora bodies in lafora disease using an antibody-enzyme fusion. Cell Metabolism. 30 (4), 689-705 (2019).
- Santelia, D., Zeeman, S. C. Progress in Arabidopsis starch research and potential biotechnological applications. Current Opinion in Biotechnology. 22 (2), 271-280 (2011).
- Raththagala, M., et al. Structural mechanism of laforin function in glycogen dephosphorylation and lafora disease. Molecular Cell. 57 (2), 261-272 (2015).
- Meekins, D. A., et al. Phosphoglucan-bound structure of starch phosphatase Starch Excess4 reveals the mechanism for C6 specificity. Proceedings of the National Academy of Sciences. 111 (20), 7272-7277 (2014).
- Vander Kooi, C. W., et al. Structural basis for the glucan phosphatase activity of Starch Excess4. Proceedings of the National Academy of Sciences. 107 (35), 15379-15384 (2010).
- Meekins, D. A., et al. Structure of the Arabidopsis glucan phosphatase like sex four2 reveals a unique mechanism for starch dephosphorylation. The Plant Cell. 25 (6), 2302-2314 (2013).
- Smith, A. M., Zeeman, S. C. Starch: A flexible, adaptable carbon store coupled to plant growth. Annual Review of Plant Biology. 71, 217-245 (2020).
- Jane, J., Kasemuwan, T., Chen, J. F., Juliano, B. O. Phosphorus in rice and other starches. Cereal Foods World. 41 (11), 827-832 (1996).
- Mak, C. A., et al. Cooperative kinetics of the glucan phosphatase starch excess4. Biochemistry. 60 (31), 2425-2435 (2021).
- Campbell, K. P., MacLennan, D. H. Purification and characterization of the 53,000-dalton glycoprotein from the sarcoplasmic reticulum. The Journal of Biological Chemistry. 256 (9), 4626-4632 (1981).
- Campbell, K. P., MacLennan, D. H., Jorgensen, A. O., Mintzer, M. C. Purification and characterization of calsequestrin from canine cardiac sarcoplasmic reticulum and identification of the 53,000 dalton glycoprotein. The Journal of Biological Chemistry. 258 (2), 1197-1204 (1983).
- Davey, M. W., Sulkowski, E., Carter, W. A. Binding of human fibroblast interferon to concanavalin A-agarose. Involvement of carbohydrate recognition and hydrophobic interaction. Biochemistry. 15 (3), 704-713 (1976).
- Meekins, D. A., et al. Mechanistic insights into glucan phosphatase activity against polyglucan substrates. The Journal of Biological Chemistry. 290 (38), 23361-23370 (2015).
- Wilkens, C., et al. Plant α-glucan phosphatases SEX4 and LSF2 display different affinity for amylopectin and amylose. FEBS Letters. 590 (1), 118-128 (2016).
- Atanasova, M., Bagdonas, H., Agirre, J. Structural glycobiology in the age of electron cryo-microscopy. Current Opinion in Structural Biology. 62, 70-78 (2020).
- Doyle, M. L. Characterization of binding interactions by isothermal titration calorimetry. Current Opinion in Biotechnology. 8 (1), 31-35 (1997).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados