È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Saggio di sedimentazione basato sulla concanavalina A per misurare il legame del substrato delle fosfatasi glucane
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo metodo descrive un saggio di sedimentazione in vitro basato sulla lectina per quantificare l'affinità di legame della fosfatasi glucana e dell'amilopectina. Questo saggio di co-sedimentazione è affidabile per misurare il legame del substrato della glucanofosfatasi e può essere applicato a vari substrati di glucano solubilizzati.
Abstract
Le fosfatasi glucane appartengono alla più ampia famiglia delle fosfatasi a doppia specificità (DSP) che defosforilate substrati di glucano, come il glicogeno negli animali e l'amido nelle piante. Le strutture cristalline della fosfatasi glucano con substrati di glucano modello rivelano distinte interfacce di legame glucano fatte di DSP e domini di legame dei carboidrati. Tuttavia, le misurazioni quantitative delle interazioni glucano-glucano-fosfatasi con substrati fisiologicamente rilevanti sono fondamentali per la comprensione biologica della famiglia di enzimi glucanofosfatasi e la regolazione del metabolismo energetico. Questo manoscritto riporta un saggio di sedimentazione in vitro basato sulla Concanavalina A (ConA) progettato per rilevare l'affinità di legame del substrato delle fosfatasi glucane contro diversi substrati glucani. Come prova del concetto, è stata determinata la costante di dissociazione (KD) della glucanofosfatasi Arabidopsis thaliana Starch Excess4 (SEX4) e dell'amilopectina. La caratterizzazione dei mutanti SEX4 e di altri membri della famiglia degli enzimi glucanofosfatasi dimostra ulteriormente l'utilità di questo test per valutare il legame differenziale delle interazioni proteina-carboidrati. Questi dati dimostrano l'idoneità di questo test a caratterizzare un'ampia gamma di proteine che interagiscono tra amido e glicogeno.
Introduzione
Le fosfatasi glucane sono membri di una sottofamiglia funzionalmente diversificata di fosfatasi a doppia specificità (DSP) all'interno dellasuperfamiglia 1 della proteina tirosina fosfatasi (PTP). Sono stati trovati nella maggior parte delle forme di vita, inclusi organismi fotosintetici ampiamente divergenti, esseri umani, vertebrati e alcuni invertebrati e protisti 2,3,4. Le piante contengono tre fosfatasi glucane note: Starch Excess4 (SEX4), Like Sex Four1 (LSF1) e Like Sex Four2 (LSF2)5,6,7. Le piante che mancano di fosfatasi glucano mostrano tassi ridotti di degradazione transitoria dell'amido e accumulo di amido nelle foglie 8,9. Laforin è il membro fondatore della famiglia delle fosfatasi glucane che defosforila il glicogeno nei vertebrati e nell'uomo 3,10. Le mutazioni della laforina provocano la malattia neurodegenerativa di Lafora, una forma autosomica recessiva fatale di epilessia11. Le fosfatasi glucane sono necessarie per il metabolismo del glicogeno e dell'amido e sono emerse come enzimi importanti per modulare il contenuto di amido nelle piante e trattare la malattia neurodegenerativa di Lafora12,13. Recenti studi di cristallografia a raggi X sulle fosfatasi glucane con substrati di glucano modello hanno fatto luce sul legame del substrato e sul meccanismo catalitico della defosforilazione del glucano14,15,16,17. Tuttavia, l'attuale comprensione di come le fosfatasi glucane si leghino ai loro substrati fisiologici è incompleta.
L'amido è un polimero insolubile di glucosio composto da 80% -90% amilopectina e 10% -20% amilosio18. I substrati per le fosfatasi glucane delle piante sono molecole di carboidrati fosforilati, come glicogeno e granuli di amido. I residui glucosilici fosforilati sono presenti con un rapporto fosfato/residuo glucosilico di 1:600. È interessante notare che i fosfati sono presenti solo sulle molecole di amilopectina19. La principale glucanofosfatasi SEX4 agisce sul granulo di amido per defosforilare le molecole di amilopectina. La struttura cristallina a raggi X di SEX4 combinata con studi di mutagenesi guidati dalla struttura ha dimostrato le specificità uniche del substrato di SEX4 per diverse posizioni all'interno di una struttura glucana15. Abbiamo recentemente dimostrato che l'attività biologicamente rilevante di SEX4 può essere osservata solo quando agisce sui suoi substrati solubilizzati di amilopectina20. Tuttavia, la comprensione delle interazioni glucano-SEX4 si è dimostrata difficile a causa della complessità strutturale del substrato, delle più ampie specificità di legame e delle basse affinità di legame tra la proteina e i suoi substrati. Questi problemi hanno ostacolato la capacità di utilizzare metodi comunemente usati nelle interazioni proteina-ligando, come la calorimetria isotermica di titolazione (ITC), la spettroscopia di risonanza magnetica nucleare (NMR) e i saggi basati su saggi basati su immunoassorbimento enzimatico (ELISA).
È interessante notare che gran parte della nostra comprensione delle interazioni carboidrati-proteine proviene dallo studio delle lectine. La concanavalina A (ConA) è una famiglia di lectine leguminose originariamente estratte dal fagiolo. ConA lega i carboidrati con elevata specificità, il che è vantaggioso per il suo uso in applicazioni di targeting e somministrazione di farmaci. Il legame di ConA ad una varietà di substrati contenenti α-D-mannosil e α-D-glucosil non riducenti è stato ampiamente studiato19,20. Le perle di selosorosio legate al ConA disponibili in commercio sono comunemente usate per purificare glicoproteine e glicolipidi21. ConA si lega a questi glucani attraverso i gruppi ossidrilici C3, C4 e C6 dei residui di glucosio. Le perle di ConA-Sepharose sono state utilizzate con successo anche per misurare il legame delle interazioni glicogeno-proteina e amido-proteina22,23. In questo studio, abbiamo utilizzato perle di ConA-Sepharose per sviluppare un test di legame per misurare le specificità di legame delle interazioni glucano-fosfatasi-amilopectina.
In precedenza, è stato utilizzato un saggio di sedimentazione basato su ConA per valutare la capacità di legame del substrato di glucanofosfatasi14,20,24. In questo studio, la stessa strategia è stata utilizzata per sviluppare un nuovo metodo per determinare l'affinità di legame delle interazioni glucano-glucano-fosfatasi e carboidrati. Questo metodo ha anche un vantaggio per studiare varie interazioni carboidrati-proteine solubilizzate.
Protocollo
1. Preparazione delle perle ConA-Sepharose
- Produrre 250 mL di un tampone legante contenente 67 mM HEPES (pH 7,5), 10 mM MgCl 2 e 0,2 mM CaCl2. Regolare il pH con una soluzione di NaOH 1 M.
- Pipettare 250 μL di sospensione di perline ConA-Sepharose in una provetta da microcentrifuga da 1,5 ml. Centrifugare il contenuto a 10.000 x g per 30 s a 4 °C. Scartare il surnatante.
NOTA: 250 μL di sfere di ConA-Sepharose in una provetta da microcentrifuga da 1,5 mL sono necessari per ogni concentrazione di amilopectina utilizzata per il test. - Aggiungere 750 μL del tampone legante a ciascun tubo contenente 250 μL di perle ConA-Sepharose. Centrifugare le provette a 10.000 x g per 1 minuto a 4 °C. Rimuovere il surnatante. Ripetere questo passaggio 2x per assicurarsi che le perline siano adeguatamente lavate ed equilibrate con il tampone di rilegatura.
2. Preparazione di soluzioni di amilopectina
- Preparare una soluzione madre di amilopectina di patate da 10 mg/ml. L'amilopectina è insolubile in acqua e può essere solubilizzata dal calore. Per solubilizzare, aggiungere 0,1 g di amilopectina di patate a 10 ml di acqua distillata. Riscaldare la sospensione a bagnomaria a 80 °C per 1 ora o fino a quando la soluzione non è più torbida.
- Lasciare che la soluzione ritorni a temperatura ambiente (RT), con vortice ripetuto per evitare l'aggregazione.
- Il trattamento alcol-alcalino è un metodo alternativo per solubilizzare i substrati di amilopectina. Per solubilizzare utilizzando questo metodo, attenersi alla seguente procedura.
- Sospendere 0,5 g di substrato di amilopectina in 5 ml di etanolo al 20% e 5 ml di 2 M NaOH. Mescolare vigorosamente il contenuto per 15-20 minuti a RT.
- Quindi, aggiungere 10 ml di acqua e regolare il pH della soluzione a 6,5 aggiungendo 2 M HCl. Portare il volume della soluzione risultante a 50 ml con acqua distillata per ottenere una soluzione di amilopectina da 10 mg / ml.
- Diluire la soluzione solubilizzata di amilopectina da 10 mg/mL per ottenere una serie di 2 mL di soluzioni diluite di amilopectina. Ad esempio, eseguire mezze diluizioni di 10 mg/ml per preparare una serie di concentrazioni di amilopectina (5 mg/ml, 2,5 mg/ml, 1,25 mg/ml, 0,625 mg/ml, 0,3125 mg/ml, 0,156 mg/ml, 0,078 mg/ml, 0,039 mg/ml, 0,019 mg/ml e 0 mg/ml).
3. Preparazione di ConA-Sepharose: perle di amilopectina
- Aggiungere 250 μL di ciascuna soluzione di amilopectina diluita in provette da microcentrifuga da 1,5 mL contenenti 250 μL di sfere di ConA-Sepharose pre-equilibrate in tampone legante. Mescolare bene il contenuto. Etichettare i tubi con la corrispondente concentrazione di amilopectina.
- Incubare il contenuto su una ruota rotante a 4 °C per 30 min.
NOTA: Non vi è alcun cambiamento nel complesso legato ConA-Sepharose:amilopectina nel tempo dopo 20 minuti. Il tempo di incubazione di 30 minuti è stato scelto variando i tempi di incubazione da 10 minuti a 1 ora per garantire il raggiungimento dell'equilibrio. - Centrifugare i tubi a 10.000 x g per 1 min. Raccogliere il surnatante in una provetta da microcentrifuga da 1,5 ml appena etichettata. Conservare queste frazioni surnatanti per eseguire il saggio D-glucosio12 (idrolisi acida dell'amilopectina seguita da determinazione UV del glucosio tramite saggio enzimatico). Questo passaggio è necessario per garantire che tutta l'amilopectina sia legata alle perline.
- Aggiungere 750 μL di tampone legante alle perle ConA-Sepharose:amilopectina. Centrifugare i tubi a 10.000 x g per 1 min. Scartare il surnatante per rimuovere eventuali molecole di amilopectina non legate.
- Ripetere il passaggio 3.4 per garantire un lavaggio sufficiente. Ogni tubo contiene ora perle di ConA-Sepharose legate a quantità variabili di substrati di amilopectina.
4. Incubazione di SEX4 con ConA-Sepharose: perle di amilopectina
- Mescolare 250 μL di conA-sefrosio:amilopectina con 100 μL del tampone legante che include 10 μg di proteina SEX4, 10 mM di ditiotreitolo (DTT) e 10 μM di cocktail inibitore della proteasi (PIC). Si noti che il volume totale in ogni tubo è di 350 μL.
NOTA: Un cocktail inibitore della proteasi viene aggiunto come misura precauzionale per evitare qualsiasi degradazione non necessaria di SEX4. Questo è un passaggio facoltativo. In questo test viene utilizzata la proteina ricombinante Arabidopsis thaliana SEX4 (AtSEX4). La proteina purificata contiene un tag di istidina N-terminale necessario per rilevare la proteina tramite chemiluminescenza. Informazioni dettagliate sulla purificazione della fosfatasi glucana sono descritte nelle pubblicazioni precedenti14,20,24. - Incubare la proteina e la sospensione di conA-salsorosio:amilopectina a 4 °C per 45 minuti con una leggera rotazione.
NOTA: Il tempo di incubazione di 45 minuti viene scelto per garantire il raggiungimento dell'equilibrio per il complesso. - Centrifugare i tubi a 10.000 x g per 1 min. Pipettare attentamente 50 μL del surnatante utilizzando una punta di carico del gel in una nuova provetta da microcentrifuga da 1,5 ml. Aggiungere 20 μL di 4x colorante SDS-PAGE e 10 μL di acqua a ciascun tubo contenente 50 μL delle frazioni surnatanti raccolte. Riscaldare i campioni a 95 °C per 10 minuti. Salvare questi campioni per eseguire i gel SDS-PAGE. Assicurarsi che 10 nuovi tubi etichettati come "surnatante (S)" abbiano le corrispondenti concentrazioni di substrato.
- Aggiungere 750 μL del tampone legante alle perle ConA-Sepharose:amilopectina: SEX4 per rimuovere qualsiasi proteina non legata dalle perle. Centrifugare i tubi a 10.000 x g per 1 min. Ripetere questo passaggio ancora una volta per garantire un corretto lavaggio. Scartare il surnatante.
- Aggiungere 20 μL di 4x colorante SDS-PAGE e 80 μL di acqua distillata nei tubi contenenti conA-Sepharose:amilopectina:SEX4 perline lavate. Riscaldare i campioni a 95 °C per 10 minuti e centrifugare a 10.000 x g per 1 minuto.
- Scartare il pellet e conservare il surnatante per l'esecuzione dei gel SDS-PAGE. Pipettare 80 μL del surnatante in nuove provette ed etichettarle come "pellet (P)".
5. Esecuzione di gel SDS-PAGE
- Caricare 40 μL dei campioni proteici non legati (realizzati nella fase 2.3, etichettata S) in pozzetti di gel di poliacrilammide prefabbricati al 4%-12% dalla concentrazione di substrato più bassa alla più alta, ma mantenere la prima corsia libera per caricare il marcatore di peso molecolare della proteina. Utilizzare un secondo gel per caricare 10 campioni di proteine legate realizzati al punto 2.5 (etichettati come P).
- Aggiungere 1x buffer di funzionamento SDS-PAGE appena preparato ad entrambe le camere dell'apparecchio. Eseguire il gel a 150 V per 35 minuti o fino a quando la parte anteriore del colorante raggiunge il fondo del gel.
- Rimuovere il gel di corsa dall'apparecchio e rimuovere i distanziatori e le lastre di vetro. Utilizzare il gel separato per eseguire un'analisi western blot.
6. Western blotting per il rilevamento di chemiluminescenza14,15
NOTA: Questo metodo può essere facilmente modificato / adattato a seconda delle apparecchiature di blotting occidentale che gli utenti hanno nei loro laboratori.
- Produrre 1 L di tampone di trasferimento contenente 5,8 g di Tris base, 2,9 g di glicina, 0,37 g di SDS e 200 mL di metanolo.
- Trasferire le proteine separate dalle dimensioni dal gel di poliacrilammide a una membrana di nitrocellulosa. Assemblare brevemente le spugne, la carta da filtro, il gel e la membrana di nitrocellulosa secondo il protocollo di trasferimento occidentale14,15. Funziona a 70 V per 1 ora.
- Per evitare il legame proteico non specifico, incubare la membrana nitrocellulosa contenente la soluzione proteica di albumina sierica bovina all'1%-5% (BSA) o proteine del latte in 50 ml di tampone TBST (20 mM Tris [pH 7,5], 150 mM NaCl, 0,1% Tween 20) per 1 ora. Lavare la membrana 3x utilizzando tampone TBST per rimuovere qualsiasi soluzione di blocco non legata.
- Incubare la membrana con un anticorpo legato alla perossidasi di rafano (HRP) specifico per la proteina His-tagged per 1 ora. Lavare la membrana 3x in tampone TBST per rimuovere eventuali anticorpi non legati. Utilizzare una diluizione 1:2.000 di anticorpi contro TBST per una riproducibilità e una sensibilità ottimali.
- L'anticorpo legato all'enzima HRP si lega specificamente al tag istidina della proteina SEX4, che produce una banda in presenza di reagenti chemiluminescenza. Per l'imaging digitale, creare una soluzione di parti uguali di soluzioni di substrato chemiluminescente (750 μL ciascuna) in un tubo da 1,5 ml. Incubare la membrana per almeno 5 minuti nella soluzione.
- Posizionare la proteina di membrana rivolta verso il basso sullo scanner blot ed eseguire il software di acquisizione per quantificare la proteina sia nella frazione pellet che in quella surnatante
7. Analisi dei dati
- Eseguire le misurazioni quantitative del segnale utilizzando il software di acquisizione con lo scanner blot. Normalizzare tutte le misurazioni quantitative nelle frazioni di surnatante e pellet al totale delle proteine caricate.
NOTA: Il software permette di quantificare l'intensità di ogni fascia proteica nelle frazioni surnatante e pellet. - Nell'esperimento di legame saturante, tracciare la percentuale di concentrazione di amilopectina legata alle proteine. Adatta i dati a Y = Bmax x X / (K D + X), utilizzando il software di analisi dei dati per calcolare KD.
NOTA: Bmax è il massimo legame specifico, l'asse Y è la percentuale di proteine legate, l'asse X e l'asse X è la concentrazione di amilopectina.
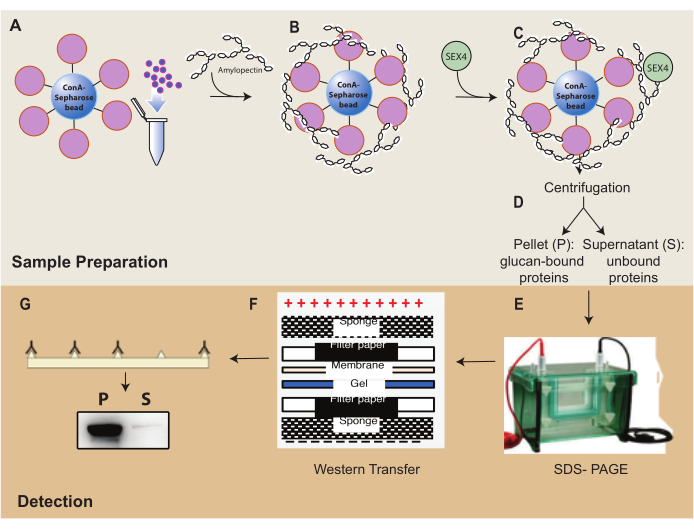
Figura 1: Panoramica del flusso di lavoro del saggio di sedimentazione ConA-Sepharose. (A) Preparazione delle perle di ConA-Sepharose. (B) Incubazione con substrato di amilopectina. (C) Incubazione con proteina SEX4. (D) Separazione delle frazioni proteiche legate e non legate mediante centrifugazione. (E) Separazione delle proteine tramite SDS-PAGE. (F) Analisi western-blot. (G) Rilevazione chemiluminescenziale della proteina SEX4 marcata con His. Fare clic qui per visualizzare una versione ingrandita di questa figura.
Risultati
Una delle caratteristiche chiave della famiglia di proteine della glucanofosfatasi è la loro capacità di legarsi ai substrati del glucano. In primo luogo, la capacità di legame di SEX4 a ConA-Sepharose:amylopectin beads è stata analizzata utilizzando SDS-PAGE (Figura 2A). L'albumina sierica bovina (BSA) è servita come controllo negativo per rilevare qualsiasi legame non specifico delle proteine alle perle ConA-Sepharose:amilopectina. L'analisi SDS-PAGE delle proteine ha mostrato la pres...
Discussione
Questo studio dimostra il successo dello sviluppo di un nuovo saggio di sedimentazione in vitro che consente di determinare l'affinità di legame delle interazioni glucano-glucano-fosfatasi. Il progetto del saggio sfrutta il legame specifico della lectina ConA ai glucani attraverso i residui idrossilici del glucosio per catturare indirettamente substrati di carboidrati solubilizzati su perle di salsorosio. Ciò consente la separazione delle frazioni proteiche legate e non legate tramite centrif...
Divulgazioni
Gli autori non dichiarano conflitti di interesse.
Riconoscimenti
Questo studio è stato supportato dal premio della National Science Foundation MCB-2012074. Gli autori ringraziano il Dr. Craig W. Vander Kooi del Dipartimento di Biochimica e Biologia Molecolare dell'Università della Florida per le preziose discussioni e supporto. Gli autori ringraziano anche il Dr. Matthew S. Gentry del Dipartimento di Biochimica e Biologia Molecolare dell'Università della Florida per il suo supporto. Vorremmo ringraziare la dottoressa Sara Lagalwar, presidente del programma di neuroscienze dello Skidmore College, per averci permesso di utilizzare lo scanner LICOR C-digit blot per l'imaging western blot.
Materiali
| Name | Company | Catalog Number | Comments |
| 6x-His Tag monoclonal antibody (HIS.H8), HRP | Therm Fisher Scientific | MA1-21315-HRP | |
| Biorad gel electrophoresis and Western blot kit | Biorad | 1703930 | |
| Calcium chloride | Sigma-Aldrich | 208291 | |
| C-Digit blot scanner | LICOR | 3600-00 | Blot scanner |
| Complete protease inhibitor cocktail | Sigma-Aldrich | 11836170001 | |
| Concanavalin A-sepharose beads | Sigma-Aldrich | C9017 | This product contains in 0.1 M acetate buffer, pH 6, containing 1 M NaCl, 1 mM CaCl2, 1 mM MnCl2, and 1 mM MgCl2 in 20% ethanol |
| Centrifuge | Eppendorf | 5425R | |
| Glycine | Fisher Scientific | BP381-5 | |
| GraphPad Prism 8.0 software | GraphPad | Version 8.0 | Data analysis software |
| HEPES | Sigma-Aldrich | H8651 | |
| Image Studio | LICOR | 3600-501 | Acquisition Software |
| Magnesium chloride | Sigma-Aldrich | M2670 | |
| Methanol | Fisher Scientific | A452SK-4 | |
| Sodium dodecyl sulfate | Fisher Scientific | PI28312 | |
| Potato amylopectin | Sigma-Aldrich | A8515 | |
| Precast SDSPAGE Gels | Genscript | M00653S | |
| Tris base | Fisher Scientific | BP154-1 | |
| Tween 20 | Fisher Scientific | MP1TWEEN201 | |
| Westernsure premium chemiluminescence substrate | LI-COR | 926-95000 |
Riferimenti
- Meekins, D. A., Vander Kooi, C. W., Gentry, M. S. Structural mechanisms of plant glucan phosphatases in starch metabolism. The FEBS Journal. 283 (13), 2427-2447 (2016).
- Gentry, M. S., et al. The phosphatase laforin crosses evolutionary boundaries and links carbohydrate metabolism to neuronal disease. The Journal of Cell Biology. 178 (3), 477-488 (2007).
- Worby, C. A., Gentry, M. S., Dixon, J. E. Laforin, a dual specificity phosphatase that dephosphorylates complex carbohydrates. The Journal of Biological Chemistry. 281 (41), 30412-30418 (2006).
- Gentry, M. S., Pace, R. M. Conservation of the glucan phosphatase laforin is linked to rates of molecular evolution and the glucan metabolism of the organism. BMC Evolutionary Biology. 9, 138 (2009).
- Niittyla, T., et al. Similar protein phosphatases control starch metabolism in plants and glycogen metabolism in mammals. The Journal of Biological Chemistry. 281 (17), 11815-11818 (2006).
- Kotting, O., et al. STARCH-EXCESS4 is a laforin-like Phosphoglucan phosphatase required for starch degradation in Arabidopsis thaliana. The Plant Cell. 21 (1), 334-346 (2009).
- Comparot-Moss, S., et al. A putative phosphatase, LSF1, is required for normal starch turnover in Arabidopsis leaves. Plant Physiology. 152 (2), 685-697 (2010).
- Zeeman, S. C., Northrop, F., Smith, A. M., Rees, T. A starch-accumulating mutant of Arabidopsis thaliana deficient in a chloroplastic starch-hydrolysing enzyme. The Plant Journal: For Cell and Molecular Biology. 15 (3), 357-365 (1998).
- Kotting, O., et al. Identification of a novel enzyme required for starch metabolism in Arabidopsis leaves. The phosphoglucan, water dikinase. Plant Physiology. 137 (1), 242-252 (2005).
- Tagliabracci, V. S., et al. Laforin is a glycogen phosphatase, deficiency of which leads to elevated phosphorylation of glycogen in vivo. Proceedings of the National Academy of Sciences. 104 (49), 19262-19266 (2007).
- Gentry, M. S., Guinovart, J. J., Minassian, B. A., Roach, P. J., Serratosa, J. M. Lafora disease offers a unique window into neuronal glycogen metabolism. The Journal of Biological Chemistry. 293 (19), 7117-7125 (2018).
- Brewer, M. K., et al. Targeting pathogenic lafora bodies in lafora disease using an antibody-enzyme fusion. Cell Metabolism. 30 (4), 689-705 (2019).
- Santelia, D., Zeeman, S. C. Progress in Arabidopsis starch research and potential biotechnological applications. Current Opinion in Biotechnology. 22 (2), 271-280 (2011).
- Raththagala, M., et al. Structural mechanism of laforin function in glycogen dephosphorylation and lafora disease. Molecular Cell. 57 (2), 261-272 (2015).
- Meekins, D. A., et al. Phosphoglucan-bound structure of starch phosphatase Starch Excess4 reveals the mechanism for C6 specificity. Proceedings of the National Academy of Sciences. 111 (20), 7272-7277 (2014).
- Vander Kooi, C. W., et al. Structural basis for the glucan phosphatase activity of Starch Excess4. Proceedings of the National Academy of Sciences. 107 (35), 15379-15384 (2010).
- Meekins, D. A., et al. Structure of the Arabidopsis glucan phosphatase like sex four2 reveals a unique mechanism for starch dephosphorylation. The Plant Cell. 25 (6), 2302-2314 (2013).
- Smith, A. M., Zeeman, S. C. Starch: A flexible, adaptable carbon store coupled to plant growth. Annual Review of Plant Biology. 71, 217-245 (2020).
- Jane, J., Kasemuwan, T., Chen, J. F., Juliano, B. O. Phosphorus in rice and other starches. Cereal Foods World. 41 (11), 827-832 (1996).
- Mak, C. A., et al. Cooperative kinetics of the glucan phosphatase starch excess4. Biochemistry. 60 (31), 2425-2435 (2021).
- Campbell, K. P., MacLennan, D. H. Purification and characterization of the 53,000-dalton glycoprotein from the sarcoplasmic reticulum. The Journal of Biological Chemistry. 256 (9), 4626-4632 (1981).
- Campbell, K. P., MacLennan, D. H., Jorgensen, A. O., Mintzer, M. C. Purification and characterization of calsequestrin from canine cardiac sarcoplasmic reticulum and identification of the 53,000 dalton glycoprotein. The Journal of Biological Chemistry. 258 (2), 1197-1204 (1983).
- Davey, M. W., Sulkowski, E., Carter, W. A. Binding of human fibroblast interferon to concanavalin A-agarose. Involvement of carbohydrate recognition and hydrophobic interaction. Biochemistry. 15 (3), 704-713 (1976).
- Meekins, D. A., et al. Mechanistic insights into glucan phosphatase activity against polyglucan substrates. The Journal of Biological Chemistry. 290 (38), 23361-23370 (2015).
- Wilkens, C., et al. Plant α-glucan phosphatases SEX4 and LSF2 display different affinity for amylopectin and amylose. FEBS Letters. 590 (1), 118-128 (2016).
- Atanasova, M., Bagdonas, H., Agirre, J. Structural glycobiology in the age of electron cryo-microscopy. Current Opinion in Structural Biology. 62, 70-78 (2020).
- Doyle, M. L. Characterization of binding interactions by isothermal titration calorimetry. Current Opinion in Biotechnology. 8 (1), 31-35 (1997).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon