Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Glukan Fosfatazların Substrat Bağlanmasını Ölçmek için Konkanavalin A Bazlı Sedimantasyon Testi
* Bu yazarlar eşit katkıda bulunmuştur
Bu Makalede
Özet
Bu yöntem, glukan fosfataz ve amilopektinin bağlanma afinitesini ölçmek için lektin bazlı in vitro sedimantasyon testini tanımlar. Bu ko-sedimantasyon testi, glukan fosfataz substrat bağlanmasını ölçmek için güvenilirdir ve çeşitli çözünür glukan substratlarına uygulanabilir.
Özet
Glukan fosfatazlar, hayvanlarda glikojen ve bitkilerde nişasta gibi glukan substratlarını defosforile eden daha büyük çift özgüllük fosfataz (DSP) ailesine aittir. Glukan fosfatazın model glukan substratları ile kristal yapıları, DSP ve karbonhidrat bağlama alanlarından yapılmış farklı glukan bağlayıcı arayüzler ortaya koymaktadır. Bununla birlikte, fizyolojik olarak ilgili substratlarla glukan-glukan fosfataz etkileşimlerinin kantitatif ölçümleri, glukan fosfataz enzim ailesinin biyolojik olarak anlaşılması ve enerji metabolizmasının düzenlenmesi için temeldir. Bu makalede, glukan fosfatazların farklı glukan substratlarına karşı substrat bağlanma afinitesini tespit etmek için tasarlanmış bir Concanavalin A (ConA) bazlı in vitro sedimantasyon testi bildirilmiştir. Kavramın bir kanıtı olarak, glukan fosfataz Arabidopsis thaliana Nişasta Fazlalığı4 (SEX4) ve amilopektinin ayrışma sabiti (KD) belirlendi. SEX4 mutantlarının ve glukan fosfataz enzim ailesinin diğer üyelerinin karakterizasyonu, protein-karbonhidrat etkileşimlerinin diferansiyel bağlanmasını değerlendirmek için bu tahlilin yararlılığını göstermektedir. Bu veriler, bu tahlilin çok çeşitli nişasta ve glikojen etkileşen proteinleri karakterize etmeye uygunluğunu göstermektedir.
Giriş
Glukan fosfatazlar, protein tirozin fosfataz (PTP) üst ailesi1 içindeki çift özgüllüklü fosfatazların (DSP'ler) fonksiyonel olarak farklı bir alt ailesinin üyeleridir. Geniş ölçüde farklı fotosentetik organizmalar, insanlar, omurgalılar ve bazı omurgasızlar ve protistler de dahil olmak üzere çoğu yaşam formunda bulunmuştur 2,3,4. Bitkiler bilinen üç glukan fosfataz içerir: Nişasta Fazlalığı4 (SEX4), Seks Dörtlüsü 1 (LSF1) ve Seks Dörtlüsü (LSF2) Gibi5,6,7. Glukan fosfatazları olmayan bitkiler, geçici nişasta bozunma ve yapraklarda nişasta birikimi oranlarının azaldığını göstermektedir 8,9. Laforin, omurgalılarda ve insanlarda glikojen defosforilleri defosforile eden glukan fosfataz ailesinin kurucu üyesidir 3,10. Laforinin mutasyonları, epilepsinin ölümcül otozomal resesif bir formu olan nörodejeneratif Lafora hastalığı ile sonuçlanır11. Glukan fosfatazlar glikojen ve nişasta metabolizması için gereklidir ve bitkilerde nişasta içeriğini modüle etmek ve nörodejeneratif Lafora hastalığının tedavisinde önemli enzimler olarak ortaya çıkmıştır12,13. Model glukan substratlı glukan fosfatazlar üzerine yapılan son X-ışını kristalografi çalışmaları, substrat bağlanmasına ve glukan defosforilasyonunun katalitik mekanizmasına ışık tutmuştur14,15,16,17. Bununla birlikte, glukan fosfatazların fizyolojik substratlarına nasıl bağlandığına dair mevcut anlayış eksiktir.
Nişasta,% 80-90 amilopektin ve% 10-20 amilozdan yapılmış çözünmez bir glikoz polimeridir18. Bitki glukan fosfatazları için substratlar, glikojen ve nişasta granülleri gibi fosforile karbonhidrat molekülleridir. Fosforile glukozil kalıntıları 1:600 fosfat:glukozil kalıntı oranında bulunur. İlginç bir şekilde, fosfatlar sadece amilopektin molekülleri19'da bulunur. Ana bitki glukan fosfataz SEX4, amilopektin moleküllerini defosforile etmek için nişasta granülü üzerinde hareket eder. SEX4'ün X-ışını kristal yapısı, yapı kılavuzlu mutajenez çalışmaları ile birleştiğinde, bir glukan yapısı15 içindeki farklı pozisyonlar için SEX4'ün benzersiz substrat özelliklerini göstermiştir. Son zamanlarda, SEX4'ün biyolojik olarak ilgili aktivitesinin sadece çözünür amilopektin substratları20 üzerinde hareket ederken gözlemlenebileceğini gösterdik. Bununla birlikte, glukan-SEX4 etkileşimlerini anlamanın, substratın yapısal karmaşıklığı, daha geniş bağlanma özgüllükleri ve protein ile substratları arasındaki düşük bağlanma afiniteleri nedeniyle zor olduğu kanıtlanmıştır. Bu sorunlar, izotermal titrasyon kalorimetrisi (ITC), nükleer manyetik rezonans (NMR) spektroskopisi ve enzime bağlı immünosorbent testi (ELISA) tabanlı testler gibi protein-ligand etkileşimlerinde yaygın olarak kullanılan yöntemleri kullanma yeteneğini engellemiştir.
İlginçtir ki, karbonhidrat-protein etkileşimleri hakkındaki anlayışımızın çoğu, lektinleri incelemekten gelmiştir. Concanavalin A (ConA), orijinal olarak jack fasulyesinden ekstrakte edilen bir baklagil lektin protein ailesidir. ConA, karbonhidratları yüksek özgüllükle bağlar, bu da ilaç hedefleme ve dağıtım uygulamalarında kullanımı için avantajlıdır. ConA'nın indirgeyici olmayan α-D-mannosil ve α-D-glukozil içeren çeşitli substratlara bağlanması kapsamlı bir şekilde incelenmiştir19,20. Ticari olarak temin edilebilen ConA'ya bağlı Sefaroz boncukları, glikoproteinleri ve glikolipidleri saflaştırmak için yaygın olarak kullanılır21. ConA, glikoz kalıntılarının C3, C4 ve C6 hidroksil grupları aracılığıyla bu glukanlara bağlanır. ConA-Sefaroz boncukları ayrıca glikojen-protein ve nişasta-protein etkileşimlerinin bağlanmasını ölçmek için başarıyla kullanılmıştır22,23. Bu çalışmada, glukan fosfataz-amilopektin etkileşimlerinin bağlanma özelliklerini ölçmek için bağlayıcı bir tahlil geliştirmek için ConA-Sefaroz boncuklarını kullandık.
Daha önce, glukan fosfataz substrat bağlanma kabiliyetini 14,20,24 değerlendirmek için ConA bazlı bir sedimantasyon testi kullanılmıştır. Bu çalışmada, aynı strateji, glukan-glukan fosfataz ve karbonhidrat etkileşimlerinin bağlanma afinitesini belirlemek için yeni bir yöntem geliştirmek için kullanılmıştır. Bu yöntem aynı zamanda çeşitli çözünür karbonhidrat-protein etkileşimlerini araştırmak için bir avantaja sahiptir.
Protokol
1. ConA-Sefaroz boncuklarının hazırlanması
- 67 mM HEPES (pH 7,5), 10 mM MgCl 2 ve0,2 mM CaCl2 içeren 250 mL'lik bir bağlayıcı tampon yapın. 1 M NaOH çözeltisi kullanarak pH'ı ayarlayın.
- Pipet, 250 μL ConA-Sefaroz boncuk süspansiyonunu 1,5 mL'lik bir mikrosantrifüj tüpüne yerleştirin. İçeriği 10.000 x g'de 4 °C'de 30 sn santrifüj edin. Supernatan'ı atın.
NOT: Tahlil için kullanılan her amilopektin konsantrasyonu için 1,5 mL'lik bir mikrosantrifüj tüpünde 250 μL ConA-Sefaroz boncuk gereklidir. - 250 μL ConA-Sefaroz boncuk içeren her tüpe 750 μL bağlayıcı tampon ekleyin. Tüpleri 10.000 x g'de 4 °C'de 1 dakika santrifüj edin. Supernatan'ı çıkarın. Boncukların uygun şekilde yıkandığından ve bağlama tamponu ile dengelendiğinden emin olmak için bu adımı 2 kez tekrarlayın.
2. Amilopektin çözeltilerinin hazırlanması
- 10 mg / mL patates amilopektin stok çözeltisi yapın. Amilopektin suda çözünmez ve ısı ile çözünür. Çözündürmek için, 10 mL damıtılmış suya 0.1 g patates amilopektin ekleyin. Süspansiyonu 80 ° C'de bir su banyosunda 1 saat boyunca veya çözelti artık bulanık kalmayıncaya kadar ısıtın.
- Topaklanmayı önlemek için çözeltinin tekrarlanan vorteksleme ile oda sıcaklığına (RT) geri dönmesine izin verin.
- Alkol-alkali tedavi, amilopektin substratlarını çözündürmek için alternatif bir yöntemdir. Bu yöntemi kullanarak çözmek için aşağıdaki adımları izleyin.
- 0.5 g amilopektin substratını 5 mL% 20 etanol ve 5 mL 2 M NaOH içinde askıya alın. İçeriği RT'de 15-20 dakika boyunca kuvvetlice karıştırın.
- Daha sonra, 10 mL su ekleyin ve 2 M HCl ekleyerek çözeltinin pH'ını 6.5'e ayarlayın. elde edilen çözeltinin hacmini, 10 mg / mL amilopektin çözeltisi yapmak için damıtılmış su ile 50 mL'ye getirin.
- 2 mL seyreltilmiş amilopektin çözeltisi serisi oluşturmak için 10 mg / mL çözünür amilopektin çözeltisini seyreltin. Örneğin, bir dizi amilopektin konsantrasyonu hazırlamak için 10 mg / mL'lik yarım seyreltme gerçekleştirin (5 mg / mL, 2.5 mg / mL, 1.25 mg / mL, 0.625 mg / mL, 0.3125 mg / mL, 0.156 mg / mL, 0.078 mg / mL, 0.039 mg / mL, 0.019 mg / mL ve 0 mg / mL).
3. ConA-Sefarozun hazırlanması: amilopektin boncukları
- Bağlama tamponunda önceden dengelenmiş 250 μL ConA-Sefaroz boncukları içeren 1,5 mL mikrosantrifüj tüplerine her seyreltilmiş amilopektin çözeltisinden 250 μL ekleyin. İçeriği iyice karıştırın. Tüpleri karşılık gelen amilopektin konsantrasyonu ile etiketleyin.
- İçeriği dönen bir tekerlek üzerinde 4 ° C'de 30 dakika boyunca inkübe edin.
NOT: ConA-Sepharose:amilopectin bağlı kompleksinde 20 dakika sonra zamanla herhangi bir değişiklik olmaz. 30 dakikalık kuluçka süresi, dengeye ulaşılmasını sağlamak için inkübasyon süreleri 10 dakika ile 1 saat arasında değiştirilerek seçildi. - Tüpleri 1 dakika boyunca 10.000 x g'de santrifüjleyin. Süpernatantı yeni etiketlenmiş 1,5 mL mikrosantrifüj tüpünde toplayın. D-glukoz testi12'yi gerçekleştirmek için bu süpernatant fraksiyonları kaydedin (amilopektinin asit hidrolizi, ardından enzimatik tahlil yoluyla glikozun UV tayini). Bu adım, tüm amilopektinin boncuklara bağlanmasını sağlamak için gereklidir.
- ConA-Sepharose:amilopectin boncuklarına 750 μL bağlama tamponu ekleyin. Tüpleri 1 dakika boyunca 10.000 x g'de santrifüjleyin. Bağlanmamış amilopektin moleküllerini çıkarmak için süpernatantı atın.
- Yeterli yıkama olduğundan emin olmak için adım 3.4'ü tekrarlayın. Her tüp şimdi değişen miktarlarda amilopektin substratlarına bağlı ConA-Sefaroz boncukları içerir.
4. SEX4'ün ConA-Sefaroz ile inkübe edilmesi: amilopektin boncukları
- 250 μL ConA-Sefaroz: amilopektin boncukları, 10 μg SEX4 proteini, 10 mM ditiyotreitol (DTT) ve 10 μM proteaz inhibitörü kokteyli (PIC) içeren 100 μL bağlayıcı tampon ile karıştırın. Her tüpteki toplam hacmin 350 μL olduğunu unutmayın.
NOT: Gereksiz SEX4 bozulmasını önlemek için ihtiyati bir adım olarak bir proteaz inhibitörü kokteyli eklenmiştir. Bu isteğe bağlı bir adımdır. Bu tahlilde rekombinant protein Arabidopsis thaliana SEX4 (AtSEX4) kullanılır. Saflaştırılmış protein, kemilüminesans yoluyla proteini tespit etmek için gerekli olan bir N-terminal histidin etiketi içerir. Glukan fosfataz saflaştırmaları hakkında ayrıntılı bilgi önceki yayınlarda açıklanmıştır14,20,24. - Proteini ve ConA-Sepharose'u inkübe edin:amilopektin boncuk süspansiyonunu 4 ° C'de 45 dakika boyunca nazik bir rotasyonla inkübe edin.
NOT: 45 dakikalık inkübasyon süresi, kompleks için dengeye ulaşılmasını sağlamak için seçilmiştir. - Tüpleri 1 dakika boyunca 10.000 x g'de santrifüjleyin. Yeni bir 1,5 mL mikrosantrifüj tüpüne bir jel yükleme ucu kullanarak süpernatantın 50 μL'lik pipetini dikkatlice alın. Toplanan süpernatant fraksiyonların 50 μL'sini içeren her tüpe 20 μL 4x SDS-PAGE boya ve 10 μL su ekleyin. Numuneleri 95 °C'de 10 dakika ısıtın. SDS-PAGE jellerini çalıştırmak için bu örnekleri kaydedin. "Süpernatant (S)" etiketli 10 yeni tüpün karşılık gelen substrat konsantrasyonlarına sahip olduğundan emin olun.
- Boncuklardan bağlanmamış proteini çıkarmak için ConA-Sepharose:amilopektin: SEX4 boncuklarına 750 μL bağlayıcı tampon ekleyin. Tüpleri 1 dakika boyunca 10.000 x g'de santrifüjleyin. Doğru yıkamayı sağlamak için bu adımı bir kez daha tekrarlayın. Supernatan'ı atın.
- Yıkanmış ConA-Sepharose:amilopektin:SEX4 boncukları içeren tüplere 20 μL 4x SDS-PAGE boya ve 80 μL damıtılmış su ekleyin. Numuneleri 95 °C'de 10 dakika ısıtın ve 10.000 x g'de 1 dakika santrifüj yapın.
- Pelet atın ve süpernatantı SDS-PAGE jellerini çalıştırmak için saklayın. Süpernatantın 80 μL'sini yeni tüplere pipet edin ve bunları "pelet (P)" olarak etiketleyin.
5. SDS-PAGE jellerinin çalıştırılması
- Bağlanmamış protein numunelerinin 40 μL'sini (adım 2.3'te yapılmış, S etiketli) en düşük substrat konsantrasyonundan en yükseğe kadar %4-%12 prekast poliakrilamid jel kuyucuklarına yükleyin, ancak protein moleküler ağırlık belirtecini yüklemek için ilk şeridi serbest bırakın. Adım 2.5'te (P olarak etiketlenmiş) yapılan 10 bağlı protein örneğini yüklemek için ikinci bir jel kullanın.
- Cihazın her iki haznesine taze hazırlanmış 1x SDS-PAGE çalışma tamponu ekleyin. Jeli 150 V'ta 35 dakika boyunca veya boya cephesi jelin dibine ulaşana kadar çalıştırın.
- Çalıştırma jelini aparattan çıkarın ve ara parçaları ve cam plakaları çıkarın. Batı lekesi analizi yapmak için ayrılmış jeli kullanın.
6. Kemilüminesans tespiti için Batı lekelenmesi14,15
NOT: Bu yöntem, kullanıcıların laboratuvarlarında sahip oldukları batı blotlama ekipmanına bağlı olarak kolayca değiştirilebilir/uyarlanabilir.
- 5.8 g Tris baz, 2.9 g glisin, 0.37 g SDS ve 200 mL metanol içeren 1 L transfer tamponu yapın.
- Boyut olarak ayrılmış proteinleri poliakrilamid jelden bir nitroselüloz membrana aktarın. Süngerleri, filtre kağıtlarını, jeli ve nitroselüloz membranını batı transfer protokolü14,15'e göre kısaca monte edin. 1 saat boyunca 70 V'ta çalıştırın.
- Spesifik olmayan protein bağlanmasını önlemek için,% 1-5 sığır serum albümini (BSA) protein çözeltisi veya süt proteini içeren nitroselüloz membranını 50 mL TBST tamponunda (20 mM Tris [pH 7.5], 150 mM NaCl,% 0.1 Tween 20) 1 saat boyunca inkübe edin. Herhangi bir bağlanmamış blokaj solüsyonunu çıkarmak için membranı TBST tamponu kullanarak 3x yıkayın.
- Zarı, 1 saat boyunca His-etiketli proteine özgü yaban turpu peroksidaz (HRP) bağlantılı bir antikor ile inkübe edin. Bağlanmamış antikorları çıkarmak için membranı TBST tamponunda 3 kat yıkayın. Optimal tekrarlanabilirlik ve duyarlılık için TBST'ye karşı 1:2.000 antikor seyreltme kullanın.
- HRP enzimine bağlı antikor, özellikle kemilüminesans reaktiflerinin varlığında bir bant veren SEX4 proteininin histidin etiketine bağlanır. Dijital görüntüleme için, 1,5 mL'lik bir tüp içinde eşit miktarda kemilüminesan substrat çözeltisi (her biri 750 μL) çözeltisi yapın. Membranı çözelti içinde en az 5 dakika inkübe edin.
- Membran proteinini leke tarayıcısının yan tarafına yerleştirin ve hem pelet hem de süpernatant fraksiyonlarındaki proteini ölçmek için edinme yazılımını çalıştırın.
7. Veri analizi
- Leke tarayıcı ile edinme yazılımını kullanarak nicel sinyal ölçümlerini gerçekleştirin. Süpernatant ve pelet fraksiyonlarındaki tüm kantitatif ölçümleri yüklenen toplam proteine normalleştirin.
NOT: Yazılım, süpernatant ve pelet fraksiyonlarındaki her protein bandının yoğunluğunu ölçmeye izin verir. - Doygunluk bağlama deneyinde, proteine bağlı ve amilopektin konsantrasyonunun yüzdesini çizin. K D'yi hesaplamak için veri analiz yazılımını kullanarak verileri Y = Bmax x X / (KD + X) olarak sığdırın.
NOT: Bmax maksimum spesifik bağlanmadır, Y ekseni proteine bağlı yüzdesidir, X ekseni amilopektin konsantrasyonudur.
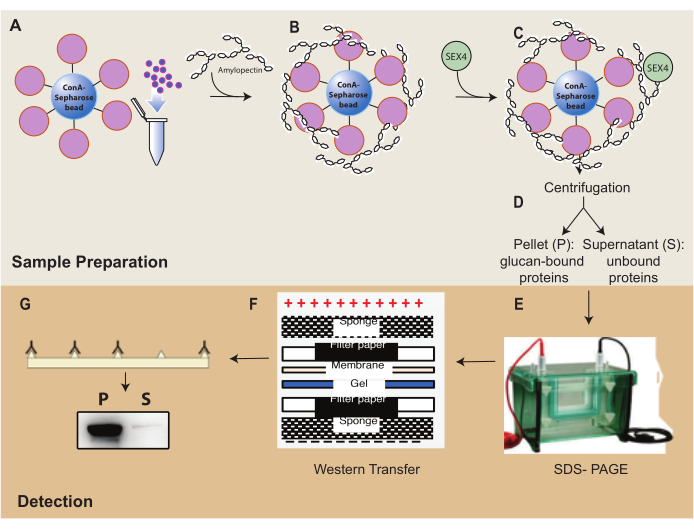
Şekil 1: ConA-Sefaroz sedimantasyon testi iş akışına genel bakış. (A) ConA-Sefaroz boncuklarının hazırlanması. (B) Amilopektin substratı ile inkübasyon. (C) SEX4 proteini ile inkübasyon. (D) Bağlı ve bağlanmamış protein fraksiyonlarının santrifüjleme yoluyla ayrılması. (E) SDS-PAGE aracılığıyla proteinin ayrılması. (F) Batı lekesi analizi. (G) His etiketli SEX4 proteininin kemilüminesans tespiti. Bu şeklin daha büyük bir versiyonunu görmek için lütfen buraya tıklayın.
Sonuçlar
Glukan fosfataz protein ailesinin temel özelliklerinden biri, glukan substratlarına bağlanma yetenekleridir. İlk olarak, SEX4'ün ConA-Sepharose:amilopectin boncuklarına bağlanma kapasitesi SDS-PAGE kullanılarak analiz edildi (Şekil 2A). Sığır serum albümini (BSA), proteinlerin ConA-Sefaroz: amilopektin boncuklarına spesifik olmayan herhangi bir bağlanmasını tespit etmek için negatif bir kontrol görevi gördü. Proteinlerin SDS-PAGE analizi, pelet fraksiyonunda SEX4 protein...
Tartışmalar
Bu çalışma, glukan-glukan fosfataz etkileşimlerinin bağlanma afinitesinin belirlenmesine izin veren yeni bir in vitro sedimantasyon testinin başarılı bir şekilde geliştirildiğini göstermektedir. Tahlil tasarımı, lektin ConA'nın glikozun hidroksil kalıntıları yoluyla glukanlara spesifik olarak bağlanmasından yararlanarak, çözünür karbonhidrat substratlarını Sefaroz boncuklarına dolaylı olarak yakalar. Bu, bağlı ve bağlanmamış protein fraksiyonlarının santrifüjleme ...
Açıklamalar
Yazarlar çıkar çatışması olmadığını beyan ederler.
Teşekkürler
Bu çalışma Ulusal Bilim Vakfı ödülü MCB-2012074 tarafından desteklenmiştir. Yazarlar, Florida Üniversitesi Biyokimya ve Moleküler Biyoloji Bölümü'nden Dr. Craig W. Vander Kooi'ye değerli tartışmalar ve destekler için teşekkür eder. Yazarlar ayrıca Florida Üniversitesi Biyokimya ve Moleküler Biyoloji Bölümü'nden Dr. Matthew S. Gentry'ye desteği için teşekkür eder. Skidmore College Neuroscience programının başkanı Dr. Sara Lagalwar'a, batı leke görüntülemesi için LICOR C basamaklı leke tarayıcısını kullanmamıza izin verdiği için teşekkür ederiz.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 6x-His Tag monoclonal antibody (HIS.H8), HRP | Therm Fisher Scientific | MA1-21315-HRP | |
| Biorad gel electrophoresis and Western blot kit | Biorad | 1703930 | |
| Calcium chloride | Sigma-Aldrich | 208291 | |
| C-Digit blot scanner | LICOR | 3600-00 | Blot scanner |
| Complete protease inhibitor cocktail | Sigma-Aldrich | 11836170001 | |
| Concanavalin A-sepharose beads | Sigma-Aldrich | C9017 | This product contains in 0.1 M acetate buffer, pH 6, containing 1 M NaCl, 1 mM CaCl2, 1 mM MnCl2, and 1 mM MgCl2 in 20% ethanol |
| Centrifuge | Eppendorf | 5425R | |
| Glycine | Fisher Scientific | BP381-5 | |
| GraphPad Prism 8.0 software | GraphPad | Version 8.0 | Data analysis software |
| HEPES | Sigma-Aldrich | H8651 | |
| Image Studio | LICOR | 3600-501 | Acquisition Software |
| Magnesium chloride | Sigma-Aldrich | M2670 | |
| Methanol | Fisher Scientific | A452SK-4 | |
| Sodium dodecyl sulfate | Fisher Scientific | PI28312 | |
| Potato amylopectin | Sigma-Aldrich | A8515 | |
| Precast SDSPAGE Gels | Genscript | M00653S | |
| Tris base | Fisher Scientific | BP154-1 | |
| Tween 20 | Fisher Scientific | MP1TWEEN201 | |
| Westernsure premium chemiluminescence substrate | LI-COR | 926-95000 |
Referanslar
- Meekins, D. A., Vander Kooi, C. W., Gentry, M. S. Structural mechanisms of plant glucan phosphatases in starch metabolism. The FEBS Journal. 283 (13), 2427-2447 (2016).
- Gentry, M. S., et al. The phosphatase laforin crosses evolutionary boundaries and links carbohydrate metabolism to neuronal disease. The Journal of Cell Biology. 178 (3), 477-488 (2007).
- Worby, C. A., Gentry, M. S., Dixon, J. E. Laforin, a dual specificity phosphatase that dephosphorylates complex carbohydrates. The Journal of Biological Chemistry. 281 (41), 30412-30418 (2006).
- Gentry, M. S., Pace, R. M. Conservation of the glucan phosphatase laforin is linked to rates of molecular evolution and the glucan metabolism of the organism. BMC Evolutionary Biology. 9, 138 (2009).
- Niittyla, T., et al. Similar protein phosphatases control starch metabolism in plants and glycogen metabolism in mammals. The Journal of Biological Chemistry. 281 (17), 11815-11818 (2006).
- Kotting, O., et al. STARCH-EXCESS4 is a laforin-like Phosphoglucan phosphatase required for starch degradation in Arabidopsis thaliana. The Plant Cell. 21 (1), 334-346 (2009).
- Comparot-Moss, S., et al. A putative phosphatase, LSF1, is required for normal starch turnover in Arabidopsis leaves. Plant Physiology. 152 (2), 685-697 (2010).
- Zeeman, S. C., Northrop, F., Smith, A. M., Rees, T. A starch-accumulating mutant of Arabidopsis thaliana deficient in a chloroplastic starch-hydrolysing enzyme. The Plant Journal: For Cell and Molecular Biology. 15 (3), 357-365 (1998).
- Kotting, O., et al. Identification of a novel enzyme required for starch metabolism in Arabidopsis leaves. The phosphoglucan, water dikinase. Plant Physiology. 137 (1), 242-252 (2005).
- Tagliabracci, V. S., et al. Laforin is a glycogen phosphatase, deficiency of which leads to elevated phosphorylation of glycogen in vivo. Proceedings of the National Academy of Sciences. 104 (49), 19262-19266 (2007).
- Gentry, M. S., Guinovart, J. J., Minassian, B. A., Roach, P. J., Serratosa, J. M. Lafora disease offers a unique window into neuronal glycogen metabolism. The Journal of Biological Chemistry. 293 (19), 7117-7125 (2018).
- Brewer, M. K., et al. Targeting pathogenic lafora bodies in lafora disease using an antibody-enzyme fusion. Cell Metabolism. 30 (4), 689-705 (2019).
- Santelia, D., Zeeman, S. C. Progress in Arabidopsis starch research and potential biotechnological applications. Current Opinion in Biotechnology. 22 (2), 271-280 (2011).
- Raththagala, M., et al. Structural mechanism of laforin function in glycogen dephosphorylation and lafora disease. Molecular Cell. 57 (2), 261-272 (2015).
- Meekins, D. A., et al. Phosphoglucan-bound structure of starch phosphatase Starch Excess4 reveals the mechanism for C6 specificity. Proceedings of the National Academy of Sciences. 111 (20), 7272-7277 (2014).
- Vander Kooi, C. W., et al. Structural basis for the glucan phosphatase activity of Starch Excess4. Proceedings of the National Academy of Sciences. 107 (35), 15379-15384 (2010).
- Meekins, D. A., et al. Structure of the Arabidopsis glucan phosphatase like sex four2 reveals a unique mechanism for starch dephosphorylation. The Plant Cell. 25 (6), 2302-2314 (2013).
- Smith, A. M., Zeeman, S. C. Starch: A flexible, adaptable carbon store coupled to plant growth. Annual Review of Plant Biology. 71, 217-245 (2020).
- Jane, J., Kasemuwan, T., Chen, J. F., Juliano, B. O. Phosphorus in rice and other starches. Cereal Foods World. 41 (11), 827-832 (1996).
- Mak, C. A., et al. Cooperative kinetics of the glucan phosphatase starch excess4. Biochemistry. 60 (31), 2425-2435 (2021).
- Campbell, K. P., MacLennan, D. H. Purification and characterization of the 53,000-dalton glycoprotein from the sarcoplasmic reticulum. The Journal of Biological Chemistry. 256 (9), 4626-4632 (1981).
- Campbell, K. P., MacLennan, D. H., Jorgensen, A. O., Mintzer, M. C. Purification and characterization of calsequestrin from canine cardiac sarcoplasmic reticulum and identification of the 53,000 dalton glycoprotein. The Journal of Biological Chemistry. 258 (2), 1197-1204 (1983).
- Davey, M. W., Sulkowski, E., Carter, W. A. Binding of human fibroblast interferon to concanavalin A-agarose. Involvement of carbohydrate recognition and hydrophobic interaction. Biochemistry. 15 (3), 704-713 (1976).
- Meekins, D. A., et al. Mechanistic insights into glucan phosphatase activity against polyglucan substrates. The Journal of Biological Chemistry. 290 (38), 23361-23370 (2015).
- Wilkens, C., et al. Plant α-glucan phosphatases SEX4 and LSF2 display different affinity for amylopectin and amylose. FEBS Letters. 590 (1), 118-128 (2016).
- Atanasova, M., Bagdonas, H., Agirre, J. Structural glycobiology in the age of electron cryo-microscopy. Current Opinion in Structural Biology. 62, 70-78 (2020).
- Doyle, M. L. Characterization of binding interactions by isothermal titration calorimetry. Current Opinion in Biotechnology. 8 (1), 31-35 (1997).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır