Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Perfil fenotípico de neuronas dopaminérgicas del mesencéfalo derivadas de células madre humanas
En este artículo
Resumen
Este protocolo describe el cultivo celular de neuronas dopaminérgicas del mesencéfalo humano, seguido de la tinción inmunológica y la generación de perfiles fenotípicos neuronales a partir de imágenes microscópicas adquiridas de alto contenido que permiten la identificación de variaciones fenotípicas debidas a modulaciones genéticas o químicas.
Resumen
La enfermedad de Parkinson (EP) está relacionada con una serie de procesos biológicos celulares que causan la pérdida de neuronas dopaminérgicas del mesencéfalo (mDA). Muchos modelos celulares actuales de EP in vitro carecen de complejidad y no tienen en cuenta múltiples fenotipos. El perfil fenotípico en neuronas mDA derivadas de células madre pluripotentes inducidas humanas (iPSC) puede abordar estas deficiencias midiendo simultáneamente una serie de fenotipos neuronales en un tipo de célula relevante para la EP en paralelo. Aquí, describimos un protocolo para obtener y analizar perfiles fenotípicos de neuronas mDA humanas disponibles comercialmente. Se utiliza un panel de tinción fluorescente específico de neurona para visualizar los fenotipos relacionados con la proteína nuclear, la α-sinucleína, la tirosina hidroxilasa (TH) y la proteína 2 asociada a microtúbulos (MAP2). El protocolo de perfil fenotípico descrito es escalable, ya que utiliza placas de 384 pocillos, manipulación automática de líquidos y microscopía de alto rendimiento. La utilidad del protocolo se ejemplifica utilizando neuronas mDA de donantes sanos y neuronas mDA portadoras de la mutación G2019S ligada a PD en el gen de la quinasa repetida 2 rica en leucina (LRRK2). Ambas líneas celulares fueron tratadas con el inhibidor de la quinasa LRRK2 PFE-360 y se midieron los cambios fenotípicos. Además, demostramos cómo se pueden analizar los perfiles fenotípicos multidimensionales utilizando métodos de clasificación supervisada basados en clústeres o aprendizaje automático. El protocolo descrito interesará especialmente a los investigadores que trabajan en el modelado de enfermedades neuronales o en el estudio de los efectos de los compuestos químicos en las neuronas humanas.
Introducción
En la enfermedad de Parkinson (EP) se alteran diversos procesos biológicos celulares. Por ejemplo, la disfunción mitocondrial, el estrés oxidativo, los defectos de degradación de proteínas, la interrupción del tráfico vesicular y la función endolisosomal se han asociado con la pérdida de neuronas dopaminérgicas del mesencéfalo (mDA), se observan comúnmente en la EP1. Por lo tanto, la EP parece implicar múltiples mecanismos de enfermedad que pueden interactuar y empeorarse entre sí. Una forma útil de investigar esta interacción mecanicista es la creación de una huella dactilar fenotípica completa o un perfil de las neuronas dopaminérgicas del mesencéfalo (mDA).
El perfil fenotípico es un enfoque que implica la creación de un perfil de una muestra basado en una colección de características medibles y, en segundo lugar, implica hacer predicciones sobre una muestra basada en este perfil 2,3. El objetivo de la elaboración de perfiles es capturar una amplia gama de características, algunas de las cuales pueden no haberse asociado previamente con una enfermedad o tratamiento3. Como resultado, la elaboración de perfiles puede revelar procesos biológicos inesperados. El perfil fenotípico generalmente se basa en células teñidas con fluorescencia, y se han desarrollado ensayos estandarizados, como Cell Painting, para crear perfiles fenotípicos4. Recientemente, el perfil fenotípico se ha aplicado, por ejemplo, para la caracterización de moléculas pequeñas o la predicción precisa de subtipos de EP basados únicamente en fibroblastos derivados del paciente 5,6. A pesar de estos avances, el perfil fenotípico rara vez se ha aplicado a células diferenciadas postmitóticas, como las neuronas mDA derivadas de células madre pluripotentes inducidas humanas (iPSC) que expresan mutaciones ligadas a la enfermedad de Parkinson, como LRRK2 G2019S. Los desafíos significativos de los modelos derivados de iPSC incluyen la presencia de características patológicas sutiles o variables en lotes de diferenciación o genotipos, y el hecho de que los fenotipos aislados de EP no capturan toda la complejidad de la enfermedad. Además, aunque los modelos neuronales de iPSC son fisiológicamente relevantes, rara vez se utilizan en los procesos de descubrimiento de fármacos en la EP debido a las preocupaciones sobre la complejidad técnica 7,8.
Previamente desarrollamos una metodología robusta para medir múltiples fenotipos fisiopatológicos relacionados con la EP en neuronas mDA humanas que son sensiblesa los cambios fenotípicos inducidos por compuestos genéticos y químicos. Este artículo describe en detalle una versión optimizada de esta metodología para crear perfiles fenotípicos a partir de neuronas mDA (Figura 1). Este protocolo tiene varias ventajas sobre los enfoques de perfiles fenotípicos descritos anteriormente, como el uso de neuronas mDA de alta calidad y la reproducibilidad técnica. Por primera vez, este protocolo permite el perfil fenotípico en neuronas mDA postmitóticas fisiológicamente relevantes después de perturbaciones químicas de una manera altamente escalable. Las neuronas mDA totalmente diferenciadas y criopreservadas están disponibles comercialmente, lo que disminuye significativamente la variabilidad de la diferenciación de un lote a otro. En segundo lugar, la variabilidad técnica puede reducirse aún más mediante el uso de un diseño experimental bien definido (es decir, la duración del cultivo o evitar los pozos de borde), la manipulación automatizada de líquidos y la microscopía automatizada. Además, aquí se describen los pasos iniciales del análisis del perfil fenotípico mediante enfoques de agrupamiento no supervisado o clasificación supervisada, lo que indica cómo se pueden analizar los datos del perfil fenotípico. Este protocolo será de utilidad para los investigadores interesados en los cambios fenotípicos de las neuronas mDA inducidos por perturbaciones genéticas o químicas, específicamente cuando se requiere una configuración de estudio altamente escalable, por ejemplo, durante las campañas de cribado o cuando se van a estudiar los efectos de un número menor de compuestos, por ejemplo, para determinar los efectos tóxicos. En resumen, se anticipa que la aplicación del perfil fenotípico de las neuronas humanas es una técnica valiosa para estudiar fenotipos complejos relacionados con enfermedades y caracterizar los efectos celulares de los candidatos a fármacos.
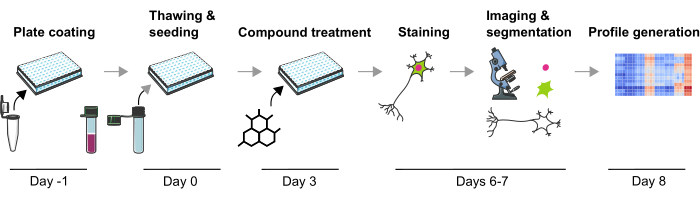
Figura 1: Representación esquemática del protocolo experimental para generar perfiles fenotípicos basados en imágenes a partir de neuronas mDA derivadas de iPSC humanas. Haga clic aquí para ver una versión más grande de esta figura.
Access restricted. Please log in or start a trial to view this content.
Protocolo
1. Preparación del medio y placas para la siembra de neuronas (Día 1)
- Para preparar las placas para la siembra de neuronas en el día 1, caliente Laminin a temperatura ambiente (RT) justo antes de usarlo. Prepare la solución de laminina diluyendo la solución madre de laminina (0,1 mg/ml) 1/10 en PBS+/+ frío (con Ca 2+ y Mg2+).
NOTA: Todos los reactivos se enumeran en la Tabla de materiales. Las composiciones de las soluciones y los tampones se describen en las Tablas 1-4. - A continuación, añadir 25 μL de la solución de laminina a cada pocillo de una placa de 384 pocillos prerrecubierta de poli-D-lisina (PDL) e incubar durante la noche a 4 °C. Las placas recubiertas pueden almacenarse a 4 °C durante un máximo de una semana.
NOTA: El protocolo se puede pausar aquí hasta por una semana. Selle las placas con una película de plástico. - Prepare los medios de mantenimiento completos y guárdelos a 4 °C durante un máximo de un mes (Tabla 1).
2. Descongelación de neuronas (Día 0)
- Para descongelar las neuronas en el día 0, precalentar un baño de agua a 37 °C y equilibrar el medio de mantenimiento completo a RT, protegido de la luz.
- Saque el vial que contiene las neuronas congeladas obtenidas comercialmente (consulte la Tabla de materiales) del tanque de nitrógeno líquido y colóquelo sobre hielo seco. A continuación, coloque el vial en el baño de agua durante 2 min. Una vez que el líquido esté completamente descongelado, desinfecte el vial con etanol al 70%.
- Aspirar las neuronas descongeladas con una pipeta P1000 (alrededor de 370 μL) y transferirlas en un tubo de centrífuga de 50 ml sin pipeteo ascendente y descendente. A continuación, enjuague el vial con 630 μL de medio de mantenimiento completo, dispense gota a gota (ángulo de 45°) en el tubo de 50 ml. Agite lentamente mientras dispensa.
NOTA: Es fundamental dispensar el medio lentamente, gota a gota, para evitar la ruptura osmótica de las células. Un ángulo de 45° y una ligera agitación minimizan la alta presión osmótica local durante el pipeteo. - Del mismo modo, agregue 1 ml de medio de mantenimiento completo en el tubo de centrífuga de 50 ml con una pipeta P1000. Luego, agregue lentamente 2 ml de medio de mantenimiento completo. Agite con cuidado mientras dispensa.
- Cuenta las celdas. Prepare un microtubo con 10 μL de Azul de Tripán y 10 μL de suspensión celular y agréguelo a un portaobjetos de la cámara de recuento (10 μL). Realice el recuento utilizando un contador de celdas automatizado (consulte Tabla de materiales) o manualmente.
- Después de contar, centrifugar el tubo de 50 ml que contiene las neuronas a 400 x g durante 5 min a RT y eliminar el sobrenadante. Vuelva a suspender cuidadosamente el gránulo con una pipeta P1000 y 1 ml de medio de mantenimiento completo. A continuación, añada el volumen necesario para alcanzar la concentración deseada (300.000 células/ml, véase también el siguiente paso).
3. Siembra de neuronas en platos preparados (Día 0)
- Para sembrar neuronas en las placas preparadas el día 0, saque las placas recubiertas del refrigerador, colóquelas debajo de la campana de cultivo celular y déjelas equilibrarse a RT durante unos 30 minutos.
- Justo antes de la siembra, aspire 15 μL de solución de recubrimiento con un manipulador de líquidos automatizado o una pipeta de 16 canales. Deje aproximadamente 10 μL por pocillo para evitar daños en el recubrimiento.
- A continuación, dispense 50 μL de la solución celular que contiene 300.000 células/ml (preparada en el paso 2.6) por pocillo con una pipeta de 16 canales, lo que da como resultado 15.000 neuronas sembradas por pocillo y un volumen final por pocillo de 60 μL.
- En una placa de 384 pocillos, evite usar las columnas 1, 2, 23, 24 y las filas A, B, O y P para minimizar los posibles efectos de borde que pueden afectar los perfiles fenotípicos. Llene los pocillos vacíos no utilizados con 80 μL de PBS. Incubar las placas a 37 °C y 5% de CO2.
4. Cambio medio o tratamiento compuesto (Día 3)
- Dependiendo del número de pocillos, precaliente un volumen apropiado de medio de mantenimiento completo en RT. Proteja de la luz.
- Si se requiere un cambio de medio, continúe con el paso 4.4. Si se desea un tratamiento compuesto, verifique la concentración de material compuesto y el disolvente utilizado (agua, DMSO, metanol, etc.).
- Prepare una solución compuesta concentrada 1,5x para todas las concentraciones deseadas que se van a probar. Prepare las diluciones compuestas utilizando un medio de mantenimiento completo.
NOTA: Como control neutro, utilice el disolvente respectivo a la misma concentración que el compuesto probado. Si se utilizan compuestos múltiples o desconocidos a diferentes concentraciones, es aconsejable realizar un experimento dosis-respuesta por separado para medir los efectos del disolvente en el perfil fenotípico. - Si se utiliza un sistema de pipeteo automático, agregue 60 μL de la solución compuesta 1.5x a una placa de almacenamiento de 384 pocillos. También puede utilizar una pipeta de 16 canales. Si se requiere un cambio de medio, agregue 60 μL de medio de mantenimiento completo en su lugar.
- Utilizando el sistema de pipeteo automático, aspire y deseche 40 μL de medio por pocillo de la placa que contiene neuronas para mantener 20 μL/pocillo. A continuación, añada 40 μL/pocillo de solución compuesta 1,5x de la placa de almacenamiento de 384 pocillos a cada pocillo para obtener la concentración final deseada.
NOTA: Es fundamental no realizar cambios completos de medio, sino dejar siempre el medio residual en el pocillo para evitar daños en la alfombra neuronal o en el revestimiento. - En caso de que el cultivo neuronal se realice durante más de los 6 días descritos en este protocolo, cambiar el medio cada 2-3 días.
5. Fijación y tinción de neuronas (días 6 y 7)
- Para fijar y teñir las neuronas en los días 6 y 7, utilice un manipulador de líquidos automatizado para todos los pasos de dispensación y lavado. También puede utilizar una pipeta de 16 canales.
- Prepare una solución de Triton X-100 al 10 % diluyendo Triton X-100 en 1x PBS. Vórtice hasta que la solución sea homogénea. Conservar a 4 °C.
- Para fijar las neuronas, dispense 20 μL/pocillo de PFA al 16%, lo que da como resultado una concentración final del 4%. Incubar la placa durante 30 minutos a temperatura media y lavarla tres veces con 1x PBS. Deje 20 μL/pocillo de PBS después del último lavado.
PRECAUCIÓN: El PFA es reconocido como una sustancia peligrosa que se sabe que causa toxicidad oral, dérmica y respiratoria. También representa una amenaza para los ojos y puede provocar mutaciones genéticas y cáncer. El manejo adecuado de PFA requiere el uso de equipos de protección personal adecuados, como protección para los ojos y la cara, además de garantizar una ventilación adecuada. Es importante evitar la liberación de PFA en el medio ambiente. - Para la permeabilización y el bloqueo, prepare una solución de bloqueo 2x (Tabla 2).
- Añadir 20 μL/pocillo de 2x solución de bloqueo (1x concentración final), incubar durante 1 h a RT y lavar una vez con PBS. Conserve 20 μL/pocillo de PBS después del lavado.
- Para la tinción de anticuerpos primarios (ver Tabla de materiales), prepare un tampón de tinción primaria 2x (Tabla 3).
- Añadir 20 μL/pocillo de tampón de tinción primario 2x (1x concentración final) e incubar durante la noche a 4 °C. A la mañana siguiente, el día 7, lávese tres veces con PBS. Deje 20 μL/pocillo de PBS después del lavado.
- Para la tinción de anticuerpos secundarios (ver Tabla de Materiales), prepare un tampón de tinción secundaria 2x (Tabla 4).
- Añadir 20 μL/pocillo de tampón de tinción secundario 2x (1x concentración final), incubar durante 2 h a RT lejos de la luz y lavar tres veces con PBS. Deje 100 μL de PBS/pocillo después del último lavado.
- Agregue un sello de aluminio en la placa para minimizar la evaporación. Alternativamente, cubra el plato con una película de plástico y papel de aluminio. Proceda a la adquisición de imágenes o guarde la placa a 4 °C.
NOTA: El protocolo se puede pausar aquí hasta por una semana. Si se observa fluorescencia de fondo, considere aumentar el tiempo de bloqueo. Si la tinción es insuficiente, intente aumentar la concentración de anticuerpos primarios o el tiempo de incubación.
- Agregue un sello de aluminio en la placa para minimizar la evaporación. Alternativamente, cubra el plato con una película de plástico y papel de aluminio. Proceda a la adquisición de imágenes o guarde la placa a 4 °C.
6. Obtención de imágenes de neuronas teñidas con fluorescencia (Día 7)
- Adquirir imágenes de las neuronas plateadas, cultivadas y teñidas en el día 7. Lo ideal es utilizar un microscopio de fluorescencia confocal automatizado (véase la tabla de materiales). También puede adquirir imágenes manualmente.
- Adquiera los canales Hoechst, TH, α-sinucleína y MAP2 utilizando láseres de 405 nm, 488 nm, 561 nm y 647 nm, respectivamente (Figura 2).
NOTA: Para generar una cantidad suficiente de datos detallados para el perfil fenotípico, utilice un objetivo de 40x y adquiera 16 campos/pocillo utilizando pilas Z que constan de 3 cortes en Z separados por 2 μm. En caso de que haya daños relacionados con el pipeteo en la alfombra neuronal, trate de evitar obtener imágenes de estas áreas. - Dependiendo del microscopio y la cámara, ajuste los tiempos de exposición y las intensidades de excitación para cada uno de los cuatro canales fluorescentes por separado para obtener un rango dinámico óptimo de las intensidades fluorescentes.
NOTA: El software de imágenes a menudo proporciona un histograma para determinar el tiempo de exposición ideal. Si el histograma se desplaza demasiado hacia la izquierda en el rango de señal baja, entonces el tiempo de exposición es demasiado corto o la intensidad de excitación es demasiado baja. Si hay un acantilado agudo en el nivel máximo de señal a la derecha, entonces el valor de la señal está saturado. En este caso, reduzca la intensidad de excitación o acorte el tiempo de exposición. - Almacene imágenes en un formato abierto y sin pérdidas, como .tif.
7. Procesamiento de imágenes (Día 8)
- La segmentación de imágenes y la extracción de características fenotípicas son necesarias para la creación de perfiles fenotípicos cuantitativos. Utilice el software PhenoLink para la segmentación de imágenes y la extracción de características (Tabla de materiales). Las instrucciones para instalar PhenoLink se pueden encontrar en el repositorio de GitHub (https://github.com/Ksilink/PhenoLink).
NOTA: La segmentación de imágenes se utiliza para identificar y separar diferentes objetos o regiones de una imagen, mientras que la extracción de características fenotípicas se utiliza para analizar y extraer información relevante de esas regiones. Varias soluciones de software alternativas, como CellProfiler 10, ImageJ/FIJI11, Napari 12 o Knime Analytics Platform13 están disponibles para extraer información cuantitativa de imágenes de fluorescencia multicanal. - Realice la segmentación de imágenes en imágenes RAW con corrección de iluminación. Determine empíricamente los respectivos umbrales de intensidad del canal fluorescente por placa para que la señal de fondo sea mínima y la señal segmentada deseada corresponda a la señal de la imagen sin procesar. Las placas procesadas y teñidas en el mismo día suelen requerir umbrales de intensidad de canal comparables para la segmentación.
- Definir el tamaño y la intensidad del núcleo para separar las células vivas de las muertas. Cuando utilice imágenes de 40x, mantenga todos los demás parámetros predeterminados y ejecute el software. Se calcularán ciento veintiséis características cuantitativas de la imagen por pocillo (Tabla suplementaria 1).
- Utilice los datos cuantitativos tabulares resultantes para construir perfiles fenotípicos y comparar perfiles fenotípicos de diferentes líneas celulares o condiciones de tratamiento. Cada fila corresponde a una condición biológica (pozo) y cada columna corresponde a una característica fenotípica determinada.
NOTA: Proporcionamos un archivo de salida de ejemplo junto con la canalización de análisis de datos para ilustrar su uso (consulte la Tabla de materiales). Además, en la Figura 3 se muestra la composición de un perfil fenotípico.
8. Generación y visualización del perfil fenotípico (Día 8)
- Si no tiene Python y Jupyter instalados en su computadora, instale Anaconda Distribution y abra el software Jupyter. Descargue el cuaderno de Jupyter Notebook proporcionado y todos los demás archivos proporcionados y guárdelos en el mismo directorio (consulte Tabla de materiales). Abra el archivo de Jupyter Notebook con el software Jupyter.
NOTA: Anaconda es una plataforma gratuita y de código abierto para lenguajes de programación como Python. Esta plataforma viene con el intérprete de Python Jupyter que puede ejecutar el cuaderno de Jupyter proporcionado para crear y analizar perfiles fenotípicos (https://github.com/Ksilink/Notebooks/tree/main/Neuro/DopaNeuronProfiling). - Ejecute el cuaderno de Jupyter Notebook celda por celda con el software de Jupyter. Los archivos .txt de datos de ejemplo y requisitos proporcionados deben ubicarse en el mismo directorio que Jupyter Notebook. Cada celda de Jupyter Notebook está anotada para explicar su funcionalidad.
NOTA: Es fundamental utilizar Jupyter Notebook en el orden correcto, empezando por la carga y el escalado de datos, y continuar celda por celda hasta el final. Todos los datos y gráficos de salida se almacenarán en una carpeta recién creada en el directorio de origen de Jupyter Notebook. En la figura 4 se muestra el flujo de trabajo y el resultado del flujo de trabajo.
Access restricted. Please log in or start a trial to view this content.
Resultados
El perfil fenotípico en neuronas mDA es una forma eficiente de cuantificar múltiples aspectos de la biología celular y sus cambios durante la modulación experimental. Para ejemplificar esta metodología, este estudio hizo uso de LRRK2 G2019S criopreservado y neuronas mDA sanas de donantes. Estas neuronas han sido diferenciadas durante aproximadamente 37 días, son marcadores neuronales postmitóticos y expresos (TUBB3 y MAP2) y marcadores neuronales dopaminérgicos, incluyendo tirosina hidroxilasa (TH) en combinació...
Access restricted. Please log in or start a trial to view this content.
Discusión
El perfil fenotípico es una técnica para medir un gran número de fenotipos en las células mediante la aplicación de tinciones fluorescentes, microscopía y análisis de imágenes3. Los perfiles fenotípicos se pueden obtener y comparar a través de líneas celulares u otras condiciones experimentales para comprender cambios complejos en la biología celular que podrían pasar desapercibidos cuando se usa una sola lectura. En este trabajo describimos la aplicación del perfil fenotípico a las...
Access restricted. Please log in or start a trial to view this content.
Divulgaciones
Todos los autores son empleados de Ksilink.
Agradecimientos
Los autores desean agradecer a todos los colegas de Ksilink por su valiosa ayuda y discusiones que condujeron al diseño del protocolo presentado.
Access restricted. Please log in or start a trial to view this content.
Materiales
| Name | Company | Catalog Number | Comments |
| Anti- chicken – Alexa 647 | Jackson ImmunoRearch | 703-605-155 | Immunofluorescence |
| Anaconda | https://www.anaconda.com/download | ||
| Anti-Map2 | Novus | NB300-213 | Immunofluorescence |
| Anti-mouse - Alexa 488 | Thermo Fisher | A11001 | Immunofluorescence |
| Anti-rabbit - Alexa 555 | Thermo Fisher | A21429 | Immunofluorescence |
| Anti-Tyrosine Hydroxylase | Merck | T2928 | Immunofluorescence |
| Anti-α-synuclein | Abcam | 138501 | Immunofluorescence |
| Bravo Automated Liquid Handling Platform with 384ST head | Agilent | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Confocal microscope | Yokogawa | CV7000 | The use of an automated confocal fluorescence microscope is recommended to ensure image quality consistency. |
| Countess Automated cell counter | Invitrogen | Cell counting before seeding. Can also be done using a manual counting chamber. | |
| DPBS +/+ | Gibco | 14040-133 | Buffer for washing |
| EL406 Washer Dispenser | BioTek (Agilent) | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Formaldehyde Solution (PFA 16 %) | Euromedex | EM-15710-S | Fixation before staining |
| Hoechst 33342 | Invitrogen | H3570 | Nuclear staining |
| iCell Base Medium 1 | Fujifilm | M1010 | Base medium for neurons |
| iCell DPN, Donor#01279, Phenotype AHN, lot#106339, 1M | Fujifilm | C1087 | Apparently healthy donor |
| iCell DPN, Donor#11299, Phenotype LRRK2 G2019S, phenotype PD lot#106139 | Fujifilm | C1149 | Donor carrying LRRK2 G2019S mutation |
| iCell Nervous System Supplement | Fujifilm | M1031 | Supplement for base medium |
| iCell Neural Supplement B | Fujifilm | M1029 | Supplement for base medium |
| Jupyter Python Notebook | In-house development | https://github.com/Ksilink/Notebooks/tree/main/Neuro/DopaNeuronProfiling | Notebook to perform phenotypic profile visualization and classification from raw data. |
| Laminin | Biolamina | LN521 | Plate coating |
| PFE-360 | MedChemExpress | HY-120085 | LRRK2 kinase inhibitor |
| PhenoLink | In-house development | https://github.com/Ksilink/PhenoLink | Software for image analysis |
| PhenoPlate 384w, PDL coated | Perkin Elmer | 6057500 | Pre-coated plate for cell culture and imaging. This plate allows imaging of all wells using all objectives of the Yokogawa CV7000 microscope. |
| Storage plates Abgene 120 µL | Thermo Scientific | AB-0781 | Necessary for compound dispensing using the Vprep pipetting system. If not available, the use of an electronic multichannel pipette is recommended. |
| Triton | Sigma | T9284 | Permeabilization before lysis |
| Trypan Blue | Sigma | T8154-20ML | Determination of living cells |
| Vprep Pipetting System | Agilent | Medium change and compound dispensing. Alternatively, an electronic multichannel pipette can be used. |
Referencias
- Panicker, N., Ge, P., Dawson, V. L., Dawson, T. M. The cell biology of Parkinson's disease. The Journal of Cell Biology. 220 (4), 202012095(2021).
- Caicedo, J. C., et al. Data-analysis strategies for image-based cell profiling. Nature Methods. 14 (9), 849-863 (2017).
- Chandrasekaran, S. N., Ceulemans, H., Boyd, J. D., Carpenter, A. E. Image-based profiling for drug discovery: due for a machine-learning upgrade. Nature Reviews Drug Discovery. 20 (2), 145-159 (2021).
- Bray, M. -A., et al. Cell Painting, a high-content image-based assay for morphological profiling using multiplexed fluorescent dyes. Nature Protocols. 11 (9), 1757-1774 (2016).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590(2022).
- Ziegler, S., Sievers, S., Waldmann, H. Morphological profiling of small molecules. Cell Chemical Biology. 28 (3), 300-319 (2021).
- Cobb, M. M., Ravisankar, A., Skibinski, G., Finkbeiner, S. iPS cells in the study of PD molecular pathogenesis. Cell and Tissue Research. 373 (1), 61-77 (2018).
- Elitt, M. S., Barbar, L., Tesar, P. J. Drug screening for human genetic diseases using iPSC models. Human Molecular Genetics. 27 (R2), 89-98 (2018).
- Vuidel, A., et al. High-content phenotyping of Parkinson's disease patient stem cell-derived midbrain dopaminergic neurons using machine learning classification. Stem Cell Reports. 17 (10), 2349-2364 (2022).
- Stirling, D. R., Swain-Bowden, M. J., Lucas, A. M., Carpenter, A. E., Cimini, B. A., Goodman, A. CellProfiler 4: improvements in speed, utility and usability. BMC Bioinformatics. 22 (1), 433(2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Sofroniew, N., et al. napari: a multi-dimensional image viewer for Python. , (2022).
- Berthold, M. R., et al. KNIME: The Konstanz Information Miner. Data Analysis, Machine Learning and Applications. , 319-326 (2008).
- Fathi, A., et al. Diverging Parkinson's Disease Pathology between patient-derived GBAN370S, LRRK2G2019S and engineered SNCAA53T iPSC-derived Dopaminergic Neurons. bioRxiv. , (2023).
- Wang, Y., Huang, H., Rudin, C., Shaposhnik, Y. Understanding How Dimension Reduction Tools Work: An Empirical Approach to Deciphering t-SNE, UMAP, TriMap, and PaCMAP for Data Visualization. Journal of Machine Learning Research. 22 (201), 1-73 (2021).
- Ke, G., et al. LightGBM: A highly efficient gradient boosting decision tree. Advances in Neural Information Processing Systems. 30, https://papers.nips.cc/paper/2017/hash/6449f44a102fde848669bdd9eb6b76fa-Abstract.html (2017).
- Avazzadeh, S., Baena, J. M., Keighron, C., Feller-Sanchez, Y., Quinlan, L. R. Modelling Parkinson's Disease: iPSCs towards Better Understanding of Human Pathology. Brain Sciences. 11 (3), 373(2021).
- Sánchez-Danés, A., et al. Disease-specific phenotypes in dopamine neurons from human iPS-based models of genetic and sporadic Parkinson's disease. EMBO Molecular Medicine. 4 (5), 380-395 (2012).
- Oosterveen, T., et al. Pluripotent stem cell derived dopaminergic subpopulations model the selective neuron degeneration in Parkinson's disease. Stem Cell Reports. 16 (11), 2718-2735 (2021).
- Hughes, R. E., et al. Multiparametric high-content cell painting identifies copper ionophores as selective modulators of esophageal cancer phenotypes. ACS Chemical Biology. 17 (7), 1876-1889 (2022).
- Akbarzadeh, M., et al. Morphological profiling by means of the Cell Painting assay enables identification of tubulin-targeting compounds. Cell Chemical Biology. 29 (6), 1053-1064 (2022).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590(2022).
- Way, G. P., et al. Morphology and gene expression profiling provide complementary information for mapping cell state. Cell Systems. 13 (11), 911-923 (2022).
- Feng, Y., Mitchison, T. J., Bender, A., Young, D. W., Tallarico, J. A. Multi-parameter phenotypic profiling: using cellular effects to characterize small-molecule compounds. Nature Reviews Drug Discovery. 8 (7), 567-578 (2009).
- Antonov, S. A., Novosadova, E. V. Current state-of-the-art and unresolved problems in using human induced pluripotent stem cell-derived dopamine neurons for parkinson's disease drug development. International Journal of Molecular Sciences. 22 (7), 3381(2021).
- Miller, J. D., et al. Human iPSC-based modeling of late-onset disease via progerin-induced aging. Cell Stem Cell. 13 (6), 691-705 (2013).
- Bezard, E., Gross, C. E., Brotchie, J. M. Presymptomatic compensation in Parkinson's disease is not dopamine-mediated. Trends in Neurosciences. 26 (4), 215-221 (2003).
- Wu, Y., Le, W., Jankovic, J. Preclinical Biomarkers of parkinson disease. Archives of Neurology. 68 (1), 22-30 (2011).
- Verstraelen, P., et al. Systematic quantification of synapses in primary neuronal culture. iScience. 23 (9), 101542(2020).
- Liu-Yesucevitz, L., et al. ALS-Linked mutations enlarge TDP-43-enriched neuronal RNA granules in the dendritic arbor. The Journal of Neuroscience. 34 (12), 4167-4174 (2014).
Access restricted. Please log in or start a trial to view this content.
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados