Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Profilage phénotypique des neurones dopaminergiques du mésencéphale dérivés de cellules souches humaines
Dans cet article
Résumé
Ce protocole décrit la culture cellulaire de neurones dopaminergiques du mésencéphale humain, suivie d’une coloration immunologique et de la génération de profils phénotypiques neuronaux à partir d’images microscopiques acquises à haut contenu permettant l’identification de variations phénotypiques dues à des modulations génétiques ou chimiques.
Résumé
La maladie de Parkinson (MP) est liée à une série de processus biologiques cellulaires qui provoquent la perte de neurones dopaminergiques du mésencéphale (mDA). De nombreux modèles cellulaires de MP in vitro actuels manquent de complexité et ne prennent pas en compte plusieurs phénotypes. Le profilage phénotypique dans les neurones mDA dérivés de cellules souches pluripotentes induites humaines (iPSC) peut remédier à ces lacunes en mesurant simultanément une gamme de phénotypes neuronaux dans un type de cellule pertinent pour la maladie de Parkinson en parallèle. Nous décrivons ici un protocole permettant d’obtenir et d’analyser des profils phénotypiques à partir de neurones mDA humains disponibles dans le commerce. Un panel de coloration fluorescente spécifique aux neurones est utilisé pour visualiser les phénotypes nucléaires, liés à la α-synucléine, à la tyrosine hydroxylase (TH) et à la protéine associée aux microtubules 2 (MAP2). Le protocole de profilage phénotypique décrit est évolutif car il utilise des plaques à 384 puits, une manipulation automatique des liquides et une microscopie à haut débit. L’utilité du protocole est illustrée par l’utilisation de neurones mDA donneurs sains et de neurones mDA porteurs de la mutation G2019S liée à la maladie de Parkinson dans le gène LRRK2 (Leucine-rich repeat kinase 2). Les deux lignées cellulaires ont été traitées avec l’inhibiteur de la kinase LRRK2 PFE-360 et les changements phénotypiques ont été mesurés. De plus, nous démontrons comment les profils phénotypiques multidimensionnels peuvent être analysés à l’aide de méthodes de classification supervisée basées sur le clustering ou l’apprentissage automatique. Le protocole décrit intéressera particulièrement les chercheurs travaillant sur la modélisation des maladies neuronales ou l’étude des effets des composés chimiques dans les neurones humains.
Introduction
Divers processus biologiques cellulaires sont perturbés dans la maladie de Parkinson (MP). Par exemple, un dysfonctionnement mitochondrial, un stress oxydatif, des défauts de dégradation des protéines, une perturbation du trafic vésiculaire et de la fonction endolysosomale ont été associés à une perte de neurones dopaminergiques du mésencéphale (mDA) sont couramment observés dans la MP1. Par conséquent, la maladie de Parkinson semble impliquer de multiples mécanismes pathologiques qui peuvent interagir et s’aggraver mutuellement. Un moyen utile d’étudier cette interaction mécanistique est la création d’une empreinte phénotypique complète ou d’un profil des neurones dopaminergiques du mésencéphale (mDA).
Le profilage phénotypique est une approche qui consiste à créer un profil d’échantillon à partir d’un ensemble de caractéristiques mesurables, et deuxièmement, à faire des prédictions sur un échantillon en fonction de ce profil 2,3. L’objectif du profilage est de saisir un large éventail de caractéristiques, dont certaines n’ont peut-être pas été associées auparavant à une maladie ou à un traitement3. Par conséquent, le profilage peut révéler des processus biologiques inattendus. Le profilage phénotypique repose généralement sur des cellules colorées par fluorescence, et des tests standardisés, tels que la peinture cellulaire, ont été développés pour créer des profils phénotypiques4. Récemment, le profilage phénotypique a, par exemple, été appliqué pour la caractérisation de petites molécules ou la prédiction précise de sous-types de MP uniquement sur la base de fibroblastes dérivés de patients 5,6. Malgré ces avancées, le profilage phénotypique a rarement été appliqué aux cellules différenciées post-mitotiques, telles que les neurones mDA dérivés de cellules souches pluripotentes induites humaines (iPSC) qui expriment des mutations liées à la maladie de Parkinson telles que LRRK2 G2019S. Les défis importants des modèles dérivés des CSPi comprennent la présence de caractéristiques pathologiques subtiles ou variables entre les lots de différenciation ou les génotypes, et le fait que les phénotypes de MP isolés ne rendent pas compte de toute la complexité de la maladie. De plus, bien que les modèles neuronaux iPSC soient physiologiquement pertinents, ils sont rarement utilisés dans les processus de découverte de médicaments contre la maladie de Parkinson en raison de préoccupations liées à la complexité technique 7,8.
Nous avons précédemment développé une méthodologie robuste pour mesurer plusieurs phénotypes physiopathologiques liés à la maladie de Parkinson dans les neurones mDA humains qui sont à la fois sensibles aux changements phénotypiques induits par les composés génétiques et chimiques9. Cet article décrit en détail une version optimisée de cette méthodologie pour créer des profils phénotypiques à partir de neurones mDA (Figure 1). Ce protocole présente plusieurs avantages par rapport aux approches de profilage phénotypique décrites précédemment, tels que l’utilisation de neurones mDA de haute qualité et la reproductibilité technique. Pour la première fois, ce protocole permet un profilage phénotypique dans les neurones mDA post-mitotiques physiologiquement pertinents après des perturbations chimiques de manière hautement évolutive. Des neurones mDA entièrement différenciés et cryoconservés sont disponibles dans le commerce, ce qui réduit considérablement la variabilité de la différenciation d’un lot à l’autre. Deuxièmement, la variabilité technique peut être encore réduite en utilisant un plan expérimental bien défini (c’est-à-dire la durée de la culture ou l’évitement des puits de bord), la manipulation automatisée des liquides et la microscopie automatisée. De plus, les étapes initiales de l’analyse du profil phénotypique à l’aide d’approches de classification ou de classification supervisées non supervisées sont décrites ici, indiquant comment les données de profilage phénotypique peuvent être analysées. Ce protocole sera utile aux chercheurs intéressés par les modifications phénotypiques des neurones mDA induites par des perturbations génétiques ou chimiques, en particulier lorsqu’un dispositif d’étude hautement évolutif est nécessaire, par exemple lors de campagnes de criblage ou lorsque les effets d’un plus petit nombre de composés doivent être étudiés, par exemple, pour déterminer les effets toxiques. En résumé, on s’attend à ce que l’application du profilage phénotypique des neurones humains soit une technique précieuse pour étudier les phénotypes complexes liés à des maladies et caractériser les effets cellulaires des candidats-médicaments.
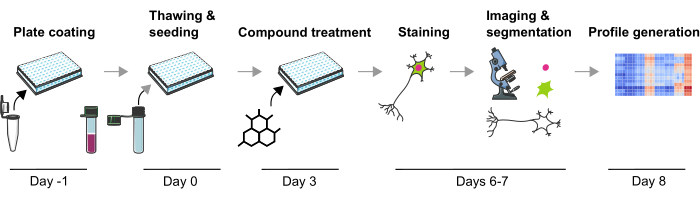
Figure 1 : Représentation schématique du protocole expérimental de génération de profils phénotypiques basés sur l’image à partir de neurones mDA humains dérivés de CSPi. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Protocole
1. Préparation du milieu et des plaques pour l’ensemencement des neurones (Jour 1)
- Pour préparer les plaques pour l’ensemencement des neurones au jour 1, réchauffez la laminine à température ambiante (RT) juste avant l’utilisation. Préparer la solution de laminine en diluant la solution mère de laminine (0,1 mg/mL) 1/10 dans du PBS+/+ froid (avec Ca 2+ et Mg2+).
REMARQUE : Tous les réactifs sont répertoriés dans le tableau des matériaux. Les compositions des solutions et des tampons sont décrites dans les tableaux 1 à 4. - Ensuite, ajoutez 25 μL de la solution de laminine dans chaque puits d’une plaque pré-enduite de Poly-D-Lysine (PDL) à 384 puits, et incubez pendant la nuit à 4 °C. Les plaques revêtues peuvent être conservées à 4 °C jusqu’à une semaine.
REMARQUE : Le protocole peut être mis en pause ici jusqu’à une semaine. Scellez les plaques à l’aide d’un film plastique. - Préparez le support d’entretien complet et conservez-le à 4 °C pendant un mois maximum (tableau 1).
2. Décongélation des neurones (Jour 0)
- Pour décongeler les neurones le jour 0, préchauffer un bain-marie à 37 °C, et équilibrer le support de maintenance complet à RT, à l’abri de la lumière.
- Sortez le flacon contenant les neurones congelés obtenus dans le commerce (voir le tableau des matériaux) du réservoir d’azote liquide et placez-le sur de la glace sèche. Placez ensuite le flacon dans le bain-marie pendant 2 min. Une fois le liquide complètement décongelé, désinfectez le flacon avec de l’éthanol à 70 %.
- Aspirer les neurones décongelés à l’aide d’une pipette P1000 (environ 370 μL) et les transférer dans un tube à centrifuger de 50 mL sans pipetage de haut en bas. Ensuite, rincez le flacon avec 630 μL de produit d’entretien complet, distribuez goutte à goutte (angle de 45°) dans le tube de 50 mL. Agiter lentement pendant la distribution.
REMARQUE : Il est essentiel de distribuer le milieu lentement, goutte à goutte, pour éviter la rupture osmotique des cellules. Un angle de 45° et une légère agitation minimisent la pression osmotique locale élevée pendant le pipetage. - De même, ajoutez 1 mL de milieu d’entretien complet dans le tube à centrifuger de 50 mL à l’aide d’une pipette P1000. Ensuite, ajoutez lentement 2 mL de Milieu d’entretien complet. Agiter soigneusement pendant la distribution.
- Comptez les cellules. Préparez un microtube avec 10 μL de Trypan Blue et 10 μL de suspension cellulaire et ajoutez-le à une lame de chambre de comptage (10 μL). Effectuez le comptage à l’aide d’un compteur de cellules automatisé (voir le tableau des matériaux) ou manuellement.
- Après le comptage, centrifuger le tube de 50 mL contenant les neurones à 400 x g pendant 5 min à RT, et retirer le surnageant. Remettre délicatement la pastille en suspension à l’aide d’une pipette P1000 et de 1 mL de milieu d’entretien complet. Ajoutez ensuite le volume nécessaire pour atteindre la concentration souhaitée (300 000 cellules/mL, voir aussi l’étape suivante).
3. Ensemencement des neurones sur des plaques préparées (Jour 0)
- Pour ensemencer les neurones sur les plaques préparées le jour 0, sortez les plaques enrobées du réfrigérateur, placez-les sous la hotte de culture cellulaire et laissez-les s’équilibrer à RT pendant environ 30 minutes.
- Juste avant l’ensemencement, aspirer une solution d’enrobage de 15 μL à l’aide d’un manipulateur de liquide automatisé ou d’une pipette à 16 canaux. Laisser environ 10 μL par puits pour éviter d’endommager le revêtement.
- Ensuite, distribuez 50 μL de la solution cellulaire contenant 300 000 cellules/mL (préparée à l’étape 2.6) par puits avec une pipette à 16 canaux, ce qui donne 15 000 neurones ensemencés par puits et un volume final par puits de 60 μL.
- Dans une plaque à 384 puits, évitez d’utiliser les colonnes 1, 2, 23, 24 et les lignes A, B, O et P afin de minimiser les effets de bord possibles qui peuvent avoir un impact sur les profils phénotypiques. Remplissez les puits vides inutilisés avec 80 μL de PBS. Incuber les plaques à 37 °C et 5% de CO2.
4. Changement moyen ou traitement composé (Jour 3)
- En fonction du nombre de puits, préchauffer un volume approprié de milieu d’entretien complet à RT. Protéger de la lumière.
- Si un changement de support est nécessaire, passez à l’étape 4.4. Si un traitement du composé est souhaité, vérifier la concentration du composé et le solvant utilisé (eau, DMSO, méthanol, etc.).
- Préparez une solution composée concentrée 1,5 fois pour toutes les concentrations souhaitées à tester. Préparez les dilutions du composé à l’aide du milieu d’entretien complet.
REMARQUE : Comme témoin neutre, utilisez le solvant respectif à la même concentration que le composé testé. Si des composés multiples ou inconnus à des concentrations différentes sont utilisés, il est conseillé d’effectuer une expérience dose-réponse distincte pour mesurer les effets du solvant sur le profil phénotypique. - Si un système de pipetage automatique est utilisé, ajouter 60 μL de la solution composée 1,5x dans une plaque de stockage à 384 puits. Vous pouvez également utiliser une pipette à 16 canaux. Si un changement de milieu est nécessaire, ajoutez plutôt 60 μL de milieu d’entretien complet.
- À l’aide du système de pipetage automatique, aspirer et éliminer 40 μL de milieu par puits de la plaque contenant les neurones pour conserver 20 μL/puits. Ajoutez ensuite 40 μL/puits de solution composée 1,5x de la plaque de stockage à 384 puits dans chaque puits pour obtenir la concentration finale souhaitée.
REMARQUE : Il est essentiel de ne pas effectuer de changements complets de milieu, mais de toujours laisser le milieu résiduel dans le puits pour éviter d’endommager le tapis neuronal ou le revêtement. - Dans le cas où la culture neuronale est effectuée pendant plus de 6 jours décrits dans ce protocole, changer de milieu tous les 2-3 jours.
5. Fixation et coloration des neurones (jours 6 à 7)
- Pour fixer et colorer les neurones aux jours 6 et 7, utilisez un manipulateur de liquide automatisé pour toutes les étapes de distribution et de lavage. Vous pouvez également utiliser une pipette à 16 canaux.
- Préparez une solution de Triton X-100 à 10 % en diluant Triton X-100 dans 1x PBS. Vortex jusqu’à ce que la solution soit homogène. Conserver à 4 °C.
- Pour fixer les neurones, distribuer 20 μL/puits de PFA à 16 %, ce qui donne une concentration finale de 4 %. Incubez l’assiette pendant 30 min à RT et lavez-la trois fois avec 1x PBS. Laisser 20 μL/puits de PBS après le dernier lavage.
ATTENTION : Le PFA est reconnu comme une substance dangereuse connue pour provoquer une toxicité orale, cutanée et respiratoire. Il constitue également une menace pour les yeux et peut entraîner des mutations génétiques et le cancer. Une bonne manipulation de l’APF nécessite l’utilisation d’équipements de protection individuelle appropriés, tels que la protection des yeux et du visage, en plus d’assurer une ventilation adéquate. Il est important d’empêcher le rejet de PFA dans l’environnement. - Pour la perméabilisation et le blocage, préparez une solution de blocage 2x (tableau 2).
- Ajouter 20 μL/puits de solution de blocage 2x (1x concentration finale), incuber pendant 1 h à RT et laver une fois avec du PBS. Conserver 20 μL/puits de PBS après le lavage.
- Pour la coloration primaire par anticorps (voir le tableau des matériaux), préparez un tampon de coloration primaire 2x (tableau 3).
- Ajouter 20 μL/puits de 2x tampon de coloration primaire (1x concentration finale) et incuber toute la nuit à 4 °C. Le lendemain matin, le jour 7, lavez-vous trois fois avec PBS. Laisser 20 μL/puits de PBS après le lavage.
- Pour la coloration par anticorps secondaire (voir le tableau des matériaux), préparez un tampon de coloration secondaire 2x (tableau 4).
- Ajouter 20 μL/puits de 2x tampon de coloration secondaire (1x concentration finale), incuber pendant 2 h à RT à l’abri de la lumière et laver trois fois avec du PBS. Laisser 100 μL PBS/puits après le dernier lavage.
- Ajoutez un joint d’étanchéité en aluminium sur la plaque pour minimiser l’évaporation. Vous pouvez également recouvrir l’assiette d’un film plastique et d’une feuille d’aluminium. Procédez à l’acquisition de l’image ou stockez la plaque à 4 °C.
REMARQUE : Le protocole peut être mis en pause ici jusqu’à une semaine. Si une fluorescence de fond est observée, envisagez d’augmenter le temps de blocage. Si la coloration est insuffisante, essayez d’augmenter la concentration d’anticorps primaires ou le temps d’incubation.
- Ajoutez un joint d’étanchéité en aluminium sur la plaque pour minimiser l’évaporation. Vous pouvez également recouvrir l’assiette d’un film plastique et d’une feuille d’aluminium. Procédez à l’acquisition de l’image ou stockez la plaque à 4 °C.
6. Imagerie de neurones colorés par fluorescence (Jour 7)
- Acquérez des images des neurones plaqués, cultivés et colorés au jour 7. Idéalement, utilisez un microscope à fluorescence confocale automatisé (voir le tableau des matériaux). Vous pouvez également acquérir des images manuellement.
- Acquérir les canaux Hoechst, TH, α-synucléine et MAP2 à l’aide de lasers de 405 nm, 488 nm, 561 nm et 647 nm, respectivement (Figure 2).
REMARQUE : Pour générer une quantité suffisante de données détaillées pour le profilage phénotypique, utilisez un objectif 40x et acquérez 16 champs/puits à l’aide d’empilements Z constitués de 3 tranches Z séparées par 2 μm. Dans le cas où il y a des dommages liés au pipetage dans le tapis neuronal, essayez d’éviter d’imager ces zones. - En fonction du microscope et de l’appareil photo, ajustez séparément les temps d’exposition et les intensités d’excitation pour chacun des quatre canaux fluorescents afin d’obtenir une plage dynamique optimale des intensités fluorescentes.
REMARQUE : Les logiciels d’imagerie fournissent souvent un histogramme pour déterminer le temps d’exposition idéal. Si l’histogramme est trop décalé vers la gauche dans la plage de signal faible, le temps d’exposition est trop court ou l’intensité de l’excitation est trop faible. S’il y a une falaise abrupte au niveau maximal du signal sur la droite, alors la valeur du signal est saturée. Dans ce cas, réduisez l’intensité de l’excitation ou raccourcissez le temps d’exposition. - Stockez les images dans un format ouvert et sans perte, tel que .tif.
7. Traitement de l’image (Jour 8)
- La segmentation d’images et l’extraction de caractéristiques phénotypiques sont nécessaires à la création de profils phénotypiques quantitatifs. Utilisez le logiciel PhenoLink pour la segmentation d’images et l’extraction de caractéristiques (Table des matériaux). Les instructions d’installation de PhenoLink se trouvent dans le dépôt GitHub (https://github.com/Ksilink/PhenoLink).
REMARQUE : La segmentation d’image est utilisée pour identifier et séparer différents objets ou régions d’une image, tandis que l’extraction de caractéristiques phénotypiques est utilisée pour analyser et extraire des informations pertinentes de ces régions. Plusieurs solutions logicielles alternatives, telles que CellProfiler10, ImageJ/FIJI 11, Napari 12 ou Knime Analytics Platform13 sont disponibles pour extraire des informations quantitatives à partir d’images de fluorescence multicanaux. - Effectuez une segmentation d’image sur des images brutes corrigées de l’éclairage. Déterminez empiriquement les seuils d’intensité des canaux fluorescents respectifs par plaque afin que le signal de fond soit minimal et que le signal segmenté souhaité corresponde au signal de l’image brute. Les plaques traitées et colorées le même jour nécessitent généralement des seuils d’intensité de canal comparables pour la segmentation.
- Définissez la taille et l’intensité du noyau pour séparer les cellules vivantes des cellules mortes. Lorsque vous utilisez des images 40x, conservez tous les autres paramètres par défaut et exécutez le logiciel. Cent vingt-six caractéristiques quantitatives de l’image seront calculées par puits (tableau supplémentaire 1).
- Utilisez les données quantitatives tabulaires qui en résultent pour construire des profils phénotypiques et comparer des profils phénotypiques de différentes lignées cellulaires ou conditions de traitement. Chaque ligne correspond à une condition biologique (puits) et chaque colonne correspond à une caractéristique phénotypique déterminée.
REMARQUE : Nous fournissons un exemple de fichier de sortie avec le pipeline d’analyse des données pour illustrer son utilisation (voir le tableau des matériaux). De plus, la figure 3 montre la composition d’un profil phénotypique.
8. Génération et visualisation de profils phénotypiques (Jour 8)
- Si Python et Jupyter ne sont pas installés sur votre ordinateur, installez la distribution Anaconda et ouvrez le logiciel Jupyter. Téléchargez le bloc-notes Jupyter fourni et tous les autres fichiers fournis et enregistrez-les dans le même répertoire (voir Table des matériaux). Ouvrez le fichier de bloc-notes Jupyter à l’aide du logiciel Jupyter.
REMARQUE : Anaconda est une plate-forme gratuite et open-source pour les langages de programmation tels que Python. Cette plate-forme est livrée avec l’interpréteur Python Jupyter qui peut exécuter le notebook Jupyter fourni pour créer et analyser des profils phénotypiques (https://github.com/Ksilink/Notebooks/tree/main/Neuro/DopaNeuronProfiling). - Exécutez le bloc-notes Jupyter cellule par cellule à l’aide du logiciel Jupyter. Les fichiers .fth et .txt des exemples de données fournis doivent se trouver dans le même répertoire que le bloc-notes Jupyter. Chaque cellule de bloc-notes Jupyter est annotée pour expliquer ses fonctionnalités.
REMARQUE : Il est essentiel d’utiliser le bloc-notes Jupyter dans le bon ordre, en commençant par le chargement et la mise à l’échelle des données, puis de procéder cellule par cellule jusqu’à la fin. Toutes les données et les graphiques de sortie seront stockés dans un dossier nouvellement créé dans le répertoire source du bloc-notes Jupyter. La figure 4 illustre le flux de travail et la sortie du flux de travail.
Résultats
Le profilage phénotypique dans les neurones mDA est un moyen efficace de quantifier de multiples aspects de la biologie cellulaire et leurs changements au cours de la modulation expérimentale. Pour illustrer cette méthodologie, cette étude a utilisé des neurones LRRK2 G2019S cryoconservés et des neurones mDA de donneurs sains. Ces neurones sont différenciés depuis environ 37 jours, sont des marqueurs neuronaux post-mitotiques et express (TUBB3 et MAP2) et des neurones dopaminergiques, y compris la tyrosine hydrox...
Discussion
Le profilage phénotypique est une technique permettant de mesurer un grand nombre de phénotypes dans les cellules en appliquant des colorations fluorescentes, la microscopie et l’analyse d’images3. Des profils phénotypiques peuvent être obtenus et comparés entre des lignées cellulaires ou d’autres conditions expérimentales pour comprendre des changements complexes dans la biologie cellulaire qui pourraient passer inaperçus lors de l’utilisation d’une seule lecture. Nous décrivon...
Déclarations de divulgation
Tous les auteurs sont employés par Ksilink.
Remerciements
Les auteurs tiennent à remercier tous les collègues de Ksilink pour leur aide précieuse et les discussions qui ont conduit à la conception du protocole présenté.
matériels
| Name | Company | Catalog Number | Comments |
| Anti- chicken – Alexa 647 | Jackson ImmunoRearch | 703-605-155 | Immunofluorescence |
| Anaconda | https://www.anaconda.com/download | ||
| Anti-Map2 | Novus | NB300-213 | Immunofluorescence |
| Anti-mouse - Alexa 488 | Thermo Fisher | A11001 | Immunofluorescence |
| Anti-rabbit - Alexa 555 | Thermo Fisher | A21429 | Immunofluorescence |
| Anti-Tyrosine Hydroxylase | Merck | T2928 | Immunofluorescence |
| Anti-α-synuclein | Abcam | 138501 | Immunofluorescence |
| Bravo Automated Liquid Handling Platform with 384ST head | Agilent | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Confocal microscope | Yokogawa | CV7000 | The use of an automated confocal fluorescence microscope is recommended to ensure image quality consistency. |
| Countess Automated cell counter | Invitrogen | Cell counting before seeding. Can also be done using a manual counting chamber. | |
| DPBS +/+ | Gibco | 14040-133 | Buffer for washing |
| EL406 Washer Dispenser | BioTek (Agilent) | If no liquid handler is available, the use of an electronic multichannel pipette is recommended. | |
| Formaldehyde Solution (PFA 16 %) | Euromedex | EM-15710-S | Fixation before staining |
| Hoechst 33342 | Invitrogen | H3570 | Nuclear staining |
| iCell Base Medium 1 | Fujifilm | M1010 | Base medium for neurons |
| iCell DPN, Donor#01279, Phenotype AHN, lot#106339, 1M | Fujifilm | C1087 | Apparently healthy donor |
| iCell DPN, Donor#11299, Phenotype LRRK2 G2019S, phenotype PD lot#106139 | Fujifilm | C1149 | Donor carrying LRRK2 G2019S mutation |
| iCell Nervous System Supplement | Fujifilm | M1031 | Supplement for base medium |
| iCell Neural Supplement B | Fujifilm | M1029 | Supplement for base medium |
| Jupyter Python Notebook | In-house development | https://github.com/Ksilink/Notebooks/tree/main/Neuro/DopaNeuronProfiling | Notebook to perform phenotypic profile visualization and classification from raw data. |
| Laminin | Biolamina | LN521 | Plate coating |
| PFE-360 | MedChemExpress | HY-120085 | LRRK2 kinase inhibitor |
| PhenoLink | In-house development | https://github.com/Ksilink/PhenoLink | Software for image analysis |
| PhenoPlate 384w, PDL coated | Perkin Elmer | 6057500 | Pre-coated plate for cell culture and imaging. This plate allows imaging of all wells using all objectives of the Yokogawa CV7000 microscope. |
| Storage plates Abgene 120 µL | Thermo Scientific | AB-0781 | Necessary for compound dispensing using the Vprep pipetting system. If not available, the use of an electronic multichannel pipette is recommended. |
| Triton | Sigma | T9284 | Permeabilization before lysis |
| Trypan Blue | Sigma | T8154-20ML | Determination of living cells |
| Vprep Pipetting System | Agilent | Medium change and compound dispensing. Alternatively, an electronic multichannel pipette can be used. |
Références
- Panicker, N., Ge, P., Dawson, V. L., Dawson, T. M. The cell biology of Parkinson's disease. The Journal of Cell Biology. 220 (4), 202012095 (2021).
- Caicedo, J. C., et al. Data-analysis strategies for image-based cell profiling. Nature Methods. 14 (9), 849-863 (2017).
- Chandrasekaran, S. N., Ceulemans, H., Boyd, J. D., Carpenter, A. E. Image-based profiling for drug discovery: due for a machine-learning upgrade. Nature Reviews Drug Discovery. 20 (2), 145-159 (2021).
- Bray, M. -. A., et al. Cell Painting, a high-content image-based assay for morphological profiling using multiplexed fluorescent dyes. Nature Protocols. 11 (9), 1757-1774 (2016).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Ziegler, S., Sievers, S., Waldmann, H. Morphological profiling of small molecules. Cell Chemical Biology. 28 (3), 300-319 (2021).
- Cobb, M. M., Ravisankar, A., Skibinski, G., Finkbeiner, S. iPS cells in the study of PD molecular pathogenesis. Cell and Tissue Research. 373 (1), 61-77 (2018).
- Elitt, M. S., Barbar, L., Tesar, P. J. Drug screening for human genetic diseases using iPSC models. Human Molecular Genetics. 27 (R2), 89-98 (2018).
- Vuidel, A., et al. High-content phenotyping of Parkinson's disease patient stem cell-derived midbrain dopaminergic neurons using machine learning classification. Stem Cell Reports. 17 (10), 2349-2364 (2022).
- Stirling, D. R., Swain-Bowden, M. J., Lucas, A. M., Carpenter, A. E., Cimini, B. A., Goodman, A. CellProfiler 4: improvements in speed, utility and usability. BMC Bioinformatics. 22 (1), 433 (2021).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Sofroniew, N., et al. . napari: a multi-dimensional image viewer for Python. , (2022).
- Berthold, M. R., et al. KNIME: The Konstanz Information Miner. Data Analysis, Machine Learning and Applications. , 319-326 (2008).
- Fathi, A., et al. Diverging Parkinson's Disease Pathology between patient-derived GBAN370S, LRRK2G2019S and engineered SNCAA53T iPSC-derived Dopaminergic Neurons. bioRxiv. , (2023).
- Wang, Y., Huang, H., Rudin, C., Shaposhnik, Y. Understanding How Dimension Reduction Tools Work: An Empirical Approach to Deciphering t-SNE, UMAP, TriMap, and PaCMAP for Data Visualization. Journal of Machine Learning Research. 22 (201), 1-73 (2021).
- Ke, G., et al. LightGBM: A highly efficient gradient boosting decision tree. Advances in Neural Information Processing Systems. 30, (2017).
- Avazzadeh, S., Baena, J. M., Keighron, C., Feller-Sanchez, Y., Quinlan, L. R. Modelling Parkinson's Disease: iPSCs towards Better Understanding of Human Pathology. Brain Sciences. 11 (3), 373 (2021).
- Sánchez-Danés, A., et al. Disease-specific phenotypes in dopamine neurons from human iPS-based models of genetic and sporadic Parkinson's disease. EMBO Molecular Medicine. 4 (5), 380-395 (2012).
- Oosterveen, T., et al. Pluripotent stem cell derived dopaminergic subpopulations model the selective neuron degeneration in Parkinson's disease. Stem Cell Reports. 16 (11), 2718-2735 (2021).
- Hughes, R. E., et al. Multiparametric high-content cell painting identifies copper ionophores as selective modulators of esophageal cancer phenotypes. ACS Chemical Biology. 17 (7), 1876-1889 (2022).
- Akbarzadeh, M., et al. Morphological profiling by means of the Cell Painting assay enables identification of tubulin-targeting compounds. Cell Chemical Biology. 29 (6), 1053-1064 (2022).
- Schiff, L., et al. Integrating deep learning and unbiased automated high-content screening to identify complex disease signatures in human fibroblasts. Nature Communications. 13 (1), 1590 (2022).
- Way, G. P., et al. Morphology and gene expression profiling provide complementary information for mapping cell state. Cell Systems. 13 (11), 911-923 (2022).
- Feng, Y., Mitchison, T. J., Bender, A., Young, D. W., Tallarico, J. A. Multi-parameter phenotypic profiling: using cellular effects to characterize small-molecule compounds. Nature Reviews Drug Discovery. 8 (7), 567-578 (2009).
- Antonov, S. A., Novosadova, E. V. Current state-of-the-art and unresolved problems in using human induced pluripotent stem cell-derived dopamine neurons for parkinson's disease drug development. International Journal of Molecular Sciences. 22 (7), 3381 (2021).
- Miller, J. D., et al. Human iPSC-based modeling of late-onset disease via progerin-induced aging. Cell Stem Cell. 13 (6), 691-705 (2013).
- Bezard, E., Gross, C. E., Brotchie, J. M. Presymptomatic compensation in Parkinson's disease is not dopamine-mediated. Trends in Neurosciences. 26 (4), 215-221 (2003).
- Wu, Y., Le, W., Jankovic, J. Preclinical Biomarkers of parkinson disease. Archives of Neurology. 68 (1), 22-30 (2011).
- Verstraelen, P., et al. Systematic quantification of synapses in primary neuronal culture. iScience. 23 (9), 101542 (2020).
- Liu-Yesucevitz, L., et al. ALS-Linked mutations enlarge TDP-43-enriched neuronal RNA granules in the dendritic arbor. The Journal of Neuroscience. 34 (12), 4167-4174 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon