Method Article
Aislamiento, expansión y nucleofección de células madre neurales de la zona subventricular murina adulta
* Estos autores han contribuido por igual
En este artículo
Resumen
Aquí, describimos un sistema de nucleofección diseñado para mejorar la eficiencia de la entrega de genes en células madre neurales expandidas (NSC) aisladas de la zona subventricular murina adulta. Los hallazgos demuestran que este método mejora significativamente la perturbación génica en las NSC, superando la efectividad de los protocolos tradicionales de transfección y mejorando la tasa de supervivencia celular.
Resumen
El aislamiento y la expansión de las células madre neurales (NSC) de la zona subventricular (SVZ) del cerebro de ratón adulto se puede lograr en un medio suplementado con factor de crecimiento básico de fibroblastos (bFGF) y factor de crecimiento epidérmico (EGF) como mitógenos, produciendo agregados clonales conocidos como neuroesferas. Este sistema in vitro es una herramienta valiosa para estudiar el potencial de NSC. La transfección de siRNAs o genes transportados en plásmidos se puede utilizar para inducir perturbaciones en la expresión génica y estudiar la biología de las NSC. Sin embargo, la entrega de ácido nucleico exógeno a los cultivos de NSC es un desafío debido a la baja eficiencia de la transfección de las células del sistema nervioso central (SNC). Aquí, presentamos un sistema de nucleofección mejorado que logra una alta eficiencia de entrega de genes en NSCs expandidas a partir de SVZ murinas adultas. Demostramos que este método relativamente simple mejora la perturbación génica en NSCs adultas, superando los protocolos tradicionales de transfección con tasas de supervivencia superiores al 80%. Además, este método también se puede aplicar en NSCs primarias aisladas, proporcionando un avance crucial en los estudios de la función génica a través de la manipulación de la expresión génica a través de la knockdown o la sobreexpresión en cultivos de neurosfera.
Introducción
Las células madre neurales (NSC, por sus siglas en inglés) son células madre multipotentes que residen en el cerebro. Estas células poseen la capacidad de autorrenovarse y diferenciarse en los tres linajes neuronales: astrocitos, oligodendrocitosy neuronas. En consecuencia, las NSC juegan un papel crucial en la neurogénesis adulta de los mamíferos, un proceso en el que se generan nuevas neuronas en el cerebro2. Las NSC residen predominantemente en dos regiones dentro del cerebro adulto denominadas nichos neurogénicos: la zona subventricular (SVZ) a lo largo de las paredes de los ventrículos laterales y la zona subgranular (SGZ) dentro del giro dentado del hipocampo 3,4. Las NSC (también conocidas como células B) en la SVZ, el nicho neurogénico más activo en el ratón adulto, se autorrenuevan y producen progenitores amplificadores de tránsito (TAP o células C) que posteriormente se diferencian en neuroblastos (células A). Estos neuroblastos migran a través de la corriente migratoria rostral (RMS) a los bulbos olfativos (OB), donde experimentan una diferenciación completa en interneuronas, integrándose en los circuitos preexistentes 3,4,5,6.
Es importante comprender la intrincada interacción de las señales y señales moleculares que regulan las NSC dentro de estos nichos para aprovechar su potencial para aplicaciones terapéuticas. Para ello, se han desarrollado diversos métodos para estudiar esta población celular, que van desde el cultivo primario selectivo de NSCs hasta la selección celular mediante marcadores de superficie 7,8,9,10. El presente manuscrito detalla el aislamiento y cultivo de NSCs SVZ in vitro utilizando un medio selectivo libre de suero que contiene ambos mitógenos: factor de crecimiento básico de fibroblastos (bFGF) y factor de crecimiento epidérmico (EGF). Este medio facilita la proliferación celular y mantiene la stemness de las NSCs obtenidas de la SVZ de cerebros de ratones adultos, formando in vitro agregados clonales, tridimensionales, no adherentes, conocidos como neuroesferas9. Los cultivos de la neurosfera sirven como una plataforma controlada para manipular y estudiar los mecanismos moleculares y los factores que afectan la proliferación, la autorrenovación, la diferenciación y la supervivencia de las NSC11. En particular, el número de neuroesferas primarias formadas en el cultivo permite estimar el número de NSCs presentes en la SVZ in vivo, lo que la convierte en una poderosa herramienta para estudiar los efectos de diferentes condiciones en el grupo de NSC adultas12,13. Además, una vez que se establece el cultivo primario, las NSC pueden generar nuevos agregados (neuroesferas secundarias) al pasar a través de divisiones simétricas en condiciones de proliferación14. Por lo tanto, la siembra de baja densidad de células secundarias de la neurosfera (ensayo clonal) se puede utilizar para evaluar la tasa de autorrenovación de estos cultivos 4,15,16,17.
A pesar del potencial de las neurosferas para descubrir los mecanismos que gobiernan la regulación de las NSC, algunos investigadores cuestionan la validez de los hallazgos in vitro, argumentando que las condiciones artificiales en las que crecen las células podrían no replicar fielmente el intrincado microambiente in vivo de los nichos neurogénicos 18,19,20,21,22. Otro punto controvertido gira en torno a la heterogeneidad observada en las neurosferas. No obstante, se cree que esta variabilidad refleja las divisiones simétricas y asimétricas de las NSC que ocurren naturalmente in vivo23,24. Además, la validación reciente ha respaldado la utilización de cultivos de NSC para predecir los mecanismos que operan dentro del nicho neurogénico de SVZ in vivo, y varios estudios han demostrado que las NSC cultivadas in vitro mantienen con precisión el perfil transcriptómico observado in vivo11,25.
Por lo tanto, los cultivos de neurosfera no solo sirven como un método para explorar las capacidades de proliferación y diferenciación de NSC, sino que también ofrecen un sistema para estudiar la influencia de los genes que gobiernan la biología de NSC. Una técnica fundamental para investigar la función génica en las NSC es la perturbación de la expresión génica. Los siRNAs o genes entregados a través de plásmidos se pueden transfectar en cultivos celulares, lo que resulta en la eliminación o la regulación positiva del gen objetivo. Este enfoque versátil reduce significativamente el tiempo y el costo en comparación con el establecimiento de cultivos con ratones knockout condicionales, lo que presenta una vía prometedora para desentrañar las bases genéticas de la neurogénesis y explorar perspectivas terapéuticas. La alteración de la expresión de genes específicos en las NSC permite la modulación de su comportamiento, influyendo en procesos biológicos cruciales como la proliferación, la diferenciación y la migración. Sin embargo, la perspectiva de transfectar NSCs, particularmente dentro de las neuroesferas de ratones, presenta desafíos notables. La estructura tridimensional de las neurosferas compromete la eficiencia de la transfección, lo que a menudo resulta en bajas tasas de administración exitosa de ácidos nucleicos exógenos, lo que limita el alcance de la manipulación genética26,27. Además, los procedimientos de transfección pueden afectar negativamente la viabilidad y funcionalidad celular27. En este contexto, presentamos el sistema de nucleofección como un método para mitigar el daño celular, logrando una alta tasa de supervivencia y asegurando una mayor eficacia en los ensayos de entrega de genes utilizados para perturbar cultivos de NSC.
Este manuscrito tiene como objetivo ilustrar el procedimiento para aislar, expandir y nucleoafectar NSCs del nicho neurogénico SVZ adulto para perturbar genes utilizando el sistema de cultivo de neurosfera. Este método supera la efectividad de los protocolos de transfección tradicionales, presentando tasas de supervivencia significativamente más altas y una mayor eficiencia en la entrega de genes entre las células objetivo.
Protocolo
Todos los experimentos realizados con animales fueron previamente aprobados por el Comité Ético de la Universitat de València y autorizados por la Conselleria de Agricultura, Ganadería y Pesca, Generalitat Valenciana (España).
1. Cultura primaria de las neuroesferas
- Preparación de reactivos
- Prepare una solución tampón de tampón de fosfato salino (DPBS) de Dulbecco en agua desionizada y esterilicela en autoclave. Alternativamente, prepare 0,1 M de PBS pH 7,4 añadiendo 137 mM de NaCl, 2,7 mM de KCl, 10 mM deNa2 HPO4 y 1,8 mM de solución de KH2PO4 en agua desionizada.
- Llene dos placas de 12 pocillos con DPBS previamente enfriados y manténgalas en hielo: una se utilizará para preservar todo el cerebro antes de la disección, y la otra conservará las SVZ disecadas hasta el procesamiento del tejido.
- Prepare la mezcla enzimática que contenga papaína como se describe a continuación.
- Prepare la solución de disociación de EDTA/L-cisteína en una solución salina equilibrada de Earl's (EBSS) hasta una concentración final de 0,2 mg/mL cada una. Para lograr la disolución completa, incubar el tubo en un baño de agua a 37 °C.
- Disuelva 12 U/mL de papaína en una solución de EDTA/L-cisteína. Hay que tener en cuenta que se utilizarán 500 μL de solución enzimática por cerebro.
- Introducir la solución de papaína en un baño de agua a 37 °C durante unos 20 minutos hasta que la enzima se disuelva completamente.
- Filtrar-esterilizar la mezcla enzimática con una jeringa y un filtro de poros de 0,22 μm y mantenerla a 4 °C hasta su uso.
NOTA: La papaína se activa después de 30 minutos a 37 °C y se puede almacenar durante 12 h a 4 °C.
- Preparar el medio de cultivo de control añadiendo la solución de mezcla de hormonas de acuerdo con la Tabla 1 y filtrar-esterilizar con filtros de nitrocelulosa de 0,22 μm de poro. Mantenga el medio a 4 °C y caliéntelo en un baño de agua a 37 °C antes de usarlo.
- Prepare el medio completo complementando el medio de control con EGF y bFGF justo antes de usarlo como se indica en la Tabla 1. No filtre el medio completo, ya que los mitógenos pueden perderse con el filtrado.
- Extracción de cerebro y disección de SVZ
NOTA: A los ratones se les permite el libre acceso a alimentos y agua antes de la eutanasia. Se utilizan ratones C57BL/6 de 8 a 16 semanas de edad. Se utilizan ambos sexos sin diferencias notables en la preparación.- Esterilice las herramientas de disección esterilizándolas en autoclave antes de la cirugía. Luego, sumerja bisturíes, pinzas, tijeras y espátulas en un vaso de precipitados lleno de etanol al 70%. Limpie a fondo todas las superficies de trabajo con etanol al 70%.
- Sacrificar el ratón por sobredosis de CO2 y confirmar por luxación cervical. Rocíe la cabeza con etanol al 70% para minimizar la posibilidad de contaminación del tejido cerebral.
- Separa la cabeza del ratón del resto del cuerpo con unas tijeras. Corta la piel por encima del cráneo con unas tijeras pequeñas a lo largo del eje rostro-caudal hasta que el cráneo quede completamente descubierto.
- Exponga el cerebro cortando inicialmente el cráneo a lo largo de la sutura sagital con tijeras pequeñas y luego, retire los huesos con pinzas finas. Tenga cuidado de no causar ningún daño al tejido cerebral debajo del cráneo durante este paso.
- Extraiga cuidadosamente el cerebro del cráneo con una espátula y colóquelo en una placa de 12 pocillos que contenga DPBS frío. Mantenga la placa en hielo hasta que se complete la disección.
- Transfiera todo el cerebro a la almohadilla de silicona donde se llevará a cabo la disección y aplique unas gotas de DPBS frío sobre el cerebro (Figura 1A).
- Utilice un bisturí para extraer el OB situado en el extremo rostral del cerebro y el cerebelo situado en la parte caudal, conservando la porción central del cerebro que contiene ambos ventrículos laterales (Figura 1B).
- Divida el cerebro a lo largo de la fisura longitudinal en dos hemisferios y luego proceda con la disección de ambos hemisferios por separado (Figura 1C).
- Trabajando con un hemisferio, reoriéntelo de modo que el área medial mire hacia arriba. Abrir el cerebro a lo largo de la línea del cuerpo calloso, separando la corteza y el cuerpo estriado del hipocampo, el tabique y el diencéfalo, exponiendo así los ventrículos laterales (Figura 1D).
NOTA: La SVZ está situada entre la cavidad del ventrículo y el cuerpo estriado, por lo que los pasos posteriores tienen como objetivo aislar la SVZ con la mayor precisión posible del tejido circundante. - Extirpar el hipocampo, el tabique y el diencéfalo siguiendo el límite ventral de los ventrículos (Figura 1E, F), el tejido más allá de los extremos rostral y caudal de la SVZ (Figura 1G) y la corteza siguiendo el cuerpo calloso (Figura 1H, I).
- Incline el tejido de modo que la SVZ mire hacia los lados (Figura 1J), lo que permite la extracción del tejido estriado debajo de la SVZ (Figura 1K) para obtener una pieza delgada de tejido que contiene las NSC (Figura 1L).
- Guarde ambos SVZ disecados del mismo ratón en una placa de 12 pocillos que contenga DPBS fríos y estériles hasta que comience el procesamiento del tejido.
NOTA: La disección de ambas SVZ de un cerebro debe durar 10 minutos. Un tiempo más prolongado puede afectar a la viabilidad de la célula. Si se necesita un mayor rendimiento de los cultivos, se pueden agrupar SVZ de varios ratones de la misma edad, sexo, antecedentes genéticos y genotipo.
- Aislamiento y siembra de NSCs
- Cortar cada SVZ en 4-5 trozos pequeños para facilitar la desagregación del tejido.
NOTA: A partir de este paso, todo el procedimiento debe realizarse en condiciones estériles en una campana de flujo laminar. - Transfiera las SVZ picadas a un tubo de centrífuga de 15 ml con una pipeta Pasteur de plástico estéril. Asegúrate de que no queden fragmentos en el plato.
- Deje que el tejido se sedimente en el fondo del tubo y elimine los DPBS restantes.
- Añadir 500 μL de mezcla enzimática filtrada que contenga papaína por cerebro (2 SVZs) e incubar los tubos en un baño de agua a 37 °C durante 30 min.
- Después de la incubación, añadir 5 mL de medio de control precalentado a 37 °C a cada muestra.
- Centrifugar las muestras a 300 x g durante 5 min. A continuación, retire con cuidado el sobrenadante con una micropipeta o una bomba de vacío.
- Añadir 1 mL de medio de control y desagregar cuidadosamente el pellet, pipeteando hacia arriba y hacia abajo de 10x a 20x, utilizando una pipeta de vidrio Pasteur pulida al fuego hasta obtener una suspensión celular homogénea.
PRECAUCIÓN: Este es uno de los pasos más críticos del protocolo, ya que una desagregación excesiva o insuficiente afectará negativamente el rendimiento de los cultivos. - Añadir 4 mL de medio control y mezclar por inversión para lavar las células. Centrifugar a 300 x g durante 5 min. A continuación, retira el sobrenadante.
- Agregue 1 mL de medio completo y vuelva a suspender el pellet pipeteando hacia arriba y hacia abajo 10 veces para homogeneizar la suspensión celular.
- Cuente la suspensión celular usando una cámara de Neubauer bajo el microscopio después de diluir 1/2 alícuota de la suspensión celular con azul de tripán o utilice un contador automático de células. Asegúrese de que los cultivos estén desagregados a nivel de una sola célula.
- Una vez preparada la suspensión celular, se siembran las células obtenidas en partes iguales en 8 pocillos de una placa de 48 pocillos en un volumen final de 500 μL de medio completo.
NOTA: Por lo general, obtenemos 20,000 celdas de dos SVZ, lo que resulta en una densidad de siembra final de aproximadamente 2,500 celdas/mL para lograr el máximo rendimiento en cultivos.
- Cortar cada SVZ en 4-5 trozos pequeños para facilitar la desagregación del tejido.
- Formación de neuroesferas
- Incubar las células durante 5 a 7 días in vitro (DIV) en una incubadora humidificada a 37 °C y 5% de CO2. En estas condiciones, las células diferenciadas mueren mientras que las NSC y sus progenitores proliferan en respuesta a la estimulación mitogénica, formando agregados clonales reconocidos como neuroesferas primarias.
NOTA: Se puede utilizar EGF o bFGF para inducir la formación de neurosfera. Sin embargo, estos dos factores de crecimiento no son totalmente sustituibles. - Verifique el tamaño de las neuroesferas usando una cámara acoplada a un microscopio invertido, y una vez que las neuroesferas alcancen un rango de tamaño de 50 a 100 μm, cuente todas las neuroesferas primarias formadas en el pocillo con un aumento de 10x. El número total de neuroesferas primarias formadas sirve como una estimación del número de NSCs presentes en el nicho SVZ.
- Después de un período de 10 días, recoja el medio de cultivo de cada pocillo para comenzar el paseo. Realice el paso posterior después de 5 a 7 DIV, aunque vale la pena señalar que las esferas primarias pueden requerir una duración ligeramente más larga para alcanzar el tamaño ideal para el subcultivo (alrededor de 100 μm).
- Incubar las células durante 5 a 7 días in vitro (DIV) en una incubadora humidificada a 37 °C y 5% de CO2. En estas condiciones, las células diferenciadas mueren mientras que las NSC y sus progenitores proliferan en respuesta a la estimulación mitogénica, formando agregados clonales reconocidos como neuroesferas primarias.
Tabla 1: Soluciones de medios de cultivo. Se describe la receta del medio de control. Se hace una solución de mezcla de hormonas premezclada. Las soluciones madre de hormonas pueden prepararse con antelación según las indicaciones y almacenarse a -20 °C. Antes del cultivo celular, suplementar el medio de control con EGF y bFGF para preparar el medio completo. Se proporcionan especificaciones de almacenamiento, existencias y concentraciones de trabajo para cada componente. Haga clic aquí para descargar esta tabla.
2. Expansión de las culturas de la neurosfera
- Paso de las neurosferas
- Recoja el medio que contiene las neuroesferas de las placas de pocillos múltiples y transfiéralas a un tubo de centrífuga de 15 ml. Asegure la recuperación de tantas células como sea posible enjuagando los pocillos con unos pocos mililitros de DPBS.
- Centrifugar las neuroesferas durante 5 min a 300 x g. A continuación, retira el sobrenadante.
- Agregue 200 μL de solución enzimática (consulte la tabla de materiales) al pellet y golpee suavemente el fondo del tubo para desalojarlo. Incubar durante 10 min a temperatura ambiente para facilitar la disociación de las neurosferas.
- Añada 1 ml de medio de control para detener la reacción enzimática y disociar mecánicamente las neuroesferas con una micropipeta P1000 pipeteando hacia arriba y hacia abajo de 10x a 20x.
- Añadir un volumen adicional de 4 mL del medio control y mezclar por inversión para lavar las células.
- Centrifugar las células durante 5 min a 300 x g. A continuación, retira el sobrenadante.
- Agregue 1 mL de medio completo y vuelva a suspender el pellet para homogeneizar la suspensión celular pipeteando hacia arriba y hacia abajo 10 veces.
NOTA: Si el número y el tamaño originales de las neuroesferas son demasiado altos, agregue un medio más completo para diluir la suspensión celular y garantizar un recuento celular confiable. - Cuente la suspensión celular usando una cámara de Neubauer después de diluir 1/2 alícuota de la suspensión celular con azul de tripán o utilice un contador automático de celdas.
- Para expandir el cultivo celular, siembre células a una densidad de 10.000 células/cm2 utilizando medio completo en una placa o matraz de cultivo apropiado (ver Tabla 2).
- Incubar las células de 5 a 7 DIV en una incubadora humidificada a 37 °C y 5% de CO2 para cada paso.
NOTA: Los cultivos de la neurosfera pueden expandirse y propagarse consistentemente a través de subcultivos repetidos, que no excedan los 10 pasajes, sin alterar sus capacidades de proliferación. Tanto el EGF como el bFGF pueden utilizarse para el cultivo; sin embargo, la expansión a largo plazo de los cultivos solo puede lograrse en presencia de EGF.
- Ensayo de autorrenovación
- Después de pasar los cultivos de neurosfera, se colocan 1.000 células individuales en una placa de 96 pocillos, completando hasta un volumen final de 200 μL con medio completo, lo que da como resultado una densidad celular final de los cultivos de 5 células/μL.
NOTA: El número de celdas sembradas debe ser lo más preciso posible. Si es necesario, prepare diluciones intermedias para garantizar un volumen de suspensión celular confiable antes de colocar el recubrimiento. Además, se recomienda colocar de 4 a 5 repeticiones por condición para reducir la variabilidad técnica. - Incubar las células a 37 °C y 5% de CO2 en una incubadora humidificada durante 5 días o hasta que las neuroesferas alcancen un tamaño adecuado (60 a 100 μm). Evite mover o agitar la placa de pocillos múltiples para evitar la agregación de la neurosfera.
- Después de la formación de la neurosfera, cuente el número de neuroesferas formadas en cada pocillo utilizando un microscopio de contraste de fase invertido equipado con una cámara adjunta. El ensayo de autorrenovación puede realizarse utilizando EGF o bFGF por separado.
- Después de pasar los cultivos de neurosfera, se colocan 1.000 células individuales en una placa de 96 pocillos, completando hasta un volumen final de 200 μL con medio completo, lo que da como resultado una densidad celular final de los cultivos de 5 células/μL.
- Criopreservación de cultivos de NSC
NOTA: Los cultivos de neurosfera pueden criopreservarse para su uso futuro, minimizando el número de ratones necesarios. Recomendamos preservar las células hasta el pasaje 10.- Para criopreservar las células, transfiera 1 mL de la suspensión celular en medio completo a un tubo criogénico debidamente marcado.
- Agregue dimetilsulfóxido (DMSO) a una concentración final del 10% a la suspensión celular y mezcle suavemente invirtiendo los tubos.
NOTA: El DMSO sirve como crioprotector, pero puede ser tóxico para las células, por lo que este paso debe realizarse rápidamente. - Coloque los tubos en un congelador de -80 °C utilizando un recipiente de congelación que permita una disminución gradual de la temperatura a 1 °C/min. Este proceso de congelación gradual evita el daño celular causado por la formación de cristales de hielo durante la criopreservación. Para el almacenamiento a largo plazo, mantenga los tubos en un tanque de nitrógeno líquido a -196 °C.
- Deshielo de los cultivos NSC
NOTA: Cuando sea necesario, las células se pueden descongelar y los cultivos se pueden volver a expandir para los experimentos.- Retire los tubos criogénicos que contienen NSC congelados del tanque de almacenamiento de nitrógeno líquido y colóquelos inmediatamente en un baño de agua a 37 °C hasta que se descongelen por completo, aproximadamente 5 min.
PRECAUCIÓN: Es crucial minimizar el tiempo de baño ya que el DMSO es tóxico y puede afectar la viabilidad celular. - Transfiera rápidamente las celdas descongeladas a un tubo de centrífuga de 15 mL que contiene 5 mL de medio de control precalentado.
- Centrifugar las células a 300 x g durante 5 min y eliminar el sobrenadante. Agregue 1 mL de medio completo y vuelva a suspender el pellet pipeteando suavemente hacia arriba y hacia abajo 10 veces para homogeneizar la suspensión celular antes de la siembra.
NOTA: La descongelación puede afectar la viabilidad de la célula; Por lo tanto, se recomienda pasar las células al menos una vez antes de sembrar para nuevos experimentos.
- Retire los tubos criogénicos que contienen NSC congelados del tanque de almacenamiento de nitrógeno líquido y colóquelos inmediatamente en un baño de agua a 37 °C hasta que se descongelen por completo, aproximadamente 5 min.
- Pruebas de micoplasma
NOTA: La contaminación por micoplasma puede comprometer la salud celular, afectando las tasas de crecimiento. Por lo tanto, las pruebas de micoplasma son cruciales para garantizar la integridad y fiabilidad de los experimentos.- Extracción de ADN
- Recoja un mínimo de 100 μL del cultivo celular para la extracción de ADN. Tanto el sobrenadante como los gránulos celulares se pueden utilizar para la extracción de ADN. Almacene esta alícuota a -20 °C hasta la extracción.
- Perforar la tapa del tubo y hervir la alícuota a 99 °C durante 15 min con un termobloque.
NOTA: La perforación de las tapas de los tubos evita que los tubos se abran durante la ebullición a altas temperaturas. - Centrífuga durante 5 min a 16.900 x g. Transfiera el sobrenadante que contiene el ADN a un nuevo tubo. Guarde el tubo con el ADN a -20 °C hasta la prueba de PCR.
- PCR para la detección de micoplasmas
- Prepare la mezcla de PCR utilizando los siguientes componentes: 8,9 μL de agua libre de nucleasas, 3 μL de tampón de reacción 5x, 1,2 μL de 25 mM MgCl2, 0,25 μL de dNTP de 2,5 mM, 0,15 μL de polimerasa Taq y 0,25 μL de cada cebador específico de micoplasma a una concentración de 10 μM (Adelante, Fw: 5'GATGTTTAGCCGGGTCGAGAG3' y Reverso, Rv: 5'GATGTCAAGAGTGGGTAAGGTT3').
- Añadir 1 μL de ADN extraído genómicamente. Prepare la mezcla de PCR en una campana de flujo laminar para evitar la contaminación del ADN.
- Incubar la reacción en un termociclador de la siguiente manera: desnaturalización inicial a 95 °C durante 5 min seguida de 35 ciclos de desnaturalización a 95 °C durante 30 s, recocido a 53 °C durante 1 min, extensión a 72 °C durante 30 s y extensión final a 72 °C durante 5 min.
- Para identificar el tamaño de la banda resultante (alrededor de 500 pb), cargue 15 μL del producto de PCR de ADN mezclado con 3 μL de solución de colorante de carga 6x sobre un gel de agarosa al 2% y ejecute la electroforesis durante 40 a 50 min a 100 V.
- Extracción de ADN
Tabla 2: Platos y frascos utilizados para el cultivo. Se proporcionan las dimensiones y volúmenes de las placas de siembra y matraces más utilizados. La tabla incluye el diámetro, el área de crecimiento y el volumen medio utilizado para cada recipiente, junto con un ejemplo del número de NSC que se sembrarán en condiciones de expansión (10.000 células/cm2). Haga clic aquí para descargar esta tabla.
3. Nucleofección de cultivos de neurosfera
- Preparación de reactivos
- Preparación del ADN
- Prepare 25 μL de células competentes de E. coli DH5α en un tubo de 1,5 mL para su transformación.
- Añada 1 μL del plásmido de interés (a 100 a 200 ng/μL) a las células y sométalas a un choque térmico a 42 °C durante 45 s. Aquí, se utiliza un plásmido que contiene un (sh)RNA de horquilla corta para silenciar la expresión de Snrpn y un plásmido de control shSCRAMBLE.
NOTA: Mantenga las células competentes en hielo durante todo el procedimiento para mantener la efectividad de la transformación. - Colocar las células transformadas en placas de agar LB con el antibiótico adecuado e incubarlas a 37 °C durante 18 h. Mantenga las placas de Petri invertidas para evitar la formación de gotas debido a la condensación, que podría perturbar las colonias bacterianas.
NOTA: Agregue antibióticos apropiados al medio de cultivo LB para permitir exclusivamente el crecimiento de bacterias transformadas. - Con una punta de pipeta estéril, recoja las colonias bacterianas individuales y transfiérelas a un matraz Erlenmeyer previamente lleno con 250 mL de LB para iniciar un cultivo líquido. Evite elegir pequeñas colonias satélite colocadas alrededor de las colonias objetivo más grandes, ya que es posible que no hayan incorporado el plásmido deseado.
- Incubar el matraz agitando vigorosamente en un agitador orbital a 37 °C durante la noche.
- Utilice un kit de maxiprep libre de endotoxinas y extraiga el ADN plásmido puro de los cultivos bacterianos siguiendo las instrucciones del fabricante. Reconstituya el ADN en el volumen adecuado de agua libre de endotoxinas para obtener una concentración de plásmido de 1 a 2 μg/μL.
NOTA: El rendimiento de maxiprep depende del crecimiento del cultivo. Por lo general, la resuspensión de la pellet de ADN plasmídico en un volumen de 200 μL dará como resultado la concentración de ADN deseada. - Guarde el tubo con el ADN plasmídico a -20 °C hasta su uso. Prepare un tubo de 1,5 mL con un volumen final de 5 μL que contenga de 2 a 6 μg del ADN plasmídico destinado a la nucleofección.
- Preparación del material
- Preparar la solución de nucleofección. Si utiliza el kit recomendado (consulte la tabla de materiales), prepare 95 μL de la solución de nucleofección en un tubo de 1,5 mL para cada nucleofección deseada. Las pipetas y cubetas suelen estar incluidas en el kit. Prepare uno de cada uno para cada condición de nucleofección.
- Prepare un matraz T25 lleno de 4 mL de medio completo precalentado por condición y manténgalo en la incubadora hasta su uso.
- Preparación del ADN
- Nucleofección de NSCs
- Utilice de 1 a 2 x 106 celdas individuales por condición para la evaluación. Centrifugar las células durante 5 min a 300 x g, luego retire el sobrenadante.
- Agregue 1 ml de DPBS y vuelva a suspender el pellet pipeteando hacia arriba y hacia abajo 10 veces para garantizar una suspensión celular homogénea.
- Repita la centrifugación (paso 2.1.6) para un segundo lavado y finalmente vuelva a suspender el pellet en 95 μL de solución de nucleofección.
NOTA: Los pasos de lavado son cruciales para eliminar los antibióticos del medio de control y garantizar una nucleofección efectiva. - Combine los 95 μL de células con una solución premezclada que contenga 5 μL de cada plásmido (2 μg en total) seleccionado para la nucleofección.
- Con una micropipeta P200, transfiera esta solución a una cubeta y colóquela en el dispositivo nucleofector.
- Nucleofecte las células con los plásmidos deseados utilizando el programa NSC optimizado del sistema nucleofector.
- Después de completar la electroporación, introduzca con cuidado el medio completo tibio en la cubeta con las pipetas Pasteur suministradas en el kit. A continuación, transfiera el contenido de la cubeta a un matraz T25 previamente preparado que contenga medio completo precalentado. Después de la electroporación, puede haber muerte celular, precipitación del ADN y la formación de un agregado viscoso. Evite transferir esto al matraz T25, ya que podría reducir la supervivencia celular.
NOTA: Este paso es crucial y debe realizarse lo más rápido posible para garantizar un alto rendimiento de nucleofección y la posterior supervivencia del cultivo. - Incubar las células nucleofectadas a 37 °C y 5% de CO2 en una incubadora humidificada durante 3 a 5 días.
- Selección de células nucleofectadas
- Estrategia A: Selección de citometría de flujo por clasificación celular activada por fluorescencia (FACS)
- Después de 3 a 5 días de nucleofección, pase los cultivos nucleofectados siguiendo el paso 2.1 y recoja todas las células individualizadas obtenidas.
- Filtre las muestras a través de un filtro de células de 40 μm para eliminar los agregados de células y analizarlas con el citómetro de flujo clasificador de células.
NOTA: Es crucial ordenar el mismo número de celdas en cada condición para obtener resultados comparativos. - Utilizando el software del citómetro, se pueden controlar las células vivas en función de dos medidas: la luz dispersada hacia adelante (FSC-A) que corresponde al tamaño de la célula y la luz dispersa lateral (SSC-A) que corresponde a la complejidad intracelular. Seleccione la población de células vivas caracterizadas por alto FSC-A y alto SSC-A.
NOTA: Opcionalmente, agregue 0.1 μg/mL de 4′,6-diamidino-2-fenilindol (DAPI) a la suspensión celular antes de clasificar. Esto permitirá descartar las células muertas y los cuerpos apoptóticos de la compuerta. - Descarte los dobletes y tripletes de celdas trazando los parámetros FSC-A frente a FSC-H. Elija los eventos incluidos en la diagonal de la gráfica, que representan singletes.
NOTA: Cuando las células pasan a través del láser, el citómetro detecta los cambios de voltaje con el tiempo. FSC-A representa el área de la señal y FSC-H significa la altura del pico. Los singletes muestran una relación proporcional entre FSC-A y FSC-H (ubicados en la diagonal de la gráfica), mientras que los agregados de celdas muestran un mayor FSC-A en relación con FSC-H debido a una señal más larga. - Seleccione las células nucleofectadas en función de la intensidad de fluorescencia del gen reportero codificado por el plásmido utilizado. Por ejemplo, cuando se utilizan células GFP+ se pueden seleccionar en función de los niveles de fluorescencia FITC-A frente a la gráfica SSC-A.
- Aísle el número deseado de células objetivo para cada condición y recójalas directamente en un tubo de recolección lleno de medio completo.
- Semillas de NSCs nucleofectadas a una densidad de 10.000 células/cm2. Incubar los cultivos celulares clasificados a 37 °C y 5% de CO2 en una incubadora humidificada.
- Estrategia B: Selección mediante resistencia a antibióticos codificada en el plásmido
- Después de 48 h de nucleofección, añadir la concentración de antibiótico adecuada para seleccionar las células resistentes.
NOTA: Antes de comenzar, se recomienda probar múltiples concentraciones del antibiótico en células no nucleofectadas. La concentración óptima de antibiótico es la cantidad más baja a la que se eliminan todas las células no resistentes. Por ejemplo, determinamos una concentración óptima de 4 μg/mL de antibiótico blasticidina para cultivos de NSC. - Incubar las células a 37 °C y 5% de CO2 en una incubadora humidificada durante 48 h.
- Después de 48 h de tratamiento antibiótico, subcultivar las células nucleofectadas de acuerdo con el paso 2.1. Si las neuroesferas siguen siendo pequeñas, retire el antibiótico del medio de cultivo mediante centrifugación a 300 x g durante 5 min. Vuelva a suspender las células y cultive en un matraz utilizando únicamente medio completo. Incubar las células a 37 °C y 5% de CO2 en una incubadora humidificada hasta que puedan expandirse siguiendo el paso 2.1.
- Después de 48 h de nucleofección, añadir la concentración de antibiótico adecuada para seleccionar las células resistentes.
- Estrategia A: Selección de citometría de flujo por clasificación celular activada por fluorescencia (FACS)
Resultados
Las condiciones óptimas de cultivo permiten el aislamiento y la expansión in vitro de las NSC derivadas de SVZ adultas
Los cultivos de NSCs derivados de la SVZ adulta han servido como un valioso método in vitro para investigar los mecanismos moleculares y las señales de nicho que regulan las NSCs dentro de sus microambientes específicos. El ensayo de neurosfera descrito en este manuscrito se empleó para examinar el recuento de NSC dentro de la SVZ adulta. Se aisló tejido SVZ de los cerebros de ratones de 3 meses de edad, se disoció y se cultivó en medio NSC completo suplementado con EGF y bFGF o cada uno por separado. Después de 10 días in vitro (DIV), el recuento total de esferas primarias formadas bajo estas tres condiciones de cultivo distintas se cuantificó utilizando microscopía de contraste de fase (Figura 2A, B). Sorprendentemente, nuestros hallazgos demuestran que la presencia de EGF condujo a la máxima formación de esferas primarias, lo que es evidente por el recuento reducido de esferas primarias observado en cultivos con solo bFGF (Figura 2A, B). Para evaluar la capacidad de autorrenovación de las NSC en diversas condiciones de medio, las células se subcultivaron y se sembraron a baja densidad (5 células/μL) en medios suplementados con las combinaciones de mitógenos antes mencionadas. La cuantificación de esferas secundarias reveló que las NSC de SVZ necesitan al menos EGF para autorrenovarse de manera eficiente y que la combinación de EGF y bFGF mejora la capacidad de autorrenovación de las células (Figura 2B). Además, para un análisis detallado de la dinámica de crecimiento en diversas condiciones de medios, se registró el número de células sembradas y obtenidas a lo largo de 7 pasajes. Las curvas de crecimiento obtenidas a partir de diferentes condiciones de medio confirmaron que los cultivos suplementados con EGF solo o en combinación con bFGF mostraron una dinámica de crecimiento mejorada en comparación con los cultivos suplementados únicamente con bFGF (Figura 2C). En conjunto, estos hallazgos corroboran que el uso simultáneo de EGF y bFGF mejora el rendimiento del cultivo de NSC in vitro.
Con el fin de investigar el recuento de NSC dentro de la SVZ a lo largo de diferentes edades postnatales y evaluar el impacto del envejecimiento de los ratones en la eficiencia del cultivo de la neurosfera, se diseccionó tejido de SVZ de ratones de 1 mes a 12 meses. En particular, nuestros hallazgos revelaron una disminución significativa en el número de esferas primarias con el aumento de la edad, mostrando la máxima eficiencia en el recuento de esferas alrededor de los 2 a 4 meses de edad (Figura 2D). Además, para evaluar la capacidad de autorrenovación de las NSC durante el subcultivo, se subcultivaron y sembraron NSC de ratones de 2 a 4 meses de edad a densidad clonal (5 células/μL) en medio completo. La cuantificación del número de esferas secundarias a lo largo de los pasajes subsiguientes indicó una disminución considerable en la eficiencia del cultivo con respecto a los pasajes (Figura 2E). Sobre la base de todas estas observaciones, se recomienda la realización de ensayos de autorrenovación durante los primeros pasos para optimizar aún más las condiciones de cultivo de NSC.
La nucleofección es una técnica muy eficaz para manipular la expresión génica en NSCs adultas
Dado que las NSCs no son fácilmente transfectables, para manipular la expresión génica, aquí presentamos un protocolo de nucleofección con una mayor tasa de entrega génica exitosa sin la necesidad de utilizar la transducción viral. Después de la disección y el cultivo de SVZ individuales de ratones de 2 a 4 meses de edad, las NSC se nucleuvieron con un plásmido portador de GFP como se describe (Figura 3A). La detección de células GFP positivas 2 días después de la nucleofección reveló una eficiencia que osciló entre el 30% y el 50%, en consonancia con la literatura previa28. Para aislar específicamente las NSC nucleofectadas con éxito, las células se clasificaron por FACS en función de la intensidad de fluorescencia de GFP de 3 a 5 días después de la nucleofección (Figura 3A-C). Aproximadamente el 40% de las células analizadas antes de FACS exhibieron altos niveles de fluorescencia GFP y posteriormente se seleccionaron por clasificación (Figura 3C). La resiembra de NSCs GFP+ clasificadas mostró que todas las células eran GFP positivas, validando el método de aislamiento celular basado en nucleofección (Figura 3D). En particular, las NSC nucleofectadas mantuvieron la viabilidad a través de los siguientes pasos, lo que confirma la viabilidad de la nucleofección para las NSC adultas. Estos resultados enfatizan la efectividad de la combinación de nucleofección y FACS para establecer un cultivo puro de NSCs modificadas.
Como ejemplo de modulación génica en NSCs adultas, se empleó la atenuación génica a través de la nucleofección de ARN de horquilla corta (sh). Específicamente, un shRNA dirigido al gen del polipéptido N (Snrpn) de la ribonucleoproteína nuclear pequeña, portador de un reportero CAG-GFP (shSNRPN) se nucleofiló en cultivos de NSC derivados de ratones de 2 meses de edad. La regulación negativa de Snrpn se confirmó mediante qPCR e inmunocitoquímica en células nucleofectadas con el plásmido shSNRPN, pero no para el plásmido shSCRAMBLE de control (Figura 3E, F)29. Para dilucidar los efectos de la regulación negativa de Snrpn en la capacidad de autorrenovación de NSC, se realizó un ensayo de baja densidad en células nucleofectadas (Figura 3F,G). La cuantificación de neuroesferas secundarias en cultivos nucleofectados reveló un aumento de la capacidad de formación de neuroesferas tras la regulación negativa de Snrpn (Figura 3G). Este ensayo subraya la capacidad de nucleofección para manipular la expresión génica en NSCs adultas y, más específicamente, identifica el papel de Snrpn en el mantenimiento de la tallo en NSCs adultas.
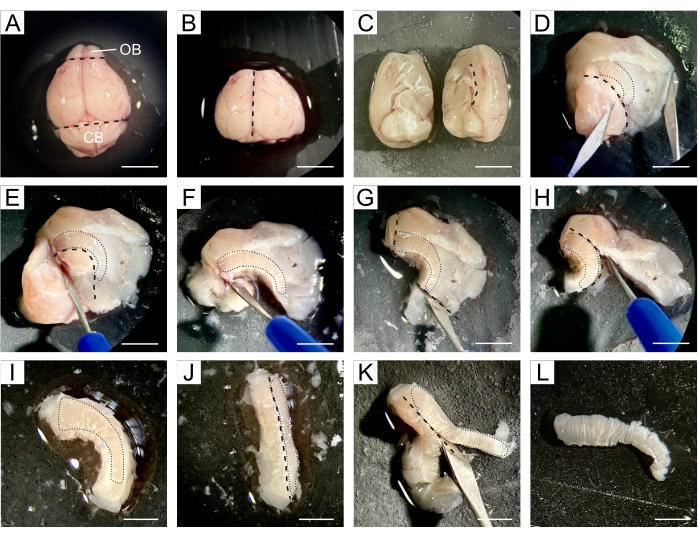
Figura 1: Descripción detallada de la disección de SVZ. (A) Después de extraer el cerebro del ratón, todo el cerebro se transfiere con DPBS a una almohadilla de silicona para su disección. (B) La OB y el cerebelo se extraen del cerebro con un bisturí. (C) El cerebro se divide en dos hemisferios para proceder a la disección por separado. (D) El cerebro se abre a lo largo de la línea del cuerpo calloso, separando la corteza y el cuerpo estriado del hipocampo, el tabique y el diencéfalo, exponiendo así los ventrículos laterales. (E-F) El hipocampo, el tabique y el diencéfalo se extirpan siguiendo el límite ventral de los ventrículos. (G-I) El tejido más allá de los extremos rostral y caudal de la SVZ y la corteza se extirpa siguiendo el cuerpo calloso. (J-K) El tejido se inclina de modo que la SVZ mira hacia los lados, lo que permite la extirpación del tejido estriado debajo de la SVZ. (L) Se obtiene una pieza delgada de tejido que contiene la SVZ. Las líneas discontinuas negras indican el sitio de corte. Las líneas punteadas negras indican la ubicación de la SVZ en cada paso. Abreviaturas: OB = bulbo olfatorio; CB = cerebelo. Barra de escala en A-H: 5 mm; en I-L: 3 mm. Haga clic aquí para ver una versión más grande de esta figura.
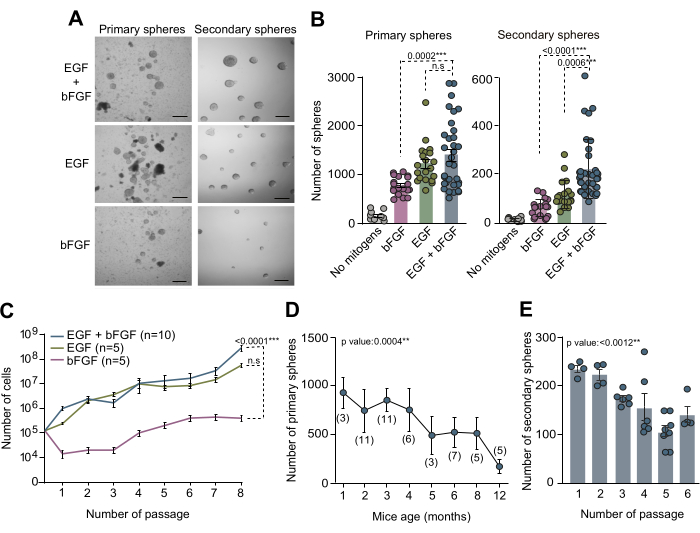
Figura 2: Ambos mitógenos EGF y bFGF son necesarios para un cultivo óptimo y la expansión de NSCs in vitro. (A) Imágenes de microscopía de contraste de fase de cultivos de neurosfera primaria y secundaria en medio NSC suplementado con EGF y bFGF o cada mitógeno por separado. (B) Número de esferas primarias obtenidas por ratón y por pocillo en función de la composición mitógena del medio de cultivo: sin mitógenos (gris, n = 9), EGF + bFGF (azul, n = 33), solo EGF (verde, n = 20) o solo bFGF (rosa, n = 19; panel izquierdo). Número de neuroesferas secundarias formadas a baja densidad (5 células/μL) por pocillo en función de la composición mitógena del medio de cultivo): sin mitógenos (gris, n=11), bFGF (rosa, n=18), EGF (verde, n=18) o EGF+bFGF (azul, n=37; panel derecho). Se utilizó la prueba de Kruskal-Wallis con análisis post hoc de Dunn. (C) Curvas de crecimiento que muestran el número total de células formadas después de 8 pasadas en distintos cultivos de neurosfera según los mitógenos presentes en el medio de cultivo: bFGF (rosa, n=5), EGF (verde, n=5) o EGF+bFGF (azul, n=10). Se utilizó un análisis de regresión lineal. (D) El número de neuroesferas primarias derivadas de ratones individuales diseccionados y disgregados de animales en varias etapas de desarrollo postnatal. Se utilizó un análisis de regresión lineal. n indicado entre paréntesis. (E) El número de neurosferas secundarias formadas a baja densidad (5 células/μL) a través de pasajes posteriores in vitro para evaluar su capacidad de autorrenovación. Se utilizó la prueba de Kruskal-Wallis y se incluyen los valores de p: *: p<0,05; **: p<0,01; : P<0,001; : P<0.0001. N.S: No significativo. Las barras de error representan el SEM. La barra de escala en A es de 100 μm. Haga clic aquí para ver una versión más grande de esta figura.
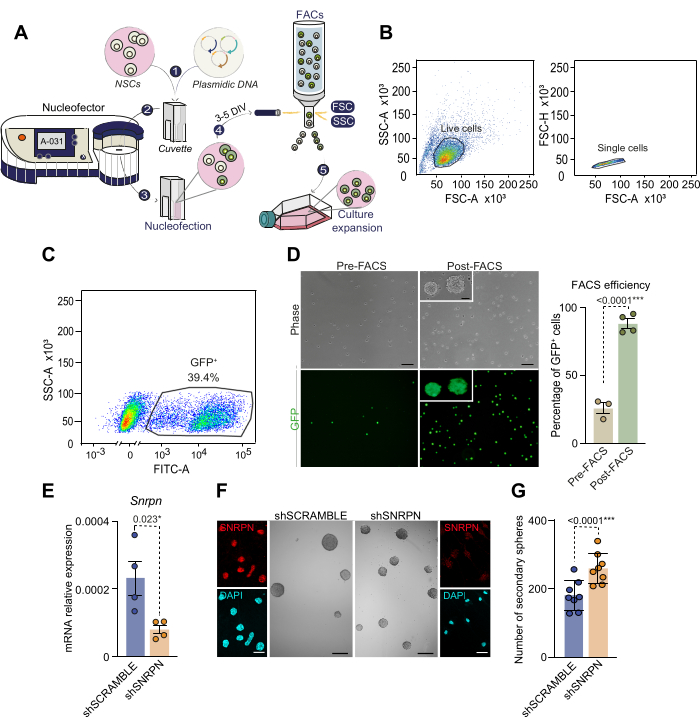
Figura 3: Esquema del proceso de nucleofección y selección celular de células nucleofectadas basada en FACS. (A) Descripción general del proceso de nucleofección y selección celular de células nucleofectadas basada en FACS. 1) La solución de ADN plasmídico se combina con la solución de nucleofección y se agrega a las células. A continuación, esta mezcla se transfiere a una cubeta para la nucleofección. 2) La cubeta que contiene el ADN y las células se inserta en el nucleofector, donde se aplica una descarga eléctrica. Para los cultivos de neurosfera aislados de la SVZ adulta, recomendamos utilizar el programa A-031 NSC. 3) Después de la nucleofección, las células se transfieren a un matraz que contiene medio completo suplementado con EGF y bFGF. 4) Las células se clasifican por FACS en función de la expresión del reportero contenido en el ADN del plásmido. 5) Las células clasificadas se someten a un cultivo adicional para experimentos posteriores. (B) Estrategia de selección basada en FACS y compuerta para NSC nucleofectadas. En primer lugar, se seleccionan las células vivas en función de un FSC-A alto y un SSC-A alto (gráfico de la izquierda) y, a continuación, se eliminan los dobletes y tripletes de células mediante el análisis de los parámetros FSC-A frente a FSC-H (gráfico de la derecha). (C) Activación de FACS para células nucleofectadas en función de su expresión de GFP (SC-A frente a fluorescencia FITC-A). (D) Microscopía de contraste de fase e imágenes de fluorescencia GFP de NSCs nucleofectadas antes (n=3) y después de (n=4) selección de FACS (panel izquierdo). El porcentaje de células GFP+ nucleofefectadas se muestra antes y después de la selección de FACS (panel derecho). Se utilizó la prueba T. (E) Análisis cuantitativo de PCR (qPCR) de la expresión de Snrpn en NSCs en proliferación nucleofectadas con shRNA específico de Snrpn (n=4). La nucleofección utilizando un shRNA SCRAMBLE sirvió como control (n=4). Se utilizó la prueba T. (F) Imágenes inmunocitoquímicas que muestran SNRPN (en rojo) en NSC 7 días después de la nucleofección de shRNA (paneles externos). Se utilizó DAPI para contrarrestar la tinción del ADN. Imágenes representativas que representan neuroesferas derivadas de NSCs bajo condiciones shSNRPN y shSCRAMBLE (paneles centrales). (G) Cuantificación del número de neuroesferas formadas en cultivos de NSC después de la nucleofección con shSNRPN (n = 8) o shSCRAMBLE (n = 8). Se utilizó la prueba T. Se incluyen los valores de p: *: p<0,05; **: p<0,01; : P<0,001; : P<0.0001. N.S: No significativo. Las barras de error representan el SEM. Barra de escala en D, 10 μm; en F, 100 μm (imágenes de gran aumento en F, 7 μm). Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Debido a la falta de marcadores definitivos para identificar la población de NSC in vivo, el análisis de las NSC se ha basado principalmente en la observación del comportamiento de células aisladas de nichos neurogénicos en condiciones ex vivo . El trabajo pionero de Reynolds y Weiss sentó las bases al establecer condiciones de cultivo precisas, lo que permitió el aislamiento y la expansión de células individuales de tejido SVZ de ratón adulto joven (2 meses de edad) en condiciones no adhesivas. Estas células generalmente se propagan en un medio libre de suero que contiene EGF y bFGF, condiciones que impiden por completo la diferenciación en neuronas y glía, al tiempo que promueven la proliferación. De hecho, en estas condicionesde cultivo 9, la mayoría de las células mueren durante los primeros días de cultivo, pero un pequeño subconjunto comienza a dividirse y forma principalmente neurosferas flotantes9. La disociación enzimática y el subcultivo de estos agregados celulares facilitan la propagación de los cultivos, demostrando la capacidad de autorrenovación de estos cultivos.
En particular, los cultivos de neurosfera muestran potencial de expansión con la adición de EGF solamente, mientras que las NSC cultivadas en un medio que contiene solo bFGF no muestran proliferación celular a largo plazo30. Ambas evidencias señalan al EGF como el principal mitógeno de autorrenovación del NSC. Sin embargo, la presencia de EGF y bFGF en el medio de cultivo mejora la capacidad de autorrenovación de las NSCs31, así como contribuye a equilibrar el potencial de diferenciación en astrocitos, neuronas y oligodendrocitos 32,33,34,35. Además, el uso de una mezcla definida de hormonas y factores en lugar de alternativas comerciales para complementar el medio garantiza la alta calidad y reproducibilidad de los cultivos de NSC. En estas condiciones, los cultivos de NSC de ratón pueden persistir como líneas celulares estables sin sufrir inmortalización. Sin embargo, una preocupación en los cultivos de la neurosfera es el potencial de las poblaciones de células proliferativas para sufrir transformaciones genéticas, eludiendo los mecanismos reguladores del ciclo celular y conduciendo a un fenotipo inmortalizado. Por lo tanto, aunque los cultivos de neurosfera son altamente expandibles, los cultivos de más de 10 pasajes in vitro generalmente se descartan para estudios, ya que pueden sufrir un envejecimiento replicativo y pueden surgir células con contenido cromosómico anormal o dinámica de crecimiento irregular. Además, el uso de cultivos de neurosfera establecidos a largo plazo debe ser monitoreado continuamente. Los cultivos de neurosfera también ofrecen la oportunidad de examinar sus características y potencial en un entorno controlado, proporcionando un ajuste más preciso y ajustable que puede modularse y monitorizarse con mayor precisión que in vivo. A través de análisis clonogénicos o poblacionales in vitro, es posible cuantificar las capacidades de autorrenovación y proliferación de estas células, lo que facilita la identificación de los mecanismos subyacentes que gobiernan estas propiedades.
Sin embargo, a pesar de la gran lista de ventajas de trabajar con NSCs in vitro, la naturaleza de este protocolo de cultivo y la delicadeza de las NSCs constituyen un desafío en el campo. Por ejemplo, el número de neuroesferas primarias generadas durante una disección típica puede variar significativamente en función de la habilidad y la precisión del experimentador. Los resultados de la neurosfera primaria ilustran esta variabilidad en el número de neurosferas primarias obtenidas de la SVZ de ratones de 2 meses de edad, que oscilan entre 500 y 3000 neurosferas. Varios factores pueden contribuir a esta variabilidad. En primer lugar, la precisión de la disección minimiza el tejido parenquimatoso no deseado, que inhibe la formación de esferas primarias. En segundo lugar, la generación de pequeños trozos de tejido SVZ permite una digestión y trituración enzimática eficientes, reduciendo así la pérdida celular. Esto pone de manifiesto la necesidad de una práctica previa suficiente del protocolo de disección y de un desarrollo afinado del procesamiento de tejidos durante el establecimiento de los cultivos de neurosfera.
Otra limitación de estos cultivos radica en el hecho de que las neuroesferas pueden comprender tanto NSC como células progenitoras, lo que dificulta la distinción entre estas dos poblaciones dentro de los cultivos primarios. Si bien se ha informado que varios marcadores como GFAP, Nestin, Musashi y SOX2 son expresados porNSCs 8, ninguno de ellos se ha asociado exclusivamente con NSCs. Las técnicas FACS emergentes basadas en la expresión de antígenos de superficie celular han permitido el aislamiento de NSCs y su progenie. Estos estudios han demostrado que los progenitores amplificadores de tránsito son incapaces de formar neuroesferas al paso 25,36. Por lo tanto, mientras que la relación entre las células SVZ y las células iniciadoras de la neurosfera requiere un mayor refinamiento 18,19,20,21,22,23, la capacidad de las neuroesferas para ser transmitidas en serie durante un período prolongado puede reflejar la presencia de NSC en los cultivos.
Este sistema de cultivo de neurosfera ha servido como un modelo robusto para investigar el impacto de las vías de señalización y la expresión génica en el mantenimiento de la capacidad de autorrenovación de NSC in vitro15,29. Un enfoque para explorar estos aspectos consiste en transfectar NSC para sobreexpresar o eliminar genes específicos. Esto se puede lograr a través de varias técnicas, incluidos los métodos virales y no virales. Si bien los vectores virales a menudo logran una alta eficiencia de transferencia de genes, tienen limitaciones importantes, como los altos requisitos de seguridad y la producción de vectores que requiere mucho tiempo26. Por el contrario, los métodos clásicos de transfección, como la lipofección y la electroporación, logran tasas de transfección muy bajas, lo que los hace inviables para las células difíciles de transfectar. La tecnología de nucleofección ofrece un enfoque fácil de usar al combinar soluciones de nucleofección específicas del tipo de célula con parámetros eléctricos únicos para cada tipo de célula37,38. Esto asegura la transferencia de ADN directamente al núcleo celular39, lo que permite la incorporación del ADN de manera independiente del ciclo celular. En consecuencia, la nucleofección surge como una técnica viable para la transfectación de células difíciles de transfectar, como las NSC de ratón28,40.
Una limitación de este método es que se recomienda y es necesario un mínimo de 2 x 106 células para cada nucleofección, aunque en condiciones óptimas, se puede utilizar un número menor de células por nucleofección (es decir, 5 × 105 células). Otro inconveniente del método es que la calidad y la concentración del ADN utilizado para la nucleofección influyen significativamente en la eficiencia de la transferencia de genes. Se recomienda encarecidamente utilizar ADN preparado libre de endotoxinas para prevenir la mortalidad celular elevada debido a la presencia de endotoxinas. Además, el uso de más de 6 μg de ADN total para la nucleofección puede reducir sustancialmente tanto la eficiencia de la transferencia de genes como la viabilidad celular. Por último, la administración de pulsos eléctricos también es fundamental para la supervivencia de las células nucleofectadas.
En este trabajo, ejemplificamos la manipulación de la expresión génica de Snrpn mediante el empleo de una estrategia que implica la regulación negativa de su expresión utilizando plásmidos episomales. Estos plásmidos no se integran en el genoma de las células, y a medida que las NSC siguen proliferando in vitro, el ADN introducido se diluirá posteriormente a lo largo de la división celular y, por lo tanto, perderá el efecto de la manipulación genética. Por lo tanto, esta estrategia es valiosa para estudiar el efecto de una alteración aguda en un corto período de tiempo o para datar el nacimiento de las células in vitro. Existen algunas alternativas para evaluar un efecto más prolongado de la perturbación génica. Por ejemplo, se podría utilizar un sistema integrador como el piggyBAC basado en transposones. Este sistema consiste en introducir la secuencia codificante deseada contenida en el plásmido flanqueada por secuencias transponibles y co-nucleoafectar las células con un plásmido que contiene la secuencia para la enzima transposasa41. Alternativamente, se podrían utilizar transposones o los sistemas CRISPR/Cas.
Un mayor desarrollo de las tecnologías de transfección será un paso importante hacia los ensayos de alto rendimiento para evaluar el papel de diferentes genes en la fisiología de las células madre neurales adultas. En combinación con métodos de purificación y expansión cada vez más sofisticados, estos estudios permitirán comprender la biología in vitro de las NSC adultas y comparar las diferencias biológicas entre las neuroesferas flotantes y las NSC in vivo.
Divulgaciones
Los autores declaran no tener intereses financieros contrapuestos.
Agradecimientos
Este trabajo ha contado con el apoyo de subvenciones del Ministerio de Ciencia e Innovación y de la Agencia Estatal de Investigación (MCIN/AEI) (PID2019-110045GB-I00; PID2022-142734OB-I00 y EUR2023-143479) a SRF. EJV (FPU20/00795) y DSL (FPU22/03797) están financiados por el programa de becas de Formación de Profesorado Universitario (FPU). LLC (PRE2020-094137) y JDM (PREP2022-000680) están financiadas por el programa de becas de la Formación de Personal Investigador (FPI). El CMM (CIACIF/2022/366) está financiado por la Generalitat Valenciana. MIL (CPI-22-481) está financiado por la beca del Programa INVESTIGO (Next Generation EU). Financiación en Acceso Abierto proporcionada por el Ministerio de Ciencia e Innovación.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.22 μm pore-filter bottles (250 mL) | VWR | 514-0330P | |
| 0.22 μm pore-filter bottles (500 mL) | VWR | 514-0332P | |
| 12-well plate | LabClinics | PLC30012 | |
| 15 mL tube | Fisher | 10738771 | |
| 24-well plate | LabClinics | PLC30024 | |
| 4-(2-hydroxyethyl)-1-piperazineethanesulfonic acid (HEPES) | BioWest | L0180-100 | |
| 4′,6-diamidino-2-phenylindole (DAPI) | Sigma | D9542 | |
| 48-well plate | ThermoFisher | 150687 | |
| 6-well plate | LabClinics | PLC30006 | |
| 96-well plate | Labclinics | PLC30096 | |
| Accutase solution | Sigma | A6964-100ML | Referred as enzymatic solution |
| Amaxa Mouse Neural Stem Cell Nucleofector Kit | Lonza | VPG-1004 | |
| Amaxa Nucleofector Iib | Lonza | 10700807 | |
| Antibiotic/Antimitotic (A/A) | Sigma | A5955 | |
| Apo-t-Transferrine | Sigma | T2252-1G | |
| Basic Fibroblast Growth Factor (bFGF) | Sigma | F0291 | |
| Blasticidin | Sigma | 203350 | |
| Bovine Serum Albumin (BSA) | Sigma | B4287-25MG | |
| Cell strainer 40 μm | LabClinics | PLC93040 | |
| Deoxynucleotide triphosphate (dNTPs) | NZYTech | MB08701 | |
| Dimethyl sulfoxide (DMSO) | Sigma | D8418 | |
| Dulbecco’s Phosphate Saline Buffer (DPBS) | Gibco | 14080-055 | |
| Dulbecco's Modified Eagle's Medium (DMEM) F12 1:1 | Gibco | 11320-074 | |
| E. coli DH5α Competent Cells | ThermoFisher | EC0112 | |
| Earles's Balanced Salt Solution (EBSS) | Gibco | 24010-043 | |
| Epidermal Growth Factor - Human recombinant (EGF) | Gibco | 53003-018 | |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma | E-6511 | |
| Fine Forceps | Fine Science Tools | 11274-20 | |
| Fine Scissors Sharp | Fine Science Tools | 14060-09 | |
| Glucose | Sigma | G-7021 | |
| GoTaq G2 Flexi DNA Polymerase | Promega | M7805 | Kit includes reagents for PCR |
| Heparin | Sigma | H-3149 | |
| Insuline | Sigma | I6634 | |
| LB agar (Lennox) | LabKem | AGLB-00P-500 | |
| LB broth (Lennox) | LabKem | LBBR-00P-500 | |
| L-Cysteine | Sigma | C-8277 | |
| L-Glutamine | Gibco | 25030-024 | |
| Neubauer chamber | Blaubrand | BR718620 | |
| Nuclease free water | Labbox | WATR-00A-10K | |
| NZYMaxiprep Endotoxin Free Kit | NZYTech | MB39901 | |
| Papain Lyophilized | Worthington | LS003119 | |
| Progesterone | Sigma | P-6149 | |
| Putrescine | Sigma | P-7505 | |
| Scalpel | Fine Science Tools | 10316-14 | |
| shSCRAMBLE | Mission (Sigma) | SHC003 | |
| shSNRPN | Mission (Sigma) | TRCN0000109285 | |
| Sodium Selenite | Sigma | S-9133 | |
| Spatula | Fine Science Tools | 10090-17 | |
| Sterile PES Syringe Filters (0.22 μm pore-filter) | Epica | SFPE-22E-050 | |
| T12.5 cm2 Flask | Biofil | TCF012025 | |
| T25 cm2 Flask | LabClinics | PLC70025 | |
| T75 cm2 Flask | LabClinics | PLC70075 | |
| Tweezers | Fine Science Tools | 91150-20 |
Referencias
- Gage, F. H. Mammalian neural stem cells. Science. 287 (5457), 1433-1438 (2000).
- Ming, G., Song, H. Adult neurogenesis in the mammalian central nervous system. Ann Rev Neurosci. 28 (1), 223-250 (2005).
- Doetsch, F., Caillé, I., Lim, D. A., García-Verdugo, J. M., Alvarez-Buylla, A. Subventricular zone astrocytes are neural stem cells in the adult mammalian brain. Cell. 97 (6), 703-716 (1999).
- Obernier, K., et al. Adult neurogenesis is sustained by symmetric self-renewal and differentiation. Cell Stem Cell. 22 (2), 221-234 (2018).
- Doetsch, F., García-Verdugo, J. M., Alvarez-Buylla, A. Regeneration of a germinal layer in the adult mammalian brain. Proc Natl Acad Sci. 96 (20), 11619-11624 (1999).
- Doetsch, F., García-Verdugo, J. M., Alvarez-Buylla, A. Cellular composition and three-dimensional organization of the subventricular germinal zone in the adult mammalian brain. J Neurosci. 17 (13), 5046-5061 (1997).
- Mamber, C., Kozareva, D. A., Kamphuis, W., Hol, E. M. Shades of gray: The delineation of marker expression within the adult rodent subventricular zone. Prog Neurobiol. 111, 1-16 (2013).
- Cebrian-Silla, A., et al. Single-cell analysis of the ventricular-subventricular zone reveals signatures of dorsal and ventral adult neurogenesis. eLife. 10, e67436(2021).
- Reynolds, B. A., Weiss, S. Generation of neurons and astrocytes from isolated cells of the adult mammalian central nervous system. Science. 255 (5052), New York, N.Y. 1707-1710 (1992).
- Azari, H., Rahman, M., Sharififar, S., Reynolds, B. A. Isolation and expansion of the adult mouse neural stem cells using the neurosphere assay. J Vis Exp. (45), e2393(2010).
- Jensen, J. B., Parmar, M. Strengths and limitations of the neurosphere culture system. Mol Neurobiol. 34 (3), 153-161 (2006).
- Ferrón, S. R., et al. Postnatal loss of Dlk1 imprinting in stem cells and niche astrocytes regulates neurogenesis. Nature. 475 (7356), 381-385 (2011).
- Gil-Perotín, S., et al. Adult neural stem cells from the subventricular zone: A review of the neurosphere assay. Anat Record. 296 (9), 1435-1452 (2013).
- Obernier, K., Alvarez-Buylla, A. Neural stem cells: origin, heterogeneity and regulation in the adult mammalian brain. Development. 146 (4), 156059(2019).
- Ferrón, S. R., et al. A combined ex/in vivo assay to detect effects of exogenously added factors in neural stem cells. Nat Prot. 2 (4), 849-859 (2007).
- Ferron, S., et al. Telomere shortening and chromosomal instability abrogates proliferation of adult but not embryonic neural stem cells. Development. 131 (16), 4059-4070 (2004).
- Singec, I., et al. Defining the actual sensitivity and specificity of the neurosphere assay in stem cell biology. Nat Meth. 3 (10), 801-806 (2006).
- Santa-Olalla, J., Baizabal, J., Fregoso, M., Cárdenas, M. D. C., Covarrubias, L. The in vivo positional identity gene expression code is not preserved in neural stem cells grown in culture. Eur J Neurosci. 18 (5), 1073-1084 (2003).
- Pastrana, E., Silva-Vargas, V., Doetsch, F. Eyes wide open: A critical review of sphere-formation as an assay for stem cells. Cell Stem Cell. 8 (5), 486-498 (2011).
- Wan, F., et al. The utility and limitations of neurosphere assay, CD133 immunophenotyping and side population assay in Glioma stem cell research. Brain Pathol. 20 (5), 877-889 (2010).
- Deleyrolle, L. P., Rietze, R. L., Reynolds, B. A. The neurosphere assay, a method under scrutiny. Acta Neuropsych. 20 (1), 2-8 (2008).
- Campos, L. S. Neurospheres: Insights into neural stem cell biology. Neurosci Res. 78 (6), 761-769 (2004).
- Reynolds, B. A., Rietze, R. L. Neural stem cells and neurospheres—re-evaluating the relationship. Nat Meth. 2 (5), 333-336 (2005).
- Parker, M. A. Expression profile of an operationally-defined neural stem cell clone. Exp Neurol. 194 (2), 320-332 (2005).
- Belenguer, G., et al. Adult neural stem cells are alerted by systemic inflammation through TNF-α receptor signaling. Cell Stem Cell. 28 (2), 285-299 (2021).
- Cruz, M. T. G., Simões, S., Lima, M. C. P. de Improving lipoplex-mediated gene transfer into C6 glioma cells and primary neurons. Exp Neurol. 187 (1), 65-75 (2004).
- Hagemann, C., et al. High efficiency transfection of glioma cell lines and primary cells for overexpression and RNAi experiments. J Neurosci Meth. 156 (12), 194-202 (2006).
- Moritz, S., Lehmann, S., Faissner, A., Holst, A. von An induction gene trap screen in neural stem cells reveals an instructive function of the niche and identifies the splicing regulator Sam68 as a Tenascin-C-regulated target gene. Stem Cells. 26 (9), 2321-2331 (2008).
- Montalbán-Loro, R., et al. TET3 prevents terminal differentiation of adult NSCs by a non-catalytic action at Snrpn. Nat Comm. 10 (1), 1726(2019).
- Gritti, A., et al. Epidermal and fibroblast growth factors behave as mitogenic regulators for a single multipotent stem cell-like population from the subventricular region of the adult mouse forebrain. J Neurosci. 19 (9), 3287-3297 (1999).
- Nieto-Estévez, V., Pignatelli, J., Araúzo-Bravo, M. J., Hurtado-Chong, A., Vicario-Abejón, C. A global transcriptome analysis reveals molecular hallmarks of neural stem cell death, survival, and differentiation in response to partial FGF-2 and EGF deprivation. PLoS One. 8 (1), 53594(2013).
- Vergaño-Vera, E., Méndez-Gómez, H. R., Hurtado-Chong, A., Cigudosa, J. C., Vicario-Abejón, C. Fibroblast growth factor-2 increases the expression of neurogenic genes and promotes the migration and differentiation of neurons derived from transplanted neural stem/progenitor cells. Neuroscience. 162 (1), 39-54 (2009).
- Gabay, L., Lowell, S., Rubin, L. L., Anderson, D. J. Deregulation of dorsoventral patterning by FGF confers trilineage differentiation capacity on CNS stem cells in vitro. Neuron. 40 (3), 485-499 (2003).
- Hack, M. A., Sugimori, M., Lundberg, C., Nakafuku, M., Götz, M. Regionalization and fate specification in neurospheres: the role of Olig2 and Pax6. Mol Cell Neurosci. 25 (4), 664-678 (2004).
- Bithell, A., Finch, S. E., Hornby, M. F., Williams, B. P. Fibroblast growth factor 2 maintains the neurogenic capacity of embryonic neural progenitor cells in vitro but changes their neuronal subtype specification. Stem Cells. 26 (6), 1565-1574 (2008).
- Pastrana, E., Cheng, L. C., Doetsch, F. Simultaneous prospective purification of adult subventricular zone neural stem cells and their progeny. Proc Natl Acad Sci. 106 (15), 6387-6392 (2009).
- Kobayashi, N., et al. Gene delivery to embryonic stem cells. Birth Def Res C. 75 (1), 10-18 (2005).
- Gresch, O., Altrogge, L. Transfection of difficult-to-transfect primary mammalian cells. Meth Mol Biol. 801, 65-74 (2011).
- Maasho, K., Marusina, A., Reynolds, N. M., Coligan, J. E., Borrego, F. Efficient gene transfer into the human natural killer cell line, NKL, using the Amaxa nucleofection system. J Immunol Meth. 284 (1-2), 133-140 (2004).
- von Holst, A., Egbers, U., Prochiantz, A., Faissner, A. Neural stem/progenitor cells express 20 tenascin C isoforms that are differentially regulated by Pax6. J Biol Chem. 282 (12), 9172-9181 (2007).
- Woodard, L. E., Wilson, M. H. piggyBac-ing models and new therapeutic strategies. Trend Biotechnol. 33 (9), 525-533 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados