Method Article
Isolamento, espansione e nucleofezione di cellule staminali neurali dalla zona subventricolare murina adulta
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Qui, descriviamo un sistema di nucleofecazione progettato per migliorare l'efficienza del rilascio genico in cellule staminali neurali espanse (NSC) isolate dalla zona subventricolare murina adulta. I risultati dimostrano che questo metodo migliora significativamente la perturbazione genica nelle NSC, superando l'efficacia dei protocolli di trasfezione tradizionali e migliorando il tasso di sopravvivenza cellulare.
Abstract
L'isolamento e l'espansione delle cellule staminali neurali (NSC) dalla zona subventricolare (SVZ) del cervello del topo adulto possono essere ottenuti in un mezzo integrato con il fattore di crescita dei fibroblasti basici (bFGF) e il fattore di crescita epidermico (EGF) come mitogeni, producendo aggregati clonali noti come neurosfere. Questo sistema in vitro è uno strumento prezioso per studiare il potenziale delle NSC. La trasfezione di siRNA o geni trasportati nei plasmidi può essere utilizzata per indurre perturbazioni nell'espressione genica e studiare la biologia delle NSC. Tuttavia, la somministrazione di acido nucleico esogeno alle colture di NSC è difficile a causa della bassa efficienza della trasfezione delle cellule del sistema nervoso centrale (SNC). Qui, presentiamo un sistema di nucleofezione migliorato che raggiunge un'elevata efficienza di consegna genica in NSC espanse da SVZ murina adulta. Dimostriamo che questo metodo relativamente semplice migliora la perturbazione genica nelle NSC adulte, superando i tradizionali protocolli di trasfezione con tassi di sopravvivenza superiori all'80%. Inoltre, questo metodo può essere applicato anche nelle NSC primarie isolate, fornendo un progresso cruciale negli studi sulla funzione genica attraverso la manipolazione dell'espressione genica tramite knockdown o sovraespressione in colture di neurosfera.
Introduzione
Le cellule staminali neurali (NSC) sono cellule staminali multipotenti residenti nel cervello. Queste cellule possiedono la capacità di auto-rinnovarsi e differenziarsi nei tre lignaggi neurali: astrociti, oligodendrociti e neuroni1. Di conseguenza, le NSC svolgono un ruolo cruciale nella neurogenesi adulta nei mammiferi, un processo in cui vengono generati nuovi neuroni nel cervello2. Le NSC risiedono prevalentemente in due regioni all'interno del cervello adulto denominate nicchie neurogene: la zona subventricolare (SVZ) lungo le pareti dei ventricoli laterali e la zona subgranulare (SGZ) all'interno del giro dentato dell'ippocampo 3,4. Le NSC (note anche come cellule B) nella SVZ, la nicchia neurogena più attiva nel topo adulto, si auto-rinnovano e producono progenitori che amplificano il transito (TAP o cellule C) che successivamente si differenziano in neuroblasti (cellule A). Questi neuroblasti migrano attraverso il flusso migratorio rostrale (RMS) fino ai bulbi olfattivi (OB), dove subiscono una differenziazione completa in interneuroni, integrandosi nei circuiti preesistenti 3,4,5,6.
Comprendere l'intricata interazione di segnali e segnali molecolari che regolano le NSC all'interno di queste nicchie è importante per sfruttare il loro potenziale per le applicazioni terapeutiche. A tal fine, sono stati sviluppati vari metodi per studiare questa popolazione cellulare, che vanno dalla coltura primaria selettiva di NSC alla selezione cellulare utilizzando marcatori di superficie 7,8,9,10. Il presente manoscritto descrive in dettaglio l'isolamento e la coltura di NSC SVZ in vitro utilizzando un terreno selettivo privo di siero contenente entrambi i mitogeni: il fattore di crescita dei fibroblasti basici (bFGF) e il fattore di crescita epidermico (EGF). Questo mezzo facilita la proliferazione cellulare e mantiene la staminalità delle NSC ottenute dalla SVZ di cervelli di topo adulto che formano aggregati clonali, tridimensionali e non aderenti in vitro noti come neurosfere9. Le colture della neurosfera fungono da piattaforma controllata per manipolare e studiare i meccanismi molecolari e i fattori che influenzano la proliferazione, l'auto-rinnovamento, la differenziazione e la sopravvivenza delle NSC11. In particolare, il numero di neurosfere primarie formate nella coltura consente una stima del numero di NSC presenti nella SVZ in vivo, rendendola un potente strumento per studiare gli effetti di diverse condizioni sul pool di NSC adulto12,13. Inoltre, una volta stabilita la coltura primaria, le NSC possono generare nuovi aggregati (neurosfere secondarie) dopo aver attraversato divisioni simmetriche in condizioni di proliferazione14. Pertanto, la semina a bassa densità di cellule della neurosfera secondaria (saggio clonale) può essere utilizzata per valutare il tasso di auto-rinnovamento di queste colture 4,15,16,17.
Nonostante il potenziale delle neurosfere nello scoprire i meccanismi che regolano la regolazione delle NSC, alcuni ricercatori mettono in dubbio la validità dei risultati in vitro, sostenendo che le condizioni artificiali in cui crescono le cellule potrebbero non replicare fedelmente l'intricato microambiente in vivo delle nicchie neurogene 18,19,20,21,22. Un altro punto controverso ruota attorno all'eterogeneità osservata nelle neurosfere. Tuttavia, si ritiene che questa variabilità rispecchi le divisioni simmetriche e asimmetriche delle NSC che si verificano naturalmente in vivo23,24. Inoltre, recenti validazioni hanno supportato l'utilizzo di colture di NSC per prevedere meccanismi che operano all'interno della nicchia neurogena SVZ in vivo, e diversi studi hanno dimostrato che le NSC coltivate in vitro mantengono accuratamente il profilo trascrittomico osservato in vivo11,25.
Pertanto, le colture della neurosfera non servono solo come metodo per esplorare la proliferazione e le capacità di differenziazione delle NSC, ma offrono anche un sistema per studiare l'influenza dei geni che governano la biologia delle NSC. Una tecnica fondamentale per studiare la funzione genica nelle NSC è la perturbazione dell'espressione genica. I siRNA o i geni somministrati attraverso i plasmidi possono essere trasfettati in colture cellulari, con conseguente knockdown o sovraregolazione del gene bersaglio. Questo approccio versatile riduce significativamente i tempi e i costi rispetto alla creazione di colture utilizzando topi knockout condizionali, presentando una strada promettente per svelare le basi genetiche della neurogenesi ed esplorare prospettive terapeutiche. L'alterazione dell'espressione di specifici geni nelle NSC consente la modulazione del loro comportamento, influenzando processi biologici cruciali come la proliferazione, la differenziazione e la migrazione. Tuttavia, la prospettiva di trasfettare le NSC, in particolare all'interno delle neurosfere di topo, presenta notevoli sfide. La struttura tridimensionale delle neurosfere compromette l'efficienza della trasfezione, spesso con conseguenti bassi tassi di successo della consegna di acido nucleico esogeno, che limita l'estensione della manipolazione genetica26,27. Inoltre, le procedure di trasfezione possono influire negativamente sulla vitalità e sulla funzionalità cellulare27. In questo contesto, presentiamo il sistema di nucleofenzione come un metodo per mitigare il danno cellulare, ottenendo un alto tasso di sopravvivenza e garantendo una maggiore efficacia nei saggi di consegna genica utilizzati per perturbare le colture di NSC.
Questo manoscritto si propone di illustrare la procedura per isolare, espandere e nucleofectare le NSC dalla nicchia neurogena SVZ adulta per perturbare i geni utilizzando il sistema di coltura della neurosfera. Questo metodo supera l'efficacia dei protocolli di trasfezione tradizionali, presentando tassi di sopravvivenza significativamente più elevati e una migliore efficienza di consegna genica tra le cellule bersaglio.
Protocollo
Tutti gli esperimenti eseguiti con gli animali sono stati preventivamente approvati dal Comitato Etico dell'Università di Valencia e autorizzati dalla Conselleria de Agricultura, Ganadería y Pesca, Generalitat Valenciana (Spagna).
1. Coltura primaria delle neurosfere
- Preparazione dei reagenti
- Preparare una soluzione tampone del tampone salino fosfato di Dulbecco (DPBS) in acqua deionizzata e sterilizzare in autoclave. In alternativa, preparare 0,1 M PBS pH 7,4 aggiungendo 137 mM di NaCl, 2,7 mM KCl, 10 mM di Na2HPO4 e 1,8 mM di KH2PO4 in acqua deionizzata.
- Riempire due piastre da 12 pozzetti con DPBS precedentemente raffreddate e tenerle in ghiaccio: una verrà utilizzata per preservare l'intero cervello prima della dissesezione e l'altra conserverà le SVZ sezionate fino al processamento tissutale.
- Preparare la miscela enzimatica contenente papaina come descritto di seguito.
- Preparare la soluzione di dissociazione EDTA/L-cisteina nella soluzione salina bilanciata di Earl (EBSS) a una concentrazione finale di 0,2 mg/mL ciascuna. Per ottenere una dissoluzione completa, incubare la provetta in un bagno d'acqua a 37 °C.
- Sciogliere 12 U/mL di papaina in una soluzione di EDTA/L-cisteina. Si consideri che verranno utilizzati 500 μL di soluzione enzimatica per cervello.
- Introdurre la soluzione di papaina a bagnomaria a 37 °C per circa 20 minuti fino a quando l'enzima non si dissolve completamente.
- Sterilizzare con filtro la miscela enzimatica con una siringa e un filtro a pori da 0,22 μm e mantenerla a 4 °C fino al momento dell'uso.
NOTA: La papaina si attiva dopo 30 minuti a 37 °C e può essere conservata per 12 ore a 4 °C.
- Preparare il terreno di coltura di controllo aggiungendo la soluzione di miscela ormonale secondo la Tabella 1 e sterilizzarlo con filtro utilizzando filtri a pori di nitrocellulosa da 0,22 μm. Mantenere il fluido a 4 °C e scaldarlo a bagnomaria a 37 °C prima dell'uso.
- Preparare il terreno completo integrando il mezzo di controllo con EGF e bFGF appena prima dell'uso come da Tabella 1. Non filtrare l'intero mezzo, poiché i mitogeni possono andare persi con il filtraggio.
- Estrazione cerebrale e dissezione SVZ
NOTA: Ai topi è consentito l'accesso gratuito a cibo e acqua prima dell'eutanasia. Vengono utilizzati topi C57BL/6 di età compresa tra 8 e 16 settimane. Entrambi i sessi vengono utilizzati senza differenze evidenti nella preparazione.- Sterilizzare gli strumenti di dissezione in autoclave prima dell'intervento chirurgico. Quindi, immergi bisturi, pinzette, forbici e spatole in un becher riempito con etanolo al 70%. Pulire accuratamente ogni superficie di lavoro con etanolo al 70%.
- Sacrificare il topo per overdose di CO2 e confermare per lussazione cervicale. Spruzzare la testina con etanolo al 70% per ridurre al minimo la possibilità di contaminazione del tessuto cerebrale.
- Separa la testa del mouse dal resto del corpo usando le forbici. Tagliare la pelle sopra il cranio con piccole forbici lungo l'asse rostro-caudale fino a quando il cranio non è completamente scoperto.
- Esporre il cervello tagliando inizialmente il cranio lungo la sutura sagittale con piccole forbici e poi, rimuovere le ossa usando una pinzetta sottile. Fare attenzione a non causare danni al tessuto cerebrale sotto il cranio durante questo passaggio.
- Estrarre con cura il cervello dal cranio usando una spatola e metterlo in una piastra a 12 pozzetti contenente DPBS freddo. Tenere la piastra sul ghiaccio fino al completamento della dissezione.
- Trasferire l'intero cervello sul cuscinetto di silicone dove avverrà la dissezione e applicare alcune gocce di DPBS freddo sul cervello (Figura 1A).
- Utilizzare un bisturi per rimuovere l'ostetricia situata all'estremità rostrale del cervello e il cervelletto situato nella parte caudale, pur mantenendo la parte centrale del cervello contenente entrambi i ventricoli laterali (Figura 1B).
- Dividere il cervello lungo la fessura longitudinale in due emisferi e quindi procedere con la dissezione di entrambi gli emisferi separatamente (Figura 1C).
- Lavorando con un emisfero, riorientarlo in modo che l'area mediale sia rivolta verso l'alto. Aprire il cervello lungo la linea del corpo calloso, separando la corteccia e lo striato dall'ippocampo, dal setto e dal diencefalo, esponendo così i ventricoli laterali (Figura 1D).
NOTA: La SVZ si trova tra la cavità del ventricolo e lo striato, quindi i passaggi successivi mirano a isolare la SVZ nel modo più accurato possibile dal tessuto circostante. - Rimuovere l'ippocampo, il setto e il diencefalo seguendo il limite ventrale dei ventricoli (Figura 1E, F), il tessuto oltre le estremità rostrale e caudale della SVZ (Figura 1G) e la corteccia che segue il corpo calloso (Figura 1H, I).
- Inclinare il tessuto in modo che la SVZ sia rivolta lateralmente (Figura 1J), consentendo la rimozione del tessuto striatale sotto la SVZ (Figura 1K) per ottenere un sottile pezzo di tessuto contenente le NSC (Figura 1L).
- Conservare entrambe le SVZ sezionate dallo stesso topo in una piastra a 12 pozzetti contenente DPBS freddo e sterile fino all'inizio del trattamento tissutale.
NOTA: La dissezione di entrambe le SVZ da un cervello dovrebbe richiedere 10 minuti. Un tempo più lungo potrebbe influire sulla vitalità cellulare. Se è necessaria una maggiore resa delle colture, è possibile raggruppare SVZ da più topi della stessa età, sesso, background genetico e genotipo.
- Isolamento e semina delle NSC
- Tagliare ogni SVZ in 4-5 piccoli pezzi per facilitare la disgregazione del tessuto.
NOTA: Da questo passaggio in poi, l'intera procedura deve essere eseguita in condizioni sterili in una cappa a flusso laminare. - Trasferire le SVZ tritate in una provetta da centrifuga da 15 ml utilizzando una pipetta Pasteur di plastica sterile. Assicurarsi che non siano rimasti frammenti nel piatto.
- Lasciare sedimentare il tessuto sul fondo della provetta e rimuovere il DPBS rimanente.
- Aggiungere 500 μl di miscela enzimatica filtrata contenente papaina per cervello (2 SVZ) e incubare le provette a bagnomaria a 37 °C per 30 minuti.
- Dopo l'incubazione, aggiungere 5 mL di terreno di controllo preriscaldato a 37 °C a ciascun campione.
- Centrifugare i campioni a 300 x g per 5 minuti. Quindi, rimuovere con cautela il surnatante utilizzando una micropipetta o una pompa a vuoto.
- Aggiungere 1 mL di terreno di controllo e disaggregare meccanicamente accuratamente il pellet, pipettando su e giù da 10 a 20 volte, utilizzando una pipetta in vetro Pasteur lucidata a fuoco fino ad ottenere una sospensione cellulare omogenea.
ATTENZIONE: Questo è uno dei passaggi più critici del protocollo, poiché una disaggregazione eccessiva o insufficiente influirà negativamente sulla resa delle colture. - Aggiungere 4 mL di terreno di controllo e mescolare per inversione per lavare le cellule. Centrifugare a 300 x g per 5 min. Quindi, rimuovere il surnatante.
- Aggiungere 1 mL di terreno completo e risospendere il pellet pipettandolo su e giù 10 volte per omogeneizzare la sospensione cellulare.
- Contare la sospensione cellulare utilizzando una camera Neubauer al microscopio dopo aver diluito 1/2 aliquota della sospensione cellulare con il blu di tripano o utilizzare un contatore automatico di cellule. Assicurarsi che le colture siano disaggregate a livello di singola cellula.
- Una volta preparata la sospensione cellulare, seminare le cellule ottenute in modo uniforme in 8 pozzetti di una piastra a 48 pozzetti in un volume finale di 500 μl di terreno completo.
NOTA: Di solito otteniamo 20.000 cellule da due SVZ, con una densità di semina finale di circa 2.500 cellule/mL per ottenere la massima resa nelle colture.
- Tagliare ogni SVZ in 4-5 piccoli pezzi per facilitare la disgregazione del tessuto.
- Formazione delle neurosfere
- Incubare le cellule per 5-7 giorni in vitro (DIV) in un incubatore umidificato a 37 °C e 5% di CO2. In queste condizioni, le cellule differenziate muoiono mentre le NSC e i progenitori proliferano in risposta alla stimolazione mitogenica, formando aggregati clonali riconosciuti come neurosfere primarie.
NOTA: Sia EGF che bFGF possono essere utilizzati per indurre la formazione di neurosfere. Tuttavia, questi due fattori di crescita non sono del tutto sostituibili. - Controlla le dimensioni delle neurosfere utilizzando una fotocamera accoppiata al microscopio invertito e, una volta che le neurosfere raggiungono un intervallo di dimensioni compreso tra 50 e 100 μm, conta tutte le neurosfere primarie formate nel pozzetto con un ingrandimento di 10x. Il numero totale di neurosfere primarie formate serve come stima del numero di NSC presenti nella nicchia SVZ.
- Dopo un periodo di 10 giorni, raccogliere il terreno di coltura da ciascun pozzetto per iniziare il passaggio. Condurre il passaggio successivo dopo 5-7 DIV, anche se vale la pena notare che le sfere primarie potrebbero richiedere una durata leggermente più lunga per raggiungere la dimensione ideale per la subcoltura (circa 100 μm).
- Incubare le cellule per 5-7 giorni in vitro (DIV) in un incubatore umidificato a 37 °C e 5% di CO2. In queste condizioni, le cellule differenziate muoiono mentre le NSC e i progenitori proliferano in risposta alla stimolazione mitogenica, formando aggregati clonali riconosciuti come neurosfere primarie.
Tabella 1: Soluzioni per i terreni di coltura. Viene descritta la ricetta del mezzo di controllo. Viene prodotta una soluzione di miscela ormonale premiscelata. Le soluzioni ormonali stock possono essere preparate in anticipo come indicato e conservate a -20 °C. Prima della coltura cellulare, integrare il terreno di controllo con EGF e bFGF per preparare il terreno completo. Per ogni componente vengono fornite le specifiche di stoccaggio, le scorte e le concentrazioni di lavoro. Clicca qui per scaricare questa tabella.
2. Espansione delle colture della neurosfera
- Passaggio delle neurosfere
- Raccogliere il terreno contenente le neurosfere dalle piastre multipozzetto e trasferirle in una provetta da centrifuga da 15 mL. Garantire il recupero del maggior numero possibile di cellule risciacquando i pozzetti con pochi millilitri di DPBS.
- Centrifugare le neurosfere per 5 minuti a 300 x g. Quindi, rimuovere il surnatante.
- Aggiungere 200 μl di soluzione enzimatica (vedere la tabella dei materiali) al pellet e picchiettare delicatamente il fondo della provetta per rimuoverlo. Incubare per 10 minuti a temperatura ambiente per facilitare la dissociazione delle neurosfere.
- Aggiungere 1 mL di terreno di controllo per arrestare la reazione enzimatica e dissociare meccanicamente le neurosfere con una micropipetta P1000 pipettando su e giù da 10x a 20x.
- Aggiungere un volume aggiuntivo di 4 mL del terreno di controllo e mescolare per inversione per lavare le cellule.
- Centrifugare le celle per 5 minuti a 300 x g. Quindi, rimuovere il surnatante.
- Aggiungere 1 mL di terreno completo e risospendere il pellet per omogeneizzare la sospensione cellulare pipettando su e giù 10 volte.
NOTA: Se il numero e la dimensione originali delle neurosfere sono troppo alti, aggiungere un terreno più completo per diluire la sospensione cellulare garantendo una conta cellulare affidabile. - Contare la sospensione cellulare utilizzando una camera Neubauer dopo aver diluito 1/2 aliquota della sospensione cellulare con il blu di tripano o utilizzare un contatore automatico di cellule.
- Per espandere la coltura cellulare, seminare le cellule a una densità di 10.000 cellule/cm2 utilizzando un terreno completo in una piastra di coltura o in un pallone appropriato (vedi Tabella 2).
- Incubare le cellule per 5-7 DIV in un incubatore umidificato a 37 °C e 5% di CO2 per ogni passaggio.
NOTA: Le colture di neurosfere possono essere costantemente espanse e propagate attraverso ripetute subcolture, non superiori a 10 passaggi, senza alterare le loro capacità di proliferazione. Per la coltura possono essere utilizzati sia EGF che bFGF; tuttavia, l'espansione a lungo termine delle colture può essere realizzata solo in presenza del FEG.
- Saggio di autorinnovamento
- Dopo aver passato le colture della neurosfera, piastrate 1.000 singole cellule in una piastra a 96 pozzetti, completando fino a un volume finale di 200 μL con terreno completo, ottenendo una densità cellulare finale delle colture di 5 cellule/μL.
NOTA: Il numero di cellule seminate deve essere il più accurato possibile. Se necessario, preparare diluizioni intermedie per garantire un volume di sospensione cellulare affidabile prima della placcatura. Inoltre, si consiglia di placcare da 4 a 5 repliche per condizione per ridurre la variabilità tecnica. - Incubare le cellule a 37 °C e 5% di CO2 in un incubatore umidificato per 5 giorni o fino a quando le neurosfere raggiungono una dimensione adeguata (da 60 a 100 μm). Evitare di spostare o scuotere la piastra multipozzetto per evitare l'aggregazione della neurosfera.
- Dopo la formazione della neurosfera, contare il numero di neurosfere formate in ciascun pozzetto utilizzando un microscopio a contrasto di fase invertito dotato di una fotocamera collegata. Il test di autorinnovo può essere condotto utilizzando EGF o bFGF separatamente.
- Dopo aver passato le colture della neurosfera, piastrate 1.000 singole cellule in una piastra a 96 pozzetti, completando fino a un volume finale di 200 μL con terreno completo, ottenendo una densità cellulare finale delle colture di 5 cellule/μL.
- Crioconservazione delle colture NSC
NOTA: Le colture di neurosfere possono essere crioconservate per un uso futuro, riducendo al minimo il numero di topi necessari. Si consiglia di conservare le celle fino al passaggio 10.- Per crioconservare le cellule, trasferire 1 mL della sospensione cellulare in un terreno completo in una crioprovetta adeguatamente etichettata.
- Aggiungere il dimetilsolfossido (DMSO) a una concentrazione finale del 10% alla sospensione cellulare e mescolare delicatamente capovolgendo le provette.
NOTA: Il DMSO funge da crioprotettore ma può essere tossico per le cellule, quindi questo passaggio deve essere eseguito rapidamente. - Porre le provette in un congelatore a -80 °C utilizzando un contenitore di congelamento che consenta una diminuzione graduale della temperatura a 1 °C/min. Questo processo di congelamento graduale previene i danni cellulari causati dalla formazione di cristalli di ghiaccio durante la crioconservazione. Per la conservazione a lungo termine, mantenere le provette in un serbatoio di azoto liquido a -196 °C.
- Scongelamento delle colture NSC
NOTA: Quando necessario, le cellule possono essere scongelate e le colture possono essere ri-espanse per gli esperimenti.- Rimuovere le crioprovette contenenti NSC congelate dal serbatoio di stoccaggio dell'azoto liquido e metterle immediatamente in un bagno d'acqua a 37 °C fino a completo scongelamento, circa 5 minuti.
ATTENZIONE: È fondamentale ridurre al minimo il tempo del bagno poiché il DMSO è tossico e può influire sulla vitalità cellulare. - Trasferire rapidamente le cellule scongelate in una provetta da centrifuga da 15 mL contenente 5 mL di terreno di controllo preriscaldato.
- Centrifugare le celle a 300 x g per 5 minuti e rimuovere il surnatante. Aggiungere 1 mL di terreno completo e risospendere il pellet pipettando delicatamente su e giù 10 volte per omogeneizzare la sospensione cellulare prima della semina.
NOTA: Lo scongelamento può influire sulla vitalità cellulare; Pertanto, si consiglia di far passare le cellule almeno una volta prima di seminare per nuovi esperimenti.
- Rimuovere le crioprovette contenenti NSC congelate dal serbatoio di stoccaggio dell'azoto liquido e metterle immediatamente in un bagno d'acqua a 37 °C fino a completo scongelamento, circa 5 minuti.
- Test del micoplasma
NOTA: La contaminazione da micoplasma può compromettere la salute delle cellule, influenzando i tassi di crescita. Pertanto, il test del micoplasma è fondamentale per garantire l'integrità e l'affidabilità degli esperimenti.- Estrazione del DNA
- Raccogliere un minimo di 100 μl di coltura cellulare per l'estrazione del DNA. Sia il surnatante che il pellet cellulare possono essere utilizzati per l'estrazione del DNA. Conservare questa aliquota a -20 °C fino all'estrazione.
- Forare il coperchio del tubo e far bollire l'aliquota a 99 °C per 15 minuti utilizzando un termoblocco.
NOTA: La perforazione dei coperchi delle provette evita l'apertura delle provette durante l'ebollizione ad alte temperature. - Centrifugare per 5 min a 16.900 x g. Trasferire il surnatante contenente il DNA in una nuova provetta. Conservare la provetta con il DNA a -20 °C fino al test PCR.
- PCR per la rilevazione di micoplasmi
- Preparare la miscela per PCR utilizzando i seguenti componenti: 8,9 μL di acqua priva di nucleasi, 3 μL di tampone di reazione 5x, 1,2 μL di 25 mM di MgCl2, 0,25 μL di 2,5 mM di dNTP, 0,15 μL di Taq polimerasi e 0,25 μL di ciascun primer specifico per micoplasma a una concentrazione di 10 μM (Forward, Fw: 5'GATGTTTAGCCGGGTCGAGAG3' e Reverse, Rv: 5'GATGTCAAGAGTGGGTAAGGTT3').
- Aggiungere 1 μL di DNA estratto genomicamente. Preparare la miscela PCR in una cappa a flusso laminare per evitare la contaminazione del DNA.
- Incubare la reazione in un termociclatore come segue: denaturazione iniziale a 95 °C per 5 minuti seguita da 35 cicli di denaturazione a 95 °C per 30 s, ricottura a 53 °C per 1 min, estensione a 72 °C per 30 s e un'estensione finale a 72 °C per 5 min.
- Per identificare la dimensione della banda risultante (circa 500 bp), caricare 15 μL del prodotto DNA PCR miscelato con 3 μL di soluzione colorante di caricamento 6x su un gel di agarosio al 2% ed eseguire l'elettroforesi per 40-50 minuti a 100 V.
- Estrazione del DNA
Tabella 2: Piastre e fiaschi utilizzati per la coltura. Vengono fornite le dimensioni e i volumi delle piastre di semina e dei palloni di semina più comunemente utilizzati. La tabella include il diametro, l'area di crescita e il volume medio utilizzato per ciascun vaso, insieme a un esempio del numero di NSC da seminare in condizioni di espansione (10.000 cellule/cm2). Clicca qui per scaricare questa tabella.
3. Nucleofeczione delle colture della neurosfera
- Preparazione dei reagenti
- Preparazione del DNA
- Preparare 25 μL di cellule DH5α di E. coli competenti in una provetta da 1,5 mL per la trasformazione.
- Aggiungere 1 μL del plasmide di interesse (a 100-200 ng/μL) alle cellule e sottoporle a uno shock termico a 42 °C per 45 s. Qui, vengono utilizzati un plasmide contenente una forcina corta (sh)RNA per silenziare l'espressione di Snrpn e un plasmide di controllo shSCRAMBLE.
NOTA: Mantenere le cellule competenti sul ghiaccio durante tutta la procedura per mantenere l'efficacia della trasformazione. - Placcare le cellule trasformate su piastre di agar LB con l'antibiotico appropriato e incubarle a 37 °C per 18 ore. Tenere le piastre di Petri capovolte per evitare la formazione di goccioline dovute alla condensa, che potrebbe disturbare le colonie batteriche.
NOTA: Aggiungere antibiotici appropriati al terreno di coltura LB per consentire esclusivamente la crescita di batteri trasformati. - Utilizzando un puntale di pipetta sterile, prelevare le singole colonie batteriche e trasferirle in un pallone di Erlenmeyer precedentemente riempito con 250 ml di LB per avviare una coltura liquida. Evita di scegliere piccole colonie satelliti posizionate intorno alle colonie bersaglio più grandi, poiché potrebbero non aver incorporato il plasmide desiderato.
- Incubare il matraccio agitando energicamente su un agitatore orbitale a 37 °C per una notte.
- Utilizza un kit maxiprep privo di endotossine ed estrai il DNA plasmidico puro dalle colture batteriche seguendo le istruzioni del produttore. Ricostituire il DNA nel volume appropriato di acqua priva di endotossine per ottenere una concentrazione plasmidica da 1 a 2 μg/μL.
NOTA: La resa del maxiprep dipende dalla crescita della coltura. Di solito, la risospensione del pellet di DNA plasmidico in un volume di 200 μL si ottiene la concentrazione di DNA desiderata. - Conservare la provetta con il DNA plasmidico a -20 °C fino all'uso. Preparare una provetta da 1,5 mL con un volume finale di 5 μl contenente da 2 a 6 μg di DNA plasmidico destinato alla nucleofezione.
- Preparazione del materiale
- Preparare la soluzione di nucleofezione. Se si utilizza il kit consigliato (vedere la Tabella dei materiali), preparare 95 μl della soluzione di nucleofecazione in una provetta da 1,5 mL per ogni nucleofeczione desiderata. Pipette e cuvette sono solitamente incluse nel kit. Preparane uno per ogni condizione di nucleofezione.
- Preparare un pallone T25 riempito con 4 mL di terreno completo preriscaldato per condizione e conservarlo nell'incubatore fino al momento dell'uso.
- Preparazione del DNA
- Nucleofeczione delle NSC
- Utilizzare da 1 a 2 x 106 celle individuali per condizione per la valutazione. Centrifugare le cellule per 5 minuti a 300 x g, quindi rimuovere il surnatante.
- Aggiungere 1 mL di DPBS e risospendere il pellet pipettando su e giù 10 volte per garantire una sospensione cellulare omogenea.
- Ripetere la centrifugazione (passaggio 2.1.6) per un secondo lavaggio e infine risospendere il pellet in 95 μl di soluzione di nucleofezione.
NOTA: Le fasi di lavaggio sono fondamentali per rimuovere gli antibiotici dal mezzo di controllo e garantire un'efficace nucleofezione. - Combinare i 95 μl di cellule con una soluzione premiscelata contenente 5 μl di ciascun plasmide (2 μg totali) selezionato per la nucleofezione.
- Utilizzando una micropipetta P200, trasferire questa soluzione in una cuvetta e posizionarla nel dispositivo nucleofettore.
- Nucleotecare le cellule con i plasmidi desiderati utilizzando il programma NSC ottimizzato del sistema nucleofettore.
- Dopo aver completato l'elettroporazione, introdurre con cura il terreno completo caldo nella cuvetta utilizzando le pipette Pasteur fornite nel kit. Successivamente, trasferire il contenuto della cuvetta in un pallone T25 precedentemente preparato contenente un mezzo completo preriscaldato. Dopo l'elettroporazione, potrebbe esserci la morte cellulare, la precipitazione del DNA e la formazione di un aggregato viscoso. Evitare di trasferirlo nel pallone T25, in quanto potrebbe ridurre la sopravvivenza cellulare.
NOTA: Questo passaggio è fondamentale e deve essere eseguito il più rapidamente possibile per garantire un'elevata resa di nucleofezione e la successiva sopravvivenza della coltura. - Incubare le cellule nucleofettate a 37 °C e 5% di CO2 in un incubatore umidificato per 3-5 giorni.
- Selezione di cellule nucleofettate
- Strategia A: Selezione della citometria a flusso mediante selezione cellulare attivata da fluorescenza (FACS)
- Dopo 3-5 giorni dalla nucleofezione, passare le colture nucleofectate seguendo la fase 2.1 e raccogliere tutte le cellule individualizzate ottenute.
- Filtrare i campioni attraverso un filtro cellulare da 40 μm per rimuovere gli aggregati cellulari e analizzarli con il citometro a flusso cell sorter.
NOTA: È fondamentale ordinare lo stesso numero di celle in ogni condizione per ottenere risultati comparativi. - Utilizzando il software del citometro, le cellule vive si basano su due misure: la luce diffusa in avanti (FSC-A) che corrisponde alla dimensione della cellula e la luce diffusa laterale (SSC-A) che corrisponde alla complessità intracellulare. Selezionare la popolazione di cellule viventi caratterizzate da alto FSC-A e alto SSC-A.
NOTA: Facoltativamente, aggiungere 0,1 μg/mL di 4′,6-diamidino-2-fenilindolo (DAPI) alla sospensione cellulare prima della selezione. Ciò consentirà di scartare le cellule morte e i corpi apoptotici dal gating. - Scartare i doppietti e le triplette cellulari tracciando i parametri FSC-A rispetto a FSC-H. Scegli gli eventi inclusi nella diagonale del grafico, che rappresentano le canottiere.
NOTA: Quando le cellule passano attraverso il laser, il citometro rileva le variazioni di tensione nel tempo. FSC-A rappresenta l'area del segnale e FSC-H indica l'altezza del picco. I singoletti mostrano una relazione proporzionale tra FSC-A e FSC-H (situati sulla diagonale del grafico), mentre gli aggregati cellulari mostrano un FSC-A maggiore rispetto a FSC-H a causa di un segnale più lungo. - Selezionare le cellule nucleofettate in base all'intensità di fluorescenza del gene reporter codificato dal plasmide utilizzato. Ad esempio, quando si utilizza GFP+, le cellule possono essere selezionate in base ai livelli di fluorescenza FITC-A rispetto al grafico SSC-A.
- Isolare il numero desiderato di cellule bersaglio per ogni condizione e raccoglierle direttamente in una provetta di raccolta riempita con terreno completo.
- NSC nucleofettate con seme a una densità di 10.000 cellule/cm2. Incubare le colture cellulari selezionate a 37 °C e 5% di CO2 in un incubatore umidificato.
- Strategia B: Selezione utilizzando la resistenza agli antibiotici codificata nel plasmide
- Dopo 48 ore di nucleofezione, aggiungere la concentrazione di antibiotico appropriata per selezionare le cellule resistenti.
NOTA: Prima di iniziare, si raccomanda di testare più concentrazioni dell'antibiotico su cellule non nucleofette. La concentrazione ottimale di antibiotici è la quantità più bassa alla quale vengono eliminate tutte le cellule non resistenti. Ad esempio, abbiamo determinato una concentrazione ottimale di 4 μg/mL di blasticidina antibiotica per le colture di NSC. - Incubare le cellule a 37 °C e 5% di CO2 in un incubatore umidificato per 48 ore.
- Dopo 48 ore di trattamento antibiotico, sottocoltivare le cellule nucleofettate secondo la fase 2.1. Se le neurosfere rimangono piccole, rimuovere l'antibiotico dal terreno di coltura mediante centrifugazione a 300 x g per 5 minuti. Risospendere le cellule e coltivarle in un pallone utilizzando solo un terreno completo. Incubare le cellule a 37 °C e 5% di CO2 in un incubatore umidificato fino a quando non possono essere espanse seguendo il passaggio 2.1.
- Dopo 48 ore di nucleofezione, aggiungere la concentrazione di antibiotico appropriata per selezionare le cellule resistenti.
- Strategia A: Selezione della citometria a flusso mediante selezione cellulare attivata da fluorescenza (FACS)
Risultati
Le condizioni ottimali di coltura consentono l'isolamento e l'espansione di NSC adulte derivate da SVZ in vitro
Le colture di NSC derivate dalla SVZ adulta sono servite come un valido metodo in vitro per studiare i meccanismi molecolari e i segnali di nicchia che regolano le NSC all'interno dei loro specifici microambienti. Il test della neurosfera descritto in questo manoscritto è stato impiegato per esaminare la conta delle NSC all'interno della SVZ adulta. Il tessuto SVZ è stato isolato dal cervello di topi di 3 mesi, dissociato e coltivato in terreno NSC completo integrato con EGF e bFGF o ciascuno separatamente. Dopo 10 giorni in vitro (DIV), la conta totale delle sfere primarie formate in queste tre distinte condizioni di coltura è stata quantificata utilizzando la microscopia a contrasto di fase (Figura 2A, B). Sorprendentemente, i nostri risultati dimostrano che la presenza di EGF ha portato alla massima formazione di sfere primarie, che è evidente dal ridotto numero di sfere primarie osservato in colture con solo bFGF (Figura 2A, B). Per valutare la capacità di auto-rinnovamento delle NSC in varie condizioni di terreno, le cellule sono state subcoltivate e piastrate a bassa densità (5 cellule/μL) in terreni integrati con le suddette combinazioni di mitogeni. La quantificazione delle sfere secondarie ha rivelato che le NSC SVZ hanno bisogno almeno di EGF per autorinnovarsi in modo efficiente e che la combinazione di EGF e bFGF migliora la capacità di auto-rinnovamento delle cellule (Figura 2B). Inoltre, per un'analisi dettagliata delle dinamiche di crescita in diverse condizioni di terreno, è stato registrato il numero di cellule seminate e ottenute in 7 passaggi. Le curve di crescita ottenute da diverse condizioni del terreno hanno confermato che le colture integrate con EGF da solo o in combinazione con bFGF hanno mostrato una migliore dinamica di crescita rispetto alle colture integrate esclusivamente con bFGF (Figura 2C). Nel complesso, questi risultati confermano che l'uso concomitante di EGF e bFGF migliora la resa di coltura di NSC in vitro.
Al fine di studiare la conta delle NSC all'interno della SVZ in diverse età postnatali e valutare l'impatto dell'invecchiamento dei topi sull'efficienza della coltura della neurosfera, è stato sezionato il tessuto SVZ di topi di età compresa tra 1 mese e 12 mesi. In particolare, i nostri risultati hanno rivelato un calo significativo del numero di sfere primarie con l'aumentare dell'età, mostrando la massima efficienza nel conteggio delle sfere a circa 2-4 mesi di età (Figura 2D). Inoltre, per valutare la capacità di auto-rinnovamento delle NSC durante la subcoltura, le NSC di topi di età compresa tra 2 e 4 mesi sono state subcoltivate e seminate a densità clonale (5 cellule/μL) in terreno completo. La quantificazione del numero di sfere secondarie nei passaggi successivi ha indicato una notevole diminuzione dell'efficienza della coltura nei passaggi (Figura 2E). Sulla base di tutte queste osservazioni, si raccomanda di condurre saggi di autorinnovamento durante i primi passaggi per ottimizzare ulteriormente le condizioni di coltura delle NSC.
La nucleofezione è una tecnica altamente efficiente per manipolare l'espressione genica nelle NSC adulte
Dato che le NSC non sono facilmente trasfecabili per manipolare l'espressione genica, qui presentiamo un protocollo di nucleofecazione con un tasso più elevato di consegna genica di successo senza la necessità di utilizzare la trasduzione virale. Dopo la dissezione e la coltura di singole SVZ da topi di 2-4 mesi, le NSC sono state nucleofettate con un plasmide portatore di GFP come descritto (Figura 3A). La rilevazione di cellule GFP-positive 2 giorni dopo la nucleofezione ha rivelato un'efficienza compresa tra il 30% e il 50%, in linea con la letteratura precedente28. Per isolare in modo specifico le NSC nucleofette, le cellule sono state ordinate mediante FACS in base all'intensità di fluorescenza della GFP da 3 a 5 giorni dopo la nucleofecazione (Figura 3A-C). Circa il 40% delle cellule analizzate prima della FACS ha mostrato alti livelli di fluorescenza GFP e sono state successivamente selezionate mediante smistamento (Figura 3C). La risemina di NSC GFP+ selezionate ha mostrato che tutte le cellule erano GFP positive, convalidando il metodo di isolamento cellulare basato sulla nucleofezione (Figura 3D). In particolare, le NSC nucleofettate hanno mantenuto la vitalità attraverso i passaggi successivi, confermando la fattibilità della nucleofezione per le NSC adulte. Questi risultati sottolineano l'efficacia della combinazione di nucleofezione e FACS per stabilire una coltura pura di NSC modificate.
Come esempio di modulazione genica nelle NSC adulte, è stata impiegata l'attenuazione genica tramite nucleofecazione a RNA a forcina corta (sh). In particolare, un shRNA che ha come bersaglio il gene N del polipeptide della ribonucleoproteina nucleare Small nuclear rikonucleoprotein , che trasporta un reporter CAG-GFP (shSNRPN), è stato nucleofettato in colture di NSC derivate da topi di 2 mesi. La sottoregolazione di Snrpn è stata confermata dalla qPCR e dall'immunocitochimica in cellule nucleofettesse con il plasmide shSNRPN, ma non per il plasmide di controllo shSCRAMBLE (Figura 3E, F)29. Per chiarire gli effetti della sottoregolazione di Snrpn sulla capacità di auto-rinnovamento delle NSC, è stato eseguito un saggio a bassa densità in cellule nucleofettate (Figura 3F, G). La quantificazione delle neurosfere secondarie in colture nucleofette, ha rivelato un aumento della capacità di formazione delle neurosfere in seguito alla sottoregolazione di Snrpn (Figura 3G). Questo saggio sottolinea la capacità della nucleofezione di manipolare l'espressione genica nelle NSC adulte e, più specificamente, identifica il ruolo di Snrpn nel mantenimento della staminalità nelle NSC adulte.
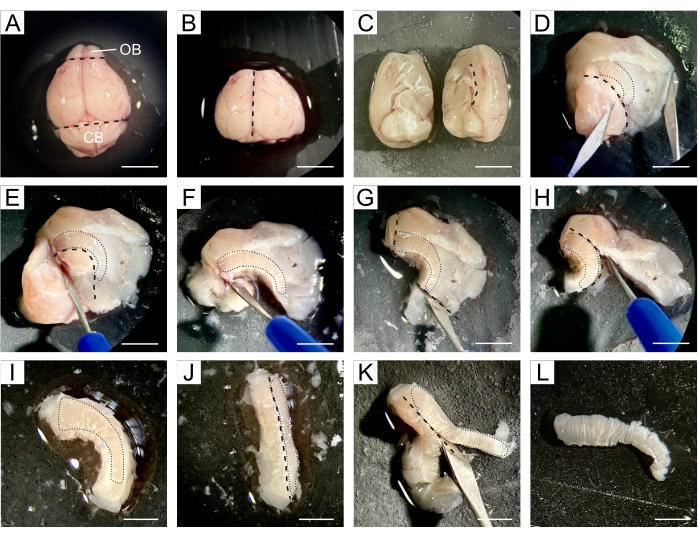
Figura 1: Descrizione dettagliata della dissezione SVZ. (A) Dopo aver rimosso il cervello del topo, l'intero cervello viene trasferito con DPBS su un tampone di silicone per la dissezione. (B) L'ostetricia e il cervelletto vengono rimossi dal cervello utilizzando un bisturi. (C) Il cervello viene diviso in due emisferi per procedere con la dissezione separatamente. (D) Il cervello viene aperto lungo la linea del corpo calloso, separando la corteccia e lo striato dall'ippocampo, dal setto e dal diencefalo, esponendo così i ventricoli laterali. (E-F) L'ippocampo, il setto e il diencefalo vengono rimossi seguendo il limite ventrale dei ventricoli. (G-I) Il tessuto oltre le estremità rostrale e caudale della SVZ e della corteccia viene rimosso seguendo il corpo calloso. (J-K) Il tessuto è inclinato in modo che la SVZ sia rivolta lateralmente, consentendo la rimozione del tessuto striatale sotto la SVZ. (L) Si ottiene un sottile pezzo di tessuto contenente la SVZ. Le linee tratteggiate nere indicano il sito di taglio. Le linee tratteggiate nere indicano la posizione della SVZ ad ogni passo. Abbreviazioni: OB = bulbo olfattivo; CB = cervelletto. Barra graduata in A-H: 5 mm; in I-L: 3 mm. Clicca qui per visualizzare una versione più grande di questa figura.
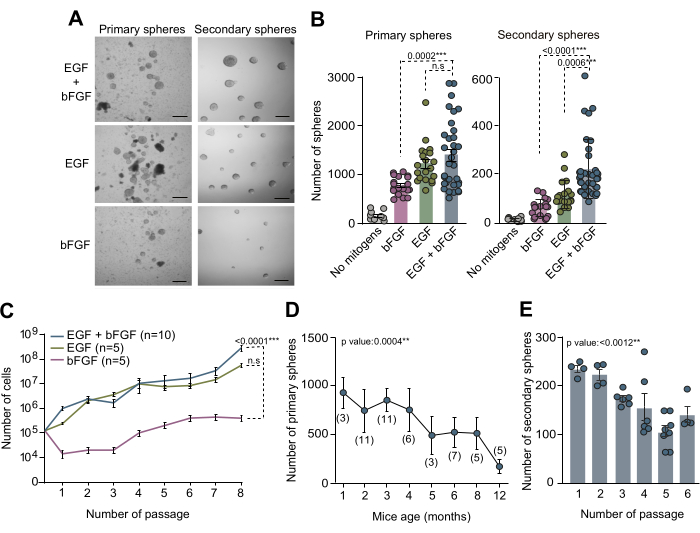
Figura 2: Entrambi i mitogeni EGF e bFGF sono necessari per una coltura ottimale e l'espansione delle NSC in vitro. (A) Immagini al microscopio a contrasto di fase di colture primarie e secondarie di neurosfere in terreno NSC integrate con EGF e bFGF o ciascun mitogeno separatamente. (B) Numero di sfere primarie ottenute per topo e per pozzetto in base alla composizione mitogena del terreno di coltura: nessun mitogeno (grigio, n=9), EGF+bFGF (blu, n=33), solo EGF (verde, n=20) o solo bFGF (rosa, n=19; pannello di sinistra). Numero di neurosfere secondarie formate a bassa densità (5 cellule/μL) per pozzetto in base alla composizione mitogena del terreno di coltura): nessun mitogeno (grigio, n=11), bFGF (rosa, n=18), EGF (verde, n=18) o EGF+bFGF (blu, n=37; pannello di destra). È stato utilizzato il test di Kruskal-Wallis con l'analisi post hoc di Dunn. (C) Curve di crescita che mostrano il numero totale di cellule formate dopo 8 passaggi in colture di neurosfere distinte in base ai mitogeni presenti nel terreno di coltura: bFGF (rosa, n=5), EGF (verde, n=5) o EGF+bFGF (blu, n=10). È stata utilizzata l'analisi di regressione lineare. (D) Il numero di neurosfere primarie derivate da singoli topi sezionati e disaggregati da animali in vari stadi di sviluppo postnatale. È stata utilizzata l'analisi di regressione lineare. n indicato tra parentesi. (E) Il numero di neurosfere secondarie formate a bassa densità (5 cellule/μL) attraverso successivi passaggi in vitro per valutare la loro capacità di auto-rinnovamento. È stato utilizzato il test di Kruskal-Wallis e i valori p sono inclusi: *: p<0,05; **: pag<0.01; : p<0.001; : pag<0,0001. N.S: Non significativo. Le barre di errore rappresentano il SEM. La barra della scala in A è di 100 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
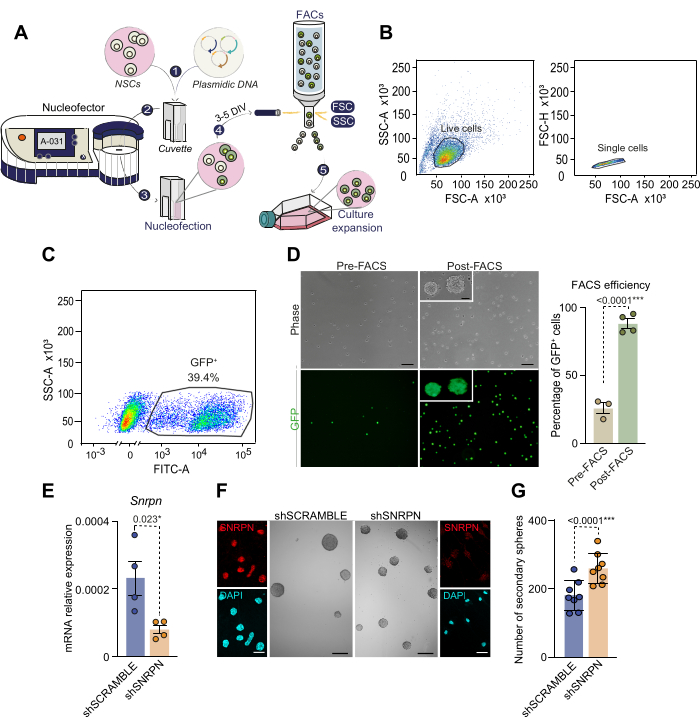
Figura 3: Schema del processo di nucleofezione e selezione cellulare basata su FACS di cellule nucleofette. (A) Cenni sul processo di nucleofecazione e sulla selezione cellulare di cellule nucleofectate basata su FACS. 1) La soluzione di DNA plasmidico viene combinata con la soluzione di nucleofezione e aggiunta alle cellule. Questa miscela viene quindi trasferita in una cuvetta per la nucleofezione. 2) La cuvetta contenente il DNA e le cellule viene inserita nel nucleoformatore, dove viene applicata una scossa elettrica. Per le colture di neurosfere isolate dalla SVZ adulta, si consiglia di utilizzare il programma A-031 NSC. 3) Dopo la nucleofezione, le cellule vengono trasferite in un pallone contenente terreno completo integrato con EGF e bFGF. 4) Le cellule vengono ordinate mediante FACS in base all'espressione del reporter contenuto nel DNA plasmidico. 5) Le cellule selezionate vengono sottoposte a un'ulteriore coltura per esperimenti successivi. (B) Strategia di selezione basata su FACS e gating per NSC nucleofettate. In primo luogo, le cellule viventi vengono selezionate in base a un alto FSC-A e ad un alto SSC-A (grafico a sinistra), quindi i doppietti e le triplette cellulari vengono eliminati analizzando i parametri FSC-A rispetto a FSC-H (grafico a destra). (C) Gating FACS per cellule nucleofectate in base alla loro espressione GFP (fluorescenza SSC-A versus FITC-A). (D) Microscopia a contrasto di fase e immagini a fluorescenza GFP di NSC nucleofettate prima (n=3) e dopo (n=4) la selezione FACS (pannello di sinistra). La percentuale di cellule GFP+ nucleofectate è illustrata prima e dopo la selezione FACS (pannello a destra). È stato utilizzato il test T. (E) Analisi quantitativa PCR (qPCR) dell'espressione di Snrpn in NSC proliferanti nucleofettesse con shRNA specifico per Snrpn (n=4). La nucleofeczione utilizzando uno shRNA SCRAMBLE è servita come controllo (n=4). È stato utilizzato il test T. (F) Immagini di immunocitochimica che mostrano SNRPN (in rosso) nelle NSC 7 giorni dopo la nucleofeczione dello shRNA (pannelli esterni). Il DAPI è stato utilizzato per contrastare la colorazione del DNA. Immagini rappresentative che ritraggono neurosfere derivate da NSC in condizioni shSNRPN e shSCRAMBLE (pannelli centrali). (G) Quantificazione del numero di neurosfere formate in colture di NSC dopo nucleofeczione con shSNRPN (n=8) o shSCRAMBLE (n=8). È stato utilizzato il test T. I valori p sono inclusi: *: p<0,05; **: pag<0.01; : p<0.001; : pag<0,0001. N.S: Non significativo. Le barre di errore rappresentano il SEM. Barra della scala in D, 10 μm; in F, 100 μm (immagini ad alto ingrandimento in F, 7 μm). Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
A causa della mancanza di marcatori definitivi per identificare la popolazione di NSC in vivo, l'analisi delle NSC si è basata principalmente sull'osservazione del comportamento di cellule isolate da nicchie neurogeniche in condizioni ex vivo . Il lavoro pionieristico di Reynolds e Weiss ha gettato le basi stabilendo condizioni di coltura precise, consentendo l'isolamento e l'espansione di singole cellule da tessuto SVZ di topo giovane adulto (2 mesi) in condizioni non adesive. Queste cellule vengono solitamente propagate in un terreno privo di siero contenente EGF e bFGF, condizioni che impediscono completamente la differenziazione in neuroni e glia promuovendo la proliferazione. Infatti, in queste condizioni di coltura9, la maggior parte delle cellule muore durante i primi giorni di coltura, ma un piccolo sottogruppo inizia a dividersi e forma principalmente neurosfere fluttuanti9. La dissociazione enzimatica e la subcoltura di questi aggregati cellulari facilitano la propagazione delle colture, dimostrando la capacità di autorinnovamento di queste colture.
In particolare, le colture di neurosfere mostrano un potenziale di espansione con l'aggiunta di solo EGF, mentre le NSC coltivate in un terreno contenente solo bFGF non mostrano una proliferazione cellulare a lungo termine30. Entrambe le prove indicano l'EGF come il principale mitogeno di autorinnovamento delle NSC. Tuttavia, la presenza di EGF e bFGF nel terreno di coltura migliora la capacità di auto-rinnovamento delle NSC31, oltre a contribuire a bilanciare il potenziale di differenziazione in astrociti, neuroni e oligodendrociti 32,33,34,35. Inoltre, l'uso di una miscela definita di ormoni e fattori invece di alternative commerciali per integrare il terreno garantisce l'alta qualità e riproducibilità delle colture di NSC. In queste condizioni, le colture di NSC di topo possono persistere come linee cellulari stabili senza subire l'immortalizzazione. Tuttavia, una preoccupazione nelle colture di neurosfere è il potenziale delle popolazioni cellulari proliferative di subire trasformazioni genetiche, bypassando i meccanismi di regolazione del ciclo cellulare e portando a un fenotipo immortalizzato. Pertanto, sebbene le colture di neurosfere siano altamente espandibili, le colture oltre i 10 passaggi in vitro vengono solitamente scartate per gli studi, poiché possono andare incontro a invecchiamento replicativo e potrebbero emergere cellule con contenuto cromosomico anormale o dinamiche di crescita irregolari. Inoltre, l'uso di colture di neurosfere consolidate a lungo termine deve essere continuamente monitorato. Le colture di neurosfere offrono anche l'opportunità di esaminare le loro caratteristiche e il loro potenziale in un ambiente controllato, fornendo un'impostazione più precisa e regolabile che può essere modulata e monitorata in modo più accurato rispetto al vivo. Attraverso analisi clonogeniche o di popolazione in vitro, è possibile quantificare le capacità di autorinnovamento e proliferazione di queste cellule, facilitando l'identificazione dei meccanismi sottostanti che regolano queste proprietà.
Tuttavia, nonostante l'ampio elenco di vantaggi derivanti dal lavorare con le NSC in vitro, la natura di questo protocollo di coltura e la delicatezza delle NSC costituiscono una sfida sul campo. Ad esempio, il numero di neurosfere primarie generate durante una dissezione tipica può variare in modo significativo in base all'abilità e alla precisione dello sperimentatore. I risultati della neurosfera primaria illustrano questa variabilità nel numero di neurosfere primarie ottenute dalla SVZ di topi di 2 mesi, che variano tra 500 e 3000 neurosfere. Vari fattori possono contribuire a questa variabilità. In primo luogo, la precisione della dissezione riduce al minimo il tessuto parenchimale indesiderato, che inibisce la formazione della sfera primaria. In secondo luogo, la generazione di piccoli pezzi di tessuto SVZ consente un'efficiente digestione e triturazione enzimatica, riducendo così la perdita cellulare. Ciò evidenzia la necessità di una sufficiente pratica preliminare del protocollo di dissezione e di uno sviluppo ottimizzato del processo tissutale durante l'istituzione delle colture della neurosfera.
Un'altra limitazione di queste colture risiede nel fatto che le neurosfere possono comprendere sia NSC che cellule progenitrici, rendendo difficile distinguere tra queste due popolazioni all'interno delle colture primarie. Sebbene sia stato riportato che vari marcatori come GFAP, nestina, Musashi e SOX2 sono espressi dalle NSC8, nessuno di questi è stato associato esclusivamente alle NSC. Le tecniche FACS emergenti basate sull'espressione dell'antigene sulla superficie cellulare hanno permesso l'isolamento delle NSC e della loro progenie. Questi studi hanno dimostrato che i progenitori che amplificano il transito non sono in grado di formare neurosfere al passaggio 25,36. Pertanto, mentre la relazione tra le cellule SVZ e le cellule che iniziano la neurosfera richiede un ulteriore raffinamento 18,19,20,21,22,23, la capacità delle neurosfere di essere passate in serie per un periodo prolungato può riflettere la presenza di NSC nelle colture.
Questo sistema di coltura della neurosfera è servito come modello robusto per studiare l'impatto delle vie di segnalazione e dell'espressione genica nel mantenimento della capacità di auto-rinnovamento delle NSC in vitro15,29. Un approccio per esplorare questi aspetti prevede la trasfetzione delle NSC per sovraesprimere o abbattere geni specifici. Ciò può essere ottenuto attraverso varie tecniche, inclusi metodi virali e non virali. Sebbene i vettori virali raggiungano spesso un'elevata efficienza di trasferimento genico, presentano importanti limitazioni, come gli elevati requisiti di sicurezza e la produzione di vettori che richiede tempo26. Al contrario, i metodi di trasfezione classici come la lipofezione e l'elettroporazione raggiungono tassi di trasfezione molto bassi, rendendoli impraticabili per le cellule difficili da trasfettare. La tecnologia di nucleofeczione offre un approccio di facile utilizzo combinando soluzioni di nucleofeczione specifiche per tipo di cella con parametri elettrici unici per ogni tipo di cella37,38. Ciò garantisce il trasferimento del DNA direttamente nel nucleo cellulare39, consentendo l'incorporazione del DNA in modo indipendente dal ciclo cellulare. Di conseguenza, la nucleofecazione emerge come una tecnica praticabile per la trasfetzione di cellule difficili da trasfettare come le NSC di topo 28,40.
Una limitazione di questo metodo è che per ogni nucleofecazione è raccomandato e necessario un minimo di 2 x 106 cellule, anche se in condizioni ottimali è possibile utilizzare un numero inferiore di cellule per singola nucleofecazione (cioè 5 × 105 cellule). Un altro svantaggio del metodo è che la qualità e la concentrazione del DNA utilizzato per la nucleofeczione influenzano in modo significativo l'efficienza del trasferimento genico. L'utilizzo di DNA preparato privo di endotossine è altamente raccomandato per prevenire l'elevata mortalità cellulare dovuta alla presenza di endotossine. Inoltre, l'utilizzo di più di 6 μg di DNA totale per la nucleofezione può ridurre sostanzialmente sia l'efficienza del trasferimento genico che la vitalità cellulare. Infine, anche la somministrazione di impulsi elettrici è fondamentale per la sopravvivenza delle cellule nucleofette.
In questo lavoro, abbiamo esemplificato la manipolazione dell'espressione genica di Snrpn impiegando una strategia che coinvolge la sottoregolazione della sua espressione utilizzando plasmidi episomiali. Questi plasmidi non sono integrati nel genoma delle cellule e, poiché le NSC continuano a proliferare in vitro, il DNA introdotto verrà successivamente diluito lungo la divisione cellulare e, quindi, perderà l'effetto della manipolazione genetica. Pertanto, questa strategia è preziosa per studiare l'effetto di un'alterazione acuta in un breve periodo o per dare alla luce cellule in vitro. Sono disponibili alcune alternative per valutare un effetto più prolungato della perturbazione genica. Ad esempio, potrebbe essere utilizzato un sistema integrativo come il piggyBAC basato su trasposone. Questo sistema consiste nell'introdurre la sequenza codificante desiderata contenuta nel plasmide affiancata da sequenze trasponibili e nel co-nucleoleggere le cellule con un plasmide contenente la sequenza per l'enzima trasposasi41. In alternativa, potrebbero essere utilizzati i trasposoni o i sistemi CRISPR/Cas.
L'ulteriore sviluppo delle tecnologie di trasfezione sarà un passo importante verso saggi ad alto rendimento per valutare il ruolo di diversi geni sulla fisiologia delle cellule staminali neurali adulte. In combinazione con metodi di purificazione ed espansione sempre più sofisticati, questi studi consentiranno la comprensione della biologia in vitro delle NSC adulte e il confronto delle differenze biologiche tra neurosfere galleggianti e NSC in vivo.
Divulgazioni
Gli autori dichiarano di non avere interessi finanziari concorrenti.
Riconoscimenti
Questo lavoro è stato sostenuto da sovvenzioni del Ministerio de Ciencia e Innovación e dell'Agencia Estatal de Investigación (MCIN/AEI) (PID2019-110045GB-I00; PID2022-142734OB-I00 e EUR2023-143479) a SRF. EJV (FPU20/00795) e DSL (FPU22/03797) sono finanziati dal programma di borse di studio spagnolo Formación de Profesorado Universitario (FPU). LLC (PRE2020-094137) e JDM (PREP2022-000680) sono finanziati dal programma di borse di studio spagnolo Formación de Personal Investigador (FPI). CMM (CIACIF/2022/366) è finanziato dalla Generalitat Valenciana. MIL (CPI-22-481) è finanziato dalla borsa di studio Programa INVESTIGO (Next Generation EU). Finanziamenti Open Access forniti dal Ministerio de Ciencia e Innovación.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.22 μm pore-filter bottles (250 mL) | VWR | 514-0330P | |
| 0.22 μm pore-filter bottles (500 mL) | VWR | 514-0332P | |
| 12-well plate | LabClinics | PLC30012 | |
| 15 mL tube | Fisher | 10738771 | |
| 24-well plate | LabClinics | PLC30024 | |
| 4-(2-hydroxyethyl)-1-piperazineethanesulfonic acid (HEPES) | BioWest | L0180-100 | |
| 4′,6-diamidino-2-phenylindole (DAPI) | Sigma | D9542 | |
| 48-well plate | ThermoFisher | 150687 | |
| 6-well plate | LabClinics | PLC30006 | |
| 96-well plate | Labclinics | PLC30096 | |
| Accutase solution | Sigma | A6964-100ML | Referred as enzymatic solution |
| Amaxa Mouse Neural Stem Cell Nucleofector Kit | Lonza | VPG-1004 | |
| Amaxa Nucleofector Iib | Lonza | 10700807 | |
| Antibiotic/Antimitotic (A/A) | Sigma | A5955 | |
| Apo-t-Transferrine | Sigma | T2252-1G | |
| Basic Fibroblast Growth Factor (bFGF) | Sigma | F0291 | |
| Blasticidin | Sigma | 203350 | |
| Bovine Serum Albumin (BSA) | Sigma | B4287-25MG | |
| Cell strainer 40 μm | LabClinics | PLC93040 | |
| Deoxynucleotide triphosphate (dNTPs) | NZYTech | MB08701 | |
| Dimethyl sulfoxide (DMSO) | Sigma | D8418 | |
| Dulbecco’s Phosphate Saline Buffer (DPBS) | Gibco | 14080-055 | |
| Dulbecco's Modified Eagle's Medium (DMEM) F12 1:1 | Gibco | 11320-074 | |
| E. coli DH5α Competent Cells | ThermoFisher | EC0112 | |
| Earles's Balanced Salt Solution (EBSS) | Gibco | 24010-043 | |
| Epidermal Growth Factor - Human recombinant (EGF) | Gibco | 53003-018 | |
| Ethylenediaminetetraacetic acid (EDTA) | Sigma | E-6511 | |
| Fine Forceps | Fine Science Tools | 11274-20 | |
| Fine Scissors Sharp | Fine Science Tools | 14060-09 | |
| Glucose | Sigma | G-7021 | |
| GoTaq G2 Flexi DNA Polymerase | Promega | M7805 | Kit includes reagents for PCR |
| Heparin | Sigma | H-3149 | |
| Insuline | Sigma | I6634 | |
| LB agar (Lennox) | LabKem | AGLB-00P-500 | |
| LB broth (Lennox) | LabKem | LBBR-00P-500 | |
| L-Cysteine | Sigma | C-8277 | |
| L-Glutamine | Gibco | 25030-024 | |
| Neubauer chamber | Blaubrand | BR718620 | |
| Nuclease free water | Labbox | WATR-00A-10K | |
| NZYMaxiprep Endotoxin Free Kit | NZYTech | MB39901 | |
| Papain Lyophilized | Worthington | LS003119 | |
| Progesterone | Sigma | P-6149 | |
| Putrescine | Sigma | P-7505 | |
| Scalpel | Fine Science Tools | 10316-14 | |
| shSCRAMBLE | Mission (Sigma) | SHC003 | |
| shSNRPN | Mission (Sigma) | TRCN0000109285 | |
| Sodium Selenite | Sigma | S-9133 | |
| Spatula | Fine Science Tools | 10090-17 | |
| Sterile PES Syringe Filters (0.22 μm pore-filter) | Epica | SFPE-22E-050 | |
| T12.5 cm2 Flask | Biofil | TCF012025 | |
| T25 cm2 Flask | LabClinics | PLC70025 | |
| T75 cm2 Flask | LabClinics | PLC70075 | |
| Tweezers | Fine Science Tools | 91150-20 |
Riferimenti
- Gage, F. H. Mammalian neural stem cells. Science. 287 (5457), 1433-1438 (2000).
- Ming, G., Song, H. Adult neurogenesis in the mammalian central nervous system. Ann Rev Neurosci. 28 (1), 223-250 (2005).
- Doetsch, F., Caillé, I., Lim, D. A., García-Verdugo, J. M., Alvarez-Buylla, A. Subventricular zone astrocytes are neural stem cells in the adult mammalian brain. Cell. 97 (6), 703-716 (1999).
- Obernier, K., et al. Adult neurogenesis is sustained by symmetric self-renewal and differentiation. Cell Stem Cell. 22 (2), 221-234 (2018).
- Doetsch, F., García-Verdugo, J. M., Alvarez-Buylla, A. Regeneration of a germinal layer in the adult mammalian brain. Proc Natl Acad Sci. 96 (20), 11619-11624 (1999).
- Doetsch, F., García-Verdugo, J. M., Alvarez-Buylla, A. Cellular composition and three-dimensional organization of the subventricular germinal zone in the adult mammalian brain. J Neurosci. 17 (13), 5046-5061 (1997).
- Mamber, C., Kozareva, D. A., Kamphuis, W., Hol, E. M. Shades of gray: The delineation of marker expression within the adult rodent subventricular zone. Prog Neurobiol. 111, 1-16 (2013).
- Cebrian-Silla, A., et al. Single-cell analysis of the ventricular-subventricular zone reveals signatures of dorsal and ventral adult neurogenesis. eLife. 10, e67436(2021).
- Reynolds, B. A., Weiss, S. Generation of neurons and astrocytes from isolated cells of the adult mammalian central nervous system. Science. 255 (5052), New York, N.Y. 1707-1710 (1992).
- Azari, H., Rahman, M., Sharififar, S., Reynolds, B. A. Isolation and expansion of the adult mouse neural stem cells using the neurosphere assay. J Vis Exp. (45), e2393(2010).
- Jensen, J. B., Parmar, M. Strengths and limitations of the neurosphere culture system. Mol Neurobiol. 34 (3), 153-161 (2006).
- Ferrón, S. R., et al. Postnatal loss of Dlk1 imprinting in stem cells and niche astrocytes regulates neurogenesis. Nature. 475 (7356), 381-385 (2011).
- Gil-Perotín, S., et al. Adult neural stem cells from the subventricular zone: A review of the neurosphere assay. Anat Record. 296 (9), 1435-1452 (2013).
- Obernier, K., Alvarez-Buylla, A. Neural stem cells: origin, heterogeneity and regulation in the adult mammalian brain. Development. 146 (4), 156059(2019).
- Ferrón, S. R., et al. A combined ex/in vivo assay to detect effects of exogenously added factors in neural stem cells. Nat Prot. 2 (4), 849-859 (2007).
- Ferron, S., et al. Telomere shortening and chromosomal instability abrogates proliferation of adult but not embryonic neural stem cells. Development. 131 (16), 4059-4070 (2004).
- Singec, I., et al. Defining the actual sensitivity and specificity of the neurosphere assay in stem cell biology. Nat Meth. 3 (10), 801-806 (2006).
- Santa-Olalla, J., Baizabal, J., Fregoso, M., Cárdenas, M. D. C., Covarrubias, L. The in vivo positional identity gene expression code is not preserved in neural stem cells grown in culture. Eur J Neurosci. 18 (5), 1073-1084 (2003).
- Pastrana, E., Silva-Vargas, V., Doetsch, F. Eyes wide open: A critical review of sphere-formation as an assay for stem cells. Cell Stem Cell. 8 (5), 486-498 (2011).
- Wan, F., et al. The utility and limitations of neurosphere assay, CD133 immunophenotyping and side population assay in Glioma stem cell research. Brain Pathol. 20 (5), 877-889 (2010).
- Deleyrolle, L. P., Rietze, R. L., Reynolds, B. A. The neurosphere assay, a method under scrutiny. Acta Neuropsych. 20 (1), 2-8 (2008).
- Campos, L. S. Neurospheres: Insights into neural stem cell biology. Neurosci Res. 78 (6), 761-769 (2004).
- Reynolds, B. A., Rietze, R. L. Neural stem cells and neurospheres—re-evaluating the relationship. Nat Meth. 2 (5), 333-336 (2005).
- Parker, M. A. Expression profile of an operationally-defined neural stem cell clone. Exp Neurol. 194 (2), 320-332 (2005).
- Belenguer, G., et al. Adult neural stem cells are alerted by systemic inflammation through TNF-α receptor signaling. Cell Stem Cell. 28 (2), 285-299 (2021).
- Cruz, M. T. G., Simões, S., Lima, M. C. P. de Improving lipoplex-mediated gene transfer into C6 glioma cells and primary neurons. Exp Neurol. 187 (1), 65-75 (2004).
- Hagemann, C., et al. High efficiency transfection of glioma cell lines and primary cells for overexpression and RNAi experiments. J Neurosci Meth. 156 (12), 194-202 (2006).
- Moritz, S., Lehmann, S., Faissner, A., Holst, A. von An induction gene trap screen in neural stem cells reveals an instructive function of the niche and identifies the splicing regulator Sam68 as a Tenascin-C-regulated target gene. Stem Cells. 26 (9), 2321-2331 (2008).
- Montalbán-Loro, R., et al. TET3 prevents terminal differentiation of adult NSCs by a non-catalytic action at Snrpn. Nat Comm. 10 (1), 1726(2019).
- Gritti, A., et al. Epidermal and fibroblast growth factors behave as mitogenic regulators for a single multipotent stem cell-like population from the subventricular region of the adult mouse forebrain. J Neurosci. 19 (9), 3287-3297 (1999).
- Nieto-Estévez, V., Pignatelli, J., Araúzo-Bravo, M. J., Hurtado-Chong, A., Vicario-Abejón, C. A global transcriptome analysis reveals molecular hallmarks of neural stem cell death, survival, and differentiation in response to partial FGF-2 and EGF deprivation. PLoS One. 8 (1), 53594(2013).
- Vergaño-Vera, E., Méndez-Gómez, H. R., Hurtado-Chong, A., Cigudosa, J. C., Vicario-Abejón, C. Fibroblast growth factor-2 increases the expression of neurogenic genes and promotes the migration and differentiation of neurons derived from transplanted neural stem/progenitor cells. Neuroscience. 162 (1), 39-54 (2009).
- Gabay, L., Lowell, S., Rubin, L. L., Anderson, D. J. Deregulation of dorsoventral patterning by FGF confers trilineage differentiation capacity on CNS stem cells in vitro. Neuron. 40 (3), 485-499 (2003).
- Hack, M. A., Sugimori, M., Lundberg, C., Nakafuku, M., Götz, M. Regionalization and fate specification in neurospheres: the role of Olig2 and Pax6. Mol Cell Neurosci. 25 (4), 664-678 (2004).
- Bithell, A., Finch, S. E., Hornby, M. F., Williams, B. P. Fibroblast growth factor 2 maintains the neurogenic capacity of embryonic neural progenitor cells in vitro but changes their neuronal subtype specification. Stem Cells. 26 (6), 1565-1574 (2008).
- Pastrana, E., Cheng, L. C., Doetsch, F. Simultaneous prospective purification of adult subventricular zone neural stem cells and their progeny. Proc Natl Acad Sci. 106 (15), 6387-6392 (2009).
- Kobayashi, N., et al. Gene delivery to embryonic stem cells. Birth Def Res C. 75 (1), 10-18 (2005).
- Gresch, O., Altrogge, L. Transfection of difficult-to-transfect primary mammalian cells. Meth Mol Biol. 801, 65-74 (2011).
- Maasho, K., Marusina, A., Reynolds, N. M., Coligan, J. E., Borrego, F. Efficient gene transfer into the human natural killer cell line, NKL, using the Amaxa nucleofection system. J Immunol Meth. 284 (1-2), 133-140 (2004).
- von Holst, A., Egbers, U., Prochiantz, A., Faissner, A. Neural stem/progenitor cells express 20 tenascin C isoforms that are differentially regulated by Pax6. J Biol Chem. 282 (12), 9172-9181 (2007).
- Woodard, L. E., Wilson, M. H. piggyBac-ing models and new therapeutic strategies. Trend Biotechnol. 33 (9), 525-533 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon