Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Generación de linfocitos T reguladores del receptor de antígeno quimérico humano
En este artículo
Resumen
Este protocolo proporciona un flujo de trabajo optimizado para generar y probar células T reguladoras del receptor de antígeno quimérico humano (CAR Tregs).
Resumen
La terapia de células T con receptor de antígeno quimérico (CAR) ha remodelado la cara del tratamiento del cáncer, lo que ha llevado a tasas de remisión récord en cánceres hematológicos previamente incurables. Estos éxitos han estimulado el interés en adaptar la plataforma CAR a un subconjunto pequeño pero fundamental de células T CD4+ responsables principalmente de regular e inhibir la respuesta inmunitaria, las células T reguladoras (Tregs). La capacidad de redirigir la actividad inmunosupresora de las Tregs a cualquier objetivo extracelular tiene enormes implicaciones para la creación de terapias celulares para enfermedades autoinmunes, rechazo de trasplantes de órganos y enfermedad de injerto contra huésped. Aquí, describimos en detalle las metodologías para el aislamiento de Treg de buena fe a partir de sangre periférica humana, la modificación genética de Tregs humanas utilizando lentivirus o CRISPR/Cas9 asistida por knock-in utilizando la entrega de plantillas de reparación dirigida homóloga (HDR) mediada por virus adenoasociados, y la expansión ex vivo de Tregs CAR humanas estables. Por último, describimos la evaluación de la estabilidad fenotípica de las Treg CAR humanas y la función supresora in vitro , que proporciona información sobre cómo se comportarán las Treg CAR humanas en aplicaciones preclínicas y clínicas.
Introducción
Las terapias de células T con receptores de antígenos quiméricos (CAR) han revolucionado el tratamiento de las neoplasias malignas hematológicas, logrando tasas de remisión notablemente altas en cánceres previamente intratables 1,2. Los resultados preliminares alentadores del uso de células T con CAR para tratar el glioblastoma 3,4,5 destacan la versatilidad y el potencial futuro de la tecnología CAR para tratar una amplia gama de neoplasias malignas. A medida que el campo explora nuevas aplicaciones de los CAR, las células T reguladoras (Tregs) han surgido como un tipo de célula prometedor. Las Tregs desempeñan un papel crucial en el mantenimiento de la homeostasis inmunitaria y en la regulación de las respuestas inmunitarias a través de varios mecanismos, como el secuestro de IL-2, la secreción de citocinas inmunosupresoras y la modulación de las células presentadoras de antígenos 6,7.
Con la tecnología CAR, las Tregs podrían aprovecharse para el tratamiento del rechazo de trasplantes de órganos, enfermedades autoinmunes y trastornos inflamatorios como alergias y asma 6,8,9. Las Tregs con CAR podrían conducir a mejoras significativas en los resultados y la calidad de vida de los pacientes al reducir el uso de fármacos inmunosupresores, que inhiben el sistema inmunitario en su conjunto y se asocian con efectos secundarios nocivos10,11. Los modelos preclínicos han mostrado resultados prometedores en la traducción de la tecnología CAR a Tregs, con aplicaciones exitosas en enfermedades como la diabetes tipo 1, la esclerosis múltiple, la enfermedad de injerto contra huésped y la enfermedad inflamatoria intestinal 9,12,13,14,15. En la clínica, actualmente se están explorando las Tregs CAR para prevenir el rechazo del trasplante de órgano sólido16.
Este artículo presenta una metodología detallada para la generación de células T reguladoras del receptor de antígeno quimérico humano (CAR Tregs). Este protocolo consiste en aislar las Tregs de la sangre periférica humana y modificarlas genéticamente mediante técnicas como la transducción lentiviral y el knock-in preciso de genes mediante la edición génica CRISPR/Cas9 y vectores de virus adenoasociados (AAV). También describimos la evaluación de la estabilidad fenotípica y la función supresora de estas Tregs modificadas, que son pasos cruciales para validar su potencial terapéutico 17,18,19. Este enfoque agiliza el diseño y las pruebas tempranas de las terapias CAR Treg, que tienen el potencial de ampliar el impacto transformador de la terapia de células T con CAR para regular el sistema inmunitario. Al compartir nuestra metodología, esperamos inspirar más investigación e innovación en el floreciente espacio de terapia CAR Treg 9,20.
Protocolo
1. Aislamiento humano de Treg
- Procesamiento de Leukopak
- Transfiera el contenido del leukopak a un tubo cónico de 50 ml. Añada un volumen igual de solución salina tamponada con fosfato (DPBS) de Dulbecco +2% de suero fetal bovino (FBS) mezclando suavemente con una pipeta.
- Centrifugar a 300 × g durante 10 min a temperatura ambiente (RT). Aspire cuidadosamente el sobrenadante. Reconstituya el pellet de la célula en 2 mL de DPBS + 2% de FBS. Añadir 8 mL de solución de cloruro de amonio por pipeta a la suspensión celular en una proporción de 4:1, mezclar por inversión suave y dejar la lisis de los glóbulos rojos restantes en hielo durante 15 min.
- Centrifugar a 300 × g durante 10 min a RT. Aspirar cuidadosamente el sobrenadante. Añadir 30 mL de DPBS + 2% FBS para lavar las células. Con el freno apagado, centrifugar las células lavadas a 150 × g durante 10 min en RT. Aspirar cuidadosamente el sobrenadante. Vuelva a suspender el pellet de celda en 30 mL de DPBS + 2% de FBS.
- Cuente las células mononucleares de sangre periférica (PBMC) ahora aisladas con azul de tripán en una proporción de 1:1.
- Debido a la alta concentración de células, diluya inicialmente 10 μL de células 1:100 con DPBS; luego, mezcle 10 μL de las celdas diluidas 1:100 con 10 μL de solución de azul de tripán para esa dilución final 1:1. Cuando utilice un contador de células automatizado, corrija el recuento de células para reflejar la dilución de 200 veces multiplicando la concentración informada por 100, ya que la mayoría de los contadores asumen una dilución de 2 veces. Anticipe producir 1-2.5 × 109 PBMC de un leukopak 1/10.
- Aislamiento de linfocitos T CD4+ (selección negativa)
- Centrifugar 108-10 9 PBMC a 500 × g durante 5 min en RT y resuspender en tampón de separación celular (DPBS + 1 mM EDTA + 2% FBS) a 5 × 107 células/mL.
NOTA: Para obtener suficientes Tregs humanas para la modificación genética, recomendamos comenzar con 1 × 109 PBMC. - Realice la separación magnética de acuerdo con las instrucciones del fabricante para el kit de enriquecimiento de células T CD4+ .
- Determine la cantidad de linfocitos T CD4+ aislados contando con azul de tripán (10 μL de células + 10 μL de azul de tripano).
- Centrifugar 108-10 9 PBMC a 500 × g durante 5 min en RT y resuspender en tampón de separación celular (DPBS + 1 mM EDTA + 2% FBS) a 5 × 107 células/mL.
- Aislamiento de linfocitos T CD8+ (selección negativa)
- Centrifugar 5 × 107 PBMC a 500 × g durante 5 min en RT. Volver a suspender en tampón de separación celular (DPBS + 1 mM EDTA + 2% FBS) a 5 × 107 células/mL.
NOTA: Recomendamos comenzar con 5 × 107 PBMC para obtener 2-5 × 106 linfocitos T CD8+ . - Realice la separación magnética de acuerdo con las instrucciones del fabricante para el kit de enriquecimiento de células T CD8+ .
- Determine el número de linfocitos T CD8+ aislados contando con azul de tripán (10 μL de células + 10 μL de azul de tripán).
- Centrifugar 5 × 107 PBMC a 500 × g durante 5 min en RT. Volver a suspender en tampón de separación celular (DPBS + 1 mM EDTA + 2% FBS) a 5 × 107 células/mL.
- Clasificación celular asistida por fluorescencia Treg (FACS)
- Aísle las células T CD4+ como se describe en el paso 1.2 y almacene durante la noche en DPBS con 2% de FBS a 4 °C (pérdida mínima en número de células y viabilidad) para FACS al día siguiente. Determine el número de linfocitos T CD4+ del paso 1.2 contando con azul de tripán (10 μL de células + 10 μL de azul de tripano).
- Centrifugar las células CD4+ a 500 × g durante 5 min. Reconstituir las células en 200 μL de DPBS.
- Por 1 × 106 células, agregue 1 μL de FITC CD4 antihumano, 1 μL de APC CD25 antihumano y 1 μL de PE CD127 antihumano. Agite suavemente y colóquelo en la nevera oscura a 4 °C durante 30 min.
- Lavar las células con 10 mL de DPBS con 2% de FBS. Girar a 500 × g durante 5 min. Vuelva a suspender suavemente las células teñidas a 1,5 × 107 células/ml en DPBS con 2% de FBS. Esta es la concentración de células recomendada para clasificar mediante la clasificación de células activadas por fluorescencia (FACS).
- Pase la suspensión celular teñida a través de una tapa de filtro de 40 μm en los tubos FACS y luego mantenga los tubos en hielo.
- Prepare tubos de recolección de 15 mL que contengan 3 mL de medio RPMI10 y colóquelos en hielo.
NOTA: Este medio consta de RPMI1640 medio basal, 10% de FBS, 1x penicilina-estreptomicina, 1x L-alanil-L-glutamina, 1x aminoácidos no esenciales, 1x piruvato de sodio y 1x HEPES. - Clasifique las células T reguladoras CD4 + CD25con altoCD127 (Tregs) y las células T convencionales CD4 + CD25con CD127+ bajo (Tconv) utilizando FACS como se muestra en la Figura 1A.
- Determinar el rendimiento y la viabilidad de las células; luego proceda con el análisis posterior o la activación de las células T.
2. Activación de células T
- Cuente las células T aisladas con azul de tripán (10 μL de células + 10 μL de azul de tripano).
- Lavar 25 μL de perlas anti-CD3/CD28 (106 perlas) por cada 1 × 106 linfocitos T obtenidos, manteniendo una proporción de 1:1 de perlas a linfocitos T. Lave las perlas anti-CD3/CD28 agregando DPBS, incubando en un imán durante 3 minutos y eliminando el tampón de perlas anti-CD3/CD28 ahora diluido, que puede ser tóxico para las células.
- Retire el tubo del imán y vuelva a suspender las perlas lavadas en el medio RPMI10 para obtener 1 × 106 perlas/1 mL de RPMI10.
- Resuspenda las células T con perlas anti-CD3/CD28 en medio RPMI10 a una concentración de 1 × 106 células T/mL. Luego, agregue 1,000 UI/mL de IL-2 a las células Treg, 100 UI/mL de IL-2 a las células Tconv CD4+ y 300 UI/mL de IL-2 a las células T CD8+ .
- Cultivo de linfocitos T a 1 × 106 células por pocillo de una placa de 24 pocillos con diferentes cantidades de perlas anti-CD3/CD28 e IL-2. Coloque la placa de 24 pocillos en una incubadora de cultivo de tejidos a 37 °C con 5% de CO2 .
- Expanda las células T activadas durante 9-12 días en presencia de IL-2 hasta que se utilicen para experimentos o se reactiven con perlas anti-CD3/CD28 para una expansión adicional. Reemplace el medio cada 2-3 días y agregue IL-2 cada vez que se agregue medio nuevo. Mantenga la densidad celular de las Tregs en 5 × 105 a 1 × 106 celdas por ml durante la expansión contando las celdas o mediante inspección visual antes de dividir.
3. Transducción lentiviral de Treg humano
- Vuelva a suspender y contar Tregs 48 h después de la activación. Centrifugar a 500 ×g durante 5 min en RT. Resuspender Tregs en RPMI10 a 1,25 × 106 células/mL con 1.000 UI/mL de IL-2.
- Precalentar la centrífuga a 32 °C.
- Descongela la construcción CAR que contiene lentivirus en hielo.
NOTA: Almacenamos alícuotas de lentivirus de un solo uso con suficiente lentivirus para transducir 2,5 × 105 células a un MOI de 1 a -80 °C. - Agregue cada alícuota de lentivirus a 2,5 × 105 Tregs en 200 μL en un tubo de microcentrífuga, cada tubo indica una reacción. Espinocular a 1.000 × g durante 1 h a 32 °C.
- Mueva cada reacción de 200 μL a una placa de 24 pocillos. Asegúrese de que el medio cubra todo el pozo. Incubar la placa de 24 pocillos con las Tregs transducidas en una incubadora de cultivo de tejidos durante la noche (16-18 h). Rellene cada pocillo hasta 2 mL con medio RPMI10 con una concentración final de IL-2 de 1.000 UI/mL.
- Continúe expandiendo las Tregs transducidas 9-12 días después de la activación dividiendo las células y complementando con RPMI10 e IL-2 recién calentadas y precalentadas según sea necesario (cada 2-3 días). La concentración ideal de células es de 5 × 105-1 × 106 células/mL.
- Evalúe la eficiencia de la modificación génica mediante citometría de flujo, como se muestra en la Figura 2.
NOTA: Nuestras construcciones lentivirales de CAR contienen una etiqueta Myc en el extremo N-terminal del gen CAR y un gen reportero de GFP unido al gen CAR por un péptido 2A, lo que permite la cuantificación de la eficiencia de la transducción sin tinción de anticuerpos (GFP) y la confirmación de la expresión de la superficie de CAR (Myc-tag). Evaluamos la eficacia de la modificación génica 5 días después de la transducción. Las eficiencias de transducción pueden variar entre el 30% y el 70% dependiendo del donante y de la construcción de CAR utilizada. Las celdas CAR+ se pueden ordenar si se desea. - Asegúrese de que las Tregs modificadas completen su ciclo de activación (9-12 días) y descansen 24 horas en ausencia de IL-2 antes de ser utilizadas en cualquier experimento.
4. Knock-in del gen CRISPR/Cas9 mediado en Tregs humanas
- Vuelva a suspender las Tregs y transfiéralas a un tubo cónico de 15 ml 48 h después de la activación. Incubar la suspensión celular en un imán durante 3 min.
- Mientras está en el imán, transfiera las células del medio a través de una pipeta a un tubo nuevo. Las perlas anti-CD3/CD28 permanecerán unidas a la pared del tubo. Deje que las Tregs desperladas descansen en RPMI10 durante 2 h después de la extracción del cordón para recuperarse del estrés inmediato del descordado, mejorando la eficiencia futura de la transducción y la recuperación de la electroporación.
- Cuenta las Tregs despeinadas con azul tripano.
- Precalentar el medio sérico reducido sin FBS a 37 °C.
- Prepare una placa de 6 pocillos con 2,5 mL de medio RPMI10 sin antibióticos (penicilina-estreptomicina) y con 1.000 UI/mL de IL-2 por pocillo. Precalentar la placa a 37 °C en una incubadora de cultivo de tejidos.
- Descongele la plantilla de reparación dirigida por homología (HDR) que contiene virus adenoasociados (AAV) CAR en hielo.
NOTA: Almacenamos alícuotas de un solo uso con suficiente AAV para infectar 4 × 106 células a un MOI de 20.000 a -80 °C. - Gira las Tregs a 500 × g durante 5 min. Después de decantar el sobrenadante, resuspender las células en un medio sérico reducido precalentado a 4 × 107 células/mL.
- Células alícuotas en 100 μL en tubos de centrífuga de 1,5 mL de baja unión a proteínas. Agregue CAR AAV a un MOI de 20,000 a cada muestra y vuelva a suspender. Incubar los tubos de reacción en la incubadora de cultivo de tejidos durante 1 h.
- Durante la incubación de 1 h, prepare y ensamble los complejos de ribonucleoproteína (RNP) CRISPR/Cas9 añadiendo lentamente 8,3 μL de proteína Cas9 (stock de 1 mg/mL) a 2,5 μL de sgRNA dirigido al locus del gen TRAC (stock de 100 μM) para una relación molar de Cas9 a sgRNA de 1:1 y un volumen total de RNP de 10,8 μL por muestra. Pipetear suavemente hacia arriba y hacia abajo para mezclar. Incubar la mezcla de RNP durante 15 min a 37°C en la incubadora de cultivo de tejidos.
NOTA: Los complejos RNP pueden permanecer en RT después hasta que estén listos para usar. - Llene un tubo de electroporación nuevo (tubo E) con 3 mL de tampón de electroporación de alta osmolaridad. Inserte el tubo E lleno en la estación de pipetas del sistema de electroporación hasta que se escuche un clic. Ajuste las condiciones de electroporación a 2.200 V, 20 ms, 1 pulso en el sistema de electroporación.
- Una vez finalizada la incubación de 1 h con AAV, centrifugar las células con AAV a 300 × g durante 5 min en RT. Aspirar cuidadosamente el sobrenadante y volver a suspender el pellet celular en 100 μL del tampón de resuspensión celular proporcionado por el sistema de electroporación por muestra.
NOTA: Trabaje rápidamente y evite dejar las células en el tampón durante más de 15 minutos. Evita crear burbujas. - Añadir 10,8 μL de complejo RNP por muestra. Mezclar bien con una pipeta sin crear burbujas.
- Inserte una punta de electroporación de 100 μL empujando la pipeta hasta su segundo tope para abrir la pinza. Coloque el cabezal superior de la pipeta en la punta de electroporación hasta que la abrazadera encaje de forma segura con el vástago de montaje del pistón. Suelte gradualmente el botón mientras mantiene la presión hacia abajo sobre la pipeta para asegurarse de que la punta encaje perfectamente sin espacios.
- Presione la pipeta hasta el primer tope y sumerja la punta de electroporación en la mezcla de celda y RNP. Tire suavemente de la muestra hacia arriba en la pipeta sin que queden burbujas.
NOTA: No puede haber burbujas dentro de la punta durante la electroporación. - Inserte la pipeta con la punta de electroporación montada que contiene la muestra verticalmente en el tubo E hasta que se escuche un clic. Evite crear burbujas.
- Confirme que se han introducido los ajustes óptimos para las Tregs humanas (2.200 V, 20 ms, 1 pulso) y pulse el botón de inicio en la pantalla táctil para electropoar las células.
- Espere a que la pantalla táctil muestre Completo al finalizar la electroporación. Retire suavemente la pipeta e inmediatamente transfiera la muestra a la placa preparada de 6 pocillos que contiene 2,5 mL de medio RPMI10 precalentado y sin antibióticos con 1.000 UI/mL de IL-2 por pocillo. Repita con las muestras restantes. Balancee suavemente la placa con movimientos lineales (de izquierda a derecha, de arriba a abajo) para asegurar una distribución uniforme de las células en cada pocillo y colóquela en la incubadora de cultivo de tejidos.
NOTA: La reutilización de una punta de electroporación es aceptable hasta 3 veces si se utilizan las mismas celdas y el mismo complejo RNP. Espere un período de recuperación de 30 minutos antes de contar las células o alterarlas de alguna manera, como agregar un potenciador HDR si lo desea. - Al día siguiente, 16-18 h después, reemplace el medio con medios que contengan antibióticos, cuente las Tregs electroporadas con azul de tripán y cultive a 106 células/mL con 1,000 UI/mL IL-2. Continúe expandiendo las Tregs electroporadas dividiendo las células y complementando RPMI10 e IL-2 frescas como se describe en el paso 2.6.
- Mantenga las muestras en hielo para su análisis por citometría de flujo.
- Configure controles de compensación de un solo color y aplíquelos al archivo de experimento.
- Lea primero la muestra sin tinción para ajustar las ganancias de FSC y SSC de modo que la población de linfocitos esté en el centro del diagrama de puntos "Todos los eventos".
- Configure la estrategia de activación de linfocitos en no desechos/células individuales en células viables en células CD4+ , como se muestra en la Figura 3C.
- Evaluar la eficiencia de la modificación génica mediante citometría de flujo.
NOTA: Nuestras construcciones AAV CAR contienen, además de los brazos de homología del locus TRAC, un gen reportero del receptor del factor de crecimiento epidérmico truncado (EGFRt) unido al gen CAR por un péptido 2A como se muestra en la Figura 3. La eficiencia de la incursión génica se determina mediante la tinción superficial para CD3, cuya pérdida indica la pérdida de la expresión superficial de TCR y, por lo tanto, la orientación exitosa del locus TRAC con CRISPR/Cas9 y EGFRt, cuya expresión indica una integración exitosa del transgén CAR. Las Tregs knock-in de CAR son células CD3-EGFRt+. Evaluamos la eficacia de la modificación génica 5 días después de la electroporación. - Asegúrese de que las Tregs modificadas completen su ciclo de activación (9-12 días) y descansen 24 horas en ausencia de IL-2 antes de ser utilizadas en cualquier experimento.
5. Activación de Treg CAR humano
- Configuración de co-cultivo (Día 0)
- Recoja la línea celular diana que expresa el antígeno CAR en un tubo cónico.
NOTA: Utilizamos células K562, una línea celular de leucemia mielógena humana que carece de expresión de HLA, CD80 y CD86 como células diana para la activación de CAR Treg. Las células parentales K562 se utilizan como control negativo y las K562 que expresan antígeno CAR se utilizan para activar las Tregs17,21 de CAR. - Irradie líneas celulares objetivo con 4.000 rad en un irradiador de cesio-137 o de rayos X. En caso de falta de acceso a un irradiador, realizar un tratamiento con mitomicina C para detener la proliferación celular mientras se mantiene la expresión del antígeno de superficie en las células diana22.
- Si aún no lo ha hecho, descornee las Tregs resuspendiendo las Tregs unidas a las perlas anti-CD3/CD28 y transfiriéndolas a un tubo cónico de 15 mL. Incubar la suspensión celular en un imán durante 3-5 min. Mientras aún está en el imán, transfiera las células del medio a través de una pipeta a un nuevo tubo; las perlas anti-CD3/CD28 permanecerán unidas a la pared del tubo.
- Determine la célula objetivo irradiada y las concentraciones de Treg de CAR debeaded con azul de tripán.
- Centrifugar las células diana irradiadas y las Tregs CAR a 500 × g durante 5 min. Vuelva a suspender con RPMI10 precalentado a 106 células/mL en sus tubos separados. Agregue IL-2 a CAR Tregs para obtener una concentración de 2,000 UI / mL.
NOTA: Esta cantidad de IL-2 se diluirá 2 veces una vez que las Tregs CAR se combinen con las células objetivo para obtener una concentración final de IL-2 de 1,000 UI / mL. - Cocultivo de 1 × 105 CAR Tregs (100 μL) con 1 × 105 ( 100 μL) células diana irradiadas negativas para el antígeno CAR (control negativo para la activación), 1 × 105 ( 100 μL) células diana irradiadas positivas para el antígeno CAR (experimento), o 2,5 μL de perlas anti-CD3/CD28 (control positivo para la activación) y 97,5 μL de medio RPMI10 en una placa inferior redonda de 96 pocillos. Colocar la placa en una incubadora de cultivo de tejidos a 37 °C y 5% CO2 durante 48 h.
NOTA: El volumen final por pocillo es de 200 μL. Asegúrese de que cada condición tenga tres réplicas.
- Recoja la línea celular diana que expresa el antígeno CAR en un tubo cónico.
- Lectura de citometría de flujo
- A menos que use un lector de placas, vuelva a suspender el contenido de cada pocillo de la placa de fondo redondo de 96 pocillos y transfiéralos a un tubo FACS. Girar a 500 x g durante 5 min. Decantar el sobrenadante y vorexear suavemente la célula pellet.
- Prepare la mezcla maestra de anticuerpos de citometría de flujo: 100 μL / muestra que contiene DPBS, CD4 PE / Cy7 1:200, CD71 PE 1:100 y colorante de viabilidad fantasma BV510 1: 2000.
NOTA: Este panel funciona bien si el gen reportero de CAR es GFP. Si un gen reportero codifica una proteína de superficie que requiere tinción de anticuerpos, como EGFRt, se puede usar un anticuerpo conjugado con FITC, por ejemplo. - Pipetear 100 μL de la mezcla maestra de anticuerpos en cada muestra, mezclar suavemente e incubar durante 30 minutos en la nevera a 4 °C.
- Lavar con 500 μL de DPBS centrifugando a 500 × g durante 5 min. Vuelva a suspender el pellet celular en 200 μL de DPBS.
- Mantenga las muestras en hielo para su análisis por citometría de flujo.
- Configure controles de compensación de un solo color y aplíquelos al archivo de flujo del experimento.
- Lea las muestras sin teñir para ajustar las ganancias de SSC y FSC de modo que la población de linfocitos esté en el centro del diagrama de puntos "Todos los eventos".
- Establezca la estrategia de activación de linfocitos en no desechos/células individuales en células viables en células CD4+ , como se muestra en la Figura 3C (Diferentes fluoróforos utilizados).
- Lea muestras a una tasa de eventos de aproximadamente 1.500 eventos/s para evaluar la activación de Treg.
NOTA: El resultado esperado es la regulación positiva de la expresión de la superficie de CD71 en las Tregs CAR en presencia del antígeno CAR, como se muestra en la Figura 4. Si no se observa una regulación positiva en presencia de antígeno CAR, entonces podría haber señalización tónica.
6. Estabilidad humana CAR Treg
- Configuración y expansión de co-cultivo (Días 0-9)
- Configure cocultivos para activar CAR Tregs como se describe en el paso 5.1.
- Después de 48 h, transfiera el cocultivo de cada pocillo de placa de fondo redondo de 96 pocillos a un pocillo de placa de 24 pocillos que contenga 2 mL precalentados de RPMI10 con 1,000 UI/mL de IL-2 para permitir la expansión celular.
- Agregue RPMI10 fresco y precalentado con 1,000 UI/mL de IL-2 y divídalo en placas adicionales de 24 pocillos según sea necesario.
- Lectura de citometría de flujo
- Vuelva a suspender y transfiera el contenido de cada réplica a un tubo cónico de 15 mL o 50 mL. Determine las concentraciones de células.
- Transfiera entre 5 × 105 y 1 × 106 celdas a un tubo FACS para cada réplica. Girar a 500 × g durante 5 min.
- Prepare la mezcla maestra de anticuerpos de citometría de flujo para la tinción de superficies: 100 μL/muestra que contiene DPBS, CD4 PE/Cy7 1:200, CD25 APC 1:200 y colorante de viabilidad fantasma BV510 1:2000. Incluya un anticuerpo conjugado con FITC para la proteína reportera CAR si es necesario.
- Decantar el sobrenadante del paso 6.2.2 y vórtice suavemente el pellet de la célula. Pipetee 100 μL de la mezcla maestra de anticuerpos que tiñe la superficie en cada tubo FACS. Coloque brevemente en un vórtice y colóquelo en una nevera a 4 °C durante 30 minutos en la oscuridad.
- Utilizando un conjunto de tampones de tinción de factores de transcripción, prepare el tampón de fijación/permeabilización añadiendo 3 volúmenes de diluyente de fijación/permeabilización a 1 volumen de concentrado de fijación/permeabilización. Cada muestra requiere 100 μL de tampón de fijación/permeabilización.
- Lave las células teñidas en la superficie con 500 μL de DPBS. Girar a 500 × g durante 5 min y decantar el sobrenadante. Pipetear 100 μL de tampón de fijación/permeabilización preparado en cada tubo. Realice un vórtice breve y permita que la fijación ocurra a 4 °C durante 30-60 min en la oscuridad.
- Usando un conjunto de tampones de tinción de factores de transcripción, prepare 1x tampón de permeabilización agregando 9 volúmenes de agua destilada a 1 volumen de concentrado de tampón de permeabilización 10x. Cada muestra requiere 1.000 μL de tampón de permeabilización para el lavado y 100 μL de tampón de permeabilización para la tinción con anticuerpos dirigidos a proteínas intracelulares.
- Lave las células fijas/permeabilizadas añadiendo 500 μL de tampón de permeabilización 1x. Girar a 500 × g durante 5 min a RT.
- Prepare la mezcla maestra de anticuerpos de tinción intracelular con tampón de permeabilización 1x, anti-humano FOXP3 eFluor 450 1:50, anti-humano HELIOS PE 1:50 y anti-humano CTLA-4 PerCP-e710 1:50. Cada muestra requerirá 100 μL de mezcla maestra de anticuerpos.
NOTA: Este panel funciona si el gen reportero de CAR es GFP o si la proteína reportera de CAR (por ejemplo, EGFRt) está teñida con un anticuerpo conjugado con FITC. - Decantar el sobrenadante desde el paso 6.2.8. Agregue 100 μL de la mezcla maestra de anticuerpos de tinción intracelular, brevemente en vórtice e incube en RT durante 30 minutos en la oscuridad.
- Lave las células fijas/permeabilizadas teñidas añadiendo 500 μL de tampón de permeabilización 1x. Centrifugar a 500 × g durante 5 min. Decantar el sobrenadante, resuspender el pellet celular en 300 μL de DPBS y almacenarlo en hielo.
- Analice por citometría de flujo como se muestra en la Figura 5. El resultado previsto es que la mayoría de las Tregs CAR serán células FOXP3+HELIOS+ . Utilice células Tconv CD4+ como control negativo para la tinción de FOXP3 y HELIOS.
7. Supresión de CAR Treg humano
- Tinción de tinte y activación nocturna de trazas de células T respondedoras (Tresp)
- Recoja las células CAR Treg expandidas, las células Tconv CD4+ recién aisladas y las células T CD8+ recién aisladas en tubos cónicos separados de 15 ml y perlas anti-CD3/CD28 debeelas si aún no lo ha hecho.
- Prepare las células diana irradiadas que expresan antígeno CAR como se describe en el paso 5.1.
- Determine las concentraciones de linfocitos T no activados y linfocitos diana irradiados.
- Combine 5 × 106 células Tconv CD4+ con 5 × 106 células T CD8+ (proporción 1:1). Estas son las células T respondedoras (Tresp) que serán inhibidas por las Tregs CAR en el ensayo. Centrifugar las células de Tresp a 500 × g durante 5 min en RT. Aspirar cuidadosamente el sobrenadante y volver a suspender las células de Tresp en 1 mL de DPBS.
- Añada 1 mL de 5 mM de colorante CellTrace Violet (CTV) reconstituido en DMSO a las 107 células Tresp en 1 mL de DPBS para una concentración final de 5 μM de CTV. Colocar al baño maría a 37 °C durante 20 min. A los 10 min, vórtice suavemente para redistribuir el CTV establecido.
- Lavar con 9 mL de medio completo RPMI10 precalentado. Girar a 500 × g durante 5 min. Vuelva a suspender en 5 mL de medio completo RPMI10 precalentado.
- Determine la concentración de células Tresp marcadas con CTV. Agregue 5 × 104 células Tresp no activadas marcadas con CTV en 200 μL de medio RPMI10 en 3-6 pocillos de una placa de fondo redondo de 96 pocillos como controles mínimos de proliferación.
- Active las células Tresp marcadas con CTV con perlas anti-CD3/CD28 en una proporción de perlas a células Tresp de 1:10 sin IL-2 en medio RPM10. Dispense 106 células Tresp marcadas con CTV en 1 mL de medio RPMI10 por pocillo de una placa de 24 pocillos en la incubadora de cultivo de tejidos durante la noche.
- Paralelamente, obtenga 106 Tregs CAR+ . Esta cantidad cubre triplicados de cuatro proporciones CAR Treg:Tresp, 1:1, 1:2, 1:4, 1:8, a 5 × 104 celdas objetivo por pocillo de placa de fondo redondo de 96 pocillos, como 5 × 104 × 3 + 2,5 × 104 × 3 + 1,25 ×4 4 × 3 + 0,625 × 104 × 3 = 2,81 × 105 CAR Tregs, a través de dos condiciones de activación de CAR Treg: células diana CAR negativas al antígeno CAR irradiadas (control negativo, sin activación) y células diana positivas para el antígeno CAR irradiadas (experimento, activación de CAR).
- Combine 3 × 105 CAR Tregs con 3 × 105 células CAR negativas irradiadas (proporción 1:1) y 3 × 105 CAR Tregs con 3 células diana CAR positivas para el antígeno CAR × 105 irradiadas (proporción 1:1), en dos tubos cónicos separados de 15 mL y centrifugar a 500 × g durante 5 min en RT. Aspire cuidadosamente el sobrenadante.
- Vuelva a suspender el pellet en cada tubo del paso 7.1.10 en 600 μL de medio RPMI10 precalentado. Esto da como resultado 200 μL para cada uno de los Treg: Tresp 1:1 triplicados. Realice la dilución en serie en una placa de fondo redondo de 96 pocillos de la siguiente manera:
- De los 600 μL preparados de Treg + suspensión de celda objetivo, pipetee 200 μL de suspensión celular en cada uno de los pocillos de relación 3 1:1.
- Agregue 100 μL de medio completo RPMI10 precalentado en cada uno de los pocillos vacíos de proporción 1:2, 1:4 y 1:8.
- Para cada uno de los triplicados, pipetee 100 μL de la suspensión celular de los pocillos 1:1 en los pocillos de relación 1:2 asociados.
- Para cada uno de los triplicados, pipetear 100 μL de la suspensión de la célula, de los pocillos 1:2, en los pocillos de proporción 1:4 asociados.
- Para cada uno de los triplicados, pipetear 100 μL de la suspensión de la célula, de los pocillos 1:4, en los pocillos de proporción 1:8 asociados.
- Pipetear los 100 μL restantes de la suspensión celular de los pocillos de proporción 1:8 en un contenedor de residuos.
NOTA: Cada pocillo debe contener 100 μL de suspensión celular con 5 × 104 (1:1), 2,5 × 104 (1:2), 1,25 × 104 (1:4) o 0,625 × 104 (1:8) Tregs CAR y un número igual de células objetivo irradiadas.
- Co-incubación de células CAR Treg y Tresp
- Después de 16-18 h después de la activación, recoja las células Tresp activadas de la placa de 24 pocillos en un tubo cónico y retire las perlas anti-CD3/CD28 mediante un imán.
- Determine los recuentos de células Tresp marcadas con CTV sin perlas .
- Lavar y centrifugar 2 × 106 células Tresp a 500 × g durante 5 min. Aspire cuidadosamente el sobrenadante. Resuspender las células en 4 mL de medio completo RPMI10.
- Agregue 100 μL de la suspensión de células T (5 × 104 células T) a cada pocillo con CAR Tregs, así como a 3-6 pocillos con 100 μL de medio RPMI10 solo para controles máximos de proliferación. Colocar la placa en una incubadora de cultivo de tejidos a 37 °C y 5% CO2 durante 72 h.
NOTA: La placa de fondo redondo de 96 pocillos ahora tiene 3-6 pocillos con células Tresp no activadas solas (control de proliferación mínimo), 3-6 pocillos con células Tresp activadas solas (control de proliferación máxima) y células Tresp activadas en presencia de un número decreciente de Tregs CAR.
- Lectura de citometría de flujo
- Vuelva a suspender y transfiera el contenido de cada plato inferior redondo de 96 pocillos a un tubo FACS etiquetado. Alternativamente, transfiera a una placa inferior en V de 96 pocillos si hay disponible un citómetro de flujo con modo de lectura de placa. Girar a 500 × g durante 5 min.
- Mientras tanto, prepare una mezcla maestra de anticuerpos con DPBS, CD4 PE/Cy7 1:200 y CD8 PerCP 1:200 antihumano. Cada muestra requerirá 100 μL de mezcla maestra de anticuerpos.
- Vórtice suavemente la célula de pellets. Pipetear 100 μL de mezcla maestra de anticuerpos en cada tubo FACS. Agitar brevemente e incubar a 4 °C durante 30 minutos en la oscuridad.
- Lavar con 500 μL de DPBS. Lave 2 veces con 100 μL de DPBS si usa un fondo en V de 96 pocillos. Centrifugar a 500 × g durante 5 min. Decantar el sobrenadante, resuspender el pellet de la célula en 200 μL de DPBS y almacenar los tubos en hielo en la oscuridad.
- Analice por citometría de flujo como se muestra en la Figura 6. El resultado esperado es que las células Tresp no activadas solas (proliferación mínima) muestren un pico de fluorescencia de CTV alto y uniforme, las células Tresp activadas solo muestren múltiples picos de intensidad de CTV, uno correspondiente a cada división celular (proliferación máxima), y las células T CD4+ y CD8+ activadas en presencia de Tregs CAR activadas que muestren una reducción en el número y la altura de los picos de CTV. por lo tanto, en proliferación.
- Calcule la supresión mediada por células Treg de la siguiente manera:
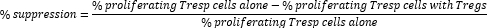
NOTA: Si se utiliza el software FlowJo, también es posible utilizar el modelado de proliferación celular para calcular el índice de división (DI) para cada muestra y calcular el porcentaje de supresión utilizando DI en lugar del porcentaje de células proliferantes en la fórmula anterior.
Resultados
El protocolo descrito aquí proporciona una línea simplificada y estandarizada para evaluar nuevas construcciones de receptores de antígenos quiméricos (CAR) en células T reguladoras humanas (Tregs), con el objetivo de crear terapias vivas para enfermedades autoinmunes, enfermedad de injerto contra huésped, rechazo de trasplante de órganos y alergia. La Figura 1 muestra cómo obtenemos Tregs humanas de alta pureza a partir de sangre periférica utiliza...
Discusión
Este protocolo proporciona una metodología simplificada y completa para generar y evaluar células T reguladoras del receptor de antígeno quimérico humano (CAR Tregs). El éxito de la tecnología CAR en el tratamiento de los cánceres hematológicos ha inspirado su aplicación al subconjunto inmunosupresor de las células T, las Tregs. A diferencia de las células T convencionales, las Tregs inhiben las respuestas inmunitarias, ofreciendo tratamientos potenciales para las afecciones q...
Divulgaciones
LMRF es un inventor y ha recibido regalías de patentes sobre células inmunitarias modificadas y es consultor para Guidepoint Global y McKesson. El resto de los autores declaran no tener intereses contrapuestos.
Agradecimientos
LMRF está financiado por la subvención de Líder Emergente en Diabetes Tipo 1 de la Red de Investigación de Islotes Humanos (HIRN) U24DK104162-07, la Subvención de Investigación Institucional de la Sociedad Americana Contra El Cáncer (ACS) IRG-19-137-20, la Subvención de Descubrimiento del Proyecto Piloto de Investigación Clínica y Traslacional de Carolina del Sur (SCTR) 1TL1TR001451-01, la Subvención de la Conexión de Investigación de la Diabetes (DRC) IPF 22-1224 y la Subvención de Swim Across America 23-1579. RWC cuenta con el apoyo de la beca de capacitación en Ciencias Celulares, Bioquímicas y Moleculares T32GM132055 y la beca de posgrado Lowvelo del Hollings Cancer Center. Este estudio fue financiado en parte por el Recurso Compartido de Citometría de Flujo y Clasificación Celular del Centro Oncológico Hollings de la Universidad Médica de Carolina del Sur (P30 CA138313). Un agradecimiento especial al Dr. Qizhi Tang de la Universidad de California, San Francisco (UCSF) por regalar amablemente los plásmidos mutantes CAR.
Materiales
| Name | Company | Catalog Number | Comments |
| Adeno-associated virus (AAV) | Charles River Laboratories | ||
| CAR target-expressing K562 cells | e.g., CD19-K562 | ||
| Cesium-137 irradiator | |||
| Anti-human CD8 PerCP (clone SK1) | Biolegend | 344708 | |
| Anti-human CD4 PE/Cy7 (clone SK3) | Biolegend | 344612 | |
| DynaMag-15 magnet | ThermoFisher | 12301D | |
| Ghost BV510 viability dye | TONBO | 13-0870-T100 | |
| K562 cells | American Type Culture Collection | CCL-243 | |
| 0.5 M EDTA, pH 8.0 | Gibco | 15575020 | |
| 1 M HEPES | Gibco | 15630080 | |
| Ammonium chloride solution | STEMCELL Technologies | 7850 | |
| Anti-human CD127 PE (clone hIL-7R-M21) | BD Biosciences | 557938 | |
| Anti-human CD25 APC (clone BC96) | Biolegend | 302610 | |
| Anti-human CD4 FITC (clone SK3) | Biolegend | 344604 | |
| Anti-human CD71 PE (clone SK1) | Biolegend | 334106 | |
| Anti-human CD8 PerCP (clone SK1) | Biolegend | 344707 | |
| Anti-human CTLA-4 PerCP-e710 | ThermoFisher | 46-1529-42 | |
| Anti-human EGFR APC (clone AY13) | Biolegend | 352905 | |
| Anti-human FOXP3 eFluor 450 | ThermoFisher | 48-4776-42 | |
| Anti-human HELIOS PE | Biolegend | 137216 | |
| Ca2+ and Mg2+ free Dulbecco’s Phosphate Buffered Saline (DPBS) | Gibco | 14190144 | |
| Cell counter (TC20 Automated Cell Counter) | Bio-Rad | 1450102 | |
| Cell Counting Slides | Bio-Rad | 1450016 | |
| CellTrace Violet Cell Proliferation Kit | ThermoFisher | C34571 | |
| DNA LoBind Tubes | Eppendorf | 22431021 | |
| Easy 50 EasySep magnet | STEMCELL Technologies | 18002 | |
| EasySep Human CD4+ T cell Enrichment Kit | STEMCELL Technologies | 19052 | |
| EasySep Human CD8+ T cell Enrichment Kit | STEMCELL Technologies | 19053 | |
| EasySep magnet | STEMCELL Technologies | 18000 | |
| eBioscience Foxp3 transcription factor staining buffer set | ThermoFisher | 00-5523-00 | |
| Falcon Round-Bottom Polystyrene Test Tubes with Cell Strainer Snap Cap, 5 mL | Fisher Scientific | 08-771-23 | 40μm |
| Fetal Bovine Serum (FBS) | Gibco | 26140079 | |
| Flow cytometer | Beckman Coulter | CytoFLEX LX U3-V5-B3-Y5-R3-I2 | |
| Fluorescence-activated cell sorter | BD Biosciences | FACS Aria III Cell Sorter | |
| GlutaMAX | Gibco | 35050061 | |
| Human CD3/28 T Cell Expansion and Activation Dynabeads | Gibco | 11131D | |
| Invitrogen Neon Transfection System | ThermoFisher | 10431915 | |
| Invitrogen Neon Transfection System 100 μL Kit | ThermoFisher | 10114334 | |
| Lentivirus | VectorBuilder | ||
| MEM Non-Essential Amino Acids Solution | Gibco | 11140050 | |
| Myc Tag antibody A647 (clone 9B11) | Cell Signaling Technologies | 2233S | |
| Opti-MEM I Reduced Serum Medium | ThermoFisher | 31985062 | |
| Penicilin-Streptomycin solution | Gibco | 15140122 | |
| Recombinant human interleukin-2 (rhIL-2) | Peprotech | 200-02 | |
| RPMI 1640 medium, no glutamine | Gibco | 11875093 | |
| Sodium pyruvate | Gibco | 11360070 | |
| Spectral Flow Cytometer | Cytek | Northern Lights | |
| TRAC gRNA | Synthego | Sequence (CAGGGTTCTGGATATCTGT) | |
| TrueCut Cas9 Protein v2 | ThermoFisher | A36496 | |
| Trypan Blue solution | Sigma | T8154-100ML | |
| 1/10 Leukopak | STEMCELL Technologies | 200-0092 | 1-2 billion PBMC |
Referencias
- Zhang, X., Zhu, L., Zhang, H., Chen, S., Xiao, Y. CAR-T cell therapy in hematological malignancies: current opportunities and challenges. Front Immunol. 13, 927153 (2022).
- Cappell, K. M., Kochenderfer, J. N. Long-term outcomes following CAR T cell therapy: what we know so far. Nat Rev Clin Oncol. 20 (6), 359-371 (2023).
- Choi, B. D., et al. Intraventricular CARv3-TEAM-E T cells in recurrent glioblastoma. N Engl J Med. 390 (14), 1290-1298 (2024).
- Brown, C. E., et al. Locoregional delivery of IL-13Ralpha2-targeting CAR-T cells in recurrent high-grade glioma: a phase 1 trial. Nat Med. 30 (4), 1001-1012 (2024).
- Bagley, S. J., et al. Intrathecal bivalent CAR T cells targeting EGFR and IL13Ralpha2 in recurrent glioblastoma: phase 1 trial interim results. Nat Med. 30 (5), 1320-1329 (2024).
- Ghobadinezhad, F., et al. The emerging role of regulatory cell-based therapy in autoimmune disease. Front Immunol. 13, 1075813 (2022).
- Sakaguchi, S., Yamaguchi, T., Nomura, T., Ono, M. Regulatory T cells and immune tolerance. Cell. 133 (5), 775-787 (2008).
- Sakaguchi, S., et al. Regulatory T cells and human disease. Annu Rev Immunol. 38, 541-566 (2020).
- Ferreira, L. M. R., Muller, Y. D., Bluestone, J. A., Tang, Q. Next-generation regulatory T cell therapy. Nat Rev Drug Discov. 18 (10), 749-769 (2019).
- Rosenblum, M. D., Gratz, I. K., Paw, J. S., Abbas, A. K. Treating human autoimmunity: current practice and future prospects. Sci Transl Med. 4 (125), 125sr121 (2012).
- Sawitzki, B., et al. Regulatory cell therapy in kidney transplantation (The ONE Study): a harmonised design and analysis of seven non-randomised, single-arm, phase 1/2A trials. Lancet. 395 (10237), 1627-1639 (2020).
- Spanier, J. A., et al. Tregs with an MHC class II peptide-specific chimeric antigen receptor prevent autoimmune diabetes in mice. J Clin Invest. 133 (18), e168601 (2023).
- Muller, Y. D., et al. Precision engineering of an anti-HLA-A2 chimeric antigen receptor in regulatory T cells for transplant immune tolerance. Front Immunol. 12, 686439 (2021).
- MacDonald, K. G., et al. Alloantigen-specific regulatory T cells generated with a chimeric antigen receptor. J Clin Invest. 126 (4), 1413-1424 (2016).
- Boardman, D. A., et al. Flagellin-specific human CAR Tregs for immune regulation in IBD. J Autoimmun. 134, 102961 (2023).
- Schreeb, K., et al. Study design: human leukocyte antigen Cclass I molecule A(*)02-chimeric antigen receptor regulatory T cells in renal transplantation. Kidney Int Rep. 7 (6), 1258-1267 (2022).
- Zimmerman, C. M., Robino, R. A., Cochrane, R. W., Dominguez, M. D., Ferreira, L. M. R. Redirecting human conventional and regulatory T cells using chimeric antigen receptors. Methods Mol Biol. 2748, 201-241 (2024).
- Tang, Q., et al. Selective decrease of donor-reactive T(regs) after liver transplantation limits T(reg) therapy for promoting allograft tolerance in humans. Sci Transl Med. 14 (669), eabo2628 (2022).
- Bender, C., et al. A phase 2 randomized trial with autologous polyclonal expanded regulatory T cells in children with new-onset type 1 diabetes. Sci Transl Med. 16 (746), eadn2404 (2024).
- Ferreira, L. M. Conference report: Advanced Therapies Week 2023. Regen Med. 18 (4), 297-299 (2023).
- Cochrane, R. W., et al. How to test human CAR T cells in solid tumors, the next frontier of CAR T cell therapy. Methods Mol Biol. 2748, 243-265 (2024).
- Roy, A., Krzykwa, E., Lemieux, R., Neron, S. Increased efficiency of gamma-irradiated versus mitomycin C-treated feeder cells for the expansion of normal human cells in long-term cultures. J Hematother Stem Cell Res. 10 (6), 873-880 (2001).
- Eyquem, J., et al. Targeting a CAR to the TRAC locus with CRISPR/Cas9 enhances tumour rejection. Nature. 543 (7643), 113-117 (2017).
- Negrini, M., Wang, G., Heuer, A., Bjorklund, T., Davidsson, M. AAV Production everywhere: a simple, fast, and reliable protocol for in-house AAV vector production based on chloroform extraction. Curr Protoc Neurosci. 93 (1), e103 (2020).
- Velasco Cardenas, R. M., et al. Harnessing CD3 diversity to optimize CAR T cells. Nat Immunol. 24 (12), 2135-2149 (2023).
- Boomer, J. S., Green, J. M. An enigmatic tail of CD28 signaling. Cold Spring Harb Perspect Biol. 2 (8), a002436 (2010).
- Suhoski, M. M., et al. Engineering artificial antigen-presenting cells to express a diverse array of co-stimulatory molecules. Mol Ther. 15 (5), 981-988 (2007).
- Fung, V. C. W., Rosado-Sanchez, I., Levings, M. K. Transduction of human T cell subsets with lentivirus. Methods Mol Biol. 2285, 227-254 (2021).
- Bailey-Bucktrout, S. L., et al. Self-antigen-driven activation induces instability of regulatory T cells during an inflammatory autoimmune response. Immunity. 39 (5), 949-962 (2013).
- Nakagawa, H., et al. Instability of Helios-deficient Tregs is associated with conversion to a T-effector phenotype and enhanced antitumor immunity. Proc Natl Acad Sci U S A. 113 (22), 6248-6253 (2016).
- Dawson, N. A. J., et al. Functional effects of chimeric antigen receptor co-receptor signaling domains in human regulatory T cells. Sci Transl Med. 12 (557), eaaz3866 (2020).
- Rana, J., et al. CAR- and TRuC-redirected regulatory T cells differ in capacity to control adaptive immunity to FVIII. Mol Ther. 29 (9), 2660-2676 (2021).
- Battaglia, M., Stabilini, A., Tresoldi, E. Expanding human T regulatory cells with the mTOR-inhibitor rapamycin. Methods Mol Biol. 821, 279-293 (2012).
- Brady, B. L., Steinel, N. C., Bassing, C. H. Antigen receptor allelic exclusion: an update and reappraisal. J Immunol. 185 (7), 3801-3808 (2010).
- Samarasinghe, S., et al. Functional characterization of alloreactive T cells identifies CD25 and CD71 as optimal targets for a clinically applicable allodepletion strategy. Blood. 115 (2), 396-407 (2010).
- Voss, K., et al. FOXP3 protects conventional human T cells from premature restimulation-induced cell death. Cell Mol Immunol. 18 (1), 194-205 (2021).
- Collison, L. W., Vignali, D. A. In vitro Treg suppression assays. Methods Mol Biol. 707, 21-37 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados