Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Erzeugung humaner chimärer Antigenrezeptor-regulatorischer T-Zellen
In diesem Artikel
Zusammenfassung
Dieses Protokoll bietet einen optimierten Arbeitsablauf zur Generierung und zum Testen von humanen chimären Antigenrezeptor-regulatorischen T-Zellen (CAR-Tregs).
Zusammenfassung
Die T-Zell-Therapie mit chimären Antigenrezeptoren (CAR) hat das Gesicht der Krebsbehandlung verändert und zu Rekordremissionsraten bei bisher unheilbaren hämatologischen Krebsarten geführt. Diese Erfolge haben das Interesse an der Anpassung der CAR-Plattform an eine kleine, aber entscheidende Untergruppe von CD4+ T-Zellen geweckt, die in erster Linie für die Regulierung und Hemmung der Immunantwort verantwortlich sind, die regulatorischen T-Zellen (Tregs). Die Fähigkeit, die immunsuppressive Aktivität von Tregs auf ein beliebiges extrazelluläres Ziel umzuleiten, hat enorme Auswirkungen auf die Entwicklung von Zelltherapien für Autoimmunerkrankungen, die Abstoßung von Organtransplantaten und die Graft-versus-Host-Krankheit. Hier beschreiben wir detailliert Methoden für die Bona-fide-Treg-Isolierung aus humanem peripherem Blut, die genetische Modifikation von humanen Tregs unter Verwendung von Lentivirus- oder CRISPR/Cas9-gestütztem Knock-in unter Verwendung von Adeno-assoziierten Virus-vermittelten homologen gerichteten Reparatur-Templates (HDR) und die ex vivo-Expansion stabiler humaner CAR-Tregs. Abschließend beschreiben wir die Bewertung der phänotypischen Stabilität und der in vitro suppressiven Funktion von humanen CAR-Tregs, die Einblicke in das Verhalten der humanen CAR-Tregs in präklinischen und klinischen Anwendungen gibt.
Einleitung
T-Zell-Therapien mit chimären Antigenrezeptoren (CAR) haben die Behandlung hämatologischer Malignome revolutioniert und bemerkenswert hohe Remissionsraten bei bisher nicht behandelbaren Krebsarten erzielt 1,2. Ermutigende erste Ergebnisse mit CAR-T-Zellen zur Behandlung von Glioblastomen 3,4,5 unterstreichen die Vielseitigkeit und das zukünftige Potenzial der CAR-Technologie, ein breites Spektrum von Malignomen zu bekämpfen. Im Zuge der Erforschung weiterer Anwendungen von CARs haben sich regulatorische T-Zellen (Tregs) als vielversprechender Zelltyp herauskristallisiert. Tregs spielen eine entscheidende Rolle bei der Aufrechterhaltung der Immunhomöostase und der Regulierung von Immunantworten durch verschiedene Mechanismen, darunter die Sequestrierung von IL-2, die Sekretion immunsuppressiver Zytokine und die Modulation antigenpräsentierender Zellen 6,7.
Mit der CAR-Technologie könnten Tregs für die Behandlung von Abstoßung von Organtransplantaten, Autoimmunerkrankungen und entzündlichen Erkrankungen wie Allergien und Asthma eingesetzt werden 6,8,9. CAR-Tregs könnten zu signifikanten Verbesserungen der Patientenergebnisse und der Lebensqualität führen, indem sie den Einsatz von Immunsuppressiva reduzieren, die das Immunsystem als Ganzes hemmen und mit schädlichen Nebenwirkungen verbunden sind10,11. Präklinische Modelle haben vielversprechende Ergebnisse bei der Übertragung der CAR-Technologie auf Tregs gezeigt, mit erfolgreichen Anwendungen bei Krankheiten wie Typ-1-Diabetes, Multipler Sklerose, Graft-versus-Host-Reaktion und entzündlichen Darmerkrankungen 9,12,13,14,15. In der Klinik werden derzeit CAR-Tregs erforscht, um die Abstoßung von Organtransplantaten zu verhindern16.
In diesem Artikel wird eine detaillierte Methodik zur Generierung von humanen chimären Antigenrezeptor-regulatorischen T-Zellen (CAR Tregs) vorgestellt. Dieses Protokoll beinhaltet die Isolierung von Tregs aus menschlichem peripherem Blut und deren genetische Modifikation mit Techniken wie lentiviraler Transduktion und präzisem Gen-Knock-in unter Verwendung von CRISPR/Cas9-Gen-Editing und Adeno-assoziierten Viren (AAV)-Vektoren. Wir beschreiben auch die Bewertung der phänotypischen Stabilität und suppressiven Funktion dieser manipulierten Tregs, die entscheidende Schritte zur Validierung ihres therapeutischen Potenzials darstellen 17,18,19. Dieser Ansatz rationalisiert das Design und die frühe Erprobung von CAR-Treg-Therapien, die das Potenzial haben, die transformative Wirkung der CAR-T-Zelltherapie zur Regulierung des Immunsystems zu erweitern. Durch den Austausch unserer Methodik hoffen wir, weitere Forschung und Innovation im aufkeimenden CAR-Treg-Therapiebereich anzuregen 9,20.
Protokoll
1. Isolation von menschlichen Treg
- Leukopak-Verarbeitung
- Übertragen Sie den Inhalt des Leukopaks in ein konisches 50-ml-Röhrchen. Fügen Sie ein gleiches Volumen von Dulbeccos phosphatgepufferter Kochsalzlösung (DPBS) +2 % fötalem Rinderserum (FBS) hinzu, indem Sie es vorsichtig mit einer Pipette mischen.
- Bei 300 × g 10 min bei Raumtemperatur (RT) schleudern. Den Überstand vorsichtig ansaugen. Rekonstituieren Sie das Zellpellet in 2 mL DPBS + 2 % FBS. Geben Sie 8 ml Ammoniumchloridlösung per Pipette im Verhältnis 4:1 in die Zellsuspension, mischen Sie durch sanfte Inversion und lassen Sie die verbleibenden roten Blutkörperchen 15 Minuten lang auf Eis lysieren.
- Bei 300 × g 10 min bei RT schleudern. Den Überstand vorsichtig ansaugen. Fügen Sie 30 ml DPBS + 2% FBS hinzu, um die Zellen zu waschen. Bei ausgeschalteter Bremse die gewaschenen Zellen 10 min lang bei RT bei 150 × g schleudern. Den Überstand vorsichtig ansaugen. Resuspendieren Sie das Zellpellet in 30 mL DPBS + 2 % FBS.
- Zählen Sie die nun isolierten mononukleären Zellen des peripheren Blutes (PBMCs) mit Trypanblau im Verhältnis 1:1.
- Aufgrund der hohen Zellkonzentration zunächst 10 μL der Zellen 1:100 mit DPBS verdünnen; Mischen Sie dann 10 μl der 1:100 verdünnten Zellen mit 10 μl Trypanblau-Lösung für diese endgültige 1:1-Verdünnung. Wenn Sie einen automatisierten Zellzähler verwenden, korrigieren Sie die Zellzahl, um die 200-fache Verdünnung widerzuspiegeln, indem Sie die angegebene Konzentration mit 100 multiplizieren, da die meisten Zähler von einer 2-fachen Verdünnung ausgehen. Erwarten Sie eine Ausbeute von 1-2,5 × 109 PBMC aus einem 1/10 Leukopak.
- CD4+ T-Zell-Isolierung (negative Selektion)
- 10 8-10 9 PBMC bei 500 × g für 5 min bei RT herunterschleudern und in Zellseparationspuffer (DPBS + 1 mM EDTA + 2 % FBS) bei 5 × 107 Zellen/ml resuspendieren.
HINWEIS: Um genügend menschliche Tregs für die genetische Veränderung zu erhalten, empfehlen wir, mit 1 × 109 PBMCs zu beginnen. - Führen Sie die magnetische Trennung gemäß den Anweisungen des Herstellers für das CD4+ T-Zellanreicherungskit durch.
- Bestimmen Sie die Menge der isolierten CD4+ T-Zellen durch Zählung mit Trypanblau (10 μl Zellen + 10 μl Trypanblau).
- 10 8-10 9 PBMC bei 500 × g für 5 min bei RT herunterschleudern und in Zellseparationspuffer (DPBS + 1 mM EDTA + 2 % FBS) bei 5 × 107 Zellen/ml resuspendieren.
- CD8+ T-Zell-Isolierung (negative Selektion)
- 5 × 107 PBMC bei 500 × g für 5 min bei RT drehen. In Zelltrennungspuffer (DPBS + 1 mM EDTA + 2 % FBS) bei 5 × 107 Zellen/ml resuspendieren.
HINWEIS: Wir empfehlen, mit 5 × 107 PBMC zu beginnen, um 2-5 × 106 CD8+ T-Zellen zu erhalten. - Führen Sie die magnetische Trennung gemäß den Anweisungen des Herstellers für das CD8+ T-Zellanreicherungskit durch.
- Bestimmen Sie die Anzahl der isolierten CD8+ T-Zellen durch Zählung mit Trypanblau (10 μl Zellen + 10 μl Trypanblau).
- 5 × 107 PBMC bei 500 × g für 5 min bei RT drehen. In Zelltrennungspuffer (DPBS + 1 mM EDTA + 2 % FBS) bei 5 × 107 Zellen/ml resuspendieren.
- Treg-Fluoreszenz-unterstützte Zellsortierung (FACS)
- CD4+ T-Zellen wie in Schritt 1.2 beschrieben isolieren und am nächsten Tag über Nacht in DPBS mit 2 % FBS bei 4 °C (minimaler Verlust an Zellzahl und Lebensfähigkeit) für FACS lagern. Bestimmen Sie die Anzahl der CD4+ T-Zellen aus Schritt 1.2, indem Sie mit Trypanblau zählen (10 μl Zellen + 10 μl Trypanblau).
- CD4+ Zellen bei 500 × g für 5 min drehen. Rekonstituieren Sie Zellen in 200 μl DPBS.
- Pro 1 × 106 Zellen 1 μl Anti-Human-CD4-FITC, 1 μL Anti-Human-CD25 APC und 1 μL Anti-Human-CD127 PE hinzufügen. Vorsichtig aufwirbeln und für 30 Min. in den 4 °C dunklen Kühlschrank stellen.
- Waschen Sie die Zellen mit 10 mL DPBS mit 2% FBS. Bei 500 × g 5 Min. schleudern. Resuspendieren Sie die gefärbten Zellen vorsichtig bei 1,5 × 107 Zellen/ml in DPBS mit 2 % FBS. Dies ist die empfohlene Zellkonzentration für die Sortierung mittels fluoreszenzaktivierter Zellsortierung (FACS).
- Führen Sie die gefärbte Zellsuspension durch eine 40-μm-Filterkappe in FACS-Röhrchen und halten Sie die Röhrchen dann auf Eis.
- Bereiten Sie 15 ml-Entnahmeröhrchen mit 3 ml RPMI10-Medium vor und legen Sie es auf Eis.
HINWEIS: Dieses Medium besteht aus RPMI1640 Basalmedium, 10% FBS, 1x Penicillin-Streptomycin, 1x L-Alanyl-L-Glutamin, 1x nicht-essentiellen Aminosäuren, 1x Natriumpyruvat und 1x HEPES. - CD4+CD25mit hohem CD127-regulatorischen T-Zellum (Tregs) und CD4+CD25mit niedrigem CD127+ konventionellerT-Zellen (Tconv) werden mit FACS sortiert, wie in Abbildung 1A gezeigt.
- Bestimmung der Zellausbeute und -lebensfähigkeit; Fahren Sie dann mit der nachgelagerten Analyse oder der T-Zell-Aktivierung fort.
2. Aktivierung der T-Zellen
- Isolierte T-Zellen mit Trypanblau zählen (10 μl Zellen + 10 μl Trypanblau).
- Waschen Sie 25 μl Anti-CD3/CD28-Kügelchen (106 Kügelchen) pro 1 × 106 erhaltenen T-Zellen, wobei ein Verhältnis von Kügelchen zu T-Zellen von 1:1 beibehalten wird. Waschen Sie Anti-CD3/CD28-Kügelchen, indem Sie DPBS hinzufügen, 3 Minuten lang auf einem Magneten inkubieren und den jetzt verdünnten Anti-CD3/CD28-Kügelchenpuffer entfernen, der für die Zellen giftig sein kann.
- Entfernen Sie das Röhrchen vom Magneten und resuspendieren Sie die gewaschenen Kügelchen in RPMI10 Medium, um 1 × 106 Kügelchen/1 ml RPMI10 zu erhalten.
- Resuspendieren Sie T-Zellen mit Anti-CD3/CD28-Kügelchen in RPMI10-Medium in einer Konzentration von 1 × 106 T-Zellen/ml. Fügen Sie dann 1.000 IE/ml IL-2 zu Treg-Zellen, 100 IE/ml IL-2 zu CD4+ Tconv-Zellen und 300 IE/ml IL-2 zu CD8+ T-Zellen hinzu.
- Kultivieren Sie T-Zellen in 1 × 106 Zellen pro Well einer 24-Well-Platte mit unterschiedlichen Mengen an Anti-CD3/CD28-Kügelchen und IL-2. Stellen Sie die 24-Well-Platte in einen 37 °C großen Inkubator mit 5 % CO2 -Gewebekultur.
- Expandieren Sie aktivierte T-Zellen für 9-12 Tage in Gegenwart von IL-2, bis sie für Experimente oder Reaktivierung mit Anti-CD3/CD28-Kügelchen für zusätzliche Expansion verwendet werden. Wechseln Sie das Medium alle 2-3 Tage und fügen Sie IL-2 jedes Mal hinzu, wenn frisches Medium hinzugefügt wird. Halten Sie die Zelldichte der Tregs während der Expansion bei 5 × 105 bis 1 × 106 Zellen pro ml durch Zählen der Zellen oder durch Sichtprüfung vor der Teilung.
3. Humane Treg-lentivirale Transduktion
- Resuspendieren und Tregs 48 h nach der Aktivierung zählen. Bei 500 ×g für 5 min bei RT drehen. Tregs in RPMI10 bei 1,25 × 106 Zellen/ml mit 1.000 IE/ml IL-2 resuspendieren.
- Die Zentrifuge auf 32 °C vorwärmen.
- Tauen Sie das Lentivirus-haltige CAR-Konstrukt auf Eis auf.
HINWEIS: Wir lagern Lentivirus-Aliquots zum Einmalgebrauch mit genügend Lentiviren, um 2,5 × 105 Zellen bei einem MOI von 1 bei -80 °C zu transduzieren. - Jedes Lentivirus-Aliquot wird mit 2,5 × 105 Tregs in 200 μl in ein Mikrozentrifugenröhrchen gegeben, wobei jedes Röhrchen eine Reaktion anzeigt. Spinokulieren Sie bei 1.000 × g für 1 h bei 32 °C.
- Verschieben Sie jede 200-μl-Reaktion auf eine 24-Well-Platte. Stellen Sie sicher, dass das Medium die gesamte Vertiefung abdeckt. Inkubieren Sie die 24-Well-Platte mit den transduzierten Tregs über Nacht (16-18 h) in einem Gewebekultur-Inkubator. Füllen Sie jede Vertiefung auf 2 mL mit RPMI10-Medium auf, wobei die endgültige IL-2-Konzentration 1.000 IE/ml beträgt.
- Setzen Sie die Expansion der transduzierten Tregs 9-12 Tage nach der Aktivierung fort, indem Sie die Zellen teilen und bei Bedarf (alle 2-3 Tage) mit frischem, vorgewärmtem RPMI10 und IL-2 ergänzen. Die ideale Konzentration der Zellen beträgt 5 × 105-1 × 106 Zellen/ml.
- Beurteilen Sie die Effizienz der Genmodifikation mithilfe der Durchflusszytometrie, wie in Abbildung 2 gezeigt.
HINWEIS: Unsere lentiviralen CAR-Konstrukte enthalten einen Myc-Tag am N-Terminus des CAR-Gens und ein GFP-Reportergen, das über ein 2A-Peptid mit dem CAR-Gen verbunden ist, was eine Quantifizierung der Transduktionseffizienz ohne Antikörperfärbung (GFP) und die Bestätigung der CAR-Oberflächenexpression (Myc-Tag) ermöglicht. Wir beurteilen die Effizienz der Genmodifikation 5 Tage nach der Transduktion. Die Transduktionseffizienz kann je nach Spender und verwendetem CAR-Konstrukt zwischen 30 % und 70 % variieren. CAR+ -Zellen können auf Wunsch sortiert werden. - Stellen Sie sicher, dass die modifizierten Tregs ihren Aktivierungszyklus (9-12 Tage) abschließen und 24 Stunden in Abwesenheit von IL-2 ruhen, bevor sie in Experimenten verwendet werden.
4. CRISPR/Cas9-vermittelter Gen-Knock-in bei menschlichen Tregs
- Resuspendieren Sie die Tregs und überführen Sie sie 48 h nach der Aktivierung in ein konisches 15-ml-Röhrchen. Inkubieren Sie die Zellsuspension 3 Minuten lang in einem Magneten.
- Während Sie sich im Magneten befinden, übertragen Sie die Zellen im Medium per Pipette in ein neues Röhrchen. Die Anti-CD3/CD28-Kügelchen bleiben an der Tubenwand haften. Lassen Sie die entbeaded Tregs nach der Raupenentfernung 2 Stunden lang in RPMI10 ruhen, um sich von der unmittelbaren Belastung durch das Abperlen zu erholen und so die zukünftige Transduktionseffizienz und die Erholung von der Elektroporation zu verbessern.
- Zählen Sie die entperlten Tregs mit Trypanblau.
- Reduziertes Serummedium ohne FBS auf 37 °C vorwärmen.
- Bereiten Sie eine 6-Well-Platte mit 2,5 mL RPMI10-Medium ohne Antibiotika (Penicillin-Streptomycin) und mit 1.000 I.E./ml IL-2 pro Well vor. Die Platte in einem Gewebekultur-Inkubator auf 37 °C vorwärmen.
- Tauen Sie die Adeno-assoziierte Viren (AAV)-haltige HDR-Vorlage (Homology-Directed Repair) auf Eis auf.
HINWEIS: Wir lagern Einweg-Aliquots mit genügend AAV, um 4 × 106 Zellen bei einem MOI von 20.000 bei -80 °C zu infizieren. - Tregs bei 500 × g 5 min drehen. Nach dem Dekantieren des Überstands werden die Zellen in einem vorgewärmten reduzierten Serummedium bei 4 × 107 Zellen/ml resuspendiert.
- Aliquot Zellen in 100 μl in 1,5 ml-Zentrifugenröhrchen mit geringer Proteinbindung. Geben Sie CAR AAV mit einem MOI von 20.000 zu jeder Probe und resuspendieren Sie. Inkubieren Sie die Reaktionsgefäße im Gewebekultur-Inkubator für 1 h.
- Während der 1-stündigen Inkubation bereiten Sie CRISPR/Cas9-Ribonukleoprotein-Komplexe (RNP) vor und assemblieren sie, indem Sie langsam 8,3 μl Cas9-Protein (1 mg/ml-Stamm) zu 2,5 μl sgRNA hinzufügen, die auf den TRAC-Genlocus (100 μM-Stamm) abzielt, für ein molares Verhältnis von Cas9 zu sgRNA von 1:1 und ein RNP-Gesamtvolumen von 10,8 μl pro Probe. Zum Mischen vorsichtig auf und ab pipettieren. Inkubieren Sie das RNP-Gemisch für 15 min bei 37°C im Gewebekultur-Inkubator.
HINWEIS: RNP-Komplexe können danach bei RT verbleiben, bis sie gebrauchsfertig sind. - Füllen Sie ein frisches Elektroporationsröhrchen (E-Rohr) mit 3 mL Elektroporationspuffer mit hoher Osmolarität. Führen Sie das gefüllte E-Rohr in die Pipettenstation des Elektroporationssystems ein, bis ein Klicken zu hören ist. Stellen Sie die Elektroporationsbedingungen im Elektroporationssystem auf 2.200 V, 20 ms, 1 Impuls ein.
- Wenn die 1-stündige Inkubation mit AAV abgeschlossen ist, drehen Sie die Zellen mit AAV bei 300 × g für 5 Minuten bei RT. Aspirieren Sie den Überstand vorsichtig und resuspendieren Sie das Zellpellet in 100 μl des Zellresuspensionspuffers, der vom Elektroporationssystem pro Probe bereitgestellt wird.
HINWEIS: Arbeiten Sie schnell und vermeiden Sie es, die Zellen länger als 15 Minuten im Puffer zu lassen. Vermeiden Sie die Bildung von Blasen. - Fügen Sie 10,8 μl RNP-Komplex pro Probe hinzu. Mit einer Pipette gut mischen, ohne Blasen zu bilden.
- Führen Sie eine 100-μl-Elektroporationsspitze ein, indem Sie die Pipette bis zum zweiten Anschlag drücken, um die Klemme zu öffnen. Positionieren Sie den oberen Kopf der Pipette in der Elektroporationsspitze, bis die Klemme sicher in den Befestigungsschaft des Kolbens einrastet. Lassen Sie den Knopf allmählich los, während Sie den Druck nach unten auf die Pipette aufrechterhalten, um sicherzustellen, dass die Spitze eng anliegt und keine Lücken aufweist.
- Drücken Sie die Pipette bis zum ersten Anschlag und tauchen Sie die Elektroporationsspitze in das Zell-RNP-Gemisch. Ziehen Sie die Probe vorsichtig und blasenfrei in die Pipette ein.
HINWEIS: Während der Elektroporation dürfen keine Blasen in der Spitze vorhanden sein. - Führen Sie die Pipette mit der montierten Elektroporationsspitze, die die Probe enthält, senkrecht in das E-Rohr ein, bis ein Klickgeräusch zu hören ist. Vermeiden Sie die Bildung von Blasen.
- Vergewissern Sie sich, dass die optimalen Einstellungen für menschliche Tregs eingegeben wurden (2.200 V, 20 ms, 1 Impuls) und drücken Sie Start auf dem Touchscreen, um die Zellen zu elektropolieren.
- Warten Sie, bis auf dem Touchscreen nach Abschluss der Elektroporation Abgeschlossen angezeigt wird. Entfernen Sie vorsichtig die Pipette und geben Sie die Probe sofort in die vorbereitete 6-Well-Platte mit 2,5 ml vorgewärmtem, antibiotikafreiem RPMI10-Medium mit 1.000 IE/ml IL-2 pro Well. Wiederholen Sie den Vorgang mit den restlichen Proben. Bewegen Sie die Platte vorsichtig in linearen Bewegungen (von links nach rechts, von oben nach unten), um eine gleichmäßige Verteilung der Zellen in jeder Vertiefung zu gewährleisten, und legen Sie sie in den Gewebekultur-Inkubator.
HINWEIS: Die Wiederverwendung einer Elektroporationsspitze ist bis zu 3x akzeptabel, wenn dieselben Zellen und RNP-Komplexe verwendet werden. Warten Sie eine Erholungsphase von 30 Minuten, bevor Sie die Zellen zählen oder sie in irgendeiner Weise stören, z. B. indem Sie auf Wunsch einen HDR-Enhancer hinzufügen. - Am nächsten Tag, 16-18 Stunden später, ersetzen Sie das Medium durch antibiotikahaltige Medien, zählen Sie die elektroporierten Tregs mit Trypanblau und kultivieren Sie bei 106 Zellen/ml mit 1.000 IE/ml IL-2. Setzen Sie die Expansion der elektroporierten Tregs fort, indem Sie die Zellen spalten und frisches RPMI10 und IL-2 ergänzen, wie in Schritt 2.6 beschrieben.
- Bewahren Sie die Proben für die Analyse durch Durchflusszytometrie auf Eis auf.
- Richten Sie Steuerelemente für die Kompensation einzelner Farben ein und wenden Sie sie auf die Experimentdatei an.
- Lesen Sie zuerst die ungefärbte Probe ab, um die FSC- und SSC-Werte so anzupassen, dass die Lymphozytenpopulation in der Mitte des Punktdiagramms "Alle Ereignisse" liegt.
- Einrichtung einer Gating-Strategie von Lymphozyten in Nicht-Trümmer/Einzelzellen in lebensfähige Zellen in CD4+ -Zellen, wie in Abbildung 3C dargestellt.
- Beurteilen Sie die Effizienz von Genmodifikationen mithilfe der Durchflusszytometrie.
HINWEIS: Unsere AAV-CAR-Konstrukte enthalten zusätzlich zu den TRAC-Locus-Homologiearmen ein verkürztes epidermaler Wachstumsfaktorrezeptor (EGFRt)-Reportergen, das durch ein 2A-Peptid mit dem CAR-Gen verbunden ist, wie in Abbildung 3 gezeigt. Die Knock-in-Effizienz von Genen wird durch Oberflächenfärbung von CD3 bestimmt, deren Verlust auf einen Verlust der TCR-Oberflächenexpression und damit auf ein erfolgreiches Targeting des TRAC-Locus mit CRISPR/Cas9 hinweist, und EGFRt, dessen Expression auf eine erfolgreiche Integration des CAR-Transgens hinweist. CAR-Knock-in-Tregs sind CD3-EGFRt+-Zellen. Wir beurteilen die Effizienz der Genmodifikation 5 Tage nach der Elektroporation. - Stellen Sie sicher, dass modifizierte Tregs ihren Aktivierungszyklus (9-12 Tage) abschließen und 24 Stunden in Abwesenheit von IL-2 ruhen, bevor sie in Experimenten verwendet werden.
5. Aktivierung des menschlichen CAR-Tregs
- Einrichtung der Co-Kultur (Tag 0)
- Sammeln Sie die CAR-Antigen-exprimierende Zielzelllinie in einem konischen Röhrchen.
HINWEIS: Wir verwenden K562-Zellen, eine humane myeloische Leukämie-Zelllinie, der die HLA-, CD80- und CD86-Expression fehlt, als Zielzellen für die CAR-Treg-Aktivierung. Als Negativkontrolle werden elterliche K562-Zellen verwendet und CAR-Antigen-exprimierendes K562 wird verwendet, um die CAR-Tregs17,21 zu aktivieren. - Bestrahlen Sie Zielzelllinien mit 4.000 rad in einem Cäsium-137- oder Röntgenbestrahlungsgerät. Falls kein Zugang zu einem Bestrahlungsgerät besteht, ist eine Mitomycin-C-Behandlung durchzuführen, um die Zellproliferation zu stoppen und gleichzeitig die Expression des Oberflächenantigens in den Zielzellen aufrechtzuerhalten22.
- Falls noch nicht geschehen, entbeaden Sie die Tregs, indem Sie die an die Anti-CD3/CD28-Kügelchen gebundenen Tregs resuspendieren und in ein konisches 15-ml-Röhrchen übertragen. Inkubieren Sie die Zellsuspension in einem Magneten für 3-5 min. Während Sie sich noch im Magneten befinden, übertragen Sie die Zellen im Medium per Pipette in ein neues Röhrchen. Die Anti-CD3/CD28-Kügelchen bleiben an der Tubenwand haften.
- Bestimmen Sie die Konzentrationen der bestrahlten Zielzelle und des entbeadten CAR Treg mit Trypanblau.
- Spin bestrahlte Zielzellen und CAR-Tregs bei 500 × g für 5 min. Resuspendieren Sie mit vorgewärmtem RPMI10 bei 106 Zellen/ml in ihren separaten Röhrchen. Fügen Sie IL-2 zu CAR-Tregs für eine Konzentration von 2.000 IE/ml hinzu.
HINWEIS: Diese Menge an IL-2 wird 2-fach verdünnt, sobald die CAR-Tregs mit den Zielzellen kombiniert werden, um eine endgültige IL-2-Konzentration von 1.000 IE/ml zu erreichen. - Co-Kultur von 1 × 105 CAR-Tregs (100 μL) mit 1 × 105 (100 μL) CAR-Antigen-negativen, bestrahlten Zielzellen (Negativkontrolle für die Aktivierung), 1 × 105 (100 μL) CAR-Antigen-positiven, bestrahlten Zielzellen (Experiment) oder 2,5 μL Anti-CD3/CD28-Kügelchen (Positivkontrolle für die Aktivierung) und 97,5 μL RPMI10-Medium in einer runden 96-Well-Bodenplatte. Stellen Sie die Platte für 48 Stunden in einen 37 °C und 5 % CO2 -Inkubator.
HINWEIS: Das endgültige Volumen pro Vertiefung beträgt 200 μl. Stellen Sie sicher, dass jede Bedingung drei Replikate hat.
- Sammeln Sie die CAR-Antigen-exprimierende Zielzelllinie in einem konischen Röhrchen.
- Auslesung der Durchflusszytometrie
- Wenn kein Plattenlesegerät verwendet wird, suspendieren Sie den Inhalt jeder Vertiefung von der 96-Well-Platte mit rundem Boden und übertragen Sie ihn in ein FACS-Röhrchen. Bei 500 x g 5 min schleudern. Dekantieren Sie den Überstand und wirbeln Sie das Zellpellet vorsichtig vortexen.
- Bereiten Sie einen Antikörper-Mastermix für die Durchflusszytometrie vor: 100 μl/Probe mit DPBS, Anti-Human-CD4 PE/Cy7 1:200, Anti-Human-CD71 PE 1:100 und Ghost Viability Dye BV510 1:2000.
HINWEIS: Dieses Panel funktioniert gut, wenn das CAR-Reportergen GFP ist. Kodiert ein Reportergen für ein Oberflächenprotein, das eine Antikörperfärbung erfordert, wie z. B. EGFRt, kann beispielsweise ein FITC-konjugierter Antikörper verwendet werden. - Pipettieren Sie 100 μl des Antikörper-Mastermixes in jede Probe, mischen Sie es vorsichtig und inkubieren Sie es 30 Minuten lang im 4 °C-Kühlschrank.
- Mit 500 μl DPBS durch Schleudern bei 500 × g für 5 min waschen. Resuspendieren Sie das Zellpellet in 200 μl DPBS.
- Bewahren Sie die Proben für die Analyse durch Durchflusszytometrie auf Eis auf.
- Richten Sie Steuerelemente für die Kompensation einzelner Farben ein und wenden Sie sie auf die Experimentablaufdatei an.
- Lesen Sie ungefärbte Proben, um die SSC- und FSC-Werte so anzupassen, dass die Lymphozytenpopulation in der Mitte des Punktdiagramms "Alle Ereignisse" liegt.
- Einrichtung einer Gating-Strategie von Lymphozyten in Nicht-Trümmer/Einzelzellen in lebensfähige Zellen in CD4+ -Zellen, wie in Abbildung 3C dargestellt (Verschiedene verwendete Fluorophore).
- Lesen Sie Beispiele mit einer Ereignisrate von etwa 1.500 Ereignissen/s, um die Treg-Aktivierung zu bewerten.
HINWEIS: Das erwartete Ergebnis ist eine Hochregulierung der CD71-Oberflächenexpression in CAR-Tregs in Gegenwart von CAR-Antigen, wie in Abbildung 4 gezeigt. Wenn in Gegenwart des CAR-Antigens keine Hochregulation beobachtet wird, könnte ein tonischer Signalweg vorliegen.
6. Human CAR Treg-Stabilität
- Aufbau und Erweiterung der Co-Kultur (Tage 0-9)
- Richten Sie Co-Kulturen ein, um CAR-Tregs zu aktivieren, wie in Schritt 5.1 beschrieben.
- Nach 48 Stunden wird die Co-Kultur aus jeder 96-Well-Plattenvertiefung mit rundem Boden in eine 24-Well-Plattenvertiefung mit vorgewärmten 2 ml RPMI10 mit 1.000 IE/ml IL-2 übertragen, um die Zellexpansion zu ermöglichen.
- Fügen Sie frisches, vorgewärmtes RPMI10 mit 1.000 IE/ml IL-2 hinzu und teilen Sie es nach Bedarf in weitere 24-Well-Platten auf.
- Auslesung der Durchflusszytometrie
- Resuspendieren Sie den Inhalt jedes Replikats und übertragen Sie ihn in ein konisches 15-ml- oder 50-ml-Röhrchen. Bestimmen Sie die Zellkonzentrationen.
- Für jedes Replikat werden zwischen 5 × 105 und 1 × 106 Zellen in ein FACS-Röhrchen übertragen. Bei 500 × g 5 Min. schleudern.
- Bereiten Sie den Durchflusszytometrie-Antikörper-Mastermix für die Oberflächenfärbung vor: 100 μl/Probe mit DPBS, Anti-Human-CD4 PE/Cy7 1:200, Anti-Human-CD25 APC 1:200 und Ghost Viability Dye BV510 1:2000. Bei Bedarf FITC-konjugierten Antikörper für das CAR-Reporterprotein einschließen.
- Dekantieren Sie den Überstand aus Schritt 6.2.2 und wirbeln Sie das Küvettenpellet vorsichtig ein. Pipettieren Sie 100 μl des oberflächenfärbenden Antikörper-Mastermixes in jedes FACS-Röhrchen. Kurz aufwirbeln und für 30 min im Dunkeln in den 4 °C Kühlschrank stellen.
- Bereiten Sie unter Verwendung eines Transkriptionsfaktor-Färbepuffers einen Fixierungs-/Permeabilisierungspuffer vor, indem Sie 3 Volumen Fixierungs-/Permeabilisierungsverdünnungsmittel zu 1 Volumen Fixierungs-/Permeabilisierungskonzentrat hinzufügen. Für jede Probe werden 100 μl Fixierungs-/Permeabilisierungspuffer benötigt.
- Oberflächengefärbte Zellen werden mit 500 μl DPBS gewaschen. Bei 500 × g 5 min schleudern und den Überstand dekantieren. Pipettieren Sie 100 μl vorbereiteten Fixierungs-/Permeabilisierungspuffer in jedes Röhrchen. Kurz vortexen und bei 4 °C für 30-60 min im Dunkeln fixieren lassen.
- Bereiten Sie unter Verwendung eines Transkriptionsfaktor-Färbepuffers 1x Permeabilisierungspuffer her, indem Sie 9 Volumen destilliertes Wasser zu 1 Volumen Permeabilization Buffer 10x Konzentrat hinzufügen. Jede Probe benötigt 1.000 μl Permeabilisierungspuffer zum Waschen und 100 μl Permeabilisierungspuffer zum Färben mit Antikörpern, die auf intrazelluläre Proteine abzielen.
- Waschen Sie die fixierten/permeabilisierten Zellen durch Zugabe von 500 μl 1x Permeabilisierungspuffer. Bei 500 × g 5 min bei RT drehen.
- Bereiten Sie einen intrazellulären Färbe-Antikörper-Mastermix mit 1x Permeabilisierungspuffer, Anti-Human-FOXP3 eFluor 450 1:50, Anti-Human-HELIOS PE 1:50 und Anti-Human-CTLA-4 PerCP-e710 1:50 vor. Für jede Probe werden 100 μl Antikörper-Mastermix benötigt.
HINWEIS: Dieses Panel funktioniert, wenn das CAR-Reportergen GFP ist oder wenn das CAR-Reporterprotein (z. B. EGFRt) mit einem FITC-konjugierten Antikörper gefärbt ist. - Dekantieren Sie den Überstand aus Schritt 6.2.8. 100 μl des intrazellulären Färbeantikörper-Mastermixes zugeben, kurz vortexen und 30 min bei RT im Dunkeln inkubieren.
- Waschen Sie die gefärbten fixierten/permeabilisierten Zellen durch Zugabe von 500 μl 1x Permeabilisierungspuffer. Bei 500 × g 5 Min. schleudern. Dekantieren Sie den Überstand, resuspendieren Sie das Zellpellet in 300 μl DPBS und lagern Sie es auf Eis.
- Analysieren Sie durch Durchflusszytometrie, wie in Abbildung 5 gezeigt. Das erwartete Ergebnis ist, dass die meisten CAR-Tregs FOXP3+HELIOS+ -Zellen sein werden. Verwenden Sie CD4+ Tconv-Zellen als Negativkontrolle für die FOXP3- und HELIOS-Färbung.
7. Menschliche CAR-Treg-Unterdrückung
- Responder-T-Zellen (Tresp)-Zellspurenfärbung und Aktivierung über Nacht
- Sammeln Sie die expandierten CAR-Treg-Zellen, frisch isolierten CD4+ Tconv-Zellen und frisch isolierten CD8+ T-Zellen in separate konische 15-ml-Röhrchen und entbeaden Sie die Anti-CD3/CD28-Kügelchen, falls dies noch nicht geschehen ist.
- Bereiten Sie bestrahlte CAR-Antigen-exprimierende Zielzellen vor, wie in Schritt 5.1 beschrieben.
- Bestimmen Sie die Konzentrationen von nicht aktivierten T-Zellen und bestrahlten Zielzellen.
- Kombinieren Sie 5 × 106 CD4+ Tconv-Zellen mit 5 × 106 CD8+ T-Zellen (Verhältnis 1:1). Dies sind die Responder-T-Zellen (Tresp), die im Assay durch CAR-Tregs gehemmt werden sollen. Tresp-Zellen bei 500 × g für 5 min bei RT drehen. Den Überstand vorsichtig aspirieren und Tresp-Zellen in 1 ml DPBS resuspendieren.
- Geben Sie 1 ml 5 mM CellTrace Violet (CTV)-Farbstoff, der in DMSO rekonstituiert wurde, zu den 107 Tresp-Zellen in 1 ml DPBS für eine Endkonzentration von 5 μM CTV. Für 20 min in ein Wasserbad bei 37 °C stellen. Nach 10 Minuten vorsichtig vortexen, um das abgesetzte CTV neu zu verteilen.
- Mit 9 ml vorgewärmtem RPMI10 Complete Medium waschen. Bei 500 × g 5 Min. schleudern. In 5 mL vorgewärmtem RPMI10 Komplettmedium resuspendieren.
- Bestimmen Sie die CTV-markierte Tresp-Zellkonzentration. 5 × 104 CTV-markierte, nicht aktivierte Tresp-Zellen in 200 μl RPMI10-Medium in 3-6 Vertiefungen einer 96-Well-Platte mit rundem Boden als minimale Proliferationskontrolle hinzufügen.
- Aktivieren Sie CTV-markierte Tresp-Zellen mit Anti-CD3/CD28-Kügelchen in einem Verhältnis von 1:10 Kügelchen zu Tresp-Zellen ohne IL-2 im RPM10-Medium . Geben Sie über Nacht 106 CTV-markierte Tresp-Zellen in 1 ml RPMI10-Medium pro Vertiefung einer 24-Well-Platte in den Gewebekultur-Inkubator.
- Parallel dazu erhältst du 106 CAR+ Tregs. Diese Menge umfasst Tripflats von vier CAR-Treg:Tresp-Verhältnissen, 1:1, 1:2, 1:4, 1:8, bei 5 × 104 Zielzellen pro 96-Well-Well-Well-Platte mit rundem Boden, als 5 × 104 × 3 + 2,5 × 104 × 3 + 1,25 × 104 × 3 + 0,625 × 104 × 3 = 2,81 × 105 CAR-Tregs, über zwei CAR-Treg-Aktivierungsbedingungen: bestrahlte CAR-Antigen-negative Zielzellen (Negativkontrolle, keine Aktivierung) und bestrahlte CAR-Antigen-positive Zielzellen (Experiment, CAR-Aktivierung).
- 3 × 105 CAR-Tregs mit 3 × 105 bestrahlten CAR-Antigen-negativen Zellen (Verhältnis 1:1) und 3 × 105 CAR-Tregs mit 3 × 105 bestrahlten CAR-Antigen-positiven Zielzellen (Verhältnis 1:1) in zwei getrennten konischen 15-ml-Röhrchen kombinieren und bei 500 × g für 5 min bei RT drehen. Den Überstand vorsichtig aspirieren.
- Das Pellet wird in jedem Röhrchen aus Schritt 7.1.10 in 600 μl vorgewärmtem RPMI10-Medium resuspendiert. Daraus ergeben sich 200 μL für jeden der 1:1 Treg: Tresp-Dreifache. Führen Sie die serielle Verdünnung auf einer 96-Well-Platte mit rundem Boden wie folgt durch:
- Aus den vorbereiteten 600 μl Treg + Zielzellsuspension pipettieren Sie 200 μl Zellsuspension in jede der 3 Vertiefungen im Verhältnis 1:1.
- Geben Sie 100 μl vorgewärmtes RPMI10-Komplettmedium in jede der leeren Vertiefungen mit den Verhältnissen 1:2, 1:4 und 1:8.
- Pipettieren Sie für jedes der Triplikate 100 μl der Zellsuspension aus den 1:1-Vertiefungen in die zugehörigen Vertiefungen im Verhältnis 1:2.
- Pipettieren Sie für jedes der Triplikate 100 μl der Zellsuspension aus den 1:2-Vertiefungen in die zugehörigen Vertiefungen im Verhältnis 1:4.
- Pipettieren Sie für jedes der Triplikate 100 μl der Zellsuspension aus den 1:4-Vertiefungen in die zugehörigen Vertiefungen im Verhältnis 1:8.
- Pipettieren Sie die restlichen 100 μl der Zellsuspension aus den Vertiefungen im Verhältnis 1:8 in einen Abfallbehälter.
HINWEIS: Jede Vertiefung sollte 100 μl der Zellsuspension mit entweder 5 × 104 (1:1), 2,5 × 104 (1:2), 1,25 × 104 (1:4) oder 0,625 × 104 (1:8) CAR-Tregs und einer gleichen Anzahl bestrahlter Zielzellen enthalten.
- Co-Inkubation von CAR-Treg- und Tresp-Zellen
- Nach 16-18 Stunden nach der Aktivierung werden aktivierte Tresp-Zellen aus der 24-Well-Platte in ein konisches Röhrchen gefüllt und die Anti-CD3/CD28-Kügelchen per Magnet entfernt.
- Bestimmen Sie die Anzahl der entperlten CTV-markierten Tresp-Zellen.
- 2 × 106 Tresp-Zellen bei 500 × g 5 Min. waschen und schleudern. Den Überstand vorsichtig ansaugen. Resuspendieren Sie die Zellen in 4 ml RPMI10 Komplettmedium.
- Geben Sie 100 μl der T-Zell-Suspension (5 × 104 T-Zellen) in jede Vertiefung mit CAR-Tregs sowie in 3-6 Vertiefungen mit 100 μl RPMI10-Medium allein, um eine maximale Proliferationskontrolle zu gewährleisten. Stellen Sie die Platte für 72 Stunden in einen 37 °C, 5 % CO2 -Gewebekultur-Inkubator.
HINWEIS: Die 96-Well-Platte mit rundem Boden verfügt nun über 3-6 Wells mit nicht aktivierten Tresp-Zellen allein (minimale Proliferationskontrolle), 3-6 Wells mit aktivierten Tresp-Zellen allein (maximale Proliferationskontrolle) und aktivierte Tresp-Zellen bei abnehmender Anzahl von CAR-Tregs.
- Auslesung der Durchflusszytometrie
- Resuspendieren Sie den Inhalt jeder 96-Well-Vertiefung mit runder Bodenplatte und überführen Sie ihn in ein beschriftetes FACS-Röhrchen. Alternativ können Sie auf eine 96-Well-V-Bodenplatte umsteigen, wenn ein Durchflusszytometer mit Plattenablesemodus verfügbar ist. Bei 500 × g 5 Min. schleudern.
- In der Zwischenzeit ist der Antikörper-Mastermix mit DPBS, Anti-Human-CD4 PE/Cy7 1:200 und Anti-Human-CD8 PerCP 1:200 vorzubereiten. Für jede Probe werden 100 μl Antikörper-Mastermix benötigt.
- Wirbeln Sie das Zellpellet vorsichtig ein. Pipettieren Sie 100 μl Antikörper-Mastermix in jedes FACS-Röhrchen. Kurz vortexen und bei 4 °C 30 min im Dunkeln inkubieren.
- Mit 500 μl DPBS waschen. Waschen Sie 2x mit 100 μl DPBS, wenn Sie einen 96-Well-V-Boden verwenden. Bei 500 × g 5 Min. schleudern. Dekantieren Sie den Überstand, resuspendieren Sie das Zellpellet in 200 μl DPBS und lagern Sie die Röhrchen im Dunkeln auf Eis.
- Analysieren Sie durch Durchflusszytometrie, wie in Abbildung 6 gezeigt. Das erwartete Ergebnis besteht darin, dass nicht aktivierte Tresp-Zellen allein (minimale Proliferation) einen einheitlich hohen CTV-Fluoreszenzpeak aufweisen, aktivierte Tresp-Zellen allein mehrere Peaks der CTV-Intensität aufweisen, von denen einer jeder Zellteilung entspricht (maximale Proliferation), und aktivierte CD4+ - und CD8+ -T-Zellen in Gegenwart von aktivierten CAR-Tregs eine Verringerung der Anzahl und Höhe der CTV-Peaks aufweisen. daher in der Proliferation.
- Berechnen Sie die Treg-Zell-vermittelte Suppression wie folgt:
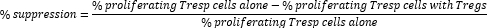
HINWEIS: Wenn Sie die FlowJo-Software verwenden, ist es auch möglich, die Zellproliferationsmodellierung zu verwenden, um den Teilungsindex (DI) für jede Probe zu berechnen und die prozentuale Suppression mit DI anstelle des Prozentsatzes der proliferierenden Zellen in der obigen Formel zu berechnen.
Ergebnisse
Das hier beschriebene Protokoll bietet eine optimierte und standardisierte Pipeline zur Bewertung neuer chimärer Antigenrezeptor (CAR)-Konstrukte in humanen regulatorischen T-Zellen (Tregs) mit dem Ziel, lebende Therapeutika für Autoimmunerkrankungen, Graft-versus-Host-Krankheit, Abstoßung von Organtransplantaten und Allergien zu entwickeln. Abbildung 1 zeigt, wie wir mit Hilfe von FACS hochreine humane Tregs aus peripherem Blut gewinnen (
Diskussion
Dieses Protokoll bietet eine optimierte und umfassende Methodik zur Generierung und Bewertung von humanen chimären Antigenrezeptor-regulatorischen T-Zellen (CAR-Tregs). Der Erfolg der CAR-Technologie bei der Behandlung hämatologischer Krebserkrankungen hat ihre Anwendung auf die immunsuppressive Untergruppe der T-Zellen, die Tregs, inspiriert. Im Gegensatz zu herkömmlichen T-Zellen hemmen Tregs Immunreaktionen und bieten potenzielle Behandlungen für Erkrankungen, die aus unerwünscht...
Offenlegungen
LMRF ist Erfinder und hat Lizenzgebühren aus Patenten auf gentechnisch veränderte Immunzellen erhalten und berät Guidepoint Global und McKesson. Die übrigen Autoren erklären keine konkurrierenden Interessen.
Danksagungen
Das LMRF wird finanziert durch das Human Islet Research Network (HIRN) Emerging Leader in Type 1 Diabetes Grant U24DK104162-07, das American Cancer Society (ACS) Institutional Research Grant IRG-19-137-20, das South Carolina Clinical and Translational Research (SCTR) Pilot Project Discovery Grant 1TL1TR001451-01, das Diabetes Research Connection (DRC) Grant IPF 22-1224 und das Swim Across America Grant 23-1579. Das RWC wird durch das Cellular, Biochemical and Molecular Sciences Training Grant T32GM132055 und das Hollings Cancer Center Lowvelo Graduate Fellowship unterstützt. Diese Studie wurde teilweise von der gemeinsamen Ressource für Durchflusszytometrie und Zellsortierung des Hollings Cancer Center der Medizinischen Universität von South Carolina (P30 CA138313) unterstützt. Besonderer Dank geht an Dr. Qizhi Tang von der University of California, San Francisco (UCSF) für die freundliche Spende der CAR-mutierten Plasmide.
Materialien
| Name | Company | Catalog Number | Comments |
| Adeno-associated virus (AAV) | Charles River Laboratories | ||
| CAR target-expressing K562 cells | e.g., CD19-K562 | ||
| Cesium-137 irradiator | |||
| Anti-human CD8 PerCP (clone SK1) | Biolegend | 344708 | |
| Anti-human CD4 PE/Cy7 (clone SK3) | Biolegend | 344612 | |
| DynaMag-15 magnet | ThermoFisher | 12301D | |
| Ghost BV510 viability dye | TONBO | 13-0870-T100 | |
| K562 cells | American Type Culture Collection | CCL-243 | |
| 0.5 M EDTA, pH 8.0 | Gibco | 15575020 | |
| 1 M HEPES | Gibco | 15630080 | |
| Ammonium chloride solution | STEMCELL Technologies | 7850 | |
| Anti-human CD127 PE (clone hIL-7R-M21) | BD Biosciences | 557938 | |
| Anti-human CD25 APC (clone BC96) | Biolegend | 302610 | |
| Anti-human CD4 FITC (clone SK3) | Biolegend | 344604 | |
| Anti-human CD71 PE (clone SK1) | Biolegend | 334106 | |
| Anti-human CD8 PerCP (clone SK1) | Biolegend | 344707 | |
| Anti-human CTLA-4 PerCP-e710 | ThermoFisher | 46-1529-42 | |
| Anti-human EGFR APC (clone AY13) | Biolegend | 352905 | |
| Anti-human FOXP3 eFluor 450 | ThermoFisher | 48-4776-42 | |
| Anti-human HELIOS PE | Biolegend | 137216 | |
| Ca2+ and Mg2+ free Dulbecco’s Phosphate Buffered Saline (DPBS) | Gibco | 14190144 | |
| Cell counter (TC20 Automated Cell Counter) | Bio-Rad | 1450102 | |
| Cell Counting Slides | Bio-Rad | 1450016 | |
| CellTrace Violet Cell Proliferation Kit | ThermoFisher | C34571 | |
| DNA LoBind Tubes | Eppendorf | 22431021 | |
| Easy 50 EasySep magnet | STEMCELL Technologies | 18002 | |
| EasySep Human CD4+ T cell Enrichment Kit | STEMCELL Technologies | 19052 | |
| EasySep Human CD8+ T cell Enrichment Kit | STEMCELL Technologies | 19053 | |
| EasySep magnet | STEMCELL Technologies | 18000 | |
| eBioscience Foxp3 transcription factor staining buffer set | ThermoFisher | 00-5523-00 | |
| Falcon Round-Bottom Polystyrene Test Tubes with Cell Strainer Snap Cap, 5 mL | Fisher Scientific | 08-771-23 | 40μm |
| Fetal Bovine Serum (FBS) | Gibco | 26140079 | |
| Flow cytometer | Beckman Coulter | CytoFLEX LX U3-V5-B3-Y5-R3-I2 | |
| Fluorescence-activated cell sorter | BD Biosciences | FACS Aria III Cell Sorter | |
| GlutaMAX | Gibco | 35050061 | |
| Human CD3/28 T Cell Expansion and Activation Dynabeads | Gibco | 11131D | |
| Invitrogen Neon Transfection System | ThermoFisher | 10431915 | |
| Invitrogen Neon Transfection System 100 μL Kit | ThermoFisher | 10114334 | |
| Lentivirus | VectorBuilder | ||
| MEM Non-Essential Amino Acids Solution | Gibco | 11140050 | |
| Myc Tag antibody A647 (clone 9B11) | Cell Signaling Technologies | 2233S | |
| Opti-MEM I Reduced Serum Medium | ThermoFisher | 31985062 | |
| Penicilin-Streptomycin solution | Gibco | 15140122 | |
| Recombinant human interleukin-2 (rhIL-2) | Peprotech | 200-02 | |
| RPMI 1640 medium, no glutamine | Gibco | 11875093 | |
| Sodium pyruvate | Gibco | 11360070 | |
| Spectral Flow Cytometer | Cytek | Northern Lights | |
| TRAC gRNA | Synthego | Sequence (CAGGGTTCTGGATATCTGT) | |
| TrueCut Cas9 Protein v2 | ThermoFisher | A36496 | |
| Trypan Blue solution | Sigma | T8154-100ML | |
| 1/10 Leukopak | STEMCELL Technologies | 200-0092 | 1-2 billion PBMC |
Referenzen
- Zhang, X., Zhu, L., Zhang, H., Chen, S., Xiao, Y. CAR-T cell therapy in hematological malignancies: current opportunities and challenges. Front Immunol. 13, 927153 (2022).
- Cappell, K. M., Kochenderfer, J. N. Long-term outcomes following CAR T cell therapy: what we know so far. Nat Rev Clin Oncol. 20 (6), 359-371 (2023).
- Choi, B. D., et al. Intraventricular CARv3-TEAM-E T cells in recurrent glioblastoma. N Engl J Med. 390 (14), 1290-1298 (2024).
- Brown, C. E., et al. Locoregional delivery of IL-13Ralpha2-targeting CAR-T cells in recurrent high-grade glioma: a phase 1 trial. Nat Med. 30 (4), 1001-1012 (2024).
- Bagley, S. J., et al. Intrathecal bivalent CAR T cells targeting EGFR and IL13Ralpha2 in recurrent glioblastoma: phase 1 trial interim results. Nat Med. 30 (5), 1320-1329 (2024).
- Ghobadinezhad, F., et al. The emerging role of regulatory cell-based therapy in autoimmune disease. Front Immunol. 13, 1075813 (2022).
- Sakaguchi, S., Yamaguchi, T., Nomura, T., Ono, M. Regulatory T cells and immune tolerance. Cell. 133 (5), 775-787 (2008).
- Sakaguchi, S., et al. Regulatory T cells and human disease. Annu Rev Immunol. 38, 541-566 (2020).
- Ferreira, L. M. R., Muller, Y. D., Bluestone, J. A., Tang, Q. Next-generation regulatory T cell therapy. Nat Rev Drug Discov. 18 (10), 749-769 (2019).
- Rosenblum, M. D., Gratz, I. K., Paw, J. S., Abbas, A. K. Treating human autoimmunity: current practice and future prospects. Sci Transl Med. 4 (125), 125sr121 (2012).
- Sawitzki, B., et al. Regulatory cell therapy in kidney transplantation (The ONE Study): a harmonised design and analysis of seven non-randomised, single-arm, phase 1/2A trials. Lancet. 395 (10237), 1627-1639 (2020).
- Spanier, J. A., et al. Tregs with an MHC class II peptide-specific chimeric antigen receptor prevent autoimmune diabetes in mice. J Clin Invest. 133 (18), e168601 (2023).
- Muller, Y. D., et al. Precision engineering of an anti-HLA-A2 chimeric antigen receptor in regulatory T cells for transplant immune tolerance. Front Immunol. 12, 686439 (2021).
- MacDonald, K. G., et al. Alloantigen-specific regulatory T cells generated with a chimeric antigen receptor. J Clin Invest. 126 (4), 1413-1424 (2016).
- Boardman, D. A., et al. Flagellin-specific human CAR Tregs for immune regulation in IBD. J Autoimmun. 134, 102961 (2023).
- Schreeb, K., et al. Study design: human leukocyte antigen Cclass I molecule A(*)02-chimeric antigen receptor regulatory T cells in renal transplantation. Kidney Int Rep. 7 (6), 1258-1267 (2022).
- Zimmerman, C. M., Robino, R. A., Cochrane, R. W., Dominguez, M. D., Ferreira, L. M. R. Redirecting human conventional and regulatory T cells using chimeric antigen receptors. Methods Mol Biol. 2748, 201-241 (2024).
- Tang, Q., et al. Selective decrease of donor-reactive T(regs) after liver transplantation limits T(reg) therapy for promoting allograft tolerance in humans. Sci Transl Med. 14 (669), eabo2628 (2022).
- Bender, C., et al. A phase 2 randomized trial with autologous polyclonal expanded regulatory T cells in children with new-onset type 1 diabetes. Sci Transl Med. 16 (746), eadn2404 (2024).
- Ferreira, L. M. Conference report: Advanced Therapies Week 2023. Regen Med. 18 (4), 297-299 (2023).
- Cochrane, R. W., et al. How to test human CAR T cells in solid tumors, the next frontier of CAR T cell therapy. Methods Mol Biol. 2748, 243-265 (2024).
- Roy, A., Krzykwa, E., Lemieux, R., Neron, S. Increased efficiency of gamma-irradiated versus mitomycin C-treated feeder cells for the expansion of normal human cells in long-term cultures. J Hematother Stem Cell Res. 10 (6), 873-880 (2001).
- Eyquem, J., et al. Targeting a CAR to the TRAC locus with CRISPR/Cas9 enhances tumour rejection. Nature. 543 (7643), 113-117 (2017).
- Negrini, M., Wang, G., Heuer, A., Bjorklund, T., Davidsson, M. AAV Production everywhere: a simple, fast, and reliable protocol for in-house AAV vector production based on chloroform extraction. Curr Protoc Neurosci. 93 (1), e103 (2020).
- Velasco Cardenas, R. M., et al. Harnessing CD3 diversity to optimize CAR T cells. Nat Immunol. 24 (12), 2135-2149 (2023).
- Boomer, J. S., Green, J. M. An enigmatic tail of CD28 signaling. Cold Spring Harb Perspect Biol. 2 (8), a002436 (2010).
- Suhoski, M. M., et al. Engineering artificial antigen-presenting cells to express a diverse array of co-stimulatory molecules. Mol Ther. 15 (5), 981-988 (2007).
- Fung, V. C. W., Rosado-Sanchez, I., Levings, M. K. Transduction of human T cell subsets with lentivirus. Methods Mol Biol. 2285, 227-254 (2021).
- Bailey-Bucktrout, S. L., et al. Self-antigen-driven activation induces instability of regulatory T cells during an inflammatory autoimmune response. Immunity. 39 (5), 949-962 (2013).
- Nakagawa, H., et al. Instability of Helios-deficient Tregs is associated with conversion to a T-effector phenotype and enhanced antitumor immunity. Proc Natl Acad Sci U S A. 113 (22), 6248-6253 (2016).
- Dawson, N. A. J., et al. Functional effects of chimeric antigen receptor co-receptor signaling domains in human regulatory T cells. Sci Transl Med. 12 (557), eaaz3866 (2020).
- Rana, J., et al. CAR- and TRuC-redirected regulatory T cells differ in capacity to control adaptive immunity to FVIII. Mol Ther. 29 (9), 2660-2676 (2021).
- Battaglia, M., Stabilini, A., Tresoldi, E. Expanding human T regulatory cells with the mTOR-inhibitor rapamycin. Methods Mol Biol. 821, 279-293 (2012).
- Brady, B. L., Steinel, N. C., Bassing, C. H. Antigen receptor allelic exclusion: an update and reappraisal. J Immunol. 185 (7), 3801-3808 (2010).
- Samarasinghe, S., et al. Functional characterization of alloreactive T cells identifies CD25 and CD71 as optimal targets for a clinically applicable allodepletion strategy. Blood. 115 (2), 396-407 (2010).
- Voss, K., et al. FOXP3 protects conventional human T cells from premature restimulation-induced cell death. Cell Mol Immunol. 18 (1), 194-205 (2021).
- Collison, L. W., Vignali, D. A. In vitro Treg suppression assays. Methods Mol Biol. 707, 21-37 (2011).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten