È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Generazione di cellule T regolatorie del recettore dell'antigene chimerico umano
In questo articolo
Riepilogo
Questo protocollo fornisce un flusso di lavoro semplificato per generare e testare cellule T regolatorie del recettore dell'antigene chimerico umano (CAR Tregs).
Abstract
La terapia con cellule T del recettore chimerico dell'antigene (CAR) ha rimodellato il volto del trattamento del cancro, portando a tassi di remissione record in tumori ematologici precedentemente incurabili. Questi successi hanno stimolato l'interesse nell'adattare la piattaforma CAR a un piccolo ma fondamentale sottogruppo di cellule T CD4+ principalmente responsabili della regolazione e dell'inibizione della risposta immunitaria, le cellule T regolatorie (Treg). La capacità di reindirizzare l'attività immunosoppressiva delle Treg a qualsiasi bersaglio extracellulare ha enormi implicazioni per la creazione di terapie cellulari per le malattie autoimmuni, il rigetto del trapianto d'organo e la malattia del trapianto contro l'ospite. Qui, descriviamo in dettaglio le metodologie per l'isolamento in buona fede delle Treg dal sangue periferico umano, la modificazione genetica delle Treg umane utilizzando lentivirus o knock-in assistito da CRISPR/Cas9 utilizzando la somministrazione di modelli di riparazione diretta omologa (HDR) mediata da virus adeno-associati e l'espansione ex vivo di Treg CAR umane stabili. Infine, descriviamo la valutazione della stabilità fenotipica delle Treg CAR umane e della funzione soppressiva in vitro , che fornisce informazioni su come si comporteranno le Treg CAR umane in applicazioni precliniche e cliniche.
Introduzione
Le terapie con cellule T del recettore chimerico dell'antigene (CAR) hanno rivoluzionato il trattamento delle neoplasie ematologiche, raggiungendo tassi di remissione notevolmente elevati in tumori precedentemente non trattabili 1,2. I primi risultati incoraggianti dell'utilizzo delle cellule T CAR per il trattamento del glioblastoma 3,4,5 evidenziano la versatilità della tecnologia CAR e il potenziale futuro per colpire un'ampia gamma di neoplasie. Mentre il campo esplora ulteriori applicazioni dei CAR, le cellule T regolatorie (Treg) sono emerse come un tipo di cellula promettente. Le Treg svolgono un ruolo cruciale nel mantenimento dell'omeostasi immunitaria e nella regolazione delle risposte immunitarie attraverso diversi meccanismi, tra cui il sequestro di IL-2, la secrezione di citochine immunosoppressive e la modulazione delle cellule presentanti l'antigene 6,7.
Con la tecnologia CAR, le Treg potrebbero essere sfruttate per il trattamento del rigetto del trapianto di organi, delle malattie autoimmuni e dei disturbi infiammatori come le allergie e l'asma 6,8,9. Le Treg CAR potrebbero portare a miglioramenti significativi degli esiti e della qualità della vita dei pazienti riducendo l'uso di farmaci immunosoppressori, che inibiscono il sistema immunitario nel suo complesso e sono associati a effetti collaterali nocivi10,11. I modelli preclinici hanno mostrato risultati promettenti nella traduzione della tecnologia CAR in Tregs, con applicazioni di successo in malattie come il diabete di tipo 1, la sclerosi multipla, la malattia del trapianto contro l'ospite e la malattia infiammatoria intestinale 9,12,13,14,15. Nella clinica, le Treg CAR sono attualmente in fase di studio per prevenire il rigetto del trapianto di organi solidi16.
Questo articolo presenta una metodologia dettagliata per la generazione di cellule T regolatorie del recettore dell'antigene chimerico umano (CAR Tregs). Questo protocollo prevede l'isolamento delle Treg dal sangue periferico umano e la loro modifica genetica utilizzando tecniche come la trasduzione lentivirale e il knock-in genico preciso utilizzando l'editing genetico CRISPR/Cas9 e vettori di virus adeno-associati (AAV). Descriviamo anche la valutazione della stabilità fenotipica e della funzione soppressiva di queste Treg ingegnerizzate, che sono passaggi cruciali per convalidare il loro potenziale terapeutico 17,18,19. Questo approccio semplifica la progettazione e la sperimentazione precoce delle terapie CAR Treg, che hanno il potenziale per estendere l'impatto trasformativo della terapia con cellule T CAR per regolare il sistema immunitario. Condividendo la nostra metodologia, speriamo di ispirare ulteriori ricerche e innovazioni nel fiorente spazio della terapia CAR Treg 9,20.
Protocollo
1. Isolamento delle Treg umane
- Lavorazione di Leukopak
- Trasferire il contenuto del leucopak in una provetta conica da 50 mL. Aggiungere un volume uguale di soluzione fisiologica tamponata con fosfato (DPBS) +2% di siero fetale bovino (FBS) di Dulbco mescolando delicatamente con una pipetta.
- Centrifugare a 300 × g per 10 minuti a temperatura ambiente (RT). Aspirare con cura il surnatante. Ricostituire il pellet cellulare in 2 mL di DPBS + 2% FBS. Aggiungere 8 mL di soluzione di cloruro di ammonio mediante pipetta alla sospensione cellulare in rapporto 4:1, mescolare per leggera inversione e lasciare la lisi dei globuli rossi rimanenti su ghiaccio per 15 minuti.
- Centrifugare a 300 × g per 10 minuti a RT. Aspirare con cura il surnatante. Aggiungere 30 ml di DPBS + 2% FBS per lavare le cellule. Con il freno disinserito, centrifugare le celle lavate a 150 × g per 10 minuti a RT. Aspirare con cautela il surnatante. Risospendere il pellet cellulare in 30 mL di DPBS + 2% FBS.
- Contare le cellule mononucleate del sangue periferico (PBMC) ora isolate con il blu di tripano in un rapporto 1:1.
- A causa dell'elevata concentrazione cellulare, diluire inizialmente 10 μL di cellule 1:100 con DPBS; quindi, mescolare 10 μl delle celle diluite 1:100 con 10 μl di soluzione di blu di tripano per la diluizione finale 1:1. Quando si utilizza un contatore automatico di cellule, correggere il numero di cellule in modo che rifletta la diluizione di 200 volte moltiplicando la concentrazione riportata per 100, poiché la maggior parte dei contatori presuppone una diluizione di 2 volte. Anticipa di produrre 1-2,5 × 109 PBMC da un 1/10 di leucopak.
- Isolamento delle cellule T CD4+ (selezione negativa)
- Centrifugare 10,8-10,9 PBMC a 500 × g per 5 minuti a RT e risospendere in tampone di separazione cellulare (DPBS + 1 mM EDTA + 2% FBS) a 5 × 10,7 cellule/mL.
NOTA: Per ottenere abbastanza Treg umane per la modificazione genetica, si consiglia di iniziare con 1 × 109 PBMC. - Eseguire la separazione magnetica secondo le istruzioni del produttore per il kit di arricchimento delle cellule T CD4+ .
- Determinare la quantità di cellule T CD4+ isolate contando con il blu di tripano (10 μL di cellule + 10 μL di blu di tripano).
- Centrifugare 10,8-10,9 PBMC a 500 × g per 5 minuti a RT e risospendere in tampone di separazione cellulare (DPBS + 1 mM EDTA + 2% FBS) a 5 × 10,7 cellule/mL.
- Isolamento delle cellule T CD8+ (selezione negativa)
- Centrifugare 5 × 107 PBMC a 500 × g per 5 minuti a RT. Risospendere in tampone di separazione cellulare (DPBS + 1 mM EDTA + 2% FBS) a 5 × 107 cellule/mL.
NOTA: Si consiglia di iniziare con 5 × 107 PBMC per ottenere 2-5 × 106 cellule T CD8+ . - Eseguire la separazione magnetica secondo le istruzioni del produttore per il kit di arricchimento delle cellule T CD8+ .
- Determinare il numero di cellule T CD8+ isolate contando con il blu di tripano (10 μL di cellule + 10 μL di blu di tripano).
- Centrifugare 5 × 107 PBMC a 500 × g per 5 minuti a RT. Risospendere in tampone di separazione cellulare (DPBS + 1 mM EDTA + 2% FBS) a 5 × 107 cellule/mL.
- Smistamento cellulare assistito da fluorescenza Treg (FACS)
- Isolare le cellule T CD4+ come descritto al punto 1.2 e conservarle per una notte in DPBS con FBS al 2% a 4 °C (perdita minima di numero e vitalità cellulare) per FACS il giorno successivo. Determinare il numero di cellule T CD4+ dal passaggio 1.2 contando con il blu di tripano (10 μL di cellule + 10 μL di blu di tripano).
- Centrifugare le cellule CD4+ a 500 × g per 5 min. Ricostituire le cellule in 200 μL di DPBS.
- Per 1 × 106 cellule, aggiungere 1 μL di FITC CD4 anti-umano, 1 μL di APC CD25 anti-umano e 1 μL di EP CD127 anti-umano. Agitare delicatamente e riporre in frigorifero buio a 4 °C per 30 min.
- Lavare le celle con 10 mL di DPBS con il 2% di FBS. Centrifugare a 500 × g per 5 min. Risospendere delicatamente le cellule colorate a 1,5 × 107 cellule/mL in DPBS con FBS al 2%. Questa è la concentrazione cellulare raccomandata per la selezione cellulare attivata dalla fluorescenza (FACS).
- Passare la sospensione cellulare colorata attraverso un tappo del filtro da 40 μm nelle provette FACS, quindi mantenere le provette sul ghiaccio.
- Preparare provette da raccolta da 15 mL contenenti 3 mL di terreno RPMI10 e metterle su ghiaccio.
NOTA: Questo terreno è costituito da RPMI1640 terreno basale, 10% FBS, 1x penicillina-streptomicina, 1x L-alanil-L-glutammina, 1x aminoacidi non essenziali, 1x piruvato di sodio e 1x HEPES. - Smistare le cellule T CD4+CD25ad altaregolazione CD127 (Tregs) e le cellule T CD4+CD25a bassoCD127+ (Tconv) utilizzando FACS come mostrato nella Figura 1A.
- Determinare la resa e la vitalità delle cellule; quindi procedere con l'analisi a valle o l'attivazione delle cellule T.
2. Attivazione delle cellule T
- Contare le cellule T isolate con blu di tripano (10 μL di cellule + 10 μL di blu di tripano).
- Lavare 25 μL di perle anti-CD3/CD28 (106 perle) per ogni 1 × 106 cellule T ottenute, mantenendo un rapporto 1:1 tra perle e cellule T. Lavare le perle anti-CD3/CD28 aggiungendo DPBS, incubando su un magnete per 3 minuti e rimuovendo il tampone anti-CD3/CD28 ora diluito, che può essere tossico per le cellule.
- Rimuovere il tubo dal magnete e risospendere le perle lavate in terreno RPMI10 per avere 1 × 106 perline/1 mL di RPMI10.
- Risospendere le cellule T con perle anti-CD3/CD28 in terreno RPMI10 a una concentrazione di 1 × 106 cellule T/mL. Quindi, aggiungere 1.000 UI/mL di IL-2 alle cellule Treg, 100 UI/mL di IL-2 alle cellule Tconv CD4+ e 300 UI/mL di IL-2 alle cellule T CD8+ .
- Coltura di cellule T a 1 × 106 cellule per pozzetto di una piastra a 24 pozzetti con diverse quantità di perle anti-CD3/CD28 e IL-2. Collocare la piastra a 24 pozzetti in un incubatore per colture tissutali a 37 °C, CO2 al 5%.
- Espandere le cellule T attivate per 9-12 giorni in presenza di IL-2 fino a quando non vengono utilizzate per esperimenti o riattivate con perline anti-CD3/CD28 per un'ulteriore espansione. Sostituire il terreno ogni 2-3 giorni e aggiungere IL-2 ogni volta che viene aggiunto un nuovo terreno. Mantenere la densità cellulare delle Treg a 5 × 10da 5 a 1 × 106 cellule per mL durante l'espansione contando le cellule o mediante ispezione visiva prima della scissione.
3. Trasduzione lentivirale delle Treg umane
- Risospendere e contare Tregs 48 h dopo l'attivazione. Centrifugare a 500 ×g per 5 minuti a RT. Risospendere le Treg in RPMI10 a 1,25 × 106 cellule/mL con 1.000 UI/mL di IL-2.
- Preriscaldare la centrifuga a 32 °C.
- Scongelare il costrutto CAR contenente lentivirus sul ghiaccio.
NOTA: Conserviamo aliquote di lentivirus monouso con una quantità di lentivirus sufficiente a trasdurre 2,5 × 105 cellule a un MOI di 1 a -80 °C. - Aggiungere ogni aliquota di lentivirus a 2,5 × 105 Treg in 200 μl in una provetta da microcentrifuga, ogni provetta indica una reazione. Spinoculato a 1.000 × g per 1 h a 32 °C.
- Spostare ogni reazione da 200 μl su una piastra a 24 pozzetti. Assicurati che il supporto copra l'intero pozzo. Incubare la piastra a 24 pozzetti con le Treg trasdotte in un incubatore di coltura tissutale per una notte (16-18 ore). Rabboccare ciascun pozzetto a 2 mL con terreno RPMI10 con la concentrazione finale di IL-2 di 1.000 UI/mL.
- Continuare ad espandere le Treg trasdotte 9-12 giorni dopo l'attivazione dividendo le cellule e integrando con RPMI10 e IL-2 freschi preriscaldati secondo necessità (ogni 2-3 giorni). La concentrazione ideale di cellule è di 5 × 10,5-1 × 10,6 cellule/mL.
- Valutare l'efficienza della modificazione genica utilizzando la citometria a flusso, come mostrato nella Figura 2.
NOTA: I nostri costrutti lentivirali CAR contengono un Myc-tag sull'N-terminale del gene CAR e un gene reporter GFP legato al gene CAR da un peptide 2A, che consente la quantificazione dell'efficienza di trasduzione senza colorazione anticorpale (GFP) e la conferma dell'espressione della superficie CAR (Myc-tag). Valutiamo l'efficienza della modificazione genica 5 giorni dopo la trasduzione. Le efficienze di trasduzione possono variare tra il 30% e il 70% a seconda del donatore e del costrutto CAR utilizzato. Le celle CAR+ possono essere ordinate se lo si desidera. - Assicurarsi che le Treg modificate completino il loro ciclo di attivazione (9-12 giorni) e riposino 24 ore in assenza di IL-2 prima di essere utilizzate in qualsiasi esperimento.
4. Knock-in genico mediato da CRISPR/Cas9 nelle Treg umane
- Risospendere le Treg e trasferirle in una provetta conica da 15 mL 48 ore dopo l'attivazione. Incubare la sospensione cellulare in un magnete per 3 minuti.
- Mentre sono nel magnete, trasferire le cellule nel mezzo tramite pipetta in una nuova provetta. Le perle anti-CD3/CD28 rimarranno attaccate alla parete del tubo. Lasciare riposare le Treg debordate in RPMI10 per 2 ore dopo la rimozione del cordone per riprendersi dallo stress immediato della debeading, migliorando l'efficienza di trasduzione futura e il recupero dall'elettroporazione.
- Conta le Treg debeaded con il tripano blu.
- Preriscaldare il terreno sierologico ridotto senza FBS a 37 °C.
- Preparare una piastra a 6 pozzetti con 2,5 mL di terreno RPMI10 senza antibiotici (penicillina-streptomicina) e con 1.000 UI/mL di IL-2 per pozzetto. Preriscaldare la piastra a 37 °C in un incubatore per colture tissutali.
- Scongelare il modello di riparazione diretta per omologia (HDR) CAR contenente virus adeno-associato (AAV) su ghiaccio.
NOTA: Conserviamo aliquote monouso con AAV sufficiente per infettare 4 × 106 cellule a un MOI di 20.000 a -80 °C. - Centrifugare le tregs a 500 × g per 5 min. Dopo aver travasato il surnatante, risospendere le cellule in un terreno sierologico ridotto preriscaldato a 4 × 107 cellule/mL.
- Aliquotare le cellule in 100 μL in provette da centrifuga da 1,5 mL a basso legame proteico. Aggiungere CAR AAV a un MOI di 20.000 a ciascun campione e risuspendare. Incubare le provette di reazione nell'incubatore per colture tissutali per 1 ora.
- Durante l'incubazione di 1 ora, preparare e assemblare i complessi ribonucleoproteici (RNP) CRISPR/Cas9 aggiungendo lentamente 8,3 μL di proteina Cas9 (1 mg/mL stock) a 2,5 μL di sgRNA che ha come bersaglio il locus del gene TRAC (100 μM stock) per un rapporto molare di Cas9 e sgRNA di 1:1 e un volume totale di RNP di 10,8 μL per campione. Pipettare delicatamente su e giù per mescolare. Incubare la miscela di RNP per 15 minuti a 37°C nell'incubatore per colture tissutali.
NOTA: I complessi RNP possono rimanere in RT in seguito fino al momento dell'uso. - Riempire una nuova provetta per elettroporazione (provetta E) con 3 mL di tampone per elettroporazione ad alta osmolarità. Inserire il tubo E riempito nella stazione di pipettatura del sistema di elettroporazione fino a quando non si sente un clic. Impostare le condizioni di elettroporazione su 2.200 V, 20 ms, 1 impulso nel sistema di elettroporazione.
- Al termine dell'incubazione di 1 ora con AAV, centrifugare le cellule con AAV a 300 × g per 5 minuti a RT. Aspirare con cautela il surnatante e risospendere il pellet cellulare in 100 μl del tampone di risospensione cellulare fornito dal sistema di elettroporazione per campione.
NOTA: Lavorare rapidamente ed evitare di lasciare le celle nel buffer per più di 15 minuti. Evita di creare bolle. - Aggiungere 10,8 μl di complesso RNP per campione. Mescolare bene con una pipetta senza creare bolle.
- Inserire un puntale per elettroporazione da 100 μl spingendo la pipetta fino al secondo arresto per aprire il morsetto. Posizionare la testa superiore della pipetta nel puntale di elettroporazione finché il morsetto non si innesta saldamente con lo stelo di montaggio del pistone. Rilasciare gradualmente il pulsante mantenendo la pressione verso il basso sulla pipetta per assicurarsi che il puntale aderisca perfettamente senza spazi vuoti.
- Premere la pipetta fino al primo arresto e immergere la punta di elettroporazione nella miscela cellula-RNP. Estrarre delicatamente il campione nella pipetta senza bolle.
NOTA: Durante l'elettroporazione non possono essere presenti bolle all'interno della punta. - Inserire verticalmente la pipetta con la punta di elettroporazione montata contenente il campione nel tubo E fino a quando non si sente un clic. Evitare la formazione di bolle.
- Verificare che siano state inserite le impostazioni ottimali per le Treg umane (2.200 V, 20 ms, 1 impulso) e premere start sul touchscreen per elettroporare le celle.
- Attendere che il touchscreen visualizzi Completo al termine dell'elettroporazione. Rimuovere delicatamente la pipetta e trasferire immediatamente il campione nella piastra a 6 pozzetti preparata contenente 2,5 mL di terreno RPMI10 preriscaldato e privo di antibiotici con 1.000 UI/mL di IL-2 per pozzetto. Ripetere con i campioni rimanenti. Agitare delicatamente la piastra con movimenti lineari (da sinistra a destra, dall'alto verso il basso) per garantire una distribuzione uniforme delle cellule in ciascun pozzetto e posizionarla nell'incubatore per colture tissutali.
NOTA: Il riutilizzo di una punta per elettroporazione è accettabile fino a 3 volte se sono in uso le stesse cellule e lo stesso complesso RNP. Attendere un periodo di recupero di 30 minuti prima di contare le celle o disturbarle in qualsiasi modo, ad esempio aggiungendo un potenziatore HDR, se lo si desidera. - Il giorno seguente, 16-18 ore dopo, sostituire il terreno con terreno contenente antibiotici, contare le Treg elettroporate con blu di tripano e coltivare a 106 cellule/mL con 1.000 UI/mL di IL-2. Continuare ad espandere le Treg elettroporate dividendo le celle e integrando RPMI10 e IL-2 freschi come descritto al punto 2.6.
- Conservare i campioni sul ghiaccio per l'analisi mediante citometria a flusso.
- Imposta i controlli di compensazione per un singolo colore e applicali al file dell'esperimento.
- Leggere prima il campione non colorato per regolare i guadagni di FSC e SSC in modo che la popolazione di linfociti si trovi al centro del dot plot "Tutti gli eventi".
- Impostare la strategia di gating dei linfociti in cellule non detritiche/singole in cellule vitali in cellule CD4+ , come mostrato nella Figura 3C.
- Valutare l'efficienza della modificazione genica utilizzando la citometria a flusso.
NOTA: I nostri costrutti AAV CAR contengono, oltre ai bracci di omologia del locus TRAC, un gene reporter del recettore del fattore di crescita epidermico troncato (EGFRt) legato al gene CAR da un peptide 2A, come mostrato nella Figura 3. L'efficienza del knock-in genico è determinata dalla colorazione superficiale per CD3, la cui perdita indica la perdita dell'espressione superficiale del TCR e, quindi, il successo del targeting del locus TRAC con CRISPR/Cas9 e EGFRt, la cui espressione indica una riuscita integrazione del transgene CAR. Le Treg knock-in CAR sono cellule CD3-EGFRt+. Valutiamo l'efficienza della modificazione genica 5 giorni dopo l'elettroporazione. - Assicurarsi che le Treg modificate completino il loro ciclo di attivazione (9-12 giorni) e riposino 24 ore in assenza di IL-2 prima di essere utilizzate in qualsiasi esperimento.
5. Attivazione delle Treg CAR umane
- Configurazione della co-cultura (giorno 0)
- Raccogliere la linea cellulare bersaglio che esprime l'antigene CAR in una provetta conica.
NOTA: Utilizziamo le cellule K562, una linea cellulare di leucemia mieloide umana priva di espressione di HLA, CD80 e CD86 come cellule bersaglio per l'attivazione delle CAR Treg. Le cellule K562 parentali sono utilizzate come controllo negativo e le cellule K562 che esprimono l'antigene CAR sono utilizzate per attivare CAR Tregs17,21. - Irradiare linee cellulari target con 4.000 rad in un irradiatore di cesio-137 o raggi X. In caso di mancanza di accesso a un irradiatore, eseguire il trattamento con mitomicina C per fermare la proliferazione cellulare mantenendo l'espressione dell'antigene di superficie nelle cellule bersaglio22.
- Se non lo avete già fatto, debordate le Treg risospendendo le Treg legate alle perle anti-CD3/CD28 e trasferendole in una provetta conica da 15 mL. Incubare la sospensione cellulare in un magnete per 3-5 minuti. Mentre sono ancora nel magnete, trasferire le cellule nel mezzo tramite pipetta in una nuova provetta; le perle anti-CD3/CD28 rimarranno attaccate alla parete del tubo.
- Determinare le concentrazioni di cellule bersaglio irradiate e di CAR Treg debeaded con blu di tripano.
- Far girare le cellule bersaglio irradiate e le CAR Treg a 500 × g per 5 minuti. Risospendere con RPMI10 preriscaldato a 106 cellule/mL nelle loro provette separate. Aggiungere IL-2 a CAR Tregs per una concentrazione di 2.000 UI/mL.
NOTA: Questa quantità di IL-2 sarà diluita di 2 volte una volta che le Treg CAR saranno combinate con le cellule bersaglio per una concentrazione finale di IL-2 di 1.000 UI/mL. - Co-coltura 1 × 105 CAR Tregs (100 μL) con 1 × 105 (100 μL) cellule bersaglio irradiate negative all'antigene CAR (controllo negativo per l'attivazione), 1 × 105 (100 μL) di cellule bersaglio irradiate positive all'antigene CAR (esperimento) o 2,5 μL di perle anti-CD3/CD28 (controllo positivo per l'attivazione) e 97,5 μL di terreno RPMI10 in una piastra inferiore rotonda a 96 pozzetti. Posizionare la piastra in un incubatore per colture tissutali a 37 °C, 5% CO2 per 48 ore.
NOTA: Il volume finale per pozzetto è di 200 μl. Assicurarsi che ogni condizione abbia tre repliche.
- Raccogliere la linea cellulare bersaglio che esprime l'antigene CAR in una provetta conica.
- Lettura della citometria a flusso
- A meno che non si utilizzi un lettore di piastre, risospendere il contenuto di ciascun pozzetto dalla piastra a fondo tondo da 96 pozzetti e trasferirlo in una provetta FACS. Centrifugare a 500 x g per 5 min. Decantare il surnatante e agitare delicatamente il pellet cellulare.
- Preparare la miscela master di anticorpi per citometria a flusso: 100 μL/campione contenente DPBS, CD4 PE/Cy7 1:200 anti-umano, CD71 PE 1:100 e colorante di vitalità Ghost BV510 1:2000.
NOTA: Questo pannello funziona bene se il gene reporter CAR è GFP. Se un gene reporter codifica per una proteina di superficie che richiede la colorazione degli anticorpi, come l'EGFRt, è possibile utilizzare un anticorpo coniugato FITC, ad esempio. - Pipettare 100 μl della miscela master di anticorpi in ciascun campione, mescolare delicatamente e incubare per 30 minuti in frigorifero a 4 °C.
- Lavare con 500 μl di DPBS centrifugando a 500 × g per 5 min. Risospendere il pellet della cella in 200 μL di DPBS.
- Conservare i campioni sul ghiaccio per l'analisi mediante citometria a flusso.
- Configura i controlli di compensazione a colore singolo e applicali al file di flusso dell'esperimento.
- Leggere i campioni non colorati per regolare i guadagni di SSC e FSC in modo che la popolazione di linfociti si trovi al centro del dot plot "Tutti gli eventi".
- Impostare la strategia di gating dei linfociti in cellule non detritiche/singole in cellule vitali in cellule CD4+ , come mostrato nella Figura 3C (Diversi fluorofori utilizzati).
- Leggere i campioni a una frequenza di circa 1.500 eventi/s per valutare l'attivazione delle Treg.
NOTA: Il risultato atteso è l'upregolazione dell'espressione di superficie di CD71 nelle CAR Tregs in presenza di antigene CAR, come mostrato nella Figura 4. Se la sovraregolazione non è osservata in presenza dell'antigene CAR, potrebbe essere presente una segnalazione tonica.
6. Stabilità delle Treg CAR umane
- Configurazione ed espansione della co-cultura (giorni 0-9)
- Configurare le impostazioni cultura condivise per attivare le reti CAR Tregs come descritto nel passaggio 5.1.
- Dopo 48 ore, trasferire la co-coltura da ciascun pozzetto a fondo tondo da 96 pozzetti in un pozzetto a 24 pozzetti contenente 2 mL di RPMI10 preriscaldati con 1.000 UI/mL di IL-2 per consentire l'espansione cellulare.
- Aggiungere RPMI10 fresco e preriscaldato con 1.000 UI/mL di IL-2 e suddividere in ulteriori piastre da 24 pozzetti, se necessario.
- Lettura della citometria a flusso
- Risospendere e trasferire il contenuto di ciascuna replica in una provetta conica da 15 mL o 50 mL. Determinare le concentrazioni cellulari.
- Trasferire tra 5 × 105 e 1 × 106 cellule in una provetta FACS per ogni replica. Centrifugare a 500 × g per 5 min.
- Preparare la miscela master di anticorpi per citometria a flusso per la colorazione superficiale: 100 μL/campione contenente DPBS, CD4 PE/Cy7 1:200 anti-umano, CD25 APC 1:200 e colorante di vitalità Ghost BV510 1:2000. Se necessario, includere l'anticorpo coniugato FITC per la proteina reporter CAR.
- Decantare il surnatante del passaggio 6.2.2 e agitare delicatamente il pellet della cella. Pipettare 100 μl della miscela master di anticorpi con colorazione superficiale in ciascuna provetta FACS. Agitare brevemente e riporre in frigorifero a 4 °C per 30 minuti al buio.
- Utilizzando un set di tamponi di colorazione del fattore di trascrizione, preparare il tampone di fissazione/permeabilizzazione aggiungendo 3 volumi di diluente di fissazione/permeabilizzazione a 1 volume di concentrato di fissazione/permeabilizzazione. Ogni campione richiede 100 μl di tampone di fissazione/permeabilizzazione.
- Lavare le celle colorate in superficie con 500 μL di DPBS. Centrifugare a 500 × g per 5 minuti e decantare il surnatante. Pipettare 100 μl di tampone di fissaggio/permeabilizzazione preparato su ciascuna provetta. Agitare brevemente e lasciare che la fissazione avvenga a 4 °C per 30-60 minuti al buio.
- Utilizzando un set di tamponi di colorazione del fattore di trascrizione, preparare 1x tampone di permeabilizzazione aggiungendo 9 volumi di acqua distillata a 1 volume di concentrato di tampone di permeabilizzazione 10x. Ogni campione richiede 1.000 μl di tampone di permeabilizzazione per il lavaggio e 100 μl di tampone di permeabilizzazione per la colorazione con anticorpi mirati alle proteine intracellulari.
- Lavare le celle fisse/permeabilizzate aggiungendo 500 μl di tampone di permeabilizzazione 1x. Centrifugare a 500 × g per 5 minuti a RT.
- Preparare la miscela master di anticorpi per la colorazione intracellulare con 1x tampone di permeabilizzazione, FOXP3 eFluor 450 1:50 anti-umano, HELIOS PE 1:50 anti-umano e CTLA-4 PerCP-e710 1:50 anti-umano. Ogni campione richiederà 100 μl di miscela master di anticorpi.
NOTA: Questo pannello funziona se il gene reporter CAR è GFP o se la proteina reporter CAR (ad esempio, EGFRt) è colorata con un anticorpo coniugato FITC. - Decantare il surnatante dal passaggio 6.2.8. Aggiungere 100 μl della miscela master di anticorpi per la colorazione intracellulare, agitare brevemente e incubare a RT per 30 minuti al buio.
- Lavare le cellule fissate/permeabilizzate colorate aggiungendo 500 μl di tampone di permeabilizzazione 1x. Centrifugare a 500 × g per 5 min. Decantare il surnatante, risospendere il pellet cellulare in 300 μl di DPBS e conservarlo in ghiaccio.
- Analizzare mediante citometria a flusso come mostrato nella Figura 5. Il risultato previsto è che la maggior parte delle Treg CAR saranno celle FOXP3+HELIOS+ . Utilizzare le cellule Tconv CD4+ come controllo negativo per la colorazione FOXP3 e HELIOS.
7. Soppressione delle Treg CAR umane
- Colorazione del colorante in tracce di cellule T responder (Tresp) e attivazione notturna
- Raccogliere le cellule CAR Treg espanse, le cellule Tconv CD4+ appena isolate e le cellule T CD8+ appena isolate in provette coniche separate da 15 mL e debeadare le perle anti-CD3/CD28, se non è già stato fatto.
- Preparare le cellule bersaglio irradiate che esprimono l'antigene CAR come descritto al punto 5.1.
- Determinare le concentrazioni delle cellule T non attivate e delle cellule bersaglio irradiate.
- Combinare 5 × 106 cellule Tconv CD4+ con 5 × 106 cellule T CD8+ (rapporto 1:1). Queste sono le cellule T responder (Tresp) che devono essere inibite dalle CAR Treg nel saggio. Far girare le cellule Tresp a 500 × g per 5 minuti a RT. Aspirare con cautela il surnatante e risospendere le cellule Tresp in 1 mL di DPBS.
- Aggiungere 1 mL di colorante CellTrace Violet (CTV) da 5 mM ricostituito in DMSO alle 10cellule Tresp da 7 in 1 mL di DPBS per una concentrazione finale di 5 μM di CTV. Mettere a bagnomaria a 37 °C per 20 min. A 10 minuti, vorticare delicatamente per ridistribuire il CTV stabilizzato.
- Lavare con 9 mL di terreno completo RPMI10 preriscaldato. Centrifugare a 500 × g per 5 min. Risospendere in 5 mL di terreno completo RPMI10 preriscaldato.
- Determinare la concentrazione di cellule Tresp marcate con CTV. Aggiungere 5 × 10 cellule Tresp non attivate marcatecon CTV in 200 μL di terreno RPMI10 in 3-6 pozzetti di una piastra a fondo tondo a 96 pozzetti come controlli di minima proliferazione.
- Attivare le cellule Tresp marcate con CTV con perle anti-CD3/CD28 con un rapporto 1:10 tra perle e cellule Tresp senza IL-2 in terreno RPM10. Erogare 10 cellule Tresp marcatecon CTV in 1 mL di terreno RPMI10 per pozzetto di una piastra da 24 pozzetti nell'incubatore di coltura tissutale durante la notte.
- In parallelo, ottieni 106 CAR+ Tregs. Questa quantità copre triplicati di quattro rapporti CAR Treg:Tresp, 1:1, 1:2, 1:4, 1:8, a 5 × 104 cellule bersaglio per pozzetto a 96 pozzetti a fondo tondo, come 5 × 104 × 3 + 2,5 × 104 × 3 + 1,25 × 104 × 3 + 0,625 × 104 × 3 = 2,81 ×10 5 Treg CAR, in due condizioni di attivazione delle Treg CAR: cellule bersaglio negative all'antigene CAR irradiate (controllo negativo, nessuna attivazione) e cellule bersaglio positive all'antigene CAR irradiate (esperimento, attivazione CAR).
- Combinare 3 × 105 CAR Treg con 3 ×10 5 cellule CAR negative irradiate (rapporto 1:1) e 3 × 105 CAR Treg con 3 × 105 cellule bersaglio CAR positive irradiate (rapporto 1:1), in due provette coniche separate da 15 ml e centrifugare a 500 × g per 5 minuti a RT. Aspirare con cautela il surnatante.
- Risospendere il pellet in ciascuna provetta dal punto 7.1.10 in 600 μl di terreno RPMI10 preriscaldato. Ciò si traduce in 200 μl per ciascuna delle triplicate 1:1 Treg: Tresp. Eseguire la diluizione in serie su una piastra a fondo tondo a 96 pozzetti come segue:
- Dai 600 μL di Treg + sospensione cellulare target, pipettare 200 μL di sospensione cellulare in ciascuno dei 3 pozzetti con rapporto 1:1.
- Aggiungere 100 μl di terreno completo RPMI10 preriscaldato in ciascuno dei pozzetti vuoti con rapporto 1:2, 1:4 e 1:8.
- Per ciascuno dei triplicati, pipettare 100 μl di sospensione cellulare dai pozzetti 1:1 nei pozzetti con rapporto 1:2 associati.
- Per ciascuno dei triplicati, pipettare 100 μl di sospensione cellulare, dai pozzetti 1:2, nei pozzetti con rapporto 1:4 associati.
- Per ciascuno dei triplicati, pipettare 100 μl di sospensione cellulare, dai pozzetti 1:4, nei pozzetti con rapporto 1:8 associati.
- Pipettare i restanti 100 μl della sospensione cellulare dai pozzetti con rapporto 1:8 in un contenitore per rifiuti.
NOTA: Ogni pozzetto deve contenere 100 μl di sospensione cellulare con 5 × 104 (1:1), 2,5 × 104 (1:2), 1,25 × 104 (1:4) o 0,625 × 104 (1:8) Treg e un numero uguale di cellule bersaglio irradiate.
- Co-incubazione di cellule CAR Treg e Tresp
- Dopo 16-18 ore dall'attivazione, raccogliere le cellule Tresp attivate dalla piastra a 24 pozzetti in un tubo conico e rimuovere le perle anti-CD3/CD28 tramite magnete.
- Determinare la conta delle cellule Tresp marcate con CTV debeaded .
- Lavare e centrifugare 2 × 10 6 celle Tresp a 500 × g per 5 min. Aspirare con cura il surnatante. Risospendere le cellule in 4 mL di terreno completo RPMI10.
- Aggiungere 100 μl della sospensione di cellule T (5 × 104 cellule T) a ciascun pozzetto con CAR Tregs, nonché a 3-6 pozzetti con 100 μl di terreno RPMI10 da solo per il massimo controllo della proliferazione. Posizionare la piastra in un incubatore per colture tissutali a 37 °C, 5% CO2 per 72 ore.
NOTA: La piastra a fondo tondo a 96 pozzetti ha ora 3-6 pozzetti con le sole cellule Tresp non attivate (controllo minimo della proliferazione), 3-6 pozzetti con le sole cellule Tresp attivate (massimo controllo della proliferazione) e le cellule Tresp attivate in presenza di un numero decrescente di Treg CAR.
- Lettura della citometria a flusso
- Risospendere e trasferire il contenuto di ciascuna piastra inferiore rotonda a 96 pozzetti in una provetta FACS etichettata. In alternativa, trasferire su una piastra con fondo a V a 96 pozzetti se è disponibile un citometro a flusso con modalità di lettura su piastra. Centrifugare a 500 × g per 5 min.
- Nel frattempo, preparare la master mix di anticorpi con DPBS, anti-umano CD4 PE/Cy7 1:200 e anti-umano CD8 PerCP 1:200. Ogni campione richiederà 100 μl di miscela master di anticorpi.
- Agitare delicatamente il pellet della cella. Pipettare 100 μl di miscela master per anticorpi in ciascuna provetta FACS. Agitare brevemente e incubare a 4 °C per 30 minuti al buio.
- Lavare con 500 μl di DPBS. Lavare 2 volte con 100 μl di DPBS se si utilizza un fondo a V a 96 pozzetti. Centrifugare a 500 × g per 5 min. Decantare il surnatante, risospendere il pellet cellulare in 200 μL di DPBS e conservare le provette su ghiaccio al buio.
- Analizzare mediante citometria a flusso come mostrato nella Figura 6. Il risultato atteso è che le cellule Tresp non attivate da sole (proliferazione minima) mostrino un picco di fluorescenza CTV uniforme e elevato, le cellule Tresp attivate da sole mostrino picchi multipli di intensità CTV, uno corrispondente a ciascuna divisione cellulare (massima proliferazione), e le cellule T CD4+ e CD8+ attivate in presenza di Treg CAR attivate mostrino una riduzione del numero e dell'altezza dei picchi CTV, quindi, in proliferazione.
- Calcola la soppressione cellulo-mediata Treg come segue:
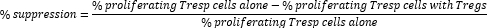
NOTA: Se si utilizza il software FlowJo, è anche possibile utilizzare la modellazione della proliferazione cellulare per calcolare l'indice di divisione (DI) per ciascun campione e calcolare la soppressione percentuale utilizzando DI anziché la percentuale di cellule proliferanti nella formula sopra.
Risultati
Il protocollo qui descritto fornisce una pipeline semplificata e standardizzata per valutare nuovi costrutti di recettori chimerici dell'antigene (CAR) nelle cellule T regolatorie umane (Treg), con l'obiettivo di creare terapie viventi per le malattie autoimmuni, la malattia del trapianto contro l'ospite, il rigetto del trapianto d'organo e l'allergia. La Figura 1 illustra come otteniamo Treg umane altamente pure dal sangue periferico utilizzando FACS (
Discussione
Questo protocollo fornisce una metodologia semplificata e completa per la generazione e la valutazione delle cellule T regolatorie del recettore dell'antigene chimerico umano (CAR Tregs). Il successo della tecnologia CAR nel trattamento dei tumori ematologici ha ispirato la sua applicazione al sottogruppo immunosoppressivo delle cellule T, le Treg. A differenza delle cellule T convenzionali, le Treg inibiscono le risposte immunitarie, offrendo potenziali trattamenti per le condizioni der...
Divulgazioni
LMRF è un inventore e ha ricevuto royalties da brevetti su cellule immunitarie ingegnerizzate e fornisce consulenza per Guidepoint Global e McKesson. Gli altri autori dichiarano di non avere interessi concorrenti.
Riconoscimenti
LMRF è finanziato dalla Human Islet Research Network (HIRN) Emerging Leader in Type 1 Diabetes grant U24DK104162-07, dall'American Cancer Society (ACS) Institutional Research Grant IRG-19-137-20, dal South Carolina Clinical and Translational Research (SCTR) Pilot Project Discovery Grant 1TL1TR001451-01, dal Diabetes Research Connection (DRC) Grant IPF 22-1224 e dal Swim Across America Grant 23-1579. RWC è supportato dalla borsa di studio per la formazione in Scienze Cellulari, Biochimiche e Molecolari T32GM132055 e dalla borsa di studio per laureati Lowvelo dell'Hollings Cancer Center. Questo studio è stato supportato in parte dalla Flow Cytometry and Cell Sorting Shared Resource, Hollings Cancer Center, Medical University of South Carolina (P30 CA138313). Un ringraziamento speciale al Dr. Qizhi Tang dell'Università della California, San Francisco (UCSF) per aver gentilmente donato i plasmidi mutanti CAR.
Materiali
| Name | Company | Catalog Number | Comments |
| Adeno-associated virus (AAV) | Charles River Laboratories | ||
| CAR target-expressing K562 cells | e.g., CD19-K562 | ||
| Cesium-137 irradiator | |||
| Anti-human CD8 PerCP (clone SK1) | Biolegend | 344708 | |
| Anti-human CD4 PE/Cy7 (clone SK3) | Biolegend | 344612 | |
| DynaMag-15 magnet | ThermoFisher | 12301D | |
| Ghost BV510 viability dye | TONBO | 13-0870-T100 | |
| K562 cells | American Type Culture Collection | CCL-243 | |
| 0.5 M EDTA, pH 8.0 | Gibco | 15575020 | |
| 1 M HEPES | Gibco | 15630080 | |
| Ammonium chloride solution | STEMCELL Technologies | 7850 | |
| Anti-human CD127 PE (clone hIL-7R-M21) | BD Biosciences | 557938 | |
| Anti-human CD25 APC (clone BC96) | Biolegend | 302610 | |
| Anti-human CD4 FITC (clone SK3) | Biolegend | 344604 | |
| Anti-human CD71 PE (clone SK1) | Biolegend | 334106 | |
| Anti-human CD8 PerCP (clone SK1) | Biolegend | 344707 | |
| Anti-human CTLA-4 PerCP-e710 | ThermoFisher | 46-1529-42 | |
| Anti-human EGFR APC (clone AY13) | Biolegend | 352905 | |
| Anti-human FOXP3 eFluor 450 | ThermoFisher | 48-4776-42 | |
| Anti-human HELIOS PE | Biolegend | 137216 | |
| Ca2+ and Mg2+ free Dulbecco’s Phosphate Buffered Saline (DPBS) | Gibco | 14190144 | |
| Cell counter (TC20 Automated Cell Counter) | Bio-Rad | 1450102 | |
| Cell Counting Slides | Bio-Rad | 1450016 | |
| CellTrace Violet Cell Proliferation Kit | ThermoFisher | C34571 | |
| DNA LoBind Tubes | Eppendorf | 22431021 | |
| Easy 50 EasySep magnet | STEMCELL Technologies | 18002 | |
| EasySep Human CD4+ T cell Enrichment Kit | STEMCELL Technologies | 19052 | |
| EasySep Human CD8+ T cell Enrichment Kit | STEMCELL Technologies | 19053 | |
| EasySep magnet | STEMCELL Technologies | 18000 | |
| eBioscience Foxp3 transcription factor staining buffer set | ThermoFisher | 00-5523-00 | |
| Falcon Round-Bottom Polystyrene Test Tubes with Cell Strainer Snap Cap, 5 mL | Fisher Scientific | 08-771-23 | 40μm |
| Fetal Bovine Serum (FBS) | Gibco | 26140079 | |
| Flow cytometer | Beckman Coulter | CytoFLEX LX U3-V5-B3-Y5-R3-I2 | |
| Fluorescence-activated cell sorter | BD Biosciences | FACS Aria III Cell Sorter | |
| GlutaMAX | Gibco | 35050061 | |
| Human CD3/28 T Cell Expansion and Activation Dynabeads | Gibco | 11131D | |
| Invitrogen Neon Transfection System | ThermoFisher | 10431915 | |
| Invitrogen Neon Transfection System 100 μL Kit | ThermoFisher | 10114334 | |
| Lentivirus | VectorBuilder | ||
| MEM Non-Essential Amino Acids Solution | Gibco | 11140050 | |
| Myc Tag antibody A647 (clone 9B11) | Cell Signaling Technologies | 2233S | |
| Opti-MEM I Reduced Serum Medium | ThermoFisher | 31985062 | |
| Penicilin-Streptomycin solution | Gibco | 15140122 | |
| Recombinant human interleukin-2 (rhIL-2) | Peprotech | 200-02 | |
| RPMI 1640 medium, no glutamine | Gibco | 11875093 | |
| Sodium pyruvate | Gibco | 11360070 | |
| Spectral Flow Cytometer | Cytek | Northern Lights | |
| TRAC gRNA | Synthego | Sequence (CAGGGTTCTGGATATCTGT) | |
| TrueCut Cas9 Protein v2 | ThermoFisher | A36496 | |
| Trypan Blue solution | Sigma | T8154-100ML | |
| 1/10 Leukopak | STEMCELL Technologies | 200-0092 | 1-2 billion PBMC |
Riferimenti
- Zhang, X., Zhu, L., Zhang, H., Chen, S., Xiao, Y. CAR-T cell therapy in hematological malignancies: current opportunities and challenges. Front Immunol. 13, 927153 (2022).
- Cappell, K. M., Kochenderfer, J. N. Long-term outcomes following CAR T cell therapy: what we know so far. Nat Rev Clin Oncol. 20 (6), 359-371 (2023).
- Choi, B. D., et al. Intraventricular CARv3-TEAM-E T cells in recurrent glioblastoma. N Engl J Med. 390 (14), 1290-1298 (2024).
- Brown, C. E., et al. Locoregional delivery of IL-13Ralpha2-targeting CAR-T cells in recurrent high-grade glioma: a phase 1 trial. Nat Med. 30 (4), 1001-1012 (2024).
- Bagley, S. J., et al. Intrathecal bivalent CAR T cells targeting EGFR and IL13Ralpha2 in recurrent glioblastoma: phase 1 trial interim results. Nat Med. 30 (5), 1320-1329 (2024).
- Ghobadinezhad, F., et al. The emerging role of regulatory cell-based therapy in autoimmune disease. Front Immunol. 13, 1075813 (2022).
- Sakaguchi, S., Yamaguchi, T., Nomura, T., Ono, M. Regulatory T cells and immune tolerance. Cell. 133 (5), 775-787 (2008).
- Sakaguchi, S., et al. Regulatory T cells and human disease. Annu Rev Immunol. 38, 541-566 (2020).
- Ferreira, L. M. R., Muller, Y. D., Bluestone, J. A., Tang, Q. Next-generation regulatory T cell therapy. Nat Rev Drug Discov. 18 (10), 749-769 (2019).
- Rosenblum, M. D., Gratz, I. K., Paw, J. S., Abbas, A. K. Treating human autoimmunity: current practice and future prospects. Sci Transl Med. 4 (125), 125sr121 (2012).
- Sawitzki, B., et al. Regulatory cell therapy in kidney transplantation (The ONE Study): a harmonised design and analysis of seven non-randomised, single-arm, phase 1/2A trials. Lancet. 395 (10237), 1627-1639 (2020).
- Spanier, J. A., et al. Tregs with an MHC class II peptide-specific chimeric antigen receptor prevent autoimmune diabetes in mice. J Clin Invest. 133 (18), e168601 (2023).
- Muller, Y. D., et al. Precision engineering of an anti-HLA-A2 chimeric antigen receptor in regulatory T cells for transplant immune tolerance. Front Immunol. 12, 686439 (2021).
- MacDonald, K. G., et al. Alloantigen-specific regulatory T cells generated with a chimeric antigen receptor. J Clin Invest. 126 (4), 1413-1424 (2016).
- Boardman, D. A., et al. Flagellin-specific human CAR Tregs for immune regulation in IBD. J Autoimmun. 134, 102961 (2023).
- Schreeb, K., et al. Study design: human leukocyte antigen Cclass I molecule A(*)02-chimeric antigen receptor regulatory T cells in renal transplantation. Kidney Int Rep. 7 (6), 1258-1267 (2022).
- Zimmerman, C. M., Robino, R. A., Cochrane, R. W., Dominguez, M. D., Ferreira, L. M. R. Redirecting human conventional and regulatory T cells using chimeric antigen receptors. Methods Mol Biol. 2748, 201-241 (2024).
- Tang, Q., et al. Selective decrease of donor-reactive T(regs) after liver transplantation limits T(reg) therapy for promoting allograft tolerance in humans. Sci Transl Med. 14 (669), eabo2628 (2022).
- Bender, C., et al. A phase 2 randomized trial with autologous polyclonal expanded regulatory T cells in children with new-onset type 1 diabetes. Sci Transl Med. 16 (746), eadn2404 (2024).
- Ferreira, L. M. Conference report: Advanced Therapies Week 2023. Regen Med. 18 (4), 297-299 (2023).
- Cochrane, R. W., et al. How to test human CAR T cells in solid tumors, the next frontier of CAR T cell therapy. Methods Mol Biol. 2748, 243-265 (2024).
- Roy, A., Krzykwa, E., Lemieux, R., Neron, S. Increased efficiency of gamma-irradiated versus mitomycin C-treated feeder cells for the expansion of normal human cells in long-term cultures. J Hematother Stem Cell Res. 10 (6), 873-880 (2001).
- Eyquem, J., et al. Targeting a CAR to the TRAC locus with CRISPR/Cas9 enhances tumour rejection. Nature. 543 (7643), 113-117 (2017).
- Negrini, M., Wang, G., Heuer, A., Bjorklund, T., Davidsson, M. AAV Production everywhere: a simple, fast, and reliable protocol for in-house AAV vector production based on chloroform extraction. Curr Protoc Neurosci. 93 (1), e103 (2020).
- Velasco Cardenas, R. M., et al. Harnessing CD3 diversity to optimize CAR T cells. Nat Immunol. 24 (12), 2135-2149 (2023).
- Boomer, J. S., Green, J. M. An enigmatic tail of CD28 signaling. Cold Spring Harb Perspect Biol. 2 (8), a002436 (2010).
- Suhoski, M. M., et al. Engineering artificial antigen-presenting cells to express a diverse array of co-stimulatory molecules. Mol Ther. 15 (5), 981-988 (2007).
- Fung, V. C. W., Rosado-Sanchez, I., Levings, M. K. Transduction of human T cell subsets with lentivirus. Methods Mol Biol. 2285, 227-254 (2021).
- Bailey-Bucktrout, S. L., et al. Self-antigen-driven activation induces instability of regulatory T cells during an inflammatory autoimmune response. Immunity. 39 (5), 949-962 (2013).
- Nakagawa, H., et al. Instability of Helios-deficient Tregs is associated with conversion to a T-effector phenotype and enhanced antitumor immunity. Proc Natl Acad Sci U S A. 113 (22), 6248-6253 (2016).
- Dawson, N. A. J., et al. Functional effects of chimeric antigen receptor co-receptor signaling domains in human regulatory T cells. Sci Transl Med. 12 (557), eaaz3866 (2020).
- Rana, J., et al. CAR- and TRuC-redirected regulatory T cells differ in capacity to control adaptive immunity to FVIII. Mol Ther. 29 (9), 2660-2676 (2021).
- Battaglia, M., Stabilini, A., Tresoldi, E. Expanding human T regulatory cells with the mTOR-inhibitor rapamycin. Methods Mol Biol. 821, 279-293 (2012).
- Brady, B. L., Steinel, N. C., Bassing, C. H. Antigen receptor allelic exclusion: an update and reappraisal. J Immunol. 185 (7), 3801-3808 (2010).
- Samarasinghe, S., et al. Functional characterization of alloreactive T cells identifies CD25 and CD71 as optimal targets for a clinically applicable allodepletion strategy. Blood. 115 (2), 396-407 (2010).
- Voss, K., et al. FOXP3 protects conventional human T cells from premature restimulation-induced cell death. Cell Mol Immunol. 18 (1), 194-205 (2021).
- Collison, L. W., Vignali, D. A. In vitro Treg suppression assays. Methods Mol Biol. 707, 21-37 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon