Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Génération de lymphocytes T régulateurs du récepteur de l’antigène chimérique humain
Dans cet article
Résumé
Ce protocole fournit un flux de travail rationalisé pour générer et tester des cellules T régulatrices du récepteur antigénique chimérique humain (CAR Tregs).
Résumé
La thérapie par lymphocytes T à récepteur antigénique chimérique (CAR) a remodelé le visage du traitement du cancer, conduisant à des taux de rémission records dans des cancers hématologiques auparavant incurables. Ces succès ont suscité l’intérêt pour l’adaptation de la plateforme CAR à un sous-ensemble restreint mais essentiel de lymphocytes T CD4+ principalement responsables de la régulation et de l’inhibition de la réponse immunitaire, les lymphocytes T régulateurs (Tregs). La capacité de rediriger l’activité immunosuppressive des Tregs vers n’importe quelle cible extracellulaire a d’énormes implications pour la création de thérapies cellulaires pour les maladies auto-immunes, le rejet de greffe d’organe et la maladie du greffon contre l’hôte. Ici, nous décrivons en détail les méthodologies pour l’isolement de bonne foi des Tregs à partir du sang périphérique humain, la modification génétique des Tregs humains à l’aide de lentivirus ou de knock-in assisté par CRISPR/Cas9 à l’aide de la réparation dirigée homologue (HDR) médiée par un virus adéno-associé, et l’expansion ex vivo de CAR Tregs humains stables. Enfin, nous décrivons l’évaluation de la stabilité phénotypique des CAR Tregs humains et de la fonction suppressive in vitro , qui fournit des informations sur la façon dont les CAR Tregs humains se comporteront dans des applications précliniques et cliniques.
Introduction
Les thérapies à base de lymphocytes T à récepteur antigénique chimérique (CAR) ont révolutionné le traitement des hémopathies malignes, atteignant des taux de rémission remarquablement élevés dans des cancers auparavant incurables 1,2. Les premiers résultats encourageants obtenus avec des cellules CAR-T pour traiter le glioblastome 3,4,5 mettent en évidence la polyvalence de la technologie CAR et son potentiel futur à cibler un large éventail de tumeurs malignes. Au fur et à mesure que le domaine explore d’autres applications des CAR, les lymphocytes T régulateurs (Tregs) sont apparus comme un type de cellule prometteur. Les Tregs jouent un rôle crucial dans le maintien de l’homéostasie immunitaire et la régulation des réponses immunitaires par plusieurs mécanismes, notamment la séquestration de l’IL-2, la sécrétion de cytokines immunosuppressives et la modulation des cellules présentatrices d’antigènes 6,7.
Avec la technologie CAR, les Tregs pourraient être exploités pour le traitement du rejet de greffe d’organe, des maladies auto-immunes et des troubles inflammatoires comme les allergies et l’asthme 6,8,9. Les CAR Tregs pourraient entraîner des améliorations significatives des résultats des patients et de la qualité de vie en réduisant l’utilisation de médicaments immunosuppresseurs, qui inhibent le système immunitaire dans son ensemble et sont associés à des effets secondaires nocifs10,11. Les modèles précliniques ont montré des résultats prometteurs dans l’application de la technologie CAR aux Tregs, avec des applications réussies dans des maladies telles que le diabète de type 1, la sclérose en plaques, la maladie du greffon contre l’hôte et les maladies inflammatoires de l’intestin 9,12,13,14,15. En clinique, les CAR Tregs sont actuellement à l’étude pour prévenir le rejet de greffe d’organe solide16.
Cet article présente une méthodologie détaillée pour générer des cellules T régulatrices du récepteur antigénique chimérique humain (CAR Tregs). Ce protocole consiste à isoler les Tregs du sang périphérique humain et à les modifier génétiquement à l’aide de techniques telles que la transduction lentivirale et l’inactivation précise de gènes à l’aide de l’édition de gènes CRISPR/Cas9 et de vecteurs de virus adéno-associés (AAV). Nous décrivons également l’évaluation de la stabilité phénotypique et de la fonction suppressive de ces Tregs modifiés, qui sont des étapes cruciales pour valider leur potentiel thérapeutique 17,18,19. Cette approche simplifie la conception et les tests précoces des thérapies CAR Treg, qui ont le potentiel d’étendre l’impact transformateur de la thérapie par cellules CAR-T pour réguler le système immunitaire. En partageant notre méthodologie, nous espérons inspirer d’autres recherches et innovations dans le domaine en plein essor de la thérapie CAR Treg 9,20.
Protocole
1. Isolement des Tregs humains
- Traitement de Leukopak
- Transférez le contenu du leukopak dans un tube conique de 50 ml. Ajoutez un volume égal de solution saline tamponnée au phosphate (DPBS) de Dulbecco + 2 % de sérum de veau fœtal (FBS) en mélangeant doucement avec une pipette.
- Essorer à 300 × g pendant 10 min à température ambiante (RT). Aspirez soigneusement le surnageant. Reconstituer la pastille cellulaire dans 2 mL de DPBS + 2 % FBS. Ajouter 8 mL de solution de chlorure d’ammonium à l’aide d’une pipette à la suspension cellulaire dans un rapport de 4:1, mélanger par inversion douce et laisser lyser les globules rouges restants sur de la glace pendant 15 min.
- Faites tourner à 300 × g pendant 10 min à RT. Aspirez soigneusement le surnageant. Ajouter 30 mL de DPBS + 2 % de FBS pour laver les cellules. Avec le frein desserré, essorez les cellules lavées à 150 × g pendant 10 min à RT. Aspirez soigneusement le surnageant. Remettre en suspension la pastille cellulaire dans 30 mL de DPBS + 2 % FBS.
- Comptez les cellules mononucléées du sang périphérique (PBMC) maintenant isolées avec du bleu trypan dans un rapport de 1:1.
- En raison de la concentration cellulaire élevée, diluer initialement 10 μL de cellules 1:100 avec DPBS ; Ensuite, mélangez 10 μL des cellules diluées à 1:100 avec 10 μL de solution de bleu de trypan pour cette dilution finale à 1:1. Lorsque vous utilisez un compteur de cellules automatisé, corrigez le nombre de cellules pour refléter la dilution de 200 fois en multipliant la concentration signalée par 100, car la plupart des compteurs supposent une dilution de 2 fois. Attendez-vous à un rendement de 1-2,5 × 109 PBMC à partir d’un leukopak de 1/10.
- Isolement des lymphocytes T CD4+ (sélection négative)
- Faire tourner 10,8-10,9 PBMC à 500 × g pendant 5 min à RT et remettre en suspension dans le tampon de séparation cellulaire (DPBS + 1 mM EDTA + 2 % FBS) à 5 × 107 cellules/mL.
REMARQUE : Pour obtenir suffisamment de Tregs humains pour la modification génétique, nous vous recommandons de commencer avec 1 × 109 PBMC. - Effectuez la séparation magnétique conformément aux instructions du fabricant pour le kit d’enrichissement des lymphocytes T CD4+ .
- Déterminer la quantité de lymphocytes T CD4+ isolés en comptant avec du bleu de trypan (10 μL de cellules + 10 μL de bleu de trypan).
- Faire tourner 10,8-10,9 PBMC à 500 × g pendant 5 min à RT et remettre en suspension dans le tampon de séparation cellulaire (DPBS + 1 mM EDTA + 2 % FBS) à 5 × 107 cellules/mL.
- Isolement des lymphocytes T CD8+ (sélection négative)
- Essorage 5 × 107 PBMC à 500 × g pendant 5 min à RT. Remise en suspension dans le tampon de séparation cellulaire (DPBS + 1 mM EDTA + 2 % FBS) à 5 × 107 cellules/mL.
REMARQUE : Nous vous recommandons de commencer par 5 × 107 PBMC pour obtenir 2 à 5 ×10 6 lymphocytes T CD8+ . - Effectuez la séparation magnétique conformément aux instructions du fabricant pour le kit d’enrichissement des lymphocytes T CD8+ .
- Déterminer le nombre de lymphocytes T CD8+ isolés en comptant avec du bleu de trypan (10 μL de cellules + 10 μL de bleu de trypan).
- Essorage 5 × 107 PBMC à 500 × g pendant 5 min à RT. Remise en suspension dans le tampon de séparation cellulaire (DPBS + 1 mM EDTA + 2 % FBS) à 5 × 107 cellules/mL.
- Tri cellulaire assisté par fluorescence Treg (FACS)
- Isolez les lymphocytes T CD4+ comme décrit à l’étape 1.2 et conservez-les pendant la nuit dans du DPBS avec 2 % de FBS à 4 °C (perte minimale du nombre de cellules et de la viabilité) pour le FACS le lendemain. Déterminer le nombre de lymphocytes T CD4+ à l’étape 1.2 en comptant avec du bleu de trypan (10 μL de cellules + 10 μL de bleu de trypan).
- Faites tourner les cellules CD4+ à 500 × g pendant 5 min. Reconstituer les cellules dans 200 μL de DPBS.
- Pour 1 × 106 cellules, ajouter 1 μL de CD4 FITC anti-humain, 1 μL de CD25 APC anti-humain et 1 μL de CD127 PE anti-humain. Agiter doucement et placer dans le réfrigérateur sombre à 4 °C pendant 30 min.
- Lavez les cellules avec 10 mL de DPBS avec 2 % de FBS. Essorer à 500 × g pendant 5 min. Remettre doucement en suspension les cellules colorées à 1,5 × 107 cellules/mL dans du DPBS avec 2 % de FBS. Il s’agit de la concentration cellulaire recommandée pour trier par tri cellulaire activé par fluorescence (FACS).
- Faites passer la suspension de cellules colorées à travers un capuchon filtrant de 40 μm dans des tubes FACS, puis maintenez les tubes sur de la glace.
- Préparez des tubes de prélèvement de 15 ml contenant 3 ml de milieu RPMI10 et placez-les sur de la glace.
REMARQUE : Ce milieu se compose d’RPMI1640 milieu basal, 10 % FBS, 1x pénicilline-streptomycine, 1x L-alanyl-L-glutamine, 1x acides aminés non essentiels, 1x pyruvate de sodium et 1x HEPES. - Triez les lymphocytes T CD4+CD25à CD127- élevés (Tregs) et les lymphocytes T conventionnels CD4+CD25à faibleCD127+ (Tconv) à l’aide de FACS, comme le montre la figure 1A.
- Déterminer le rendement et la viabilité des cellules ; puis procéder à l’analyse en aval ou à l’activation des lymphocytes T.
2. Activation des lymphocytes T
- Comptez les lymphocytes T isolés avec du bleu de trypan (10 μL de cellules + 10 μL de bleu de trypan).
- Laver 25 μL de billes anti-CD3/CD28 (106 billes) pour 1 × 106 cellules T obtenues, en maintenant un rapport de 1:1 entre les billes et les cellules T. Lavez les billes anti-CD3/CD28 en ajoutant du DPBS, en les incubant sur un aimant pendant 3 minutes et en retirant le tampon à billes anti-CD3/CD28 maintenant dilué, qui peut être toxique pour les cellules.
- Retirez le tube de l’aimant et remettez en suspension les billes lavées dans le milieu RPMI10 pour obtenir 1 × 106 billes/1 ml de RPMI10.
- Remettre en suspension les lymphocytes T avec des billes anti-CD3/CD28 dans le milieu RPMI10 à une concentration de 1 × 106 lymphocytes T/mL. Ensuite, ajoutez 1 000 UI/ml d’IL-2 aux cellules Treg, 100 UI/ml d’IL-2 aux cellules Tconv CD4+ et 300 UI/ml d’IL-2 aux cellules T CD8+ .
- Cultivez des lymphocytes T à 1 × 10à 6 cellules par puits d’une plaque de 24 puits avec différentes quantités de billes anti-CD3/CD28 et d’IL-2. Placez la plaque à 24 puits dans un incubateur de culture tissulaire à 37 °C, 5 % de CO2 .
- Développer les lymphocytes T activés pendant 9 à 12 jours en présence d’IL-2 jusqu’à ce qu’ils soient utilisés pour des expériences ou réactivés avec des billes anti-CD3/CD28 pour une expansion supplémentaire. Remplacez le milieu tous les 2-3 jours et ajoutez de l’IL-2 chaque fois que du milieu frais est ajouté. Maintenez la densité cellulaire des Tregs à 5 × 105 à 1 × 106 cellules par mL pendant l’expansion en comptant les cellules ou par inspection visuelle avant la séparation.
3. Transduction lentivirale Treg humaine
- Remettez en suspension et comptez les Tregs 48 h après l’activation. Essorage à 500 ×g pendant 5 min à RT. Remettre en suspension les Tregs dans RPMI10 à 1,25 × 106 cellules/mL avec 1 000 UI/mL d’IL-2.
- Préchauffez la centrifugeuse à 32 °C.
- Décongeler la construction CAR contenant des lentivirus sur de la glace.
REMARQUE : Nous stockons des aliquotes de lentivirus à usage unique avec suffisamment de lentivirus pour transduire 2,5 × 105 cellules à un moment d’inertie de 1 à -80 °C. - Ajouter chaque aliquote de lentivirus à 2,5 × 105 Tregs dans 200 μL dans un tube de microcentrifugation, chaque tube indiquant une réaction. Spinoculé à 1 000 × g pendant 1 h à 32 °C.
- Déplacer chaque réaction de 200 μL sur une plaque de 24 puits. Assurez-vous que le support couvre tout le puits. Incuber la plaque de 24 puits avec les Tregs transduits dans un incubateur de culture tissulaire pendant la nuit (16-18 h). Compléter chaque puits à 2 mL avec du milieu RPMI10, la concentration finale d’IL-2 étant de 1 000 UI/mL.
- Continuez à développer les Tregs transduits 9 à 12 jours après l’activation en divisant les cellules et en complétant avec du RPMI10 et de l’IL-2 fraîchement préchauffés si nécessaire (tous les 2-3 jours). La concentration idéale de cellules est de 5 × 105-1 × 106 cellules/mL.
- Évaluez l’efficacité de la modification génique à l’aide de la cytométrie en flux, comme le montre la figure 2.
REMARQUE : Nos constructions CAR lentivirales contiennent un marqueur Myc sur l’extrémité N-terminale du gène CAR et un gène rapporteur GFP lié au gène CAR par un peptide 2A, permettant de quantifier l’efficacité de la transduction sans coloration des anticorps (GFP) et de confirmer l’expression de surface du CAR (Myc-tag). Nous évaluons l’efficacité de la modification génique 5 jours après la transduction. L’efficacité de la transduction peut varier entre 30 % et 70 % selon le donateur et la construction CAR utilisée. Les cellules CAR+ peuvent être triées si vous le souhaitez. - Assurez-vous que les Tregs modifiés terminent leur cycle d’activation (9 à 12 jours) et se reposent 24 h en l’absence d’IL-2 avant d’être utilisés dans des expériences.
4. Knock-in du gène médié par CRISPR/Cas9 chez les Tregs humains
- Remettre les Tregs en suspension et les transférer dans un tube conique de 15 mL 48 h après l’activation. Incuber la suspension cellulaire dans un aimant pendant 3 min.
- Pendant qu’ils sont dans l’aimant, transférez les cellules dans le milieu à l’aide d’une pipette dans un nouveau tube. Les billes anti-CD3/CD28 resteront fixées à la paroi du tube. Laissez les Tregs déperlés reposer dans RPMI10 pendant 2 h après l’enlèvement du talon pour récupérer de la contrainte immédiate du débourdonnage, améliorant ainsi l’efficacité future de la transduction et la récupération de l’électroporation.
- Comptez les Tregs perlés avec du bleu trypan.
- Milieu sérique réduit préchauffé sans FBS à 37 °C.
- Préparez une plaque à 6 puits avec 2,5 ml de milieu RPMI10 sans antibiotiques (pénicilline-streptomycine) et avec 1 000 UI/ml d’IL-2 par puits. Préchauffez la plaque à 37 °C dans un incubateur de culture tissulaire.
- Décongelez le modèle de réparation dirigée par homologie CAR (HDR) contenant un virus adéno-associé (AAV) sur de la glace.
REMARQUE : Nous stockons des aliquotes à usage unique avec suffisamment d’AAV pour infecter 4 × 106 cellules à un moment d’inertie de 20 000 à -80 °C. - Faites tourner les Tregs à 500 × g pendant 5 min. Après avoir transvasé le surnageant, remettre les cellules en suspension dans un milieu sérique réduit préchauffé à 4 × 107 cellules/mL.
- Cellules aliquotes dans 100 μL dans des tubes à centrifuger de 1,5 mL à faible liaison protéique. Ajouter l’AAV CAR à un moment d’inertie de 20 000 à chaque échantillon et remettre en suspension. Incuber les tubes de réaction dans l’incubateur de culture tissulaire pendant 1 h.
- Au cours de l’incubation de 1 h, préparez et assemblez les complexes de ribonucléoprotéine (RNP) CRISPR/Cas9 en ajoutant lentement 8,3 μL de protéine Cas9 (stock de 1 mg/mL) à 2,5 μL d’ARNsg ciblant le locus du gène TRAC (stock de 100 μM) pour un rapport molaire de Cas9 à l’ARNg de 1:1 et un volume total de RNP de 10,8 μL par échantillon. Pipeter doucement de haut en bas pour mélanger. Incuber le mélange RNP pendant 15 min à 37°C dans l’incubateur de culture tissulaire.
REMARQUE : Les complexes RNP peuvent rester à RT par la suite jusqu’à ce qu’ils soient prêts à l’emploi. - Remplissez un tube d’électroporation frais (tube E) avec 3 mL de tampon d’électroporation à haute osmolarité. Insérez le tube E rempli dans la station de pipette du système d’électroporation jusqu’à ce qu’un clic se fasse entendre. Réglez les conditions d’électroporation à 2 200 V, 20 ms, 1 impulsion dans le système d’électroporation.
- Lorsque l’incubation de 1 h avec AAV est terminée, faites tourner les cellules avec AAV à 300 × g pendant 5 min à RT. Aspirez soigneusement le surnageant et remettez en suspension la pastille cellulaire dans 100 μL du tampon de remise en suspension cellulaire fourni par le système d’électroporation par échantillon.
REMARQUE : Travaillez rapidement et évitez de laisser les cellules dans la mémoire tampon pendant plus de 15 minutes. Évitez de créer des bulles. - Ajouter 10,8 μL de complexe RNP par échantillon. Bien mélanger à l’aide d’une pipette sans créer de bulles.
- Insérez une pointe d’électroporation de 100 μL en poussant la pipette jusqu’à sa deuxième butée pour ouvrir la pince. Positionnez la tête supérieure de la pipette dans la pointe d’électroporation jusqu’à ce que la pince s’engage solidement avec la tige de montage du piston. Relâchez progressivement le bouton tout en maintenant une pression vers le bas sur la pipette pour vous assurer que la pointe s’adapte parfaitement sans aucun espace.
- Appuyez sur la pipette jusqu’à la première butée et plongez l’embout d’électroporation dans le mélange cellule-RNP. Tirez doucement l’échantillon dans la pipette sans aucune bulle.
REMARQUE : Aucune bulle ne doit être présente à l’intérieur de la pointe pendant l’électroporation. - Insérez la pipette avec l’embout d’électroporation monté contenant l’échantillon verticalement dans le tube E jusqu’à ce qu’un clic se fasse entendre. Évitez de créer des bulles.
- Vérifiez que les paramètres optimaux pour les Tregs humains ont été saisis (2 200 V, 20 ms, 1 impulsion) et appuyez sur start sur l’écran tactile pour électropoler les cellules.
- Attendez que l’écran tactile affiche Terminé à la fin de l’électroporation. Retirer délicatement la pipette et transférer immédiatement l’échantillon dans la plaque à 6 puits préparée contenant 2,5 ml de milieu RPMI10 préchauffé et sans antibiotique avec 1 000 UI/mL d’IL-2 par puits. Répétez l’opération avec les échantillons restants. Balancez doucement la plaque en mouvements linéaires (de gauche à droite, de haut en bas) pour assurer une répartition uniforme des cellules dans chaque puits et placez-la dans l’incubateur de culture tissulaire.
REMARQUE : La réutilisation d’un embout d’électroporation est acceptable jusqu’à 3 fois si les mêmes cellules et le même complexe RNP sont utilisés. Prévoyez une période de récupération de 30 minutes avant de compter les cellules ou de les perturber de quelque manière que ce soit, par exemple en ajoutant un amplificateur HDR si vous le souhaitez. - Le lendemain, 16 à 18 h plus tard, remplacer le milieu par un milieu contenant des antibiotiques, compter les Tregs électroporés avec du bleu de trypan et cultiver à 106 cellules/ml avec 1 000 UI/ml d’IL-2. Continuez à développer les Tregs électroporés en divisant les cellules et en complétant les RPMI10 et IL-2 frais, comme décrit à l’étape 2.6.
- Conservez les échantillons sur de la glace pour les analyser par cytométrie en flux.
- Configurez les commandes de compensation d’une seule couleur et appliquez-les au fichier de test.
- Lisez d’abord l’échantillon non coloré pour ajuster les gains de FSC et de SSC de sorte que la population lymphocytaire se situe au milieu du graphique à points « Tous les événements ».
- Configurez la stratégie de contrôle des lymphocytes en non-débris/cellules uniques en cellules viables en cellules CD4+ , comme illustré à la figure 3C.
- Évaluez l’efficacité de la modification génique à l’aide de la cytométrie en flux.
REMARQUE : Nos constructions CAR AAV contiennent, en plus des bras d’homologie locus TRAC, un gène rapporteur du récepteur du facteur de croissance épidermique tronqué (EGFRt) lié au gène CAR par un peptide 2A, comme le montre la figure 3. L’efficacité de l’inhibition du gène est déterminée par la coloration de surface du CD3, dont la perte indique une perte d’expression de surface du TCR et, par conséquent, un ciblage réussi du locus TRAC avec CRISPR/Cas9 et EGFRt, dont l’expression indique une intégration réussie du transgène CAR. Les Tregs knock-in CAR sont des cellules CD3-EGFRt+. Nous évaluons l’efficacité de la modification génique 5 jours après l’électroporation. - Assurez-vous que les Tregs modifiés terminent leur cycle d’activation (9 à 12 jours) et se reposent 24 h en l’absence d’IL-2 avant d’être utilisés dans des expériences.
5. Activation humaine du CAR Treg
- Mise en place de la co-culture (Jour 0)
- Collectez la lignée cellulaire cible exprimant l’antigène CAR dans un tube conique.
REMARQUE : Nous utilisons des cellules K562, une lignée cellulaire de leucémie myéloïde humaine qui n’exprime pas HLA, CD80 et CD86 comme cellules cibles pour l’activation des CAR Treg. Les cellules parentales K562 sont utilisées comme contrôle négatif et les cellules K562 exprimant l’antigène CAR sont utilisées pour activer les CAR Tregs 17,21. - Irradiez des lignées cellulaires cibles avec 4 000 rad dans un irradiateur au césium 137 ou à rayons X. En cas de manque d’accès à un irradiateur, effectuer un traitement à la mitomycine C pour stopper la prolifération cellulaire tout en maintenant l’expression de l’antigène de surface dans les cellules cibles22.
- Si ce n’est pas déjà fait, dégrainez les Tregs en les remettant en suspension liés aux billes anti-CD3/CD28 et en les transférant dans un tube conique de 15 ml. Incuber la suspension cellulaire dans un aimant pendant 3 à 5 min. Pendant qu’ils sont encore dans l’aimant, transférez les cellules dans le milieu à l’aide d’une pipette dans un nouveau tube ; les billes anti-CD3/CD28 resteront fixées à la paroi du tube.
- Déterminer les concentrations de cellules cibles irradiées et de CAR Treg perlés avec du bleu trypan.
- Essorez les cellules cibles irradiées et les CAR Tregs à 500 × g pendant 5 min. Remettre en suspension avec du RPMI10 préchauffé à 106 cellules/mL dans leurs tubes séparés. Ajouter de l’IL-2 aux CAR-Tregs pour obtenir une concentration de 2 000 UI/mL.
REMARQUE : Cette quantité d’IL-2 sera diluée 2 fois une fois que les CAR Tregs seront combinés avec les cellules cibles pour une concentration finale d’IL-2 de 1 000 UI/mL. - Co-culture de 1 × 10 5 cellules cibles irradiées à antigène CAR (100 μL) avec 1 × 105 (100 μL) de cellules cibles irradiées négatives pour l’antigène CAR (contrôle négatif pour l’activation), 1 × 105 (100 μL) de cellules cibles irradiées positives pour l’antigène CAR (expérience), ou 2,5 μL de billes anti-CD3/CD28 (contrôle positif pour l’activation) et 97,5 μL de milieu RPMI10 dans une plaque inférieure ronde à 96 puits. Placez la plaque dans un incubateur de culture tissulaire à 37 °C, 5 % de CO2 pendant 48 h.
REMARQUE : Le volume final par puits est de 200 μL. Assurez-vous que chaque condition comporte trois répétitions.
- Collectez la lignée cellulaire cible exprimant l’antigène CAR dans un tube conique.
- Lecture de la cytométrie en flux
- À moins d’utiliser un lecteur de plaques, remettre en suspension le contenu de chaque puits à partir de la plaque à fond rond de 96 puits et le transférer dans un tube FACS. Essorer à 500 x g pendant 5 min. Décantez le surnageant et vortex doucement la pastille cellulaire.
- Préparez le mélange maître d’anticorps de cytométrie en flux : 100 μL/échantillon contenant du DPBS, du CD4 PE/Cy7 1:200 anti-humain, du CD71 PE 1:100 et du colorant de viabilité fantôme BV510 1:2000.
REMARQUE : Ce panneau fonctionne bien si le gène rapporteur CAR est GFP. Si un gène rapporteur code pour une protéine de surface qui nécessite une coloration par anticorps, comme l’EGFRt, un anticorps conjugué à la FITC peut être utilisé, par exemple. - Pipeter 100 μL du mélange maître d’anticorps dans chaque échantillon, mélanger doucement et incuber pendant 30 min dans le réfrigérateur à 4 °C.
- Laver avec 500 μL de DPBS en essorant à 500 × g pendant 5 min. Remettre en suspension la pastille cellulaire dans 200 μL de DPBS.
- Conservez les échantillons sur de la glace pour les analyser par cytométrie en flux.
- Configurez des commandes de compensation monochromes et appliquez-les au fichier de flux d’expérience.
- Lire des échantillons non colorés pour ajuster les gains de SSC et de FSC de sorte que la population lymphocytaire se situe au milieu du graphique à points « Tous événements ».
- Mettre en place une stratégie de contrôle des lymphocytes en non-débris/cellules uniques en cellules viables en cellules CD4+ , comme illustré à la figure 3C (Différents fluorophores utilisés).
- Lisez des échantillons à un taux d’événements d’environ 1 500 événements/s pour évaluer l’activation des Tregs.
REMARQUE : Le résultat attendu est la régulation positive de l’expression de surface de CD71 dans les CAR Tregs en présence de l’antigène CAR, comme le montre la figure 4. Si la régulation positive n’est pas observée en présence de l’antigène CAR, alors une signalisation tonique pourrait être présente.
6. Stabilité humaine des Treg CAR
- Mise en place et expansion de la co-culture (jours 0-9)
- Mettre en place des co-cultures pour activer les Tregs CAR comme décrit à l’étape 5.1.
- Après 48 h, transférer la coculture de chaque puits à fond rond de 96 puits dans un puits à plaque de 24 puits contenant 2 mL de RPMI10 préchauffé avec 1 000 UI/mL d’IL-2 pour permettre l’expansion cellulaire.
- Ajouter du RPMI10 fraîchement préchauffé avec 1 000 UI/mL d’IL-2 et diviser en plaques supplémentaires à 24 puits au besoin.
- Lecture de la cytométrie en flux
- Remettre en suspension et transférer le contenu de chaque réplication dans un tube conique de 15 ml ou 50 ml. Déterminez les concentrations cellulaires.
- Transférez entre 5 × 105 et 1 × 106 cellules dans un tube FACS pour chaque répétition. Essorage à 500 × g pendant 5 min.
- Préparez le mélange maître d’anticorps de cytométrie en flux pour la coloration de surface : 100 μL/échantillon contenant du DPBS, du CD4 PE/Cy7 1:200 anti-humain, du CD25 APC 1:200 et du colorant de viabilité fantôme BV510 1:2000. Incluez l’anticorps conjugué à la FITC pour la protéine rapporteure CAR si nécessaire.
- Décantez le surnageant à partir de l’étape 6.2.2 et agitez doucement la pastille cellulaire. Pipeter 100 μL du mélange maître d’anticorps de coloration de surface dans chaque tube FACS. Agiter brièvement et placer au réfrigérateur à 4 °C pendant 30 min dans l’obscurité.
- À l’aide d’un ensemble de tampons de coloration du facteur de transcription, préparer le tampon de fixation/perméabilisation en ajoutant 3 volumes de diluant de fixation/perméabilisation à 1 volume de concentré de fixation/perméabilisation. Chaque échantillon nécessite 100 μL de tampon de fixation/perméabilisation.
- Laver les cellules colorées en surface avec 500 μL de DPBS. Essorer à 500 × g pendant 5 min et décanter le surnageant. Pipette 100 μL de tampon de fixation/perméabilisation préparé dans chaque tube. Agiter brièvement et laisser la fixation se produire à 4 °C pendant 30 à 60 minutes dans l’obscurité.
- À l’aide d’un ensemble de tampons de coloration de facteur de transcription, préparez 1x tampon de perméabilisation en ajoutant 9 volumes d’eau distillée à 1 volume de concentré de tampon de perméabilisation 10x. Chaque échantillon nécessite 1 000 μL de tampon de perméabilisation pour le lavage et 100 μL de tampon de perméabilisation pour la coloration avec des anticorps ciblant les protéines intracellulaires.
- Lavez les cellules fixes/perméabilisées en ajoutant 500 μL de 1x tampon de perméabilisation. Faites tourner à 500 × g pendant 5 min à RT.
- Préparez un mélange maître d’anticorps de coloration intracellulaire avec 1x tampon de perméabilisation, FOXP3 eFluor 450 1:50, HELIOS PE 1:50 et CTLA-4 PerCP-e710 1:50 anti-humain. Chaque échantillon nécessitera 100 μL de mélange maître d’anticorps.
REMARQUE : Ce panel fonctionne si le gène rapporteur CAR est GFP ou si la protéine rapporteure CAR (par exemple, EGFRt) est colorée avec un anticorps conjugué à FITC. - Décanter le surnageant à partir de l’étape 6.2.8. Ajouter 100 μL du mélange maître d’anticorps de coloration intracellulaire, agiter brièvement le vortex et incuber à RT pendant 30 minutes dans l’obscurité.
- Lavez les cellules fixes/perméabilisées colorées en ajoutant 500 μL de 1x tampon de perméabilisation. Essorer à 500 × g pendant 5 min. Décantez le surnageant, remettez en suspension la pastille cellulaire dans 300 μL de DPBS et stockez-la sur de la glace.
- Analysez par cytométrie en flux comme indiqué à la figure 5. Le résultat attendu est que la plupart des Tregs CAR seront des cellules FOXP3+HELIOS+ . Utilisez les cellules CD4+ Tconv comme contrôle négatif pour la coloration FOXP3 et HELIOS.
7. Suppression des Tregs CAR humains
- Coloration à l’état de traces de cellules T répondeur (Tresp) et activation nocturne
- Rassemblez les cellules CAR Treg expansées, les cellules Tconv CD4+ fraîchement isolées et les cellules T CD8+ fraîchement isolées dans des tubes coniques séparés de 15 ml et débilles anti-CD3/CD28 si ce n’est pas déjà fait.
- Préparez des cellules cibles irradiées exprimant l’antigène CAR comme décrit à l’étape 5.1.
- Déterminer les concentrations de lymphocytes T non activés et de cellules cibles irradiées.
- Combinez 5 × 106 cellules Tconv CD4+ avec 5 cellules T × 106 CD8+ (rapport 1:1). Ce sont les lymphocytes T répondeurs (Tresp) à inhiber par les CAR Tregs dans le test. Faites tourner les cellules Tresp à 500 × g pendant 5 min à RT. Aspirez soigneusement le surnageant et remettez en suspension les cellules Tresp dans 1 mL de DPBS.
- Ajouter 1 mL de colorant CellTrace Violet (CTV) 5 mM reconstitué dans du DMSO aux 10 cellules7 Tresp dans 1 mL de DPBS pour une concentration finale de 5 μM CTV. Mettre au bain-marie à 37 °C pendant 20 min. À 10 min, agiter doucement pour redistribuer la CTV stabilisée.
- Laver avec 9 ml de milieu complet RPMI10 préchauffé. Essorer à 500 × g pendant 5 min. Remettre en suspension dans 5 mL de milieu complet RPMI10 préchauffé.
- Déterminez la concentration des cellules Tresp marquées CTV. Ajouter 5 × 104 cellules Tresp non activées marquées CTV dans 200 μL de milieu RPMI10 dans 3 à 6 puits d’une plaque à fond rond de 96 puits comme contrôles minimaux de la prolifération.
- Activez les cellules Tresp marquées CTV avec des billes anti-CD3/CD28 à un rapport de 1:10 entre les billes et les cellules Tresp sans IL-2 dans un milieu RPM10. Distribuer pendant la nuit 106 cellules Tresp marquées au CTV dans 1 mL de milieu RPMI10 par puits d’une plaque à 24 puits dans l’incubateur de culture tissulaire.
- En parallèle, obtenez 106 CAR+ Tregs. Ce montant couvre les triplés de quatre rapports CAR Treg :Tresp, 1:1, 1:2, 1:4, 1:8, à 5 × 104 cellules cibles par puits de plaque à fond rond de 96 puits, comme 5 × 104 × 3 + 2,5 × 104 × 3 + 1,25 × 104 × 3 + 0,625 × 104 × 3 = 2,81 × 105 CAR Tregs, dans deux conditions d’activation des CAR Treg : cellules cibles irradiées à antigène CAR négatif (contrôle négatif, pas d’activation) et des cellules cibles positives à l’antigène CAR irradiées (expérience, activation CAR).
- Combinez 3 × 105 CAR Tregs avec 3 cellules × 105 irradiées à antigène CAR négatif (rapport 1:1) et 3 × 105 CAR Tregs avec 3 × 105 cellules cibles irradiées à antigène CAR positif (rapport 1:1), dans deux tubes coniques séparés de 15 mL et tournez à 500 × g pendant 5 min à RT. Aspirez soigneusement le surnageant.
- Remettre la pastille en suspension dans chaque tube à partir de l’étape 7.1.10 dans 600 μL de milieu RPMI10 préchauffé. Cela donne 200 μL pour chacun des Treg 1:1 : Tresp triplée. Effectuez une dilution en série sur une plaque à fond rond de 96 puits comme suit :
- À partir des 600 μL de suspension de cellules Treg + cible, pipetez 200 μL de suspension cellulaire dans chacun des 3 puits de rapport 1:1.
- Ajoutez 100 μL de milieu complet RPMI10 préchauffé dans chacun des puits vides à rapport 1:2, 1:4 et 1:8.
- Pour chacun des triples, pipeter 100 μL de suspension cellulaire des puits 1:1 dans les puits de rapport 1:2 associés.
- Pour chacun des triples, pipeter 100 μL de la suspension cellulaire, à partir des puits 1:2, dans les puits de rapport 1:4 associés.
- Pour chacun des triples, pipeter 100 μL de suspension cellulaire, à partir des puits 1:4, dans les puits de rapport 1:8 associés.
- Pipetez les 100 μL restants de la suspension cellulaire à partir des puits de rapport 1:8 dans un conteneur à déchets.
REMARQUE : Chaque puits doit contenir 100 μL de suspension cellulaire avec 5 × 104 (1:1), 2,5 × 104 (1:2), 1,25 × 104 (1:4) ou 0,625 × 104 (1:8) CAR Tregs et un nombre égal de cellules cibles irradiées.
- Co-incubation de cellules CAR Treg et Tresp
- Après 16 à 18 heures après l’activation, collectez les cellules Tresp activées de la plaque à 24 puits dans un tube conique et retirez les billes anti-CD3/CD28 à l’aide d’un aimant.
- Déterminez le nombre de cellules Tresp marquées CTV débilées .
- Laver et essorer 2 × 106 cellules Tresp à 500 × g pendant 5 min. Aspirez soigneusement le surnageant. Remettre les cellules en suspension dans 4 mL de milieu complet RPMI10.
- Ajouter 100 μL de suspension de lymphocytes T (5 × 10 4 lymphocytes T) à chaque puits avec des Tregs CAR, ainsi qu’à 3-6 puits avec 100 μL de milieu RPMI10 seul pour des contrôles maximaux de la prolifération. Placez la plaque dans un incubateur de culture tissulaire à 37 °C, 5 % de CO2 pendant 72 h.
REMARQUE : La plaque à fond rond de 96 puits comporte maintenant 3 à 6 puits avec des cellules Tresp non activées seules (contrôle de la prolifération minimale), 3 à 6 puits avec des cellules Tresp activées seules (contrôle de la prolifération maximale) et des cellules Tresp activées en présence d’un nombre décroissant de Treg CAR.
- Lecture de la cytométrie en flux
- Remettez en suspension et transférez le contenu de chaque puits à fond rond de 96 puits dans un tube FACS étiqueté. Vous pouvez également transférer sur une plaque à fond en V de 96 puits si un cytomètre en flux avec mode de lecture de plaque est disponible. Essorer à 500 × g pendant 5 min.
- Entre-temps, préparez un mélange maître d’anticorps avec du DPBS, du CD4 PE/Cy7 1:200 anti-humain et du CD8 PerCP 1:200 humain. Chaque échantillon nécessitera 100 μL de mélange maître d’anticorps.
- Agitez doucement la pastille cellulaire. Pipeter 100 μL de mélange maître d’anticorps dans chaque tube FACS. Agiter brièvement et incuber à 4 °C pendant 30 min dans l’obscurité.
- Laver avec 500 μL de DPBS. Lavez 2 fois avec 100 μL de DPBS si vous utilisez un fond en V de 96 puits. Essorer à 500 × g pendant 5 min. Décantez le surnageant, remettez en suspension la pastille cellulaire dans 200 μL de DPBS et stockez les tubes sur de la glace dans l’obscurité.
- Analysez par cytométrie en flux comme indiqué à la figure 6. Le résultat attendu est que les cellules Tresp non activées seules (prolifération minimale) présentent un pic de fluorescence CTV élevé uniforme, que les cellules Tresp activées seules présentent plusieurs pics d’intensité CTV, un correspondant à chaque division cellulaire (prolifération maximale), et que les lymphocytes T CD4+ et CD8+ activés en présence de CAR Tregs activés présentent une réduction du nombre et de la hauteur des pics CTV, d’où leur prolifération.
- Calculez la suppression à médiation cellulaire Treg comme suit :
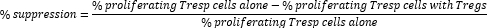
REMARQUE : Si vous utilisez le logiciel FlowJo, il est également possible d’utiliser la modélisation de la prolifération cellulaire pour calculer l’indice de division (ID) pour chaque échantillon et de calculer le pourcentage de suppression à l’aide de l’ID au lieu du pourcentage de cellules proliférantes dans la formule ci-dessus.
Résultats
Le protocole décrit ici fournit un pipeline rationalisé et standardisé pour évaluer les nouvelles constructions de récepteurs antigéniques chimériques (CAR) dans les cellules T régulatrices humaines (Tregs), dans le but de créer des thérapies vivantes pour les maladies auto-immunes, la maladie du greffon contre l’hôte, le rejet de greffe d’organe et l’allergie. La figure 1 illustre comment nous obtenons des Tregs humains hautement purs à pa...
Discussion
Ce protocole fournit une méthodologie rationalisée et complète pour la génération et l’évaluation des cellules T régulatrices du récepteur de l’antigène chimérique humain (CAR Tregs). Le succès de la technologie CAR dans le traitement des cancers hématologiques a inspiré son application au sous-ensemble immunosuppresseur des lymphocytes T, les Tregs. Contrairement aux lymphocytes T conventionnels, les Tregs inhibent les réponses immunitaires, offrant des traitements pot...
Déclarations de divulgation
LMRF est un inventeur et a reçu des redevances sur des brevets sur des cellules immunitaires modifiées et est consultant pour Guidepoint Global et McKesson. Les autres auteurs ne déclarent aucun intérêt concurrent.
Remerciements
LMRF est financé par la subvention U24DK104162-07 du Human Islet Research Network (HIRN) pour le leader émergent du diabète de type 1, la subvention de recherche institutionnelle IRG-19-137-20 de l’American Cancer Society (ACS), la subvention de découverte du projet pilote SCTR (South Carolina Clinical and Translational Research) 1TL1TR001451-01, la subvention IPF 22-1224 de la Diabetes Research Connection (DRC) et la subvention 23-1579 pour Swim Across America. Le RWC est soutenu par la bourse de formation en sciences cellulaires, biochimiques et moléculaires T32GM132055 et la bourse d’études supérieures Lowvelo du Hollings Cancer Center. Cette étude a été financée en partie par la ressource partagée Cytométrie en flux et tri cellulaire, Hollings Cancer Center, Université médicale de Caroline du Sud (P30 CA138313). Un merci spécial au Dr Qizhi Tang de l’Université de Californie à San Francisco (UCSF) pour avoir aimablement offert les plasmides mutants CAR.
matériels
| Name | Company | Catalog Number | Comments |
| Adeno-associated virus (AAV) | Charles River Laboratories | ||
| CAR target-expressing K562 cells | e.g., CD19-K562 | ||
| Cesium-137 irradiator | |||
| Anti-human CD8 PerCP (clone SK1) | Biolegend | 344708 | |
| Anti-human CD4 PE/Cy7 (clone SK3) | Biolegend | 344612 | |
| DynaMag-15 magnet | ThermoFisher | 12301D | |
| Ghost BV510 viability dye | TONBO | 13-0870-T100 | |
| K562 cells | American Type Culture Collection | CCL-243 | |
| 0.5 M EDTA, pH 8.0 | Gibco | 15575020 | |
| 1 M HEPES | Gibco | 15630080 | |
| Ammonium chloride solution | STEMCELL Technologies | 7850 | |
| Anti-human CD127 PE (clone hIL-7R-M21) | BD Biosciences | 557938 | |
| Anti-human CD25 APC (clone BC96) | Biolegend | 302610 | |
| Anti-human CD4 FITC (clone SK3) | Biolegend | 344604 | |
| Anti-human CD71 PE (clone SK1) | Biolegend | 334106 | |
| Anti-human CD8 PerCP (clone SK1) | Biolegend | 344707 | |
| Anti-human CTLA-4 PerCP-e710 | ThermoFisher | 46-1529-42 | |
| Anti-human EGFR APC (clone AY13) | Biolegend | 352905 | |
| Anti-human FOXP3 eFluor 450 | ThermoFisher | 48-4776-42 | |
| Anti-human HELIOS PE | Biolegend | 137216 | |
| Ca2+ and Mg2+ free Dulbecco’s Phosphate Buffered Saline (DPBS) | Gibco | 14190144 | |
| Cell counter (TC20 Automated Cell Counter) | Bio-Rad | 1450102 | |
| Cell Counting Slides | Bio-Rad | 1450016 | |
| CellTrace Violet Cell Proliferation Kit | ThermoFisher | C34571 | |
| DNA LoBind Tubes | Eppendorf | 22431021 | |
| Easy 50 EasySep magnet | STEMCELL Technologies | 18002 | |
| EasySep Human CD4+ T cell Enrichment Kit | STEMCELL Technologies | 19052 | |
| EasySep Human CD8+ T cell Enrichment Kit | STEMCELL Technologies | 19053 | |
| EasySep magnet | STEMCELL Technologies | 18000 | |
| eBioscience Foxp3 transcription factor staining buffer set | ThermoFisher | 00-5523-00 | |
| Falcon Round-Bottom Polystyrene Test Tubes with Cell Strainer Snap Cap, 5 mL | Fisher Scientific | 08-771-23 | 40μm |
| Fetal Bovine Serum (FBS) | Gibco | 26140079 | |
| Flow cytometer | Beckman Coulter | CytoFLEX LX U3-V5-B3-Y5-R3-I2 | |
| Fluorescence-activated cell sorter | BD Biosciences | FACS Aria III Cell Sorter | |
| GlutaMAX | Gibco | 35050061 | |
| Human CD3/28 T Cell Expansion and Activation Dynabeads | Gibco | 11131D | |
| Invitrogen Neon Transfection System | ThermoFisher | 10431915 | |
| Invitrogen Neon Transfection System 100 μL Kit | ThermoFisher | 10114334 | |
| Lentivirus | VectorBuilder | ||
| MEM Non-Essential Amino Acids Solution | Gibco | 11140050 | |
| Myc Tag antibody A647 (clone 9B11) | Cell Signaling Technologies | 2233S | |
| Opti-MEM I Reduced Serum Medium | ThermoFisher | 31985062 | |
| Penicilin-Streptomycin solution | Gibco | 15140122 | |
| Recombinant human interleukin-2 (rhIL-2) | Peprotech | 200-02 | |
| RPMI 1640 medium, no glutamine | Gibco | 11875093 | |
| Sodium pyruvate | Gibco | 11360070 | |
| Spectral Flow Cytometer | Cytek | Northern Lights | |
| TRAC gRNA | Synthego | Sequence (CAGGGTTCTGGATATCTGT) | |
| TrueCut Cas9 Protein v2 | ThermoFisher | A36496 | |
| Trypan Blue solution | Sigma | T8154-100ML | |
| 1/10 Leukopak | STEMCELL Technologies | 200-0092 | 1-2 billion PBMC |
Références
- Zhang, X., Zhu, L., Zhang, H., Chen, S., Xiao, Y. CAR-T cell therapy in hematological malignancies: current opportunities and challenges. Front Immunol. 13, 927153 (2022).
- Cappell, K. M., Kochenderfer, J. N. Long-term outcomes following CAR T cell therapy: what we know so far. Nat Rev Clin Oncol. 20 (6), 359-371 (2023).
- Choi, B. D., et al. Intraventricular CARv3-TEAM-E T cells in recurrent glioblastoma. N Engl J Med. 390 (14), 1290-1298 (2024).
- Brown, C. E., et al. Locoregional delivery of IL-13Ralpha2-targeting CAR-T cells in recurrent high-grade glioma: a phase 1 trial. Nat Med. 30 (4), 1001-1012 (2024).
- Bagley, S. J., et al. Intrathecal bivalent CAR T cells targeting EGFR and IL13Ralpha2 in recurrent glioblastoma: phase 1 trial interim results. Nat Med. 30 (5), 1320-1329 (2024).
- Ghobadinezhad, F., et al. The emerging role of regulatory cell-based therapy in autoimmune disease. Front Immunol. 13, 1075813 (2022).
- Sakaguchi, S., Yamaguchi, T., Nomura, T., Ono, M. Regulatory T cells and immune tolerance. Cell. 133 (5), 775-787 (2008).
- Sakaguchi, S., et al. Regulatory T cells and human disease. Annu Rev Immunol. 38, 541-566 (2020).
- Ferreira, L. M. R., Muller, Y. D., Bluestone, J. A., Tang, Q. Next-generation regulatory T cell therapy. Nat Rev Drug Discov. 18 (10), 749-769 (2019).
- Rosenblum, M. D., Gratz, I. K., Paw, J. S., Abbas, A. K. Treating human autoimmunity: current practice and future prospects. Sci Transl Med. 4 (125), 125sr121 (2012).
- Sawitzki, B., et al. Regulatory cell therapy in kidney transplantation (The ONE Study): a harmonised design and analysis of seven non-randomised, single-arm, phase 1/2A trials. Lancet. 395 (10237), 1627-1639 (2020).
- Spanier, J. A., et al. Tregs with an MHC class II peptide-specific chimeric antigen receptor prevent autoimmune diabetes in mice. J Clin Invest. 133 (18), e168601 (2023).
- Muller, Y. D., et al. Precision engineering of an anti-HLA-A2 chimeric antigen receptor in regulatory T cells for transplant immune tolerance. Front Immunol. 12, 686439 (2021).
- MacDonald, K. G., et al. Alloantigen-specific regulatory T cells generated with a chimeric antigen receptor. J Clin Invest. 126 (4), 1413-1424 (2016).
- Boardman, D. A., et al. Flagellin-specific human CAR Tregs for immune regulation in IBD. J Autoimmun. 134, 102961 (2023).
- Schreeb, K., et al. Study design: human leukocyte antigen Cclass I molecule A(*)02-chimeric antigen receptor regulatory T cells in renal transplantation. Kidney Int Rep. 7 (6), 1258-1267 (2022).
- Zimmerman, C. M., Robino, R. A., Cochrane, R. W., Dominguez, M. D., Ferreira, L. M. R. Redirecting human conventional and regulatory T cells using chimeric antigen receptors. Methods Mol Biol. 2748, 201-241 (2024).
- Tang, Q., et al. Selective decrease of donor-reactive T(regs) after liver transplantation limits T(reg) therapy for promoting allograft tolerance in humans. Sci Transl Med. 14 (669), eabo2628 (2022).
- Bender, C., et al. A phase 2 randomized trial with autologous polyclonal expanded regulatory T cells in children with new-onset type 1 diabetes. Sci Transl Med. 16 (746), eadn2404 (2024).
- Ferreira, L. M. Conference report: Advanced Therapies Week 2023. Regen Med. 18 (4), 297-299 (2023).
- Cochrane, R. W., et al. How to test human CAR T cells in solid tumors, the next frontier of CAR T cell therapy. Methods Mol Biol. 2748, 243-265 (2024).
- Roy, A., Krzykwa, E., Lemieux, R., Neron, S. Increased efficiency of gamma-irradiated versus mitomycin C-treated feeder cells for the expansion of normal human cells in long-term cultures. J Hematother Stem Cell Res. 10 (6), 873-880 (2001).
- Eyquem, J., et al. Targeting a CAR to the TRAC locus with CRISPR/Cas9 enhances tumour rejection. Nature. 543 (7643), 113-117 (2017).
- Negrini, M., Wang, G., Heuer, A., Bjorklund, T., Davidsson, M. AAV Production everywhere: a simple, fast, and reliable protocol for in-house AAV vector production based on chloroform extraction. Curr Protoc Neurosci. 93 (1), e103 (2020).
- Velasco Cardenas, R. M., et al. Harnessing CD3 diversity to optimize CAR T cells. Nat Immunol. 24 (12), 2135-2149 (2023).
- Boomer, J. S., Green, J. M. An enigmatic tail of CD28 signaling. Cold Spring Harb Perspect Biol. 2 (8), a002436 (2010).
- Suhoski, M. M., et al. Engineering artificial antigen-presenting cells to express a diverse array of co-stimulatory molecules. Mol Ther. 15 (5), 981-988 (2007).
- Fung, V. C. W., Rosado-Sanchez, I., Levings, M. K. Transduction of human T cell subsets with lentivirus. Methods Mol Biol. 2285, 227-254 (2021).
- Bailey-Bucktrout, S. L., et al. Self-antigen-driven activation induces instability of regulatory T cells during an inflammatory autoimmune response. Immunity. 39 (5), 949-962 (2013).
- Nakagawa, H., et al. Instability of Helios-deficient Tregs is associated with conversion to a T-effector phenotype and enhanced antitumor immunity. Proc Natl Acad Sci U S A. 113 (22), 6248-6253 (2016).
- Dawson, N. A. J., et al. Functional effects of chimeric antigen receptor co-receptor signaling domains in human regulatory T cells. Sci Transl Med. 12 (557), eaaz3866 (2020).
- Rana, J., et al. CAR- and TRuC-redirected regulatory T cells differ in capacity to control adaptive immunity to FVIII. Mol Ther. 29 (9), 2660-2676 (2021).
- Battaglia, M., Stabilini, A., Tresoldi, E. Expanding human T regulatory cells with the mTOR-inhibitor rapamycin. Methods Mol Biol. 821, 279-293 (2012).
- Brady, B. L., Steinel, N. C., Bassing, C. H. Antigen receptor allelic exclusion: an update and reappraisal. J Immunol. 185 (7), 3801-3808 (2010).
- Samarasinghe, S., et al. Functional characterization of alloreactive T cells identifies CD25 and CD71 as optimal targets for a clinically applicable allodepletion strategy. Blood. 115 (2), 396-407 (2010).
- Voss, K., et al. FOXP3 protects conventional human T cells from premature restimulation-induced cell death. Cell Mol Immunol. 18 (1), 194-205 (2021).
- Collison, L. W., Vignali, D. A. In vitro Treg suppression assays. Methods Mol Biol. 707, 21-37 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon