Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Detección de alcaloides de cornezuelo de centeno regulados en matrices alimentarias por cromatografía líquida-espectrometría de movilidad iónica atrapada-espectrometría de masas de tiempo de vuelo
En este artículo
Resumen
Este protocolo presenta un método validado de cromatografía líquida-movilidad iónica-espectrometría de masas de alta resolución para determinar la presencia de alcaloides del cornezuelo de centeno en los alimentos en cumplimiento del Reglamento (UE) 2023/915 de la Comisión recientemente publicado.
Resumen
La espectrometría de masas de movilidad iónica (IMS) actúa como una dimensión de separación adicional cuando se integra en los flujos de trabajo de cromatografía líquida-espectrometría de masas (LC-MS). Los métodos LC-IMS-MS proporcionan una resolución máxima más alta, una separación mejorada de compuestos isobáricos e isoméricos y una mejor relación señal-ruido (S/N) en comparación con los métodos LC-MS tradicionales. IMS proporciona otra característica molecular para la identificación de analitos, a saber, el parámetro de sección transversal de colisión (CCS), lo que reduce los resultados falsos positivos. Por lo tanto, los métodos LC-IMS-MS abordan importantes desafíos analíticos en el campo de la seguridad alimentaria (es decir, la detección de compuestos a niveles traza en matrices alimentarias complejas y la identificación inequívoca de moléculas isobáricas e isoméricas).
Los alcaloides del cornezuelo de centeno (EA) son una familia de micotoxinas producidas por hongos que atacan a una amplia variedad de especies de gramíneas, incluidos granos pequeños como el centeno, el triticale, el trigo, la cebada, el mijo y la avena. Se han establecido niveles máximos (MLs) de estas micotoxinas en varios productos alimenticios, tal y como se detalla en el Reglamento CE/2023/915 de la Comisión. Esta nueva legislación incluye seis EA principales y sus correspondientes epímeros, por lo que se requiere una metodología eficiente para distinguir adecuadamente estas moléculas isoméricas considerando su co-ocurrencia.
Por lo tanto, el objetivo de este protocolo es mostrar cómo la integración de IMS en los flujos de trabajo de LC-MS contribuye a la separación de EA isoméricos, mejorando la selectividad del método analítico. Además, ilustra cómo la generación de bibliotecas de CCS a través de la caracterización de estándares analíticos proporciona una mayor confianza para la identificación de micotoxinas. Este protocolo está diseñado para explicar de forma clara los beneficios de la implementación de IMS en la inocuidad de los alimentos, tomando como ejemplo la determinación de EAs en cereales. Una extracción basada en QuEChERS seguida de un análisis de espectrometría de movilidad iónica atrapada en LC (TIMS)-MS proporcionó límites de cuantificación que oscilaron entre 0,65 y 2,6 ng/g con una precisión aceptable (aunque baja recuperación para la ergotaminina) a 1,5x, 1x y 0,5x el ML y exhibió un efecto de matriz insignificante.
Introducción
La espectrometría de masas por movilidad iónica (IMS) se está convirtiendo en una técnica analítica cada vez más utilizada, que a menudo se presenta como una dimensión de separación adicional integrada en la cromatografía líquida/gaseosa tradicional (LC/GC) acoplada a los flujos de trabajo de MS. El IMS consiste en la separación de moléculas a lo largo de una célula de movilidad, llena de un gas tampón, bajo un campo eléctrico y a presión atmosférica1. Dependiendo de la relación masa-carga (m/z) y de la conformación geométrica, una molécula ionizada interactuará con el gas tampón a medida que se mueve a través de la célula de movilidad, lo que se refleja en el parámetro de movilidad iónica (K)2 y se calcula mediante la siguiente ecuación:
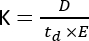
donde D representa la longitud total de la deriva, td es el tiempo total de deriva y E es el campo eléctrico. Por lo tanto, K se mide en m2 V−1 s−1, aunque por razones prácticas a menudo se expresa como cm2 V−1 s−1. La capacidad intrínseca de moverse a través de la célula de movilidad puede medirse por el tiempo de deriva y luego convertirse en el llamado valor de la sección transversal de colisión (CCS), que es un parámetro altamente reproducible para cada molécula independientemente del instrumento IMS3. El CCS se puede derivar de la movilidad siguiendo esta ecuación:
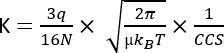
q es la carga del ion; N la densidad del número de gases tampón; μ la masa reducida de los socios de colisión amortiguan el iones de gas; kB la constante de Boltzmann; y T la temperatura del gas tampón. Por lo tanto, IMS proporciona información adicional complementaria a los datos analíticos resultantes de los análisis de cromatografía y MS.
Se ha demostrado que la implementación de IMS en plataformas LC-MS aumenta la fiabilidad de las determinaciones analíticas, especialmente cuando se trabaja con compuestos que se encuentran en concentraciones traza. Varios estudios han reportado que los métodos LC-IMS-MS mejoran la calidad de los espectros de masas al reducir el ruido de fondo, que en última instancia afecta la sensibilidad del método, y reduce la tasa de falsos positivos y negativos proporcionados por las metodologías LC-MS multirresiduo 4,5,6. Además, la reproducibilidad de los valores de CCS permite la comparación no solo entre diferentes instrumentos que utilizan la misma tecnología, sino también entre diferentes tecnologías de movilidad iónica, a saber, espectrometría de movilidad de iones de onda viajera (TWIMS), espectrometría de movilidad de iones atrapados (TIMS) y espectrometría de movilidad de iones de tubo de deriva (DTIMS)2,7, que son los sistemas más utilizados 1. Así, una consecuencia notable del potencial de la CCS como parámetro de identificación radica en la posibilidad de construir bibliotecas de CCS, reflejada en su aplicabilidad en estudios de metabolómica8. No obstante, una de las características más poderosas de IMS es la capacidad de separar compuestos isoméricos e isobáricos que pueden no resolverse suficientemente con métodos LC-MS. Este puede ser el caso cuando se trabaja con grandes conjuntos de analitos de interés en matrices complejas, lo cual es una situación común en el análisis ambiental y de alimentos. En este contexto, se han propuesto métodos LC-IMS-MS para el monitoreo de plaguicidas y, en menor medida, de medicamentos veterinarios y micotoxinas en alimentos9.
Debido a su alto poder de resolución y selectividad, las plataformas LC/GC-IMS-MS emergen como las herramientas más útiles para abordar algunos de los desafíos actuales en seguridad alimentaria, especialmente aquellos relacionados con mezclas isoméricas. La preocupación por la salud relacionada con las mezclas isoméricas como contaminantes alimentarios se ha reflejado en la legislación europea vigente, que, por ejemplo, limita la concentración máxima de seis alcaloides principales del cornezuelo de centeno (EA) y sus correspondientes seis epímeros en varios productos alimenticios10.
Los EA constituyen una familia de metabolitos secundarios tóxicos producidos por una amplia gama de hongos, principalmente de la familia Clavicipitaceae (por ejemplo, Claviceps purpurea, el productor más importante de EA debido a su amplia gama de huéspedes), pero también Trichocomaceae, que pueden parasitar la cabeza de la semilla de plantas vivas (como el centeno, la cebada, el trigo y la avena) en el momento de la floración11, 12. En condiciones específicas, especialmente la temperatura y la actividad del agua, los hongos Claviceps pueden producir EA que se acumulan en los cuerpos fructíferos, conocidos como esclerocios o cornezuelo de centeno, en el cultivo huésped. Hasta cierto punto, los EA pueden soportar el procesamiento de la materia prima hasta llegar al producto final; por lo tanto, irrumpiendo en la cadena alimentaria. La ingestión de alimentos contaminados puede llevar a la intoxicación por EA, conocida como ergotismo, que se presenta con síntomas agudos como dolor abdominal, vómitos, sensación de ardor en la piel, insomnio y alucinaciones13. Para reducir el impacto de los EA en la salud humana, la Comisión Europea estableció niveles máximos (ML) en varios alimentos para la suma de los principales EA: los Epímeros R ergometrina (Em), ergotamina (Et), ergosina (Es), ergocristina (Ecr), ergokriptina (Ekr) y ergocornina (Eco) y sus correspondientes S-epímeros: ergometrinina (Emn), ergosinina (Esn), ergotaminina (Etn), ergocorninina (Econ), ergokriptinina (Ekrn), y ergocristinina (Ecrn). Estos compuestos pueden epimerizar de las formas R a S y viceversa, especialmente bajo la exposición a luz intensa, almacenamiento prolongado o contacto con algunos solventes a pH 12 alto o bajo. Aunque la proporción de formas R y S puede variar en diferentes condiciones, la Comisión Técnica CONTAM de la EFSA informó de una mayor incidencia de formas R que de formas S tras examinar la bibliografía disponible sobre los EA en los productos alimenticios14. Por lo tanto, los ML varían en función de varios factores, como la susceptibilidad del cultivo, el grado de procesamiento o la frecuencia de consumo. En el marco de la UE, los ML para los productos elaborados de cebada, trigo, espelta y avena se han fijado en 50 o 150 μg/kg (en función del contenido de cenizas inferior o superior a 900 mg/100 g, respectivamente), mientras que los cereales destinados directamente al consumo humano están sujetos a un ML de 150 μg/kg, excepto los alimentos infantiles a base de cereales. en el que el ML se reduce a 20 μg/kg10.
Esta legislación estricta requiere metodologías analíticas lo suficientemente sensibles como para determinar los niveles de concentración de trazas (μg/kg) y, al mismo tiempo, identificar adecuadamente los EA regulados y sus epímeros correspondientes, ya que ambas formas, isómeros R y S, se pueden encontrar juntas en muestras contaminadas. Esta tarea representa un gran desafío, ya que cada par toxina-epímero comparte exactamente el mismo patrón de masa y fragmentación. Además, una adecuada separación cromatográfica entre ambos compuestos puede resultar compleja. Por lo tanto, se requieren gradientes de LC bien optimizados para evitar una cuantificación errónea cuando los epímeros de EA coexisten en muestras de alimentos. Aunque varios estudios han reportado métodos de LC-MS para la determinación inequívoca de EA 15,16,17,18, el método cromatográfico debe ser estudiado extensamente para lograr una separación adecuada de los picos cromatográficos para identificar inequívocamente los EAs. Sin embargo, esto no suele ser factible para métodos multiclase en los que se determinan simultáneamente contaminantes pertenecientes a diferentes familias químicas. En este contexto, un estudio reciente realizado por Carbonell-Rozas, Hernández-Mesa, et al.19 reportó un método LC-IMS-MS para la cuantificación de EAs en muestras de trigo y cebada, utilizando dos instrumentos TWIMS diferentes que proporcionaron valores de CCS reproducibles y límites bajos de cuantificación (LOQs) para detectar cualquier incumplimiento de acuerdo con la legislación vigente. Por lo tanto, el objetivo de este protocolo es mostrar cómo la integración de IMS en los flujos de trabajo de LC-MS contribuye a la separación de EA isoméricos, mejorando la selectividad del método analítico. Además, ilustra cómo la generación de bibliotecas de CCS a través de la caracterización de estándares analíticos proporciona una mayor confianza para la identificación de micotoxinas. Este protocolo está diseñado para explicar claramente los beneficios de implementar IMS en el análisis de seguridad alimentaria, tomando como ejemplo la determinación de EAs en cereales. Este protocolo aborda el tratamiento de la muestra basado en un procedimiento QuEChERS, el análisis de la muestra mediante LC-TIMS-MS y la extracción e interpretación de datos IMS.
Protocolo
1. Preparación de soluciones de stock, intermedios y estándar de trabajo
NOTA: Use guantes de nitrilo, bata de laboratorio y gafas de seguridad.
- Prepare soluciones madre individuales de los 12 EA (ver Tabla de Materiales) a 10.000 ng/mL en viales de vidrio ámbar de 4 mL utilizando acetonitrilo. Las formas R (-ine) se alícuota previamente en porciones de 25.000 ng, mientras que las formas S (-inina) se distribuyeron en porciones de 10.000 ng. Este estudio se inició con estas alícuotas, alcanzando concentraciones de 10.000 ng/mL diluyendo las formas R y S en 2,5 y 1 mL de acetonitrilo, respectivamente, seguido de vórtice durante 2 min.
- Prepare una solución madre intermedia a 1.000 ng/mL de EA totales (83,33 ng/mL de cada uno) en un vial de vidrio ámbar de 12 mL añadiendo 65 μL de cada solución alcaloide individual y 7,02 mL de acetonitrilo.
NOTA: Una vez preparada la solución madre intermedia, las soluciones madre individuales deben secarse y almacenarse a -20 °C cubiertas con papel de aluminio para evitar la epimerización. - Prepare soluciones estándar de trabajo a partir de la solución madre intermedia pipeteando 2,7, 1,8 y 0,9 mL en tubos de vidrio ámbar de 4 mL. Llévelos a la sequedad bajo un suave chorro de nitrógeno y vuelva a suspenderlos en 600 μL de acetonitrilo.
NOTA: Se estableció que las concentraciones resultantes alcanzaban 1,5, 1 y 0,5 veces el mL (150 ng/g) al agregar 50 μL a 1 g de la muestra. Estos se seleccionan como niveles de concentración para los estudios de validación (es decir, recuperaciones, repetibilidad, etc.). - Divida los 600 μL de cada solución estándar de trabajo en tres viales de vidrio ámbar de 2 mL pipeteando 200 μL y llévelos a secar.
NOTA: Los pasos 1.2 a 1.4 se explican esquemáticamente en la Figura 1. Al dividir y secar las soluciones estándar de trabajo, es posible fortificar por triplicado en tres días diferentes, según lo requiera el estudio de validación, sin perder la integridad química de los EA. - En cada día de validación, vuelva a suspender un vial de vidrio ámbar de 2 ml de cada nivel de fortificación en 200 μl de acetonitrilo.
NOTA: Cada nivel de fortificación se evaluará por triplicado utilizando 50 μL por muestra; por lo tanto, se utilizará un total de 150 μL de la solución.
2. Preparación de reactivos y soluciones
NOTA: Use guantes de nitrilo, bata de laboratorio y gafas de seguridad.
- Prepare una solución de carbonato de amonio de 5 mM pesando 24,02 mg de carbonato de amonio y disolviéndola en 50 mL de agua. Pesar 24,02 mg de carbonato de amonio en un vaso de precipitados y añadir aproximadamente 5 ml de agua. Disuelva el carbonato de amonio agitando suavemente la solución manualmente, luego transfiérala a un matraz aforado de 50 mL. Para alcanzar el volumen final de 50 ml, enjuague el vaso de precipitados con agua adicional y transfiera el fermento al matraz aforado.
- Prepare 250 mL de la solución de extracción [acetonitrilo:5 mM de carbonato de amonio (85:15, v/v)] mezclando 212,5 mL de acetonitrilo y 37,5 mL de la solución de amonio preparada en el paso 2.1.
- Prepare 100 mL de una mezcla de metanol y agua (1:1, v/v) para resuspender los extractos de muestra (paso 8.8.).
- Para cada muestra, pesar 150 mg de una mezcla de sorbente a base de circonio C18:material dispersivo en fase sólida (1:1, p/p) en tubos de centrífuga de 15 ml para la etapa de limpieza dispersiva (paso 8.5.).
- Prepare los disolventes para la separación cromatográfica en dos frascos diferentes, a saber, 1 L de agua ultrapura (disolvente A) y metanol (disolvente B), ambos con un 0,3% (v/v) de ácido fórmico. Para lograr esta concentración, se añadieron 3,03 mL de ácido fórmico (99% de pureza) a los frascos.
NOTA: Los disolventes para la fase móvil deben prepararse justo antes de ejecutar los análisis LC-IMS-MS.
3. Ajuste de los parámetros instrumentales
NOTA: El instrumento utilizado para realizar este estudio LC-IMS-MS fue un UHPLC acoplado a un IM-HRMS, equipado con una fuente de ionización por electrospray calentada con sonda aislada al vacío (VIP-HESI). El instrumento funcionó en modo positivo.
- Cree un método cromatográfico utilizando el software de adquisición para la separación de los analitos ajustando el siguiente gradiente de elución: 0 min, 10% B; 2 min, 10% B; 4,5 min, 40% B; 9 min, 45% B; 11 min, 95% B; 12 min, 95% B; 13 min, 10% B; 16 min, 10% B.
- Ajuste el caudal de la fase móvil a 0,4 mL/min, la temperatura de la columna a 35 °C y el volumen de inyección a 5 μL. Si es posible, mantenga una temperatura fija de 10 °C dentro del muestreador automático.
- Ajuste los parámetros IMS de la siguiente manera: valores de movilidad (1/K0), de 0,1 a 1,5 V·s/cm2; tiempo de rampa, 100 ms; tiempo de precisión, 10 ms; ciclo de trabajo, 10%; y velocidad de rampa, 9,05 Hz.
- Configure los parámetros de la fuente de iones para la detección de MS de la siguiente manera: voltaje capilar, 2.500 V; presión del nebulizador, 2,5 bar; caudal de gas seco, 8 L/min; temperatura del gas seco, 200 °C; temperatura del gas de la vaina, 450 °C; caudal de gas de la vaina, 4 L/min. Para la adquisición de MS, seleccione los siguientes parámetros, teniendo en cuenta dos eventos de exploración: exploración completa de todo el espectro de masas con un m/z de 20 a 1.300 seguido de bbCID (disociación inducida por colisión de banda ancha), que genera iones producto a partir de cualquier precursor a tres energías de colisión fijas de 24, 36 y 50 eV. Realice la ionización y la adquisición de datos en modo de polaridad positiva.
- Guarde este archivo de método de adquisición para su uso posterior.
4. Adquisición de datos de los estándares analíticos de los EA
NOTA: Utilice guantes de nitrilo, bata de laboratorio y gafas de seguridad solo para el paso 4.1.
- Preparar dos viales ámbar de 1,5 mL con 500 μL de solución intermedia a 1.000 ng/mL y otro vial con una mezcla de disolventes A y B (1:1, v/v) como solución de lavado.
- Calibre el instrumento timsTOF según lo indicado por el fabricante y seleccione la polaridad positiva para las adquisiciones de IMS y MS.
- Coloque la columna C18 (ver Tabla de Materiales) dentro del horno LC, con la temperatura ajustada a 35 °C como se indica en el archivo del método de adquisición guardado en el paso 3.5.
NOTA: Asegúrese de que ambos extremos estén bien apretados; de lo contrario, se observará una fuga de fase móvil. - Acondicione la columna LC para permitir un caudal de fase móvil de 0,1 mL/min y seleccione una relación de mezcla de disolvente A y B de 90:10 (%) como relación de composición inicial de la fase móvil.
- Espere hasta que la presión de la columna sea estable (~130 bar).
- Aumente el caudal de la fase móvil a 0,2 mL/min, espere hasta que la presión se estabilice y vuelva a aumentarlo a 0,3 mL/min.
- Una vez que la presión se estabilice nuevamente, ajuste el caudal a 0,4 mL, que será el caudal de trabajo como se detalla en el archivo del método de adquisición guardado en el paso 3.5, y espere hasta que la presión se estabilice alrededor de 350 bar.
NOTA: Es importante no aumentar directamente el caudal de 0,2 a 0,4 mL/min. Un cambio fuerte en el flujo provoca picos de alta presión que pueden dañar la columna de cromatografía. - Mientras se acondiciona la columna, escriba la lista de trabajo, incluyendo dos muestras correspondientes a los dos viales que contienen la solución intermedia de EA a 1.000 ng/mL, como se indica en el paso 4.1, y el vial de lavado. Analice el vial de lavado dos veces, al principio y al final del lote de muestras.
- En la columna Archivo de método del pool de trabajo, cargue el archivo de método de adquisición previamente guardado en el paso 3.5.
- Cree una carpeta para guardar todos los archivos de datos adquiridos y enrutar todas las muestras seleccionando la carpeta en la columna Ruta de la muestra .
NOTA: Asegúrese de utilizar la misma carpeta para guardar todos los archivos generados durante el estudio de validación para su posterior procesamiento. Durante el acondicionamiento de la columna, los valores en los que la presión se estabiliza pueden variar ligeramente. No obstante, asegúrese de que la presión sea estable antes de volver a aumentar el caudal para preservar la integridad de la columna. Además, cuando se alcanza el caudal de trabajo, la presión también debe ser estable para lograr una separación cromatográfica reproducible. - Vaya a la barra de herramientas superior del software y ejecute la lista de trabajo.
5. Tratamiento de datos para la creación de un método de cuantificación
- Una vez ejecutado el pool de trabajo, abra el software cualitativo.
- Cargue todas las muestras analizadas haciendo clic en Archivo | Abrir pool de trabajo.
- Haga clic con el botón derecho en una muestra correspondiente a la solución intermedia de EA a 1.000 ng/mL, vaya a Editar cromatograma y, a continuación, seleccione Tipo | Cromatograma iónico extraído, e introduzca la masa molecular teórica del ion protonado relacionado con cada EA. A continuación, haga clic en Agregar y, por último, en Aceptar. Dado que el conjunto de analitos está formado por seis EA y sus epímeros relacionados que comparten la misma masa exacta, aparecerán seis cromatogramas iónicos extraídos.
NOTA: El parámetro Anchura cambia la estrechez de la ventana m/z . Se recomienda seleccionar una ventana m/z suficientemente amplia en caso de dificultades para encontrar el pico cromatográfico del analito de interés. Sin embargo, es aconsejable establecer ventanas m/z estrechas para evitar posibles interferencias (por debajo de 0,05 unidades). - En cada cromatograma iónico extraído aparecerán dos picos cromatográficos relacionados con el EA principal y su epímero. Haga clic con el botón derecho mientras selecciona toda el área de un pico. En consecuencia, el espectro de movilidad iónica aparecerá justo debajo.
- Vaya al eje x del espectro de movilidad iónica, haga clic con el botón derecho y luego haga clic en Sección transversal de colisión. Anote el valor de CCS.
NOTA: Si los valores de CCS no aparecen automáticamente, vaya a Calibrar en la barra de herramientas superior, luego a Calculadora de movilidad y escriba manualmente el valor de movilidad para obtener su CCS correspondiente. - Seleccione el área correspondiente al otro pico cromatográfico dentro del mismo cromatograma iónico y repita los pasos 5.4. y 5.5.
- Del mismo modo, el patrón de fragmentación también aparecerá después del paso 5.4. Con base en el patrón de fragmentación reportado en la literatura y las bases de datos, compare y elija el ion producto más intenso como punto de identificación complementario.
- Anote el tiempo de retención, el valor CCS y la masa exacta del aducto de iones principal (generalmente el ion protonillado) de cada EA para crear un método de cuantificación.
- Repita los pasos 5.4 a 5.8. para los analitos restantes.
- Seleccione todas las masas moleculares generadas en el paso 5.3 haciendo clic en ellas mientras presiona Ctrl. A continuación, haga clic con el botón derecho y seleccione Copiar. Vaya a la otra muestra correspondiente a la solución intermedia del EA, haga clic derecho sobre ella y seleccione Pegar. Los cromatogramas iónicos y los espectros de movilidad se extraerán automáticamente.
- Vuelva a comprobar los parámetros de identificación de todos los analitos de esta segunda muestra.
6. Creación de un método de procesamiento de datos para la determinación rutinaria de alcaloides del cornezuelo de centeno
- Abra el software y haga clic en la pestaña Gestión de métodos .
- Haga clic en Configuración de analito e introduzca el nombre de cada EA junto con su tiempo de retención, el valor CCS y la masa de los aductos protonados recopilados en los pasos 5.4. al 5.9. Cambie la tolerancia de cada punto de identificación según sea necesario, aunque se recomiendan los ajustes predeterminados para la aplicación de este protocolo.
NOTA: Cuanto más altos sean los valores de tolerancia establecidos, mayor será la posibilidad de tratar con falsos positivos, ya que se acepta una mayor desviación de los valores teóricos. Del mismo modo, los valores de tolerancia demasiado estrictos pueden dar lugar a falsos negativos. Esto sucede cuando la desviación del valor teórico es causada únicamente por el rendimiento del instrumento. - Guarde el método de cuantificación para su uso posterior.
7. Muestreo
- Adquirir muestras de trigo y avena en supermercados, preferiblemente en envases de 1 kg, siguiendo las directrices para el muestreo establecidas por la Comisión Europea20. Si no dispone de paquetes de 1 kg, asegúrese de tener, al menos, 50 g de muestras para completar todo el análisis.
- Muele las muestras con un molinillo. En este estudio, se utilizó una trituradora de cuchillas para moler todo el contenido de los paquetes, que luego se devolvieron a sus paquetes originales para su homogeneización.
NOTA: Limpie a fondo el molinillo entre muestras para evitar la contaminación cruzada. Usa una bata de laboratorio. - Alicuente las muestras homogeneizadas en tubos de centrífuga de 50 mL y almacene en condiciones frescas y secas.
8. Preparación de la muestra
- Preparar un cronograma para realizar el estudio de validación durante tres días no consecutivos (para evaluar la reproducibilidad interna). Para cada día, ensaye tres niveles de concentración fortificando muestras a 225 ng/g (1,5 mL), 150 ng/g (ML) y 75 ng/g (0,5 mL). Estudie cada nivel por triplicado, representando nueve muestras con picos más otras tres muestras en blanco por día.
NOTA: Teniendo en cuenta los 3 días de validación, se requerirán un total de 36 muestras junto con otras 8 muestras en blanco destinadas a preparar la curva de calibración emparejada con la matriz. Use guantes de nitrilo, bata de laboratorio y gafas de seguridad para toda esta sección del protocolo. - Pesar 1 g de muestra en un tubo de centrífuga de 50 mL.
NOTA: Al realizar el estudio de validación, fortalezca las muestras justo después del pesaje con 50 μL de la solución estándar de trabajo correspondiente detallada en los pasos 1.3. y 1.5. - Añadir 4 mL de la solución de extracción [acetonitrilo:5 mM de solución acuosa de carbonato de amonio (85:15, v/v)]. Agite la muestra durante 1 min.
- Centrifugar la muestra durante 5 min a 9.750 × g y 4 °C.
NOTA: La capacidad de la centrífuga puede ser un factor limitante en el procedimiento, por lo que se recomienda extraer simultáneamente solo el número exacto de muestras que caben en la centrífuga. - Transfiera todo el sobrenadante al tubo de centrífuga de 15 ml que contiene 150 mg de la mezcla de sorbente de limpieza (1:1, p/p). Agite la muestra durante 1 min.
- Centrifugar el tubo durante 5 min a 9.750 × g y 4 °C.
- Recoja el sobrenadante y colóquelo en un frasco de vidrio ámbar de 4 ml. Evapore el extracto bajo un suave chorro de nitrógeno.
NOTA: Las muestras deben mantenerse secas a -20 °C si no se analizan el mismo día. - Reconstituya la muestra en 750 μL de la mezcla de metanol:agua (1:1, v/v).
- Transfiera el extracto a una jeringa de 2 ml y fíltrelo a través de un filtro de nailon de 0,22 μm en un vial cromatográfico ámbar de 1,5 ml.
- Recoja los extractos reconstituidos de las ocho muestras en blanco destinadas a la curva de calibración emparejada con la matriz en un vial de vidrio ámbar de 12 ml (cada muestra se resuspende en 750 μl, lo que representa un volumen total de 6 ml).
- Pipetear 1 mL de la solución intermedia a 1.000 ng/mL (83,33 ng/mL por cada alcaloide de cornezuelo) (paso 1.2) en un vial de vidrio ámbar de 2 mL, secar el disolvente bajo un suave chorro de nitrógeno y volver a suspender en 1 mL del extracto de muestra en blanco recogido anteriormente.
NOTA: Esta mezcla a una concentración de 1.000 ng/mL (83,33 ng/mL para cada EA) se utilizará para preparar la curva de calibración emparejada con la matriz. - Prepare diez viales de vidrio ámbar de 2 mL y coloque 450 μL de extracto de muestra en blanco (paso 8.10) en cada uno y márquelos con números del 1 al 10. Cada vial será un punto de la curva de calibración.
NOTA: En este protocolo, se utilizaron 450 μL ya que era el valor más bajo que el dispositivo LC podía alcanzar dentro del vial sin utilizar insertos. Si la aguja no llega tan lejos en el vial, aumente el volumen y, en consecuencia, calcule cuántas muestras en blanco adicionales deben extraerse considerando que cada una se resuspende en 750 μL de mezcla de metanol:agua (1/1, v:v). - Construya la curva de calibración por dilución en serie. Transfiera 450 μL de la mezcla preparada en el paso 8.11. en el vial 1, que ya contiene 450 μL de extracto de muestra en blanco, para alcanzar una concentración de 41,67 ng/mL (el punto de calibración más alto). Agite el vial durante 2 min.
NOTA: El volumen de la mezcla debe coincidir con el volumen del extracto de muestra en blanco para diluir dos veces en cada paso. - Transfiera 450 μL del vial 1 al vial 2, que ya contiene 450 μL de extracto de muestra en blanco, para alcanzar una concentración de 20,83 ng/mL (el segundo punto de calibración más alto).
- Repita el paso 8.14 (es decir, del vial 2 al vial 3, y así sucesivamente) y continúe posteriormente hasta obtener los diez puntos de calibración. El rango de concentración para cada EA será: 41,67, 20,83, 10,42, 5,21, 2,60, 1,30, 0,65, 0,33, 0,16 y 0,08 ng/mL.
- Prepare una mezcla estándar a 1.000 ng/mL (83,33 ng/mL para cada EA) como se detalla en el paso 8.11 pero utilizando 1 mL de mezcla de metanol:agua (1:1, v/v).
- Prepare una curva de calibración estándar como se detalla en los pasos 8.13 a 8.15, pero utilizando 450 μL de mezcla de metanol:agua (1:1, v/v) para las diluciones en serie. Esta curva de calibración se utilizará para evaluar el efecto de la matriz comparándola con la curva de calibración emparejada con la matriz.
9. Tratamiento cuantitativo de los datos
- Inicialice y ejecute el instrumento siguiendo las instrucciones descritas en los pasos 4.1. al 4.11. Consulte la Figura 2 para ver la lista de trabajo utilizada en este protocolo.
NOTA: Al escribir la lista de trabajo para las curvas de calibración, inyéctelas antes de las muestras enriquecidas comenzando con la curva de calibración estándar de la concentración más baja a la más alta, un vial de lavado que contenga metanol:agua (1:1, v/v) y la curva de calibración emparejada con la matriz de la concentración más baja a la más alta. - Abra el software cuantitativo y vaya a la pestaña rápida de TASQ | Importar lote en la parte inferior izquierda.
- Seleccione la carpeta creada en el paso 4.10. que contiene todos los datos brutos (archivos relacionados con la información adquirida después del análisis LC-IMS-MS para todas las muestras incluidas en el pool de trabajo).
- Una vez importado al software, vaya a Procesar lote, justo debajo de Importar lote.
- Elija las muestras a procesar (es decir, las 27 muestras con picos, 9 blancos y ambas curvas de calibración) y el método de procesamiento de datos creado anteriormente en los pasos 6.1. al 6.3. Aparecerá una ventana con diferentes parámetros para configurar (es decir, el tipo de integrador para evaluar los picos cromatográficos o el tipo de señal analítica a considerar, entre otros). Cambie esos parámetros como desee, aunque los valores predeterminados son adecuados para la ejecución de este protocolo.
- Vaya a Revisar el cribado una vez finalizado el procesamiento de datos y compruebe si hay algún problema relacionado con la integración automática, como la selección de picos cromatográficos incorrectos que pueden estar demasiado cerca unos de otros. Si es así, limite los valores referidos a la tolerancia de tiempo de retención, tolerancia de valores CCS y/o error de masa establecidos inicialmente en el archivo de método cuantitativo y vuelva a procesar los datos. Si la integración automática de picos sigue seleccionando picos incorrectos, integre manualmente el pico seleccionando el área en el cromatograma que aparece en la ventana Revisar cribado (para ello, tenga en cuenta los parámetros analíticos obtenidos para los estándares de EA en la sección 5).
NOTA: Es muy importante revisar cada muestra, ya que algunos pares principales de epímeros EA tienen tiempos de retención muy similares y pueden ser identificados erróneamente por el software. - Cuando todos los picos se hayan integrado con éxito para todas las muestras, haga clic en la pestaña Gestión de lotes en la esquina superior izquierda e indique el tipo de muestra asociada a cada análisis: disolvente, blanco, control de calidad, muestra o punto de calibración. En este último caso, asigne a cada punto de calibración un nivel con un número.
- En la misma pestaña Gestión de lotes , vaya a la ventana inferior denominada Concentración de lotes y especifique un valor de concentración para cada nivel establecido en el paso anterior. Una vez hecho esto, haga clic en Guardar concentraciones.
- Haga clic en Cuantificar lote y cambie los parámetros como desee (es decir, el modelo para que se ajuste a los datos de calibración, ponderación, si el modelo se ve obligado a cruzar cero, etc.). Los valores predeterminados son adecuados para la ejecución de este protocolo, excepto para la ponderación de las curvas de calibración, que se cambió a 1/x.
NOTA: Dado que el software solo puede considerar una curva de calibración a la vez, es necesario repetir el paso 9.9 dos veces para tener información sobre las curvas de calibración estándar y coincidentes con la matriz. En este protocolo, se utilizaron curvas de calibración emparejadas por matrices para la cuantificación de analitos, por lo que primero se evaluaron las curvas de calibración del solvente, se extrajeron los datos de calibración como se explica más adelante en el paso 9.13 y, finalmente, se seleccionaron las curvas de calibración emparejadas con matrices para realizar la cuantificación de las muestras. - Una vez realizada la cuantificación de los alcaloides del cornezuelo de centeno según la ficha del método de cuantificación, haga clic en la pestaña Cuantificación en la parte superior de la pantalla.
- Si los parámetros predeterminados establecidos en el paso 9.9 no proporcionan una buena linealidad y precisión, ajuste las curvas de calibración cambiando el ajuste y el peso haciendo clic derecho en el gráfico con el objetivo de tener un buen modelo lineal (R2 > 0,99) con la precisión adecuada en cada nivel de calibración (desviación inferior al 20%).
NOTA: El software marcará en rojo las celdas de aquellos niveles de calibración que no proporcionen la precisión adecuada. - Una vez que haya terminado, haga clic con el botón derecho en la carpeta del lote en el extremo izquierdo de la pantalla y seleccione Generar informe. A continuación, seleccione Cuantificación de analitos para exportar el informe al formato deseado.
Resultados
En primer lugar, se inyectaron soluciones estándar de trabajo en el instrumento LC-IMS-MS para obtener todas las características de identificación (es decir, tiempo de retención, CCS y espectros de masas) de cada EA analizado aquí. Dado que inicialmente se desconocían los parámetros de identificación, excepto la masa exacta, el método de adquisición se basó en un evento de dos exploraciones, comenzando con un escaneo completo de todo el espectro de masas seguido de un bbCID. L...
Discusión
El uso exitoso de este protocolo se basa en la optimización del procedimiento de extracción, previamente realizada por Carbonell-Rozas et al.17, quienes implementaron el uso de un solvente de extracción lo suficientemente efectivo para extraer EAs de matrices alimentarias complejas como la cebada y el trigo, y una limpieza que proporcionó valores de SSE relativamente bajos. La elección del disolvente de extracción representa un paso crítico teniendo en cuen...
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Esta investigación ha sido financiada por la Consejería de Universidad, Investigación e Innovación - Junta de Andalucía (PROYEXCEL_00195) y la beca postdoctoral concedida por la Generalitat Valenciana y el Fondo Social Europeo+ (CIAPOS/2022/049). Los autores agradecen al Centro de Instrumentación Científica (CIC) de la Universidad de Granada por facilitar el acceso a la instrumentación analítica utilizada en este protocolo.
Materiales
| Name | Company | Catalog Number | Comments |
| Acetonitrile | VWR | 83640.32 | |
| Amber glass tubes 4 mL | VWR | 548-0052 | |
| Amber glass tubes 12 mL | VWR | 548-0903 | |
| Amber vials 1.5 mL | Agilent | 5190-9063 | |
| Ammonium carbonate | Fluka | 9716 | |
| Analytical balance BAS 31 | Boeco | 4400519 | |
| Balance CP 323 S | Sartorius | 23-84182 | |
| C18 | Supelco | 52604-U | |
| Centrifuge tubes, 15 mL | VWR | 525-1082 | |
| Centrifuge tubes, 50 mL | VWR | 525-0155 | |
| Centrifuge Universal 320 R | Hettich | 1406 | |
| Compass HyStar | Bruker | Acquisition software | |
| DataAnalysis | Bruker | Qualitative software | |
| Elute PLUS UHPLC | Bruker | ||
| EVA EC-S evaporator | VLM | V830.012.12 | |
| Formic acid GR for analysis ACS, Reag. Ph Eur | Merck | 100264 | |
| Grinder TitanMill300 | Cecotec | 1559 | |
| Methanol | VWR | 83638.32 | |
| Milli-Q water purification system (18.2 MΩ cm) | Millipore | ZD5211584 | |
| Pipette tips 1- 5 mL | Labortecnic | 162005 | |
| Pipette tips 100 - 1000 µL | Labortecnic | 1622222 | |
| Pipette tips 5 - 200 µL | Labortecnic | 162001 | |
| Pippette Transferpette S variable, DE-M 10 - 100 µL | BRAND | 704774 | |
| Pippette Transferpette S variable, DE-M 100 - 1000 µL | BRAND | 704780 | |
| Pippette Transferpette S variable, DE-M 500 - 5000 µL | BRAND | 704782 | |
| Syringe 2 mL | VWR | 613-2003 | |
| Syringe Filter 13 mm, 0.22µm | Phenomenex | AF-8-7707-12 | |
| TASQ | Bruker | Quantitative software | |
| timsTOFPro2 IM-HRMS | Bruker | ||
| Vortex Genie 2 | Scientific Industries | 15547335 | |
| Zorbax Eclipse Plus RRHD C18 column (50 x 2.1 mm, 1.8 µm particle size) | Agilent | 959757-902 | |
| Z-Sep+ | Supelco | 55299-U | Zirconia-based sorbent |
| Ergot alkaloids | CAS registry sorbent | ||
| Ergocornine (Eco) | Techno Spec | E178 | 564-36-3 |
| Ergocorninine (Econ) | Techno Spec | E130 | 564-37-4 |
| Ergocristine (Ecr) | Techno Spec | E180 | 511-08-0 |
| Ergocristinine (Ecrn) | Techno Spec | E188 | 511-07-9 |
| Ergokryptine (Ekr) | Techno Spec | E198 | 511-09-1 |
| Ergopkryptinine (Ekrn) | Techno Spec | E190 | 511-10-4 |
| Ergometrine (Em) | Romer Labs | "002067" | 60-79-7 |
| Ergometrinine (Emn) | Romer Labs | LMY-090-5ML | 479-00-5 |
| Ergosine (Es) | Techno Spec | E184 | 561-94-4 |
| Ergosinine (Esn) | Techno Spec | E194 | 596-88-3 |
| Ergotamine (Et) | Romer Labs | "002069" | 113-15-5 |
| Ergotaminine (Etn) | Romer Labs | "002075" | 639-81-6 |
Referencias
- Kanu, A. B., Dwivedi, P., Tam, M., Matz, L., Hill, H. H. Ion mobility-mass spectrometry. J Mass Spectrom. 43 (1), 1-22 (2008).
- Gabelica, V., et al. Recommendations for reporting ion mobility Mass spectrometry measurements. Mass Spectrom Rev. 38 (3), 291-320 (2019).
- Feuerstein, M. L., et al. Comparability of steroid collision cross sections using three different IM-HRMS technologies: An interplatform study. J Am Soc Mass Spectrom. 33 (10), 1951-1959 (2022).
- Regueiro, J., Negreira, N., Berntssen, M. H. G. Ion-mobility-derived collision cross section as an additional identification point for multiresidue screening of pesticides in fish feed. Anal Chem. 88 (22), 11169-11177 (2016).
- Regueiro, J., Negreira, N., Hannisdal, R., Berntssen, M. H. G. Targeted approach for qualitative screening of pesticides in salmon feed by liquid chromatography coupled to traveling-wave ion mobility/quadrupole time-of-flight mass spectrometry. Food Control. 78, 116-125 (2017).
- Hernández-Mesa, M., Monteau, F., Le Bizec, B., Dervilly-Pinel, G. Potential of ion mobility-mass spectrometry for both targeted and non-targeted analysis of phase II steroid metabolites in urine. Anal Chim Acta: X. 1, 100006 (2019).
- George, A. C., et al. Interplatform comparison between three ion mobility techniques for human plasma lipid collision cross sections. Anal Chim Acta. 1304, 342535 (2024).
- Mairinger, T., Causon, T. J., Hann, S. The potential of ion mobility-mass spectrometry for non-targeted metabolomics. Curr Opin Chem Biol. 42, 9-15 (2018).
- Hernández-Mesa, M., Escourrou, A., Monteau, F., Le Bizec, B., Dervilly-Pinel, G. Current applications and perspectives of ion mobility spectrometry to answer chemical food safety issues. TrAC Trend Anal Chem. 94, 39-53 (2017).
- European, C. Commission Regulation (EU) 2023/915 of 25 April 2023 on maximum levels for certain contaminants in food and repealing Regulation (EC) No 1881/2006. Off J Eur Union. 119, 103-157 (2023).
- Carbonell-Rozas, L., et al. Occurrence of egot alkaloids in major and minor cereals from Northern Italy: A three harvesting years scenario. J Agricl Food Chem. 71 (42), 15821-15828 (2023).
- Arroyo-Manzanares, N., Gámiz-Gracia, L., García-Campaña, A. M., Diana Di Mavungu, J., De Saeger, S., Jean-Michel Mérillon, J. -. M., Ramawat, K. G. . Fungal Metabolites. , (2016).
- van Dongen, P., de Groot, A. History of ergot alkaloids from ergotism to ergometrine. Eur J Obstet Gynecol Reprod Biol. 60 (2), 109-116 (1995).
- European Food Safety Authority. Human and animal dietary exposure to ergot alkaloids. EFSA J. 15 (7), e04902 (2017).
- Nam, M., Kim, D., Kim, M. -. S. Simultaneous determination of total ergot alkaloids in wheat flour by Orbitrap mass spectrometry. Food Chem. 441, 138363 (2024).
- García-Juan, A., León, N., Armenta, S., Pardo, O. Development and validation of an analytical method for the simultaneous determination of 12 ergot, 2 tropane, and 28 pyrrolizidine alkaloids in cereal-based food by LC-MS/MS. Food Res Int. 174 (Pt 1), 113614 (2023).
- Carbonell-Rozas, L., Mahdjoubi, C. K., Arroyo-Manzanares, N., García-Campaña, A. M., Gámiz-Gracia, L. Occurrence of ergot alkaloids in barley and wheat from Algeria. Toxins. 13 (5), 316 (2021).
- Carbonell-Rozas, L., Gámiz-Gracia, L., Lara, F. J., García-Campaña, A. M. Determination of the main ergot alkaloids and their epimers in oat-based functional foods by ultra-high performance liquid chromatography tandem mass spectrometry. Molecules. 26 (12), 3717 (2021).
- Carbonell-Rozas, L., et al. Ion mobility-mass spectrometry to extend analytical performance in the determination of ergot alkaloids in cereal samples. J Chromatogr A. 1682, 463502 (2022).
- European Commission. Commission Implementing Regulation (EU) 2023/2782 of 14 December 2023 laying down the methods of sampling and analysis for the control of the levels of mycotoxins in food and repealing Regulation (EC) No 401/2006. Off J Eur Union. , (2023).
- Carbonell-Rozas, L., Vander Cruyssen, L., Dall'Asta, C., Leggieri, M. C., Battilani, P. Fit-for-purpose method development to determine co-occurring multiclass mycotoxins in apple and apple puree samples. Food Anal Methods. 16 (8), 1403-1412 (2023).
- Laouni, C., et al. Emerging mycotoxin occurrence in chicken feed and eggs from Algeria. Mycotoxin Res. 40, 447-456 (2024).
- Ben Hassouna, K., et al. Mycotoxin occurrence in milk and durum wheat samples from Tunisia using dispersive liquid-liquid microextraction and liquid chromatography with fluorescence detection. Toxins. 15 (11), 633 (2023).
- Narváez, A., et al. Occurrence and exposure assessment of mycotoxins in ready-to-eat tree nut products through ultra-high performance liquid chromatography coupled with high resolution Q-orbitrap mass spectrometry. Metabolites. 10 (9), 344 (2020).
- Arroyo-Manzanares, N., Rodríguez-Estévez, V., García-Campaña, A. M., Castellón-Rendón, E., Gámiz-Gracia, L. Determination of principal ergot alkaloids in swine feeding. J Sci Food Agric. 101 (12), 5214-5224 (2021).
- Pereira, V. L., Fernandes, J. O., Cunha, S. C. Comparative assessment of three cleanup procedures after QuEChERS extraction for determination of trichothecenes (type A and type B) in processed cereal-based baby foods by GC-MS. Food Chem. 182, 143-149 (2015).
- Tuzimski, T., Szubartowski, S. Method development for selected bisphenols analysis in sweetened condensed milk from a can and breast milk samples by HPLC-DAD and HPLC-QqQ-MS: Comparison of sorbents (Z-SEP, Z-SEP Plus, PSA, C18, chitin and EMR-lipid) for clean-up of QuEChERS extract. Molecules. 24 (11), 2093 (2019).
- Łozowicka, B., Rutkowska, E., Jankowska, M. Influence of QuEChERS modifications on recovery and matrix effect during the multi-residue pesticide analysis in soil by GC/MS/MS and GC/ECD/NPD. Environ Sci Pollut Res. 24 (8), 7124-7138 (2017).
- Schummer, C., Xandonella, I., van Nieuwenhuyse, A., Moris, G. Epimerization of ergot alkaloids in feed. Heliyon. 6 (6), e04336 (2020).
- Cherewyk, J. E., Grusie-Ogilvie, T. J., Parker, S. E., Blakley, B. R., Al-Dissi, A. M. The impact of storage temperature and time on ergot alkaloid concentrations. Toxins. 15 (8), 497 (2023).
- Silva, &. #. 1. 9. 4. ;., Mateus, A. R., Barros, S. C., Silva, A. S. Ergot alkaloids on cereals and seeds: Analytical methods, occurrence, and future perspectives. Molecules. 28 (20), 7233 (2023).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados