Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Nachweis von regulierten Mutterkornalkaloiden in Lebensmittelmatrizen mittels Flüssigchromatographie-Mobilitätsspektrometrie-Flugzeitspektrometrie
In diesem Artikel
Zusammenfassung
Dieses Protokoll stellt eine validierte Massenspektrometrie mit Flüssigchromatographie, Ionenmobilität und hoher Auflösung vor, um das Vorhandensein von Mutterkornalkaloiden in Lebensmitteln in Übereinstimmung mit der kürzlich veröffentlichten Verordnung (EU) 2023/915 der Kommission zu bestimmen.
Zusammenfassung
Die Ionenmobilitäts-Massenspektrometrie (IMS) fungiert als zusätzliche Trenndimension, wenn sie in Arbeitsabläufe der Flüssigchromatographie-Massenspektrometrie (LC-MS) integriert wird. LC-IMS-MS-Methoden bieten im Vergleich zu herkömmlichen LC-MS-Methoden eine höhere Peakauflösung, eine verbesserte Trennung von isobaren und isomeren Verbindungen und ein verbessertes Signal-Rausch-Verhältnis (S/N). IMS bietet eine weitere molekulare Eigenschaft für die Identifizierung von Analyten, nämlich den Kollisionsquerschnittsparameter (CCS), der falsch positive Ergebnisse reduziert. Daher adressieren LC-IMS-MS-Methoden wichtige analytische Herausforderungen im Bereich der Lebensmittelsicherheit (d.h. Nachweis von Verbindungen in Spurenmengen in komplexen Lebensmittelmatrizen und eindeutige Identifizierung von isobaren und isomeren Molekülen).
Mutterkornalkaloide (EAs) sind eine Familie von Mykotoxinen, die von Pilzen produziert werden und eine Vielzahl von Grasarten befallen, darunter kleine Getreidesorten wie Roggen, Triticale, Weizen, Gerste, Hirse und Hafer. In mehreren Lebensmitteln wurden Höchstgehalte (ML) dieser Mykotoxine festgelegt, wie in der Verordnung (EG) 2023/915 der Kommission festgelegt. Diese neue Gesetzgebung umfasst sechs Haupt-EAs und ihre entsprechenden Epimere, so dass eine effiziente Methodik erforderlich ist, um diese isomeren Moleküle unter Berücksichtigung ihres gemeinsamen Auftretens richtig zu unterscheiden.
Daher ist es das Ziel dieses Protokolls zu zeigen, wie die Integration von IMS in LC-MS-Arbeitsabläufe zur Trennung isomerer EAs beiträgt und die Selektivität der Analysemethode erhöht. Darüber hinaus wird veranschaulicht, wie die Generierung von CCS-Bibliotheken durch die Charakterisierung analytischer Standards eine höhere Zuverlässigkeit bei der Identifizierung von Mykotoxinen bietet. Mit diesem Protokoll sollen die Vorteile der Umsetzung von IMS im Bereich der Lebensmittelsicherheit am Beispiel der Bestimmung von EA in Getreide klar erläutert werden. Eine QuEChERS-basierte Extraktion, gefolgt von einer LC-gefangenen Ionenmobilitätsspektrometrie (TIMS)-MS-Analyse, ergab Bestimmungsgrenzen von 0,65 bis 2,6 ng/g mit akzeptabler Genauigkeit (wenn auch geringe Wiederfindung für Ergotaminin) bei 1,5x, 1x und 0,5x der ML und zeigte einen vernachlässigbaren Matrixeffekt.
Einleitung
Die Ionenmobilitäts-Massenspektrometrie (IMS) wird zu einer zunehmend eingesetzten Analysetechnik, die oft als zusätzliche Trenndimension in die traditionelle Flüssigkeits-/Gaschromatographie (LC/GC) integriert und an MS-Arbeitsabläufe gekoppelt ist. IMS besteht aus der Trennung von Molekülen entlang einer Mobilitätszelle, die mit einem Puffergas gefüllt ist, unter einem elektrischen Feld und bei Atmosphärendruck1. Abhängig vom Masse-Ladungs-Verhältnis (m/z) und der geometrischen Konformation interagiert ein ionisiertes Molekül mit dem Puffergas, während es sich durch die Mobilitätszelle bewegt, was sich im Ionenmobilitätsparameter (K)2 widerspiegelt und durch die folgende Gleichung berechnet wird:
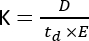
Dabei steht D für die Gesamtdriftlänge, td für die Gesamtdriftzeit und E für das elektrische Feld. Daher wird K in m2 V−1 s−1 gemessen, obwohl es aus praktischen Gründen oft als cm2 V−1 s−1 ausgedrückt wird. Die intrinsische Fähigkeit, sich durch die Mobilitätszelle zu bewegen, kann anhand der Driftzeit gemessen und später in den sogenannten Kollisionsquerschnitt (CCS)-Wert umgerechnet werden, der für jedes Molekül unabhängig vom IMS-Instrument3 ein hochgradig reproduzierbarer Parameter ist. Die CCS kann aus der Mobilität nach folgender Gleichung abgeleitet werden:
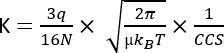
q ist die Ladung des Ions; N die Dichte der Puffergaszahl; μ die reduzierte Masse der Kollisionspartner puffert Gas-Ionen; kB die Boltzmann-Konstante; und T die Temperatur des Puffergases. Daher bietet IMS zusätzliche Informationen, die die analytischen Daten aus der Chromatographie und MS-Analyse ergänzen.
Es hat sich gezeigt, dass die Implementierung von IMS in LC-MS-Plattformen die Zuverlässigkeit analytischer Bestimmungen erhöht, insbesondere bei der Arbeit mit Verbindungen, die in Spurenkonzentrationen vorliegen. Mehrere Studien haben berichtet, dass LC-IMS-MS-Methoden die Qualität der Massenspektren verbessern, indem sie das Hintergrundrauschen reduzieren, was sich letztendlich auf die Empfindlichkeit der Methode auswirkt, und die Rate falsch positiver und negativer Ergebnisse durch LC-MS-Methoden mit mehreren Resten reduziert 4,5,6. Darüber hinaus ermöglicht die Reproduzierbarkeit der CCS-Werte den Vergleich nicht nur zwischen verschiedenen Instrumenten, die dieselbe Technologie verwenden, sondern auch zwischen verschiedenen Ionenmobilitätstechnologien, nämlich der Wanderwellen-Ionenmobilitätsspektrometrie (TWIMS), der Mobilitätsspektrometrie gefangener Ionen (TIMS) und der Driftröhren-Ionenmobilitätsspektrometrie (DTIMS)2,7, die die am häufigsten verwendeten Systeme sind 1. Eine bemerkenswerte Konsequenz des Potenzials von CCS als Identifikationsparameter liegt daher in der Möglichkeit, CCS-Bibliotheken aufzubauen, was sich in seiner Anwendbarkeit in Metabolomik-Studien widerspiegelt8. Nichtsdestotrotz ist eine der leistungsfähigsten Eigenschaften von IMS die Fähigkeit, isomere und isobare Verbindungen zu trennen, die mit LC-MS-Methoden möglicherweise nicht ausreichend aufgelöst werden können. Dies kann der Fall sein, wenn mit großen Mengen von Analyten von Interesse in komplexen Matrices gearbeitet wird, was in der Umwelt- und Lebensmittelanalytik häufig der Fall ist. In diesem Zusammenhang wurden LC-IMS-MS-Methoden für die Überwachung von Pestiziden und in geringerem Maße von Tierarzneimitteln und Mykotoxinen in Lebensmitteln vorgeschlagen9.
Aufgrund ihres hohen Auflösungsvermögens und ihrer Selektivität erweisen sich LC/GC-IMS-MS-Plattformen als die nützlichsten Werkzeuge, um einige der aktuellen Herausforderungen in der Lebensmittelsicherheit zu bewältigen, insbesondere im Zusammenhang mit isomeren Gemischen. Die gesundheitlichen Bedenken in Bezug auf isomere Gemische als Lebensmittelkontaminanten spiegeln sich in den geltenden europäischen Rechtsvorschriften wider, die beispielsweise die Höchstkonzentration von sechs Hauptmutterkornalkaloiden (EA) und den entsprechenden sechs Epimeren in mehreren Lebensmitteln begrenzen10.
EAs stellen eine Familie toxischer Sekundärmetaboliten dar, die von einer Vielzahl von Pilzen produziert werden, hauptsächlich aus der Familie der Clavicipitaceae (z. B. Claviceps purpurea, der aufgrund seines breiten Wirtsspektrums der wichtigste EA-Produzent ist), aber auch von Trichocomaceae, die den Samenkopf lebender Pflanzen (wie Roggen, Gerste, Weizen und Hafer) zum Zeitpunkt der Blüte parasitieren können11. 12. Schadenfreude Unter bestimmten Bedingungen, insbesondere bei Temperatur und Wasseraktivität, können Clavizeps-Pilze EAs produzieren, die sich in Fruchtkörpern, den sogenannten Sklerotien oder Mutterkorn, in der Wirtskultur anreichern. Bis zu einem gewissen Grad können EAs der Verarbeitung des Rohstoffs bis zum Erreichen des Endprodukts standhalten; daher in die Nahrungskette einzubrechen. Die Einnahme kontaminierter Lebensmittel kann zu einer EA-Vergiftung führen, die als Ergotismus bekannt ist und sich mit akuten Symptomen wie Bauchschmerzen, Erbrechen, Brennen der Haut, Schlaflosigkeit und Halluzinationen äußert13. Um die Auswirkungen von EA auf die menschliche Gesundheit zu verringern, hat die Europäische Kommission in mehreren Lebensmitteln Höchstgehalte für die Summe der wichtigsten EA festgelegt: die R-Epimere Ergometrin (Em), Ergotamin (Et), Ergosin (Es), Ergocristin (Ecr), Ergokryptin (Ekr) und Ergocornin (Eco) und die entsprechenden S-Epimere: Ergometrinin (Emn), Ergosinin (Esn), Ergotaminin (Etn), Ergocorninin (Econ), Ergokryptinin (Ekrn), und Ergocristinin (Ecrn). Diese Verbindungen können von R- in S-Formen und umgekehrt epimerisieren, insbesondere bei starker Lichteinwirkung, längerer Lagerung oder Kontakt mit einigen Lösungsmitteln bei hohem oder niedrigem pH-Wert von 12. Obwohl der Anteil der R- und S-Formen unter verschiedenen Bedingungen variieren kann, stellte das CONTAM-Gremium der EFSA nach Überprüfung der verfügbaren Literatur zu EA in Lebensmitteln ein höheres Vorkommen von R-Formen als von S-Formen fest 14. Daher variieren die MLs in Abhängigkeit von mehreren Faktoren, wie z. B. der Anfälligkeit der Pflanze, dem Verarbeitungsgrad oder der Häufigkeit des Verbrauchs. Im EU-Rahmen wurden die Mindestwertmengen für gemahlene Erzeugnisse aus Gerste, Weizen, Dinkel und Hafer auf 50 bzw. 150 μg/kg (je nach Aschegehalt von weniger oder mehr als 900 mg/100 g) festgesetzt, während für Getreide, das direkt für den menschlichen Verzehr bestimmt ist, ein Mindestwert von 150 μg/kg erhoben wird, mit Ausnahme von Säuglingsnahrung auf Getreidebasis. in denen das ML auf 20 μg/kgreduziert wird 10.
Diese strenge Gesetzgebung erfordert Analysemethoden, die empfindlich genug sind, um die Spurenkonzentration (μg/kg) zu bestimmen und gleichzeitig regulierte EAs und ihre entsprechenden Epimere ordnungsgemäß zu identifizieren, da beide Formen, R- und S-Isomere, zusammen in kontaminierten Proben gefunden werden können. Diese Aufgabe stellt eine große Herausforderung dar, da jedes Toxin-Epimer-Paar genau die gleiche Masse und das gleiche Fragmentierungsmuster aufweist. Darüber hinaus kann eine ordnungsgemäße chromatographische Trennung zwischen beiden Verbindungen komplex sein. Daher sind gut optimierte LC-Gradienten erforderlich, um Fehlquantifizierungen zu vermeiden, wenn EA-Epimere in Lebensmittelproben gleichzeitig auftreten. Obwohl mehrere Studien über LC-MS-Methoden zur eindeutigen Bestimmung von EAs berichtet haben 15,16,17,18, muss die chromatographische Methode umfassend untersucht werden, um eine angemessene Trennung der chromatographischen Peaks zu erreichen und EAs eindeutig zu identifizieren. Dies ist jedoch bei Mehrklassenmethoden, bei denen Kontaminanten, die zu verschiedenen chemischen Familien gehören, gleichzeitig bestimmt werden, in der Regel nicht realisierbar. In diesem Zusammenhang berichtete eine kürzlich von Carbonell-Rozas, Hernández-Mesa et al.19 durchgeführte Studie über eine LC-IMS-MS-Methode zur Quantifizierung von EAs in Weizen- und Gerstenproben, bei der zwei verschiedene TWIMS-Instrumente verwendet wurden, die reproduzierbare CCS-Werte und niedrige Bestimmungsgrenzen (LOQs) lieferten, um eine Nichteinhaltung in Übereinstimmung mit den geltenden Rechtsvorschriften zu erkennen. Daher ist es das Ziel dieses Protokolls zu zeigen, wie die Integration von IMS in LC-MS-Arbeitsabläufe zur Trennung isomerer EAs beiträgt und die Selektivität der Analysemethode erhöht. Darüber hinaus wird veranschaulicht, wie die Generierung von CCS-Bibliotheken durch die Charakterisierung analytischer Standards eine höhere Zuverlässigkeit bei der Identifizierung von Mykotoxinen bietet. Mit diesem Protokoll sollen die Vorteile der Implementierung von IMS in der Lebensmittelsicherheitsanalyse am Beispiel der Bestimmung von EA in Getreide klar erläutert werden. Dieses Protokoll befasst sich mit der Probenbehandlung auf der Grundlage eines QuEChERS-Verfahrens, der Probenanalyse mittels LC-TIMS-MS und der IMS-Datenextraktion und -interpretation.
Protokoll
1. Vorbereitung von Lager-, Zwischen- und Arbeitsstandardlösungen
HINWEIS: Verwenden Sie Nitrilhandschuhe, einen Laborkittel und eine Schutzbrille.
- Bereiten Sie einzelne Stammlösungen der 12 EAs (siehe Materialtabelle) bei 10.000 ng/ml in 4-ml-Braunglasfläschchen mit Acetonitril her. Die R-Formen (-ine) wurden zuvor in 25.000 ng-Portionen aliquotiert, während die S-Formen (-inin) in 10.000 ng-Portionen verteilt waren. Diese Studie begann mit diesen Aliquoten und erreichte Konzentrationen von 10.000 ng/ml, indem die R- und S-Formen in 2,5 bzw. 1 ml Acetonitril verdünnt und anschließend 2 Minuten lang vortexiert wurden.
- Bereiten Sie eine Zwischenlösung mit 1.000 ng/ml Gesamt-EAs (jeweils 83,33 ng/ml) in einem 12-ml-Braunglasfläschchen vor, indem Sie 65 μl jeder einzelnen Alkaloidlösung und 7,02 mL Acetonitril hinzufügen.
HINWEIS: Sobald die Zwischenlösung hergestellt ist, müssen die einzelnen Stammlösungen getrocknet und bei -20 °C mit Aluminiumfolie abgedeckt gelagert werden, um eine Epimerisierung zu vermeiden. - Bereiten Sie funktionierende Standardlösungen aus der Zwischenlösung her, indem Sie 2,7, 1,8 und 0,9 ml in 4-ml-Braunglasröhrchen pipettieren. Bringen Sie sie unter einen sanften Stickstoffstrahl zum Trocknen und resuspendieren Sie sie in 600 μl Acetonitril.
HINWEIS: Die resultierenden Konzentrationen erreichten das 1,5-, 1- und 0,5-fache des ml-Gehalts (150 ng/g), wenn 50 μl zu 1 g der Probe hinzugefügt wurden. Diese werden als Konzentrationsniveaus für Validierungsstudien (d. h. Wiederfindungen, Wiederholbarkeit usw.) ausgewählt. - Teilen Sie die 600 μl jeder Arbeitsstandardlösung in drei 2-ml-Braunglasfläschchen auf, indem Sie 200 μl pipettieren und bis zum Trocknen bringen.
HINWEIS: Die Schritte 1.2 bis 1.4 sind in Abbildung 1 schematisch dargestellt. Durch die Teilung und Trocknung der Arbeitsstandardlösungen ist es möglich, an drei verschiedenen Tagen, wie in der Validierungsstudie gefordert, dreifach anzureichern, ohne die chemische Integrität der EAs zu verlieren. - An jedem Validierungstag resuspendieren Sie ein 2-ml-Braunglasfläschchen jeder Anreicherungsstufe in 200 μl Acetonitril.
HINWEIS: Jeder Anreicherungsgrad wird in dreifacher Ausfertigung mit 50 μl pro Probe bewertet; Daher werden insgesamt 150 μl der Lösung verwendet.
2. Vorbereitung der Reagenzien und Lösungen
HINWEIS: Verwenden Sie Nitrilhandschuhe, einen Laborkittel und eine Schutzbrille.
- Bereiten Sie eine 5 mM Ammoniumcarbonatlösung her, indem Sie 24,02 mg Ammoniumcarbonat wiegen und in 50 mL Wasser auflösen. Wiegen Sie 24,02 mg Ammoniumcarbonat in ein Becherglas und fügen Sie ca. 5 ml Wasser hinzu. Lösen Sie das Ammoniumcarbonat auf, indem Sie die Lösung vorsichtig manuell umrühren, und geben Sie sie dann in einen 50-ml-Messkolben. Um das endgültige Volumen von 50 mL zu erreichen, spülen Sie das Becherglas mit zusätzlichem Wasser aus und übertragen Sie den Auftrieb in den Messkolben.
- 250 ml der Extraktionslösung [Acetonitril: 5 mM Ammoniumcarbonat (85:15, v/v)] werden durch Mischen von 212,5 ml Acetonitril und 37,5 ml der in Schritt 2.1 hergestellten Ammoniumlösung hergestellt.
- Bereiten Sie 100 ml eines Methanol-Wasser-Gemisches (1:1, v/v) vor, um die Probenextrakte zu resuspendieren (Schritt 8.8).
- Für jede Probe werden 150 mg eines dispersiven Festphasenmaterials C18:Zirkonoxid-basiertes Sorptionsmittelgemisch (1:1, w/w) in 15-ml-Zentrifugenröhrchen für die dispersive Reinigungsstufe (Schritt 8.5.) gewogen.
- Die Lösungsmittel werden für die chromatographische Trennung in zwei verschiedenen Flaschen vorbereitet, nämlich 1 l Reinstwasser (Lösungsmittel A) und Methanol (Lösungsmittel B), die beide 0,3 % (v/v) Ameisensäure enthalten. Um diese Konzentration zu erreichen, wurden 3,03 mL Ameisensäure (99 % Reinheit) in die Flaschen gegeben.
HINWEIS: Lösungsmittel für die mobile Phase sollten kurz vor der Durchführung der LC-IMS-MS-Analysen vorbereitet werden.
3. Einstellen von instrumentellen Parametern
HINWEIS: Das Instrument, das zur Durchführung dieser LC-IMS-MS-Studie verwendet wurde, war ein UHPLC-Gerät, das mit einem IM-HRMS gekoppelt war und mit einer vakuumisolierten sonde mit beheizter Elektrospray-Ionisation (VIP-HESI) ausgestattet war. Das Gerät wurde im positiven Modus betrieben.
- Erstellen Sie mit der Erfassungssoftware eine chromatographische Methode für die Trennung der Analyten, indem Sie den folgenden Elutionsgradienten einstellen: 0 min, 10% B; 2 min, 10% B; 4,5 Minuten, 40% B; 9 min, 45% B; 11 min, 95% B; 12 min, 95% B; 13 Minuten, 10% B; 16 Minuten, 10% B.
- Stellen Sie die Flussrate der mobilen Phase auf 0,4 mL/min, die Säulentemperatur auf 35 °C und das Injektionsvolumen auf 5 μl ein. Wenn möglich, halten Sie eine feste Temperatur von 10 °C im Autosampler.
- Stellen Sie die IMS-Parameter wie folgt ein: Mobilitätswerte (1/K0) von 0,1 bis 1,5 Vμs/cm2; Rampenzeit, 100 ms; Genauigkeitszeit, 10 ms; Einschaltdauer, 10%; und Rampenrate von 9,05 Hz.
- Stellen Sie die Parameter der Ionenquelle für die MS-Detektion wie folgt ein: Kapillarspannung, 2.500 V; Verneblerdruck, 2,5 bar; Durchfluss bei trockenem Gas, 8 l/min; Temperatur des trockenen Gases, 200 °C; Temperatur des Mantelgases, 450 °C; Durchflussmenge des Mantelgases, 4 L/min. Für die MS-Erfassung wählen Sie die folgenden Parameter unter Berücksichtigung von zwei Scan-Ereignissen: vollständige Abtastung des gesamten Massenspektrums mit einem m/z von 20 bis 1.300 , gefolgt von bbCID (Broadband Collision-Induced Dissociation), die Produktionsionen aus einem beliebigen Vorläufer bei drei festen Kollisionsenergien von 24, 36 und 50 eV erzeugt. Führen Sie Ionisation und Datenerfassung im Modus mit positiver Polarität durch.
- Speichern Sie diese Datei mit der Erfassungsmethode zur weiteren Verwendung.
4. Datenerfassung aus analytischen Standards der Umweltverträglichkeitsprüfungen
HINWEIS: Verwenden Sie Nitrilhandschuhe, Laborkittel und Schutzbrille nur für Schritt 4.1.
- Bereiten Sie zwei braune 1,5-ml-Fläschchen mit 500 μl Zwischenlösung bei 1.000 ng/mL und ein weiteres Fläschchen mit einer Mischung aus Lösungsmitteln A und B (1:1, v/v) als Waschlösung vor.
- Kalibrieren Sie das timsTOF-Gerät wie vom Hersteller angegeben und wählen Sie eine positive Polarität sowohl für IMS- als auch für MS-Erfassungen.
- Setzen Sie die C18-Säule (siehe Materialtabelle) in den LC-Ofen ein, wobei die Temperatur auf 35 °C eingestellt ist, wie in der in Schritt 3.5 gespeicherten Datei mit der Erfassungsmethode angegeben.
HINWEIS: Stellen Sie sicher, dass beide Enden gut angezogen sind. Andernfalls wird eine Leckage der mobilen Phase beobachtet. - Konditionieren Sie die LC-Säule so, dass eine Flussrate der mobilen Phase von 0,1 ml/min möglich ist, und wählen Sie ein Lösungsmittel-A- und B-Mischungsverhältnis von 90:10 (%) als anfängliches Zusammensetzungsverhältnis der mobilen Phase.
- Warten Sie, bis der Säulendruck stabil ist (~130 bar).
- Erhöhen Sie die Durchflussrate der mobilen Phase auf 0,2 mL/min, warten Sie, bis sich der Druck stabilisiert hat, und erhöhen Sie sie erneut auf 0,3 mL/min.
- Sobald sich der Druck wieder stabilisiert hat, stellen Sie die Durchflussrate auf 0,4 ml ein, was der Arbeitsdurchflussrate entspricht, wie in der in Schritt 3.5 gespeicherten Erfassungsmethodendatei beschrieben, und warten Sie, bis sich der Druck bei etwa 350 bar stabilisiert hat.
HINWEIS: Es ist wichtig, den Durchfluss nicht direkt von 0,2 auf 0,4 ml/min zu erhöhen. Eine starke Änderung der Strömung provoziert Hochdruckspitzen, die die Chromatographiesäule beschädigen können. - Während die Säule konditioniert wird, ist die Arbeitsliste zu schreiben, die zwei Proben enthält, die den beiden Fläschchen entsprechen, die die EA-Zwischenlösung bei 1.000 ng/ml enthalten, wie in Schritt 4.1 beschrieben, und das Waschfläschchen. Analysieren Sie das Waschfläschchen zweimal, zu Beginn und am Ende der Probencharge.
- Laden Sie in der Spalte Methodendatei des Arbeitsvorrats die zuvor in Schritt 3.5 gespeicherte Erfassungsmethodendatei.
- Erstellen Sie einen Ordner, in dem Sie alle erfassten Datendateien speichern und alle Samples weiterleiten können, indem Sie den Ordner in der Spalte Sample-Pfad auswählen.
HINWEIS: Stellen Sie sicher, dass Sie denselben Ordner verwenden, um alle während der Validierungsstudie generierten Dateien für die weitere Verarbeitung zu speichern. Während der Säulenkonditionierung können die Werte, bei denen sich der Druck stabilisiert, leicht variieren. Stellen Sie jedoch sicher, dass der Druck stabil ist, bevor Sie die Durchflussrate erneut erhöhen, um die Integrität der Säule zu erhalten. Darüber hinaus muss bei Erreichen des Arbeitsdurchflusses auch der Druck stabil sein, um eine reproduzierbare chromatographische Trennung zu erreichen. - Gehen Sie in die obere Symbolleiste der Software und führen Sie den Arbeitsvorrat aus.
5. Datenaufbereitung zur Erstellung einer Quantifizierungsmethode
- Sobald der Arbeitsvorrat ausgeführt ist, öffnen Sie die qualitative Software.
- Laden Sie alle analysierten Proben, indem Sie auf Datei | Arbeitsvorrat öffnen.
- Klicken Sie mit der rechten Maustaste auf eine Probe, die der Zwischenlösung des EAs bei 1.000 ng/ml entspricht, gehen Sie zu Chromatogramm bearbeiten und wählen Sie dann Typ | Extrahiertes Ionenchromatogramm und Eingabe der theoretischen Molekülmasse des protonierten Ions in Bezug auf jedes EA. Klicken Sie dann auf Hinzufügen und schließlich auf OK. Da der Satz von Analyten aus sechs EAs und ihren verwandten Epimeren besteht, die genau die gleiche Masse haben, erscheinen sechs extrahierte Ionenchromatogramme.
HINWEIS: Mit dem Parameter "Breite" wird geändert, wie schmal das m/z-Fenster ist. Es wird empfohlen, ein ausreichend breites m/z-Fenster zu wählen, falls es schwierig ist, den chromatographischen Peak des interessierenden Analyten zu finden. Es ist jedoch ratsam, schmale m/z-Fenster einzustellen, um mögliche Interferenzen zu vermeiden (unter 0,05 Einheiten). - In jedem extrahierten Ionenchromatogramm erscheinen zwei chromatographische Peaks, die sich auf den Haupt-EA und sein Epimer beziehen. Klicken Sie mit der rechten Maustaste, während Sie den gesamten Bereich eines Peaks auswählen. Folglich wird das Ionenmobilitätsspektrum direkt darunter auftauchen.
- Wechseln Sie zur x-Achse des Ionenmobilitätsspektrums, klicken Sie mit der rechten Maustaste, und klicken Sie dann auf Kollisionsquerschnitt. Notieren Sie sich den CCS-Wert.
HINWEIS: Wenn die CCS-Werte nicht automatisch angezeigt werden, gehen Sie in der oberen Symbolleiste zu Kalibrieren , dann zu Mobilitätsrechner und geben Sie den Mobilitätswert manuell ein, um den entsprechenden CCS-Wert zu erhalten. - Wählen Sie den Bereich aus, der dem anderen chromatographischen Peak innerhalb desselben Ionenchromatogramms entspricht, und wiederholen Sie die Schritte 5.4. und 5.5.
- In ähnlicher Weise wird das Fragmentierungsmuster auch nach Schritt 5.4 angezeigt. Auf der Grundlage des in der Literatur und in Datenbanken berichteten Fragmentierungsmusters ist das intensivste Produktionsion als ergänzender Identifikationspunkt zu vergleichen und auszuwählen.
- Notieren Sie die Retentionszeit, den CCS-Wert und die genaue Masse des Hauptionenaddukts (in der Regel das protonierte Ion) jedes EA, um eine Quantifizierungsmethode zu erstellen.
- Wiederholen Sie die Schritte 5.4 bis 5.8. für die übrigen Analyten.
- Wählen Sie alle in Schritt 5.3 erzeugten Molekülmassen aus, indem Sie sie bei gedrückter Strg-Taste anklicken. Klicken Sie dann mit der rechten Maustaste, und wählen Sie Kopieren aus. Wechseln Sie zu dem anderen Beispiel, das der Zwischenlösung des EAs entspricht, klicken Sie mit der rechten Maustaste darauf und wählen Sie Einfügen aus. Die Ionenchromatogramme und Mobilitätsspektren werden automatisch extrahiert.
- Überprüfen Sie die Identifizierungsparameter für alle Analyten in dieser zweiten Probe.
6. Erstellung eines Datenverarbeitungsverfahrens zur routinemäßigen Bestimmung von Mutterkornalkaloiden
- Öffnen Sie die Software und klicken Sie auf die Registerkarte Methodenverwaltung .
- Klicken Sie auf Analyt-Einstellungen und geben Sie den Namen jedes EA zusammen mit seiner Retentionszeit, dem CCS-Wert und der Masse der protonierten Addukte ein, die in Schritt 5.4 gesammelt wurden. bis 5.9. Ändern Sie die Toleranz jedes Identifikationspunkts nach Bedarf, obwohl die Standardeinstellungen für die Anwendung dieses Protokolls empfohlen werden.
HINWEIS: Je höher die eingestellten Toleranzwerte sind, desto höher ist die Wahrscheinlichkeit, dass es zu falsch positiven Ergebnissen kommt, da eine größere Abweichung von den theoretischen Werten akzeptiert wird. Ebenso können zu strenge Toleranzwerte zu falsch negativen Ergebnissen führen. Dies ist der Fall, wenn die Abweichung vom theoretischen Wert ausschließlich durch die Leistung des Instruments verursacht wird. - Speichern Sie die Quantifizierungsmethode zur weiteren Verwendung.
7. Probenahme
- Sammeln Sie Proben von Weizen und Hafer in Supermärkten, vorzugsweise in 1-kg-Behältern, gemäß den von der Europäischen Kommission festgelegten Richtlinien für die Probenahme20. Wenn keine 1-kg-Packungen verfügbar sind, stellen Sie sicher, dass Sie mindestens 50 g Proben haben, um die gesamte Analyse abzuschließen.
- Mahlen Sie die Proben mit einer Mühle. In dieser Studie wurde eine Messermühle verwendet, um den gesamten Inhalt der Verpackungen zu mahlen, die dann zur Homogenisierung in ihre ursprünglichen Verpackungen zurückgeführt wurden.
HINWEIS: Reinigen Sie den Zerkleinerer zwischen den Proben gründlich, um Kreuzkontaminationen zu vermeiden. Verwenden Sie einen Laborkittel. - Aliquotieren Sie die homogenisierten Proben in 50-ml-Zentrifugenröhrchen und lagern Sie sie unter kühlen und trockenen Bedingungen.
8. Vorbereitung der Probe
- Erstellen Sie einen Zeitplan für die Durchführung der Validierungsstudie an drei nicht aufeinanderfolgenden Tagen (um die interne Reproduzierbarkeit zu bewerten). Bestimmen Sie jeden Tag drei Konzentrationsstufen, indem Sie die Proben mit 225 ng/g (1,5 ml), 150 ng/g (ML) und 75 ng/g (0,5 ml) anreichern. Untersuchen Sie jede Ebene in dreifacher Ausfertigung, was neun dotierten Proben plus weiteren drei Blindproben pro Tag entspricht.
HINWEIS: Unter Berücksichtigung der 3 Validierungstage werden insgesamt 36 Proben benötigt, zusammen mit weiteren 8 Leerproben, die für die Erstellung der matrixangepassten Kalibrierkurve bestimmt sind. Verwenden Sie Nitrilhandschuhe, einen Laborkittel und eine Schutzbrille für diesen gesamten Abschnitt des Protokolls. - 1 g der Probe wird in ein 50-ml-Zentrifugenröhrchen gewogen.
HINWEIS: Bei der Durchführung der Validierungsstudie sind die Proben unmittelbar nach dem Wiegen mit 50 μl der entsprechenden Arbeitsstandardlösung gemäß Schritt 1.3 anzureichern. und 1.5. - Fügen Sie 4 mL der Extraktionslösung hinzu [Acetonitril: 5 mM Ammoniumcarbonat wässrige Lösung (85:15, v/v)]. Die Probe 1 Minute lang vortexen.
- Die Probe wird 5 Minuten lang bei 9.750 × g und 4 °C zentrifugiert.
HINWEIS: Die Kapazität der Zentrifuge kann ein limitierender Faktor im Verfahren sein, daher wird empfohlen, gleichzeitig nur die genaue Anzahl von Proben zu extrahieren, die in die Zentrifuge passen. - Der gesamte Überstand wird in das 15-ml-Zentrifugenröhrchen überführt, das 150 mg des Reinigungssorbens (1:1, w/w) enthält. Die Probe 1 Minute lang vortexen.
- Das Röhrchen wird 5 min lang bei 9.750 × g und 4 °C zentrifugiert.
- Sammeln Sie den Überstand und geben Sie ihn in ein 4-ml-Braunglasfläschchen. Verdampfen Sie den Extrakt unter einem sanften Stickstoffstrahl.
HINWEIS: Die Proben müssen bei -20 °C getrocknet werden, wenn sie nicht am selben Tag analysiert werden. - Die Probe wird in 750 μl des Methanol-Wasser-Gemisches (1:1, v/v) rekonstituiert.
- Übertragen Sie den Extrakt in eine 2-ml-Spritze und filtrieren Sie ihn durch einen 0,22-μm-Nylonfilter in ein 1,5-ml-Bernstein-Chromatographiefläschchen.
- Sammeln Sie die rekonstituierten Extrakte der acht Blindproben, die für die matrixangepasste Kalibrierkurve vorgesehen sind, in ein 12-ml-Braunglasfläschchen (jede Probe wird in 750 μl resuspendiert, was einem Gesamtvolumen von 6 mL entspricht).
- 1 ml der Zwischenlösung mit 1.000 ng/ml (83,33 ng/ml für jedes Mutterkornalkaloid) (Schritt 1.2) in ein 2-ml-Braunglasfläschchen pipettieren, das Lösungsmittel unter einem sanften Stickstoffstrahl trocknen und den zuvor entnommenen Blindprobenextrakt in 1 ml resuspendieren.
HINWEIS: Diese Mischung in einer Konzentration von 1.000 ng/ml (83,33 ng/ml für jeden EA) wird verwendet, um die matrixangepasste Kalibrierkurve zu erstellen. - Bereiten Sie zehn 2-ml-Braunglasfläschchen vor und geben Sie jeweils 450 μl Blindprobenextrakt (Schritt 8.10) hinein und markieren Sie sie mit Ziffern von 1 bis 10. Jedes Fläschchen ist ein Punkt der Kalibrierkurve.
HINWEIS: In diesem Protokoll wurden 450 μl verwendet, da dies der niedrigste Wert war, den das LC-Gerät ohne Verwendung von Einsätzen im Inneren des Fläschchens erreichen konnte. Wenn die Nadel nicht so weit in das Fläschchen hineinreicht, vergrößern Sie das Volumen und berechnen Sie daher, wie viele zusätzliche Blindproben entnommen werden sollten, wobei zu berücksichtigen ist, dass jede einzelne in 750 μl Methanol:Wasser-Gemisch (1/1, v:v) resuspendiert wird. - Erstellen Sie die Kalibrierkurve durch serielle Verdünnung. 450 μl der in Schritt 8.11 hergestellten Mischung werden übertragen. in Fläschchen 1, das bereits 450 μl Blindprobenextrakt enthält, um eine Konzentration von 41,67 ng/mL (den höchsten Kalibrierpunkt) zu erreichen. Das Fläschchen 2 Min. lang einreiben.
HINWEIS: Das Volumen der Mischung muss mit dem Volumen des Leerprobenextrakts übereinstimmen, um in jedem Schritt zweimal verdünnt zu werden. - Übertragen Sie 450 μl aus Fläschchen 1 in Fläschchen 2, das bereits 450 μl Blindprobenextrakt enthält, um eine Konzentration von 20,83 ng/ml (den zweithöchsten Kalibrierpunkt) zu erreichen.
- Wiederholen Sie Schritt 8.14 (d. h. von Fläschchen 2 zu Fläschchen 3 usw.) und fahren Sie anschließend fort, bis die zehn Kalibrierpunkte erreicht sind. Der Konzentrationsbereich für jeden EA beträgt dann: 41,67, 20,83, 10,42, 5,21, 2,60, 1,30, 0,65, 0,33, 0,16 und 0,08 ng/ml.
- Eine Standardmischung mit 1.000 ng/ml (83,33 ng/ml für jeden EA) wird wie in Schritt 8.11 beschrieben, jedoch unter Verwendung von 1 ml Methanol:Wasser-Gemisch (1:1, v/v) hergestellt.
- Bereiten Sie eine Standardkalibrierkurve vor, wie in den Schritten 8.13 bis 8.15 beschrieben, jedoch unter Verwendung von 450 μl Methanol:Wasser-Gemisch (1:1, v/v) für die seriellen Verdünnungen. Diese Kalibrierkurve wird verwendet, um den Matrixeffekt zu bewerten, indem sie mit der matrixangepassten Kalibrierkurve verglichen wird.
9. Quantitative Datenaufbereitung
- Initialisieren Sie das Gerät und führen Sie es gemäß den in Schritt 4.1 beschriebenen Anweisungen aus. bis 4.11. In Abbildung 2 finden Sie den Arbeitsvorrat, der in diesem Protokoll verwendet wird.
HINWEIS: Wenn Sie die Arbeitsliste für Kalibrierkurven schreiben, injizieren Sie diese vor den angereicherten Proben, beginnend mit der Standard-Kalibrierkurve von der niedrigsten zur höchsten Konzentration, einem Waschfläschchen mit Methanol:Wasser (1:1, v/v) und der matrixangepassten Kalibrierkurve von der niedrigsten zur höchsten Konzentration. - Öffnen Sie die quantitative Software und navigieren Sie zum TASQ-Quick-Tab | Stapel importieren unten links.
- Wählen Sie den Ordner aus, der in Schritt 4.10 erstellt wurde. mit allen Rohdaten (Dateien, die sich auf die Informationen beziehen, die nach der LC-IMS-MS-Analyse für alle in der Arbeitsliste enthaltenen Proben erfasst wurden).
- Gehen Sie nach dem Import in die Software zu Stapel verarbeiten, direkt unter Stapel importieren.
- Wählen Sie die zu verarbeitenden Proben (d. h. die 27 dotierten Proben, 9 Leerproben und beide Kalibrierungskurven) und die Datenverarbeitungsmethode, die zuvor in Schritt 6.1 erstellt wurde. bis 6.3. Es öffnet sich ein Fenster mit verschiedenen Parametern, die eingestellt werden können (z. B. die Art des Integrators zur Bewertung der chromatographischen Peaks oder die Art des zu berücksichtigenden analytischen Signals). Ändern Sie diese Parameter wie gewünscht, obwohl die Standardwerte für die Ausführung dieses Protokolls geeignet sind.
- Gehen Sie nach Abschluss der Datenverarbeitung zu Screening überprüfen und prüfen Sie, ob es Probleme im Zusammenhang mit der automatischen Integration gibt, z. B. die Auswahl falscher chromatographischer Peaks, die möglicherweise zu nahe beieinander liegen. Wenn dies der Fall ist, schränken Sie die Werte ein, die sich auf die Toleranz der Verweildauer, die Toleranz der CCS-Werte und/oder den Massenfehler beziehen, die ursprünglich in der Datei mit der quantitativen Methode festgelegt wurden, und verarbeiten Sie die Daten erneut. Wenn die automatische Peakintegration immer noch falsche Peaks auswählt, integrieren Sie den Peak manuell, indem Sie den Bereich im Chromatogramm auswählen, der im Fenster "Screening überprüfen " angezeigt wird (berücksichtigen Sie dazu die analytischen Parameter, die für die EA-Standards in Abschnitt 5 erhalten wurden).
HINWEIS: Es ist sehr wichtig, jede Probe zu überprüfen, da einige Haupt-EA-Epimer-Paare sehr ähnliche Retentionszeiten haben und von der Software falsch identifiziert werden können. - Wenn alle Peaks für alle Proben erfolgreich integriert wurden, klicken Sie auf die Registerkarte Chargenverwaltung in der oberen linken Ecke und geben Sie die Art der Probe an, die mit jeder Analyse verbunden ist: Lösungsmittel, Leerprobe, Qualitätskontrolle, Probe oder Kalibrierpunkt. Im letzteren Fall weisen Sie jedem Kalibrierungspunkt eine Ebene mit einer Zahl zu.
- Navigieren Sie auf derselben Registerkarte "Chargenverwaltung " zum unteren Fenster mit dem Namen "Chargenkonzentration" und geben Sie einen Konzentrationswert für jede im vorherigen Schritt festgelegte Stufe an. Sobald dies erledigt ist, klicken Sie auf Konzentrationen speichern.
- Klicken Sie auf Batch quantifizieren und ändern Sie die Parameter wie gewünscht (d. h. das Modell, das an die Kalibrierungsdaten angepasst werden soll, die Gewichtung, ob das Modell gezwungen ist, Null zu überschreiten usw.). Für die Ausführung dieses Protokolls eignen sich die Standardwerte, mit Ausnahme der Gewichtung der Kalibrierkurven, die auf 1/x umgestellt wurde.
HINWEIS: Da die Software jeweils nur eine Kalibrierkurve berücksichtigen kann, ist es notwendig, Schritt 9.9 zweimal zu wiederholen, um Informationen über Standard- und Matrix-angepasste Kalibrierkurven zu erhalten. In diesem Protokoll wurden matrixangepasste Kalibrierkurven für die Quantifizierung von Analyten verwendet, so dass zuerst die Lösungsmittel-Kalibrierkurven bewertet, Kalibrierdaten extrahiert wurden, wie später in Schritt 9.13 erläutert, und schließlich die matrixangepassten Kalibrierkurven ausgewählt wurden, um die Quantifizierung der Proben durchzuführen. - Sobald die Quantifizierung der Mutterkornalkaloide gemäß der Datei mit der Quantifizierungsmethode durchgeführt wurde, klicken Sie auf die Registerkarte Quantifizierung im oberen Teil des Bildschirms.
- Wenn die in Schritt 9.9 eingestellten Standardparameter keine gute Linearität und Genauigkeit bieten, passen Sie die Kalibrierkurven an, indem Sie die Anpassung und das Gewicht ändern, indem Sie mit der rechten Maustaste auf das Diagramm klicken, mit dem Ziel, ein gutes lineares Modell (R2 > 0,99) mit ausreichender Genauigkeit auf jeder Kalibrierstufe (Abweichung unter 20%) zu erhalten.
HINWEIS: Die Software markiert die Zellen der Kalibrierungsstufen, die nicht die richtige Genauigkeit bieten, rot. - Wenn Sie fertig sind, klicken Sie mit der rechten Maustaste auf den Batch-Ordner ganz links auf dem Bildschirm und wählen Sie Bericht generieren. Wählen Sie dann Quantifizierung analysieren , um den Bericht in das gewünschte Format zu exportieren.
Ergebnisse
Zunächst wurden funktionierende Standardlösungen in das LC-IMS-MS-Instrument injiziert, um alle Identifikationsmerkmale (d.h. Retentionszeit, CCS und Massenspektren) jedes hier analysierten EA zu erhalten. Da die Identifikationsparameter, mit Ausnahme der genauen Masse, zunächst unbekannt waren, basierte die Erfassungsmethode auf einem Two-Scan-Ereignis, beginnend mit einem vollständigen Scan des gesamten Massenspektrums, gefolgt von einer bbCID. Die retrospektive Herangehensweise an...
Diskussion
Die erfolgreiche Anwendung dieses Protokolls basiert auf der Optimierung des Extraktionsverfahrens, die zuvor von Carbonell-Rozas et al.17 durchgeführt wurde, die die Verwendung eines Extraktionslösungsmittels implementierten, das wirksam genug ist, um EAs aus komplexen Lebensmittelmatrizes wie Gerste und Weizen zu extrahieren, und einer Reinigung, die relativ niedrige SSE-Werte lieferte. Die Wahl des Extraktionslösungsmittels stellt einen kritischen Schritt da...
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Diese Forschung wurde von der Consejería de Universidad, Investigación e Innovación - Junta de Andalucía (PROYEXCEL_00195) und dem Postdoktorandenstipendium der Generalitat Valenciana und des Europäischen Sozialfonds+ (CIAPOS/2022/049) finanziert. Die Autoren danken dem "Centro de Instrumentación Científica (CIC)" an der Universität Granada für den Zugang zu den in diesem Protokoll verwendeten analytischen Instrumenten.
Materialien
| Name | Company | Catalog Number | Comments |
| Acetonitrile | VWR | 83640.32 | |
| Amber glass tubes 4 mL | VWR | 548-0052 | |
| Amber glass tubes 12 mL | VWR | 548-0903 | |
| Amber vials 1.5 mL | Agilent | 5190-9063 | |
| Ammonium carbonate | Fluka | 9716 | |
| Analytical balance BAS 31 | Boeco | 4400519 | |
| Balance CP 323 S | Sartorius | 23-84182 | |
| C18 | Supelco | 52604-U | |
| Centrifuge tubes, 15 mL | VWR | 525-1082 | |
| Centrifuge tubes, 50 mL | VWR | 525-0155 | |
| Centrifuge Universal 320 R | Hettich | 1406 | |
| Compass HyStar | Bruker | Acquisition software | |
| DataAnalysis | Bruker | Qualitative software | |
| Elute PLUS UHPLC | Bruker | ||
| EVA EC-S evaporator | VLM | V830.012.12 | |
| Formic acid GR for analysis ACS, Reag. Ph Eur | Merck | 100264 | |
| Grinder TitanMill300 | Cecotec | 1559 | |
| Methanol | VWR | 83638.32 | |
| Milli-Q water purification system (18.2 MΩ cm) | Millipore | ZD5211584 | |
| Pipette tips 1- 5 mL | Labortecnic | 162005 | |
| Pipette tips 100 - 1000 µL | Labortecnic | 1622222 | |
| Pipette tips 5 - 200 µL | Labortecnic | 162001 | |
| Pippette Transferpette S variable, DE-M 10 - 100 µL | BRAND | 704774 | |
| Pippette Transferpette S variable, DE-M 100 - 1000 µL | BRAND | 704780 | |
| Pippette Transferpette S variable, DE-M 500 - 5000 µL | BRAND | 704782 | |
| Syringe 2 mL | VWR | 613-2003 | |
| Syringe Filter 13 mm, 0.22µm | Phenomenex | AF-8-7707-12 | |
| TASQ | Bruker | Quantitative software | |
| timsTOFPro2 IM-HRMS | Bruker | ||
| Vortex Genie 2 | Scientific Industries | 15547335 | |
| Zorbax Eclipse Plus RRHD C18 column (50 x 2.1 mm, 1.8 µm particle size) | Agilent | 959757-902 | |
| Z-Sep+ | Supelco | 55299-U | Zirconia-based sorbent |
| Ergot alkaloids | CAS registry sorbent | ||
| Ergocornine (Eco) | Techno Spec | E178 | 564-36-3 |
| Ergocorninine (Econ) | Techno Spec | E130 | 564-37-4 |
| Ergocristine (Ecr) | Techno Spec | E180 | 511-08-0 |
| Ergocristinine (Ecrn) | Techno Spec | E188 | 511-07-9 |
| Ergokryptine (Ekr) | Techno Spec | E198 | 511-09-1 |
| Ergopkryptinine (Ekrn) | Techno Spec | E190 | 511-10-4 |
| Ergometrine (Em) | Romer Labs | "002067" | 60-79-7 |
| Ergometrinine (Emn) | Romer Labs | LMY-090-5ML | 479-00-5 |
| Ergosine (Es) | Techno Spec | E184 | 561-94-4 |
| Ergosinine (Esn) | Techno Spec | E194 | 596-88-3 |
| Ergotamine (Et) | Romer Labs | "002069" | 113-15-5 |
| Ergotaminine (Etn) | Romer Labs | "002075" | 639-81-6 |
Referenzen
- Kanu, A. B., Dwivedi, P., Tam, M., Matz, L., Hill, H. H. Ion mobility-mass spectrometry. J Mass Spectrom. 43 (1), 1-22 (2008).
- Gabelica, V., et al. Recommendations for reporting ion mobility Mass spectrometry measurements. Mass Spectrom Rev. 38 (3), 291-320 (2019).
- Feuerstein, M. L., et al. Comparability of steroid collision cross sections using three different IM-HRMS technologies: An interplatform study. J Am Soc Mass Spectrom. 33 (10), 1951-1959 (2022).
- Regueiro, J., Negreira, N., Berntssen, M. H. G. Ion-mobility-derived collision cross section as an additional identification point for multiresidue screening of pesticides in fish feed. Anal Chem. 88 (22), 11169-11177 (2016).
- Regueiro, J., Negreira, N., Hannisdal, R., Berntssen, M. H. G. Targeted approach for qualitative screening of pesticides in salmon feed by liquid chromatography coupled to traveling-wave ion mobility/quadrupole time-of-flight mass spectrometry. Food Control. 78, 116-125 (2017).
- Hernández-Mesa, M., Monteau, F., Le Bizec, B., Dervilly-Pinel, G. Potential of ion mobility-mass spectrometry for both targeted and non-targeted analysis of phase II steroid metabolites in urine. Anal Chim Acta: X. 1, 100006 (2019).
- George, A. C., et al. Interplatform comparison between three ion mobility techniques for human plasma lipid collision cross sections. Anal Chim Acta. 1304, 342535 (2024).
- Mairinger, T., Causon, T. J., Hann, S. The potential of ion mobility-mass spectrometry for non-targeted metabolomics. Curr Opin Chem Biol. 42, 9-15 (2018).
- Hernández-Mesa, M., Escourrou, A., Monteau, F., Le Bizec, B., Dervilly-Pinel, G. Current applications and perspectives of ion mobility spectrometry to answer chemical food safety issues. TrAC Trend Anal Chem. 94, 39-53 (2017).
- European, C. Commission Regulation (EU) 2023/915 of 25 April 2023 on maximum levels for certain contaminants in food and repealing Regulation (EC) No 1881/2006. Off J Eur Union. 119, 103-157 (2023).
- Carbonell-Rozas, L., et al. Occurrence of egot alkaloids in major and minor cereals from Northern Italy: A three harvesting years scenario. J Agricl Food Chem. 71 (42), 15821-15828 (2023).
- Arroyo-Manzanares, N., Gámiz-Gracia, L., García-Campaña, A. M., Diana Di Mavungu, J., De Saeger, S., Jean-Michel Mérillon, J. -. M., Ramawat, K. G. . Fungal Metabolites. , (2016).
- van Dongen, P., de Groot, A. History of ergot alkaloids from ergotism to ergometrine. Eur J Obstet Gynecol Reprod Biol. 60 (2), 109-116 (1995).
- European Food Safety Authority. Human and animal dietary exposure to ergot alkaloids. EFSA J. 15 (7), e04902 (2017).
- Nam, M., Kim, D., Kim, M. -. S. Simultaneous determination of total ergot alkaloids in wheat flour by Orbitrap mass spectrometry. Food Chem. 441, 138363 (2024).
- García-Juan, A., León, N., Armenta, S., Pardo, O. Development and validation of an analytical method for the simultaneous determination of 12 ergot, 2 tropane, and 28 pyrrolizidine alkaloids in cereal-based food by LC-MS/MS. Food Res Int. 174 (Pt 1), 113614 (2023).
- Carbonell-Rozas, L., Mahdjoubi, C. K., Arroyo-Manzanares, N., García-Campaña, A. M., Gámiz-Gracia, L. Occurrence of ergot alkaloids in barley and wheat from Algeria. Toxins. 13 (5), 316 (2021).
- Carbonell-Rozas, L., Gámiz-Gracia, L., Lara, F. J., García-Campaña, A. M. Determination of the main ergot alkaloids and their epimers in oat-based functional foods by ultra-high performance liquid chromatography tandem mass spectrometry. Molecules. 26 (12), 3717 (2021).
- Carbonell-Rozas, L., et al. Ion mobility-mass spectrometry to extend analytical performance in the determination of ergot alkaloids in cereal samples. J Chromatogr A. 1682, 463502 (2022).
- European Commission. Commission Implementing Regulation (EU) 2023/2782 of 14 December 2023 laying down the methods of sampling and analysis for the control of the levels of mycotoxins in food and repealing Regulation (EC) No 401/2006. Off J Eur Union. , (2023).
- Carbonell-Rozas, L., Vander Cruyssen, L., Dall'Asta, C., Leggieri, M. C., Battilani, P. Fit-for-purpose method development to determine co-occurring multiclass mycotoxins in apple and apple puree samples. Food Anal Methods. 16 (8), 1403-1412 (2023).
- Laouni, C., et al. Emerging mycotoxin occurrence in chicken feed and eggs from Algeria. Mycotoxin Res. 40, 447-456 (2024).
- Ben Hassouna, K., et al. Mycotoxin occurrence in milk and durum wheat samples from Tunisia using dispersive liquid-liquid microextraction and liquid chromatography with fluorescence detection. Toxins. 15 (11), 633 (2023).
- Narváez, A., et al. Occurrence and exposure assessment of mycotoxins in ready-to-eat tree nut products through ultra-high performance liquid chromatography coupled with high resolution Q-orbitrap mass spectrometry. Metabolites. 10 (9), 344 (2020).
- Arroyo-Manzanares, N., Rodríguez-Estévez, V., García-Campaña, A. M., Castellón-Rendón, E., Gámiz-Gracia, L. Determination of principal ergot alkaloids in swine feeding. J Sci Food Agric. 101 (12), 5214-5224 (2021).
- Pereira, V. L., Fernandes, J. O., Cunha, S. C. Comparative assessment of three cleanup procedures after QuEChERS extraction for determination of trichothecenes (type A and type B) in processed cereal-based baby foods by GC-MS. Food Chem. 182, 143-149 (2015).
- Tuzimski, T., Szubartowski, S. Method development for selected bisphenols analysis in sweetened condensed milk from a can and breast milk samples by HPLC-DAD and HPLC-QqQ-MS: Comparison of sorbents (Z-SEP, Z-SEP Plus, PSA, C18, chitin and EMR-lipid) for clean-up of QuEChERS extract. Molecules. 24 (11), 2093 (2019).
- Łozowicka, B., Rutkowska, E., Jankowska, M. Influence of QuEChERS modifications on recovery and matrix effect during the multi-residue pesticide analysis in soil by GC/MS/MS and GC/ECD/NPD. Environ Sci Pollut Res. 24 (8), 7124-7138 (2017).
- Schummer, C., Xandonella, I., van Nieuwenhuyse, A., Moris, G. Epimerization of ergot alkaloids in feed. Heliyon. 6 (6), e04336 (2020).
- Cherewyk, J. E., Grusie-Ogilvie, T. J., Parker, S. E., Blakley, B. R., Al-Dissi, A. M. The impact of storage temperature and time on ergot alkaloid concentrations. Toxins. 15 (8), 497 (2023).
- Silva, &. #. 1. 9. 4. ;., Mateus, A. R., Barros, S. C., Silva, A. S. Ergot alkaloids on cereals and seeds: Analytical methods, occurrence, and future perspectives. Molecules. 28 (20), 7233 (2023).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten