É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Detecção de Alcalóides Ergot Regulados em Matrizes de Alimentos por Cromatografia Líquida - Espectrometria de Mobilidade Iônica Aprisionada - Espectrometria de Massa por Tempo de Voo
Neste Artigo
Resumo
Este protocolo apresenta um método validado de cromatografia líquida-mobilidade iónica e espectrometria de massa de alta resolução para determinar a presença de alcaloides da cravagem nos alimentos, em conformidade com o Regulamento (UE) 2023/915 da Comissão, recentemente publicado.
Resumo
A espectrometria de massa de mobilidade iônica (IMS) atua como uma dimensão de separação adicional quando integrada aos fluxos de trabalho de cromatografia líquida-espectrometria de massa (LC-MS). Os métodos LC-IMS-MS fornecem maior resolução de pico, separação aprimorada de compostos isobáricos e isoméricos e melhor relação sinal-ruído (S/N) em comparação com os métodos tradicionais de LC-MS. O IMS fornece outra característica molecular para a identificação de analitos, ou seja, o parâmetro de seção transversal de colisão (CCS), reduzindo os resultados falsos positivos. Portanto, os métodos LC-IMS-MS abordam importantes desafios analíticos no campo da segurança alimentar (ou seja, detecção de compostos em níveis de traços em matrizes alimentares complexas e identificação inequívoca de moléculas isobáricas e isoméricas).
Os alcalóides da cravagem (EAs) são uma família de micotoxinas produzidas por fungos que atacam uma ampla variedade de espécies de gramíneas, incluindo pequenos grãos como centeio, triticale, trigo, cevada, painço e aveia. Foram estabelecidos teores máximos (ML) destas micotoxinas em vários géneros alimentícios, tal como especificado no Regulamento (CE/2023/915) da Comissão. Esta nova legislação inclui seis EAs principais e seus epímeros correspondentes, portanto, é necessária uma metodologia eficiente para distinguir adequadamente essas moléculas isoméricas considerando sua co-ocorrência.
Portanto, o objetivo deste protocolo é mostrar como a integração do IMS em fluxos de trabalho LC-MS contribui para a separação de EAs isoméricos, aumentando a seletividade do método analítico. Além disso, ilustra como a geração de bibliotecas de CCS por meio da caracterização de padrões analíticos fornece maior confiança para a identificação de micotoxinas. Este protocolo foi concebido para explicar claramente os benefícios da implementação do SGI na segurança alimentar, tomando como exemplo a determinação de EAs em cereais. Uma extração baseada em QuEChERS seguida por uma análise de espectrometria de mobilidade iônica com aprisionamento por LC (TIMS)-MS forneceu limites de quantificação variando de 0,65 a 2,6 ng/g com precisão aceitável (embora baixa recuperação para ergotaminina) em 1,5x, 1x e 0,5x o ML e exibiu um efeito de matriz insignificante.
Introdução
A espectrometria de massa de mobilidade iônica (IMS) está se tornando uma técnica analítica cada vez mais usada, muitas vezes apresentada como uma dimensão de separação adicional integrada à cromatografia líquida/gasosa tradicional (LC/GC) acoplada a fluxos de trabalho de MS. O IMS consiste na separação de moléculas ao longo de uma célula de mobilidade, preenchida com um gás tampão, sob um campo elétrico e à pressão atmosférica1. Dependendo da relação massa-carga (m/z) e da conformação geométrica, uma molécula ionizada interagirá com o gás tampão à medida que se move pela célula de mobilidade, o que é refletido no parâmetro de mobilidade iônica (K)2 e calculado através da seguinte equação:
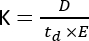
onde D representa o comprimento total da deriva, td é o tempo total da deriva e E é o campo elétrico. Portanto, K é medido em m2 V−1 s−1, embora por razões práticas seja frequentemente expresso como cm2 V−1 s−1. A capacidade intrínseca de se mover através da célula de mobilidade pode ser medida pelo tempo de deriva e posteriormente convertida para o chamado valor da seção transversal de colisão (CCS), que é um parâmetro altamente reprodutível para cada molécula, independentemente do instrumento IMS3. O CCS pode ser derivado da mobilidade seguindo esta equação:
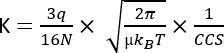
q sendo a carga do íon; N a densidade do número de gases tampão; μ a massa reduzida dos parceiros de colisão tampão de íons de gás; kB a constante de Boltzmann; e T a temperatura do gás tampão. Portanto, o IMS fornece informações adicionais complementares aos dados analíticos resultantes das análises de cromatografia e MS.
A implementação do IMS em plataformas LC-MS demonstrou aumentar a confiabilidade das determinações analíticas, especialmente ao trabalhar com compostos que estão em concentrações vestigiais. Vários estudos relataram que os métodos LC-IMS-MS melhoram a qualidade dos espectros de massa, reduzindo o ruído de fundo, o que afeta a sensibilidade do método, e reduz a taxa de falsos positivos e negativos fornecidos pelas metodologias LC-MS multirresíduos 4,5,6. Além disso, a reprodutibilidade dos valores de CCS permite a comparação não apenas entre diferentes instrumentos usando a mesma tecnologia, mas também entre diferentes tecnologias de mobilidade iônica, a saber, espectrometria de mobilidade iônica de onda viajante (TWIMS), espectrometria de mobilidade iônica de aprisionamento (TIMS) e espectrometria de mobilidade iônica de tubo de deriva (DTIMS)2,7, que são os sistemas mais usados1. Assim, uma consequência marcante do potencial do CCS como parâmetro de identificação reside na possibilidade de construção de bibliotecas de CCS, refletida em sua aplicabilidade em estudos metabolômicos8. No entanto, uma das características mais poderosas do IMS é a capacidade de separar compostos isoméricos e isobáricos que podem não ser suficientemente resolvidos por métodos LC-MS. Este pode ser o caso ao trabalhar com grandes conjuntos de analitos de interesse em matrizes complexas, o que é uma situação comum em análises ambientais e de alimentos. Nesse contexto, métodos LC-IMS-MS têm sido propostos para o monitoramento de pesticidas e, em menor grau, medicamentos veterinários e micotoxinas em alimentos9.
Devido ao seu alto poder de resolução e seletividade, as plataformas LC/GC-IMS-MS surgem como as ferramentas mais úteis para enfrentar alguns dos desafios atuais em segurança alimentar, especialmente aqueles relacionados a misturas isoméricas. A preocupação com a saúde relacionada com as misturas isoméricas como contaminantes alimentares reflectiu-se na actual legislação europeia, que, por exemplo, limita a concentração máxima de seis principais alcaloides da cravagem (EA) e dos seis epímeros correspondentes em vários produtos alimentares10.
Os EAs constituem uma família de metabólitos secundários tóxicos produzidos por uma ampla gama de fungos, principalmente da família Clavicipitaceae (por exemplo, Claviceps purpurea, o produtor de EA mais importante devido à sua ampla gama de hospedeiros), mas também Trichocomaceae, que podem parasitar a cabeça da semente de plantas vivas (como centeio, cevada, trigo e aveia) na época da floração11, 12. Sob condições específicas, especialmente temperatura e atividade de água, os fungos Claviceps podem produzir EAs que se acumulam em corpos frutíferos, conhecidos como escleródios ou ergot, na cultura hospedeira. Até certo ponto, os EAs podem suportar o processamento da matéria-prima até chegar ao produto final; portanto, invadindo a cadeia alimentar. A ingestão de alimentos contaminados pode levar à intoxicação por EA, conhecida como ergotismo, que se apresenta com sintomas agudos, como dor abdominal, vômitos, sensação de queimação na pele, insônia e alucinações13. Para reduzir o impacto das EA na saúde humana, a Comissão Europeia fixou teores máximos (ML) em vários alimentos para a soma das principais EAs: os epímeros R ergometrina (Em), ergotamina (Et), ergosina (Es), ergocristina (Ecr), ergocriptina (Ekr) e ergocornina (Eco) e seus epímeros S correspondentes: ergometrina (Emn), ergosinina (Esn), ergotamina (Etn), ergocorninina (Econ), ergocriptinina (Ekrn), e ergocristinina (Ecrn). Esses compostos podem epimerizar das formas R a S e vice-versa, especialmente sob exposição a luz forte, armazenamento prolongado ou contato com alguns solventes em pH alto ou baixo 12. Embora a proporção de formas R e S possa variar em diferentes condições, o Painel CONTAM da EFSA relatou uma maior ocorrência de formas R do que de formas S após revisão da literatura disponível sobre EAs em produtos alimentícios14. Portanto, os MLs variam dependendo de vários fatores, como a suscetibilidade da cultura, o grau de processamento ou a frequência de consumo. No quadro da UE, os ML para os produtos moídos de cevada, trigo, espelta e aveia foram fixados em 50 ou 150 μg/kg (dependendo do teor de cinzas inferior ou superior a 900 mg/100 g, respetivamente), enquanto os cereais destinados diretamente ao consumo humano estão sujeitos a um ML de 150 μg/kg, com exceção dos alimentos para bebés à base de cereais, em que o ML é reduzido para 20 μg/kg10.
Esta legislação rigorosa requer metodologias analíticas sensíveis o suficiente para determinar os níveis de concentração de traços (μg/kg), identificando adequadamente os EAs regulamentados e seus epímeros correspondentes, já que ambas as formas, isômeros R e S, podem ser encontradas juntas em amostras contaminadas. Essa tarefa representa um grande desafio, pois cada par toxina-epímero compartilha exatamente o mesmo padrão de massa e fragmentação. Além disso, uma separação cromatográfica adequada entre os dois compostos pode ser complexa. Portanto, gradientes de LC bem otimizados são necessários para evitar quantificação incorreta quando os epímeros de EA co-ocorrem em amostras de alimentos. Embora vários estudos tenham relatado métodos de LC-MS para determinação inequívoca de EAs 15,16,17,18, o método cromatográfico deve ser estudado extensivamente para obter a separação adequada dos picos cromatográficos para identificar inequivocamente os EAs. No entanto, isso geralmente não é viável para métodos multiclasse nos quais contaminantes pertencentes a diferentes famílias químicas são determinados simultaneamente. Nesse contexto, um estudo recente conduzido por Carbonell-Rozas, Hernández-Mesa, et al.19 relatou um método LC-IMS-MS para a quantificação de EAs em amostras de trigo e cevada, usando dois instrumentos TWIMS diferentes que forneceram valores de CCS reprodutíveis e baixos limites de quantificação (LOQs) para detectar qualquer não conformidade de acordo com a legislação vigente. Portanto, o objetivo deste protocolo é mostrar como a integração do IMS em fluxos de trabalho LC-MS contribui para a separação de EAs isoméricos, aumentando a seletividade do método analítico. Além disso, ilustra como a geração de bibliotecas de CCS por meio da caracterização de padrões analíticos fornece maior confiança para a identificação de micotoxinas. Este protocolo foi elaborado para explicar claramente os benefícios da implementação do SGI na análise de segurança alimentar, tomando como exemplo a determinação de EAs em cereais. Este protocolo aborda o tratamento da amostra com base em um procedimento QuEChERS, análise de amostra por LC-TIMS-MS e extração e interpretação de dados IMS.
Protocolo
1. Preparação de soluções padrão, intermediárias e de trabalho
NOTA: Use luvas de nitrilo, jaleco e óculos de segurança.
- Prepare soluções de estoque individuais dos 12 EAs (consulte a Tabela de Materiais) a 10.000 ng / mL em frascos de vidro âmbar de 4 mL usando acetonitrila. As formas R (-ine) foram previamente aliquotadas em porções de 25.000 ng, enquanto as formas S (-inine) foram distribuídas em porções de 10.000 ng. Este estudo iniciou-se com essas alíquotas, atingindo concentrações de 10.000 ng/mL diluindo as formas R e S em 2,5 e 1 mL de acetonitrila, respectivamente, seguidas de vórtice por 2 min.
- Prepare uma solução de estoque intermediária a 1.000 ng / mL de EAs totais (83,33 ng / mL de cada) em um frasco de vidro âmbar de 12 mL, adicionando 65 μL de cada solução alcalóide individual e 7,02 mL de acetonitrila.
NOTA: Uma vez preparada a solução-mãe intermédia, as soluções-mãe individuais devem ser secas e armazenadas a -20 °C cobertas com folha de alumínio para evitar a epimerização. - Prepare as soluções-padrão de trabalho a partir da solução de estoque intermediária pipetando 2,7, 1,8 e 0,9 mL em tubos de vidro âmbar de 4 mL. Leve-os à secura sob um fluxo suave de nitrogênio e ressuspenda em 600 μL de acetonitrila.
NOTA: As concentrações resultantes foram estabelecidas para atingir 1,5, 1 e 0,5x o mL (150 ng/g) ao adicionar 50 μL a 1 g da amostra. Estes são selecionados como níveis de concentração para estudos de validação (ou seja, recuperações, repetibilidade, etc.). - Divida os 600 μL de cada solução-padrão de trabalho em três frascos de vidro âmbar de 2 mL pipetando 200 μL e leve-os até a secura.
NOTA: As etapas 1.2 a 1.4 são explicadas esquematicamente na Figura 1. Ao dividir e secar as soluções-padrão de trabalho, é possível fortificar em triplicado em três dias diferentes, conforme exigido pelo estudo de validação, sem perder a integridade química dos EAs. - Em cada dia de validação, ressuspenda um frasco de vidro âmbar de 2 mL de cada nível de fortificação em 200 μL de acetonitrila.
NOTA: Cada nível de fortificação será avaliado em triplicata usando 50 μL por amostra; portanto, um total de 150 μL da solução será usado.
2. Preparação de reagentes e soluções
NOTA: Use luvas de nitrilo, jaleco e óculos de segurança.
- Prepare uma solução de carbonato de amônio 5 mM pesando 24,02 mg de carbonato de amônio e dissolvendo-a em 50 mL de água. Pesar 24,02 mg de carbonato de amônio em um béquer e adicionar aproximadamente 5 mL de água. Dissolver o carbonato de amónio agitando suavemente a solução manualmente e, em seguida, transferi-lo para um balão volumétrico de 50 ml. Para atingir o volume final de 50 ml, lavar o copo com água adicional e transferir a subida para o balão volumétrico.
- Preparar 250 ml da solução de extracção [acetonitrila:5 mM de carbonato de amónio (85:15, v/v)] misturando 212,5 ml de acetonitrilo e 37,5 ml da solução de amónio preparada no passo 2.1.
- Preparar 100 ml de uma mistura metanol:água (1:1, v/v) para ressuspender os extractos da amostra (passo 8.8).
- Para cada amostra, pesar 150 mg de um material dispersivo em fase sólida C18:mistura de adsorventes à base de zircónia (1:1, p/p) em tubos de centrífuga de 15 ml para a fase de limpeza dispersiva (passo 8.5).
- Preparar os solventes para separação cromatográfica em dois frascos diferentes, a saber, 1 L de água ultrapura (solvente A) e metanol (solvente B), ambos contendo 0,3% (v/v) de ácido fórmico. Para atingir essa concentração, 3,03 mL de ácido fórmico (99% de pureza) foram adicionados aos frascos.
NOTA: Os solventes para a fase móvel devem ser preparados imediatamente antes de executar as análises LC-IMS-MS.
3. Configurando parâmetros instrumentais
NOTA: O instrumento usado para realizar este estudo LC-IMS-MS foi um UHPLC acoplado a um IM-HRMS, equipado com uma fonte de ionização por eletrospray aquecida por sonda isolada a vácuo (VIP-HESI). O instrumento foi operado em modo positivo.
- Criar um método cromatográfico utilizando o software de aquisição para a separação dos analitos, definindo o seguinte gradiente de eluição: 0 min, 10% B; 2 min, 10% B; 4,5 min, 40% B; 9 min, 45% B; 11 min, 95% B; 12 min, 95% B; 13 min, 10% B; 16 min, 10% B.
- Defina a vazão da fase móvel em 0,4 mL/min, a temperatura da coluna em 35 °C e o volume de injeção em 5 μL. Se possível, mantenha uma temperatura fixa de 10 °C dentro do amostrador automático.
- Defina os parâmetros IMS da seguinte forma: valores de mobilidade (1/K0), de 0,1 a 1,5 V·s/cm2; tempo de rampa, 100 ms; tempo de precisão, 10 ms; ciclo de trabalho, 10%; e taxa de rampa, 9,05 Hz.
- Defina os parâmetros da fonte de íons para detecção de MS da seguinte forma: tensão capilar, 2.500 V; pressão do nebulizador, 2,5 bar; vazão de gás seco, 8 L/min; temperatura do gás seco, 200 °C; temperatura do gás da bainha, 450 °C; vazão de gás da bainha, 4 L/min. Para aquisição de MS, selecione os seguintes parâmetros, levando em consideração dois eventos de varredura: varredura completa de todo o espectro de massa com um m/z de 20 a 1.300 seguido por bbCID (dissociação induzida por colisão de banda larga), que gera íons de produto de qualquer precursor em três energias de colisão fixas de 24, 36 e 50 eV. Realize ionização e aquisição de dados no modo de polaridade positiva.
- Salve este arquivo de método de aquisição para uso posterior.
4. Aquisição de dados a partir de padrões analíticos de EAs
NOTA: Use luvas de nitrilo, jaleco de laboratório e óculos de segurança apenas para a etapa 4.1.
- Prepare dois frascos âmbar de 1,5 mL com 500 μL de solução intermediária a 1.000 ng / mL e outro frasco com uma mistura de solventes A e B (1: 1, v / v) como solução de lavagem.
- Calibre o instrumento timsTOF conforme indicado pelo fabricante e selecione a polaridade positiva para aquisições IMS e MS.
- Coloque a coluna C18 (consulte Tabela de Materiais) dentro do forno LC, com a temperatura ajustada para 35 °C conforme indicado no método de aquisição file salvo na etapa 3.5.
NOTA: Certifique-se de que ambas as extremidades estejam bem apertadas; caso contrário, vazamento de fase móvel será observado. - Condicione a coluna LC para permitir uma taxa de fluxo de fase móvel de 0.1 mL/min e selecione uma proporção de mistura de solvente A e B 90:10 (%) como a proporção inicial de composição da fase móvel.
- Aguarde até que a pressão da coluna esteja estável (~130 bar).
- Aumente a taxa de fluxo da fase móvel para 0.2 mL/min, espere até que a pressão se estabilize e aumente novamente para 0.3 mL/min.
- Assim que a pressão se estabilizar novamente, defina a vazão para 0.4 mL, que será a vazão de trabalho conforme detalhado no método de aquisição file salvo na etapa 3.5, e aguarde até que a pressão se estabilize em torno de 350 bar.
NOTA: É importante não aumentar diretamente o fluxo de 0,2 para 0,4 mL/min. Uma forte mudança no fluxo provoca picos de alta pressão que podem prejudicar a coluna de cromatografia. - Enquanto a coluna está sendo condicionada, escreva a lista de trabalho, incluindo duas amostras correspondentes aos dois frascos contendo a solução intermediária dos EAs a 1.000 ng / mL, conforme indicado na etapa 4.1, e o frasco de lavagem. Analise o frasco de lavagem duas vezes, no início e no final do lote de amostra.
- Na coluna Arquivo de método da lista de trabalho, carregar o arquivo de método de aquisição gravado anteriormente na etapa 3.5.
- Crie uma pasta para salvar todos os arquivos de dados adquiridos e rotear todas as amostras selecionando a pasta na coluna Caminho da amostra .
NOTA: Certifique-se de usar a mesma pasta para salvar todos os arquivos gerados durante o estudo de validação para processamento posterior. Durante o condicionamento da coluna, os valores nos quais a pressão se estabiliza podem variar ligeiramente. No entanto, certifique-se de que a pressão esteja estável antes de aumentar a vazão novamente para preservar a integridade da coluna. Além disso, quando a vazão de trabalho é atingida, a pressão também deve ser estável para obter uma separação cromatográfica reprodutível. - Vá para a barra de ferramentas superior do software e execute a lista de trabalho.
5. Tratamento de dados para a criação de um método de quantificação
- Depois que a lista de trabalho for executada, abra o software qualitativo.
- Carregue todas as amostras analisadas clicando em Arquivo | Abra a lista de trabalho.
- Clique com o botão direito do mouse em uma amostra correspondente à solução intermediária do EA a 1.000 ng/mL, vá para Editar cromatograma e selecione Tipo | Extraído do cromatograma de íons e insira a massa molecular teórica do íon protonado relacionado a cada EA. Em seguida, clique em Adicionar e, finalmente, em OK. Como o conjunto de analitos é formado por seis EAs e seus epímeros relacionados que compartilham a mesma massa exata, seis cromatogramas de íons extraídos aparecerão.
NOTA: O parâmetro Width altera o quão estreita é a janela m/z . Recomenda-se selecionar uma janela m/z suficientemente larga em caso de dificuldades em encontrar o pico cromatográfico da substância a analisar em causa. No entanto, é aconselhável definir janelas m/z estreitas para evitar possíveis interferências (abaixo de 0,05 unidades). - Em cada cromatograma de íons extraído, aparecerão dois picos cromatográficos relacionados ao EA principal e seu epímero. Clique com o botão direito do mouse enquanto seleciona toda a área de um pico. Consequentemente, o espectro de mobilidade iônica aparecerá logo abaixo.
- Vá para o eixo x do espectro de mobilidade iônica, clique com o botão direito do mouse e clique em Seção Transversal de Colisão. Anote o valor CCS.
NOTA: Se os valores CCS não aparecerem automaticamente, vá para Calibrar na barra de ferramentas superior, depois Calculadora de mobilidade e digite manualmente o valor de mobilidade para obter o CCS correspondente. - Selecionar a área correspondente ao outro pico cromatográfico dentro do mesmo cromatograma de íons e repetir as etapas 5.4. e 5.5.
- Da mesma forma, o padrão de fragmentação também aparecerá após a etapa 5.4. Com base no padrão de fragmentação relatado na literatura e em bancos de dados, comparar e escolher o íon produto mais intenso como ponto de identificação complementar.
- Anote o tempo de retenção, o valor CCS e a massa exata do aduto de íons principal (geralmente o íon protonado) de cada EA para criar um método de quantificação.
- Repita as etapas 5.4 a 5.8. para os restantes analitos.
- Selecione todas as massas moleculares geradas na etapa 5.3 clicando nelas enquanto pressiona Ctrl. Em seguida, clique com o botão direito do mouse e selecione Copiar. Vá para o outro exemplo correspondente à solução intermediária do EA, clique com o botão direito do mouse e selecione Colar. Os cromatogramas de íons e os espectros de mobilidade serão extraídos automaticamente.
- Verificar novamente os parâmetros de identificação de todas as substâncias a analisar nesta segunda amostra.
6. Criação de um método de processamento de dados para a determinação rotineira de alcaloides da cravagem
- Abra o software e clique na guia Gerenciamento de métodos .
- Clique em Configurações do analito e insira o nome de cada EA junto com seu tempo de retenção, valor CCS e a massa dos adutos protonados coletados nas etapas 5.4. para 5.9. Altere a tolerância de cada ponto de identificação conforme necessário, embora as configurações padrão sejam recomendadas para a aplicação deste protocolo.
NOTA: Quanto maiores os valores de tolerância definidos, maior a possibilidade de lidar com falsos positivos, uma vez que um desvio maior dos valores teóricos é aceito. Da mesma forma, valores de tolerância muito rigorosos podem levar a falsos negativos. Isso acontece quando o desvio do valor teórico é causado exclusivamente pelo desempenho do instrumento. - Guarde o método de quantificação para uso posterior.
7. Amostragem
- Adquirir amostras de trigo e aveia em supermercados, preferencialmente em embalagens de 1 kg, seguindo as diretrizes de amostragem estabelecidas pela Comissão Europeia20. Se não estiverem disponíveis embalagens de 1 kg, certifique-se de ter, pelo menos, 50 g de amostras para completar toda a análise.
- Moa as amostras usando um moedor. Neste estudo, foi utilizado um moedor de lâminas para moer todo o conteúdo das embalagens, que foram devolvidas às suas embalagens originais para homogeneização.
NOTA: Limpe bem o moedor entre as amostras para evitar contaminação cruzada. Use um jaleco de laboratório. - Alicotar as amostras homogeneizadas em tubos de centrifugação de 50 mL e armazená-las em condições frescas e secas.
8. Preparação da amostra
- Preparar um cronograma para realizar o estudo de validação por três dias não consecutivos (para avaliar a reprodutibilidade interna). Para cada dia, analise três níveis de concentração fortificando amostras a 225 ng/g (1,5 mL), 150 ng/g (ML) e 75 ng/g (0,5 mL). Estude cada nível em triplicado, representando nove amostras enriquecidas mais outras três amostras em branco por dia.
NOTA: Considerando os 3 dias de validação, serão necessárias um total de 36 amostras juntamente com outras 8 amostras em branco destinadas à preparação da curva de calibração pareada com matriz. Use luvas de nitrilo, jaleco de laboratório e óculos de segurança para toda esta seção do protocolo. - Pesar 1 g de amostra em um tubo de centrífuga de 50 mL.
NOTA: Ao realizar o estudo de validação, fortificar as amostras logo após a pesagem com 50 μL da solução-padrão de trabalho correspondente detalhada nas etapas 1.3. e 1.5. - Adicionar 4 ml da solução de extracção [acetonitrila:5 mM de solução aquosa de carbonato de amónio (85:15, v/v)]. Vortex a amostra por 1 min.
- Centrifugue a amostra por 5 min a 9.750 × g e 4 °C.
NOTA: A capacidade da centrífuga pode ser um fator limitante no procedimento, por isso é recomendável extrair simultaneamente apenas o número exato de amostras que caberão na centrífuga. - Transferir todo o sobrenadante para o tubo de centrifugação de 15 ml contendo 150 mg da mistura de adsorvente de limpeza (1:1, p/p). Vortex a amostra por 1 min.
- Centrifugar o tubo durante 5 min a 9.750 × g e 4 °C.
- Recolha o sobrenadante e coloque-o num frasco para injetáveis de vidro âmbar de 4 ml. Evaporar o extracto sob uma corrente suave de azoto.
NOTA: As amostras devem ser mantidas secas a -20 °C se não forem analisadas no mesmo dia. - Reconstituir a amostra em 750 μl da mistura metanol:água (1:1, v/v).
- Transferir o extracto para uma seringa de 2 ml e filtrá-lo através de um filtro de nylon de 0,22 μm para um frasco para injetáveis cromatográficos âmbar de 1,5 ml.
- Recolher os extractos reconstituídos das oito amostras em branco destinadas à curva de calibração compatível com a matriz num frasco para injectáveis de vidro âmbar de 12 ml (cada amostra é ressuspensa em 750 μL, representando um volume total de 6 ml).
- Pipete 1 mL da solução intermediária a 1.000 ng / mL (83,33 ng / mL para cada alcalóide da cravagem) (etapa 1.2) em um frasco de vidro âmbar de 2 mL, seque o solvente sob um fluxo suave de nitrogênio e ressuspenda em 1 mL do extrato de amostra em branco coletado anteriormente.
NOTA: Esta mistura a uma concentração de 1,000 ng/mL (83.33 ng/mL para cada EA) será usada para preparar a curva de calibração combinada com a matriz. - Prepare dez frascos de vidro âmbar de 2 mL e coloque 450 μL de extrato de amostra em branco (etapa 8.10) em cada um e marque-os com números de 1 a 10. Cada frasco para injetáveis será um ponto da curva de calibração.
NOTA: Neste protocolo, foi utilizado 450 μL, pois era o valor mais baixo que o dispositivo LC poderia atingir dentro do frasco sem o uso de insertos. Se a agulha não chegar tão longe no frasco, aumente o volume e, consequentemente, calcule quantas amostras extras em branco devem ser extraídas, considerando que cada uma é ressuspensa em 750 μL de mistura metanol:água (1/1, v:v). - Construir a curva de calibração por diluição serial. Transferir 450 μL da mistura preparada na etapa 8.11. no frasco 1, que já contém 450 μL de extrato de amostra em branco, para atingir uma concentração de 41,67 ng/mL (o ponto de calibração mais alto). Vortex o frasco para injetáveis durante 2 min.
NOTA: O volume da mistura deve corresponder ao volume do extracto da amostra em branco a diluir duas vezes em cada fase. - Transfira 450 μL do frasco 1 para o frasco 2, que já contém 450 μL de extrato de amostra em branco, para atingir uma concentração de 20,83 ng/mL (o segundo ponto de calibração mais alto).
- Repita a etapa 8.14 (ou seja, do frasco 2 para o frasco 3 e assim por diante) e continue subsequentemente até que os dez pontos de calibração sejam obtidos. A faixa de concentração para cada EA será: 41,67, 20,83, 10,42, 5,21, 2,60, 1,30, 0,65, 0,33, 0,16 e 0,08 ng/mL.
- Prepare uma mistura padrão a 1.000 ng/mL (83,33 ng/mL para cada EA) conforme detalhado na etapa 8.11, mas usando 1 mL de mistura metanol:água (1:1, v/v).
- Preparar uma curva de calibração normalizada, conforme descrito nos passos 8.13 a 8.15, mas utilizando 450 μL de mistura metanol:água (1:1, v/v) para as diluições em série. Esta curva de calibração será usada para avaliar o efeito da matriz, comparando-o com a curva de calibração pareada com a matriz.
9. Tratamento de dados quantitativos
- Inicialize e execute o instrumento seguindo as instruções descritas nas etapas 4.1. para 4.11. Consulte a Figura 2 para obter a lista de trabalho usada neste protocolo.
NOTA: Ao escrever a lista de trabalho para curvas de calibração, injete-as antes das amostras fortificadas, começando com a curva de calibração padrão da concentração mais baixa para a mais alta, um frasco de lavagem contendo metanol: água (1:1, v/v) e a curva de calibração correspondente à matriz da concentração mais baixa para a mais alta. - Abra o software quantitativo e navegue até a guia TASQ Quick | Lote de importação no canto inferior esquerdo.
- Selecione a pasta criada na etapa 4.10. contendo todos os dados brutos (arquivos relacionados às informações adquiridas após a análise LC-IMS-MS para todas as amostras incluídas na lista de trabalho).
- Depois de importado para o software, vá para Processar lote, logo abaixo de Importar lote.
- Escolha as amostras a serem processadas (ou seja, as 27 amostras enriquecidas, 9 blanks e ambas as curvas de calibração) e o método de processamento de dados criado anteriormente nas etapas 6.1. para 6.3. Uma janela aparecerá com diferentes parâmetros a serem definidos (ou seja, o tipo de integrador para avaliar os picos cromatográficos ou o tipo de sinal analítico a ser considerado, entre outros). Altere esses parâmetros conforme desejado, embora os valores padrão sejam adequados para a execução deste protocolo.
- Vá para Revisar triagem após a conclusão do processamento de dados e verifique se há algum problema relacionado à integração automática, como a seleção de picos cromatográficos incorretos que podem estar muito próximos uns dos outros. Em caso afirmativo, restrinja os valores referentes à tolerância do tempo de retenção, tolerância dos valores CCS e/ou erro de massa inicialmente estabelecidos no arquivo do método quantitativo e reprocesse os dados. Se a integração automática de picos ainda selecionar picos incorretos, integre manualmente o pico selecionando a área no cromatograma que aparece na janela Review Screening (para fazer isso, leve em consideração os parâmetros analíticos obtidos para os padrões EA na seção 5).
NOTA: É muito importante revisar cada amostra, pois alguns pares principais de EA-epímero têm tempos de retenção muito semelhantes e podem ser identificados incorretamente pelo software. - Quando todos os picos tiverem sido integrados com sucesso para todas as amostras, clique na guia Gerenciamento de lotes no canto superior esquerdo e indique o tipo de amostra associada a cada análise: solvente, branco, controle de qualidade, amostra ou ponto de calibração. Neste último caso, atribua cada ponto de calibração a um nível com um número.
- Na mesma guia Gerenciamento de lotes , navegue até a janela inferior chamada Concentração de lotes e especifique um valor de concentração para cada nível estabelecido na etapa anterior. Feito isso, clique em Salvar concentrações.
- Clique em Quantificar lote e altere os parâmetros conforme desejado (ou seja, o modelo para ajustar os dados de calibração, ponderação, se o modelo é forçado a cruzar zero, etc.). Os valores padrão são adequados para a execução deste protocolo, exceto para a ponderação das curvas de calibração, que foi alterada para 1/x.
NOTA: Como o software só pode considerar uma curva de calibração por vez, é necessário repetir a etapa 9.9 duas vezes para ter informações sobre as curvas de calibração padrão e com correspondência de matriz. Neste protocolo, curvas de calibração pareadas com matriz foram utilizadas para a quantificação dos analitos, de modo que as curvas de calibração do solvente foram avaliadas primeiro, os dados de calibração foram extraídos conforme explicado posteriormente na etapa 9.13 e, finalmente, as curvas de calibração pareadas com a matriz foram selecionadas para realizar a quantificação das amostras. - Uma vez realizada a quantificação dos alcaloides da cravagem de acordo com o ficheiro do método de quantificação, clique no separador Quantificação na parte superior do ecrã.
- Se os parâmetros padrão definidos na etapa 9.9 não fornecerem boa linearidade e precisão, ajuste as curvas de calibração alterando o encaixe e o peso clicando com o botão direito do mouse no gráfico com o objetivo de ter um bom modelo linear (R2 > 0,99) com precisão adequada em cada nível de calibração (desvio abaixo de 20%).
NOTA: O software marcará em vermelho as células dos níveis de calibração que não fornecem a precisão adequada. - Quando terminar, clique com o botão direito do mouse na pasta de lote na extremidade esquerda da tela e selecione Gerar relatório. Em seguida, selecione Quantificação do analito para exportar o relatório para o formato desejado.
Resultados
Primeiro, soluções padrão de trabalho foram injetadas no instrumento LC-IMS-MS para obter todos os recursos de identificação (ou seja, tempo de retenção, CCS e espectros de massa) de cada EA analisado aqui. Como os parâmetros de identificação, exceto a massa exata, eram inicialmente desconhecidos, o método de aquisição foi baseado em um evento de duas varreduras, começando com uma varredura completa de todo o espectro de massa seguida por um bbCID. A forma retrospectiva de ...
Discussão
O sucesso do uso desse protocolo baseia-se na otimização do procedimento de extração, previamente realizada por Carbonell-Rozas et al.17, que implementaram o uso de um solvente de extração eficaz o suficiente para extrair EAs de matrizes alimentares complexas, como cevada e trigo, e uma limpeza que forneceu valores de SSE relativamente baixos. A escolha do solvente de extração representa uma etapa crítica considerando as características químicas dos ana...
Divulgações
Os autores não têm conflitos de interesse a divulgar.
Agradecimentos
Esta investigação foi financiada pela Consejería de Universidad, Investigación e Innovación - Junta de Andalucía (PROYEXCEL_00195) e a bolsa de pós-doutoramento atribuída pela Generalitat Valenciana e Fundo Social Europeu+ (CIAPOS/2022/049). Os autores agradecem ao "Centro de Instrumentación Científica (CIC)" da Universidade de Granada por fornecer acesso à instrumentação analítica usada neste protocolo.
Materiais
| Name | Company | Catalog Number | Comments |
| Acetonitrile | VWR | 83640.32 | |
| Amber glass tubes 4 mL | VWR | 548-0052 | |
| Amber glass tubes 12 mL | VWR | 548-0903 | |
| Amber vials 1.5 mL | Agilent | 5190-9063 | |
| Ammonium carbonate | Fluka | 9716 | |
| Analytical balance BAS 31 | Boeco | 4400519 | |
| Balance CP 323 S | Sartorius | 23-84182 | |
| C18 | Supelco | 52604-U | |
| Centrifuge tubes, 15 mL | VWR | 525-1082 | |
| Centrifuge tubes, 50 mL | VWR | 525-0155 | |
| Centrifuge Universal 320 R | Hettich | 1406 | |
| Compass HyStar | Bruker | Acquisition software | |
| DataAnalysis | Bruker | Qualitative software | |
| Elute PLUS UHPLC | Bruker | ||
| EVA EC-S evaporator | VLM | V830.012.12 | |
| Formic acid GR for analysis ACS, Reag. Ph Eur | Merck | 100264 | |
| Grinder TitanMill300 | Cecotec | 1559 | |
| Methanol | VWR | 83638.32 | |
| Milli-Q water purification system (18.2 MΩ cm) | Millipore | ZD5211584 | |
| Pipette tips 1- 5 mL | Labortecnic | 162005 | |
| Pipette tips 100 - 1000 µL | Labortecnic | 1622222 | |
| Pipette tips 5 - 200 µL | Labortecnic | 162001 | |
| Pippette Transferpette S variable, DE-M 10 - 100 µL | BRAND | 704774 | |
| Pippette Transferpette S variable, DE-M 100 - 1000 µL | BRAND | 704780 | |
| Pippette Transferpette S variable, DE-M 500 - 5000 µL | BRAND | 704782 | |
| Syringe 2 mL | VWR | 613-2003 | |
| Syringe Filter 13 mm, 0.22µm | Phenomenex | AF-8-7707-12 | |
| TASQ | Bruker | Quantitative software | |
| timsTOFPro2 IM-HRMS | Bruker | ||
| Vortex Genie 2 | Scientific Industries | 15547335 | |
| Zorbax Eclipse Plus RRHD C18 column (50 x 2.1 mm, 1.8 µm particle size) | Agilent | 959757-902 | |
| Z-Sep+ | Supelco | 55299-U | Zirconia-based sorbent |
| Ergot alkaloids | CAS registry sorbent | ||
| Ergocornine (Eco) | Techno Spec | E178 | 564-36-3 |
| Ergocorninine (Econ) | Techno Spec | E130 | 564-37-4 |
| Ergocristine (Ecr) | Techno Spec | E180 | 511-08-0 |
| Ergocristinine (Ecrn) | Techno Spec | E188 | 511-07-9 |
| Ergokryptine (Ekr) | Techno Spec | E198 | 511-09-1 |
| Ergopkryptinine (Ekrn) | Techno Spec | E190 | 511-10-4 |
| Ergometrine (Em) | Romer Labs | "002067" | 60-79-7 |
| Ergometrinine (Emn) | Romer Labs | LMY-090-5ML | 479-00-5 |
| Ergosine (Es) | Techno Spec | E184 | 561-94-4 |
| Ergosinine (Esn) | Techno Spec | E194 | 596-88-3 |
| Ergotamine (Et) | Romer Labs | "002069" | 113-15-5 |
| Ergotaminine (Etn) | Romer Labs | "002075" | 639-81-6 |
Referências
- Kanu, A. B., Dwivedi, P., Tam, M., Matz, L., Hill, H. H. Ion mobility-mass spectrometry. J Mass Spectrom. 43 (1), 1-22 (2008).
- Gabelica, V., et al. Recommendations for reporting ion mobility Mass spectrometry measurements. Mass Spectrom Rev. 38 (3), 291-320 (2019).
- Feuerstein, M. L., et al. Comparability of steroid collision cross sections using three different IM-HRMS technologies: An interplatform study. J Am Soc Mass Spectrom. 33 (10), 1951-1959 (2022).
- Regueiro, J., Negreira, N., Berntssen, M. H. G. Ion-mobility-derived collision cross section as an additional identification point for multiresidue screening of pesticides in fish feed. Anal Chem. 88 (22), 11169-11177 (2016).
- Regueiro, J., Negreira, N., Hannisdal, R., Berntssen, M. H. G. Targeted approach for qualitative screening of pesticides in salmon feed by liquid chromatography coupled to traveling-wave ion mobility/quadrupole time-of-flight mass spectrometry. Food Control. 78, 116-125 (2017).
- Hernández-Mesa, M., Monteau, F., Le Bizec, B., Dervilly-Pinel, G. Potential of ion mobility-mass spectrometry for both targeted and non-targeted analysis of phase II steroid metabolites in urine. Anal Chim Acta: X. 1, 100006 (2019).
- George, A. C., et al. Interplatform comparison between three ion mobility techniques for human plasma lipid collision cross sections. Anal Chim Acta. 1304, 342535 (2024).
- Mairinger, T., Causon, T. J., Hann, S. The potential of ion mobility-mass spectrometry for non-targeted metabolomics. Curr Opin Chem Biol. 42, 9-15 (2018).
- Hernández-Mesa, M., Escourrou, A., Monteau, F., Le Bizec, B., Dervilly-Pinel, G. Current applications and perspectives of ion mobility spectrometry to answer chemical food safety issues. TrAC Trend Anal Chem. 94, 39-53 (2017).
- European, C. Commission Regulation (EU) 2023/915 of 25 April 2023 on maximum levels for certain contaminants in food and repealing Regulation (EC) No 1881/2006. Off J Eur Union. 119, 103-157 (2023).
- Carbonell-Rozas, L., et al. Occurrence of egot alkaloids in major and minor cereals from Northern Italy: A three harvesting years scenario. J Agricl Food Chem. 71 (42), 15821-15828 (2023).
- Arroyo-Manzanares, N., Gámiz-Gracia, L., García-Campaña, A. M., Diana Di Mavungu, J., De Saeger, S., Jean-Michel Mérillon, J. -. M., Ramawat, K. G. . Fungal Metabolites. , (2016).
- van Dongen, P., de Groot, A. History of ergot alkaloids from ergotism to ergometrine. Eur J Obstet Gynecol Reprod Biol. 60 (2), 109-116 (1995).
- European Food Safety Authority. Human and animal dietary exposure to ergot alkaloids. EFSA J. 15 (7), e04902 (2017).
- Nam, M., Kim, D., Kim, M. -. S. Simultaneous determination of total ergot alkaloids in wheat flour by Orbitrap mass spectrometry. Food Chem. 441, 138363 (2024).
- García-Juan, A., León, N., Armenta, S., Pardo, O. Development and validation of an analytical method for the simultaneous determination of 12 ergot, 2 tropane, and 28 pyrrolizidine alkaloids in cereal-based food by LC-MS/MS. Food Res Int. 174 (Pt 1), 113614 (2023).
- Carbonell-Rozas, L., Mahdjoubi, C. K., Arroyo-Manzanares, N., García-Campaña, A. M., Gámiz-Gracia, L. Occurrence of ergot alkaloids in barley and wheat from Algeria. Toxins. 13 (5), 316 (2021).
- Carbonell-Rozas, L., Gámiz-Gracia, L., Lara, F. J., García-Campaña, A. M. Determination of the main ergot alkaloids and their epimers in oat-based functional foods by ultra-high performance liquid chromatography tandem mass spectrometry. Molecules. 26 (12), 3717 (2021).
- Carbonell-Rozas, L., et al. Ion mobility-mass spectrometry to extend analytical performance in the determination of ergot alkaloids in cereal samples. J Chromatogr A. 1682, 463502 (2022).
- European Commission. Commission Implementing Regulation (EU) 2023/2782 of 14 December 2023 laying down the methods of sampling and analysis for the control of the levels of mycotoxins in food and repealing Regulation (EC) No 401/2006. Off J Eur Union. , (2023).
- Carbonell-Rozas, L., Vander Cruyssen, L., Dall'Asta, C., Leggieri, M. C., Battilani, P. Fit-for-purpose method development to determine co-occurring multiclass mycotoxins in apple and apple puree samples. Food Anal Methods. 16 (8), 1403-1412 (2023).
- Laouni, C., et al. Emerging mycotoxin occurrence in chicken feed and eggs from Algeria. Mycotoxin Res. 40, 447-456 (2024).
- Ben Hassouna, K., et al. Mycotoxin occurrence in milk and durum wheat samples from Tunisia using dispersive liquid-liquid microextraction and liquid chromatography with fluorescence detection. Toxins. 15 (11), 633 (2023).
- Narváez, A., et al. Occurrence and exposure assessment of mycotoxins in ready-to-eat tree nut products through ultra-high performance liquid chromatography coupled with high resolution Q-orbitrap mass spectrometry. Metabolites. 10 (9), 344 (2020).
- Arroyo-Manzanares, N., Rodríguez-Estévez, V., García-Campaña, A. M., Castellón-Rendón, E., Gámiz-Gracia, L. Determination of principal ergot alkaloids in swine feeding. J Sci Food Agric. 101 (12), 5214-5224 (2021).
- Pereira, V. L., Fernandes, J. O., Cunha, S. C. Comparative assessment of three cleanup procedures after QuEChERS extraction for determination of trichothecenes (type A and type B) in processed cereal-based baby foods by GC-MS. Food Chem. 182, 143-149 (2015).
- Tuzimski, T., Szubartowski, S. Method development for selected bisphenols analysis in sweetened condensed milk from a can and breast milk samples by HPLC-DAD and HPLC-QqQ-MS: Comparison of sorbents (Z-SEP, Z-SEP Plus, PSA, C18, chitin and EMR-lipid) for clean-up of QuEChERS extract. Molecules. 24 (11), 2093 (2019).
- Łozowicka, B., Rutkowska, E., Jankowska, M. Influence of QuEChERS modifications on recovery and matrix effect during the multi-residue pesticide analysis in soil by GC/MS/MS and GC/ECD/NPD. Environ Sci Pollut Res. 24 (8), 7124-7138 (2017).
- Schummer, C., Xandonella, I., van Nieuwenhuyse, A., Moris, G. Epimerization of ergot alkaloids in feed. Heliyon. 6 (6), e04336 (2020).
- Cherewyk, J. E., Grusie-Ogilvie, T. J., Parker, S. E., Blakley, B. R., Al-Dissi, A. M. The impact of storage temperature and time on ergot alkaloid concentrations. Toxins. 15 (8), 497 (2023).
- Silva, &. #. 1. 9. 4. ;., Mateus, A. R., Barros, S. C., Silva, A. S. Ergot alkaloids on cereals and seeds: Analytical methods, occurrence, and future perspectives. Molecules. 28 (20), 7233 (2023).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados