È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Rilevazione di alcaloidi regolati dell'ergot in matrici alimentari mediante cromatografia liquida-spettrometria di mobilità ionica intrappolata-spettrometria di massa a tempo di volo
In questo articolo
Riepilogo
Questo protocollo presenta un metodo convalidato di cromatografia liquida-spettrometria di massa a mobilità ionica ad alta risoluzione per determinare la presenza di alcaloidi dell'ergot negli alimenti in conformità con il Regolamento della Commissione (UE) 2023/915 recentemente pubblicato.
Abstract
La spettrometria di massa a mobilità ionica (IMS) funge da dimensione di separazione aggiuntiva quando integrata nei flussi di lavoro di cromatografia liquida-spettrometria di massa (LC-MS). I metodi LC-IMS-MS offrono una risoluzione di picco più elevata, una migliore separazione dei composti isobarici e isomerici e un migliore rapporto segnale/rumore (S/N) rispetto ai tradizionali metodi LC-MS. L'IMS fornisce un'altra caratteristica molecolare per l'identificazione degli analiti, ovvero il parametro della sezione d'urto di collisione (CCS), riducendo i risultati falsi positivi. Pertanto, i metodi LC-IMS-MS affrontano importanti sfide analitiche nel campo della sicurezza alimentare (ad esempio, la rilevazione di composti a livelli di tracce in matrici alimentari complesse e l'identificazione univoca di molecole isobariche e isomeriche).
Gli alcaloidi della segale cornuta (EA) sono una famiglia di micotossine prodotte da funghi che attaccano un'ampia varietà di specie erbacee, tra cui piccoli cereali come segale, triticale, grano, orzo, miglio e avena. I livelli massimi (ML) di queste micotossine sono stati stabiliti in diversi prodotti alimentari, come specificato nel regolamento (CE) 2023/915 della Commissione. Questa nuova legislazione include sei EA principali e i relativi epimeri, quindi è necessaria una metodologia efficiente per distinguere correttamente queste molecole isomeriche considerando la loro co-occorrenza.
Pertanto, l'obiettivo di questo protocollo è quello di mostrare come l'integrazione dell'IMS nei flussi di lavoro LC-MS contribuisca alla separazione degli EA isomeri, migliorando la selettività del metodo analitico. Inoltre, illustra come la generazione di librerie CCS attraverso la caratterizzazione di standard analitici fornisca una maggiore sicurezza per l'identificazione delle micotossine. Questo protocollo è concepito per spiegare chiaramente i vantaggi dell'attuazione dell'IMS nella sicurezza alimentare, prendendo come esempio la determinazione degli EA nei cereali. Un'estrazione basata su QuEChERS seguita da un'analisi TIMS-MS con spettrometria di mobilità ionica intrappolata LC (LC-Trapped Ion Mobility Spectre) ha fornito limiti di quantificazione compresi tra 0,65 e 2,6 ng/g con un'accuratezza accettabile (anche se un recupero basso per l'ergotaminina) a 1,5x, 1x e 0,5x il ML e ha mostrato un effetto matrice trascurabile.
Introduzione
La spettrometria di massa a mobilità ionica (IMS) sta diventando una tecnica analitica sempre più utilizzata, spesso presentata come una dimensione di separazione aggiuntiva integrata nella tradizionale cromatografia liquido/gassosa (LC/GC) accoppiata ai flussi di lavoro MS. L'IMS consiste nella separazione di molecole lungo una cella di mobilità, riempita con un gas tampone, sotto un campo elettrico e a pressione atmosferica1. A seconda del rapporto massa/carica (m/z) e della conformazione geometrica, una molecola ionizzata interagirà con il gas tampone mentre si muove attraverso la cella di mobilità, che si riflette nel parametro2 della mobilità ionica (K) e viene calcolato attraverso la seguente equazione:
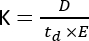
dove D rappresenta la lunghezza totale della deriva, td è il tempo totale di deriva ed E è il campo elettrico. Pertanto, K è misurato in m2 V−1 s−1, anche se per ragioni pratiche è spesso espresso come cm2 V−1 s−1. La capacità intrinseca di muoversi attraverso la cella di mobilità può essere misurata dal tempo di deriva e successivamente convertita nel cosiddetto valore della sezione d'urto di collisione (CCS), che è un parametro altamente riproducibile per ciascuna molecola indipendentemente dallo strumento IMS3. La CCS può essere derivata dalla mobilità seguendo questa equazione:
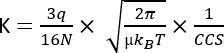
q è la carica dello ione; N la densità numerica del gas tampone; μ la massa ridotta dei partner di collisione tampona gli ioni gas; kB la costante di Boltzmann; e T la temperatura del gas tampone. Pertanto, l'IMS fornisce informazioni aggiuntive complementari ai dati analitici risultanti dalla cromatografia e dalle analisi MS.
È stato dimostrato che l'implementazione dell'IMS nelle piattaforme LC-MS aumenta l'affidabilità delle determinazioni analitiche, soprattutto quando si lavora con composti che si trovano a concentrazioni di tracce. Diversi studi hanno riportato che i metodi LC-IMS-MS migliorano la qualità degli spettri di massa riducendo il rumore di fondo, che in ultima analisi influisce sulla sensibilità del metodo, e riduce il tasso di falsi positivi e negativi forniti dalle metodologie LC-MS multi-residuo 4,5,6. Inoltre, la riproducibilità dei valori CCS consente il confronto non solo tra diversi strumenti che utilizzano la stessa tecnologia, ma anche tra diverse tecnologie di mobilità ionica, vale a dire la spettrometria di mobilità ionica a onde viaggianti (TWIMS), la spettrometria di mobilità ionica intrappolata (TIMS) e la spettrometria di mobilità ionica a tubo di deriva (DTIMS)2,7, che sono i sistemi più frequentemente utilizzati 1. Pertanto, una notevole conseguenza del potenziale della CCS come parametro di identificazione risiede nella possibilità di costruire librerie di CCS, che si riflette nella sua applicabilità negli studi di metabolomica8. Tuttavia, una delle caratteristiche più potenti dell'IMS è la capacità di separare i composti isomerici e isobarici che potrebbero non essere sufficientemente risolti dai metodi LC-MS. Questo può essere il caso quando si lavora con grandi insiemi di analiti di interesse in matrici complesse, che è una situazione comune nell'analisi ambientale e alimentare. In questo contesto, sono stati proposti metodi LC-IMS-MS per il monitoraggio dei pesticidi e, in misura minore, dei farmaci veterinari e delle micotossine negli alimenti9.
Grazie al loro elevato potere risolutivo e selettività, le piattaforme LC/GC-IMS-MS emergono come gli strumenti più utili per affrontare alcune delle attuali sfide nella sicurezza alimentare, in particolare quelle relative alle miscele isomeriche. La preoccupazione per la salute legata alle miscele isomeriche come contaminanti alimentari è stata riflessa nell'attuale legislazione europea, che, ad esempio, limita la concentrazione massima di sei alcaloidi principali dell'ergot (EA) e dei loro corrispondenti sei epimeri in diversi prodotti alimentari10.
Gli EA costituiscono una famiglia di metaboliti secondari tossici prodotti da un'ampia gamma di funghi, principalmente della famiglia delle Clavicipitaceae (ad esempio, Claviceps purpurea, il più importante produttore di EA grazie alla sua ampia gamma di ospiti), ma anche Trichocomaceae, che possono parassitare il seme di piante viventi (come segale, orzo, grano e avena) al momento della fioritura11, 12. In condizioni specifiche, in particolare la temperatura e l'attività dell'acqua, i funghi Claviceps possono produrre EA che si accumulano nei corpi fruttiferi, noti come sclerozi o segale cornuta, nella coltura ospite. In una certa misura, gli EA possono resistere alla lavorazione della materia prima fino a raggiungere il prodotto finale; quindi, irrompendo nella catena alimentare. L'ingestione di cibo contaminato può portare all'intossicazione da EA, nota come ergotismo, che si presenta con sintomi acuti come dolore addominale, vomito, sensazione di bruciore della pelle, insonnia e allucinazioni13. Per ridurre l'impatto degli EA sulla salute umana, la Commissione europea ha fissato i livelli massimi (ML) in diversi alimenti per la somma dei principali EA: gli epimeri R ergometrina (Em), ergotamina (Et), ergosina (Es), ergocristina (Ecr), ergokryptina (Ekr) ed ergocornina (Eco) e i loro corrispondenti epimeri S: ergometrinina (Emn), ergosinina (Esn), ergotaminina (Etn), ergocorninina (Econ), ergokryptinine (Ekrn), ed ergocristinina (Ecrn). Questi composti possono epimerizzare da forme R a S e viceversa, soprattutto in caso di esposizione a luce intensa, conservazione prolungata o contatto con alcuni solventi ad alto o basso pH 12. Sebbene la percentuale di forme R e S possa variare a seconda delle condizioni, il gruppo CONTAM dell'EFSA ha segnalato una maggiore presenza di forme R rispetto alle forme S dopo aver esaminato la letteratura disponibile sugli EA nei prodotti alimentari14. Pertanto, i ML variano a seconda di diversi fattori, come la suscettibilità della coltura, il grado di lavorazione o la frequenza di consumo. Nel quadro dell'UE, i valori limite di colesterolo per i prodotti lavorati di orzo, frumento, farro e avena sono stati fissati a 50 o 150 μg/kg (a seconda del tenore di ceneri inferiore o superiore a 900 mg/100 g, rispettivamente), mentre i cereali destinati direttamente al consumo umano sono soggetti a un limite massimo di 150 μg/kg, ad eccezione degli alimenti per la prima infanzia a base di cereali, in cui il ML è ridotto a 20 μg/kg10.
Questa legislazione rigorosa richiede metodologie analitiche sufficientemente sensibili da determinare i livelli di concentrazione in tracce (μg/kg) e identificare correttamente gli EA regolati e i loro epimeri corrispondenti, poiché entrambe le forme, gli isomeri R e S, possono essere trovate insieme in campioni contaminati. Questo compito rappresenta una grande sfida poiché ogni coppia tossina-epimero condivide lo stesso identico modello di massa e frammentazione. Inoltre, una corretta separazione cromatografica tra i due composti può essere complessa. Pertanto, sono necessari gradienti LC ben ottimizzati per evitare un'errata quantificazione quando gli epimeri EA si verificano in concomitanza nei campioni alimentari. Sebbene diversi studi abbiano riportato metodi LC-MS per la determinazione univoca degli EA 15,16,17,18, il metodo cromatografico deve essere studiato ampiamente per ottenere un'adeguata separazione dei picchi cromatografici per identificare inequivocabilmente gli EA. Tuttavia, questo non è solitamente fattibile per i metodi multiclasse in cui vengono determinati contemporaneamente contaminanti appartenenti a diverse famiglie chimiche. In questo contesto, un recente studio condotto da Carbonell-Rozas, Hernández-Mesa, et al.19 ha riportato un metodo LC-IMS-MS per la quantificazione degli EA in campioni di frumento e orzo, utilizzando due diversi strumenti TWIMS che hanno fornito valori CCS riproducibili e bassi limiti di quantificazione (LOQ) per rilevare eventuali non conformità in conformità con la normativa vigente. Pertanto, l'obiettivo di questo protocollo è quello di mostrare come l'integrazione dell'IMS nei flussi di lavoro LC-MS contribuisca alla separazione degli EA isomeri, migliorando la selettività del metodo analitico. Inoltre, illustra come la generazione di librerie CCS attraverso la caratterizzazione di standard analitici fornisca una maggiore affidabilità per l'identificazione delle micotossine. Questo protocollo è stato concepito per spiegare chiaramente i vantaggi dell'implementazione dell'IMS nell'analisi della sicurezza alimentare, prendendo come esempio la determinazione degli EA nei cereali. Questo protocollo riguarda il trattamento del campione basato su una procedura QuEChERS, l'analisi del campione mediante LC-TIMS-MS e l'estrazione e l'interpretazione dei dati IMS.
Protocollo
1. Preparazione di soluzioni stock, intermedie e standard di lavoro
NOTA: Utilizzare guanti in nitrile, camice da laboratorio e occhiali di sicurezza.
- Preparare le singole soluzioni madre dei 12 EA (vedere la Tabella dei materiali) a 10.000 ng/mL in flaconcini di vetro ambrato da 4 mL utilizzando acetonitrile. Le forme R (-ine) erano precedentemente aliquotate in porzioni da 25.000 ng, mentre le forme S (-inine) erano distribuite in porzioni da 10.000 ng. Questo studio è iniziato con queste aliquote, raggiungendo concentrazioni di 10.000 ng/mL diluendo le forme R e S in 2,5 e 1 mL di acetonitrile, rispettivamente, seguito da vorticing per 2 minuti.
- Preparare una soluzione madre intermedia a 1.000 ng/mL di EA totali (83,33 ng/mL di ciascuno) in un flaconcino di vetro ambrato da 12 mL aggiungendo 65 μL di ogni singola soluzione alcaloide e 7,02 mL di acetonitrile.
NOTA: Una volta preparata la soluzione madre intermedia, le singole soluzioni madre devono essere essiccate e conservate a -20 °C coperte da un foglio di alluminio per evitare l'epimerizzazione. - Preparare le soluzioni standard di lavoro dalla soluzione madre intermedia pipettando 2,7, 1,8 e 0,9 mL in provette di vetro ambrato da 4 mL. Portarli a secco sotto una leggera corrente di azoto e risospenderli in 600 μL di acetonitrile.
NOTA: Le concentrazioni risultanti sono state stabilite per raggiungere 1,5, 1 e 0,5 volte l'mL (150 ng/g) quando si aggiungono 50 μL a 1 g del campione. Questi sono selezionati come livelli di concentrazione per gli studi di convalida (ad esempio, recuperi, ripetibilità, ecc.). - Dividere i 600 μl di ciascuna soluzione standard di lavoro in tre fiale di vetro ambrato da 2 mL pipettando 200 μl e portarle ad asciugare.
NOTA: I passaggi da 1.2 a 1.4 sono spiegati schematicamente nella Figura 1. Dividendo ed essiccando le soluzioni standard di lavoro, è possibile fortificare in triplicato in tre giorni diversi, come richiesto dallo studio di validazione, senza perdere l'integrità chimica degli EA. - In ogni giorno di convalida, risospendere un flaconcino di vetro ambrato da 2 mL di ciascun livello di fortificazione in 200 μL di acetonitrile.
NOTA: Ogni livello di fortificazione sarà valutato in triplicato utilizzando 50 μl per campione; pertanto, verranno utilizzati un totale di 150 μL della soluzione.
2. Preparazione dei reagenti e delle soluzioni
NOTA: Utilizzare guanti in nitrile, camice da laboratorio e occhiali di sicurezza.
- Preparare una soluzione di carbonato di ammonio da 5 mM pesando 24,02 mg di carbonato di ammonio e sciogliendola in 50 mL di acqua. Pesare 24,02 mg di carbonato di ammonio in un becher e aggiungere circa 5 ml di acqua. Sciogliere il carbonato di ammonio agitando delicatamente la soluzione manualmente, quindi trasferirla in un matraccio tarato da 50 mL. Per raggiungere il volume finale di 50 ml, sciacquare il becher con altra acqua e trasferire la lievitazione nel matraccio tarato.
- Preparare 250 mL della soluzione di estrazione [acetonitrile:5 mM di carbonato di ammonio (85:15, v/v)] mescolando 212,5 mL di acetonitrile e 37,5 mL della soluzione di ammonio preparata al punto 2.1.
- Preparare 100 mL di una miscela metanolo:acqua (1:1, v/v) per risospendere gli estratti del campione (fase 8.8.).
- Per ciascun campione, pesare 150 mg di una miscela assorbente a base di zirconia C18:materiale dispersivo in fase solida (1:1, p/p) in provette da centrifuga da 15 mL per la fase di purificazione dispersiva (fase 8.5.).
- Preparare i solventi per la separazione cromatografica in due bottiglie diverse, ovvero 1 L di acqua ultrapura (solvente A) e metanolo (solvente B), entrambi contenenti lo 0,3% (v/v) di acido formico. Per raggiungere questa concentrazione, sono stati aggiunti 3,03 ml di acido formico (purezza del 99%) alle bottiglie.
NOTA: I solventi per la fase mobile devono essere preparati appena prima di eseguire le analisi LC-IMS-MS.
3. Impostazione dei parametri strumentali
NOTA: Lo strumento utilizzato per eseguire questo studio LC-IMS-MS era un UHPLC accoppiato con un IM-HRMS, dotato di una sorgente di ionizzazione elettrospray riscaldata con sonda isolata in vuoto (VIP-HESI). Lo strumento è stato azionato in modalità positiva.
- Creare un metodo cromatografico utilizzando il software di acquisizione per la separazione degli analiti impostando il seguente gradiente di eluizione: 0 min, 10% B; 2 min, 10% B; 4,5 min, 40% B; 9 min, 45% B; 11 min, 95% B; 12 min, 95% B; 13 min, 10% B; 16 min, 10% B.
- Impostare la velocità di flusso della fase mobile a 0,4 mL/min, la temperatura della colonna a 35 °C e il volume di iniezione a 5 μL. Se possibile, mantenere una temperatura fissa di 10 °C all'interno dell'autocampionatore.
- Impostare i parametri IMS come segue: valori di mobilità (1/K0), da 0,1 a 1,5 V·s/cm2; tempo di rampa, 100 ms; tempo di precisione, 10 ms; ciclo di lavoro, 10%; e velocità di rampa, 9,05 Hz.
- Impostare i parametri della sorgente ionica per il rilevamento MS come segue: tensione capillare, 2.500 V; pressione del nebulizzatore, 2,5 bar; portata di gas secco, 8 L/min; temperatura del gas secco, 200 °C; temperatura del gas della guaina, 450 °C; portata del gas della guaina, 4 L/min. Per l'acquisizione MS, selezionare i seguenti parametri, tenendo conto di due eventi di scansione: scansione completa dell'intero spettro di massa con un m/z da 20 a 1.300 seguita da bbCID (broadband collision-induced dissociation), che genera ioni prodotti da qualsiasi precursore a tre energie di collisione fisse di 24, 36 e 50 eV. Eseguire la ionizzazione e l'acquisizione dei dati in modalità a polarità positiva.
- Salvare questo file del metodo di acquisizione per un ulteriore utilizzo.
4. Acquisizione dei dati dagli standard analitici degli EA
NOTA: Utilizzare guanti in nitrile, camice da laboratorio e occhiali di sicurezza solo per il passaggio 4.1.
- Preparare due fiale di ambra da 1,5 mL con 500 μL di soluzione intermedia a 1.000 ng/mL e un'altra fiala con una miscela di solventi A e B (1:1, v/v) come soluzione di lavaggio.
- Calibrare lo strumento timsTOF come indicato dal produttore e selezionare la polarità positiva sia per le acquisizioni IMS che MS.
- Posizionare la colonna C18 (vedi Tabella Materiali) all'interno del forno LC, con la temperatura impostata a 35 °C come indicato nel file del metodo di acquisizione salvato al punto 3.5.
NOTA: Assicurarsi che entrambe le estremità siano ben serrate; In caso contrario, si osserverà una dispersione di fase mobile. - Condizionare la colonna LC in modo da consentire una velocità di flusso in fase mobile di 0,1 mL/min e selezionare un rapporto di miscelazione del solvente A e B di 90:10 (%) come rapporto iniziale di composizione della fase mobile.
- Attendere che la pressione della colonna sia stabile (~130 bar).
- Aumentare la portata della fase mobile a 0,2 mL/min, attendere che la pressione si stabilizzi e aumentarla nuovamente a 0,3 mL/min.
- Una volta che la pressione si stabilizza di nuovo, impostare la portata a 0,4 mL, che sarà la portata di lavoro come descritto nel file del metodo di acquisizione salvato nel passaggio 3.5, e attendere che la pressione si stabilizzi intorno ai 350 bar.
NOTA: È importante non aumentare direttamente il flusso da 0,2 a 0,4 mL/min. Una forte variazione del flusso provoca picchi di alta pressione che possono danneggiare la colonna cromatografica. - Durante il condizionamento della colonna, scrivere la lista di lavoro, includendo due campioni corrispondenti alle due fiale contenenti la soluzione intermedia di EA a 1.000 ng/mL, come indicato al punto 4.1, e la fiala di lavaggio. Analizzare la fiala di lavaggio due volte, all'inizio e alla fine del lotto di campioni.
- Nella colonna File metodo della lista di lavoro, caricare il file del metodo di acquisizione precedentemente salvato nel passaggio 3.5.
- Creare una cartella per salvare tutti i file di dati acquisiti e instradare tutti i campioni selezionando la cartella nella colonna Percorso campione .
NOTA: Assicurarsi di utilizzare la stessa cartella per salvare tutti i file generati durante lo studio di convalida per un'ulteriore elaborazione. Durante il condizionamento della colonna, i valori ai quali la pressione si stabilizza possono variare leggermente. Tuttavia, assicurarsi che la pressione sia stabile prima di aumentare nuovamente la portata per preservare l'integrità della colonna. Inoltre, quando viene raggiunta la portata di lavoro, anche la pressione deve essere stabile per ottenere una separazione cromatografica riproducibile. - Vai alla barra degli strumenti superiore del software ed esegui l'elenco di lavoro.
5. Trattamento dei dati per la creazione di un metodo di quantificazione
- Una volta eseguita la lista di lavoro, apri il software qualitativo.
- Caricare tutti i campioni analizzati facendo clic su File | Apri la lista di lavoro.
- Fare clic con il pulsante destro del mouse su un campione corrispondente alla soluzione intermedia degli EA a 1.000 ng/mL, andare su Modifica cromatogramma, quindi selezionare Tipo | Cromatogramma ionico estratto e inserire la massa molecolare teorica dello ione protonato correlato a ciascun EA. Quindi, fai clic su Aggiungi e infine su OK. Poiché l'insieme degli analiti è formato da sei EA e dai loro epimeri correlati che condividono la stessa identica massa, appariranno sei cromatogrammi ionici estratti.
NOTA: Il parametro Larghezza modifica la stretta della finestra m/z . Si consiglia di selezionare una finestra m/z sufficientemente ampia in caso di difficoltà nel trovare il picco cromatografico dell'analita di interesse. Tuttavia, si consiglia di impostare finestre m/z strette per evitare possibili interferenze (inferiori a 0,05 unità). - In ogni cromatogramma ionico estratto, appariranno due picchi cromatografici relativi all'EA principale e al suo epimero. Fare clic con il pulsante destro del mouse mentre si seleziona l'intera area di un picco. Di conseguenza, lo spettro della mobilità ionica apparirà proprio sotto.
- Vai all'asse x dello spettro di mobilità ionica, fai clic con il pulsante destro del mouse, quindi fai clic su Sezione trasversale di collisione. Annotare il valore CCS.
NOTA: Se i valori CCS non vengono visualizzati automaticamente, andare su Calibra nella barra degli strumenti superiore, quindi su Calcolatore di mobilità e digitare manualmente il valore di mobilità per ottenere il CCS corrispondente. - Selezionare l'area corrispondente all'altro picco cromatografico all'interno dello stesso cromatogramma ionico e ripetere i passaggi 5.4. e 5.5.
- Analogamente, il modello di frammentazione verrà visualizzato anche dopo il passaggio 5.4. Sulla base del modello di frammentazione riportato in letteratura e nei database, confrontare e scegliere lo ione prodotto più intenso come punto di identificazione complementare.
- Annotare il tempo di ritenzione, il valore CCS e la massa esatta dell'addotto ionico principale (di solito lo ione protonato) di ciascun EA per creare un metodo di quantificazione.
- Ripetere i passaggi da 5.4 a 5.8. per gli altri analiti.
- Seleziona tutte le masse molecolari generate nel passaggio 5.3 facendo clic su di esse mentre premi Ctrl. Quindi, fai clic con il pulsante destro del mouse e seleziona Copia. Vai all'altro esempio corrispondente alla soluzione intermedia degli EA, fai clic con il pulsante destro del mouse su di esso e seleziona Incolla. I cromatogrammi ionici e gli spettri di mobilità verranno estratti automaticamente.
- Ricontrollare i parametri di identificazione di tutti gli analiti in questo secondo campione.
6. Creazione di un metodo di elaborazione dei dati per la determinazione di routine degli alcaloidi della segale cornuta
- Apri il software e fai clic sulla scheda Gestione metodi .
- Fare clic su Impostazioni analita e inserire il nome di ciascun EA insieme al tempo di ritenzione, al valore CCS e alla massa degli addotti protonati raccolti nei passaggi 5.4. a 5.9. Modificare la tolleranza di ciascun punto di identificazione in base alle esigenze, anche se per l'applicazione di questo protocollo si consigliano le impostazioni predefinite.
NOTA: Più alti sono i valori di tolleranza impostati, maggiore è la possibilità di gestire falsi positivi, poiché è accettato uno scostamento maggiore dai valori teorici. Allo stesso modo, valori di tolleranza troppo rigorosi possono portare a falsi negativi. Ciò accade quando lo scostamento dal valore teorico è causato esclusivamente dalle prestazioni dello strumento. - Conservare il metodo di quantificazione per un ulteriore utilizzo.
7. Campionamento
- Acquisire campioni di grano e avena nei supermercati, preferibilmente in contenitori da 1 kg, seguendo le linee guida per il campionamento stabilite dalla Commissione Europea20. Se non sono disponibili confezioni da 1 kg, assicurati di avere almeno 50 g di campioni per completare l'intera analisi.
- Macinare i campioni utilizzando un macinatore. In questo studio, è stata utilizzata una smerigliatrice a lame per macinare l'intero contenuto delle confezioni, che sono state poi riportate nelle loro confezioni originali per l'omogeneizzazione.
NOTA: Pulire accuratamente il macinatore tra i campioni per evitare contaminazioni incrociate. Utilizzare un camice da laboratorio. - Aliquotare i campioni omogeneizzati in provette da centrifuga da 50 mL e conservarli in condizioni fresche e asciutte.
8. Preparazione del campione
- Preparare un programma per eseguire lo studio di convalida per tre giorni non consecutivi (per valutare la riproducibilità interna). Per ogni giorno, saggiare tre livelli di concentrazione fortificando i campioni a 225 ng/g (1,5 mL), 150 ng/g (ML) e 75 ng/g (0,5 mL). Studia ogni livello in triplice copia, che rappresenta nove campioni a spillo più altri tre campioni in bianco al giorno.
NOTA: Considerando i 3 giorni di convalida, saranno necessari un totale di 36 campioni insieme ad altri 8 campioni in bianco destinati alla preparazione della curva di calibrazione a matrice. Utilizzare guanti in nitrile, camice da laboratorio e occhiali di sicurezza per l'intera sezione del protocollo. - Pesare 1 g di campione in una provetta da centrifuga da 50 mL.
NOTA: Quando si esegue lo studio di convalida, fortificare i campioni subito dopo la pesata con 50 μl della soluzione standard di lavoro corrispondente descritta nei passaggi 1.3. e 1.5. - Aggiungere 4 mL della soluzione di estrazione [acetonitrile:5 mM soluzione acquosa di carbonato di ammonio (85:15, v/v)]. Agitare il campione per 1 minuto.
- Centrifugare il campione per 5 minuti a 9,750 × g e 4 °C.
NOTA: La capacità della centrifuga può essere un fattore limitante nella procedura, quindi si consiglia di estrarre contemporaneamente solo il numero esatto di campioni che si adattano alla centrifuga. - Trasferire l'intero surnatante nella provetta da centrifuga da 15 mL contenente 150 mg della miscela di assorbente di purificazione (1:1, p/p). Agitare il campione per 1 minuto.
- Centrifugare la provetta per 5 minuti a 9,750 × g e 4 °C.
- Raccogliere il surnatante e metterlo in un flaconcino di vetro ambrato da 4 mL. Far evaporare l'estratto sotto un leggero getto di azoto.
NOTA: I campioni devono essere mantenuti asciutti a -20 °C se non analizzati lo stesso giorno. - Ricostituire il campione in 750 μl della miscela metanolo:acqua (1:1, v/v).
- Trasferire l'estratto in una siringa da 2 ml e filtrarlo attraverso un filtro di nylon da 0,22 μm in una fiala cromatografica ambrata da 1,5 ml.
- Raccogliere gli estratti ricostituiti degli otto campioni bianchi destinati alla curva di calibrazione corrispondente alla matrice in un flaconcino di vetro ambrato da 12 mL (ogni campione viene risospeso in 750 μL, per un volume totale di 6 mL).
- Pipettare 1 mL della soluzione intermedia a 1.000 ng/mL (83,33 ng/mL per ciascun alcaloide della segale cornuta) (fase 1.2) in un flaconcino di vetro ambrato da 2 mL, asciugare il solvente sotto un leggero getto di azoto e risospendere in 1 mL dell'estratto del campione bianco precedentemente raccolto.
NOTA: Questa miscela a una concentrazione di 1.000 ng/mL (83,33 ng/mL per ogni EA) verrà utilizzata per preparare la curva di calibrazione a matrice. - Preparare dieci fiale di vetro ambrato da 2 mL e inserire 450 μL di estratto di campione bianco (passaggio 8.10) in ciascuna di esse e contrassegnarle con numeri da 1 a 10. Ogni fiala sarà un punto della curva di calibrazione.
NOTA: In questo protocollo, è stato utilizzato 450 μL poiché era il valore più basso che il dispositivo LC poteva raggiungere all'interno della fiala senza l'uso di inserti. Se l'ago non arriva così in profondità nella fiala, aumentare il volume e, di conseguenza, calcolare quanti campioni bianchi in più devono essere estratti considerando che ognuno di essi viene risospeso in 750 μL di miscela metanolo:acqua (1/1, v:v). - Costruire la curva di calibrazione mediante diluizione seriale. Trasferire 450 μl della miscela preparata al punto 8.11. nella fiala 1, che contiene già 450 μL di estratto di campione bianco, per raggiungere una concentrazione di 41,67 ng/mL (il punto di calibrazione più alto). Agitare la fiala per 2 min.
NOTA: Il volume della miscela deve corrispondere al volume dell'estratto del campione bianco per diluire due volte in ogni fase. - Trasferire 450 μl dalla fiala 1 alla fiala 2, che contiene già 450 μl di estratto di campione bianco, per raggiungere una concentrazione di 20,83 ng/mL (il secondo punto di calibrazione più alto).
- Ripetere il passaggio 8.14 (cioè dalla fiala 2 alla fiala 3 e così via) e continuare successivamente fino a ottenere i dieci punti di calibrazione. L'intervallo di concentrazione per ciascun EA sarà quindi: 41,67, 20,83, 10,42, 5,21, 2,60, 1,30, 0,65, 0,33, 0,16 e 0,08 ng/mL.
- Preparare una miscela standard a 1.000 ng/mL (83,33 ng/mL per ogni EA) come descritto al punto 8.11 ma utilizzando 1 mL di miscela metanolo:acqua (1:1, v/v).
- Preparare una curva di calibrazione standard come descritto nei passaggi da 8.13 a 8.15, ma utilizzando 450 μL di miscela metanolo:acqua (1:1, v/v) per le diluizioni seriali. Questa curva di calibrazione verrà utilizzata per valutare l'effetto della matrice confrontandolo con la curva di calibrazione abbinata alla matrice.
9. Trattamento quantitativo dei dati
- Inizializzare ed eseguire lo strumento seguendo le istruzioni descritte nei passaggi 4.1. al punto 4.11. Vedere la Figura 2 per l'elenco di lavoro utilizzato in questo protocollo.
NOTA: Quando si scrive l'elenco di lavoro per le curve di calibrazione, iniettarle prima dei campioni fortificati iniziando con la curva di calibrazione standard dalla concentrazione più bassa a quella più alta, una fiala di lavaggio contenente metanolo:acqua (1:1, v/v) e la curva di calibrazione corrispondente alla matrice dalla concentrazione più bassa a quella più alta. - Apri il software quantitativo e vai alla scheda rapida TASQ | Importa batch in basso a sinistra.
- Selezionare la cartella creata nel passaggio 4.10. contenente tutti i dati grezzi (file relativi alle informazioni acquisite dopo l'analisi LC-IMS-MS per tutti i campioni inclusi nella worklist).
- Una volta importato nel software, vai su Elabora batch, proprio sotto Importa batch.
- Scegliere i campioni da elaborare (ad esempio, i 27 campioni addizionati, i 9 bianchi ed entrambe le curve di calibrazione) e il metodo di elaborazione dei dati precedentemente creato nei passaggi 6.1. al punto 6.3. Apparirà una finestra con diversi parametri da impostare (ad esempio, il tipo di integratore per valutare i picchi cromatografici o il tipo di segnale analitico da considerare, tra gli altri). Modificare questi parametri come desiderato, anche se i valori predefiniti sono adatti per l'esecuzione di questo protocollo.
- Vai a Rivedi screening al termine dell'elaborazione dei dati e verifica se ci sono problemi relativi all'integrazione automatica, come la selezione di picchi cromatografici errati che potrebbero essere troppo vicini tra loro. In caso affermativo, restringere i valori riferiti alla tolleranza al tempo di ritenzione, alla tolleranza ai valori CCS e/o all'errore di massa inizialmente stabiliti nel file del metodo quantitativo e rielaborare i dati. Se l'integrazione automatica dei picchi continua a selezionare picchi errati, integrare manualmente il picco selezionando l'area nel cromatogramma che appare nella finestra Review Screening (a tale scopo, tenere conto dei parametri analitici ottenuti per gli standard EA nella sezione 5).
NOTA: È molto importante rivedere ogni campione, poiché alcune coppie EA-epimero principali hanno tempi di ritenzione molto simili e possono essere identificate erroneamente dal software. - Quando tutti i picchi sono stati integrati correttamente per tutti i campioni, fare clic sulla scheda Gestione batch nell'angolo in alto a sinistra e indicare il tipo di campione associato a ciascuna analisi: solvente, bianco, controllo qualità, campione o punto di calibrazione. In quest'ultimo caso, assegnare a ciascun punto di calibrazione un livello con un numero.
- Nella stessa scheda Gestione batch , accedere alla finestra inferiore denominata Concentrazione batch e specificare un valore di concentrazione per ogni livello stabilito nel passaggio precedente. Fatto ciò, fai clic su Salva concentrazioni.
- Fare clic su Quantifica batch e modificare i parametri come desiderato (ad esempio, il modello per adattarsi ai dati di calibrazione, la ponderazione, se il modello è costretto a superare lo zero, ecc.). I valori predefiniti sono adatti per l'esecuzione di questo protocollo, ad eccezione della ponderazione delle curve di calibrazione, che è stata commutata su 1/x.
NOTA: Poiché il software può considerare solo una curva di calibrazione alla volta, è necessario ripetere il passaggio 9.9 due volte per avere informazioni sulle curve di calibrazione standard e a matrice abbinata. In questo protocollo, sono state utilizzate curve di calibrazione a matrice abbinata per la quantificazione degli analiti, quindi le curve di calibrazione del solvente sono state valutate per prime, i dati di calibrazione sono stati estratti come spiegato più avanti nel passaggio 9.13 e, infine, le curve di calibrazione a matrice abbinata sono state selezionate per eseguire la quantificazione dei campioni. - Una volta eseguita la quantificazione degli alcaloidi dell'ergot secondo il file del metodo di quantificazione, fare clic sulla scheda Quantificazione nella parte superiore dello schermo.
- Se i parametri di default impostati al punto 9.9 non forniscono una buona linearità e precisione, regolare le curve di calibrazione modificando l'adattamento e il peso cliccando con il tasto destro del mouse sul grafico con l'obiettivo di avere un buon modello lineare (R2 > 0,99) con una precisione adeguata su ogni livello di calibrazione (deviazione inferiore al 20%).
NOTA: Il software segnerà in rosso le celle di quei livelli di calibrazione che non forniscono la precisione adeguata. - Una volta terminato, fai clic con il pulsante destro del mouse sulla cartella batch all'estrema sinistra dello schermo e seleziona Genera report. Quindi, selezionare Quantificazione analita per esportare il report nel formato desiderato.
Risultati
In primo luogo, le soluzioni standard funzionanti sono state iniettate nello strumento LC-IMS-MS per ottenere tutte le caratteristiche di identificazione (ad esempio, tempo di ritenzione, CCS e spettri di massa) di ciascun EA analizzato qui. Poiché i parametri di identificazione, ad eccezione della massa esatta, erano inizialmente sconosciuti, il metodo di acquisizione si è basato su un evento a due scansioni, iniziando con una scansione completa dell'intero spettro di massa seguita da...
Discussione
Il successo dell'utilizzo di questo protocollo si basa sull'ottimizzazione della procedura di estrazione, precedentemente effettuata da Carbonell-Rozas et al.17, che ha implementato l'uso di un solvente di estrazione abbastanza efficace da estrarre EA da matrici alimentari complesse come orzo e grano, e una bonifica che ha fornito valori di SSE relativamente bassi. La scelta del solvente da estrazione rappresenta un passaggio critico considerando le caratteristich...
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Questa ricerca è stata finanziata dalla Consejería de Universidad, Investigación e Innovación - Junta de Andalucía (PROYEXCEL_00195) e dalla borsa di studio post-dottorato della Generalitat Valenciana e del Fondo Sociale Europeo+ (CIAPOS/2022/049). Gli autori ringraziano il "Centro de Instrumentación Científica (CIC)" dell'Università di Granada per aver fornito l'accesso alla strumentazione analitica utilizzata in questo protocollo.
Materiali
| Name | Company | Catalog Number | Comments |
| Acetonitrile | VWR | 83640.32 | |
| Amber glass tubes 4 mL | VWR | 548-0052 | |
| Amber glass tubes 12 mL | VWR | 548-0903 | |
| Amber vials 1.5 mL | Agilent | 5190-9063 | |
| Ammonium carbonate | Fluka | 9716 | |
| Analytical balance BAS 31 | Boeco | 4400519 | |
| Balance CP 323 S | Sartorius | 23-84182 | |
| C18 | Supelco | 52604-U | |
| Centrifuge tubes, 15 mL | VWR | 525-1082 | |
| Centrifuge tubes, 50 mL | VWR | 525-0155 | |
| Centrifuge Universal 320 R | Hettich | 1406 | |
| Compass HyStar | Bruker | Acquisition software | |
| DataAnalysis | Bruker | Qualitative software | |
| Elute PLUS UHPLC | Bruker | ||
| EVA EC-S evaporator | VLM | V830.012.12 | |
| Formic acid GR for analysis ACS, Reag. Ph Eur | Merck | 100264 | |
| Grinder TitanMill300 | Cecotec | 1559 | |
| Methanol | VWR | 83638.32 | |
| Milli-Q water purification system (18.2 MΩ cm) | Millipore | ZD5211584 | |
| Pipette tips 1- 5 mL | Labortecnic | 162005 | |
| Pipette tips 100 - 1000 µL | Labortecnic | 1622222 | |
| Pipette tips 5 - 200 µL | Labortecnic | 162001 | |
| Pippette Transferpette S variable, DE-M 10 - 100 µL | BRAND | 704774 | |
| Pippette Transferpette S variable, DE-M 100 - 1000 µL | BRAND | 704780 | |
| Pippette Transferpette S variable, DE-M 500 - 5000 µL | BRAND | 704782 | |
| Syringe 2 mL | VWR | 613-2003 | |
| Syringe Filter 13 mm, 0.22µm | Phenomenex | AF-8-7707-12 | |
| TASQ | Bruker | Quantitative software | |
| timsTOFPro2 IM-HRMS | Bruker | ||
| Vortex Genie 2 | Scientific Industries | 15547335 | |
| Zorbax Eclipse Plus RRHD C18 column (50 x 2.1 mm, 1.8 µm particle size) | Agilent | 959757-902 | |
| Z-Sep+ | Supelco | 55299-U | Zirconia-based sorbent |
| Ergot alkaloids | CAS registry sorbent | ||
| Ergocornine (Eco) | Techno Spec | E178 | 564-36-3 |
| Ergocorninine (Econ) | Techno Spec | E130 | 564-37-4 |
| Ergocristine (Ecr) | Techno Spec | E180 | 511-08-0 |
| Ergocristinine (Ecrn) | Techno Spec | E188 | 511-07-9 |
| Ergokryptine (Ekr) | Techno Spec | E198 | 511-09-1 |
| Ergopkryptinine (Ekrn) | Techno Spec | E190 | 511-10-4 |
| Ergometrine (Em) | Romer Labs | "002067" | 60-79-7 |
| Ergometrinine (Emn) | Romer Labs | LMY-090-5ML | 479-00-5 |
| Ergosine (Es) | Techno Spec | E184 | 561-94-4 |
| Ergosinine (Esn) | Techno Spec | E194 | 596-88-3 |
| Ergotamine (Et) | Romer Labs | "002069" | 113-15-5 |
| Ergotaminine (Etn) | Romer Labs | "002075" | 639-81-6 |
Riferimenti
- Kanu, A. B., Dwivedi, P., Tam, M., Matz, L., Hill, H. H. Ion mobility-mass spectrometry. J Mass Spectrom. 43 (1), 1-22 (2008).
- Gabelica, V., et al. Recommendations for reporting ion mobility Mass spectrometry measurements. Mass Spectrom Rev. 38 (3), 291-320 (2019).
- Feuerstein, M. L., et al. Comparability of steroid collision cross sections using three different IM-HRMS technologies: An interplatform study. J Am Soc Mass Spectrom. 33 (10), 1951-1959 (2022).
- Regueiro, J., Negreira, N., Berntssen, M. H. G. Ion-mobility-derived collision cross section as an additional identification point for multiresidue screening of pesticides in fish feed. Anal Chem. 88 (22), 11169-11177 (2016).
- Regueiro, J., Negreira, N., Hannisdal, R., Berntssen, M. H. G. Targeted approach for qualitative screening of pesticides in salmon feed by liquid chromatography coupled to traveling-wave ion mobility/quadrupole time-of-flight mass spectrometry. Food Control. 78, 116-125 (2017).
- Hernández-Mesa, M., Monteau, F., Le Bizec, B., Dervilly-Pinel, G. Potential of ion mobility-mass spectrometry for both targeted and non-targeted analysis of phase II steroid metabolites in urine. Anal Chim Acta: X. 1, 100006 (2019).
- George, A. C., et al. Interplatform comparison between three ion mobility techniques for human plasma lipid collision cross sections. Anal Chim Acta. 1304, 342535 (2024).
- Mairinger, T., Causon, T. J., Hann, S. The potential of ion mobility-mass spectrometry for non-targeted metabolomics. Curr Opin Chem Biol. 42, 9-15 (2018).
- Hernández-Mesa, M., Escourrou, A., Monteau, F., Le Bizec, B., Dervilly-Pinel, G. Current applications and perspectives of ion mobility spectrometry to answer chemical food safety issues. TrAC Trend Anal Chem. 94, 39-53 (2017).
- European, C. Commission Regulation (EU) 2023/915 of 25 April 2023 on maximum levels for certain contaminants in food and repealing Regulation (EC) No 1881/2006. Off J Eur Union. 119, 103-157 (2023).
- Carbonell-Rozas, L., et al. Occurrence of egot alkaloids in major and minor cereals from Northern Italy: A three harvesting years scenario. J Agricl Food Chem. 71 (42), 15821-15828 (2023).
- Arroyo-Manzanares, N., Gámiz-Gracia, L., García-Campaña, A. M., Diana Di Mavungu, J., De Saeger, S., Jean-Michel Mérillon, J. -. M., Ramawat, K. G. . Fungal Metabolites. , (2016).
- van Dongen, P., de Groot, A. History of ergot alkaloids from ergotism to ergometrine. Eur J Obstet Gynecol Reprod Biol. 60 (2), 109-116 (1995).
- European Food Safety Authority. Human and animal dietary exposure to ergot alkaloids. EFSA J. 15 (7), e04902 (2017).
- Nam, M., Kim, D., Kim, M. -. S. Simultaneous determination of total ergot alkaloids in wheat flour by Orbitrap mass spectrometry. Food Chem. 441, 138363 (2024).
- García-Juan, A., León, N., Armenta, S., Pardo, O. Development and validation of an analytical method for the simultaneous determination of 12 ergot, 2 tropane, and 28 pyrrolizidine alkaloids in cereal-based food by LC-MS/MS. Food Res Int. 174 (Pt 1), 113614 (2023).
- Carbonell-Rozas, L., Mahdjoubi, C. K., Arroyo-Manzanares, N., García-Campaña, A. M., Gámiz-Gracia, L. Occurrence of ergot alkaloids in barley and wheat from Algeria. Toxins. 13 (5), 316 (2021).
- Carbonell-Rozas, L., Gámiz-Gracia, L., Lara, F. J., García-Campaña, A. M. Determination of the main ergot alkaloids and their epimers in oat-based functional foods by ultra-high performance liquid chromatography tandem mass spectrometry. Molecules. 26 (12), 3717 (2021).
- Carbonell-Rozas, L., et al. Ion mobility-mass spectrometry to extend analytical performance in the determination of ergot alkaloids in cereal samples. J Chromatogr A. 1682, 463502 (2022).
- European Commission. Commission Implementing Regulation (EU) 2023/2782 of 14 December 2023 laying down the methods of sampling and analysis for the control of the levels of mycotoxins in food and repealing Regulation (EC) No 401/2006. Off J Eur Union. , (2023).
- Carbonell-Rozas, L., Vander Cruyssen, L., Dall'Asta, C., Leggieri, M. C., Battilani, P. Fit-for-purpose method development to determine co-occurring multiclass mycotoxins in apple and apple puree samples. Food Anal Methods. 16 (8), 1403-1412 (2023).
- Laouni, C., et al. Emerging mycotoxin occurrence in chicken feed and eggs from Algeria. Mycotoxin Res. 40, 447-456 (2024).
- Ben Hassouna, K., et al. Mycotoxin occurrence in milk and durum wheat samples from Tunisia using dispersive liquid-liquid microextraction and liquid chromatography with fluorescence detection. Toxins. 15 (11), 633 (2023).
- Narváez, A., et al. Occurrence and exposure assessment of mycotoxins in ready-to-eat tree nut products through ultra-high performance liquid chromatography coupled with high resolution Q-orbitrap mass spectrometry. Metabolites. 10 (9), 344 (2020).
- Arroyo-Manzanares, N., Rodríguez-Estévez, V., García-Campaña, A. M., Castellón-Rendón, E., Gámiz-Gracia, L. Determination of principal ergot alkaloids in swine feeding. J Sci Food Agric. 101 (12), 5214-5224 (2021).
- Pereira, V. L., Fernandes, J. O., Cunha, S. C. Comparative assessment of three cleanup procedures after QuEChERS extraction for determination of trichothecenes (type A and type B) in processed cereal-based baby foods by GC-MS. Food Chem. 182, 143-149 (2015).
- Tuzimski, T., Szubartowski, S. Method development for selected bisphenols analysis in sweetened condensed milk from a can and breast milk samples by HPLC-DAD and HPLC-QqQ-MS: Comparison of sorbents (Z-SEP, Z-SEP Plus, PSA, C18, chitin and EMR-lipid) for clean-up of QuEChERS extract. Molecules. 24 (11), 2093 (2019).
- Łozowicka, B., Rutkowska, E., Jankowska, M. Influence of QuEChERS modifications on recovery and matrix effect during the multi-residue pesticide analysis in soil by GC/MS/MS and GC/ECD/NPD. Environ Sci Pollut Res. 24 (8), 7124-7138 (2017).
- Schummer, C., Xandonella, I., van Nieuwenhuyse, A., Moris, G. Epimerization of ergot alkaloids in feed. Heliyon. 6 (6), e04336 (2020).
- Cherewyk, J. E., Grusie-Ogilvie, T. J., Parker, S. E., Blakley, B. R., Al-Dissi, A. M. The impact of storage temperature and time on ergot alkaloid concentrations. Toxins. 15 (8), 497 (2023).
- Silva, &. #. 1. 9. 4. ;., Mateus, A. R., Barros, S. C., Silva, A. S. Ergot alkaloids on cereals and seeds: Analytical methods, occurrence, and future perspectives. Molecules. 28 (20), 7233 (2023).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon