Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Détection d’alcaloïdes d’ergot régulés dans les matrices alimentaires par chromatographie en phase liquide, spectrométrie de mobilité ionique piégée-spectrométrie de masse à temps de vol
Dans cet article
Résumé
Ce protocole présente une méthode validée de chromatographie en phase liquide, de mobilité ionique et de spectrométrie de masse à haute résolution pour déterminer la présence d’alcaloïdes de l’ergot dans les aliments, conformément au règlement (UE) 2023/915 de la Commission récemment publié.
Résumé
La spectrométrie de masse à mobilité ionique (IMS) agit comme une dimension de séparation supplémentaire lorsqu’elle est intégrée dans les flux de travail de chromatographie liquide-spectrométrie de masse (LC-MS). Les méthodes LC-IMS-MS offrent une résolution de crête plus élevée, une séparation améliorée des composés isobares et isomères et un meilleur rapport signal/bruit (S/N) par rapport aux méthodes LC-MS traditionnelles. L’IMS fournit une autre caractéristique moléculaire pour l’identification des analytes, à savoir le paramètre de section efficace de collision (CCS), réduisant ainsi les résultats faussement positifs. Par conséquent, les méthodes LC-IMS-MS relèvent d’importants défis analytiques dans le domaine de la sécurité alimentaire (c’est-à-dire la détection de composés à l’état de traces dans des matrices alimentaires complexes et l’identification sans ambiguïté des molécules isobares et isomères).
Les alcaloïdes de l’ergot (EA) sont une famille de mycotoxines produites par des champignons qui attaquent une grande variété d’espèces de graminées, y compris les petites céréales telles que le seigle, le triticale, le blé, l’orge, le millet et l’avoine. Des teneurs maximales (CM) de ces mycotoxines ont été établies dans plusieurs denrées alimentaires, comme détaillé dans le règlement (CE/2023/915) de la Commission. Cette nouvelle législation comprend six EA principaux et leurs épimères correspondants, de sorte qu’une méthodologie efficace est nécessaire pour bien distinguer ces molécules isomères compte tenu de leur cooccurrence.
Par conséquent, l’objectif de ce protocole est de montrer comment l’intégration de l’IMS dans les flux de travail LC-MS contribue à la séparation des EA isomériques, améliorant ainsi la sélectivité de la méthode analytique. De plus, il illustre comment la génération de banques de CSC par la caractérisation d’étalons analytiques offre une plus grande confiance pour l’identification des mycotoxines. Ce protocole est conçu pour expliquer clairement les avantages de la mise en œuvre de l’EMI en matière de salubrité des aliments, en prenant comme exemple la détermination des EA dans les céréales. Une extraction basée sur QuEChERS suivie d’une analyse par spectrométrie de mobilité ionique piégée par LC (TIMS)-MS a fourni des limites de quantification allant de 0,65 à 2,6 ng/g avec une précision acceptable (bien qu’une faible récupération pour l’ergotaminine) à 1,5x, 1x et 0,5x le ML et a montré un effet de matrice négligeable.
Introduction
La spectrométrie de masse à mobilité ionique (IMS) est en train de devenir une technique analytique de plus en plus utilisée, souvent présentée comme une dimension de séparation supplémentaire intégrée à la chromatographie liquide/gazeuse traditionnelle (LC/GC) couplée aux flux de travail MS. L’IMS consiste en la séparation de molécules le long d’une cellule de mobilité, remplie d’un gaz tampon, sous un champ électrique et à la pression atmosphérique1. En fonction du rapport masse/charge (m/z) et de la conformation géométrique, une molécule ionisée interagira avec le gaz tampon lorsqu’elle se déplacera à travers la cellule de mobilité, ce qui est reflété dans le paramètre de mobilité ionique (K)2 et calculé à l’aide de l’équation suivante :
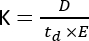
où D représente la longueur totale de la dérive, td est le temps total de dérive et E est le champ électrique. Par conséquent, K est mesuré en m2 V−1 s−1, bien que pour des raisons pratiques, il soit souvent exprimé en cm2 V−1 s−1. La capacité intrinsèque de se déplacer à travers la cellule de mobilité peut être mesurée par le temps de dérive, puis convertie en valeur de section efficace de collision (CCS), qui est un paramètre hautement reproductible pour chaque molécule indépendamment de l’instrument IMS3. Le CCS peut être dérivé de la mobilité en suivant cette équation :
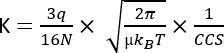
q étant la charge de l’ion ; N la densité en nombre du gaz tampon ; μ la masse réduite des partenaires de collision tampon gaz-ion ; kB la constante de Boltzmann ; et T , la température du gaz tampon. Par conséquent, l’IMS fournit des informations supplémentaires en complément des données analytiques résultant des analyses par chromatographie et MS.
Il a été démontré que la mise en œuvre de l’IMS dans les plateformes LC-MS augmente la fiabilité des déterminations analytiques, en particulier lorsque l’on travaille avec des composés à l’état de traces. Plusieurs études ont montré que les méthodes LC-IMS-MS améliorent la qualité des spectres de masse en réduisant le bruit de fond, ce qui affecte en fin de compte la sensibilité de la méthode et réduit le taux de faux positifs et négatifs fourni par les méthodologies LC-MS multi-résidus 4,5,6. De plus, la reproductibilité des valeurs CCS permet de comparer non seulement différents instruments utilisant la même technologie, mais aussi entre différentes technologies de mobilité ionique, à savoir la spectrométrie de mobilité ionique à ondes progressives (TWIMS), la spectrométrie de mobilité d’ions piégés (TIMS) et la spectrométrie de mobilité d’ions de tube dérivant (DTIMS)2,7, qui sont les systèmes les plus fréquemment utilisés1. Ainsi, une conséquence remarquable du potentiel du CCS en tant que paramètre d’identification réside dans la possibilité de construire des bibliothèques CCS, ce qui se reflète dans son applicabilité dans les études métabolomiques8. Néanmoins, l’une des caractéristiques les plus puissantes de l’IMS est la capacité de séparer les composés isomères et isobares qui peuvent ne pas être suffisamment résolus par les méthodes LC-MS. Cela peut être le cas lorsque l’on travaille avec de grands ensembles d’analytes d’intérêt dans des matrices complexes, ce qui est une situation courante dans l’analyse environnementale et alimentaire. Dans ce contexte, des méthodes LC-IMS-MS ont été proposées pour la surveillance des pesticides et, dans une moindre mesure, des médicaments vétérinaires et des mycotoxines dans les aliments9.
En raison de leur pouvoir de résolution et de leur sélectivité élevés, les plateformes LC/GC-IMS-MS apparaissent comme les outils les plus utiles pour relever certains des défis actuels en matière de sécurité alimentaire, en particulier ceux liés aux mélanges isomères. Les préoccupations sanitaires liées aux mélanges isomères en tant que contaminants alimentaires ont été reflétées dans la législation européenne actuelle, qui, par exemple, limite la concentration maximale de six principaux alcaloïdes de l’ergot (EA) et de leurs six épimères correspondants dans plusieurs produits alimentaires10.
Les EA constituent une famille de métabolites secondaires toxiques produits par un large éventail de champignons, principalement de la famille des Clavicipitaceae (par exemple, Claviceps purpurea, le plus important producteur d’EA en raison de sa large gamme d’hôtes), mais aussi Trichocomaceae, qui peuvent parasiter l’épi des graines des plantes vivantes (comme le seigle, l’orge, le blé et l’avoine) au moment de la floraison11, 12. Épisode 12 Dans des conditions spécifiques, en particulier la température et l’activité de l’eau, les champignons Claviceps peuvent produire des EA qui s’accumulent dans les fructifications, appelées sclérotes ou ergot, dans la culture hôte. Dans une certaine mesure, les EA peuvent résister au traitement de la matière première jusqu’à l’obtention du produit final ; par conséquent, entrer dans la chaîne alimentaire. L’ingestion d’aliments contaminés peut entraîner une intoxication à l’EA, connue sous le nom d’ergotisme, qui se manifeste par des symptômes aigus tels que des douleurs abdominales, des vomissements, une sensation de brûlure de la peau, de l’insomnie et des hallucinations13. Afin de réduire l’impact des EA sur la santé humaine, la Commission européenne a fixé des teneurs maximales (CM) dans plusieurs aliments pour la somme des principaux EA : les épimères R ergométrine (Em), ergotamine (Et), ergosine (Es), ergocristine (Ecr), ergokryptine (Ekr) et ergocornine (Eco) et leurs S-épimères correspondants : ergométrinine (Emn), ergosinine (Esn), ergotaminine (Etn), ergocorninine (Econ), ergokryptine (Ekrn), et l’ergocristinine (Ecrn). Ces composés peuvent s’épimériser des formes R à S et vice versa, en particulier en cas d’exposition à une lumière forte, de stockage prolongé ou de contact avec certains solvants à pH 12 élevé ou faible. Bien que la proportion de formes R et S puisse varier dans différentes conditions, le groupe scientifique CONTAM de l’EFSA a signalé une occurrence plus élevée de formes R que de formes S après avoir examiné la littérature disponible sur les EA dans les produits alimentaires14. Par conséquent, les CM varient en fonction de plusieurs facteurs, tels que la sensibilité de la culture, le degré de transformation ou la fréquence de consommation. Dans le cadre de l’UE, les CM pour les produits moulus d’orge, de blé, d’épeautre et d’avoine ont été fixées à 50 ou 150 μg/kg (en fonction de la teneur en cendres inférieure ou supérieure à 900 mg/100 g, respectivement), tandis que les céréales destinées directement à la consommation humaine sont soumises à une CM de 150 μg/kg, à l’exception des aliments pour bébés à base de céréales. dans lequel la CM est réduite à 20 μg/kg10.
Cette législation stricte exige des méthodologies analytiques suffisamment sensibles pour déterminer les concentrations à l’état de traces (μg/kg) tout en identifiant correctement les EA réglementés et leurs épimères correspondants, car les deux formes, les isomères R et S, peuvent être trouvées ensemble dans les échantillons contaminés. Cette tâche représente un défi majeur puisque chaque paire toxine-épimère partage exactement la même masse et le même schéma de fragmentation. De plus, une séparation chromatographique correcte entre les deux composés peut être complexe. Par conséquent, des gradients de LC bien optimisés sont nécessaires pour éviter les erreurs de quantification lorsque les épimères EA coexistent dans des échantillons alimentaires. Bien que plusieurs études aient rapporté des méthodes LC-MS pour la détermination sans ambiguïté des EA15, 16, 17, 18, la méthode chromatographique doit être étudiée de manière approfondie pour obtenir une séparation adéquate des pics chromatographiques afin d’identifier sans équivoque les EA. Cependant, cela n’est généralement pas possible pour les méthodes à plusieurs classes dans lesquelles les contaminants appartenant à différentes familles chimiques sont déterminés simultanément. Dans ce contexte, une étude récente menée par Carbonell-Rozas, Hernández-Mesa et al.19 a fait état d’une méthode LC-IMS-MS pour la quantification des EA dans les échantillons de blé et d’orge, à l’aide de deux instruments TWIMS différents qui ont fourni des valeurs CCS reproductibles et des limites basses de quantification (LOQ) pour détecter toute non-conformité conformément à la législation en vigueur. Par conséquent, l’objectif de ce protocole est de montrer comment l’intégration de l’IMS dans les flux de travail LC-MS contribue à la séparation des EA isomériques, améliorant ainsi la sélectivité de la méthode analytique. De plus, il illustre comment la génération de banques de CSC par la caractérisation des étalons analytiques offre une plus grande confiance pour l’identification des mycotoxines. Ce protocole est conçu pour expliquer clairement les avantages de la mise en œuvre de l’IMS dans l’analyse de la sécurité alimentaire, en prenant comme exemple la détermination des EA dans les céréales. Ce protocole traite du traitement des échantillons sur la base d’une procédure QuEChERS, de l’analyse des échantillons par LC-TIMS-MS et de l’extraction et de l’interprétation des données IMS.
Protocole
1. Préparation des solutions mères, intermédiaires et standard de travail
REMARQUE : Utilisez des gants en nitrile, une blouse de laboratoire et des lunettes de sécurité.
- Préparer des solutions mères individuelles des 12 EA (voir le tableau des matières) à 10 000 ng/mL dans des flacons en verre ambré de 4 mL à l’aide d’acétonitrile. Les formes R (-ine) étaient auparavant aliquotes en portions de 25 000 ng, tandis que les formes S (-inine) étaient distribuées en portions de 10 000 ng. Cette étude a commencé avec ces aliquotes, atteignant des concentrations de 10 000 ng/mL en diluant les formes R et S dans 2,5 et 1 mL d’acétonitrile, respectivement, suivie d’un vortex pendant 2 min.
- Préparer une solution mère intermédiaire à raison de 1 000 ng/mL d’EA totaux (83,33 ng/mL de chaque) dans un flacon en verre ambré de 12 mL en ajoutant 65 μL de chaque solution d’alcaloïde individuelle et 7,02 mL d’acétonitrile.
REMARQUE : Une fois la solution mère intermédiaire préparée, les solutions mères individuelles doivent être séchées et entreposées à -20 °C recouvertes d’une feuille d’aluminium pour éviter l’épimérisation. - Préparez des solutions étalons de travail à partir de la solution mère intermédiaire en pipetant 2,7, 1,8 et 0,9 ml dans des tubes en verre ambré de 4 ml. Mettez-les à sec sous un léger jet d’azote et mettez-les en suspension dans 600 μL d’acétonitrile.
REMARQUE : Les concentrations résultantes ont été établies de manière à atteindre 1,5, 1 et 0,5 fois le mL (150 ng/g) en ajoutant 50 μL à 1 g de l’échantillon. Ceux-ci sont choisis comme niveaux de concentration pour les études de validation (c.-à-d. récupérations, répétabilité, etc.). - Répartissez les 600 μL de chaque solution étalon de travail dans trois flacons en verre ambré de 2 mL en pipetant 200 μL et portez-les à sec.
REMARQUE : Les étapes 1.2 à 1.4 sont expliquées schématiquement à la figure 1. En divisant et en séchant les solutions étalons fonctionnelles, il est possible de fortifier en trois exemplaires sur trois jours différents, comme l’exige l’étude de validation, sans perdre l’intégrité chimique des EA. - Chaque jour de validation, remettre en suspension un flacon en verre ambré de 2 mL de chaque niveau de fortification dans 200 μL d’acétonitrile.
REMARQUE : Chaque niveau d’enrichissement sera évalué en trois exemplaires à l’aide de 50 μL par échantillon ; par conséquent, un total de 150 μL de la solution sera utilisé.
2. Préparation des réactifs et des solutions
REMARQUE : Utilisez des gants en nitrile, une blouse de laboratoire et des lunettes de sécurité.
- Préparez une solution de carbonate d’ammonium de 5 mM en pesant 24,02 mg de carbonate d’ammonium et en la dissolvant dans 50 mL d’eau. Peser 24,02 mg de carbonate d’ammonium dans un bécher et ajouter environ 5 ml d’eau. Dissoudre le carbonate d’ammonium en agitant doucement la solution manuellement, puis la transférer dans une fiole jaugée de 50 ml. Pour atteindre le volume final de 50 ml, rincer le bécher avec de l’eau supplémentaire et transférer la levée dans la fiole jaugée.
- Préparez 250 mL de la solution d’extraction [acétonitrile : 5 mM de carbonate d’ammonium (85:15, v/v)] en mélangeant 212,5 mL d’acétonitrile et 37,5 mL de la solution d’ammonium préparée à l’étape 2.1.
- Préparez 100 mL d’un mélange méthanol/eau (1:1, v/v) pour remettre en suspension les extraits d’échantillons (étape 8.8.).
- Pour chaque échantillon, peser 150 mg d’un mélange de sorbant à base de C18 :zircone (1:1, p/p) dans des tubes de centrifugation de 15 mL pour l’étape de nettoyage dispersif (étape 8.5.).
- Préparez les solvants pour la séparation chromatographique dans deux flacons différents, à savoir 1 L d’eau ultrapure (solvant A) et de méthanol (solvant B), tous deux contenant 0,3 % (v/v) d’acide formique. Pour atteindre cette concentration, 3,03 mL d’acide formique (pureté de 99 %) ont été ajoutés aux bouteilles.
REMARQUE : Les solvants pour la phase mobile doivent être préparés juste avant d’effectuer les analyses LC-IMS-MS.
3. Réglage des paramètres instrumentaux
REMARQUE : L’instrument utilisé pour réaliser cette étude LC-IMS-MS était un UHPLC couplé à un IM-HRMS, équipé d’une source d’ionisation par électronébulisation par sonde chauffée par sonde isolée sous vide (VIP-HESI). L’instrument fonctionnait en mode positif.
- Créer une méthode chromatographique à l’aide du logiciel d’acquisition pour la séparation des analytes en réglant le gradient d’élution suivant : 0 min, 10 % B ; 2 min, 10 % B ; 4,5 min, 40 % B ; 9 min, 45 % B ; 11 min, 95 % B ; 12 min, 95 % B ; 13 min, 10 % B ; 16 min, 10 % B.
- Réglez le débit de la phase mobile à 0,4 mL/min, la température de la colonne à 35 °C et le volume d’injection à 5 μL. Si possible, maintenez une température fixe de 10 °C à l’intérieur de l’échantillonneur automatique.
- Réglez les paramètres IMS comme suit : valeurs de mobilité (1/K0), de 0,1 à 1,5 V·s/cm2 ; temps de rampe, 100 ms ; temps de précision, 10 ms ; cycle de service, 10 % ; et taux de rampe, 9,05 Hz.
- Réglez les paramètres de la source ionique pour la détection MS comme suit : tension capillaire, 2 500 V ; pression du nébuliseur, 2,5 bar ; débit de gaz sec, 8 L/min ; température des gaz secs, 200 °C ; température du gaz de la gaine, 450 °C ; débit de gaz de gaine, 4 L/min. Pour l’acquisition MS, sélectionnez les paramètres suivants, en tenant compte de deux événements de balayage : le balayage complet de l’ensemble du spectre de masse avec un m/z de 20 à 1 300 suivi de la bbCID (dissociation induite par collision à large bande), qui génère des ions produits à partir de n’importe quel précurseur à trois énergies de collision fixes de 24, 36 et 50 eV. Effectuez l’ionisation et l’acquisition de données en mode de polarité positive.
- Enregistrez ce fichier de méthode d’acquisition pour une utilisation ultérieure.
4. Acquisition de données à partir des normes analytiques des EA
REMARQUE : Utilisez des gants en nitrile, une blouse de laboratoire et des lunettes de sécurité pour l’étape 4.1 uniquement.
- Préparez deux fioles ambrées de 1,5 mL avec 500 μL de solution intermédiaire à 1 000 ng/mL et une autre fiole avec un mélange de solvants A et B (1:1, v/v) comme solution de lavage.
- Étalonnez l’instrument timsTOF comme indiqué par le fabricant et sélectionnez la polarité positive pour les acquisitions IMS et MS.
- Placez la colonne C18 (voir tableau des matériaux) à l’intérieur du four LC, avec la température réglée sur 35 °C comme indiqué dans le fichier de méthode d’acquisition enregistré à l’étape 3.5.
REMARQUE : Assurez-vous que les deux extrémités sont bien serrées ; Sinon, une fuite de phase mobile sera observée. - Conditionnez la colonne LC de manière à ce qu’elle permette un débit de phase mobile de 0,1 mL/min et choisissez un rapport de mélange de solvant A et B de 90:10 ( %) comme rapport initial de composition de la phase mobile.
- Attendez que la pression de la colonne soit stable (~130 bar).
- Augmentez le débit de la phase mobile à 0,2 mL/min, attendez que la pression se stabilise et augmentez-le à nouveau à 0,3 mL/min.
- Une fois que la pression se stabilise à nouveau, réglez le débit à 0,4 mL, ce qui sera le débit de travail comme indiqué dans le fichier de méthode d’acquisition enregistré à l’étape 3.5, et attendez que la pression se stabilise autour de 350 bars.
REMARQUE : Il est important de ne pas augmenter directement le débit de 0,2 à 0,4 mL/min. Un fort changement dans le débit provoque des pics de haute pression qui peuvent endommager la colonne de chromatographie. - Pendant le conditionnement de la colonne, rédiger la liste de travail, y compris deux échantillons correspondant aux deux flacons contenant la solution intermédiaire d’EAs à 1 000 ng/mL, comme indiqué à l’étape 4.1, et au flacon de lavage. Analysez le flacon de lavage deux fois, au début et à la fin du lot d’échantillons.
- Dans la colonne Fichier de méthode de la liste de travail, chargez le fichier de méthode d’acquisition précédemment enregistré à l’étape 3.5.
- Créez un dossier pour enregistrer tous les fichiers de données acquis et acheminer tous les échantillons en sélectionnant le dossier dans la colonne Chemin d’accès à l’échantillon .
REMARQUE : Assurez-vous d’utiliser le même dossier pour enregistrer tous les fichiers générés au cours de l’étude de validation en vue d’un traitement ultérieur. Lors du conditionnement de la colonne, les valeurs auxquelles la pression se stabilise peuvent varier légèrement. Néanmoins, assurez-vous que la pression est stable avant d’augmenter à nouveau le débit pour préserver l’intégrité de la colonne. De plus, lorsque le débit de travail est atteint, la pression doit également être stable pour obtenir une séparation chromatographique reproductible. - Allez dans la barre d’outils supérieure du logiciel et exécutez la liste de travail.
5. Traitement des données pour la création d’une méthode de quantification
- Une fois la liste de travail exécutée, ouvrez le logiciel qualitatif.
- Chargez tous les échantillons analysés en cliquant sur Fichier | Ouvrez la liste de travail.
- Faites un clic droit sur un échantillon correspondant à la solution intermédiaire d’EAs à 1 000 ng/mL, allez dans Modifier le chromatogramme, puis sélectionnez Type | Chromatogramme ionique extrait, et entrez la masse moléculaire théorique de l’ion protoné lié à chaque EA. Ensuite, cliquez sur Ajouter, et enfin, OK. Étant donné que l’ensemble des analytes est formé de six EA et de leurs épimères apparentés qui partagent exactement la même masse, six chromatogrammes ioniques extraits apparaîtront.
REMARQUE : Le paramètre Largeur modifie l’étroitesse de la fenêtre m/z . Il est recommandé de sélectionner une fenêtre m/z suffisamment large en cas de difficultés à trouver le pic chromatographique de l’analyte d’intérêt. Cependant, il est conseillé de définir des fenêtres m/z étroites pour éviter d’éventuelles interférences (inférieures à 0,05 unités). - Dans chaque chromatogramme ionique extrait, deux pics chromatographiques liés à l’EA principal et à son épimère apparaîtront. Faites un clic droit tout en sélectionnant toute la zone d’un pic. Par conséquent, le spectre de mobilité ionique apparaîtra juste en dessous.
- Allez sur l’axe des x du spectre de mobilité ionique, cliquez avec le bouton droit de la souris, puis cliquez sur Section transversale de collision. Notez la valeur CCS.
REMARQUE : Si les valeurs CCS n’apparaissent pas automatiquement, allez dans Calibrer dans la barre d’outils supérieure, puis Calculatrice de mobilité, et tapez manuellement la valeur de mobilité pour obtenir leur CCS correspondant. - Sélectionnez la zone correspondant à l’autre pic chromatographique dans le même chromatogramme ionique et répétez les étapes 5.4. et 5.5.
- De même, le modèle de fragmentation apparaîtra également après l’étape 5.4. Sur la base du modèle de fragmentation rapporté dans la littérature et les bases de données, comparez et choisissez l’ion produit le plus intense comme point d’identification complémentaire.
- Notez le temps de rétention, la valeur CCS et la masse exacte de l’adduit ionique principal (généralement l’ion protoné) de chaque EA pour créer une méthode de quantification.
- Répétez les étapes 5.4 à 5.8. pour les analytes restants.
- Sélectionnez toutes les masses moléculaires générées à l’étape 5.3 en cliquant dessus tout en appuyant sur Ctrl. Ensuite, cliquez avec le bouton droit de la souris et sélectionnez Copier. Allez dans l’autre exemple correspondant à la solution intermédiaire EAs, faites un clic droit dessus et sélectionnez Coller. Les chromatogrammes ioniques et les spectres de mobilité seront automatiquement extraits.
- Vérifiez les paramètres d’identification de tous les analytes de ce deuxième échantillon.
6. Création d’une méthode de traitement de données pour le dosage de routine des alcaloïdes de l’ergot de seigle
- Ouvrez le logiciel et cliquez sur l’onglet Gestion des méthodes .
- Cliquez sur Paramètres de l’analyte et entrez le nom de chaque EA ainsi que son temps de rétention, sa valeur CCS et la masse des adduits protonés collectés à l’étape 5.4. à 5.9. Modifiez la tolérance de chaque point d’identification si nécessaire, bien que les paramètres par défaut soient recommandés pour l’application de ce protocole.
REMARQUE : Plus les valeurs de tolérance définies sont élevées, plus la possibilité de traiter des faux positifs est élevée, car un écart plus important par rapport aux valeurs théoriques est accepté. De même, des valeurs de tolérance trop strictes peuvent conduire à des faux négatifs. Cela se produit lorsque l’écart par rapport à la valeur théorique est uniquement causé par les performances de l’instrument. - Conservez la méthode de quantification pour une utilisation ultérieure.
7. Échantillonnage
- Acquérir des échantillons de blé et d’avoine dans les supermarchés, de préférence dans des récipients de 1 kg, en suivant les directives d’échantillonnage établies par la Commission européenne20. Si des colis de 1 kg ne sont pas disponibles, assurez-vous simplement d’avoir, au moins, 50 g d’échantillons pour compléter l’ensemble de l’analyse.
- Broyez les échantillons à l’aide d’un broyeur. Dans cette étude, une meuleuse à lames a été utilisée pour fraiser tout le contenu des emballages, qui ont ensuite été retournés dans leurs emballages d’origine pour homogénéisation.
REMARQUE : Nettoyez soigneusement le broyeur entre les échantillons pour éviter la contamination croisée. Utilisez une blouse de laboratoire. - Aliquote les échantillons homogénéisés dans des tubes à centrifuger de 50 mL et stockez-les dans des conditions fraîches et sèches.
8. Préparation de l’échantillon
- Préparez un calendrier pour réaliser l’étude de validation pendant trois jours non consécutifs (afin d’évaluer la reproductibilité à l’interne). Pour chaque jour, dosez trois niveaux de concentration en fortifiant les échantillons à 225 ng/g (1,5 mL), 150 ng/g (ML) et 75 ng/g (0,5 mL). Étudiez chaque niveau en trois exemplaires, ce qui représente neuf échantillons enrichis et trois autres échantillons vierges par jour.
REMARQUE : Compte tenu des 3 jours de validation, un total de 36 échantillons sera nécessaire ainsi que 8 autres échantillons vierges destinés à la préparation de la courbe d’étalonnage appariée à la matrice. Utilisez des gants en nitrile, une blouse de laboratoire et des lunettes de sécurité pour toute cette section du protocole. - Peser 1 g de l’échantillon dans un tube à centrifuger de 50 ml.
REMARQUE : Lors de l’exécution de l’étude de validation, renforcez les échantillons juste après la pesée avec 50 μL de la solution étalon de travail correspondante détaillée aux étapes 1.3. et 1.5. - Ajouter 4 mL de la solution d’extraction [acétonitrile : 5 mM de solution aqueuse de carbonate d’ammonium (85:15, v/v)]. Vortex l’échantillon pendant 1 min.
- Centrifuger l’échantillon pendant 5 min à 9 750 × g et 4 °C.
REMARQUE : La capacité de la centrifugeuse peut être un facteur limitant dans la procédure, il est donc recommandé d’extraire simultanément uniquement le nombre exact d’échantillons qui tiendront dans la centrifugeuse. - Transvasez le surnageant entier dans le tube à centrifuger de 15 mL contenant 150 mg du mélange de sorbant de nettoyage (1:1, p/p). Vortex l’échantillon pendant 1 min.
- Centrifugez le tube pendant 5 min à 9 750 × g et 4 °C.
- Prélever le surnageant et le placer dans un flacon en verre ambré de 4 ml. Évaporez l’extrait sous un léger jet d’azote.
REMARQUE : Les échantillons doivent être conservés au sec à -20 °C s’ils ne sont pas analysés le même jour. - Reconstituer l’échantillon dans 750 μL du mélange méthanol/eau (1:1, v/v).
- Transférez l’extrait dans une seringue de 2 mL et filtrez-le à travers un filtre en nylon de 0,22 μm dans un flacon chromatographique ambré de 1,5 mL.
- Recueillir les extraits reconstitués des huit échantillons à blanc destinés à la courbe d’étalonnage matricielle appariée dans un flacon en verre ambré de 12 mL (chaque échantillon est remis en suspension dans 750 μL, ce qui représente un volume total de 6 mL).
- Pipeter 1 mL de la solution intermédiaire à 1 000 ng/mL (83,33 ng/mL pour chaque alcaloïde de l’ergot) (étape 1.2) dans un flacon en verre ambré de 2 mL, sécher le solvant sous un léger jet d’azote et remettre en suspension dans 1 mL l’extrait d’échantillon blanc précédemment prélevé.
REMARQUE : Ce mélange à une concentration de 1 000 ng/mL (83,33 ng/mL pour chaque EA) sera utilisé pour préparer la courbe d’étalonnage appariée à la matrice. - Préparez dix flacons en verre ambré de 2 ml et placez 450 μL d’extrait d’échantillon blanc (étape 8.10) dans chacun d’eux et marquez-les avec des chiffres de 1 à 10. Chaque flacon sera un point de la courbe d’étalonnage.
REMARQUE : Dans ce protocole, 450 μL a été utilisé car il s’agissait de la valeur la plus basse que le dispositif LC pouvait atteindre à l’intérieur du flacon sans utiliser d’inserts. Si l’aiguille n’atteint pas aussi loin dans le flacon, augmentez le volume et, par conséquent, calculez le nombre d’échantillons à blanc supplémentaires à extraire en tenant compte du fait que chacun est remis en suspension dans 750 μL de mélange méthanol :eau (1/1, v :v). - Construire la courbe d’étalonnage par dilution en série. Transvasez 450 μL du mélange préparé à l’étape 8.11. dans le flacon 1, qui contient déjà 450 μL d’extrait d’échantillon à blanc, pour atteindre une concentration de 41,67 ng/mL (le point d’étalonnage le plus élevé). Agitez le flacon pendant 2 min.
REMARQUE : Le volume du mélange doit correspondre au volume de l’extrait de l’échantillon à blanc à diluer deux fois à chaque étape. - Transférez 450 μL du flacon 1 dans le flacon 2, qui contient déjà 450 μL d’extrait d’échantillon à blanc, pour atteindre une concentration de 20,83 ng/mL (le deuxième point d’étalonnage le plus élevé).
- Répétez l’étape 8.14 (c’est-à-dire du flacon 2 au flacon 3, et ainsi de suite) et continuez ensuite jusqu’à ce que les dix points d’étalonnage soient obtenus. La plage de concentration pour chaque EA sera alors de : 41,67, 20,83, 10,42, 5,21, 2,60, 1,30, 0,65, 0,33, 0,16 et 0,08 ng/mL.
- Préparer un mélange étalon à 1 000 ng/mL (83,33 ng/mL pour chaque EA) comme indiqué à l’étape 8.11, mais en utilisant 1 mL de mélange méthanol :eau (1:1, v/v).
- Préparez une courbe d’étalonnage standard comme indiqué aux étapes 8.13 à 8.15, mais en utilisant 450 μL de mélange méthanol/eau (1:1, v/v) pour les dilutions en série. Cette courbe d’étalonnage sera utilisée pour évaluer l’effet de matrice en le comparant à la courbe d’étalonnage appariée à la matrice.
9. Traitement des données quantitatives
- Initialisez et exécutez l’instrument en suivant les instructions décrites à l’étape 4.1. à 4.11. Voir la figure 2 pour la liste de travail utilisée dans ce protocole.
REMARQUE : Lorsque vous rédigez la liste de travail pour les courbes d’étalonnage, injectez-les avant les échantillons enrichis en commençant par la courbe d’étalonnage standard de la concentration la plus faible à la concentration la plus élevée, un flacon de lavage contenant du méthanol :eau (1:1, v/v) et la courbe d’étalonnage appariée à la matrice de la concentration la plus basse à la concentration la plus élevée. - Ouvrez le logiciel quantitatif et accédez à l’onglet TASQ Quick | Importer le lot en bas à gauche.
- Sélectionnez le dossier créé à l’étape 4.10. contenant toutes les données brutes (fichiers relatifs aux informations acquises après l’analyse LC-IMS-MS pour tous les échantillons inclus dans la liste de travail).
- Une fois importé dans le logiciel, allez dans Traiter le lot, juste en dessous de Importer le lot.
- Choisissez les échantillons à traiter (c’est-à-dire les 27 échantillons enrichis, les 9 blancs et les deux courbes d’étalonnage) et la méthode de traitement des données précédemment créée aux étapes 6.1. à 6.3. Une fenêtre s’affichera avec différents paramètres à définir (c’est-à-dire le type d’intégrateur pour évaluer les pics chromatographiques ou le type de signal analytique à prendre en compte, entre autres). Modifiez ces paramètres comme vous le souhaitez, bien que les valeurs par défaut conviennent à l’exécution de ce protocole.
- Allez à Vérifier le filtrage une fois le traitement des données terminé et vérifiez s’il y a des problèmes liés à l’intégration automatique, tels que la sélection de pics chromatographiques incorrects qui peuvent être trop proches les uns des autres. Si c’est le cas, limitez les valeurs relatives à la tolérance de temps de rétention, à la tolérance des valeurs CCS et/ou à l’erreur de masse initialement établies dans le fichier de méthode quantitative et retraitez les données. Si l’intégration automatique des pics sélectionne toujours des pics incorrects, intégrez manuellement le pic en sélectionnant la zone du chromatogramme qui apparaît dans la fenêtre de vérification du criblage (pour ce faire, tenez compte des paramètres analytiques obtenus pour les étalons EA dans la section 5).
REMARQUE : Il est très important d’examiner chaque échantillon, car certaines paires EA-épimères principales ont des temps de rétention très similaires et peuvent être mal identifiées par le logiciel. - Lorsque tous les pics ont été intégrés avec succès pour tous les échantillons, cliquez sur l’onglet Gestion des lots dans le coin supérieur gauche et indiquez le type d’échantillon associé à chaque analyse : solvant, blanc, contrôle qualité, échantillon ou point d’étalonnage. Dans ce dernier cas, attribuez à chaque point d’étalonnage un numéro à un niveau.
- Dans le même onglet Gestion des lots , accédez à la fenêtre inférieure appelée Concentration des lots et spécifiez une valeur de concentration pour chaque niveau établi à l’étape précédente. Une fois cela fait, cliquez sur Enregistrer les concentrations.
- Cliquez sur Quantifier le lot et modifiez les paramètres comme vous le souhaitez (c’est-à-dire le modèle pour s’adapter aux données d’étalonnage, la pondération, si le modèle est forcé de franchir zéro, etc.). Les valeurs par défaut sont adaptées à l’exécution de ce protocole, à l’exception de la pondération des courbes d’étalonnage, qui a été basculée à 1/x.
REMARQUE : Étant donné que le logiciel ne peut prendre en compte qu’une seule courbe d’étalonnage à la fois, il est nécessaire de répéter l’étape 9.9 deux fois pour obtenir des informations sur les courbes d’étalonnage standard et matricielle. Dans ce protocole, des courbes d’étalonnage appariées à la matrice ont été utilisées pour la quantification des analytes, de sorte que les courbes d’étalonnage du solvant ont été évaluées en premier, les données d’étalonnage ont été extraites comme expliqué plus tard à l’étape 9.13 et, enfin, les courbes d’étalonnage appariées à la matrice ont été sélectionnées pour effectuer la quantification des échantillons. - Une fois que la quantification des alcaloïdes de l’ergot a été effectuée selon le fichier de méthode de quantification, cliquez sur l’onglet Quantification dans la partie supérieure de l’écran.
- Si les paramètres par défaut définis à l’étape 9.9 n’offrent pas une bonne linéarité et précision, ajustez les courbes d’étalonnage en modifiant l’ajustement et le poids en faisant un clic droit sur le graphique dans le but d’avoir un bon modèle linéaire (R2 > 0,99) avec une précision adéquate sur chaque niveau d’étalonnage (écart inférieur à 20 %).
REMARQUE : Le logiciel marquera en rouge les cellules de ces niveaux d’étalonnage qui n’offrent pas une précision appropriée. - Une fois terminé, cliquez avec le bouton droit de la souris sur le dossier de lot à l’extrême gauche de l’écran et sélectionnez Générer un rapport. Ensuite, sélectionnez Quantification de l’analyte pour exporter le rapport au format souhaité.
Résultats
Tout d’abord, des solutions standard fonctionnelles ont été injectées dans l’instrument LC-IMS-MS pour obtenir toutes les caractéristiques d’identification (c’est-à-dire le temps de rétention, le CCS et les spectres de masse) de chaque EA analysé ici. Comme les paramètres d’identification, à l’exception de la masse exacte, étaient initialement inconnus, la méthode d’acquisition a été basée sur un événement à deux balayages, commençant par un balayage compl...
Discussion
L’utilisation réussie de ce protocole repose sur l’optimisation de la procédure d’extraction, précédemment réalisée par Carbonell-Rozas et al.17, qui ont mis en œuvre l’utilisation d’un solvant d’extraction suffisamment efficace pour extraire les EA de matrices alimentaires complexes telles que l’orge et le blé, et un nettoyage qui a fourni des valeurs SSE relativement faibles. Le choix du solvant d’extraction représente une étape critiqu...
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Cette recherche a été financée par la Consejería de Universidad, Investigación e Innovación - Junta de Andalucía (PROYEXCEL_00195) et la bourse postdoctorale accordée par la Generalitat Valenciana et le Fonds social européen+ (CIAPOS/2022/049). Les auteurs remercient le « Centro de Instrumentación Científica (CIC) » de l’Université de Grenade de m’avoir donné accès à l’instrumentation analytique utilisée dans ce protocole.
matériels
| Name | Company | Catalog Number | Comments |
| Acetonitrile | VWR | 83640.32 | |
| Amber glass tubes 4 mL | VWR | 548-0052 | |
| Amber glass tubes 12 mL | VWR | 548-0903 | |
| Amber vials 1.5 mL | Agilent | 5190-9063 | |
| Ammonium carbonate | Fluka | 9716 | |
| Analytical balance BAS 31 | Boeco | 4400519 | |
| Balance CP 323 S | Sartorius | 23-84182 | |
| C18 | Supelco | 52604-U | |
| Centrifuge tubes, 15 mL | VWR | 525-1082 | |
| Centrifuge tubes, 50 mL | VWR | 525-0155 | |
| Centrifuge Universal 320 R | Hettich | 1406 | |
| Compass HyStar | Bruker | Acquisition software | |
| DataAnalysis | Bruker | Qualitative software | |
| Elute PLUS UHPLC | Bruker | ||
| EVA EC-S evaporator | VLM | V830.012.12 | |
| Formic acid GR for analysis ACS, Reag. Ph Eur | Merck | 100264 | |
| Grinder TitanMill300 | Cecotec | 1559 | |
| Methanol | VWR | 83638.32 | |
| Milli-Q water purification system (18.2 MΩ cm) | Millipore | ZD5211584 | |
| Pipette tips 1- 5 mL | Labortecnic | 162005 | |
| Pipette tips 100 - 1000 µL | Labortecnic | 1622222 | |
| Pipette tips 5 - 200 µL | Labortecnic | 162001 | |
| Pippette Transferpette S variable, DE-M 10 - 100 µL | BRAND | 704774 | |
| Pippette Transferpette S variable, DE-M 100 - 1000 µL | BRAND | 704780 | |
| Pippette Transferpette S variable, DE-M 500 - 5000 µL | BRAND | 704782 | |
| Syringe 2 mL | VWR | 613-2003 | |
| Syringe Filter 13 mm, 0.22µm | Phenomenex | AF-8-7707-12 | |
| TASQ | Bruker | Quantitative software | |
| timsTOFPro2 IM-HRMS | Bruker | ||
| Vortex Genie 2 | Scientific Industries | 15547335 | |
| Zorbax Eclipse Plus RRHD C18 column (50 x 2.1 mm, 1.8 µm particle size) | Agilent | 959757-902 | |
| Z-Sep+ | Supelco | 55299-U | Zirconia-based sorbent |
| Ergot alkaloids | CAS registry sorbent | ||
| Ergocornine (Eco) | Techno Spec | E178 | 564-36-3 |
| Ergocorninine (Econ) | Techno Spec | E130 | 564-37-4 |
| Ergocristine (Ecr) | Techno Spec | E180 | 511-08-0 |
| Ergocristinine (Ecrn) | Techno Spec | E188 | 511-07-9 |
| Ergokryptine (Ekr) | Techno Spec | E198 | 511-09-1 |
| Ergopkryptinine (Ekrn) | Techno Spec | E190 | 511-10-4 |
| Ergometrine (Em) | Romer Labs | "002067" | 60-79-7 |
| Ergometrinine (Emn) | Romer Labs | LMY-090-5ML | 479-00-5 |
| Ergosine (Es) | Techno Spec | E184 | 561-94-4 |
| Ergosinine (Esn) | Techno Spec | E194 | 596-88-3 |
| Ergotamine (Et) | Romer Labs | "002069" | 113-15-5 |
| Ergotaminine (Etn) | Romer Labs | "002075" | 639-81-6 |
Références
- Kanu, A. B., Dwivedi, P., Tam, M., Matz, L., Hill, H. H. Ion mobility-mass spectrometry. J Mass Spectrom. 43 (1), 1-22 (2008).
- Gabelica, V., et al. Recommendations for reporting ion mobility Mass spectrometry measurements. Mass Spectrom Rev. 38 (3), 291-320 (2019).
- Feuerstein, M. L., et al. Comparability of steroid collision cross sections using three different IM-HRMS technologies: An interplatform study. J Am Soc Mass Spectrom. 33 (10), 1951-1959 (2022).
- Regueiro, J., Negreira, N., Berntssen, M. H. G. Ion-mobility-derived collision cross section as an additional identification point for multiresidue screening of pesticides in fish feed. Anal Chem. 88 (22), 11169-11177 (2016).
- Regueiro, J., Negreira, N., Hannisdal, R., Berntssen, M. H. G. Targeted approach for qualitative screening of pesticides in salmon feed by liquid chromatography coupled to traveling-wave ion mobility/quadrupole time-of-flight mass spectrometry. Food Control. 78, 116-125 (2017).
- Hernández-Mesa, M., Monteau, F., Le Bizec, B., Dervilly-Pinel, G. Potential of ion mobility-mass spectrometry for both targeted and non-targeted analysis of phase II steroid metabolites in urine. Anal Chim Acta: X. 1, 100006 (2019).
- George, A. C., et al. Interplatform comparison between three ion mobility techniques for human plasma lipid collision cross sections. Anal Chim Acta. 1304, 342535 (2024).
- Mairinger, T., Causon, T. J., Hann, S. The potential of ion mobility-mass spectrometry for non-targeted metabolomics. Curr Opin Chem Biol. 42, 9-15 (2018).
- Hernández-Mesa, M., Escourrou, A., Monteau, F., Le Bizec, B., Dervilly-Pinel, G. Current applications and perspectives of ion mobility spectrometry to answer chemical food safety issues. TrAC Trend Anal Chem. 94, 39-53 (2017).
- European, C. Commission Regulation (EU) 2023/915 of 25 April 2023 on maximum levels for certain contaminants in food and repealing Regulation (EC) No 1881/2006. Off J Eur Union. 119, 103-157 (2023).
- Carbonell-Rozas, L., et al. Occurrence of egot alkaloids in major and minor cereals from Northern Italy: A three harvesting years scenario. J Agricl Food Chem. 71 (42), 15821-15828 (2023).
- Arroyo-Manzanares, N., Gámiz-Gracia, L., García-Campaña, A. M., Diana Di Mavungu, J., De Saeger, S., Jean-Michel Mérillon, J. -. M., Ramawat, K. G. . Fungal Metabolites. , (2016).
- van Dongen, P., de Groot, A. History of ergot alkaloids from ergotism to ergometrine. Eur J Obstet Gynecol Reprod Biol. 60 (2), 109-116 (1995).
- European Food Safety Authority. Human and animal dietary exposure to ergot alkaloids. EFSA J. 15 (7), e04902 (2017).
- Nam, M., Kim, D., Kim, M. -. S. Simultaneous determination of total ergot alkaloids in wheat flour by Orbitrap mass spectrometry. Food Chem. 441, 138363 (2024).
- García-Juan, A., León, N., Armenta, S., Pardo, O. Development and validation of an analytical method for the simultaneous determination of 12 ergot, 2 tropane, and 28 pyrrolizidine alkaloids in cereal-based food by LC-MS/MS. Food Res Int. 174 (Pt 1), 113614 (2023).
- Carbonell-Rozas, L., Mahdjoubi, C. K., Arroyo-Manzanares, N., García-Campaña, A. M., Gámiz-Gracia, L. Occurrence of ergot alkaloids in barley and wheat from Algeria. Toxins. 13 (5), 316 (2021).
- Carbonell-Rozas, L., Gámiz-Gracia, L., Lara, F. J., García-Campaña, A. M. Determination of the main ergot alkaloids and their epimers in oat-based functional foods by ultra-high performance liquid chromatography tandem mass spectrometry. Molecules. 26 (12), 3717 (2021).
- Carbonell-Rozas, L., et al. Ion mobility-mass spectrometry to extend analytical performance in the determination of ergot alkaloids in cereal samples. J Chromatogr A. 1682, 463502 (2022).
- European Commission. Commission Implementing Regulation (EU) 2023/2782 of 14 December 2023 laying down the methods of sampling and analysis for the control of the levels of mycotoxins in food and repealing Regulation (EC) No 401/2006. Off J Eur Union. , (2023).
- Carbonell-Rozas, L., Vander Cruyssen, L., Dall'Asta, C., Leggieri, M. C., Battilani, P. Fit-for-purpose method development to determine co-occurring multiclass mycotoxins in apple and apple puree samples. Food Anal Methods. 16 (8), 1403-1412 (2023).
- Laouni, C., et al. Emerging mycotoxin occurrence in chicken feed and eggs from Algeria. Mycotoxin Res. 40, 447-456 (2024).
- Ben Hassouna, K., et al. Mycotoxin occurrence in milk and durum wheat samples from Tunisia using dispersive liquid-liquid microextraction and liquid chromatography with fluorescence detection. Toxins. 15 (11), 633 (2023).
- Narváez, A., et al. Occurrence and exposure assessment of mycotoxins in ready-to-eat tree nut products through ultra-high performance liquid chromatography coupled with high resolution Q-orbitrap mass spectrometry. Metabolites. 10 (9), 344 (2020).
- Arroyo-Manzanares, N., Rodríguez-Estévez, V., García-Campaña, A. M., Castellón-Rendón, E., Gámiz-Gracia, L. Determination of principal ergot alkaloids in swine feeding. J Sci Food Agric. 101 (12), 5214-5224 (2021).
- Pereira, V. L., Fernandes, J. O., Cunha, S. C. Comparative assessment of three cleanup procedures after QuEChERS extraction for determination of trichothecenes (type A and type B) in processed cereal-based baby foods by GC-MS. Food Chem. 182, 143-149 (2015).
- Tuzimski, T., Szubartowski, S. Method development for selected bisphenols analysis in sweetened condensed milk from a can and breast milk samples by HPLC-DAD and HPLC-QqQ-MS: Comparison of sorbents (Z-SEP, Z-SEP Plus, PSA, C18, chitin and EMR-lipid) for clean-up of QuEChERS extract. Molecules. 24 (11), 2093 (2019).
- Łozowicka, B., Rutkowska, E., Jankowska, M. Influence of QuEChERS modifications on recovery and matrix effect during the multi-residue pesticide analysis in soil by GC/MS/MS and GC/ECD/NPD. Environ Sci Pollut Res. 24 (8), 7124-7138 (2017).
- Schummer, C., Xandonella, I., van Nieuwenhuyse, A., Moris, G. Epimerization of ergot alkaloids in feed. Heliyon. 6 (6), e04336 (2020).
- Cherewyk, J. E., Grusie-Ogilvie, T. J., Parker, S. E., Blakley, B. R., Al-Dissi, A. M. The impact of storage temperature and time on ergot alkaloid concentrations. Toxins. 15 (8), 497 (2023).
- Silva, &. #. 1. 9. 4. ;., Mateus, A. R., Barros, S. C., Silva, A. S. Ergot alkaloids on cereals and seeds: Analytical methods, occurrence, and future perspectives. Molecules. 28 (20), 7233 (2023).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon