Method Article
Imágenes tridimensionales de tejido tumoral mediante el enfoque de blanqueamiento iterativo que extiende la multiplexidad
En este artículo
Resumen
En este artículo se describe un protocolo de inmunofluorescencia multiplex optimizado para caracterizar la arquitectura tridimensional de las metástasis peritoneales.
Resumen
La heterogeneidad espacial del microambiente tumoral (TME) es un determinante crítico de la respuesta terapéutica, particularmente para los agentes inmunooncológicos, donde el éxito depende de la distribución de subpoblaciones específicas de células inmunitarias. Durante la última década, se han introducido múltiples tecnologías sofisticadas para lograr una resolución detallada del TME utilizando secciones bidimensionales de tejidos fijados en formol, incluidos en parafina (FFPE) o congelados fijos. Si bien estas secciones delgadas son más fáciles de obtener y analizar, carecen de la arquitectura tridimensional necesaria para caracterizar un tumor de manera confiable y completa. Para abordar esta limitación, se desarrolló una técnica de montaje de tejido e imagen que permitió el análisis tridimensional de las lesiones tumorales en su estado nativo in vivo . Este protocolo describe la obtención de tejido tumoral humano, el montaje de muestras en plataformas impresas a medida y los procedimientos de tinción para muestras posteriores a la fijación. La técnica de inmunofluorescencia multiplexada, IBEX (Iterative Bleaching Extends Multiplexity), se adaptó para caracterizar el TME tridimensional con hasta 15 marcadores de células tumorales, inmunitarias y estromales utilizando anticuerpos disponibles en el mercado. Se lograron profundidades de imagen de hasta 100 μm utilizando un microscopio confocal láser de luz blanca invertido con un adaptador de imagen impreso a medida y placas comerciales con fondo de vidrio para garantizar una orientación óptima del tejido. Este protocolo pone de manifiesto el potencial del método IBEX para ampliar los estudios de inmunofluorescencia multiplexada, proporcionando una comprensión más completa de la composición de TME.
Introducción
Los tumores sólidos son muy variables, con una composición compleja de tipos celulares (malignos y no malignos), proteínas de la matriz extracelular y factores solubles 1,2. En última instancia, este microambiente tumoral (TME) altamente complejo y heterogéneo determina la susceptibilidad a tratamientos como la inmunoterapia 3,4. Como tal, se han aplicado tecnologías como la inmunofluorescenciaaltamente multiplexada 5,6 y la transcriptómica espacial7 para caracterizar la complejidad del TME en secciones de espesor de capa celular (~4 μM).
A diferencia de los enfoques de dispersión celular, como la secuenciación de una sola célula y la citometría de flujo 8,9,10, estas técnicas preservan las relaciones espaciales para aportar información crítica sobre la diafonía celular y pueden emplearse para identificar biomarcadores potenciales para la predicción de la respuesta al tratamiento 11,12,13. Sin embargo, las lesiones tumorales son estructuras inherentemente tridimensionales (3D), y los enfoques 2D como los mencionados anteriormente no capturan adecuadamente el complejo panorama celular. La importancia de la biología 3D ha sido apreciada en el campo, como lo demuestra la amplia utilización de cultivos de organoides tumorales derivados de pacientes14. Sin embargo, los organoides no conservan la complejidad celular del TME original in vivo, lo que limita la aplicabilidad para su uso con fármacos inmunomoduladores o para la caracterización de las diversas poblaciones de células TME.
Aunque puede ser posible realizar análisis en secciones 2D en serie y "unir" computacionalmente las imágenes para una representación 3D, esta técnica es prohibitiva y difícil de lograr con alta fidelidad15. Con el fin de caracterizar de manera rentable los tejidos 3D (por ejemplo, tumores) de manera reproducible, se desarrolló y demostró un método único para la preparación y la tinción/análisis de inmunofluorescencia múltiple utilizando lesiones tumorales peritoneales de un paciente con cáncer peritoneal primario.
Basado en una adaptación de la técnica IBEX (Iterative Bleaching Extends Multiplexity)16,17, este protocolo detalla la impresión 3D de un soporte de tejido y un adaptador de imagen, la obtención y el montaje de tejido peritoneal, múltiples ciclos de tinción con varios marcadores de tumores y tipos celulares, y la obtención de imágenes confocales en 3D de alta resolución.
Protocolo
El estudio se llevó a cabo con la aprobación de la Junta de Revisión Institucional de los Institutos Nacionales de Salud bajo el protocolo NCT01915225. El tejido humano se obtuvo con consentimiento informado por escrito en el momento de la laparoscopia diagnóstica. Todo el tejido era portador de tumor, según se determinó mediante inspección visible y se confirmó mediante el examen histopatológico final. Los detalles de todos los reactivos y equipos utilizados se enumeran en la Tabla de Materiales. En la Figura 1 se proporciona una ilustración esquemática de todo el flujo de trabajo.
1. Plataformas de impresión, adaptador de imagen y placa de incubación
- Procedimientos de corte/impresión 3D/posprocesamiento para la plataforma y las piezas de la placa de 9 pocillos (descritos en la Figura 2A-C)
- Corte: Abra el software de corte e importe los archivos de diseño en formato . Formato STL (Fichero Complementario 1 y Fichero Complementario 2). Orientar los modelos para una impresión óptima y añadir soportes. Seleccione la impresora de grado médico, seleccione la resina transparente y la altura de capa de 50 μm . Compruebe la imprimibilidad y envíe el trabajo a la impresora.
- Impresión 3D: Comprueba que la impresora esté lista (cartucho de resina, tanque y plataforma correctos en su lugar). En la pantalla táctil de la impresora, seleccione el trabajo cargado en el software de corte para iniciar la impresión. Una vez completada la impresión, utilice la herramienta de extracción para separar la pieza impresa de la plataforma.
- Post-procesamiento: Enjuague la pieza en la unidad de lavado llena con alcohol isopropílico (IPA) al 99% durante 15 min. Retire las piezas de la unidad de lavado y sumérjalas en alcohol isopropílico fresco durante 5 minutos.
- Fotocure la pieza durante 60 minutos a 60 °C. Retire los soportes con cortadores al ras. Lija las marcas de soporte con papel de lija de grano fino si es necesario.
- Procedimientos de corte/impresión 3D/posprocesamiento para el ajustador de altura y la tapa exterior (descritos en la Figura 2A-C)
- Corte: Abra el software de impresión e importe los archivos de diseño en formato . Formato STL (Fichero Complementario 3 y Fichero Complementario 4). Orientar los modelos y asignar materiales. Seleccione el modo de impresión deseado (Alta mezcla, alta velocidad) y establezca la configuración del modelo para finalizar en Mate. Compruebe la imprimibilidad y envíe el trabajo a la impresora.
- Impresión 3D: Asegúrese de que la impresora tenga los materiales correctos cargados y que la bandeja de impresión esté limpia. En la interfaz del monitor de la impresora, seleccione el trabajo cargado para iniciar la impresión. Una vez completada la impresión, utiliza la herramienta rascadora para retirar suavemente la pieza impresa de la bandeja de impresión.
- Post-procesado: Colocar las piezas impresas en un limpiador de chorro de agua o en un sistema de limpieza por agitación para eliminar el material de soporte. Realice cualquier extracción manual del soporte, si es necesario, utilizando herramientas como pinzas o alicates.
- Corte por láser del deslizador de la plataforma (mostrado en la Figura 2A, C)
- Preparando el diseño: Importe el archivo *. Archivo de diseño DWG en el software vectorial (Archivo complementario 5). Establezca los colores de línea en rojo (RGB) para el corte vectorial. Envíe el trabajo a la cortadora láser.
- Configuración de la cortadora láser: Abra el software de la cortadora láser e importe el archivo de diseño. Seleccione el tipo de material y ajuste la configuración de potencia, velocidad y resolución para acrílico de 1/32" de grosor. Coloque el material en la cama láser, asegúrese de que esté correctamente alineado y enfoque el láser.
- Corte por láser: Inicie el proceso de corte por láser y supervise para asegurarse de que el material se corte correctamente.
2. Montaje de tisú en plataformas impresas
- Preparación del medio de cosecha y mesa de preparación
- Complemente 100 mL de DMEM (glucosa baja) con Anti-Anti y colóquelo en una incubadora a 37 °C hasta que lo necesite (medio de cosecha).
- Instale una "mesa de preparación" en el quirófano con los siguientes elementos: plataformas impresas en 3D esterilizadas en autoclave, paquete de 2-0 lazos de suturas de seda, placa calefactora, taza de muestra con medio de cosecha precalentado de 50 mL, placa de cultivo de 15 cm que contiene 30 mL de medio de cosecha precalentado y una placa de 24 pocillos con 4 pocillos cada uno que contiene 1,5 mL de medio de cosecha (coloque todo en una placa calefactora), 1 par de pinzas y 1 par de tijeras quirúrgicas.
- Resección y montaje de tejido
NOTA: El tejido se obtiene del quirófano. Las áreas del peritoneo con focos tumorales de menos de 0,5 mm de grosor y diámetro se identifican en la inspección visual (Figura 2D). Cabe destacar que se necesitan ~ 2cm2 de tejido (peritoneo) para preparar una plataforma.- Separe cuidadosamente el peritoneo portador del tumor de la pared abdominal, la musculatura y la fascia subyacentes (realizado por el cirujano). Transfiera las hojas de papel resultantes a un recipiente de muestra que contenga medio de cosecha tibio y colóquelas en una placa calefactora (mesa de preparación).
- Monta el pañuelo en plataformas en la placa de 15 cm que contiene el medio de recolección. Coloque cuidadosamente el tejido portador del tumor (con el lado mesotelial hacia arriba) sobre el lado del orificio pequeño de la plataforma y asegúrelo en su lugar con una sutura de seda 2-0. Coloque la plataforma de tejido preparada inversamente (sumergida) en una placa de 24 pocillos que contenga medio de cosecha (Figura 2E-G).
NOTA: Montar el tejido lo suficientemente enseñado para evitar pliegues en la superficie de la plataforma, pero no genere tensión ya que esto inducirá cambios biomecánicos y fisiológicos no deseados.
3. Procedimiento de fijación/tinción/blanqueo
NOTA: En la Figura 3 se describen los cambios con respecto al protocolo original adaptado a FFPE de 4 μm de grosor o secciones congeladas fijas. La Tabla 1 proporciona una lista de anticuerpos (con diluciones) utilizados para las rondas 1-6 de IBEX.
- Fijación
- Prepare 40 mL de tampón de fijación añadiendo 10 mL de material fijador a un tubo cónico de 50 mL que contenga 30 mL de PBS frío 1x.
- Con ayuda de unas pinzas, transfiera el tejido montado en plataformas al tampón de fijación e incube durante 24 h a 4 °C.
- Decantar el fijador y sustituirlo por 40 mL de PBS frío 1x e incubar durante 10 min a 4 °C.
- Tinción
- Agregue 1 mL de tampón de bloqueo (1% BSA, 0.3% Triton-X-100, 1% Fc Block en 1x PBS filtrado con filtro de jeringa de 0.22 μm) a cada pocillo e inserte plataformas de tejido. Bloquee durante al menos 2 h a temperatura ambiente en un balancín ajustado a baja velocidad (30 rpm).
NOTA: Asegúrese de que la muesca de la plataforma y la ranura del pozo estén alineadas. Este paso no es sensible al tiempo y se puede realizar durante la noche a 4 °C. - Prepare 0,8 mL de solución de anticuerpo/colorante en un tubo de microcentrífuga y centrifugue a 10.000 x g durante 5 minutos a temperatura ambiente. Transfiera 0,75 mL a un pocillo limpio de la placa de incubación de 9 pocillos e inserte una plataforma lavada.
- Envuelva la placa en papel de aluminio e incube durante al menos 2 h a temperatura ambiente en un balancín ajustado a baja velocidad (30 rpm).
- Lave la plataforma en un tubo cónico de 50 mL con 40 mL de 1x PBS durante 10 min a 4 °C. La plataforma ya está lista para ser insertada en el adaptador de imagen.
- Agregue 1 mL de tampón de bloqueo (1% BSA, 0.3% Triton-X-100, 1% Fc Block en 1x PBS filtrado con filtro de jeringa de 0.22 μm) a cada pocillo e inserte plataformas de tejido. Bloquee durante al menos 2 h a temperatura ambiente en un balancín ajustado a baja velocidad (30 rpm).
- Blanqueo
- Después de cada ronda de tinción e imagen, blanquee los fluoróforos con el agente reductor de ésteres borohidruro de litio (LiBH4). Este tratamiento eliminará las señales de fluorescencia de Alexa Fluor (AF)488, AF647 y AF750, pero no de AF594 y el colorante nuclear Hoechst.
PRECAUCIÓN: El borohidruro de litio es extremadamente reactivo con el agua. Los gases de hidrógeno inflamables se liberan al entrar en contacto con el agua; El borohidruro de litio gastado y los desechables contaminados con borohidruro de litio deben eliminarse como residuos peligrosos. - Prepare 5 mL de solución de LiBH4 de 1,5 mg/mL en agua destilada (en una campana química) e incube durante 30 min a temperatura ambiente.
NOTA: La solución de LiBH4 perderá su eficacia reductora/blanqueadora cuando se prepare >4 h antes de su uso. - Transfiera 1 mL de solución LiBH4 a un pocillo limpio de la placa de incubación de 9 pocillos e inserte la plataforma lavada durante 60 min a temperatura ambiente.
- Lave brevemente la plataforma con 20 ml de PBS 1x en un tubo cónico y compruebe la señal de fluorescencia restante utilizando la configuración láser más alta para la compensación Z en la ronda de imágenes anterior antes de pasar a las siguientes rondas de tinción.
- Repita el bloqueo de la muestra durante 2 h a temperatura ambiente antes de pasar al siguiente paso de incubación de anticuerpos.
- Después de cada ronda de tinción e imagen, blanquee los fluoróforos con el agente reductor de ésteres borohidruro de litio (LiBH4). Este tratamiento eliminará las señales de fluorescencia de Alexa Fluor (AF)488, AF647 y AF750, pero no de AF594 y el colorante nuclear Hoechst.
4. Procedimiento de diagnóstico por imágenes
- Montaje de adaptadores/plataformas de imagen
- Agregue 0,1 ml de 1x PBS al centro del fondo de vidrio de un plato de imágenes de 3,5 cm y reserve. Atornille el anillo de ajuste de altura sin apretar en el sentido de las agujas del reloj en la tapa exterior y monte la plataforma en el soporte de plástico interior (alineación de muesca-ranura). Asegure la plataforma con el control deslizante.
- Coloque el adaptador de imagen ensamblado en el plato con fondo de vidrio y baje con cuidado el anillo de ajuste de altura hasta que el tejido toque el tampón. Evite girar la plataforma sobre el fondo de vidrio.
- Una vez que se alcanza la distancia óptima desde el vidrio, agregue 0,2 mL de 1x PBS en el pañuelo (ahora la parte inferior hacia arriba) para evitar la deshidratación del tejido durante la toma de imágenes.
- Imágenes confocales
- Encienda todos los componentes del microscopio confocal invertido, incluida la computadora, el microscopio, el láser y la fuente de luz LED para el uso del ocular. Inicie el software de adquisición de imágenes .
- Seleccione el objetivo apropiado (por ejemplo, 20x o 40x) y agregue el medio de inmersión apropiado. Por ejemplo, en este estudio se utiliza un objetivo de agua 20x; Se añaden ~200 μL de agua al objetivo para evitar que se seque durante las adquisiciones de imágenes largas.
- Coloque el plato de imágenes ensamblado en el soporte de muestras en la platina del microscopio y asegúrese de que la muestra esté segura (no se mueva).
NOTA: Al colocar la muestra ensamblada en la platina del microscopio, tome nota de la posición de la muesca y utilice la misma posición para los ciclos iterativos para facilitar la alineación de la imagen. Es importante mantener el adaptador de imagen en la altura óptima para rondas consecutivas. - Centre la muestra con el control X-Y y acerque el objetivo al cubreobjetos. Mirando a través del ocular y utilizando la señal de Hoechst como referencia, mueva el objetivo para obtener un enfoque preciso en el tejido.
- Seleccione las líneas láser apropiadas o ajuste la WLL a los espectros de excitación y emisión de los fluoróforos utilizados para teñir la muestra de tejido. Elimine la superposición entre canales ajustando la configuración y agregue secuencias si los fluoróforos tienen espectros de excitación similares.
- Elija los siguientes parámetros de adquisición de imágenes: velocidad de escaneo de 600 Hz, resolución XY de 1024 x 1024, promedios de 3 líneas y escaneo bidireccional .
- Elija los siguientes parámetros de pila Z: (1) use un tamaño de paso Z optimizado para el objetivo que se está utilizando, (2) defina el principio y el final de la pila, (3) ajuste los parámetros de compensación Z para lograr un brillo igual en toda la pila y (4) adquiera una pila de prueba de 1 mosaico con un promedio.
- Defina la región de la que se va a crear una imagen y adquiera una pila Z de mosaico de alta resolución.
Resultados
El peritoneo es una estructura membranosa de menos de 1 mm de espesor que recubre la superficie de la pared abdominal y es contigua a las superficies de los órganos abdominales. Está compuesto por mesotelio superficial y tejido conectivo subyacente que se intercala con células adiposas, linfocitos, macrófagos y fibroblastos, así como vasos sanguíneos y linfáticos. Las neoplasias malignas peritoneales suelen provenir de metástasis tumorales abdominales (p. ej., cáncer de ovario o cáncer gástrico)18 o, con menor frecuencia, pueden ser tumores primarios del peritoneo (p. ej., mesotelioma)19. Estas lesiones tumorales, a menudo pequeñas (<1 cm), son muy complejas, pero se pueden caracterizar en 3D mediante la técnica descrita. Se utiliza una proyección máxima de cinco secciones confocales para ver toda la muestra montada en una plataforma (Figura 4, recuadro en la esquina superior izquierda). Se utilizaron tres anticuerpos primarios conjugados (AF488-CD44, AF594-CD45 y AF647-Podoplanin) junto con el colorante nuclear Hoechst para la primera ronda de IBEX (IBEX1, Figura 4). Las lesiones tumorales se hacen evidentes por las estructuras en forma de roseta que son doblemente positivas para CD44 y Podoplanin y muestran una disposición de núcleos característica del mesotelioma papilar.
Las células tumorales se identificaron utilizando la morfología celular y dos anticuerpos, ya que tanto la anti-Podoplanina como la anti-CD44 reconocen a las células no tumorales, como lo demuestra la detección de vasos linfáticos a través de la anti-Podoplanina y la unión del anticuerpo CD44 a subconjuntos de células inmunitarias. A continuación, se capturan las regiones de interés (ROI) más pequeñas mediante un formato de escaneo de mosaicos de mayor resolución en tamaños de paso Z optimizados para representar proyecciones 3D (Figura 5A). Las plataformas se sometieron a los procedimientos de tinción iterativos y se obtuvieron imágenes de los mismos ROI en los ciclos 1-6 de IBEX. En la Figura 5B-M se reúnen imágenes representativas de las interfaces tumoral (recuadro amarillo) y de la estructura linfoide terciaria tumoral (TLS) (recuadro azul).
La presencia de TLS y la densidad de células CD3 positivas indican una infiltración significativa de células inmunitarias20 y una posible respuesta a los fármacos inmunomoduladores21. Cabe destacar que la TLS y la mayor parte de las lesiones tumorales están presentes en diferentes profundidades Z del tejido, como se demostró al comparar las proyecciones de pila de diferentes secciones ópticas en la Figura 6, lo que destaca los beneficios de las imágenes 3D y la posible pérdida de biología cuando las imágenes 2D se aplican a estructuras 3D. El carácter 3D de estas lesiones se hace más evidente en los archivos de la película (Película 1-12), que muestran representaciones en volumen de las lesiones individuales en la Figura 5.
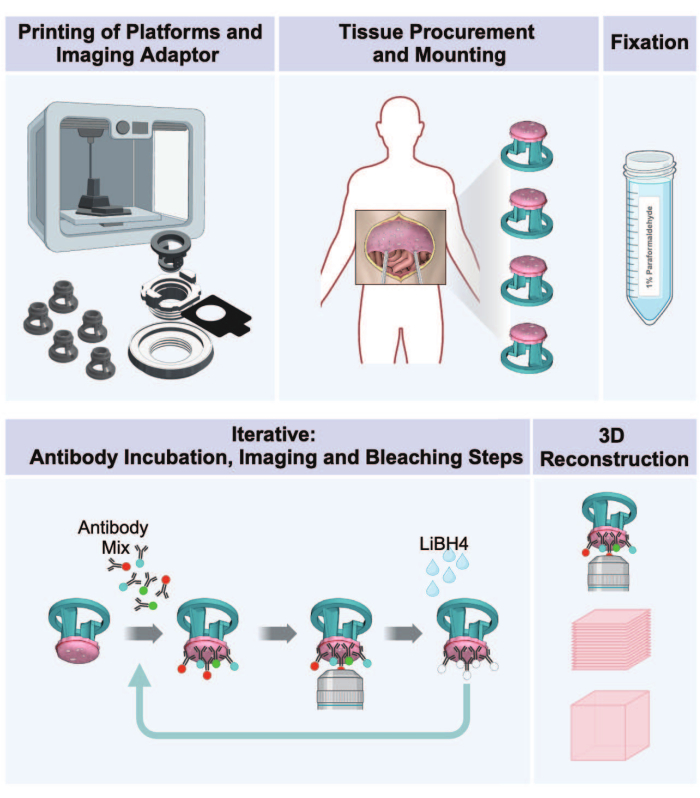
Figura 1: Flujo de trabajo desde la impresión 3D hasta la obtención de imágenes en 3D de tumores peritoneales. El protocolo consiste en una secuencia de cinco pasos para preparar imágenes multiplexadas en 3D a partir de tejido peritoneal portador de tumores. (1) Impresión de plataformas receptoras de tejidos, piezas de adaptadores de imágenes y placa de incubación. (2) Procedimientos de obtención y montaje de tejidos. (3) Fijación con paraformaldehído. (4) Ciclos de tinción e imagen de IBEX. (5) Reconstrucción 3D mediante software de imagen. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Componentes de hardware con interfaz de tejido para imágenes 3D. (A) Diagrama esquemático del ensamblaje de la plataforma dentro del adaptador de imágenes. (B) Una placa de incubación de 9 pocillos. (C) Fotografías de los componentes del adaptador de imagen con el montaje. (D) Peritoneo portador de mesotelioma observado durante la laparoscopia diagnóstica. (E) Montaje de tejido en la plataforma. (F,G) Peritoneo portador de tumores montado sobre plataformas. Haga clic aquí para ver una versión más grande de esta figura.
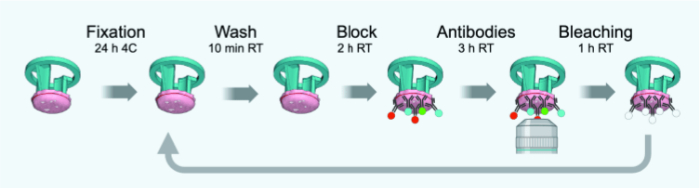
Figura 3: Esquema de la línea de tiempo de los ciclos del IBEX. Haga clic aquí para ver una versión más grande de esta figura.
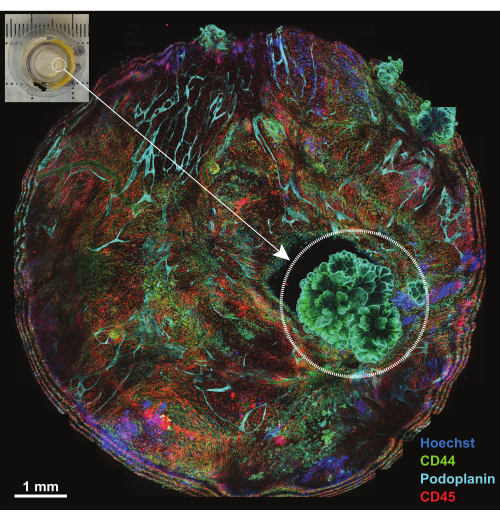
Figura 4: Vista general de la plataforma después de la incubación con el panel IBEX1. Máxima proyección de toda la plataforma tras la incubación con panel IBEX1 (Hoechst, AF488-CD44, AF594-CD45 y AF647-Podoplanin). El círculo discontinuo resalta el tumor. Recuadro: Fotografía del peritoneo portador de tumores montada en la plataforma de imagen. Barra de escala: 1 mm. Haga clic aquí para ver una versión más grande de esta figura.
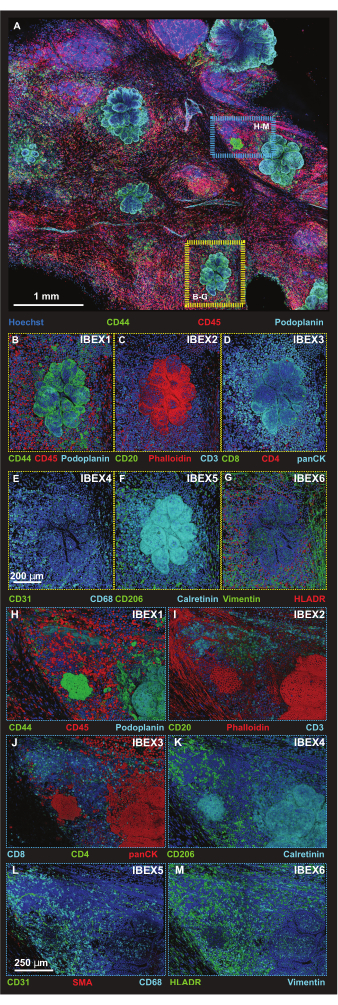
Figura 5: Imágenes de inmunofluorescencia de alta resolución de regiones seleccionadas de la plataforma. (A) Proyección máxima de la imagen combinada de 56 mosaicos Z-stack adquirida después de la incubación con el panel IBEX1 (Hoechst, AF488-CD44, AF594-CD45 y AF647-Podoplanin). Rectángulo amarillo: lesión tumoral mostrada en paneles (B-G). Rectángulo azul: interfaz entre el tumor y la estructura linfoide terciaria mostrada en (H-M). (B-G) Una lesión tumoral individual con panel IBEX 1-6 en representación volumétrica. (B) IBEX1, verde: CD44, rojo: CD45, cian: Podoplanin. (C) IBEX2, verde: CD20, rojo: F-actina, cian: CD3. (D) IBEX3, verde: CD8, rojo: CD4, cian: panCK. (E) IBEX4, verde: CD31, cian: CD68. (F) IBEX5, verde: CD206, cian: Calretinina. (G) IBEX6, verde: Vimentina, rojo: HLADR. (H-M) Una interfaz de estructura linfoide terciaria tumoral individual con el panel IBEX 1-6 en representación de volumen. (H) IBEX1, verde: CD44, rojo: CD45, cian: Podoplanin. (I) IBEX2, verde: CD20, rojo: F-actina, cian: CD3. (J) IBEX3, verde: CD4, rojo: panCK, cian: CD8. (K) IBEX4, verde: CD31, rojo: SMA, cian: CD68. (L) IBEX5, verde: CD206, cian: calretinina. (M) IBEX6, verde: HLADR, cian: Vimentina. Todos los paneles, azul: Hoechst. Barras de escala: (A, 1 mm); (B-M, 250 μm). Haga clic aquí para ver una versión más grande de esta figura.
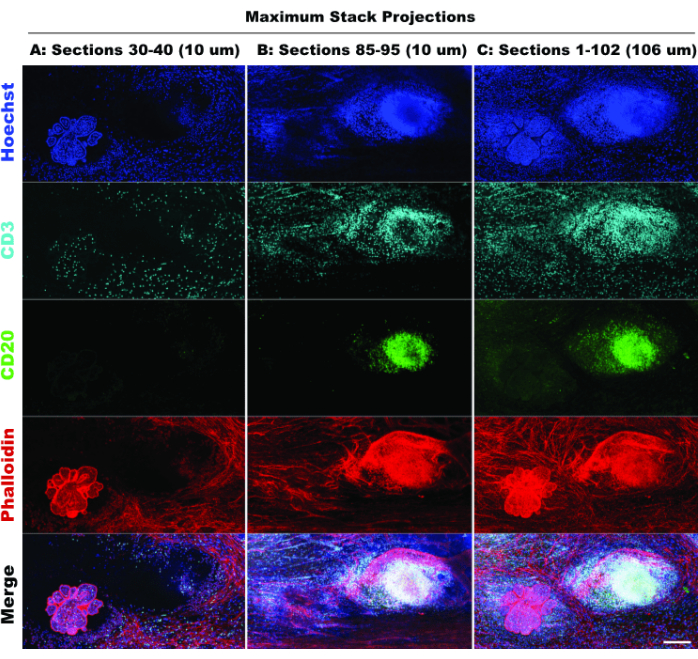
Figura 6: Imágenes 2D simuladas. La comparación de proyecciones limitadas (10 secciones ópticas) y de pila completa (102 secciones ópticas) confirma la pérdida de contexto tisular en la simulación 2D. (A) Proyección máxima de las secciones 30-40 (10 μm). (B) Proyección máxima de las secciones 85-95 (10 μm). (C) Proyección máxima de las secciones 1-102 (~100 μm). Todos los paneles, IBEX2 (Hoechst, AF488-CD20, AF647-CD3, AF790-faloidina). Barra de escala: 200 μm. Haga clic aquí para ver una versión más grande de esta figura.
Tabla 1: Anticuerpos utilizados para los paneles individuales de IBEX. Haga clic aquí para descargar esta tabla.
Vídeo 1: Representación animada del volumen de la lesión individual con el panel IBEX 1 (azul: Hoechst, verde: CD44, rojo: CD45, cian: Podoplanin). Por favor, haga clic aquí para descargar esta película.
Vídeo 2: Representación animada del volumen de la lesión individual con el panel IBEX 2 (azul: Hoechst, verde: CD20, rojo: F-actina, cian: CD3). Por favor, haga clic aquí para descargar esta película.
Vídeo 3: Representación animada del volumen de la lesión individual con el panel IBEX 3 (azul: Hoechst, verde: CD8, rojo: CD4, cian: panCK). Por favor, haga clic aquí para descargar esta película.
Vídeo 4: Representación animada del volumen de la lesión individual con el panel IBEX 4 (azul: Hoechst, verde: CD31, cian: CD68). Por favor, haga clic aquí para descargar esta película.
Vídeo 5: Representación animada del volumen de la lesión individual con el panel 5 del IBEX (azul: Hoechst, verde: CD206, cian: calretinina). Por favor, haga clic aquí para descargar esta película.
Vídeo 6: Representación animada en volumen de la lesión individual con el panel IBEX 6 (azul: Hoechst, verde: Vimentin, rojo: HLADR). Por favor, haga clic aquí para descargar esta película.
Vídeo 7: Renderizado animado en volumen de la interfaz estructura-lesión linfoide terciaria con el panel 1 de IBEX (azul: Hoechst, verde: CD44, rojo: CD45, cian: Podoplanin). Por favor, haga clic aquí para descargar esta película.
Vídeo 8: Representación animada en volumen de la interfaz estructura-lesión linfoide terciaria con el panel 2 de IBEX (azul: Hoechst, verde: CD20, rojo: F-actina, cian: CD3). Por favor, haga clic aquí para descargar esta película.
Vídeo 9: Renderizado animado en volumen de la interfaz estructura-lesión linfoide terciaria con el panel 3 de IBEX (azul: Hoechst, verde: CD8, rojo: CD4, cian: panCK). Por favor, haga clic aquí para descargar esta película.
Vídeo 10: Renderizado animado en volumen de la interfaz estructura-lesión linfoide terciaria con el panel 4 de IBEX (azul: Hoechst, verde: CD31, rojo: SMA, cian: CD68). Por favor, haga clic aquí para descargar esta película.
Vídeo 11: Representación animada en volumen de la interfaz estructura-lesión linfoide terciaria con el panel 5 de IBEX (azul: Hoechst, verde: CD206, cian: calretinina). Por favor, haga clic aquí para descargar esta película.
Vídeo 12: Renderizado animado en volumen de la interfaz estructura-lesión linfoide terciaria con el panel 6 de IBEX (azul: Hoechst, verde: Vimentin, rojo: HLADR). Por favor, haga clic aquí para descargar esta película.
Archivo complementario 1: Archivo de impresión de la placa de incubación. Haga clic aquí para descargar este archivo.
Archivo complementario 2: Archivo de impresión para las 36 plataformas individuales. Haga clic aquí para descargar este archivo.
Archivo complementario 3: Archivo de impresión para el anillo de ajuste de altura. Haga clic aquí para descargar este archivo.
Archivo complementario 4: Archivo de impresión para la tapa exterior. Haga clic aquí para descargar este archivo.
Archivo complementario 5: Archivo cortado con láser para el control deslizante. Haga clic aquí para descargar este archivo.
Discusión
Las técnicas multiplex actuales para la obtención de imágenes de fluorescencia están restringidas a secciones delgadas y no proporcionan contexto 3D. El presente estudio describe un protocolo para aplicar el método de imagen IBEX16,17 a tumores intactos montados en plataformas diseñadas a medida. Las lesiones tumorales peritoneales fueron elegidas para resaltar la técnica, ya que los pacientes comúnmente presentan múltiples depósitos tumorales de tamaño ideal que se toman junto con el peritoneo normal circundante como parte de la cirugía de citorreducción estándar realizada en la mayoría de los centros terciarios de referencia en todo el mundo22,23. No obstante, el protocolo es aplicable tanto a las metástasis parenquimatosas como a los tumores en modelo murino, utilizando cortes tumorales, como se ha descrito previamente24,25. Además, el diámetro del anillo de montaje en las plataformas se puede ajustar a las necesidades experimentales/del tejido, aunque las áreas más grandes son propensas a la flacidez del tejido y a tiempos de imagen prolongados. Independientemente de la fuente y el tamaño del tejido, el tiempo entre la obtención y la conservación debe ser mínimo para garantizar una calidad óptima26. La instalación de una mesa de preparación en el quirófano garantiza un procesamiento rápido cuando hay pacientes involucrados. Debe anotarse el tiempo transcurrido desde la obtención hasta la fijación del tejido para garantizar la comparabilidad entre los experimentos.
Las muestras de tejido se toman a través del protocolo IBEX a medida que se realizan montajes completos, es decir, etapas de fijación e imagen, así como de blanqueo, con el tejido intacto adherido a la plataforma. Esto garantiza la conservación de la muestra a través de los ciclos iterativos de tinción/imagen/blanqueo, ya que no hay una interfaz de vidrio de sección semipermanente y no es necesario colocar/quitar repetidamente un cubreobjetos del tejido, lo que puede provocar la pérdida de tejido. Para ahorrar en reactivos, la placa de 9 pocillos diseñada a medida garantiza un ajuste óptimo de la plataforma con un "volumen muerto" mínimo. Todos los anticuerpos utilizados para la tinción son anticuerpos conjugados primarios, y todos los conjugados de fluoróforos utilizados (excepto el colorante de Hoechst) son colorantes Alexa Fluor, que fueron elegidos por su excelente brillo27. Otros fluoróforos Los paneles de anticuerpos se diseñaron de acuerdo con los datos depositados por la comunidad de imágenes IBEX, un repositorio abierto y global que recopila conjuntos de datos, protocolos y comentarios de un grupo internacional de científicos que utilizan IBEX (https://ibeximagingcommunity.github.io/ibex_imaging_knowledge_base/). La secuencia de paneles de anticuerpos se eligió cuidadosamente. En general, los blancos con baja abundancia se ubicaron en ciclos más tempranos. El ciclo 1 de IBEX (CD45, CD44 y Podoplanin) es una excepción, ya que estos tres marcadores se utilizaron para identificar lesiones tumorales en la ronda de tinción inicial.
Los paneles de anticuerpos son comparativamente pequeños para reducir la diafonía de canales a configuraciones de alta intensidad láser, lo cual es necesario para obtener imágenes más profundas del tejido utilizando un microscopio confocal. En la medida de lo posible, se incluyeron conjugados AF750 para ampliar el panel de 2 a 3 anticuerpos. Cabe destacar que no hay muchos conjugados AF750 disponibles comercialmente, y el hecho de que el objetivo de este canal sea muy abundante limita el uso de este fluoróforo. Un ejemplo es el anticuerpo anti-SMA conjugado AF750 que se utilizó en este protocolo. Aunque se trata de un clon que funciona de forma fiable conjugado con AF488 o AF594, la intensidad de la señal de este anticuerpo conjugado con AF750 es significativamente menor, lo que dificulta la generación de pilas Z de alta calidad.
Las incubaciones de anticuerpos de 3 h a temperatura ambiente son cortas en comparación con el protocolo original y reflejan muy probablemente la menor densidad celular en las muestras de tejido peritoneal. Por el contrario, la etapa de blanqueo con borohidruro de litio 1,5 mg/mL durante 60 min fue más estricta. Estos parámetros se determinaron empíricamente y pueden variar según el tejido y los anticuerpos utilizados. Cabe destacar que, debido a la incubación en la solución blanqueadora reactiva, pueden quedar atrapadas pequeñas burbujas de aire en el tejido. Cada vez que esto se hizo evidente durante la etapa de imagen del procedimiento, las muestras se desgasificaron exponiendo la plataforma en la placa de incubación de 9 pocillos al vacío usando un recipiente de succión. Los registros de muestras simples de ITK, a menudo aplicados en los estudios de IBEX, no se pudieron realizar debido a la abrumadora entrada de datos al software de análisis de imágenes. No obstante, las poblaciones de células idénticas se identifican fácilmente comparando conjuntos de datos individuales. En general, los ciclos iterativos se cronometraron de tal manera que un ciclo podía completarse en un día, y las imágenes ocupaban la mayor parte de un ciclo completo (~ 8 h).
La técnica tiene limitaciones que merecen ser mencionadas. El requisito de tiempo del protocolo hace que sea poco probable que este método se adopte en términos generales, sino más bien en determinadas circunstancias. Imaginamos que este protocolo se utilizará mejor para caracterizar los cambios en el microambiente inmunológico del tumor antes y después del tratamiento (por ejemplo, anticuerpos biespecíficos para la inmunoterapia contra el cáncer) o para predecir los resultados generales del tratamiento. A pesar de que este protocolo ofrece caracterización 3D, no se sabe cuánta área debe ser interrogada para describir efectivamente un tumor, incluyendo sus complejas poblaciones celulares28. El acceso a tejido humano fresco también puede ser difícil en ciertos centros, pero prevemos que este protocolo también es aplicable a modelos animales29.
Las aplicaciones de este protocolo son diversas, desde la investigación básica que analiza la composición de TME en múltiples tumores sólidos hasta las biopsias de tejido previas y posteriores a los pacientes inscritos en ensayos clínicos. Aunque los datos que se pueden obtener de una sección de tejido grueso de una sola capa celular son inmensos, la biología 3D puede pasarse por alto fácilmente. Vemos las imágenes 3D como un complemento de otras técnicas de biología espacial, probablemente ayudando en la selección de secciones 2D para análisis posteriores. Además, se está llevando a cabo activamente un protocolo para incorporar un componente de imágenes en vivo durante varias horas utilizando el montaje de tejido y la configuración de imágenes descritos.
Divulgaciones
Ninguno.
Agradecimientos
Esta investigación fue apoyada por el Programa de Investigación Intramuros de los Institutos Nacionales de Salud y el Instituto Nacional del Cáncer (NCI). Esta investigación también fue parcialmente apoyada por el CAT-I, una colaboración de investigación entre el NIAID y el NCI, dirigida por Ronald Germain. Nos gustaría expresar nuestro agradecimiento a Andrea Radtke por sus esfuerzos entusiastas y colaborativos. Su experiencia benefició enormemente este estudio.
Materiales
| Name | Company | Catalog Number | Comments |
| Alexa Fluor Plus 750 Phalloidin | Invitrogen | A30105 | F-actin dye |
| Alexa Fluor 488 anti-human CD206 (MMR) Antibody | BioLegend | 321114 | antibody |
| Alexa Fluor 488 anti-human CD31 Antibody | BioLegend | 303110 | antibody |
| Alexa Fluor 488 anti-human CD4 Antibody | BioLegend | 300519 | antibody |
| Alexa Fluor 488 anti-human HLA-DR Antibody | BioLegend | 307656 | antibody |
| Alexa Fluor 488 anti-mouse/human CD44 Antibody | BioLegend | 103016 | antibody |
| Alexa Fluor 594 anti-human CD45 Antibody | BioLegend | 304060 | antibody |
| Alexa Fluor 647 Anti-Calretinin antibody [EP1798] | Abcam | ab214244 | antibody |
| Alexa Fluor 647 anti-human CD3 Antibody | BioLegend | 300416 | antibody |
| Alexa Fluor 647 anti-human CD8 Antibody | BioLegend | 344726 | antibody |
| Alexa Fluor 647 anti-human Podoplanin Antibody | BioLegend | 337007 | antibody |
| Alexa Fluor 647 anti-Vimentin Antibody | BioLegend | 677807 | antibody |
| Alexa Fluor 647 CD68 Antibody (KP1) | Santa Cruz | sc-20060AF647 | antibody |
| Alexa Fluor 750 Cytokeratin, pan Antibody (AE-1/AE-3) | Novus | NBP2-33200AF750 | antibody |
| Alexa Fluor 750 Human alpha-Smooth Muscle Actin Antibody | R&D | IC1420S | antibody |
| Alexa Fluor 488 CD20 Monoclonal Antibody (L26) eBioscience | Thermo Fisher | 53-0202-82 | antibody |
| Antibiotic-Antimycotic | Gibco | 15240096 | supplement harvest medium |
| BioMed Clear Resin (Form 3) | FormLabs | RS-F2-BMCL-01 | resin used for platform + incubation plate |
| BSA | Sigma | A7906-500g | blocking solution component |
| Cast acrylic 1/32" thick | material used for cutting slider | ||
| CleanStation DT3 | Stratasys | DT3 | post processing for height adjuster & outer lid |
| Container, Specimen | McKesson | 870203 | transfer of tissue from perating table to prep table |
| CorelDraw | CorelDRAW | Software to prepare the vector-based design file for laser cutting | |
| Cytofix | BD Bioscience | 554655 | fixative |
| Dish 15 cm | Falcon | 353025 | dish used during mounting |
| Dish 35 mm No. 1.5 Coverslip 14 mm Glass Diameter Uncoated | Matek | P35G-1.5-14-C | imaging dish |
| DMEM (no glucose) | Gibco | 11966025 | harvest medium |
| Fc Block | BD Bioscience | 564220 | blocking solution component |
| Form 3B+ | Formlabs | Form 3B+ | printer used for platform + incubation plate |
| Form Cure | Formlabs | FH-CU-01 | post processing for platform + incubation plate |
| Form Wash | Formlabs | FH-WA-01 | post processing for platform + incubation plate |
| GrabCAD Print | GrabCAD | GrabCAD Print | Software to prepare models for Stratasys printers |
| Hoechst 33342 10 mg/mL | Biotium | 40046 | nuclear dye |
| J826 Prime 3D Printer | Stratasys | J826 | printer used for height adjuster & outer lid |
| LAS X | Leica | LAS X | Confocal software |
| Laser cutting system | Universal Laser Systems | ULS PLS6.150D | CO2 Laser cutting used for slider |
| Lithium Borohydride | STREM Chemicals | 93-0397 | bleaching chemical |
| PBS, pH 7.4 | Gibco | 10010023 | base Buffer for washing, blocking, staining |
| PreForm | FormLabs | PreForm | Software to prepare models for Formlab printers |
| Silk sutures 2-0 | Ethicon | A305.O35 | affix tissue to platform |
| Stellaris 8 WLL confocal microscope | Leica | STELLARIS 8 | Confocal Imaging |
| Syringe filter | filter ab solution | ||
| Triton X-100 | American Bio | AB02025-00100 | permeabelizing reagent, blocking solution component |
| Vero ContactClear | Stratasys | CTT610, 4 KG | resin used for height adjuster & outer lid |
| Warming Tray | Spring USA | ST-1220 | keep media and tissue warm |
| Water, distilled | Gibco | 15230-170 | diluent for LiBH4 |
Referencias
- Anderson, N. M., Simon, M. C. The tumor microenvironment. Curr Biol. 30 (16), R921-R925 (2020).
- Li, Z., Li, J., Bai, X., Huang, X., Wang, Q. Tumor microenvironment as a complex milieu driving cancer progression: A mini-review. Clin Transl Oncol. , (2024).
- Fridman, W. H., Zitvogel, L., Sautes-Fridman, C., Kroemer, G. The immune contexture in cancer prognosis and treatment. Nat Rev Clin Oncol. 14 (12), 717-734 (2017).
- Roma-Rodrigues, C., Mendes, R., Baptista, P. V., Fernandes, A. R. Targeting tumor microenvironment for cancer therapy. Int J Mol Sci. 20 (4), 840 (2019).
- Sheng, W., et al. Multiplex immunofluorescence: A powerful tool in cancer immunotherapy. Int J Mol Sci. 24 (4), 3086 (2023).
- Bollhagen, A., Bodenmiller, B. Highly multiplexed tissue imaging in precision oncology and translational cancer research. Cancer Discov. 14 (11), 2071-2088 (2024).
- Jin, Y., et al. Advances in spatial transcriptomics and its applications in cancer research. Mol Cancer. 23 (1), 129 (2024).
- Kashyap, A., et al. Quantification of tumor heterogeneity: From data acquisition to metric generation. Trends Biotechnol. 40 (6), 647-676 (2022).
- Chang, Q., Hedley, D. Emerging applications of flow cytometry in solid tumor biology. Methods. 57 (3), 359-367 (2012).
- Zhang, L., et al. Single-cell analyses inform mechanisms of myeloid-targeted therapies in colon cancer. Cell. 181 (2), 442-459.e29 (2020).
- Wang, X. Q., et al. Spatial predictors of immunotherapy response in triple-negative breast cancer. Nature. 621 (7980), 868-876 (2023).
- Maestri, E., et al. Spatial proximity of tumor-immune interactions predicts patient outcome in hepatocellular carcinoma. Hepatology. 79 (4), 768-779 (2024).
- Wang, Q., et al. Role of tumor microenvironment in cancer progression and therapeutic strategy. Cancer Med. 12 (10), 11149-11165 (2023).
- Veninga, V., Voest, E. E. Tumor organoids: Opportunities and challenges to guide precision medicine. Cancer Cell. 39 (9), 1190-1201 (2021).
- Lin, J. R., et al. Multiplexed 3d atlas of state transitions and immune interaction in colorectal cancer. Cell. 186 (2), 363-381.e19 (2023).
- Radtke, A. J., et al. Ibex: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Radtke, A. J., et al. Ibex: A versatile multiplex optical imaging approach for deep phenotyping and spatial analysis of cells in complex tissues. Proc Natl Acad Sci U S A. 117 (52), 33455-33465 (2020).
- Van Baal, J. O., et al. The histophysiology and pathophysiology of the peritoneum. Tissue Cell. 49 (1), 95-105 (2017).
- Bridda, A., Padoan, I., Mencarelli, R., Frego, M. Peritoneal mesothelioma: A review. MedGenMed. 9 (2), 32 (2007).
- Demuytere, J., Ernst, S., Van Ovost, J., Cosyns, S., Ceelen, W. The tumor immune microenvironment in peritoneal carcinomatosis. Int Rev Cell Mol Biol. 371, 63-95 (2022).
- Wang, Q., et al. Heterogeneity of tertiary lymphoid structures predicts the response to neoadjuvant therapy and immune microenvironment characteristics in triple-negative breast cancer. Br J Cancer. , (2024).
- Kepenekian, V., et al. Peritoneal mesothelioma: Systematic review of hyperthermic intraperitoneal chemotherapy (HIPEC) protocol outcomes. Indian J Surg Oncol. 14 (Suppl 1), 39-59 (2023).
- Mcquade, C., Renton, M., Chouhan, A., Macdermott, R., O'brien, C. Review of imaging peritoneal disease and treatment. Can Assoc Radiol J. , (2024).
- Kenerson, H. L., et al. Tumor slice culture as a biologic surrogate of human cancer. Ann Transl Med. 8 (4), 114 (2020).
- Arrizabalaga, L., et al. Tumor slice culture system for ex vivo immunotherapy studies. Methods Cell Biol. 189, 55-69 (2024).
- Neumeister, V. M., et al. Quantitative assessment of effect of preanalytic cold ischemic time on protein expression in breast cancer tissues. J Natl Cancer Inst. 104 (23), 1815-1824 (2012).
- Panchuk-Voloshina, N., et al. Alexa dyes: A series of new fluorescent dyes that yield exceptionally bright, photostable conjugates. J Histochem Cytochem. 47 (9), 1179-1188 (1999).
- Rajaram, S., et al. Sampling strategies to capture single-cell heterogeneity. Nat Methods. 14 (10), 967-970 (2017).
- Bella, A., et al. Mouse models of peritoneal carcinomatosis to develop clinical applications. Cancers (Basel). 13 (5), 963 (2021).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados