Method Article
L'imaging tridimensionale di tessuti tumorali utilizzando lo sbiancamento iterativo estende l'approccio alla multiplexità
In questo articolo
Riepilogo
Questo articolo descrive un protocollo per l'immunofluorescenza multiplex ottimizzato per caratterizzare l'architettura tridimensionale delle metastasi peritoneali.
Abstract
L'eterogeneità spaziale del microambiente tumorale (TME) è un determinante critico della risposta terapeutica, in particolare per gli agenti immuno-oncologici, il cui successo dipende dalla distribuzione di specifiche sottopopolazioni di cellule immunitarie. Nell'ultimo decennio, sono state introdotte diverse tecnologie sofisticate per ottenere una risoluzione dettagliata del TME utilizzando sezioni bidimensionali di tessuti fissati in formalina, inclusi in paraffina (FFPE) o fissati congelati. Sebbene queste sezioni sottili siano più facili da reperire e analizzare, mancano dell'architettura tridimensionale necessaria per caratterizzare in modo affidabile e completo un tumore. Per ovviare a questa limitazione, è stata sviluppata una tecnica di montaggio e imaging tissutale per consentire l'analisi tridimensionale delle lesioni tumorali nel loro stato nativo in vivo . Questo protocollo delinea l'approvvigionamento di tessuto tumorale umano, il montaggio di campioni su piattaforme stampate su misura e le procedure di colorazione per i campioni post-fissazione. La tecnica di immunofluorescenza multiplexata, IBEX (Iterative Bleaching Extends Multiplexity), è stata adattata per caratterizzare il TME tridimensionale con un massimo di 15 marcatori per cellule tumorali, immunitarie e stromali utilizzando anticorpi disponibili in commercio. Le profondità di imaging fino a 100 μm sono state raggiunte utilizzando un microscopio confocale laser a luce bianca invertita con un adattatore di imaging stampato su misura e piastre commerciali con fondo in vetro per garantire un orientamento ottimale dei tessuti. Questo protocollo evidenzia il potenziale del metodo IBEX per ampliare gli studi di immunofluorescenza multiplex, fornendo una comprensione più completa della composizione del TME.
Introduzione
I tumori solidi sono altamente variabili, con un'intricata composizione di tipi di cellule (maligne e non maligne), proteine della matrice extracellulare e fattori solubili 1,2. In definitiva, questo microambiente tumorale (TME) altamente complesso ed eterogeneo determina la suscettibilità a trattamenti come l'immunoterapia 3,4. Pertanto, tecnologie come l'immunofluorescenza altamente multiplexata 5,6 e la trascrittomica spaziale7 sono state applicate per caratterizzare la complessità del TME in sezioni spesse uno strato cellulare (~4 μM).
A differenza degli approcci di dispersione cellulare come il sequenziamento di singole cellule e la citometria a flusso 8,9,10, queste tecniche preservano le relazioni spaziali per fornire informazioni critiche sul crosstalk cellulare e possono essere impiegate per identificare potenziali biomarcatori per la previsione della risposta al trattamento 11,12,13. Tuttavia, le lesioni tumorali sono strutture intrinsecamente tridimensionali (3D) e gli approcci 2D come quelli sopra menzionati non catturano adeguatamente il complesso panorama cellulare. L'importanza della biologia 3D è stata apprezzata nel campo, come evidenziato dall'ampio utilizzo di colture di organoidi tumorali derivate da pazienti14. Tuttavia, gli organoidi non conservano la complessità cellulare del TME originale in vivo, il che ne limita l'applicabilità per l'uso con farmaci immunomodulatori o per la caratterizzazione delle diverse popolazioni di cellule TME.
Sebbene sia possibile eseguire analisi su sezioni 2D seriali e "cucire" computazionalmente le immagini per un rendering 3D, questa tecnica è proibitiva in termini di costi e difficile da realizzare con l'alta fedeltà15. Per caratterizzare in modo economico i tessuti 3D (ad es. tumori) in modo riproducibile, è stato sviluppato e dimostrato un metodo unico per la preparazione e la colorazione/analisi in immunofluorescenza multiplex utilizzando lesioni tumorali peritoneali di un paziente con carcinoma peritoneale primario.
Basato su un adattamento della consolidata tecnica IBEX (Iterative Bleaching Extends Multiplexity)16,17, questo protocollo descrive in dettaglio la stampa 3D di un supporto per tessuti e di un adattatore per imaging, l'approvvigionamento e il montaggio del tessuto peritoneale, cicli multipli di colorazione con vari marcatori di tumore e tipo cellulare e imaging 3D confocale ad alta risoluzione.
Protocollo
Lo studio è stato condotto con l'approvazione del National Institutes of Health Institutional Review Board secondo il protocollo NCT01915225. Il tessuto umano è stato ottenuto con il consenso informato scritto al momento della laparoscopia diagnostica. Tutto il tessuto era portatore di tumore, come determinato dall'ispezione visibile e confermato dall'esame istopatologico finale. I dettagli di tutti i reagenti e le attrezzature utilizzate sono elencati nella tabella dei materiali. Nella Figura 1 viene fornita un'illustrazione schematica dell'intero flusso di lavoro.
1. Piattaforme di stampa, adattatore di imaging e piastra di incubazione
- Procedure di slicing/stampa 3D/post-elaborazione per la piattaforma e le parti della piastra a 9 pozzetti (descritte nella Figura 2A-C)
- Slicing: Aprire il software di slicing e importare i file di disegno in formato . Formato STL (File Supplementare 1 e File Supplementare 2). Orienta i modelli per una stampa ottimale e aggiungi supporti. Seleziona la stampante per uso medico, seleziona la resina trasparente e l'altezza dello strato di 50 μm . Verificare la stampabilità e inviare il lavoro alla stampante.
- Stampa 3D: verifica che la stampante sia pronta (cartuccia di resina, serbatoio e piattaforma corretti in posizione). Sul touchscreen della stampante, selezionare il lavoro caricato dal software slicer per avviare la stampa. Una volta completata la stampa, utilizzare lo strumento di rimozione per staccare la parte stampata dalla piattaforma.
- Post-elaborazione: Sciacquare la parte nell'unità di lavaggio riempita con alcol isopropilico (IPA) al 99% per 15 minuti. Rimuovere le parti dall'unità di lavaggio e immergerle in alcool isopropilico fresco per 5 minuti.
- Polimerizzare la parte per 60 minuti a 60 °C. Rimuovere i supporti utilizzando tronchesi. Se necessario, carteggiare eventuali segni di supporto con carta vetrata a grana fine.
- Procedure di affettatura/stampa 3D/post-elaborazione per il regolatore di altezza e il coperchio esterno (descritti nella Figura 2A-C)
- Slicing: apri il software di stampa e importa i file di disegno in formato . Formato STL (File Supplementare 3 e File Supplementare 4). Orientare i modelli e assegnare i materiali. Selezionare la modalità di stampa desiderata (Miscelazione alta, alta velocità) e impostare le impostazioni del modello per la finitura su Opaco. Verificare la stampabilità e inviare il lavoro alla stampante.
- Stampa 3D: assicurati che la stampante abbia caricato i materiali corretti e che il vassoio di costruzione sia pulito. Sull'interfaccia del monitor della stampante, selezionare il lavoro caricato per avviare la stampa. Una volta completata la stampa, utilizzare lo strumento raschietto per rimuovere delicatamente la parte stampata dal vassoio di stampa.
- Post-elaborazione: Posizionare i pezzi stampati in un pulitore a getto d'acqua o in un sistema di pulizia ad agitazione per rimuovere il materiale di supporto. Eseguire l'eventuale rimozione manuale del supporto, se necessario, utilizzando strumenti come pinzette o pinze.
- Taglio laser del cursore della piattaforma (mostrato nella Figura 2A, C)
- Preparazione del design: Importare il file *. File di progettazione DWG nel software vettoriale (File supplementare 5). Imposta i colori delle linee su rosso (RGB) per il taglio vettoriale. Invia il lavoro al laser cutter.
- Configurazione del laser cutter: apri il software del laser cutter e importa il file di progettazione. Seleziona il tipo di materiale e regola le impostazioni per potenza, velocità e risoluzione per acrilico spesso 1/32". Posizionare il materiale sul letto laser, assicurarsi che sia allineato correttamente e mettere a fuoco il laser.
- Taglio laser: avviare il processo di taglio laser e monitorare per assicurarsi che il materiale venga tagliato correttamente.
2. Montaggio di tessuti su piattaforme stampate
- Preparazione del terreno di raccolta e della tabella di preparazione
- Integrare 100 ml di DMEM (basso contenuto di glucosio) con Anti-Anti e mettere in un'incubatrice a 37 °C fino al momento del bisogno (terreno di raccolta).
- Allestire un "tavolo di preparazione" in sala operatoria con i seguenti elementi: piattaforme stampate in 3D autoclavate, confezione da 2-0 fascette per suture in seta, piastra riscaldante, tazza per campioni con 50 ml di terreno di coltura preriscaldato, piastra di coltura da 15 cm contenente 30 ml di terreno di coltura preriscaldato e una piastra da 24 pozzetti con 4 pozzetti ciascuno contenente 1,5 ml di terreno di coltura (posizionare il tutto sulla piastra riscaldante), 1 paio di pinze e 1 paio di forbici chirurgiche.
- Resezione e montaggio dei tessuti
NOTA: Il tessuto viene prelevato dalla sala operatoria. Le aree del peritoneo con focolai di tumore di spessore e diametro inferiori a 0,5 mm sono identificate all'ispezione visiva (Figura 2D). Da notare, ~ 2 cm2 di tessuto (peritoneo) sono necessari per preparare una piattaforma.- Separare con cura il peritoneo portatore del tumore dalla muscolatura e dalla fascia della parete addominale sottostante (eseguita dal chirurgo). Trasferire i fogli di tessuto risultanti in una tazza per campioni contenente un terreno di coltura caldo e posizionarli su una piastra riscaldante (tavolo di preparazione).
- Montare il tessuto su piattaforme nella piastra da 15 cm contenente il terreno di raccolta. Avvolgere con cura il tessuto tumorale (lato mesoteliale rivolto verso l'alto) sul lato del piccolo orifizio della piattaforma e fissarlo in posizione con una sutura di seta 2-0. Posizionare la piattaforma di tessuto preparata inversamente (sommersa) in una piastra a 24 pozzetti contenente il terreno di raccolta (Figura 2E-G).
NOTA: Montare il tessuto abbastanza teso da evitare pieghe sulla superficie della piattaforma, ma non generare tensione in quanto ciò indurrà cambiamenti biomeccanici e fisiologici indesiderati.
3. Procedura di fissaggio/colorazione/sbiancamento
NOTA: Le modifiche rispetto al protocollo originale su misura per FFPE di 4 μm di spessore o sezioni congelate fisse sono descritte nella Figura 3. La Tabella 1 fornisce un elenco di anticorpi (con diluizioni) utilizzati per i cicli IBEX 1-6.
- Fissazione
- Preparare 40 mL di tampone di fissazione aggiungendo 10 mL di stock di fissativo a una provetta conica da 50 mL contenente 30 mL di 1x PBS freddo.
- Utilizzando un paio di pinze, trasferire il tessuto montato sulle piattaforme nel tampone di fissazione e incubare per 24 ore a 4 °C.
- Decantare il fissativo e sostituirlo con 40 mL di 1x PBS freddo e incubare per 10 minuti a 4 °C.
- Macchiatura
- Aggiungere 1 mL di tampone bloccante (1% BSA, 0,3% Triton-X-100, 1% Fc Block in 1x PBS filtrato con filtro per siringa da 0,22 μm) a ciascun pozzetto e inserire le piattaforme tissutali. Bloccare per almeno 2 ore a temperatura ambiente su un bilanciere impostato a bassa velocità (30 giri/min).
NOTA: Assicurarsi che la tacca sulla piattaforma e la scanalatura nel pozzo siano allineate. Questo passaggio non è sensibile al fattore tempo e può essere eseguito durante la notte a 4 °C. - Preparare 0,8 mL di soluzione anticorpo/colorante in una provetta da microcentrifuga e centrifugare a 10.000 x g per 5 minuti a temperatura ambiente. Trasferire 0,75 mL in un pozzetto pulito della piastra di incubazione a 9 pozzetti e inserire una piattaforma lavata.
- Avvolgere la piastra in un foglio di alluminio e incubare per almeno 2 ore a temperatura ambiente su un bilanciere impostato a bassa velocità (30 giri/min).
- Lavare la piattaforma in una provetta conica da 50 mL con 40 mL di 1x PBS per 10 minuti a 4 °C. La piattaforma è ora pronta per essere inserita nell'adattatore di imaging.
- Aggiungere 1 mL di tampone bloccante (1% BSA, 0,3% Triton-X-100, 1% Fc Block in 1x PBS filtrato con filtro per siringa da 0,22 μm) a ciascun pozzetto e inserire le piattaforme tissutali. Bloccare per almeno 2 ore a temperatura ambiente su un bilanciere impostato a bassa velocità (30 giri/min).
- Candeggio
- Dopo ogni ciclo di colorazione e imaging, sbiancare i fluorofori utilizzando l'agente riducente dell'estere boroidruro di litio (LiBH4). Questo trattamento eliminerà i segnali di fluorescenza da Alexa Fluor (AF)488, AF647 e AF750, ma non da AF594 e dal colorante nucleare Hoechst.
ATTENZIONE: Il boroidruro di litio è estremamente reattivo con l'acqua. L'idrogeno gassoso infiammabile viene rilasciato a contatto con l'acqua; Il boroidruro di litio esaurito e i prodotti monouso contaminati da boroidruro di litio devono essere smaltiti come rifiuti pericolosi. - Preparare 5 mL di soluzione di LiBH4 da 1,5 mg/mL in acqua distillata (in una cappa chimica) e incubare per 30 minuti a temperatura ambiente.
NOTA: La soluzione LiBH4 perde la sua efficacia riducente/sbiancante se preparata >4 ore prima dell'uso. - Trasferire 1 mL di soluzione di LiBH4 in un pozzetto pulito della piastra di incubazione a 9 pozzetti e inserire la piattaforma lavata per 60 minuti a temperatura ambiente.
- Lavare brevemente la piattaforma in 20 mL di 1x PBS in una provetta conica e verificare la presenza del segnale di fluorescenza rimanente utilizzando l'impostazione laser più alta per la compensazione Z nel ciclo di imaging precedente prima di procedere ai successivi cicli di colorazione.
- Ripetere il blocco del campione per 2 ore a temperatura ambiente prima di procedere alla successiva fase di incubazione degli anticorpi.
- Dopo ogni ciclo di colorazione e imaging, sbiancare i fluorofori utilizzando l'agente riducente dell'estere boroidruro di litio (LiBH4). Questo trattamento eliminerà i segnali di fluorescenza da Alexa Fluor (AF)488, AF647 e AF750, ma non da AF594 e dal colorante nucleare Hoechst.
4. Procedura di imaging
- Assemblaggio di adattatori/piattaforme di imaging
- Aggiungere 0,1 ml di 1x PBS al centro del fondo di vetro di una piastra per immagini da 3,5 cm e mettere da parte. Avvitare l'anello di regolazione dell'altezza in senso orario nel coperchio esterno e montare la piattaforma nel supporto interno in plastica (allineamento tacca-scanalatura). Fissare la piattaforma con il cursore.
- Posizionare l'adattatore di imaging assemblato sul piatto inferiore in vetro e abbassare con cautela l'anello di regolazione dell'altezza finché il tessuto non tocca il tampone. Evitare di ruotare la piattaforma sul fondo in vetro.
- Una volta raggiunta la distanza ottimale dal vetro, aggiungere 0,2 mL di 1x PBS sul tessuto (ora con il lato inferiore rivolto verso l'alto) per evitare la disidratazione dei tessuti durante l'imaging.
- Imaging confocale
- Accendere tutti i componenti del microscopio confocale invertito, inclusi il computer, il microscopio, il laser e la sorgente luminosa a LED per l'uso dell'oculare. Avviare il software di acquisizione delle immagini .
- Selezionare l'obiettivo appropriato (ad es. 20x o 40x) e aggiungere il mezzo di immersione appropriato. Ad esempio, in questo studio viene utilizzato un obiettivo d'acqua 20x; ~200 μL di acqua vengono aggiunti all'obiettivo per evitare l'essiccazione durante le lunghe acquisizioni di immagini.
- Posizionare la capsula di imaging assemblata nel portacampioni sul tavolino del microscopio e assicurarsi che il campione sia sicuro (non si muova).
NOTA: Quando si posiziona il campione assemblato sul tavolino del microscopio, prendere nota della posizione della tacca e utilizzare la stessa posizione per i cicli iterativi per facilitare l'allineamento dell'immagine. È importante mantenere l'adattatore di imaging all'altezza ottimale per cicli consecutivi. - Centrare il campione utilizzando il controllo X-Y e portare l'obiettivo fino al vetro di copertura. Guardando attraverso l'oculare e utilizzando il segnale di Hoechst come riferimento, spostare l'obiettivo per ottenere una messa a fuoco fine sul tessuto.
- Selezionare le linee laser appropriate o regolare WLL in base agli spettri di eccitazione ed emissione dei fluorofori utilizzati per colorare il campione di tessuto. Elimina la sovrapposizione tra i canali regolando le impostazioni e aggiungi sequenze se i fluorofori hanno spettri di eccitazione simili.
- Scegli i seguenti parametri di acquisizione delle immagini: velocità di scansione 600 Hz, risoluzione XY 1024 x 1024, medie a 3 linee e scansione bidirezionale .
- Scegliere i seguenti parametri Z-stack: (1) utilizzare le dimensioni Z-step ottimizzate per l'obiettivo utilizzato, (2) definire l'inizio e la fine dello stack, (3) regolare i parametri di compensazione Z per ottenere la stessa luminosità in tutto lo stack e (4) acquisire uno stack di test da 1 riquadro con una media.
- Definisci la regione di cui creare l'immagine e acquisisci uno stack tile-Z ad alta risoluzione.
Risultati
Il peritoneo è una struttura membranosa di spessore inferiore a 1 mm che riveste la superficie della parete addominale ed è contigua alle superfici degli organi addominali. È composto da mesotelio superficiale e tessuto connettivo sottostante intervallato da cellule adipose, linfociti, macrofagi e fibroblasti, nonché da vasi sanguigni e linfatici. Le neoplasie peritoneali derivano più spesso da metastasi tumorali addominali (ad esempio, carcinoma ovarico o carcinoma gastrico)18 o, meno frequentemente, possono essere tumori primari del peritoneo (ad esempio, mesotelioma)19. Queste lesioni tumorali, spesso piccole (<1 cm), sono molto complesse ma sono suscettibili di caratterizzazione 3D utilizzando la tecnica descritta. Una proiezione massima di cinque sezioni confocali viene utilizzata per visualizzare l'intero campione montato su una piattaforma (Figura 4, riquadro nell'angolo in alto a sinistra). Per il primo ciclo di IBEX sono stati utilizzati tre anticorpi coniugati primari (AF488-CD44, AF594-CD45 e AF647-Podoplanina) insieme al colorante nucleare Hoechst (IBEX1, Figura 4). Le lesioni tumorali diventano evidenti dalle strutture a rosetta che sono doppiamente positive per CD44 e Podoplanina e mostrano una disposizione dei nuclei caratteristica del mesotelioma papillare.
Le cellule tumorali sono state identificate utilizzando la morfologia cellulare e due anticorpi poiché sia l'anti-Podoplanina che l'anti-CD44 riconoscono le cellule non tumorali, come evidenziato dal rilevamento di vasi linfatici tramite anti-Podoplanina e dal legame dell'anticorpo CD44 a sottogruppi di cellule immunitarie. Le regioni di interesse (ROI) più piccole vengono quindi acquisite utilizzando un formato di scansione delle tessere a risoluzione più elevata con dimensioni ottimizzate del passo Z per il rendering di proiezioni 3D (Figura 5A). Le piattaforme sono state sottoposte alle procedure di colorazione iterativa e le stesse ROI sono state visualizzate nei cicli IBEX 1-6. Le immagini rappresentative per le interfacce del tumore (riquadro giallo) e della struttura linfoide tumorale-terziaria (TLS) (riquadro blu) sono assemblate nella Figura 5B-M.
La presenza di TLS e la densità delle cellule CD3-positive indicano una significativa infiltrazione di cellule immunitarie20 e possibilmente risposta ai farmaci immunomodulatori21. Da notare che la TLS e la maggior parte delle lesioni tumorali sono presenti in diverse profondità Z del tessuto, come dimostrato confrontando le proiezioni stack di diverse sezioni ottiche nella Figura 6, evidenziando i vantaggi dell'imaging 3D e la potenziale biologia mancata quando l'imaging 2D viene applicato a strutture 3D. Il carattere 3D di queste lesioni diventa più evidente nei file dei filmati (Filmati 1-12), che raffigurano le rappresentazioni volumetriche delle singole lesioni nella Figura 5.
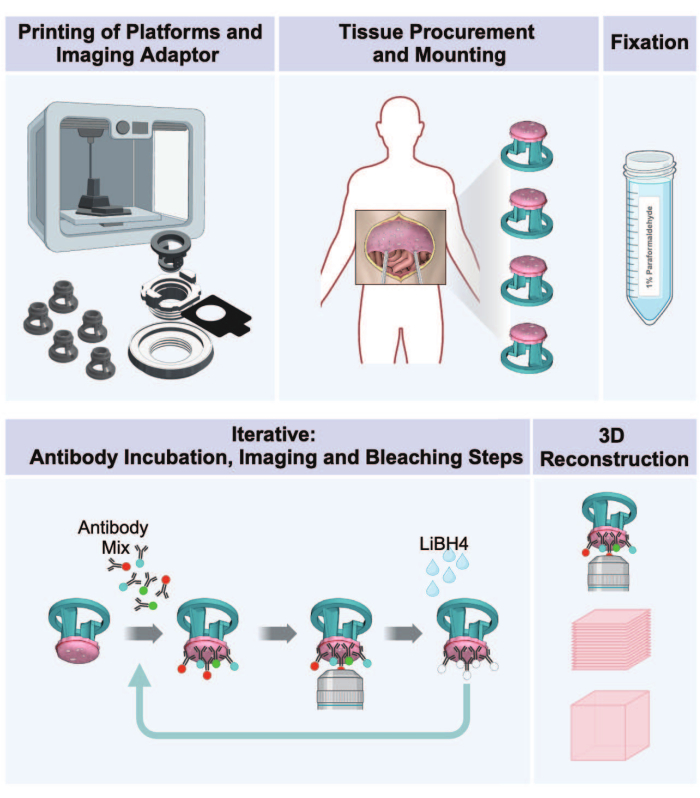
Figura 1: Flusso di lavoro dalla stampa 3D all'imaging 3D dei tumori peritoneali. Il protocollo consiste in una sequenza di cinque passaggi per preparare immagini multiplex 3D dal tessuto peritoneale portatore di tumore. (1) Stampa di piattaforme riceventi di tessuti, parti di adattatori di imaging e piastre di incubazione. (2) Procedure di approvvigionamento e montaggio dei tessuti. (3) Fissazione con paraformaldeide. (4) Cicli di colorazione e imaging IBEX. (5) Ricostruzione 3D utilizzando software di imaging. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Componenti hardware con interfaccia tissutale per l'imaging 3D. (A) Diagramma schematico dell'assemblaggio della piattaforma all'interno dell'adattatore di imaging. (B) Una piastra di incubazione a 9 pozzetti. (C) Fotografie dei componenti dell'adattatore di immagine con assemblaggio. (D) Peritoneo portatore di mesotelioma come osservato durante la laparoscopia diagnostica. (E) Montaggio del tessuto sulla piattaforma. (F,G) Peritoneo tumorale montato su piattaforme. Clicca qui per visualizzare una versione più grande di questa figura.
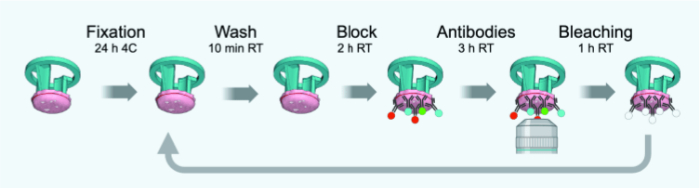
Figura 3: Schema cronologico dei cicli IBEX. Clicca qui per visualizzare una versione più grande di questa figura.
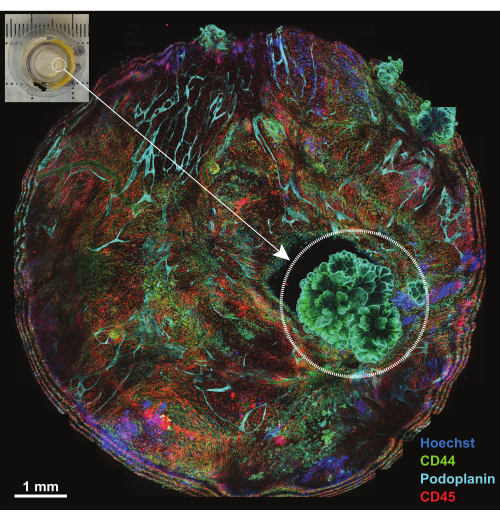
Figura 4: Panoramica della piattaforma dopo l'incubazione con il pannello IBEX1. Proiezione massima dell'intera piattaforma dopo l'incubazione con il pannello IBEX1 (Hoechst, AF488-CD44, AF594-CD45 e AF647-Podoplanin). Il cerchio tratteggiato evidenzia il tumore. Riquadro: Fotografia del peritoneo tumorale montato sulla piattaforma di imaging. Barra della scala: 1 mm. Clicca qui per visualizzare una versione più grande di questa figura.
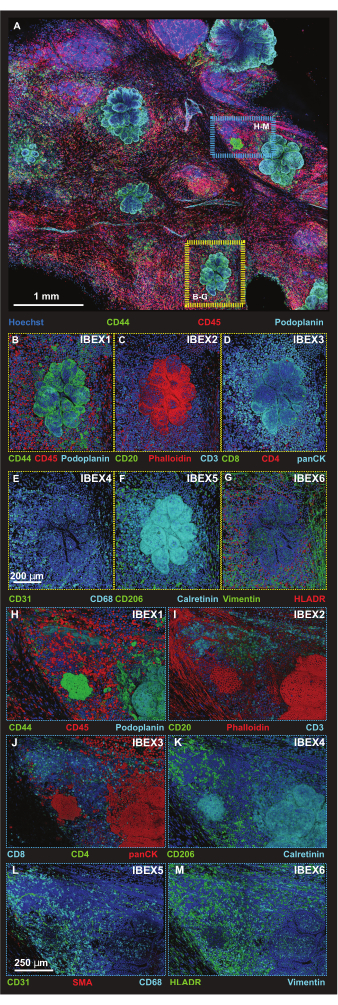
Figura 5: Immagini in immunofluorescenza ad alta risoluzione di regioni selezionate della piattaforma. (A) Proiezione massima di un'immagine Z-stack a 56 tile acquisita dopo l'incubazione con il pannello IBEX1 (Hoechst, AF488-CD44, AF594-CD45 e AF647-Podoplanin). Rettangolo giallo: lesione tumorale mostrata nei pannelli (B-G). Rettangolo blu: interfaccia della struttura linfoide tumorale-terziaria mostrata in (H-M). (B-G) Una singola lesione tumorale con pannello IBEX 1-6 in rendering di volume. (B) IBEX1, verde: CD44, rosso: CD45, ciano: Podoplanina. (C) IBEX2, verde: CD20, rosso: F-actina, ciano: CD3. (D) IBEX3, verde: CD8, rosso: CD4, ciano: panCK. (E) IBEX4, verde: CD31, ciano: CD68. (F) IBEX5, verde: CD206, ciano: calretitina. (G) IBEX6, verde: Vimentina, rosso: HLADR. (H-M) Una singola struttura linfoide tumorale-terziaria si interfaccia con il pannello IBEX 1-6 nel rendering del volume. (H) IBEX1, verde: CD44, rosso: CD45, ciano: Podoplanina. (I) IBEX2, verde: CD20, rosso: F-actina, ciano: CD3. (J) IBEX3, verde: CD4, rosso: panCK, ciano: CD8. (K) IBEX4, verde: CD31, rosso: SMA, ciano: CD68. (L) IBEX5, verde: CD206, ciano: calretitina. (M) IBEX6, verde: HLADR, ciano: Vimentina. Tutti i pannelli, blu: Hoechst. Barre della scala: (A, 1 mm); (B-M, 250 μm). Clicca qui per visualizzare una versione più grande di questa figura.
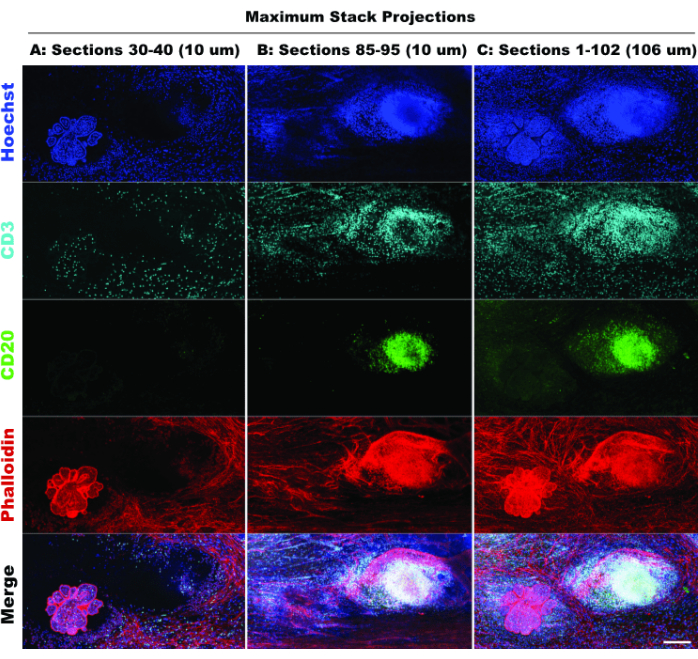
Figura 6: Imaging 2D simulato. Il confronto tra proiezioni limitate (10 sezioni ottiche) e full stack (102 sezioni ottiche) conferma la perdita di contesto tissutale nella simulazione 2D. (A) Proiezione massima delle sezioni 30-40 (10 μm). (B) Proiezione massima delle sezioni 85-95 (10 μm). (C) Proiezione massima delle sezioni 1-102 (~100 μm). Tutti i pannelli, IBEX2 (Hoechst, AF488-CD20, AF647-CD3, AF790-falloidina). Barra della scala: 200 μm. Fare clic qui per visualizzare una versione più grande di questa figura.
Tabella 1: Anticorpi utilizzati per i singoli pannelli IBEX. Clicca qui per scaricare questa tabella.
Filmato 1: Rendering animato a volume della singola lesione con pannello IBEX 1 (blu: Hoechst, verde: CD44, rosso: CD45, ciano: Podoplanina). Clicca qui per scaricare questo filmato.
Filmato 2: Rendering animato del volume della singola lesione con pannello IBEX 2 (blu: Hoechst, verde: CD20, rosso: F-actina, ciano: CD3). Clicca qui per scaricare questo filmato.
Filmato 3: Rendering animato a volume della singola lesione con pannello IBEX 3 (blu: Hoechst, verde: CD8, rosso: CD4, ciano: panCK). Clicca qui per scaricare questo filmato.
Filmato 4: Rendering animato a volume della singola lesione con pannello IBEX 4 (blu: Hoechst, verde: CD31, ciano: CD68). Clicca qui per scaricare questo filmato.
Filmato 5: Rendering animato del volume della singola lesione con pannello IBEX 5 (blu: Hoechst, verde: CD206, ciano: Calretinina). Clicca qui per scaricare questo filmato.
Filmato 6: Rendering animato in volume della singola lesione con pannello IBEX 6 (blu: Hoechst, verde: Vimentina, rosso: HLADR). Clicca qui per scaricare questo filmato.
Filmato 7: Rendering animato del volume dell'interfaccia struttura-lesione linfoide terziaria con pannello IBEX 1 (blu: Hoechst, verde: CD44, rosso: CD45, ciano: Podoplanina). Clicca qui per scaricare questo filmato.
Filmato 8: Rendering animato del volume dell'interfaccia struttura-lesione linfoide terziaria con pannello IBEX 2 (blu: Hoechst, verde: CD20, rosso: F-actina, ciano: CD3). Clicca qui per scaricare questo filmato.
Filmato 9: Rendering animato del volume dell'interfaccia struttura-lesione linfoide terziaria con pannello IBEX 3 (blu: Hoechst, verde: CD8, rosso: CD4, ciano: panCK). Clicca qui per scaricare questo filmato.
Filmato 10: Rendering animato del volume dell'interfaccia struttura-lesione linfoide terziaria con il pannello IBEX 4 (blu: Hoechst, verde: CD31, rosso: SMA, ciano: CD68). Clicca qui per scaricare questo filmato.
Filmato 11: Rendering animato del volume dell'interfaccia struttura-lesione linfoide terziaria con il pannello IBEX 5 (blu: Hoechst, verde: CD206, ciano: Calretinina). Clicca qui per scaricare questo filmato.
Filmato 12: Rendering animato del volume dell'interfaccia struttura-lesione linfoide terziaria con pannello IBEX 6 (blu: Hoechst, verde: Vimentina, rosso: HLADR). Clicca qui per scaricare questo filmato.
File supplementare 1: File di stampa per la piastra di incubazione. Clicca qui per scaricare questo file.
File supplementare 2: file di stampa per le 36 singole piattaforme. Clicca qui per scaricare questo file.
File supplementare 3: File di stampa per l'anello di regolazione dell'altezza. Clicca qui per scaricare questo file.
File supplementare 4: File di stampa per il coperchio esterno. Clicca qui per scaricare questo file.
File supplementare 5: File tagliato al laser per il cursore. Clicca qui per scaricare questo file.
Discussione
Le attuali tecniche multiplex per l'imaging a fluorescenza sono limitate a sezioni sottili e non forniscono un contesto 3D. Il presente studio descrive un protocollo per applicare il metodo di imaging IBEX16,17 a tumori intatti montati su piattaforme progettate su misura. Le lesioni tumorali peritoneali sono state scelte per evidenziare la tecnica in quanto i pazienti presentano comunemente depositi tumorali multipli di dimensioni ideali che vengono prelevati insieme al peritoneo normale circostante come parte della chirurgia di citoriduzione standard intrapresa nella maggior parte dei centri di riferimento terziari in tutto il mondo22,23. Il protocollo è comunque applicabile sia alle metastasi parenchimali che ai tumori modello murino utilizzando fette tumorali come è stato precedentemente descritto24,25. Inoltre, il diametro dell'anello di montaggio sulle piattaforme può essere regolato in base alle esigenze sperimentali/tissutali, sebbene le aree più grandi siano soggette a cedimenti dei tessuti e tempi di imaging prolungati. Indipendentemente dalla fonte e dalle dimensioni del tessuto, il tempo tra l'approvvigionamento e la conservazione deve essere ridotto al minimo per garantire una qualità ottimale26. L'allestimento di un tavolo di preparazione in sala operatoria garantisce una pronta elaborazione quando sono coinvolti i pazienti. Il tempo che intercorre tra l'approvvigionamento e la fissazione del tessuto deve essere annotato per garantire la comparabilità tra gli esperimenti.
I campioni di tessuto vengono prelevati attraverso il protocollo IBEX mentre le montature intere, ovvero la fissazione e l'imaging, nonché le fasi di sbiancamento, vengono eseguite con il tessuto intatto fissato alla piattaforma. Ciò garantisce la conservazione del campione attraverso i cicli iterativi di colorazione/imaging/sbiancamento, poiché non esiste un'interfaccia sezione semipermanente e non è necessario posizionare/rimuovere ripetutamente un vetro di copertura dal tessuto, entrambi fattori che possono portare alla perdita di tessuto. Per risparmiare sui reagenti, la piastra a 9 pozzetti progettata su misura garantisce un adattamento ottimale della piattaforma con un "volume morto" minimo. Tutti gli anticorpi utilizzati per la colorazione sono anticorpi coniugati primari e tutti i coniugati fluorofori utilizzati (ad eccezione del colorante Hoechst) sono coloranti Alexa Fluor, che sono stati scelti per la loro eccellente luminosità27. Altri pannelli di anticorpi fluorofori sono stati progettati in base ai dati depositati dalla comunità di imaging IBEX, un repository globale aperto che raccoglie set di dati, protocolli e feedback da un gruppo internazionale di scienziati che utilizzano IBEX (https://ibeximagingcommunity.github.io/ibex_imaging_knowledge_base/). La sequenza dei pannelli anticorpali è stata scelta con cura. In generale, i bersagli con bassa abbondanza sono stati collocati nei cicli precedenti. Il ciclo IBEX 1 (CD45, CD44 e Podoplanina) è un'eccezione poiché questi tre marcatori sono stati utilizzati per identificare le lesioni tumorali nel ciclo di colorazione iniziale.
I pannelli di anticorpi sono relativamente piccoli per ridurre la diafonia dei canali con impostazioni di alta intensità laser, necessarie per visualizzare il tessuto più in profondità utilizzando un microscopio confocale. I coniugati AF750 sono stati inclusi ove possibile per espandere il pannello da 2 a 3 anticorpi. Da notare che non ci sono molti coniugati AF750 disponibili in commercio, e il fatto che il bersaglio per questo canale dovrebbe essere molto abbondante limita l'uso di questo fluoroforo. Un esempio è l'anticorpo anti-SMA coniugato AF750 che è stato utilizzato in questo protocollo. Sebbene si tratti di un clone che funziona in modo affidabile coniugato con AF488 o AF594, l'intensità del segnale per questo anticorpo coniugato con AF750 è significativamente inferiore, il che rende difficile generare Z-stack di alta qualità.
Le incubazioni degli anticorpi di 3 ore a temperatura ambiente sono brevi rispetto al protocollo originale e riflettono molto probabilmente la minore densità cellulare nei campioni di tessuto peritoneale. Al contrario, la fase di sbiancamento con 1,5 mg/mL di boroidruro di litio per 60 minuti è stata più rigorosa. Questi parametri sono stati determinati empiricamente e possono variare a seconda dei tessuti e degli anticorpi utilizzati. Da notare che a causa dell'incubazione nella soluzione sbiancante reattiva, piccole bolle d'aria possono rimanere intrappolate nel tessuto. Ogni volta che ciò è diventato evidente durante la fase di imaging della procedura, i campioni sono stati degassati esponendo la piattaforma nella piastra di incubazione a 9 pozzetti per sottovuoto utilizzando un contenitore di aspirazione. Non è stato possibile eseguire semplici registrazioni di campioni ITK, spesso applicate negli studi IBEX, a causa dell'eccessivo inserimento di dati nel software di analisi delle immagini. Tuttavia, le popolazioni cellulari identiche possono essere facilmente identificate confrontando i singoli set di dati. Nel complesso, i cicli iterativi sono stati cronometrati in modo tale che un ciclo potesse essere completato in un giorno, con l'imaging che occupava la maggior parte di un intero ciclo (~ 8 ore).
La tecnica ha dei limiti che meritano di essere menzionati. Il requisito temporale del protocollo rende improbabile l'adozione di questo metodo su larga scala, ma piuttosto in circostanze selezionate. Immaginiamo che questo protocollo sia utilizzato al meglio per caratterizzare i cambiamenti nel microambiente immunitario tumorale prima e dopo il trattamento (ad esempio, anticorpi bispecifici per l'immunoterapia del cancro) o per prevedere i risultati generali del trattamento. Sebbene questo protocollo offra una caratterizzazione 3D, non è noto quanta area debba essere interrogata per descrivere efficacemente un tumore, comprese le sue complesse popolazioni cellulari28. L'accesso a tessuti umani freschi può anche essere difficile in alcuni centri, ma prevediamo che questo protocollo sia applicabile anche ai modelli animali29.
Le applicazioni di questo protocollo sono diverse, dalla ricerca di base che analizza la composizione del TME in più tumori solidi alle biopsie pre/post-tissutali per i pazienti arruolati in studi clinici. Sebbene i dati ottenibili da una singola sezione di tessuto spesso in uno strato cellulare siano immensi, la biologia 3D può essere facilmente trascurata. Vediamo l'imaging 3D come un'aggiunta ad altre tecniche di biologia spaziale, probabilmente aiutando nella selezione di sezioni 2D per le analisi successive. Inoltre, è in corso un protocollo per incorporare un componente di imaging dal vivo per più ore utilizzando il montaggio dei tessuti e la configurazione di imaging descritta.
Divulgazioni
Nessuno.
Riconoscimenti
Questa ricerca è stata supportata dal Programma di ricerca intramurale del National Institutes of Health e dal National Cancer Institute (NCI). Questa ricerca è stata anche parzialmente supportata dal CAT-I, una collaborazione di ricerca tra NIAID e NCI, guidata da Ronald Germain. Desideriamo esprimere la nostra gratitudine ad Andrea Radtke per il suo impegno entusiasta e collaborativo. La sua esperienza ha giovato notevolmente a questo studio.
Materiali
| Name | Company | Catalog Number | Comments |
| Alexa Fluor Plus 750 Phalloidin | Invitrogen | A30105 | F-actin dye |
| Alexa Fluor 488 anti-human CD206 (MMR) Antibody | BioLegend | 321114 | antibody |
| Alexa Fluor 488 anti-human CD31 Antibody | BioLegend | 303110 | antibody |
| Alexa Fluor 488 anti-human CD4 Antibody | BioLegend | 300519 | antibody |
| Alexa Fluor 488 anti-human HLA-DR Antibody | BioLegend | 307656 | antibody |
| Alexa Fluor 488 anti-mouse/human CD44 Antibody | BioLegend | 103016 | antibody |
| Alexa Fluor 594 anti-human CD45 Antibody | BioLegend | 304060 | antibody |
| Alexa Fluor 647 Anti-Calretinin antibody [EP1798] | Abcam | ab214244 | antibody |
| Alexa Fluor 647 anti-human CD3 Antibody | BioLegend | 300416 | antibody |
| Alexa Fluor 647 anti-human CD8 Antibody | BioLegend | 344726 | antibody |
| Alexa Fluor 647 anti-human Podoplanin Antibody | BioLegend | 337007 | antibody |
| Alexa Fluor 647 anti-Vimentin Antibody | BioLegend | 677807 | antibody |
| Alexa Fluor 647 CD68 Antibody (KP1) | Santa Cruz | sc-20060AF647 | antibody |
| Alexa Fluor 750 Cytokeratin, pan Antibody (AE-1/AE-3) | Novus | NBP2-33200AF750 | antibody |
| Alexa Fluor 750 Human alpha-Smooth Muscle Actin Antibody | R&D | IC1420S | antibody |
| Alexa Fluor 488 CD20 Monoclonal Antibody (L26) eBioscience | Thermo Fisher | 53-0202-82 | antibody |
| Antibiotic-Antimycotic | Gibco | 15240096 | supplement harvest medium |
| BioMed Clear Resin (Form 3) | FormLabs | RS-F2-BMCL-01 | resin used for platform + incubation plate |
| BSA | Sigma | A7906-500g | blocking solution component |
| Cast acrylic 1/32" thick | material used for cutting slider | ||
| CleanStation DT3 | Stratasys | DT3 | post processing for height adjuster & outer lid |
| Container, Specimen | McKesson | 870203 | transfer of tissue from perating table to prep table |
| CorelDraw | CorelDRAW | Software to prepare the vector-based design file for laser cutting | |
| Cytofix | BD Bioscience | 554655 | fixative |
| Dish 15 cm | Falcon | 353025 | dish used during mounting |
| Dish 35 mm No. 1.5 Coverslip 14 mm Glass Diameter Uncoated | Matek | P35G-1.5-14-C | imaging dish |
| DMEM (no glucose) | Gibco | 11966025 | harvest medium |
| Fc Block | BD Bioscience | 564220 | blocking solution component |
| Form 3B+ | Formlabs | Form 3B+ | printer used for platform + incubation plate |
| Form Cure | Formlabs | FH-CU-01 | post processing for platform + incubation plate |
| Form Wash | Formlabs | FH-WA-01 | post processing for platform + incubation plate |
| GrabCAD Print | GrabCAD | GrabCAD Print | Software to prepare models for Stratasys printers |
| Hoechst 33342 10 mg/mL | Biotium | 40046 | nuclear dye |
| J826 Prime 3D Printer | Stratasys | J826 | printer used for height adjuster & outer lid |
| LAS X | Leica | LAS X | Confocal software |
| Laser cutting system | Universal Laser Systems | ULS PLS6.150D | CO2 Laser cutting used for slider |
| Lithium Borohydride | STREM Chemicals | 93-0397 | bleaching chemical |
| PBS, pH 7.4 | Gibco | 10010023 | base Buffer for washing, blocking, staining |
| PreForm | FormLabs | PreForm | Software to prepare models for Formlab printers |
| Silk sutures 2-0 | Ethicon | A305.O35 | affix tissue to platform |
| Stellaris 8 WLL confocal microscope | Leica | STELLARIS 8 | Confocal Imaging |
| Syringe filter | filter ab solution | ||
| Triton X-100 | American Bio | AB02025-00100 | permeabelizing reagent, blocking solution component |
| Vero ContactClear | Stratasys | CTT610, 4 KG | resin used for height adjuster & outer lid |
| Warming Tray | Spring USA | ST-1220 | keep media and tissue warm |
| Water, distilled | Gibco | 15230-170 | diluent for LiBH4 |
Riferimenti
- Anderson, N. M., Simon, M. C. The tumor microenvironment. Curr Biol. 30 (16), R921-R925 (2020).
- Li, Z., Li, J., Bai, X., Huang, X., Wang, Q. Tumor microenvironment as a complex milieu driving cancer progression: A mini-review. Clin Transl Oncol. , (2024).
- Fridman, W. H., Zitvogel, L., Sautes-Fridman, C., Kroemer, G. The immune contexture in cancer prognosis and treatment. Nat Rev Clin Oncol. 14 (12), 717-734 (2017).
- Roma-Rodrigues, C., Mendes, R., Baptista, P. V., Fernandes, A. R. Targeting tumor microenvironment for cancer therapy. Int J Mol Sci. 20 (4), 840 (2019).
- Sheng, W., et al. Multiplex immunofluorescence: A powerful tool in cancer immunotherapy. Int J Mol Sci. 24 (4), 3086 (2023).
- Bollhagen, A., Bodenmiller, B. Highly multiplexed tissue imaging in precision oncology and translational cancer research. Cancer Discov. 14 (11), 2071-2088 (2024).
- Jin, Y., et al. Advances in spatial transcriptomics and its applications in cancer research. Mol Cancer. 23 (1), 129 (2024).
- Kashyap, A., et al. Quantification of tumor heterogeneity: From data acquisition to metric generation. Trends Biotechnol. 40 (6), 647-676 (2022).
- Chang, Q., Hedley, D. Emerging applications of flow cytometry in solid tumor biology. Methods. 57 (3), 359-367 (2012).
- Zhang, L., et al. Single-cell analyses inform mechanisms of myeloid-targeted therapies in colon cancer. Cell. 181 (2), 442-459.e29 (2020).
- Wang, X. Q., et al. Spatial predictors of immunotherapy response in triple-negative breast cancer. Nature. 621 (7980), 868-876 (2023).
- Maestri, E., et al. Spatial proximity of tumor-immune interactions predicts patient outcome in hepatocellular carcinoma. Hepatology. 79 (4), 768-779 (2024).
- Wang, Q., et al. Role of tumor microenvironment in cancer progression and therapeutic strategy. Cancer Med. 12 (10), 11149-11165 (2023).
- Veninga, V., Voest, E. E. Tumor organoids: Opportunities and challenges to guide precision medicine. Cancer Cell. 39 (9), 1190-1201 (2021).
- Lin, J. R., et al. Multiplexed 3d atlas of state transitions and immune interaction in colorectal cancer. Cell. 186 (2), 363-381.e19 (2023).
- Radtke, A. J., et al. Ibex: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Radtke, A. J., et al. Ibex: A versatile multiplex optical imaging approach for deep phenotyping and spatial analysis of cells in complex tissues. Proc Natl Acad Sci U S A. 117 (52), 33455-33465 (2020).
- Van Baal, J. O., et al. The histophysiology and pathophysiology of the peritoneum. Tissue Cell. 49 (1), 95-105 (2017).
- Bridda, A., Padoan, I., Mencarelli, R., Frego, M. Peritoneal mesothelioma: A review. MedGenMed. 9 (2), 32 (2007).
- Demuytere, J., Ernst, S., Van Ovost, J., Cosyns, S., Ceelen, W. The tumor immune microenvironment in peritoneal carcinomatosis. Int Rev Cell Mol Biol. 371, 63-95 (2022).
- Wang, Q., et al. Heterogeneity of tertiary lymphoid structures predicts the response to neoadjuvant therapy and immune microenvironment characteristics in triple-negative breast cancer. Br J Cancer. , (2024).
- Kepenekian, V., et al. Peritoneal mesothelioma: Systematic review of hyperthermic intraperitoneal chemotherapy (HIPEC) protocol outcomes. Indian J Surg Oncol. 14 (Suppl 1), 39-59 (2023).
- Mcquade, C., Renton, M., Chouhan, A., Macdermott, R., O'brien, C. Review of imaging peritoneal disease and treatment. Can Assoc Radiol J. , (2024).
- Kenerson, H. L., et al. Tumor slice culture as a biologic surrogate of human cancer. Ann Transl Med. 8 (4), 114 (2020).
- Arrizabalaga, L., et al. Tumor slice culture system for ex vivo immunotherapy studies. Methods Cell Biol. 189, 55-69 (2024).
- Neumeister, V. M., et al. Quantitative assessment of effect of preanalytic cold ischemic time on protein expression in breast cancer tissues. J Natl Cancer Inst. 104 (23), 1815-1824 (2012).
- Panchuk-Voloshina, N., et al. Alexa dyes: A series of new fluorescent dyes that yield exceptionally bright, photostable conjugates. J Histochem Cytochem. 47 (9), 1179-1188 (1999).
- Rajaram, S., et al. Sampling strategies to capture single-cell heterogeneity. Nat Methods. 14 (10), 967-970 (2017).
- Bella, A., et al. Mouse models of peritoneal carcinomatosis to develop clinical applications. Cancers (Basel). 13 (5), 963 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon