Method Article
Dreidimensionale Bildgebung von tumortragendem Gewebe mit dem iterativen Bleaching erweitert den Multiplexitätsansatz
In diesem Artikel
Zusammenfassung
Dieser Artikel beschreibt ein Protokoll für Multiplex-Immunfluoreszenz, das optimiert wurde, um die dreidimensionale Architektur von Peritonealmetastasen zu charakterisieren.
Zusammenfassung
Die räumliche Heterogenität der Tumormikroumgebung (TME) ist eine entscheidende Determinante für das therapeutische Ansprechen, insbesondere bei immunonkologischen Wirkstoffen, bei denen der Erfolg von der Verteilung spezifischer Immunzell-Subpopulationen abhängt. In den letzten zehn Jahren wurden mehrere ausgefeilte Technologien eingeführt, um eine detaillierte Auflösung der TME unter Verwendung von zweidimensionalen Schnitten von formalinfixiertem, paraffineingebettetem (FFPE) oder fixiertem gefrorenem Gewebe zu erreichen. Diese Dünnschliffe sind zwar einfacher zu beschaffen und zu analysieren, aber es fehlt ihnen die dreidimensionale Architektur, die für eine zuverlässige und umfassende Charakterisierung eines Tumors erforderlich ist. Um diese Einschränkung zu beheben, wurde ein Gewebemontage- und Bildgebungsverfahren entwickelt, das die dreidimensionale Analyse von Tumorläsionen in ihrem nativen in vivo-Zustand ermöglicht. Dieses Protokoll beschreibt die Beschaffung von menschlichem Tumorgewebe, die Befestigung von Proben auf individuell bedruckten Plattformen und die Färbeverfahren für Proben nach der Fixierung. Die Multiplex-Immunfluoreszenztechnik IBEX (Iterative Bleaching Extends Multiplexity) wurde angepasst, um die dreidimensionale TME mit bis zu 15 Markern für Tumor-, Immun- und Stromazellen unter Verwendung kommerziell erhältlicher Antikörper zu charakterisieren. Bildgebungstiefen von bis zu 100 μm wurden mit einem konfokalen Invers-Weißlicht-Lasermikroskop mit einem speziell bedruckten Bildgebungsadapter und handelsüblichen Glasbodenschalen erreicht, um eine optimale Gewebeorientierung zu gewährleisten. Dieses Protokoll unterstreicht das Potenzial der IBEX-Methode, Multiplex-Immunfluoreszenzstudien zu erweitern und ein umfassenderes Verständnis der TME-Zusammensetzung zu ermöglichen.
Einleitung
Solide Tumoren sind sehr variabel und weisen eine komplizierte Zusammensetzung von Zelltypen (maligne und nicht-maligne Zelltypen), extrazelluläre Matrixproteine und lösliche Faktorenauf 1,2. Letztendlich bestimmt diese hochkomplexe, heterogene Tumormikroumgebung (TME) die Anfälligkeit für Behandlungen wie Immuntherapien 3,4. Daher wurden Technologien wie die hochmultiplexierte Immunfluoreszenz 5,6 und die räumliche Transkriptomik7 eingesetzt, um die Komplexität der TME in zellschichtdicken (~4 μM) Schnitten zu charakterisieren.
Im Gegensatz zu Zelldispersionsansätzen wie Einzelzellsequenzierung und Durchflusszytometrie 8,9,10 bewahren diese Techniken räumliche Beziehungen, um wichtige Informationen über die zelluläre Wechselwirkung zu liefern, und können zur Identifizierung potenzieller Biomarker für die Vorhersage des Ansprechens auf die Behandlung eingesetzt werden 11,12,13. Tumorläsionen sind jedoch von Natur aus dreidimensionale (3D) Strukturen, und 2D-Ansätze wie die oben genannten erfassen die komplexe zelluläre Landschaft nicht ausreichend. Die Bedeutung der 3D-Biologie wurde in diesem Bereich erkannt, wie die breite Verwendung von patientengewonnenen Tumororganoidkulturen zeigt14. Organoide behalten jedoch nicht die zelluläre Komplexität der ursprünglichen in vivo TME bei, was die Anwendbarkeit für die Verwendung mit immunmodulatorischen Arzneimitteln oder für die Charakterisierung der verschiedenen TME-Zellpopulationen einschränkt.
Obwohl es möglich sein kann, Analysen an seriellen 2D-Schnitten durchzuführen und die Bilder für ein 3D-Rendering rechnerisch zusammenzufügen, ist diese Technik kostspielig und mit hoher Genauigkeit schwer zu bewerkstelligen15. Um 3D-Gewebe (z.B. Tumore) kostengünstig reproduzierbar zu charakterisieren, wurde eine einzigartige Methode zur Präparation und Multiplex-Immunfluoreszenzfärbung/-analyse entwickelt und anhand von Peritonealtumorläsionen eines Patienten mit primärem Peritonealkarzinom demonstriert.
Basierend auf einer Adaption der etablierten IBEX-Technik (Iterative Bleaching Extends Multiplexity)16,17 beschreibt dieses Protokoll den 3D-Druck eines Gewebehalters und eines Bildgebungsadapters, die Entnahme und Montage von peritonealem Gewebe, mehrere Färbezyklen mit verschiedenen Tumor- und Zelltypmarkern und hochauflösende konfokale 3D-Bildgebung.
Protokoll
Die Studie wurde mit Genehmigung des National Institutes of Health Institutional Review Board gemäß Protokoll NCT01915225 durchgeführt. Menschliches Gewebe wurde mit schriftlicher Einverständniserklärung zum Zeitpunkt der diagnostischen Laparoskopie entnommen. Alle Gewebe waren tumortragend, was durch sichtbare Inspektion festgestellt und durch histopathologische abschließende Untersuchung bestätigt wurde. Einzelheiten zu allen verwendeten Reagenzien und Geräten sind in der Materialtabelle aufgeführt. Eine schematische Darstellung des gesamten Arbeitsablaufs ist in Abbildung 1 dargestellt.
1. Druckplattformen, Bildgebungsadapter und Inkubationsplatte
- Schneiden/3D-Druck/Nachbearbeitungsverfahren für die Plattform und 9-Well-Plattenteile (dargestellt in Abbildung 2A-C)
- Slicing: Öffnen Sie die Slicing-Software und importieren Sie die Designdateien in . STL-Format (Ergänzungsdatei 1 und Ergänzungsdatei 2). Richten Sie die Modelle für einen optimalen Druck aus und fügen Sie Stützen hinzu. Wählen Sie den medizinischen Drucker, das klare Harz und die Schichthöhe von 50 μm aus. Überprüfen Sie die Druckbarkeit und senden Sie den Auftrag an den Drucker.
- 3D-Druck: Überprüfen Sie, ob der Drucker bereit ist (richtige Harzpatrone, Tank und Plattform). Wählen Sie auf dem Touchscreen des Druckers den hochgeladenen Auftrag aus der Slicer-Software aus, um den Druck zu starten. Sobald der Druck abgeschlossen ist, verwenden Sie das Entfernungswerkzeug, um das gedruckte Teil von der Plattform zu lösen.
- Nachbearbeitung: Spülen Sie das Teil in der mit 99%igem Isopropylalkohol (IPA) gefüllten Wascheinheit für 15 min. Teile aus der Wascheinheit nehmen und 5 min in frischem IPA einweichen.
- Härten Sie das Teil 60 Minuten lang bei 60 °C nach. Entfernen Sie die Stützen mit bündigen Fräsern. Schleifen Sie ggf. Stützpunkte mit feinkörnigem Schleifpapier ab.
- Schneiden/3D-Druck/Nachbearbeitungsverfahren für Höhenversteller und Außendeckel (dargestellt in Abbildung 2A-C)
- Slicing: Öffnen Sie die Drucksoftware und importieren Sie die Designdateien in . STL-Format (Zusatzdatei 3 und Zusatzdatei 4). Richten Sie die Modelle aus, und weisen Sie Materialien zu. Wählen Sie den gewünschten Druckmodus (Hohe Mischung, hohe Geschwindigkeit) und stellen Sie die Modelleinstellungen auf Matt ein. Überprüfen Sie die Druckbarkeit und senden Sie den Auftrag an den Drucker.
- 3D-Druck: Stellen Sie sicher, dass der Drucker die richtigen Materialien geladen hat und das Baufach sauber ist. Wählen Sie auf der Monitoroberfläche des Druckers den hochgeladenen Auftrag aus, um den Druck zu starten. Sobald der Druck abgeschlossen ist, verwenden Sie das Schaberwerkzeug, um das gedruckte Teil vorsichtig aus dem Baufach zu entfernen.
- Nachbearbeitung: Legen Sie die gedruckten Teile in einen Wasserstrahlreiniger oder ein Rührreinigungssystem, um das Stützmaterial zu entfernen. Führen Sie bei Bedarf ein manuelles Entfernen der Stützen mit Werkzeugen wie einer Pinzette oder Zange durch.
- Laserschneiden eines Plattformschiebers (siehe Abbildung 2A,C)
- Vorbereiten des Designs: Importieren Sie die *. DWG-Designdatei in der Vektorsoftware (Ergänzungsdatei 5). Stellen Sie die Linienfarben für das Vektorschneiden auf Rot (RGB) ein. Senden Sie den Auftrag an den Lasercutter.
- Einrichten des Lasercutters: Öffnen Sie die Lasercutter-Software und importieren Sie die Designdatei. Wählen Sie den Materialtyp aus und passen Sie die Einstellungen für Leistung, Geschwindigkeit und Auflösung für 1/32" dickes Acryl an. Platzieren Sie das Material auf dem Laserbett, stellen Sie sicher, dass es richtig ausgerichtet ist, und fokussieren Sie den Laser.
- Laserschneiden: Starten Sie den Laserschneidprozess und überwachen Sie, um sicherzustellen, dass das Material korrekt geschnitten wird.
2. Tissue-Montage auf gedruckten Plattformen
- Vorbereitung des Erntemediums und der Zubereitungstabelle
- Ergänzen Sie 100 mL DMEM (niedriger Glukosespiegel) mit Anti-Anti und stellen Sie ihn bis zur Verwendung in einen 37 °C heißen Inkubator (Erntemedium).
- Richten Sie im Operationssaal einen "Vorbereitungstisch" mit folgenden Gegenständen ein: autoklavierte 3D-gedruckte Plattformen, Packung mit 2-0 Seidenfäden, Heizplatte, Probenbecher mit 50 mL vorgewärmtem Erntemedium, 15 cm Kulturplatte mit 30 mL vorgewärmtem Erntemedium und eine 24-Well-Platte mit 4 Wells mit je 1,5 mL Erntemedium (alles auf Heizplatte legen), 1 Paar Pinzetten und 1 Paar chirurgische Scheren.
- Geweberesektion und -befestigung
HINWEIS: Das Gewebe wird aus dem Operationssaal entnommen. Bereiche des Peritoneums mit Tumorherden von weniger als 0,5 mm Dicke und Durchmesser werden bei der visuellen Inspektion identifiziert (Abbildung 2D). Bemerkenswert ist, dass ~ 2 cm2 Gewebe (Peritoneum) benötigt werden, um eine Plattform vorzubereiten.- Trennen Sie das tumortragende Bauchfell vorsichtig von der darunter liegenden Bauchwandmuskulatur und Faszie (durchgeführt vom Chirurgen). Füllen Sie die resultierenden Gewebeblätter in einen Probenbecher mit warmem Erntemedium und legen Sie sie auf eine Heizplatte (Vorbereitungstisch).
- Montieren Sie das Gewebe auf Plattformen in der 15 cm dicken Platte, die das Erntemedium enthält. Legen Sie das tumortragende Gewebe (mesotheliale Seite nach oben) vorsichtig über die kleine Öffnungsseite der Plattform und fixieren Sie es mit einer 2-0 Seidennaht. Legen Sie die vorbereitete Gewebeplattform invers (getaucht) in eine 24-Well-Platte mit Erntemedium (Abbildung 2E-G).
HINWEIS: Montieren Sie das Gewebe so gelehrt, dass Falten auf der Plattformoberfläche vermieden werden, aber erzeugen Sie keine Spannung, da dies zu unerwünschten biomechanischen und physiologischen Veränderungen führt.
3. Fixierungs-/Färbe-/Bleichverfahren
HINWEIS: Änderungen gegenüber dem ursprünglichen Protokoll, die auf 4 μm dünne FFPE- oder fixierte Fixierschnitte zugeschnitten sind, sind in Abbildung 3 dargestellt. Tabelle 1 enthält eine Liste der Antikörper (mit Verdünnungen), die für die IBEX-Runden 1-6 verwendet wurden.
- Fixierung
- Bereiten Sie 40 mL Fixationspuffer vor, indem Sie 10 mL Fixiermittel in ein konisches 50-ml-Röhrchen mit 30 mL kaltem 1x PBS geben.
- Das auf Plattformen montierte Gewebe mit einer Pinzette in den Fixierpuffer überführen und 24 Stunden bei 4 °C inkubieren.
- Dekantieren Sie das Fixiermittel und ersetzen Sie es durch 40 mL kaltes 1x PBS und inkubieren Sie es für 10 min bei 4 °C.
- Färbung
- Geben Sie 1 ml Blockierungspuffer (1 % BSA, 0,3 % Triton-X-100, 1 % Fc-Block in 1x PBS, gefiltert mit 0,22 μm Spritzenfilter) in jede Vertiefung und setzen Sie Gewebeplattformen ein. Mindestens 2 h bei Raumtemperatur auf einer Wippe mit niedriger Drehzahl (30 U/min) blockieren.
HINWEIS: Stellen Sie sicher, dass die Kerbe auf der Plattform und die Nut in der Vertiefung ausgerichtet sind. Dieser Schritt ist nicht zeitkritisch und kann über Nacht bei 4 °C durchgeführt werden. - Bereiten Sie 0,8 ml Antikörper-/Farbstofflösung in einem Mikrozentrifugenröhrchen vor und zentrifugieren Sie sie 5 Minuten lang bei Raumtemperatur bei 10.000 x g . Übertragen Sie 0,75 mL in eine saubere Vertiefung der 9-Well-Inkubationsplatte und setzen Sie eine gewaschene Plattform ein.
- Wickeln Sie die Platte in Alufolie ein und inkubieren Sie sie mindestens 2 h bei Raumtemperatur auf einer Wippe, die auf niedrige Geschwindigkeit (30 U/min) eingestellt ist.
- Waschen Sie die Plattform in einem konischen 50-ml-Röhrchen mit 40 mL 1x PBS für 10 min bei 4 °C. Die Plattform kann nun in den Bildgebungsadapter eingesetzt werden.
- Geben Sie 1 ml Blockierungspuffer (1 % BSA, 0,3 % Triton-X-100, 1 % Fc-Block in 1x PBS, gefiltert mit 0,22 μm Spritzenfilter) in jede Vertiefung und setzen Sie Gewebeplattformen ein. Mindestens 2 h bei Raumtemperatur auf einer Wippe mit niedriger Drehzahl (30 U/min) blockieren.
- Bleiche
- Nach jeder Färbe- und Bildgebungsrunde werden die Fluorophore mit dem Esterreduktionsmittel Lithiumborhydrid (LiBH4) gebleicht. Diese Behandlung eliminiert die Fluoreszenzsignale von Alexa Fluor (AF)488, AF647 und AF750, nicht jedoch von AF594 und dem Kernfarbstoff Hoechst.
ACHTUNG: Lithiumborhydrid ist extrem reaktiv mit Wasser. Bei Kontakt mit Wasser werden brennbare Wasserstoffgase freigesetzt; Verbrauchtes Lithiumborhydrid und mit Lithiumborhydrid verunreinigte Einwegartikel müssen als Sondermüll entsorgt werden. - 5 ml 1,5 mg/ml LiBH4-Lösung in destilliertem Wasser (in einer chemischen Haube) zubereiten und 30 Minuten bei Raumtemperatur inkubieren.
HINWEIS: Die LiBH4-Lösung verliert ihre reduzierende/bleichende Wirkung, wenn sie >4 h vor der Verwendung zubereitet wird. - Übertragen Sie 1 ml LiBH4-Lösung in eine saubere Vertiefung der 9-Well-Inkubationsplatte und setzen Sie die gewaschene Plattform 60 Minuten lang bei Raumtemperatur ein.
- Waschen Sie die Plattform kurz in 20 mL 1x PBS in einem konischen Röhrchen und prüfen Sie, ob das verbleibende Fluoreszenzsignal mit der höchsten Lasereinstellung für die Z-Kompensation in der vorangegangenen Bildgebungsrunde vorhanden ist, bevor Sie mit den nachfolgenden Färberunden fortfahren.
- Wiederholen Sie die Blockierung der Probe für 2 Stunden bei Raumtemperatur, bevor Sie mit dem nächsten Antikörper-Inkubationsschritt fortfahren.
- Nach jeder Färbe- und Bildgebungsrunde werden die Fluorophore mit dem Esterreduktionsmittel Lithiumborhydrid (LiBH4) gebleicht. Diese Behandlung eliminiert die Fluoreszenzsignale von Alexa Fluor (AF)488, AF647 und AF750, nicht jedoch von AF594 und dem Kernfarbstoff Hoechst.
4. Bildgebendes Verfahren
- Montage von bildgebenden Adaptern/Plattformen
- 0,1 mL 1x PBS in die Mitte des Glasbodens einer 3,5 cm großen Bildgebungsschale geben und beiseite stellen. Schrauben Sie den Höhenverstellring locker im Uhrzeigersinn in den Außendeckel und montieren Sie die Plattform in die innere Kunststoffhalterung (Kerb-Nut-Ausrichtung). Sichern Sie die Plattform mit dem Schieber.
- Setzen Sie den zusammengebauten Bildgebungsadapter auf die Glasbodenschale und senken Sie den Höhenverstellring vorsichtig ab, bis das Gewebe den Puffer berührt. Vermeiden Sie es, die Plattform auf dem Glasboden zu schwenken.
- Sobald der optimale Abstand zum Glas erreicht ist, geben Sie 0,2 ml 1x PBS auf das Gewebe (jetzt mit der Unterseite nach oben), um eine Dehydrierung des Gewebes während der Bildgebung zu vermeiden.
- Konfokale Bildgebung
- Schalten Sie alle Komponenten des inversen konfokalen Mikroskops ein, einschließlich des Computers, des Mikroskops, des Lasers und der LED-Lichtquelle für die Verwendung des Okulars. Starten Sie die Bildaufnahmesoftware .
- Wählen Sie das passende Objektiv (z. B. 20x oder 40x) und fügen Sie das entsprechende Immersionsmedium hinzu. Zum Beispiel wird in dieser Studie ein 20-faches Wasserobjektiv verwendet; Dem Objektiv werden ~200 μl Wasser zugesetzt, um ein Austrocknen bei langen Bildaufnahmen zu vermeiden.
- Setzen Sie die zusammengebaute Bildgebungsschale in den Probenhalter auf dem Mikroskoptisch ein und stellen Sie sicher, dass die Probe sicher ist (sich nicht bewegt).
HINWEIS: Wenn Sie die zusammengesetzte Probe auf den Mikroskoptisch legen, beachten Sie die Kerbposition und verwenden Sie dieselbe Position für iterative Zyklen, um die Bildausrichtung zu erleichtern. Es ist wichtig, den Bildgebungsadapter für aufeinanderfolgende Runden auf der optimalen Höheneinstellung zu halten. - Zentrieren Sie die Probe mit der X-Y-Steuerung und bringen Sie das Objektiv an das Deckglas. Wenn Sie durch das Okular schauen und das Hoechst-Signal als Referenz verwenden, bewegen Sie das Objektiv, um eine Feinfokussierung auf das Gewebe zu erhalten.
- Wählen Sie geeignete Laserlinien aus oder passen Sie die WLL an die Anregungs- und Emissionsspektren der Fluorophore an, die zur Färbung der Gewebeprobe verwendet werden. Beseitigen Sie Überlappungen zwischen den Kanälen, indem Sie die Einstellungen anpassen und Sequenzen hinzufügen, wenn Fluorophore ähnliche Anregungsspektren aufweisen.
- Wählen Sie die folgenden Bildaufnahmeparameter aus: Scangeschwindigkeit 600 Hz, XY-Auflösung 1024 x 1024, 3-Linien-Mittelwerte und bidirektionaler Scan.
- Wählen Sie die folgenden Z-Stack-Parameter aus: (1) Verwenden Sie eine Z-Schrittgröße, die für das verwendete Ziel optimiert ist, (2) definieren Sie den Anfang und das Ende des Stacks, (3) passen Sie die Z-Kompensationsparameter an, um eine gleichmäßige Helligkeit im gesamten Stack zu erreichen, und (4) erfassen Sie einen 1-Kachel-Teststack mit einem Mittelwert.
- Definieren Sie den Bereich, der abgebildet werden soll, und erstellen Sie einen Kachel-Z-Stapel mit hoher Auflösung.
Ergebnisse
Das Peritoneum ist eine membranöse Struktur von weniger als 1 mm Dicke, die die Oberfläche der Bauchdecke auskleidet und an die Oberflächen der Bauchorgane angrenzt. Es besteht aus oberflächlichem Mesothel und darunter liegendem Bindegewebe, das mit Fettzellen, Lymphozyten, Makrophagen und Fibroblasten sowie Blut- und Lymphgefäßen durchsetzt ist. Peritoneale Malignome gehen meist auf metastasen von abdominalen Tumoren zurück (z. B. Eierstockkrebs oder Magenkrebs)18 oder seltener können Primärtumoren des Peritoneums (z. B. Mesotheliom) sein19. Diese oft kleinen (<1 cm) Tumorläsionen sind hochkomplex, können aber mit der beschriebenen Technik in 3D charakterisiert werden. Eine maximale Projektion von fünf konfokalen Schnitten wird verwendet, um die gesamte Probe auf einer Plattform zu betrachten (Abbildung 4, Einschub in der oberen linken Ecke). Drei primäre konjugierte Antikörper (AF488-CD44, AF594-CD45 und AF647-Podoplanin) wurden zusammen mit dem nukleären Farbstoff Hoechst für die erste Runde von IBEX verwendet (IBEX1, Abbildung 4). Tumorläsionen werden durch die rosettenartigen Strukturen deutlich, die für CD44 und Podoplanin doppelt positiv sind und eine für das papilläre Mesotheliom charakteristische Anordnung von Kernen aufweisen.
Tumorzellen wurden anhand der Zellmorphologie und zweier Antikörper identifiziert, da sowohl Anti-Podoplanin als auch Anti-CD44 Nicht-Tumorzellen erkennen, wie der Nachweis von Lymphgefäßen über Anti-Podoplanin und die Bindung des CD44-Antikörpers an Untergruppen von Immunzellen zeigt. Kleinere Regions of Interest (ROIs) werden dann mit einem Kachelscanformat in höherer Auflösung und optimierten Z-Schrittgrößen für das Rendern von 3D-Projektionen erfasst (Abbildung 5A). Die Plattformen wurden durch die iterativen Färbeverfahren geführt, und die gleichen ROIs wurden in den IBEX-Zyklen 1-6 abgebildet. Repräsentative Bilder für Tumor (gelber Kasten) und Tumor-tertiäre lymphoide Struktur (TLS) Grenzflächen (blauer Kasten) sind in Abbildung 5B-M zusammengestellt.
Das Vorhandensein von TLS und die Dichte von CD3-positiven Zellen deuten auf eine signifikante Infiltration von Immunzellen20 und möglicherweise auf ein Ansprechen auf immunmodulatorische Arzneimittelhin 21. Bemerkenswert ist, dass TLS und der Großteil der Tumorläsionen in unterschiedlichen Z-Tiefen des Gewebes vorhanden sind, wie der Vergleich von Stapelprojektionen verschiedener optischer Schnitte in Abbildung 6 zeigt, was die Vorteile der 3D-Bildgebung und die potenziell übersehene Biologie hervorhebt, wenn die 2D-Bildgebung auf 3D-Strukturen angewendet wird. Der 3D-Charakter dieser Läsionen wird in den Filmdateien (Film 1-12) deutlicher, die die Volumendarstellungen der einzelnen Läsionen in Abbildung 5 darstellen.
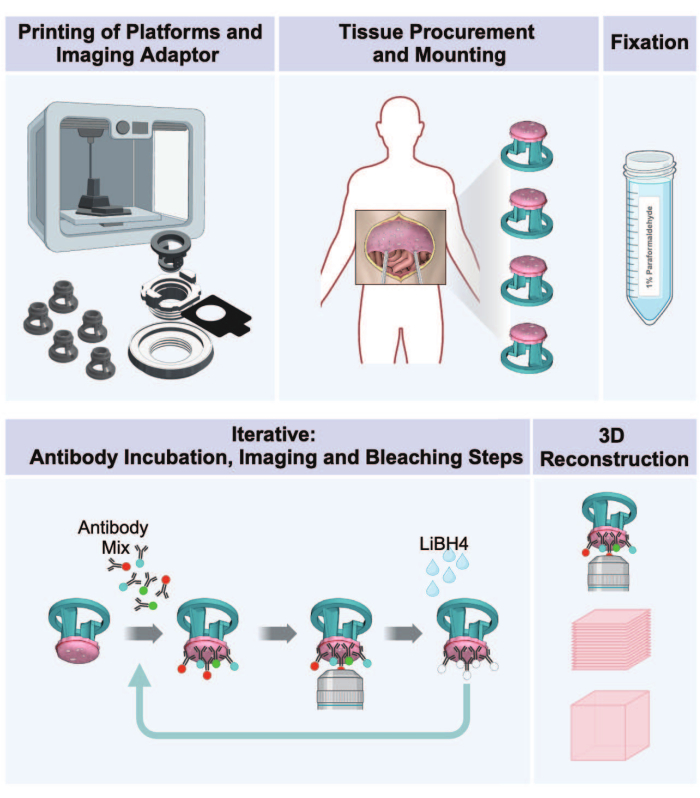
Abbildung 1: Arbeitsablauf vom 3D-Druck zur 3D-Bildgebung von Peritonealtumoren. Das Protokoll besteht aus einer Abfolge von fünf Schritten zur Erstellung von 3D-Multiplexbildern aus tumortragendem Peritonealgewebe. (1) Drucken von Gewebeaufnahmeplattformen, Teilen von Bildgebungsadaptern und Inkubationsplatte. (2) Verfahren zur Beschaffung und Montage von Gewebe. (3) Fixierung mit Paraformaldehyd. (4) IBEX-Färbe- und Bildgebungszyklen. (5) 3D-Rekonstruktion mit Bildgebungssoftware. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Hardware-Komponenten mit Gewebeschnittstelle für die 3D-Bildgebung. (A) Schematische Darstellung der Plattformanordnung innerhalb des Bildgebungsadapters. (B) Eine 9-Well-Inkubationsplatte. (C) Fotos von Bildadapterkomponenten mit Montage. (D) Mesotheliom-tragendes Peritoneum, wie es bei der diagnostischen Laparoskopie beobachtet wurde. (E) Montage des Gewebes auf der Plattform. (F,G) Tumortragendes Peritoneum, das auf Plattformen montiert ist. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
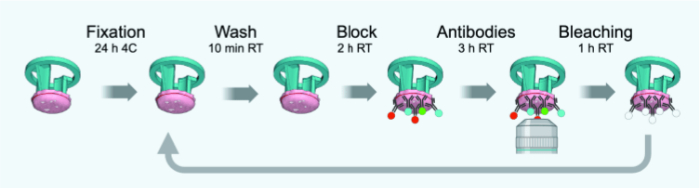
Abbildung 3: Zeitleiste der IBEX-Zyklen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
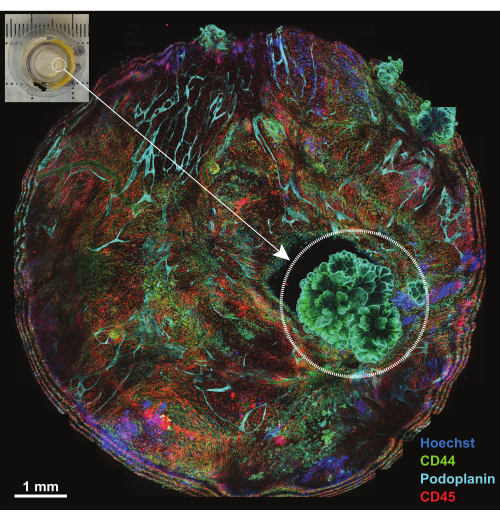
Abbildung 4: Überblick über die Plattform nach der Inkubation mit dem IBEX1-Panel. Maximale Projektion der gesamten Plattform nach der Inkubation mit IBEX1-Panel (Hoechst, AF488-CD44, AF594-CD45 und AF647-Podoplanin). Der gestrichelte Kreis markiert den Tumor. Einschub: Foto des tumortragenden Peritoneums, das auf der Bildgebungsplattform montiert ist. Maßstab: 1 mm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
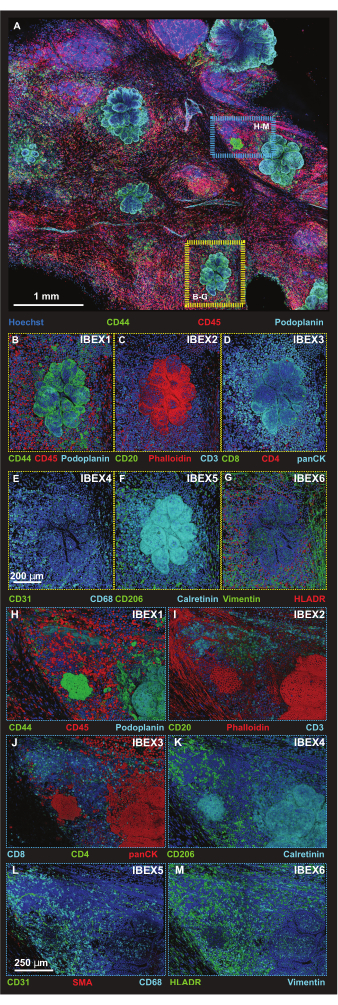
Abbildung 5: Hochauflösende Immunfluoreszenzbilder ausgewählter Plattformregionen. (A) Maximale Projektion des zusammengefügten 56-Kachel-Z-Stapel-Bildes, das nach der Inkubation mit dem IBEX1-Panel (Hoechst, AF488-CD44, AF594-CD45 und AF647-Podoplanin) aufgenommen wurde. Gelbes Rechteck: Tumorläsion in Panels (B-G). Blaues Rechteck: Schnittstelle zwischen Tumor und tertiärer lymphatischer Struktur, dargestellt in (H-M). (B-G) Eine einzelne Tumorläsion mit IBEX-Panel 1-6 im Volumen-Rendering. (B) IBEX1, grün: CD44, rot: CD45, cyan: Podoplanin. (C) IBEX2, grün: CD20, rot: F-Aktin, cyan: CD3. (D) IBEX3, grün: CD8, rot: CD4, cyan: panCK. (E) IBEX4, grün: CD31, cyan: CD68. (F) IBEX5, grün: CD206, cyan: Calretinin. (G) IBEX6, grün: Vimentin, rot: HLADR. (H-M) Eine individuelle Schnittstelle zwischen Tumor und tertiärer lymphatärer Struktur und IBEX-Panel 1-6 im Volumen-Rendering. (H) IBEX1, grün: CD44, rot: CD45, cyan: Podoplanin. (I) IBEX2, grün: CD20, rot: F-Aktin, cyan: CD3. (J) IBEX3, grün: CD4, rot: panCK, cyan: CD8. (K) IBEX4, grün: CD31, rot: SMA, cyan: CD68. (L) IBEX5, grün: CD206, cyan: Calretinin. (M) IBEX6, grün: HLADR, cyan: Vimentin. Alle Blenden, blau: Hoechst. Maßstabsleisten: (A, 1 mm); (B-M, 250 μm). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
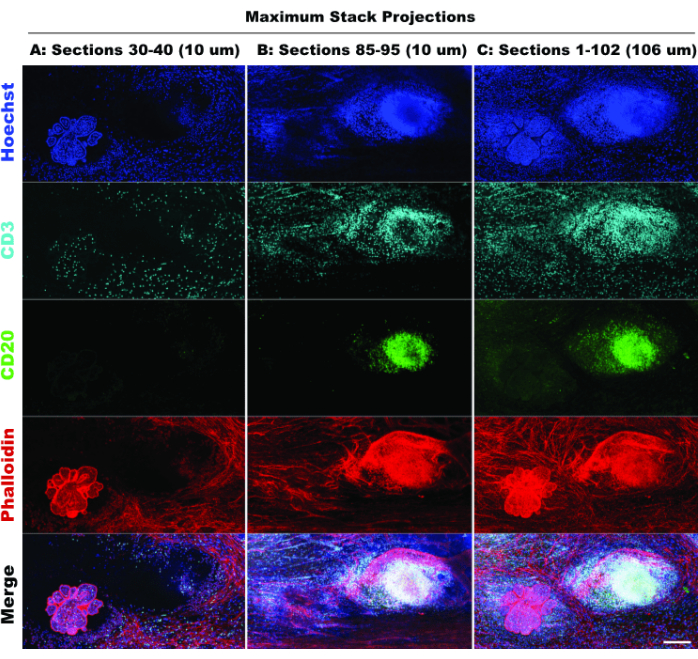
Abbildung 6: Simulierte 2D-Bildgebung. Der Vergleich von begrenzten (10 optische Schnitte) und Full-Stack-Projektionen (102 optische Schnitte) bestätigt den Verlust des Gewebekontexts in der 2D-Simulation. (A) Maximale Projektion der Abschnitte 30-40 (10 μm). (B) Maximale Projektion der Abschnitte 85-95 (10 μm). (C) Maximale Projektion der Abschnitte 1-102 (~100 μm). Alle Panels, IBEX2 (Hoechst, AF488-CD20, AF647-CD3, AF790-phalloidin). Maßstab: 200 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Tabelle 1: Antikörper, die für die einzelnen IBEX-Panels verwendet wurden. Bitte klicken Sie hier, um diese Tabelle herunterzuladen.
Film 1: Animiertes Volumen-Rendering der einzelnen Läsion mit IBEX-Panel 1 (blau: Hoechst, grün: CD44, rot: CD45, cyan: Podoplanin). Bitte klicken Sie hier, um diesen Film herunterzuladen.
Film 2: Animiertes Volumen-Rendering der einzelnen Läsion mit IBEX-Panel 2 (blau: Hoechst, grün: CD20, rot: F-Aktin, cyan: CD3). Bitte klicken Sie hier, um diesen Film herunterzuladen.
Film 3: Animiertes Volumen-Rendering der einzelnen Läsion mit IBEX-Panel 3 (blau: Hoechst, grün: CD8, rot: CD4, cyan: panCK). Bitte klicken Sie hier, um diesen Film herunterzuladen.
Film 4: Animiertes Volumen-Rendering der einzelnen Läsion mit IBEX-Panel 4 (blau: Hoechst, grün: CD31, cyan: CD68). Bitte klicken Sie hier, um diesen Film herunterzuladen.
Film 5: Animiertes Volumen-Rendering der einzelnen Läsion mit IBEX-Panel 5 (blau: Hoechst, grün: CD206, cyan: Calretinin). Bitte klicken Sie hier, um diesen Film herunterzuladen.
Film 6: Animiertes Volumen-Rendering der einzelnen Läsion mit IBEX-Panel 6 (blau: Hoechst, grün: Vimentin, rot: HLADR). Bitte klicken Sie hier, um diesen Film herunterzuladen.
Film 7: Animiertes Volumen-Rendering der tertiären lymphoiden Struktur-Läsions-Grenzfläche mit IBEX-Panel 1 (blau: Hoechst, grün: CD44, rot: CD45, cyan: Podoplanin). Bitte klicken Sie hier, um diesen Film herunterzuladen.
Film 8: Animiertes Volumen-Rendering der tertiären lymphoiden Struktur-Läsionen-Grenzfläche mit IBEX-Panel 2 (blau: Hoechst, grün: CD20, rot: F-Aktin, cyan: CD3). Bitte klicken Sie hier, um diesen Film herunterzuladen.
Film 9: Animiertes Volumen-Rendering der tertiären lymphoiden Struktur-Läsions-Grenzfläche mit IBEX-Panel 3 (blau: Hoechst, grün: CD8, rot: CD4, cyan: panCK). Bitte klicken Sie hier, um diesen Film herunterzuladen.
Film 10: Animiertes Volumen-Rendering der tertiären lymphoiden Struktur-Läsions-Grenzfläche mit IBEX-Panel 4 (blau: Hoechst, grün: CD31, rot: SMA, cyan: CD68). Bitte klicken Sie hier, um diesen Film herunterzuladen.
Film 11: Animiertes Volumen-Rendering der tertiären lymphoiden Struktur-Läsions-Grenzfläche mit IBEX-Panel 5 (blau: Hoechst, grün: CD206, cyan: Calretinin). Bitte klicken Sie hier, um diesen Film herunterzuladen.
Film 12: Animiertes Volumen-Rendering der tertiären lymphoiden Struktur-Läsions-Grenzfläche mit IBEX-Panel 6 (blau: Hoechst, grün: Vimentin, rot: HLADR). Bitte klicken Sie hier, um diesen Film herunterzuladen.
Ergänzungsdatei 1: Druckdatei für die Inkubationsplatte. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzungsdatei 2: Druckdatei für die 36 einzelnen Plattformen. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 3: Druckdatei für den Höhenverstellring. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzungsdatei 4: Druckdatei für den Außendeckel. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 5: Lasergeschnittene Datei für den Schieberegler. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Aktuelle Multiplex-Techniken für die Fluoreszenzbildgebung sind auf Dünnschliffe beschränkt und bieten keinen 3D-Kontext. Die vorliegende Studie beschreibt ein Protokoll zur Anwendung des IBEX-Bildgebungsverfahrens16,17 auf intakte Tumore, die auf speziell entwickelten Plattformen montiert sind. Peritoneale Tumorläsionen wurden ausgewählt, um die Technik hervorzuheben, da Patienten häufig mehrere, ideal große Tumorablagerungen aufweisen, die zusammen mit dem umgebenden normalen Peritoneum als Teil der Standard-Zytoreduktionsoperation entnommen werden, die in den meisten tertiären Überweisungszentren auf der ganzen Welt durchgeführt wird22,23. Nichtsdestotrotz ist das Protokoll sowohl auf parenchymale Metastasen als auch auf Mausmodelltumoren anwendbar, wobei Tumorschnitte verwendet werden, wie sie zuvor beschrieben wurden24,25. Darüber hinaus kann der Durchmesser des Montagerings auf den Plattformen an die Bedürfnisse von Experimenten/Geweben angepasst werden, obwohl größere Bereiche anfällig für Gewebeerschlaffung und verlängerte Bildgebungszeiten sind. Unabhängig von der Gewebequelle und -größe muss die Zeit zwischen Beschaffung und Konservierung so gering wie möglich gehalten werden, um eine optimale Qualität zu gewährleisten26. Die Aufstellung eines Vorbereitungstisches im Operationssaal garantiert eine zeitnahe Bearbeitung, wenn Patienten beteiligt sind. Die Zeit von der Beschaffung bis zur Fixierung des Gewebes sollte notiert werden, um die Vergleichbarkeit zwischen den Versuchen zu gewährleisten.
Die Gewebeproben werden durch das IBEX-Protokoll entnommen, da ganze Halterungen, d. h. Fixations- und Bildgebungs- und Bleichschritte durchgeführt werden, wobei das intakte Gewebe auf der Plattform befestigt wird. Dies garantiert die Konservierung der Probe durch die iterativen Färbe-/Bildgebungs-/Bleichzyklen, da es keine semi-permanente Schnittglas-Schnittstelle gibt und kein wiederholtes Platzieren/Entfernen eines Deckglases vom Gewebe erforderlich ist, was beides zu Gewebeverlust führen kann. Um Reagenzien zu sparen, garantiert die speziell entwickelte 9-Well-Platte einen optimalen Sitz der Plattform mit minimalem "Totvolumen". Alle Antikörper, die für die Färbung verwendet werden, sind primäre konjugierte Antikörper, und alle verwendeten Fluorophor-Konjugate (mit Ausnahme des Hoechst-Farbstoffs) sind Alexa-Fluor-Farbstoffe, die aufgrund ihrer hervorragenden Helligkeitausgewählt wurden 27. Andere Fluorophor-Antikörper-Panels wurden auf der Grundlage von Daten aus der IBEX-Bildgebungsgemeinschaft entwickelt, einem offenen, globalen Repositorium, das Datensätze, Protokolle und Rückmeldungen einer internationalen Gruppe von Wissenschaftlern sammelt, die IBEX (https://ibeximagingcommunity.github.io/ibex_imaging_knowledge_base/) verwenden. Die Sequenz der Antikörper-Panels wurde sorgfältig ausgewählt. Im Allgemeinen wurden Ziele mit geringer Abundanz in früheren Zyklen platziert. Der IBEX-Zyklus 1 (CD45, CD44 und Podoplanin) stellt eine Ausnahme dar, da diese drei Marker zur Identifizierung von Tumorläsionen in der ersten Färberunde verwendet wurden.
Antikörper-Panels sind vergleichsweise klein, um das Kanalübersprechen bei hohen Laserintensitätseinstellungen zu reduzieren, was notwendig ist, um das Gewebe mit einem konfokalen Mikroskop tiefer abzubilden. Wo immer möglich, wurden AF750-Konjugate eingeschlossen, um das Panel von 2 auf 3 Antikörper zu erweitern. Bemerkenswert ist, dass es nicht viele AF750-Konjugate gibt, die im Handel erhältlich sind, und die Tatsache, dass das Ziel für diesen Kanal sehr häufig vorhanden sein sollte, schränkt die Verwendung dieses Fluorophors ein. Ein Beispiel ist der AF750-konjugierte Anti-SMA-Antikörper, der in diesem Protokoll verwendet wurde. Obwohl es sich um einen Klon handelt, der zuverlässig an AF488 oder AF594 konjugiert arbeitet, ist die Signalintensität für diesen Antikörper, der mit AF750 konjugiert ist, deutlich geringer, was es schwierig macht, qualitativ hochwertige Z-Stacks zu erzeugen.
Antikörperinkubationen von 3 h bei Raumtemperatur sind im Vergleich zum ursprünglichen Protokoll kurz und spiegeln höchstwahrscheinlich die geringere Zelldichte in peritonealen Gewebeproben wider. Im Gegensatz dazu war der Bleichschritt mit 1,5 mg/mL Lithiumborhydrid für 60 min strenger. Diese Parameter wurden empirisch ermittelt und können je nach verwendetem Gewebe und Antikörper variieren. Zu beachten ist, dass durch die Inkubation in der reaktiven Bleichlösung kleine Luftbläschen im Gewebe eingeschlossen werden können. Wann immer sich dies während des bildgebenden Schritts des Verfahrens zeigte, wurden die Proben entgast, indem die Plattform in der 9-Well-Inkubationsplatte mit einem Saugbehälter einem Vakuum ausgesetzt wurde. Einfache ITK-Probenregistrierungen, die häufig in IBEX-Studien angewendet werden, konnten aufgrund der überwältigenden Dateneingabe in die Bildanalysesoftware nicht durchgeführt werden. Nichtsdestotrotz lassen sich identische Zellpopulationen durch den Vergleich einzelner Datensätze leicht identifizieren. Insgesamt wurden die iterativen Zyklen so getaktet, dass ein Zyklus an einem Tag abgeschlossen werden konnte, wobei die Bildgebung den größten Teil eines gesamten Zyklus (~ 8 h) in Anspruch nahm.
Die Technik hat Einschränkungen, die Erwähnung verdienen. Aufgrund des Zeitbedarfs des Protokolls ist es unwahrscheinlich, dass diese Methode auf breiter Basis, sondern eher unter bestimmten Umständen angewendet wird. Wir stellen uns vor, dass dieses Protokoll am besten zur Charakterisierung von Veränderungen in der Immunmikroumgebung des Tumors vor und nach der Behandlung (z. B. bispezifische Antikörper für die Krebsimmuntherapie) oder zur Vorhersage allgemeiner Behandlungsergebnisse eingesetzt wird. Obwohl dieses Protokoll eine 3D-Charakterisierung bietet, ist nicht bekannt, wie viel Fläche abgefragt werden muss, um einen Tumor einschließlich seiner komplexen Zellpopulationen effektiv zu beschreiben28. Der Zugang zu frischem menschlichem Gewebe kann in bestimmten Zentren ebenfalls schwierig sein, aber wir gehen davon aus, dass dieses Protokoll auch auf Tiermodelle anwendbar ist29.
Die Anwendungen für dieses Protokoll sind vielfältig und reichen von der Grundlagenforschung zur Analyse der TME-Zusammensetzung bei mehreren soliden Tumoren bis hin zu Prä-/Post-Gewebebiopsien für Patienten, die an klinischen Studien teilnehmen. Obwohl die Daten, die aus einem einzigen Zellschicht-dicken Gewebeschnitt gewonnen werden können, immens sind, kann die 3D-Biologie leicht übersehen werden. Wir sehen die 3D-Bildgebung als Ergänzung zu anderen Techniken der räumlichen Biologie, die wahrscheinlich bei der Auswahl von 2D-Schnitten für spätere Analysen hilfreich sind. Darüber hinaus wird ein Protokoll zur Integration einer Live-Bildgebungskomponente über mehrere Stunden unter Verwendung des beschriebenen Gewebemontage- und Bildgebungsaufbaus aktiv durchgeführt.
Offenlegungen
Nichts.
Danksagungen
Diese Forschung wurde durch das Intramural Research Program der National Institutes of Health und des National Cancer Institute (NCI) unterstützt. Diese Forschung wurde teilweise auch vom CAT-I unterstützt, einer Forschungskooperation zwischen NIAID und NCI unter der Leitung von Ronald Germain. Wir danken Andrea Radtke für ihren engagierten und kooperativen Einsatz. Ihre Expertise kam dieser Studie sehr zugute.
Materialien
| Name | Company | Catalog Number | Comments |
| Alexa Fluor Plus 750 Phalloidin | Invitrogen | A30105 | F-actin dye |
| Alexa Fluor 488 anti-human CD206 (MMR) Antibody | BioLegend | 321114 | antibody |
| Alexa Fluor 488 anti-human CD31 Antibody | BioLegend | 303110 | antibody |
| Alexa Fluor 488 anti-human CD4 Antibody | BioLegend | 300519 | antibody |
| Alexa Fluor 488 anti-human HLA-DR Antibody | BioLegend | 307656 | antibody |
| Alexa Fluor 488 anti-mouse/human CD44 Antibody | BioLegend | 103016 | antibody |
| Alexa Fluor 594 anti-human CD45 Antibody | BioLegend | 304060 | antibody |
| Alexa Fluor 647 Anti-Calretinin antibody [EP1798] | Abcam | ab214244 | antibody |
| Alexa Fluor 647 anti-human CD3 Antibody | BioLegend | 300416 | antibody |
| Alexa Fluor 647 anti-human CD8 Antibody | BioLegend | 344726 | antibody |
| Alexa Fluor 647 anti-human Podoplanin Antibody | BioLegend | 337007 | antibody |
| Alexa Fluor 647 anti-Vimentin Antibody | BioLegend | 677807 | antibody |
| Alexa Fluor 647 CD68 Antibody (KP1) | Santa Cruz | sc-20060AF647 | antibody |
| Alexa Fluor 750 Cytokeratin, pan Antibody (AE-1/AE-3) | Novus | NBP2-33200AF750 | antibody |
| Alexa Fluor 750 Human alpha-Smooth Muscle Actin Antibody | R&D | IC1420S | antibody |
| Alexa Fluor 488 CD20 Monoclonal Antibody (L26) eBioscience | Thermo Fisher | 53-0202-82 | antibody |
| Antibiotic-Antimycotic | Gibco | 15240096 | supplement harvest medium |
| BioMed Clear Resin (Form 3) | FormLabs | RS-F2-BMCL-01 | resin used for platform + incubation plate |
| BSA | Sigma | A7906-500g | blocking solution component |
| Cast acrylic 1/32" thick | material used for cutting slider | ||
| CleanStation DT3 | Stratasys | DT3 | post processing for height adjuster & outer lid |
| Container, Specimen | McKesson | 870203 | transfer of tissue from perating table to prep table |
| CorelDraw | CorelDRAW | Software to prepare the vector-based design file for laser cutting | |
| Cytofix | BD Bioscience | 554655 | fixative |
| Dish 15 cm | Falcon | 353025 | dish used during mounting |
| Dish 35 mm No. 1.5 Coverslip 14 mm Glass Diameter Uncoated | Matek | P35G-1.5-14-C | imaging dish |
| DMEM (no glucose) | Gibco | 11966025 | harvest medium |
| Fc Block | BD Bioscience | 564220 | blocking solution component |
| Form 3B+ | Formlabs | Form 3B+ | printer used for platform + incubation plate |
| Form Cure | Formlabs | FH-CU-01 | post processing for platform + incubation plate |
| Form Wash | Formlabs | FH-WA-01 | post processing for platform + incubation plate |
| GrabCAD Print | GrabCAD | GrabCAD Print | Software to prepare models for Stratasys printers |
| Hoechst 33342 10 mg/mL | Biotium | 40046 | nuclear dye |
| J826 Prime 3D Printer | Stratasys | J826 | printer used for height adjuster & outer lid |
| LAS X | Leica | LAS X | Confocal software |
| Laser cutting system | Universal Laser Systems | ULS PLS6.150D | CO2 Laser cutting used for slider |
| Lithium Borohydride | STREM Chemicals | 93-0397 | bleaching chemical |
| PBS, pH 7.4 | Gibco | 10010023 | base Buffer for washing, blocking, staining |
| PreForm | FormLabs | PreForm | Software to prepare models for Formlab printers |
| Silk sutures 2-0 | Ethicon | A305.O35 | affix tissue to platform |
| Stellaris 8 WLL confocal microscope | Leica | STELLARIS 8 | Confocal Imaging |
| Syringe filter | filter ab solution | ||
| Triton X-100 | American Bio | AB02025-00100 | permeabelizing reagent, blocking solution component |
| Vero ContactClear | Stratasys | CTT610, 4 KG | resin used for height adjuster & outer lid |
| Warming Tray | Spring USA | ST-1220 | keep media and tissue warm |
| Water, distilled | Gibco | 15230-170 | diluent for LiBH4 |
Referenzen
- Anderson, N. M., Simon, M. C. The tumor microenvironment. Curr Biol. 30 (16), R921-R925 (2020).
- Li, Z., Li, J., Bai, X., Huang, X., Wang, Q. Tumor microenvironment as a complex milieu driving cancer progression: A mini-review. Clin Transl Oncol. , (2024).
- Fridman, W. H., Zitvogel, L., Sautes-Fridman, C., Kroemer, G. The immune contexture in cancer prognosis and treatment. Nat Rev Clin Oncol. 14 (12), 717-734 (2017).
- Roma-Rodrigues, C., Mendes, R., Baptista, P. V., Fernandes, A. R. Targeting tumor microenvironment for cancer therapy. Int J Mol Sci. 20 (4), 840 (2019).
- Sheng, W., et al. Multiplex immunofluorescence: A powerful tool in cancer immunotherapy. Int J Mol Sci. 24 (4), 3086 (2023).
- Bollhagen, A., Bodenmiller, B. Highly multiplexed tissue imaging in precision oncology and translational cancer research. Cancer Discov. 14 (11), 2071-2088 (2024).
- Jin, Y., et al. Advances in spatial transcriptomics and its applications in cancer research. Mol Cancer. 23 (1), 129 (2024).
- Kashyap, A., et al. Quantification of tumor heterogeneity: From data acquisition to metric generation. Trends Biotechnol. 40 (6), 647-676 (2022).
- Chang, Q., Hedley, D. Emerging applications of flow cytometry in solid tumor biology. Methods. 57 (3), 359-367 (2012).
- Zhang, L., et al. Single-cell analyses inform mechanisms of myeloid-targeted therapies in colon cancer. Cell. 181 (2), 442-459.e29 (2020).
- Wang, X. Q., et al. Spatial predictors of immunotherapy response in triple-negative breast cancer. Nature. 621 (7980), 868-876 (2023).
- Maestri, E., et al. Spatial proximity of tumor-immune interactions predicts patient outcome in hepatocellular carcinoma. Hepatology. 79 (4), 768-779 (2024).
- Wang, Q., et al. Role of tumor microenvironment in cancer progression and therapeutic strategy. Cancer Med. 12 (10), 11149-11165 (2023).
- Veninga, V., Voest, E. E. Tumor organoids: Opportunities and challenges to guide precision medicine. Cancer Cell. 39 (9), 1190-1201 (2021).
- Lin, J. R., et al. Multiplexed 3d atlas of state transitions and immune interaction in colorectal cancer. Cell. 186 (2), 363-381.e19 (2023).
- Radtke, A. J., et al. Ibex: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Radtke, A. J., et al. Ibex: A versatile multiplex optical imaging approach for deep phenotyping and spatial analysis of cells in complex tissues. Proc Natl Acad Sci U S A. 117 (52), 33455-33465 (2020).
- Van Baal, J. O., et al. The histophysiology and pathophysiology of the peritoneum. Tissue Cell. 49 (1), 95-105 (2017).
- Bridda, A., Padoan, I., Mencarelli, R., Frego, M. Peritoneal mesothelioma: A review. MedGenMed. 9 (2), 32 (2007).
- Demuytere, J., Ernst, S., Van Ovost, J., Cosyns, S., Ceelen, W. The tumor immune microenvironment in peritoneal carcinomatosis. Int Rev Cell Mol Biol. 371, 63-95 (2022).
- Wang, Q., et al. Heterogeneity of tertiary lymphoid structures predicts the response to neoadjuvant therapy and immune microenvironment characteristics in triple-negative breast cancer. Br J Cancer. , (2024).
- Kepenekian, V., et al. Peritoneal mesothelioma: Systematic review of hyperthermic intraperitoneal chemotherapy (HIPEC) protocol outcomes. Indian J Surg Oncol. 14 (Suppl 1), 39-59 (2023).
- Mcquade, C., Renton, M., Chouhan, A., Macdermott, R., O'brien, C. Review of imaging peritoneal disease and treatment. Can Assoc Radiol J. , (2024).
- Kenerson, H. L., et al. Tumor slice culture as a biologic surrogate of human cancer. Ann Transl Med. 8 (4), 114 (2020).
- Arrizabalaga, L., et al. Tumor slice culture system for ex vivo immunotherapy studies. Methods Cell Biol. 189, 55-69 (2024).
- Neumeister, V. M., et al. Quantitative assessment of effect of preanalytic cold ischemic time on protein expression in breast cancer tissues. J Natl Cancer Inst. 104 (23), 1815-1824 (2012).
- Panchuk-Voloshina, N., et al. Alexa dyes: A series of new fluorescent dyes that yield exceptionally bright, photostable conjugates. J Histochem Cytochem. 47 (9), 1179-1188 (1999).
- Rajaram, S., et al. Sampling strategies to capture single-cell heterogeneity. Nat Methods. 14 (10), 967-970 (2017).
- Bella, A., et al. Mouse models of peritoneal carcinomatosis to develop clinical applications. Cancers (Basel). 13 (5), 963 (2021).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten