Method Article
L’imagerie tridimensionnelle de tissus porteurs de tumeurs à l’aide du blanchiment itératif étend l’approche de multiplexité
Dans cet article
Résumé
Cet article décrit un protocole d’immunofluorescence multiplex optimisé pour caractériser l’architecture tridimensionnelle des métastases péritonéales.
Résumé
L’hétérogénéité spatiale du microenvironnement tumoral (TME) est un déterminant essentiel de la réponse thérapeutique, en particulier pour les agents immuno-oncologiques, où le succès dépend de la distribution de sous-populations spécifiques de cellules immunitaires. Au cours de la dernière décennie, de multiples technologies sophistiquées ont été introduites pour obtenir une résolution détaillée de l’EUT à l’aide de coupes bidimensionnelles provenant de tissus fixés au formol, inclus en paraffine (FFPE) ou congelés fixes. Bien que ces lames minces soient plus faciles à obtenir et à analyser, elles ne disposent pas de l’architecture tridimensionnelle nécessaire pour caractériser de manière fiable et complète une tumeur. Pour remédier à cette limitation, une technique de montage et d’imagerie tissulaire a été développée pour permettre l’analyse tridimensionnelle des lésions tumorales dans leur état natif in vivo . Ce protocole décrit l’obtention de tissus tumoraux humains, le montage d’échantillons sur des plates-formes imprimées sur mesure et les procédures de coloration des échantillons post-fixation. La technique d’immunofluorescence multiplexée, IBEX (Iterative Bleaching Extends Multiplexity), a été adaptée pour caractériser l’EUT tridimensionnel avec jusqu’à 15 marqueurs pour les cellules tumorales, immunitaires et stromales à l’aide d’anticorps disponibles dans le commerce. Des profondeurs d’imagerie allant jusqu’à 100 μm ont été obtenues à l’aide d’un microscope confocal laser à lumière blanche inversée avec un adaptateur d’imagerie imprimé sur mesure et des antennes paraboliques commerciales à fond de verre pour assurer une orientation optimale des tissus. Ce protocole met en évidence le potentiel de la méthode IBEX pour étendre les études d’immunofluorescence multiplexée, fournissant une compréhension plus complète de la composition des EUT.
Introduction
Les tumeurs solides sont très variables, avec une composition complexe de types de cellules (malignes et non malignes), de protéines de la matrice extracellulaire et de facteurs solubles 1,2. En fin de compte, ce microenvironnement tumoral (TME) très complexe et hétérogène détermine la susceptibilité à des traitements tels que l’immunothérapie 3,4. Ainsi, des technologies telles que l’immunofluorescence hautement multiplexée 5,6 et la transcriptomique spatiale7 ont été appliquées pour caractériser la complexité de l’EUT dans des coupes épaisses de couche cellulaire (~4 μM).
Contrairement aux approches de dispersion cellulaire telles que le séquençage unicellulaire et la cytométrie en flux 8,9,10, ces techniques préservent les relations spatiales pour fournir des informations essentielles sur la diaphonie cellulaire et peuvent être utilisées pour identifier des biomarqueurs potentiels pour la prédiction de la réponse au traitement 11,12,13. Cependant, les lésions tumorales sont intrinsèquement des structures tridimensionnelles (3D), et les approches 2D comme celles mentionnées ci-dessus ne capturent pas de manière adéquate le paysage cellulaire complexe. L’importance de la biologie 3D a été appréciée dans le domaine, comme en témoigne l’utilisation généralisée des cultures d’organoïdes tumoraux dérivés de patients14. Cependant, les organoïdes ne conservent pas la complexité cellulaire de l’EUT in vivo d’origine, ce qui limite l’applicabilité à l’utilisation avec des médicaments immunomodulateurs ou à la caractérisation des diverses populations cellulaires d’EUT.
Bien qu’il soit possible d’effectuer des analyses sur des sections 2D en série et d’assembler les images par calcul pour un rendu 3D, cette technique est d’un coût prohibitif et difficile à réaliser avec une haute fidélité15. Afin de caractériser de manière rentable les tissus 3D (par exemple, les tumeurs) de manière reproductible, une méthode unique de préparation et de coloration/analyse par immunofluorescence multiplex a été développée et démontrée à l’aide de lésions tumorales péritonéales d’un patient atteint d’un cancer péritonéal primitif.
Basé sur une adaptation de la technique bien établie IBEX (Iterative Bleaching Extends Multiplexity)16,17, ce protocole détaille l’impression 3D d’un support de tissu et d’un adaptateur d’imagerie, l’approvisionnement et le montage de tissus péritonéaux, plusieurs cycles de coloration avec divers marqueurs de type tumoral et cellulaire, et l’imagerie 3D confocale à haute résolution.
Protocole
L’étude a été menée avec l’approbation du National Institutes of Health Institutional Review Board en vertu du protocole NCT01915225. Des tissus humains ont été obtenus avec un consentement éclairé écrit au moment de la laparoscopie diagnostique. Tous les tissus étaient porteurs d’une tumeur, comme l’a déterminé l’inspection visible et confirmé par l’examen histopathologique final. Les détails de tous les réactifs et équipements utilisés sont répertoriés dans la table des matériaux. Une illustration schématique de l’ensemble du flux de travail est fournie à la figure 1.
1. Plates-formes d’impression, adaptateur d’imagerie et plaque d’incubation
- Procédures de tranchage/impression 3D/post-traitement de la plate-forme et des pièces de plaque à 9 puits (illustrées à la figure 2A-C)
- Découpage : Ouvrez le logiciel de découpage et importez les fichiers de conception dans . STL (Fichier supplémentaire 1 et Fichier supplémentaire 2). Orientez les modèles pour une impression optimale et ajoutez des supports. Sélectionnez l’imprimante de qualité médicale, sélectionnez la résine transparente et la hauteur de couche de 50 μm . Vérifiez l’imprimabilité et envoyez le travail à l’imprimante.
- Impression 3D : Vérifiez que l’imprimante est prête (cartouche de résine, réservoir et plate-forme corrects en place). Sur l’écran tactile de l’imprimante, sélectionnez la tâche téléchargée dans le logiciel de découpe pour démarrer l’impression. Une fois l’impression terminée, utilisez l’outil de retrait pour détacher la pièce imprimée de la plate-forme.
- Post-traitement : Rincez la pièce dans l’unité de lavage remplie d’alcool isopropylique à 99 % (IPA) pendant 15 min. Retirez les pièces de l’unité de lavage et faites-les tremper dans de l’IPA fraîche pendant 5 min.
- Post-polymérisez la pièce pendant 60 min à 60 °C. Retirez les supports à l’aide d’un cutter affleurant. Poncez les marques de support à l’aide d’un papier de verre à grain fin si nécessaire.
- Procédures de tranchage/impression 3D/post-traitement pour le réglage de la hauteur et le couvercle extérieur (illustrés à la figure 2A-C)
- Découpage : Ouvrez le logiciel d’impression et importez les fichiers de conception dans . STL (Fichier supplémentaire 3 et Fichier supplémentaire 4). Orientez les modèles et attribuez les matériaux. Sélectionnez le mode d’impression souhaité (High mix, high speed) et réglez les paramètres du modèle pour finir sur Mat. Vérifiez l’imprimabilité et envoyez le travail à l’imprimante.
- Impression 3D : Assurez-vous que l’imprimante a les bons matériaux chargés et que le plateau de construction est propre. Sur l’interface du moniteur de l’imprimante, sélectionnez la tâche téléchargée pour démarrer l’impression. Une fois l’impression terminée, utilisez le grattoir pour retirer délicatement la pièce imprimée du plateau de construction.
- Post-traitement : Placez les pièces imprimées dans un nettoyeur à jet d’eau ou un système de nettoyage par agitation pour retirer le matériau de support. Effectuez tout retrait manuel du support, si nécessaire, à l’aide d’outils tels qu’une pince à épiler ou une pince.
- Découpe laser du curseur de plate-forme (illustré sur la figure 2A, C)
- Préparation du design : importez le fichier *. Fichier de conception DWG dans le logiciel vectoriel (Fichier supplémentaire 5). Réglez les couleurs des lignes sur rouge (RVB) pour la découpe vectorielle. Envoyez le travail à la découpeuse laser.
- Configuration de la découpeuse laser : Ouvrez le logiciel de découpe laser et importez le fichier de conception. Sélectionnez le type de matériau et ajustez les paramètres de puissance, de vitesse et de résolution pour l’acrylique de 1/32 po d’épaisseur. Placez le matériau sur le lit laser, assurez-vous qu’il est correctement aligné et faites la mise au point du laser.
- Découpe laser : Démarrez le processus de découpe laser et surveillez pour vous assurer que le matériau est correctement coupé.
2. Montage des tissus sur des plates-formes imprimées
- Préparation du milieu de récolte et de la table de préparation
- Compléter 100 ml de DMEM (faible teneur en glucose) avec Anti-Anti et placer dans un incubateur à 37 °C jusqu’à ce que nécessaire (milieu de récolte).
- Installez une « table de préparation » dans la salle d’opération avec les éléments suivants : plates-formes imprimées en 3D autoclavées, paquet de 2-0 attaches de suture en soie, plaque chauffante, tasse d’échantillon avec 50 ml de milieu de récolte préchauffé, plaque de culture de 15 cm contenant 30 ml de milieu de récolte préchauffé et une plaque de 24 puits avec 4 puits contenant chacun 1,5 ml de milieu de récolte (placer le tout sur une plaque chauffante), 1 paire de pinces et 1 paire de ciseaux chirurgicaux.
- Résection et montage tissulaire
REMARQUE : Le tissu est prélevé dans la salle d’opération. Les zones du péritoine avec des foyers tumoraux de moins de 0,5 mm d’épaisseur et de diamètre sont identifiées par inspection visuelle (Figure 2D). Il convient de noter que ~ 2 cm2 de tissu (péritoine) sont nécessaires pour préparer une plate-forme.- Séparez soigneusement le péritoine porteur de la tumeur de la musculature de la paroi abdominale sous-jacente et du fascia (effectué par le chirurgien). Transférez les feuilles de tissu obtenues dans un gobelet d’échantillonnage contenant du substrat de récolte chaud et placez-les sur une plaque chauffante (table de préparation).
- Montez le tissu sur des plates-formes dans la plaque de 15 cm contenant le milieu de récolte. Drapez soigneusement le tissu porteur de la tumeur (côté mésothélial vers le haut) sur le petit côté orifice de la plate-forme et fixez-le en place avec une suture en soie 2-0. Placer la plate-forme tissulaire préparée à l’envers (immergée) dans une plaque de 24 puits contenant le milieu de récolte (figure 2E-G).
REMARQUE : Montez le tissu suffisamment enseigné pour éviter les plis sur la surface de la plate-forme, mais ne générez pas de tension car cela induirait des changements biomécaniques et physiologiques indésirables.
3. Procédure de fixation/coloration/blanchiment
REMARQUE : Les modifications par rapport au protocole original adapté à des sections FFPE de 4 μm d’épaisseur ou à des sections congelées fixes sont décrites à la figure 3. Le tableau 1 fournit une liste des anticorps (avec dilutions) utilisés pour les rondes IBEX 1 à 6.
- Fixation
- Préparez 40 ml de tampon de fixation en ajoutant 10 ml de produit fixateur dans un tube conique de 50 ml contenant 30 ml de PBS froid.
- À l’aide d’une paire de pinces, transférez le tissu monté sur les plates-formes dans le tampon de fixation et incubez pendant 24 h à 4 °C.
- Décanter le fixateur et le remplacer par 40 mL de 1x PBS froid et incuber pendant 10 min à 4 °C.
- Coloration
- Ajouter 1 mL de tampon de blocage (1 % BSA, 0,3 % Triton-X-100, 1 % Fc Block dans 1x PBS filtré avec un filtre à seringue de 0,22 μm) dans chaque puits et insérer les plates-formes tissulaires. Bloquer pendant au moins 2 h à température ambiante sur une bascule réglée à basse vitesse (30 tr/min).
REMARQUE : Assurez-vous que l’encoche sur la plate-forme et la rainure dans le puits sont alignées. Cette étape n’est pas urgente et peut être effectuée toute la nuit à 4 °C. - Préparez 0,8 mL de solution d’anticorps/colorant dans un tube de microcentrifugation et centrifugez à 10 000 x g pendant 5 min à température ambiante. Transférez 0,75 mL dans un puits propre de la plaque d’incubation à 9 puits et insérez une plate-forme lavée.
- Enveloppez la plaque dans du papier d’aluminium et incubez pendant au moins 2 h à température ambiante sur une bascule réglée à basse vitesse (30 tr/min).
- Lavez la plate-forme dans un tube conique de 50 mL avec 40 mL de 1x PBS pendant 10 min à 4 °C. La plate-forme est maintenant prête à être insérée dans l’adaptateur d’imagerie.
- Ajouter 1 mL de tampon de blocage (1 % BSA, 0,3 % Triton-X-100, 1 % Fc Block dans 1x PBS filtré avec un filtre à seringue de 0,22 μm) dans chaque puits et insérer les plates-formes tissulaires. Bloquer pendant au moins 2 h à température ambiante sur une bascule réglée à basse vitesse (30 tr/min).
- Blanchiment
- Après chaque cycle de coloration et d’imagerie, blanchir les fluorophores à l’aide de l’agent réducteur d’esters borohydrure de lithium (LiBH4). Ce traitement éliminera les signaux de fluorescence d’Alexa Fluor (AF)488, AF647 et AF750, mais pas de l’AF594 et du colorant nucléaire Hoechst.
ATTENTION : Le borohydrure de lithium est extrêmement réactif avec l’eau. Des gaz d’hydrogène inflammables sont libérés au contact de l’eau ; Le borohydrure de lithium usagé et les produits jetables contaminés par le borohydrure de lithium doivent être éliminés comme déchets dangereux. - Préparez 5 ml de solution de LiBH4 à 1,5 mg/mL dans de l’eau distillée (dans une hotte chimique) et incubez pendant 30 minutes à température ambiante.
REMARQUE : La solution LiBH4 perdra son efficacité réductrice/blanchissante lorsqu’elle est préparée >4 h avant utilisation. - Transvaser 1 mL de solution de LiBH4 dans un puits propre de la plaque d’incubation à 9 puits et insérer la plate-forme lavée pendant 60 minutes à température ambiante.
- Lavez brièvement la plate-forme dans 20 ml de 1x PBS dans un tube conique et vérifiez le signal de fluorescence restant à l’aide du réglage laser le plus élevé pour la compensation Z lors du cycle d’imagerie précédent avant de passer aux cycles de coloration suivants.
- Répétez le blocage de l’échantillon pendant 2 h à température ambiante avant de passer à l’étape suivante d’incubation de l’anticorps.
- Après chaque cycle de coloration et d’imagerie, blanchir les fluorophores à l’aide de l’agent réducteur d’esters borohydrure de lithium (LiBH4). Ce traitement éliminera les signaux de fluorescence d’Alexa Fluor (AF)488, AF647 et AF750, mais pas de l’AF594 et du colorant nucléaire Hoechst.
4. Procédure d’imagerie
- Assemblage d’adaptateurs/plates-formes d’imagerie
- Ajouter 0,1 ml de 1x PBS au centre du fond en verre d’une parabole d’imagerie de 3,5 cm et réserver. Vissez la bague de réglage de la hauteur dans le sens des aiguilles d’une montre dans le couvercle extérieur et montez la plate-forme dans le support intérieur en plastique (alignement encoche-rainure). Fixez la plate-forme avec le curseur.
- Placez l’adaptateur d’imagerie assemblé sur la parabole à fond en verre et abaissez soigneusement la bague de réglage de la hauteur jusqu’à ce que le tissu touche le tampon. Évitez de faire pivoter la plate-forme sur le fond en verre.
- Une fois que la distance optimale du verre est atteinte, ajoutez 0,2 ml de 1x PBS sur le tissu (maintenant le dessous vers le haut) pour éviter la déshydratation des tissus pendant l’imagerie.
- Imagerie confocale
- Allumez tous les composants du microscope confocal inversé, y compris l’ordinateur, le microscope, le laser et la source lumineuse LED pour l’utilisation de l’oculaire. Démarrez le logiciel d’acquisition d’images .
- Sélectionnez l’objectif approprié (par exemple, 20x ou 40x) et ajoutez le support d’immersion approprié. Par exemple, un objectif d’eau de 20x est utilisé dans cette étude ; ~200 μL d’eau sont ajoutés à l’objectif pour éviter le dessèchement lors de longues acquisitions d’images.
- Placez la boîte d’imagerie assemblée dans le porte-échantillon sur la platine du microscope et assurez-vous que l’échantillon est bien fixé (ne bouge pas).
REMARQUE : Lorsque vous placez l’échantillon assemblé sur la platine du microscope, notez la position de l’encoche et utilisez la même position pour les cycles itératifs afin de faciliter l’alignement de l’image. Il est important de maintenir l’adaptateur d’imagerie à la hauteur optimale pour des tours consécutifs. - Centrez l’échantillon à l’aide de la commande X-Y et amenez l’objectif jusqu’à la vitre de protection. En regardant à travers l’oculaire et en utilisant le signal de Hoechst comme référence, déplacez l’objectif pour obtenir une mise au point fine sur le tissu.
- Sélectionnez les lignes laser appropriées ou ajustez la CMU aux spectres d’excitation et d’émission des fluorophores utilisés pour colorer l’échantillon de tissu. Éliminez le chevauchement entre les canaux en ajustant les paramètres et ajoutez des séquences si les fluorophores ont des spectres d’excitation similaires.
- Choisissez les paramètres d’acquisition d’image suivants : vitesse de balayage 600 Hz, résolution XY 1024 x 1024, moyennes sur 3 lignes et balayage bidirectionnel .
- Choisissez les paramètres de pile Z suivants : (1) utilisez la taille du pas Z optimisée pour l’objectif utilisé, (2) définissez le début et la fin de la pile, (3) ajustez les paramètres de compensation Z pour obtenir une luminosité égale dans toute la pile et (4) acquérez une pile de test à 1 tuile avec une moyenne.
- Définissez la région à imager et acquérez une pile Tile-Z haute résolution.
Résultats
Le péritoine est une structure membraneuse de moins de 1 mm d’épaisseur qui tapisse la surface de la paroi abdominale et qui est contiguë aux surfaces des organes abdominaux. Il est composé d’un mésothélium de surface et d’un tissu conjonctif sous-jacent qui est entrecoupé de cellules adipeuses, de lymphocytes, de macrophages et de fibroblastes, ainsi que de vaisseaux sanguins et lymphatiques. Les tumeurs malignes du péritoine proviennent le plus souvent de métastases tumorales abdominales (par exemple, le cancer de l’ovaire ou le cancer gastrique)18 ou, moins fréquemment, peuvent être des tumeurs primitives du péritoine (par exemple, le mésothéliome)19. Ces lésions tumorales souvent petites (<1 cm) sont très complexes mais se prêtent à une caractérisation 3D à l’aide de la technique décrite. Une projection maximale de cinq sections confocales est utilisée pour visualiser l’ensemble de l’échantillon monté sur une plate-forme (figure 4, encastrée dans le coin supérieur gauche). Trois anticorps conjugués primaires (AF488-CD44, AF594-CD45 et AF647-Podoplanin) ainsi que le colorant nucléaire Hoechst ont été utilisés pour la première série d’études IBEX (IBEX1, figure 4). Les lésions tumorales deviennent évidentes par les structures en forme de rosette qui sont doublement positives pour CD44 et la podoplanine et montrent un arrangement de noyaux caractéristique du mésothéliome papillaire.
Les cellules tumorales ont été identifiées à l’aide de la morphologie cellulaire et de deux anticorps, car l’anti-Podoplanin et l’anti-CD44 reconnaissent les cellules non tumorales, comme en témoigne la détection des vaisseaux lymphatiques via l’anti-Podoplanine et la liaison de l’anticorps CD44 à des sous-ensembles de cellules immunitaires. Les régions d’intérêt plus petites sont ensuite capturées à l’aide d’un format de balayage de tuiles à une résolution plus élevée à des tailles de pas Z optimisées pour le rendu des projections 3D (Figure 5A). Les plates-formes ont été soumises aux procédures de coloration itérative, et les mêmes ROI ont été imagés dans les cycles IBEX 1 à 6. Des images représentatives des interfaces tumeur (boîte jaune) et tumeur-structure lymphoïde tertiaire (TLS) (boîte bleue) sont assemblées dans la figure 5B-M.
La présence de TLS et la densité de cellules CD3 positives indiquent une infiltration significative des cellules immunitaires20 et peut-être une réponse aux médicaments immunomodulateurs21. Il convient de noter que le TLS et la majeure partie des lésions tumorales sont présents dans différentes profondeurs Z du tissu, comme le montre la comparaison des projections d’empilement de différentes sections optiques dans la figure 6, mettant en évidence les avantages de l’imagerie 3D et le manque potentiel de biologie lorsque l’imagerie 2D est appliquée aux structures 3D. Le caractère 3D de ces lésions devient plus évident dans les fichiers vidéo (vidéo 1-12), qui représentent les rendus volumiques des lésions individuelles dans la figure 5.
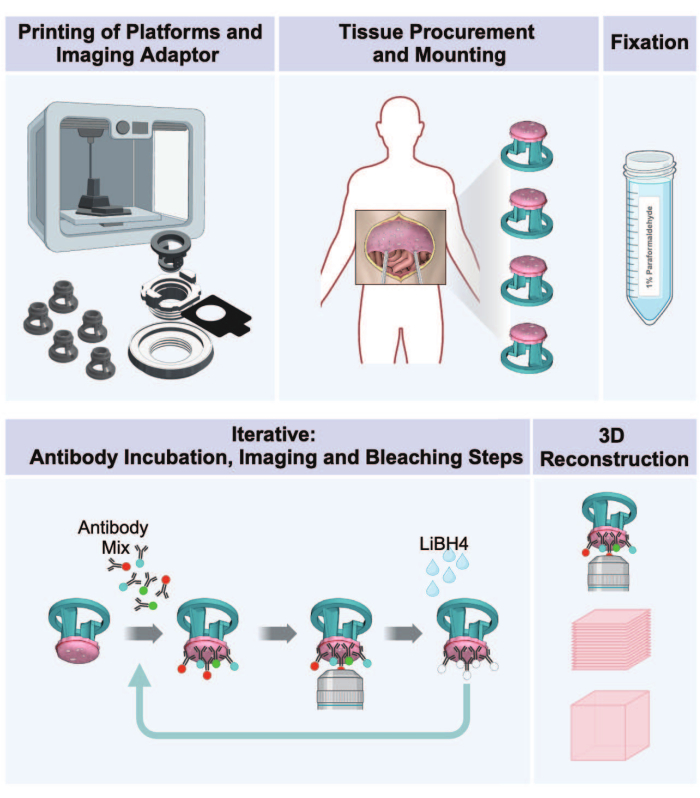
Figure 1 : Flux de travail de l’impression 3D à l’imagerie 3D des tumeurs péritonéales. Le protocole consiste en une séquence de cinq étapes pour préparer des images multiplexées 3D à partir de tissus péritonéaux porteurs de tumeurs. (1) Impression de plates-formes réceptrices de tissus, de pièces d’adaptateur d’imagerie et de plaques d’incubation. (2) Procédures d’approvisionnement et de montage des tissus. (3) Fixation avec du paraformaldéhyde. (4) Cycles de coloration et d’imagerie IBEX. (5) Reconstruction 3D à l’aide d’un logiciel d’imagerie. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Composants matériels avec interface tissulaire pour l’imagerie 3D. (A) Schéma de principe de l’assemblage de la plate-forme dans l’adaptateur d’imagerie. (B) Une plaque d’incubation à 9 puits. (C) Photographies des composants de l’adaptateur d’image avec assemblage. (D) Péritoine porteur de mésothéliome observé lors de la laparoscopie diagnostique. (E) Montage du tissu sur la plate-forme. (F,G) Péritoine porteur de tumeur monté sur des plates-formes. Veuillez cliquer ici pour voir une version agrandie de cette figure.
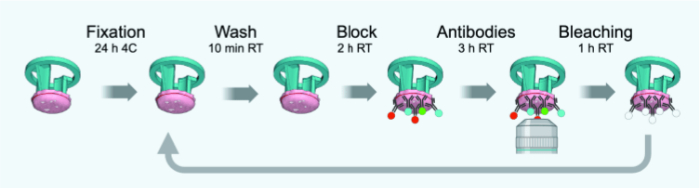
Figure 3 : Schéma chronologique des cycles IBEX. Veuillez cliquer ici pour voir une version agrandie de cette figure.
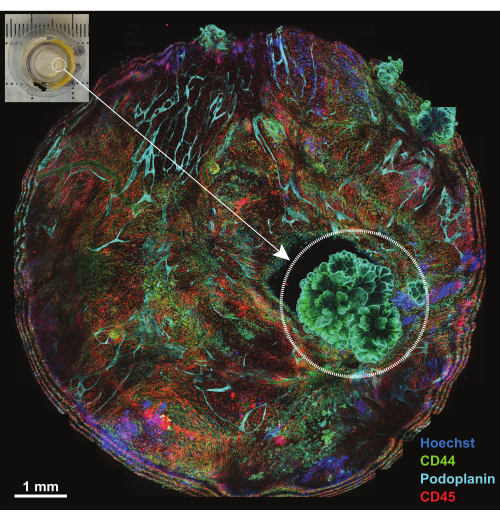
Figure 4 : Vue d’ensemble de la plateforme après l’incubation avec le panel IBEX1. Projection maximale de l’ensemble de la plateforme après l’incubation avec le panel IBEX1 (Hoechst, AF488-CD44, AF594-CD45 et AF647-Podoplanin). Le cercle pointillé met en évidence la tumeur. Encadré : Photographie d’un péritoine porteur d’une tumeur monté sur la plate-forme d’imagerie. Barre d’échelle : 1 mm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
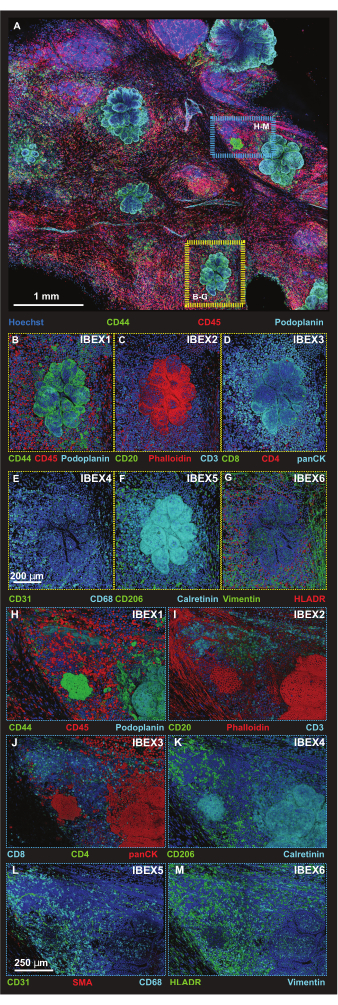
Figure 5 : Images d’immunofluorescence à haute résolution de certaines régions de la plateforme. (A) Projection maximale de l’image fusionnée de la pile Z de 56 tuiles acquise après incubation avec le panel IBEX1 (Hoechst, AF488-CD44, AF594-CD45 et AF647-Podoplanin). Rectangle jaune : lésion tumorale représentée sur des panneaux (B-G). Rectangle bleu : interface tumeur-structure lymphoïde tertiaire représentée en (H-M). (B-G) Une lésion tumorale individuelle avec un panel IBEX de 1 à 6 en rendu de volume. (B) IBEX1, vert : CD44, rouge : CD45, cyan : podoplanine. (C) IBEX2, vert : CD20, rouge : F-actine, cyan : CD3. (D) IBEX3, vert : CD8, rouge : CD4, cyan : panCK. (E) IBEX4, vert : CD31, cyan : CD68. (F) IBEX5, vert : CD206, cyan : calrétinine. (G) IBEX6, vert : Vimentin, rouge : HLADR. (H-M) Une interface individuelle de structure lymphoïde tumeur-tertiaire avec le panel IBEX 1-6 en rendu de volume. (H) IBEX1, vert : CD44, rouge : CD45, cyan : podoplanine. (I) IBEX2, vert : CD20, rouge : F-actine, cyan : CD3. (J) IBEX3, vert : CD4, rouge : panCK, cyan : CD8. (K) IBEX4, vert : CD31, rouge : SMA, cyan : CD68. (L) IBEX5, vert : CD206, cyan : calrétinine. (M) IBEX6, vert : HLADR, cyan : Vimentin. Tous les panneaux, bleus : Hoechst. Barres d’échelle : (A, 1 mm) ; (B-M, 250 μm). Veuillez cliquer ici pour voir une version agrandie de cette figure.
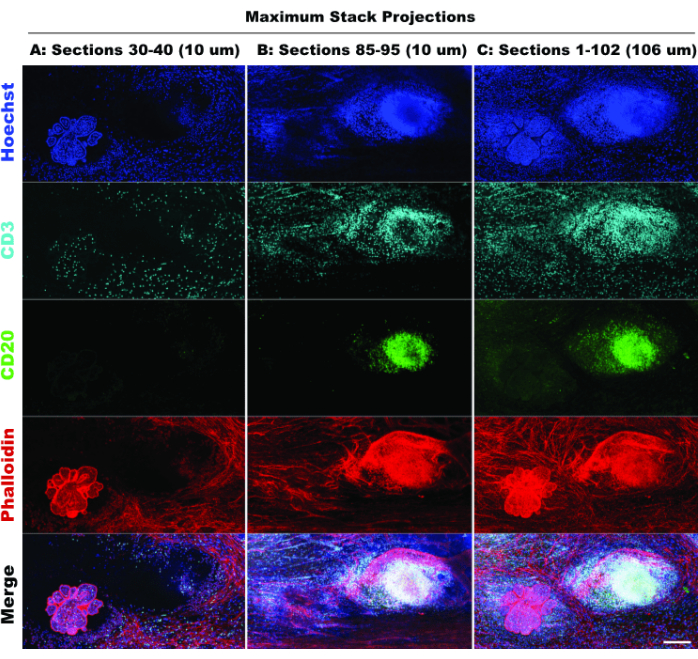
Figure 6 : Imagerie 2D simulée. La comparaison de projections limitées (10 sections optiques) et de projections complètes (102 sections optiques) confirme la perte de contexte tissulaire dans la simulation 2D. (A) Projection maximale des sections 30-40 (10 μm). (B) Projection maximale des sections 85-95 (10 μm). (C) Projection maximale des sections 1-102 (~100 μm). Tous les panneaux, IBEX2 (Hoechst, AF488-CD20, AF647-CD3, AF790-phalloidin). Barre d’échelle : 200 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau 1 : Anticorps utilisés pour les différents panels IBEX. Veuillez cliquer ici pour télécharger ce tableau.
Vidéo 1 : Rendu volumique animé de la lésion individuelle avec le panneau IBEX 1 (bleu : Hoechst, vert : CD44, rouge : CD45, cyan : Podoplanin). Veuillez cliquer ici pour télécharger ce film.
Vidéo 2 : Rendu volumique animé de la lésion individuelle avec le panneau IBEX 2 (bleu : Hoechst, vert : CD20, rouge : F-actin, cyan : CD3). Veuillez cliquer ici pour télécharger ce film.
Vidéo 3 : Rendu volumique animé de la lésion individuelle avec le panneau IBEX 3 (bleu : Hoechst, vert : CD8, rouge : CD4, cyan : panCK). Veuillez cliquer ici pour télécharger ce film.
Vidéo 4 : Rendu volumique animé de la lésion individuelle avec le panneau IBEX 4 (bleu : Hoechst, vert : CD31, cyan : CD68). Veuillez cliquer ici pour télécharger ce film.
Vidéo 5 : Rendu volumique animé de la lésion individuelle avec le panneau IBEX 5 (bleu : Hoechst, vert : CD206, cyan : Calrétinine). Veuillez cliquer ici pour télécharger ce film.
Vidéo 6 : Rendu volumique animé de la lésion individuelle avec le panneau IBEX 6 (bleu : Hoechst, vert : Vimentin, rouge : HLADR). Veuillez cliquer ici pour télécharger ce film.
Vidéo 7 : Rendu volumique animé de l’interface structure-lésion lymphoïde tertiaire avec le panneau IBEX 1 (bleu : Hoechst, vert : CD44, rouge : CD45, cyan : Podoplanin). Veuillez cliquer ici pour télécharger ce film.
Vidéo 8 : Rendu volumique animé de l’interface structure-lésion lymphoïde tertiaire avec le panneau IBEX 2 (bleu : Hoechst, vert : CD20, rouge : F-actin, cyan : CD3). Veuillez cliquer ici pour télécharger ce film.
Vidéo 9 : Rendu volumique animé de l’interface structure-lésion lymphoïde tertiaire avec le panneau IBEX 3 (bleu : Hoechst, vert : CD8, rouge : CD4, cyan : panCK). Veuillez cliquer ici pour télécharger ce film.
Vidéo 10 : Rendu volumique animé de l’interface structure-lésion lymphoïde tertiaire avec le panneau IBEX 4 (bleu : Hoechst, vert : CD31, rouge : SMA, cyan : CD68). Veuillez cliquer ici pour télécharger ce film.
Vidéo 11 : Rendu volumique animé de l’interface structure-lésion lymphoïde tertiaire avec le panneau IBEX 5 (bleu : Hoechst, vert : CD206, cyan : Calrétinine). Veuillez cliquer ici pour télécharger ce film.
Vidéo 12 : Rendu volumique animé de l’interface structure-lésion lymphoïde tertiaire avec le panneau IBEX 6 (bleu : Hoechst, vert : Vimentin, rouge : HLADR). Veuillez cliquer ici pour télécharger ce film.
Fichier supplémentaire 1 : Fichier d’impression de la plaque d’incubation. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 2 : Fichier d’impression pour les 36 plateformes individuelles. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 3 : Fichier d’impression pour la bague de réglage de la hauteur. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 4 : Imprimer le fichier pour le couvercle extérieur. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 5 : Fichier découpé au laser pour le curseur. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Les techniques multiplex actuelles pour l’imagerie par fluorescence sont limitées aux lames minces et ne fournissent pas de contexte 3D. La présente étude décrit un protocole permettant d’appliquer la méthode d’imagerie IBEX16,17 à des tumeurs intactes montées sur des plates-formes conçues sur mesure. Les lésions tumorales péritonéales ont été choisies pour mettre en évidence la technique, car les patients présentent souvent plusieurs dépôts tumoraux de taille idéale qui sont prélevés avec le péritoine normal environnant dans le cadre de la chirurgie de cytoréduction standard entreprise dans la plupart des centres de référence tertiaires à travers le monde22,23. Le protocole est néanmoins applicable aux métastases parenchymateuses et aux tumeurs modèles murins en utilisant des coupes tumorales comme cela a été décrit précédemment24,25. De plus, le diamètre de l’anneau de montage sur les plates-formes peut être ajusté aux besoins expérimentaux/tissulaires, bien que les zones plus grandes soient sujettes à l’affaissement des tissus et aux temps d’imagerie prolongés. Quelles que soient la source et la taille des tissus, le temps entre l’obtention et la conservation doit être réduit au minimum pour garantir une qualité optimale26. L’installation d’une table de préparation dans la salle d’opération garantit un traitement rapide lorsque des patients sont impliqués. Le temps écoulé entre l’obtention et la fixation du tissu doit être noté pour assurer la comparabilité entre les expériences.
Les échantillons de tissus sont prélevés selon le protocole IBEX au fur et à mesure que des montages entiers, c’est-à-dire la fixation et l’imagerie ainsi que les étapes de blanchiment, sont effectués avec le tissu intact fixé à la plate-forme. Cela garantit la conservation de l’échantillon tout au long des cycles itératifs de coloration/imagerie/blanchiment, car il n’y a pas d’interface semi-permanente entre la section et le verre et il n’est pas nécessaire de placer/retirer à plusieurs reprises une vitre de protection du tissu, ce qui peut entraîner une perte de tissu. Pour économiser sur les réactifs, la plaque à 9 puits conçue sur mesure garantit un ajustement optimal de la plate-forme avec un « volume mort » minimal. Tous les anticorps utilisés pour la coloration sont des anticorps conjugués primaires, et tous les conjugués fluorophores utilisés (à l’exception du colorant Hoechst) sont des colorants Alexa Fluor, qui ont été choisis pour leur excellente brillance27. D’autres panels d’anticorps ont été conçus sur la base des données déposées par la communauté d’imagerie IBEX, un référentiel mondial ouvert rassemblant des ensembles de données, des protocoles et des commentaires d’un groupe international de scientifiques utilisant IBEX (https://ibeximagingcommunity.github.io/ibex_imaging_knowledge_base/). La séquence des panels d’anticorps a été soigneusement choisie. En général, les cibles peu abondantes ont été placées dans des cycles plus précoces. Le cycle 1 d’IBEX (CD45, CD44 et Podoplanin) est une exception puisque ces trois marqueurs ont été utilisés pour identifier les lésions tumorales lors du cycle initial de coloration.
Les panels d’anticorps sont relativement petits pour réduire la diaphonie des canaux à haute intensité laser, ce qui est nécessaire pour imager les tissus plus profondément à l’aide d’un microscope confocal. Les conjugués AF750 ont été inclus dans la mesure du possible pour étendre le panel de 2 à 3 anticorps. Il convient de noter qu’il n’y a pas beaucoup de conjugués AF750 disponibles dans le commerce, et le fait que la cible de ce canal devrait être très abondante limite l’utilisation de ce fluorophore. Un exemple est l’anticorps anti-SMA conjugué à l’AF750 qui a été utilisé dans ce protocole. Bien qu’il s’agisse d’un clone qui fonctionne de manière fiable conjugué à AF488 ou AF594, l’intensité du signal de cet anticorps conjugué à AF750 est nettement inférieure, ce qui rend difficile la génération d’empilements Z de haute qualité.
Les incubations d’anticorps de 3 h à température ambiante sont courtes par rapport au protocole original et reflètent très probablement la densité cellulaire plus faible dans les échantillons de tissu péritonéal. En revanche, l’étape de blanchiment avec 1,5 mg/mL de borohydrure de lithium pendant 60 minutes était plus stricte. Ces paramètres ont été déterminés empiriquement et peuvent varier selon les tissus et les anticorps utilisés. Il convient de noter qu’en raison de l’incubation dans la solution de blanchiment réactive, de petites bulles d’air peuvent être piégées dans les tissus. Chaque fois que cela devenait évident au cours de l’étape d’imagerie de la procédure, les échantillons étaient dégazés en exposant la plate-forme dans la plaque d’incubation à 9 puits à un vide à l’aide d’une cartouche d’aspiration. Des enregistrements d’échantillons ITK simples, souvent appliqués dans les études IBEX, n’ont pas pu être effectués en raison de la saisie écrasante de données dans le logiciel d’analyse d’images. Néanmoins, des populations cellulaires identiques sont facilement identifiées en comparant des ensembles de données individuels. Dans l’ensemble, les cycles itératifs ont été chronométrés de manière à ce qu’un cycle puisse être complété en une journée, l’imagerie occupant la majeure partie d’un cycle entier (~ 8 h).
La technique a des limites qui méritent d’être mentionnées. En raison du temps prescrit par le protocole, il est peu probable que cette méthode soit adoptée à grande échelle, mais plutôt dans certaines circonstances. Nous envisageons que ce protocole soit mieux utilisé pour caractériser les changements dans le microenvironnement immunitaire tumoral avant et après le traitement (par exemple, les anticorps bispécifiques pour l’immunothérapie du cancer) ou pour prédire les résultats généraux du traitement. Bien que ce protocole offre une caractérisation 3D, on ne sait pas quelle zone doit être interrogée pour décrire efficacement une tumeur, y compris ses populations cellulaires complexes28. L’accès à des tissus humains frais peut également être difficile dans certains centres, mais nous prévoyons que ce protocole s’applique également aux modèlesanimaux29.
Les applications de ce protocole sont diverses, de la recherche fondamentale analysant la composition des EUT dans plusieurs tumeurs solides aux biopsies pré/post-tissulaires pour les patients inscrits à des essais cliniques. Bien que les données disponibles à partir d’une coupe de tissu épaisse d’une seule couche cellulaire soient immenses, la biologie 3D peut être facilement négligée. Nous considérons l’imagerie 3D comme un complément à d’autres techniques de biologie spatiale, ce qui aidera probablement à sélectionner des sections 2D pour les analyses ultérieures. De plus, un protocole permettant d’incorporer un composant d’imagerie en direct sur plusieurs heures à l’aide du montage et de la configuration d’imagerie tissulaires décrits est actuellement en cours.
Déclarations de divulgation
Aucun.
Remerciements
Cette recherche a été soutenue par le programme de recherche intra-muros des National Institutes of Health et du National Cancer Institute (NCI). Cette recherche a également été partiellement soutenue par le CAT-I, une collaboration de recherche entre le NIAID et le NCI, dirigée par Ronald Germain. Nous tenons à exprimer notre gratitude à Andrea Radtke pour ses efforts enthousiastes et collaboratifs. Son expertise a grandement profité à cette étude.
matériels
| Name | Company | Catalog Number | Comments |
| Alexa Fluor Plus 750 Phalloidin | Invitrogen | A30105 | F-actin dye |
| Alexa Fluor 488 anti-human CD206 (MMR) Antibody | BioLegend | 321114 | antibody |
| Alexa Fluor 488 anti-human CD31 Antibody | BioLegend | 303110 | antibody |
| Alexa Fluor 488 anti-human CD4 Antibody | BioLegend | 300519 | antibody |
| Alexa Fluor 488 anti-human HLA-DR Antibody | BioLegend | 307656 | antibody |
| Alexa Fluor 488 anti-mouse/human CD44 Antibody | BioLegend | 103016 | antibody |
| Alexa Fluor 594 anti-human CD45 Antibody | BioLegend | 304060 | antibody |
| Alexa Fluor 647 Anti-Calretinin antibody [EP1798] | Abcam | ab214244 | antibody |
| Alexa Fluor 647 anti-human CD3 Antibody | BioLegend | 300416 | antibody |
| Alexa Fluor 647 anti-human CD8 Antibody | BioLegend | 344726 | antibody |
| Alexa Fluor 647 anti-human Podoplanin Antibody | BioLegend | 337007 | antibody |
| Alexa Fluor 647 anti-Vimentin Antibody | BioLegend | 677807 | antibody |
| Alexa Fluor 647 CD68 Antibody (KP1) | Santa Cruz | sc-20060AF647 | antibody |
| Alexa Fluor 750 Cytokeratin, pan Antibody (AE-1/AE-3) | Novus | NBP2-33200AF750 | antibody |
| Alexa Fluor 750 Human alpha-Smooth Muscle Actin Antibody | R&D | IC1420S | antibody |
| Alexa Fluor 488 CD20 Monoclonal Antibody (L26) eBioscience | Thermo Fisher | 53-0202-82 | antibody |
| Antibiotic-Antimycotic | Gibco | 15240096 | supplement harvest medium |
| BioMed Clear Resin (Form 3) | FormLabs | RS-F2-BMCL-01 | resin used for platform + incubation plate |
| BSA | Sigma | A7906-500g | blocking solution component |
| Cast acrylic 1/32" thick | material used for cutting slider | ||
| CleanStation DT3 | Stratasys | DT3 | post processing for height adjuster & outer lid |
| Container, Specimen | McKesson | 870203 | transfer of tissue from perating table to prep table |
| CorelDraw | CorelDRAW | Software to prepare the vector-based design file for laser cutting | |
| Cytofix | BD Bioscience | 554655 | fixative |
| Dish 15 cm | Falcon | 353025 | dish used during mounting |
| Dish 35 mm No. 1.5 Coverslip 14 mm Glass Diameter Uncoated | Matek | P35G-1.5-14-C | imaging dish |
| DMEM (no glucose) | Gibco | 11966025 | harvest medium |
| Fc Block | BD Bioscience | 564220 | blocking solution component |
| Form 3B+ | Formlabs | Form 3B+ | printer used for platform + incubation plate |
| Form Cure | Formlabs | FH-CU-01 | post processing for platform + incubation plate |
| Form Wash | Formlabs | FH-WA-01 | post processing for platform + incubation plate |
| GrabCAD Print | GrabCAD | GrabCAD Print | Software to prepare models for Stratasys printers |
| Hoechst 33342 10 mg/mL | Biotium | 40046 | nuclear dye |
| J826 Prime 3D Printer | Stratasys | J826 | printer used for height adjuster & outer lid |
| LAS X | Leica | LAS X | Confocal software |
| Laser cutting system | Universal Laser Systems | ULS PLS6.150D | CO2 Laser cutting used for slider |
| Lithium Borohydride | STREM Chemicals | 93-0397 | bleaching chemical |
| PBS, pH 7.4 | Gibco | 10010023 | base Buffer for washing, blocking, staining |
| PreForm | FormLabs | PreForm | Software to prepare models for Formlab printers |
| Silk sutures 2-0 | Ethicon | A305.O35 | affix tissue to platform |
| Stellaris 8 WLL confocal microscope | Leica | STELLARIS 8 | Confocal Imaging |
| Syringe filter | filter ab solution | ||
| Triton X-100 | American Bio | AB02025-00100 | permeabelizing reagent, blocking solution component |
| Vero ContactClear | Stratasys | CTT610, 4 KG | resin used for height adjuster & outer lid |
| Warming Tray | Spring USA | ST-1220 | keep media and tissue warm |
| Water, distilled | Gibco | 15230-170 | diluent for LiBH4 |
Références
- Anderson, N. M., Simon, M. C. The tumor microenvironment. Curr Biol. 30 (16), R921-R925 (2020).
- Li, Z., Li, J., Bai, X., Huang, X., Wang, Q. Tumor microenvironment as a complex milieu driving cancer progression: A mini-review. Clin Transl Oncol. , (2024).
- Fridman, W. H., Zitvogel, L., Sautes-Fridman, C., Kroemer, G. The immune contexture in cancer prognosis and treatment. Nat Rev Clin Oncol. 14 (12), 717-734 (2017).
- Roma-Rodrigues, C., Mendes, R., Baptista, P. V., Fernandes, A. R. Targeting tumor microenvironment for cancer therapy. Int J Mol Sci. 20 (4), 840 (2019).
- Sheng, W., et al. Multiplex immunofluorescence: A powerful tool in cancer immunotherapy. Int J Mol Sci. 24 (4), 3086 (2023).
- Bollhagen, A., Bodenmiller, B. Highly multiplexed tissue imaging in precision oncology and translational cancer research. Cancer Discov. 14 (11), 2071-2088 (2024).
- Jin, Y., et al. Advances in spatial transcriptomics and its applications in cancer research. Mol Cancer. 23 (1), 129 (2024).
- Kashyap, A., et al. Quantification of tumor heterogeneity: From data acquisition to metric generation. Trends Biotechnol. 40 (6), 647-676 (2022).
- Chang, Q., Hedley, D. Emerging applications of flow cytometry in solid tumor biology. Methods. 57 (3), 359-367 (2012).
- Zhang, L., et al. Single-cell analyses inform mechanisms of myeloid-targeted therapies in colon cancer. Cell. 181 (2), 442-459.e29 (2020).
- Wang, X. Q., et al. Spatial predictors of immunotherapy response in triple-negative breast cancer. Nature. 621 (7980), 868-876 (2023).
- Maestri, E., et al. Spatial proximity of tumor-immune interactions predicts patient outcome in hepatocellular carcinoma. Hepatology. 79 (4), 768-779 (2024).
- Wang, Q., et al. Role of tumor microenvironment in cancer progression and therapeutic strategy. Cancer Med. 12 (10), 11149-11165 (2023).
- Veninga, V., Voest, E. E. Tumor organoids: Opportunities and challenges to guide precision medicine. Cancer Cell. 39 (9), 1190-1201 (2021).
- Lin, J. R., et al. Multiplexed 3d atlas of state transitions and immune interaction in colorectal cancer. Cell. 186 (2), 363-381.e19 (2023).
- Radtke, A. J., et al. Ibex: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Radtke, A. J., et al. Ibex: A versatile multiplex optical imaging approach for deep phenotyping and spatial analysis of cells in complex tissues. Proc Natl Acad Sci U S A. 117 (52), 33455-33465 (2020).
- Van Baal, J. O., et al. The histophysiology and pathophysiology of the peritoneum. Tissue Cell. 49 (1), 95-105 (2017).
- Bridda, A., Padoan, I., Mencarelli, R., Frego, M. Peritoneal mesothelioma: A review. MedGenMed. 9 (2), 32 (2007).
- Demuytere, J., Ernst, S., Van Ovost, J., Cosyns, S., Ceelen, W. The tumor immune microenvironment in peritoneal carcinomatosis. Int Rev Cell Mol Biol. 371, 63-95 (2022).
- Wang, Q., et al. Heterogeneity of tertiary lymphoid structures predicts the response to neoadjuvant therapy and immune microenvironment characteristics in triple-negative breast cancer. Br J Cancer. , (2024).
- Kepenekian, V., et al. Peritoneal mesothelioma: Systematic review of hyperthermic intraperitoneal chemotherapy (HIPEC) protocol outcomes. Indian J Surg Oncol. 14 (Suppl 1), 39-59 (2023).
- Mcquade, C., Renton, M., Chouhan, A., Macdermott, R., O'brien, C. Review of imaging peritoneal disease and treatment. Can Assoc Radiol J. , (2024).
- Kenerson, H. L., et al. Tumor slice culture as a biologic surrogate of human cancer. Ann Transl Med. 8 (4), 114 (2020).
- Arrizabalaga, L., et al. Tumor slice culture system for ex vivo immunotherapy studies. Methods Cell Biol. 189, 55-69 (2024).
- Neumeister, V. M., et al. Quantitative assessment of effect of preanalytic cold ischemic time on protein expression in breast cancer tissues. J Natl Cancer Inst. 104 (23), 1815-1824 (2012).
- Panchuk-Voloshina, N., et al. Alexa dyes: A series of new fluorescent dyes that yield exceptionally bright, photostable conjugates. J Histochem Cytochem. 47 (9), 1179-1188 (1999).
- Rajaram, S., et al. Sampling strategies to capture single-cell heterogeneity. Nat Methods. 14 (10), 967-970 (2017).
- Bella, A., et al. Mouse models of peritoneal carcinomatosis to develop clinical applications. Cancers (Basel). 13 (5), 963 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon