Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Vecteurs d'ingénierie et de l'évolution de la synthèse Adeno-Associated Virus (AAV) thérapie génique par la famille de réarrangement d'ADN
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Nous démontrons la technique de base pour concevoir et faire évoluer moléculaire synthétiques viral adéno-associé (AAV) vecteurs de thérapie génique par l'intermédiaire de la famille réarrangement d'ADN. En outre, nous fournir des directives générales et des exemples représentatifs de sélection et d'analyse des différents capsides chimériques avec des propriétés améliorées sur les cellules cibles en culture ou chez la souris.
Résumé
Viral adéno-associé (AAV) représentent quelques-uns des véhicules les plus puissants et prometteurs pour le transfert de gène thérapeutique humaine due à une combinaison unique de propriétés bénéfiques 1. Il s'agit notamment de l'apathogenicity des virus de type sauvage sous-jacents et les méthodologies les plus avancés pour la production de haute-titre, de haute pureté et de qualité clinique des vecteurs recombinants 2. Un autre avantage particulier du système de AAV sur d'autres virus est la disponibilité d'une richesse naturelle de sérotypes qui diffèrent dans les propriétés essentielles mais peuvent tous être facilement conçue en tant que vecteurs en utilisant un protocole commun 1,2. En outre, un certain nombre de groupes, y compris notre propre ont récemment mis au point des stratégies pour utiliser ces virus naturels comme modèles pour la création de vecteurs synthétiques qui combinent des actifs soit des sérotypes d'entrée multiples, ou qui améliorent les propriétés d'un seul isolat. Les technologies respectives pour atteindre ces objectifs are soit la famille réarrangement d'ADN 3, c.-à-la fragmentation de divers gènes de capside AAV suivis par leur ré-assemblage basée sur des homologies partielles (typiquement> 80% pour les sérotypes les plus AAV), ou d'affichage, insertion peptide 4,5 c.-à-habituellement de sept acides aminés dans une boucle exposée de la capside virale, où le peptide idéalement médie re-ciblage à un type cellulaire souhaité. Pour le maximum de succès, les deux méthodes sont appliquées de façon à haut débit dans lequel les protocoles sont en place-mise à l'échelle pour obtenir des bibliothèques de près d'un million distinctes variantes de capside. Chaque clone est alors composé d'une combinaison unique de nombreux virus parentaux (DNA shuffling approche) ou contient un peptide particulier au sein de l'épine dorsale même virale (approche affichage peptide). L'étape finale ultérieure est itératif de sélection d'une telle bibliothèque sur les cellules cibles afin d'enrichir de capsides individuels répondant à la plupart ou idéalement à toutes les exigences du processus de sélection. Ce dernier préférence peigneines pression positive, tels que la croissance sur un type de cellule certain intérêt, avec une sélection négative, pour l'élimination de toutes les capsides exemple réagissant avec des anticorps anti-AAV. Cette combinaison augmente les chances que les capsides synthétiques survivants de la sélection correspondent aux besoins de l'application donnée d'une manière qui n'aurait probablement pas été trouvée dans aucun naturellement AAV isoler. Ici, nous nous concentrons sur la méthode de la famille de réarrangement d'ADN que l'théoriquement et expérimentalement plus difficile des deux technologies. Nous décrire et démontrer toutes les étapes essentielles pour la génération et la sélection des bibliothèques AAV mélangées (Fig. 1), puis de discuter les pièges et les aspects critiques des protocoles que l'on doit être au courant afin de réussir avec AAV l'évolution moléculaire.
Protocole
1. Préparation des jeux de gènes plasmidiques codant AAV capside
- Pour faciliter la préparation de routine de quantités suffisantes des divers AAV capside (cap) des gènes de l'ADN après brassage, d'abord sous-cloner ces gènes dans une infrastructure commune plasmide. Il faut inclure identiques séquences flanquantes de plus de 20 nucléotides de côté utilisation en tant que sites de liaison d'amorces pour PCR imbriquée et pour le clonage (fig. 2).
- En utilisant des amorces appropriées (voir le tableau pour les amorces exemplaires pour AAV5), amplifier par PCR des gènes cap souhaités à partir des plasmides AAV couramment disponibles qui contiennent généralement le gène AAV2 représentant à côté de la gène cap de choix. Parce que le produit de PCR sera utilisée pour le clonage standard, ~ 1 ug de produit purifié est déjà suffisante, et tout protocole PCR conventionnelle peut donc être utilisé.
- Digérer le produit de PCR purifié (par exemple, utilisez un gel d'agarose de purification ou d'un kit de purification PCR standard) etdestinataire plasmide avec des enzymes de restriction dont la reconnaissance des sites sont présents dans les amorces utilisées pour l'amplification de capuchon (1,1) ainsi que dans le plasmide receveur. Dans notre laboratoire, nous utilisons Pac I et Asc les sites I (Fig. 2) car ils sont absents dans la plupart des AAV.
2. DNase basée sur la fragmentation Gene Cap
- Amplifier par PCR des gènes de plafonnement de choix à partir des plasmides générés dans les étapes 1.1-1.3. Une réaction comme décrit ci-dessous donnera ~ 3 pg de produit de PCR. Selon le nombre de gènes de caches pour être inclus dans la bibliothèque, cela suffit pour un maximum de six réactions traînants.
- Pour le PCR, mis en place une réaction de 50 ul contenant 200 ng bouchon plasmide, chaque amorce une concentration de 2 uM final, 10 pl de tampon 5x Hifi et 1 ul polymérase Hifi. Commencez avec 5 min à 95 ° C, puis exécutez 40 cycles de 15 sec à 94 ° C, 30 sec 57 ° C et 3 min 68 ° C, suivie d'une étape finale de 10 min à72 ° C. Purifier les produits de PCR par un gel ou un kit, puis mettre en place un contrôle DNase condensé pour créer des fragments de gènes de plafonnement pour le remontage en chimères.
- Par conséquent, tout aussi mélanger les différents produits de PCR capitalisation d'un montant total de 4 pg dans 54 ul de H 2 O. Ajouter 6 pi de tampon de réaction DNase et 0,5 ul DNase I de la réaction, soigneusement feuilleter trois fois, centrifuger brièvement et immédiatement mis sur un bloc de 25 ° C de chauffage. Incuber entre 1 et 2 min (mis en place plusieurs réactions parallèles et y mettre fin par incréments de 15 sec), puis arrêter la réaction en ajoutant 6 pi 25 mM EDTA et par une brève vortex et incuber 10 min à 75 ° C.
- Purifier les fragments de coiffure sur un gel d'agarose standard de 1%. Idéalement, un frottis doit être visible entre 100 et 500 paires de bases. Depuis la DNase I est une enzyme très puissant, la manutention et le calendrier sont essentiels à cette étape, et de multiples variations dans le temps d'incubation dans l'étape 2.3 pourrait être nécessaire pour res optimalesPHP sous (fig. 3). Purifier l'ADN éluée en utilisant un kit standard et déterminer sa concentration.
3. Famille d'ADN Shuffling
- Tout d'abord, ré-assembler les fragments de plafonnement en pleine longueur des séquences via un PCR dans laquelle ils s'auto-Premier basées sur des homologies partielles. Par conséquent, mettre en place une réaction de 50 ul avec 500 ng de fragments purifiés (étape 2.4), 10 pi de tampon Phusion 10x, 1 pi dNTPs 10 mM, 1,5 ul de DMSO et 0,5 Phusion ul polymérase II. Incuber 30 sec à 98 ° C, puis exécutez 40 cycles de 10 sec 98 ° C, 30 sec 42 ° C et 45 sec 72 ° C, suivie d'une finale de 10 min étape à 72 ° C.
- Dans une seconde qui a suivi PCR, amplifier les gènes capuchon ré-assemblées pour le clonage ultérieur, en utilisant des amorces qui se lient à des séquences conservées flanquantes (fig. 2). Par conséquent, mettre en place une réaction de 50 ul contenant 2 pl de la première PCR (étape 3.1), chaque amorce à une concentration finale de 2 uM, 0,5 ul de MgCl2,10 ul de tampon 5x Hifi et 1 ul polymérase Hifi. Nous vous recommandons d'exécuter 16-24 PCR afin d'assurer des rendements suffisamment élevés pour le clonage ultérieur. Piscine des RAP et purifier la bande bouchon sur toute la longueur (gel ou kit).
- Digérer la piscine bouchon purifié gène avec Pac I et Asc I pour le clonage dans une capable de se répliquer AAV plasmide portant AAV RTI (répétitions terminales inversées; réplication et les signaux d'emballage) ainsi que le gène rep AAV2. Celui-ci devrait être suivie par les mêmes sites pour accueillir la piscine gène cap "dans le cadre" (voir fig. 2 pour les détails). Pour atteindre la digestion complète du produit de PCR, incuber pendant la nuit avec l'enzyme en excès.
- Ligaturer les fragments de capitalisation et de la façon appropriée coupé ITR de AAV / rep backbone à un rapport molaire de 3:1. Faire un 40 ul volume total mastermix (suffisant pour 20 transformations) avec une concentration finale d'ADN de 50 ng / ul et incuber une nuit à 16 ° C.
- Transformez réaction de ligature en mélangeant (sur de la glace) 2 pl avec 30 ul électro-compétentes E. coli (à partir de cellules cultivées commerciales et rendue compétente en utilisant n'importe quel protocole standard). Ajouter dans des cuvettes d'électroporation pré-refroidi (1 écart mm) sur la glace. Électroporer à 1,8 kV, 200 Ω et 25 uF. La constante de temps devrait être proche de 5 ms. Immédiatement ajouter 1 ml préchauffé milieu SOC et le transfert à un flacon de 250 ml. 20 électroporations ces donnera une bibliothèque avec une grande diversité d'environ 1x10 6 clones différents.
- Ajouter 1 volume préchauffé milieu SOC aux transformations mises en commun et agiter à 37 ° C et 180 rpm pendant 1 h. Ensuite, mettre en place le volume total à 800 ml avec du milieu LB plus ampicilline (concentration finale de 50 pg / ml) et incuber pendant une autre h 16 dans les mêmes conditions. Purifier l'ADN plasmidique en utilisant par exemple la bibliothèque. une Qiagen Mega kit de préparation.
Option à l'étape 3.6: Pour calculer la diversité bibliothèque exacte, plaque un aliquOT de la solution 800 ml (avant l'incubation 16 h) sur LB-ampicilline plaques (par exemple, 10 pi sur une plaque de 10 cm) et les colonies du jour suivant. En outre, afin de valider les hautes efficacités traînants, par exemple la séquence 24 clones et de les aligner sur les gènes parentaux capitalisation (voir aussi Fig. 8). Enfin, pour confirmer la vitalité de bibliothèque et une grande diversité fonctionnelle, sous-clone choisi au hasard des gènes cap dans un plasmide helper AAV et les utiliser pour produire et analyser des vecteurs recombinants à petite échelle (fig. 4-5) (voir aussi l'étape 5.4 ci-dessous).
4. La production de la Bibliothèque virale
- Graine 10 15 cm 2 plats de cellules HEK293T (4.5x10 6 cellules / boîte) et 48 h plus tard, avec 220 ug transfecter AAV bibliothèque et 220 ug de plasmide adénoviral (nécessaire pour la propagation AAV). Par conséquent, préchauffer l'Î (polyéthylènimine) et 300 mM de NaCl à 37 ° C. Puis mélanger 7,9 ml de NaCl et de l'ADN, et d'ajouter H 2 O pour un total volume de 15,8 ml. Dans un tube, mélanger 3,52 ml Prince-Édouard, 7,9 ml de NaCl et 4,38 ml H 2 O (tous les volumes pour dix transfections). Combinez les mélanges (vortex) et incuber pendant 10 min à température ambiante, avant de distribuer la solution uniformément dans tous les plats (3 ml par boîte).
- Après 48 h, racler les cellules dans le milieu et les spin down à 1200 tpm pendant 15 min. Remettre en suspension le culot cellulaire dans 6 ml de tampon de lyse (50 mM Tris-HCl pH 8,5, 50 mM NaHCO 3) et sous réserve de 5 cycles gel-dégel (-80/37 ° C). Incuber avec 50 U benzonase par ml pendant 1 h à 37 ° C, avant le filage bas les débris cellulaires à 3750 rpm pendant 20 min.
- Préparer 15%, 25% et 40% (dilutions de 2,5 ul / ml PHENOLRED) à partir d'un iodixanol 60% (OptiPrep dans du PBS-MK) en stock au PBS-MK (1x PBS, MgCl 2 1 mM, 2,5 mM KCl).
- Mettre en place un gradient de purification d'AAV en utilisant une pipette Pasteur pour ajouter 5 ml suspension de virus dans un tube de centrifugeuse Beckman Quick-Seal (14x89 mm), suivie par 1,5 ml each de 15%, 25% et la solution iodixanol 40%. Terminez le dégradé avec un tampon de lyse.
- Ultracentrifugeuse à 50.000 K pendant 2 h à 4 ° C dans un Beckman Ti70.1 rotor. Ensuite, nettoyer l'extérieur du tube avec de l'éthanol 70%, enfoncer une aiguille dans la partie supérieure du tube de ventilation et d'en tirer 1,2 ml de la fraction iodixanol 40% à l'aide d'une aiguille. Prenez soin d'éviter d'attirer de la fraction de 25% car il contient des vides capsides AAV.
5. Présélection et de sélection
- À ce stade, on peut soit itérative amplifier la bibliothèque entière dans des cellules cultivées ou chez les animaux jusqu'à ce que capsides chimériques présentant des propriétés désirées ont émergé (fig. 6-11).
- Pour sélectionner dans des cellules cultivées, co-infecter les aliquotes différentes de la bibliothèque (par exemple, 1, 10 et 100 pi) et l'adénovirus-5 à soutenir la croissance AAV. Test de multiples variations de la bibliothèque et d'un assistant adénoviral est essentiel que l'infectiosité de bibliothèque dans un type cellulaire donné ne peut pas être prédit. Récolter lescellules après ~ 3 jours, extraire amplifié par l'intermédiaire d'AAV congélation-décongélation, inactiver l'adénovirus pendant 30 min à 56 ° C et re-infecter de nouvelles cellules. Répétez jusqu'à 5 fois jusqu'à ce que capsides distinctes s'enrichir (valider par séquençage).
- Pour la sélection des animaux, infecter avec la bibliothèque et d'en extraire le type désiré tissus ou de cellules après ~ 1 semaine. Ne pas co-infecter avec l'adénovirus car cela provoquerait la toxicité chez les animaux indésirables. Sauvetage d'ADN viral par PCR en utilisant les mêmes amorces que précédemment (Fig. 2), re-cloner la piscine casquette, produire une bibliothèque fraîche et répéter jusqu'à ce que capsides distinctes s'enrichir (valider par séquençage).
- Les conditions exactes (volume, le titre, route) pour la sélection in vivo dépendra sur le tissu cible d'intérêt. Pour le foie, les souris sont généralement infectées par le 1x10 11 à 1x10 12 particules virales dans un volume total de 200 pi de PBS par injection veine de la queue (IV). Le facteur limitant est généralement le titre viral de l'originala préparation l, parce que l'injection de 1x10 12 AAV dans 200 pi exige un titre d'au moins 5x10 12 / ml, ce qui n'est pas tous les laboratoires peuvent obtenir régulièrement. Ainsi, alors que un titre maximal est bénéfique pour la première infection (parce que la bibliothèque n'a pas encore été enrichi pour capsides efficaces dans un tissu donné et pourraient donc avoir une infectivité relativement faible dans l'ensemble), nous vous recommandons d'utiliser au moins 1x10 11 particules par souris pour la sélection du foie.
- Si plusieurs souris sont disponibles, il est très utile pour injecter le nombre de particules différentes qui s'apparentent à la sélection dans les cellules cultivées, et d'utiliser plusieurs souris par groupe (et puis de mettre en commun les foies recueillies au sein de chaque groupe) afin de minimiser la variabilité et à accroître la réussite taux, par exemple, 3 souris à 1x10 11 et 3 souris à 1x10 12 particules.
- Depuis AAV est un virus non pathogène et la réplication incompétent (sans helpervirus), il n'ya pas de réactions indésirables à rechercher dans l'absence d'Adenovirus.
- Pour l'euthanasie, les animaux ont été anesthésiés à l'aide un vaporisateur d'isoflurane et ensuite euthanasiés par dislocation cervicale.
- Les tissus (foie, dans ce cas) ont été récoltés après que les animaux ont été euthanasiés confirmé. En général, il n'est pas nécessaire aussi pour d'autres tissus pour perfuser les animaux ou de récolter AAV-infectés organes avant son décès, car l'ADN d'AAV est stable et peut facilement être sauvé à partir de cellules congelées et des tissus.
- Pour étudier capsides simples (à partir de la bibliothèque (étape 3.6) ou après la sélection (étapes 5.2-5.3)), la production de vecteurs AAV codant pour un gène rapporteur (par exemple, gfp 6) en suivant les étapes 4.1 à 4.5. Par conséquent, cloner le gène cap d'intérêt dans un helper norme AAV plasmide 2. A l'étape 4.1, triple-transfecter des cellules avec 14,7 ug chacun de vecteur AAV (codant pour le journaliste), AAV aide et d'assistance adénoviral. Infecter les cellules en culture ou des animaux atteints du virus purifié à des montants différents et déterminationpar exemple, ne efficacité de la transduction par FACS ou la microscopie.
6. Les résultats représentatifs
Utilisation du protocole décrit ici se traduit généralement par des bibliothèques virales avec une diversité d'environ 1x10 6 capsides uniques qui peuvent ensuite être testés pour la plupart des particules individuelles d'affichage ou toutes les propriétés désirées sur une lignée cellulaire donnée ou chez les animaux. Dans ce qui suit, nous allons donner des exemples représentatifs des résultats de telles in vitro ou in vivo projections.
Avant cela, cependant, nous considérons qu'il est important de signaler à nouveau l'utilité de l'analyse des clones individuels de la bibliothèque d'origine plasmidique pour leur fonctionnalité et de la diversité (en option à l'étape 3.6). C'est parce que les deux derniers paramètres sont des conditions préalables maximum de critiques pour le succès de la sélection de la bibliothèque réelle virale qui est fabriqué à partir de la banque de plasmides. Par conséquent, on peut choisir au hasard seul mélangéesgènes de capitalisation et les utilisent pour produire des vecteurs recombinants AAV (extraits bruts ou des particules purifiées, selon le degré de précision souhaité) exprimant un gène rapporteur facilement détectable et quantifiable. Un essai typique pour comparer les différentes variantes de capside est alors microscopie à fluorescence, comme indiqué dans l'exemple représentatif à la Fig. 4.
Une autre méthode consiste FACS basée sur la mesure de expression du gène rapporteur fluorescent qui détient les avantages supplémentaires qu'elle permet également pour la détermination de l'expression des gènes par cellule, plus que cela fonctionne pour les cellules en suspension. Fig. La figure 5 montre un résultat typique d'une telle analyse FACS à base de pétrole brut lysats vecteurs AAV dans divers types cellulaires.
Parce que la sélection ci-dessus décrit des chimères capitalisation individuels est aléatoire et restreint, il n'est bien sûr pas utile comme une approche réelle pour l'enrichissement des candidats désirés. Au lieu de cela, la sélection de la vibibliothèque ral dans les cellules ou tissus cibles chez les animaux est plus approprié. Comme les conditions et les paramètres varient avec chaque application, nous allons seulement mettre en évidence quelques lignes directrices plus représentatives et les résultats.
Le moyen le plus la sélection est itératif d'amplification bibliothèque sur des lignées cellulaires en culture ou des cellules primaires (étape 5.2). Depuis AAV nécessite Adenovirus la co-infection pour sa propagation, les cellules cibles doit être susceptible d'adénovirus. On peut alors se développer et d'infecter les par exemple dans des plaques de 6 puits qui contiennent un nombre de cellules suffisant par puits pour assurer une couverture complète de la bibliothèque. Une deuxième notion importante est que l'équilibre de l'AAV et l'adénovirus est délicat, trop AAV va inhiber l'adénovirus, alors qu'un excès de celle-ci va tuer les cellules (à la différence AAV, adénovirus provoque des infections lytiques) avant que les AAV pourrait répliquer.
Toutefois, étant donné que l'infectiosité bibliothèque sur un type de cellule certaine est inconnue, trouver "bonne" AAV: ratios adénovirus auqui peut se propager à la fois exige parallèlement des tests de différentes combinaisons de doses de la bibliothèque et l'assistance adénoviral (par exemple, 10:1, 1:1 et 1:10). Une bonne mesure de l'infection adénovirus puissant est l'apparition d'effets cytopathiques trois jours après l'inoculation du virus, comme en témoigne l'arrondissement des cellules et à détacher comme on le voit dans la figure. Vice-versa, un 6. Utiles lecture de l'infection AAV et l'amplification est la détection des protéines de la capside par Western blot, en utilisant l'anticorps B1 qui reconnaît une capside AAV hautement conservé épitope (Fig. 7) 7.
Une décision importante qui est alors extrait brut AAV à choisir pour ré-infection ultérieure des cellules fraîches (étape 5.2). Idéalement, on prendra ceux où les bandes sont moins AAV capside notable (Fig. 7), car ceux-ci suggèrent une étroite liaison génotype-phénotype. Ce dernier décrit la situation dans laquelle un génome codant pour un variant certaine capside esteffectivement encapsidé dans la capside correspondante. Pour atteindre et maintenir un virage serré génotype-phénotype de liaison est la clé de la sélection réussie de candidats individuels virales comme il à son tour veille à ce que capsides ayant des propriétés souhaitées livrer le modèle apparenté génétique dans les cellules au cours des cycles d'infection successifs. Par conséquent, nous recommandons de choisir les extraits bruts avec une expression modérée de capside ré-infection et d'utiliser des quantités minimes. Ensemble, ces deux mesures permettront d'éviter la surcharge des cellules nouvellement infectées avec différents capside / génome combinaisons qui pourraient autrement perturber la liaison génotype-phénotype une fois que les virus réinfectés commencer la réplication et de ré-emballage de leurs génomes dans la non-apparentés capsides.
En outre, il est essentiel de surveiller la diversité bibliothèque au cours des cycles d'infections à répétition par séquençage de l'ADN. Idéalement, on remarquera des changements dans la composition de clones individuels indicatifs de sélection avec succès, à savoir accumlation de fragments distincts sérotype et les pertes des autres, comme en témoigne la figure. 8. Dans le cas idéal, seuls quelques-uns ou même un seul clone sera finalement détecté qui peut ensuite être analysé comme décrit ci-dessus. Si, cependant, aucun changement n'est observé après quatre ou cinq passages, il faut soit augmenter la pression de sélection (voir Discussion) ou considérer que la lignée cellulaire utilisée peut être inapproprié, car il peut être trop sensibles aux sérotypes un trop grand nombre.
Pour l'amplification de bibliothèque in vivo (étape 5.3), les mêmes règles et les considérations s'appliquent, avec deux différences essentielles: d'abord, on fait pas des clones choisis au hasard dans l'écran des animaux en raison des coûts associés et les considérations éthiques. Deuxièmement, on ne va pas co-infecter avec adénovirus auxiliaire car elle provoque des effets toxiques ou mortels chez les animaux, ainsi que le tropisme adénoviral à limiter la sélection aux cellules sensibles au virus helper. Au lieu de cela, la bibliothèque AAV est infusée dans ee, les animaux secourus à partir des cellules / tissus cibles par PCR (Fig. 9), puis re-cloné et ré-emballé pour un tour de nouvelles infections. Comme avec la sélection dans la culture, ce processus est répété jusqu'à ce que les candidats individuels ont émergé.
Quelle que soit la procédure de sélection, la dernière étape est la validation des variantes de capside enrichis dans les systèmes appropriés. Figures. 10 et 11 montrent des exemples représentatifs de notre sélection précédente propres chimères AAV qui effectuent exceptionnellement bien dans la culture de tissus ou dans le foie de souris. Comme on le voit dans la figure. 10, un clone particulier (AAV-DJ 3) surpasse en effet une collection de huit wildtypes AAV naturelles dans un large éventail de lignées cellulaires. Enfin, un autre clone sélectionné dans lignées d'hépatomes cultivées présente un tropisme plus pour le foie de souris et en conséquence moins hors de ciblage quand périphérie infusé dans des souris adultes que le puissant vecteur de contrôle AAV8 testé en com directeparaison (fig. 11). Notez que bien d'être plus précis pour le foie, le clone AAV-DN transduit effectivement cet organe un peu moins efficace que AAV8. À cet égard, l'AAV-DN est un exemple représentatif bon pour le résultat de l'évolution et la sélection AAV où les candidats finaux présentent généralement un certain nombre de propriétés désirées, mais ne sont pas nécessairement parfaite dans tous les aspects.
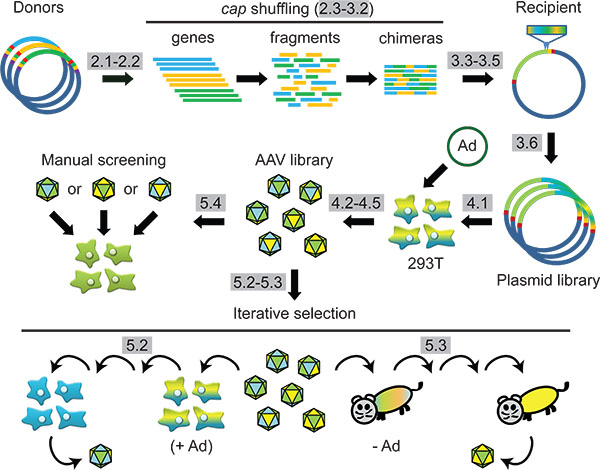
Figure 1 Schéma:. Synthétique AAV capside d'ingénierie via réarrangement d'ADN de la famille et la sélection subséquente dans les cellules ou chez l'animal. Étapes du protocole sont surlignées en gris.
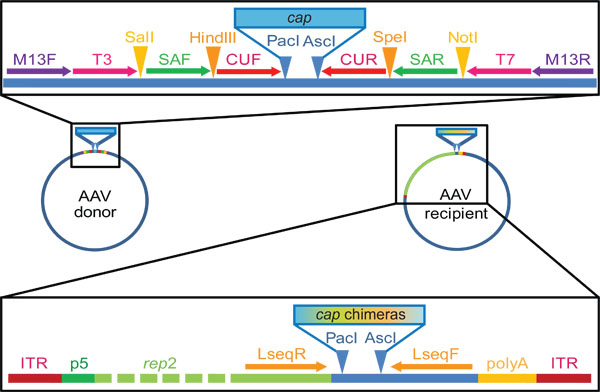
Figure 2. Donateurs AAV gène cap et plasmides utilisés bénéficiaires dans notre laboratoire pour la famille réarrangement d'ADN et la génération de la bibliothèque. Notez que ce sont uniquement des exemples représentatifs, et que les emplacements exacts et les séquencespeuvent être personnalisés. Le plasmide donneur base est dérivé du disponible dans le commerce KS pBlueScript II (+) vecteur qui nous conçu pour contenir la liaison d'amorce représenté, ainsi que des sites de restriction, représenté par des flèches ou des triangles, respectivement. Nous avons ensuite cloné les gènes cap des sérotypes d'AAV 1-9 (amplifié avec des amorces CAPF / R) dans ce plasmide, pour devenir flanqué de Pac I et les sites de restriction Asc I. Amorces T3 et T7 sont utilisées pour l'isolement bouchon à l'étape 2.2, tandis que l'amplification ultérieure de re-assemblés séquences chimériques (étape 3.2) peut être accompli en utilisant des paires d'amorces soit SAF / R ou CUF / R, ou les deux dans une PCR nichée. Le destinataire capable de se répliquer plasmide porte répétitions terminales inversées (AAV RTI, la réplication et les signaux d'emballage) flanquant le gène rep AAV2 sous le contrôle du promoteur p5 AAV. Pac I et les sites de restriction Asc I en aval de permettre représentant "dans la trame" clonage de la piscine de gènes mélangées capitalisation. Laindiquées sites d'amorce LSEQ contraignantes sont utiles pour le séquençage des gènes bouchon (étape 3.6).

Figure 3. Exemple de DNase I condensé de gènes d'assemblage (AAV2, 8 et 9). Montré est une analyse électrophorèse sur gel d'agarose des produits de gène cap digère en utilisant les temps indiqués incubation différentes (en minutes: secondes). Lane U montre la piscine d'entrée non digérés bouchon fragment de contrôle. Tailles de bandes de marqueur d'ADN dans les voies M sont en kilobases. Dans cet exemple, idéal digère ont été obtenus avec des temps d'incubation de 1h45 ou 2:00 min, ce qui a donné le pic préféré prédominante autour de 100 à 500 paires de bases (boîte jaune).

Figure 4. Exemple pour la microscopie basée sur l'analyse de trois lignées de cellules humaines infectées par cinq différents recombinant exprimant YFP avez AAVctors (noms sur le dessus) à base de gènes capuchon mélangées de façon aléatoire choisi parmi une bibliothèque d'origine sur la base de AAV2, 8 et 9.

Figure 5. Exemple pour FACS-analyse basée sur des quatre types de cellules infectées (à série de dix dilutions) avec 18 différents vecteurs recombinants AAV YFP-exprimant (noms sur le dessus, y compris ceux de la Fig. 4) faite avec mélangées gènes capitalisation choisies au hasard à partir d'une bibliothèque d'origine (AAV2, 8 et 9). L'expression a été YFP précodés facile visualisation. Pourcentages représentés sont des cellules transduites, pour qui le noir indique toujours 0% et blanc le plus grand nombre mesuré dans chaque type de cellule. B2 Clone (rouge) est un exemple d'un clone avec une efficacité globale des pauvres, comme on peut s'y attendre de capsides non sélectionnés. Notez que l'analyse FACS est plus sensible et peut également être utilisé pour les cellules en suspension (comme SupT1) qui sont moins susceptibles à la microscopie. hu, Humaine; mu, murin.
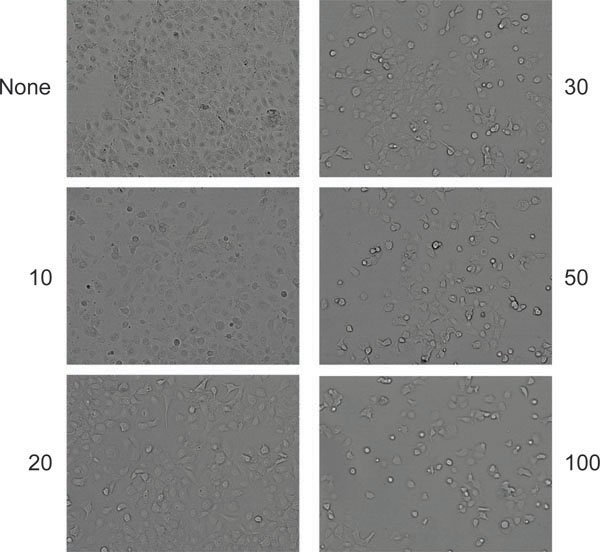
Figure 6. Aspect typique d'effets cytopathiques dans les cellules (cellules HeLa dans ce cas) après productive co-infection par l'AAV et l'adénovirus. Les cellules ont été soit non infecté gauche (en haut à gauche) ou infectés par des montants indiqués (particules par cellule) de l'adénovirus.

Figure 7. Détection d'expression protéine de capside d'AAV par Western blot en tant que mesure de l'infection bibliothèque et d'amplification. Le blot gauche montre des cellules co-infectées avec différents volumes (en ul) d'une bibliothèque AAV et l'adénovirus helper. Les deux premières voies sont de bons exemples de coopérationnditions cédant à peine détectable expression de la protéine d'AAV, indiquant suffisante mais non excessive infection AAV et d'amplification et par conséquent la désirée étanche génotype-phénotype liaison. Ainsi, pl 0,1, 1 ou 10 de ces surnageants ont été utilisés pour ré-infection de cellules fraîches (blot à droite). Les cellules de la voie C ont été infectées par AAV seul comme un contrôle négatif (pas d'expression détectable en raison de l'absence de l'helpervirus).
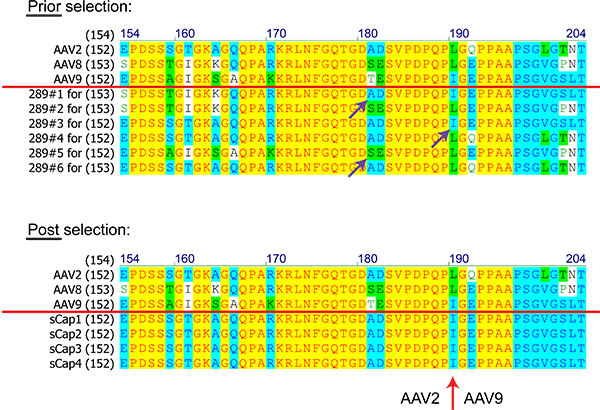
Comparaisons Figure 8. De séquences de protéines (numéros sont des acides aminés) de clones d'une bibliothèque d'AAV sur la base de AAV2, 8 et 9 avant et après la sélection. Séquences ci-dessus de la ligne rouge montre les AAV parentales. Flèches pourpres indiquent événements de recombinaison homologue. La flèche rouge indique un cross-over entre les AAV2 et AAV9 noté dans tous les clones sélectionnés.
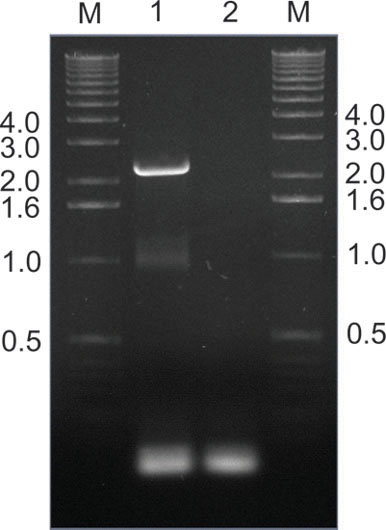
Figure 9. </ Sauvetage strong> de succès infectés clones AAV à partir de tissus de souris par PCR. Dans cet exemple, les gènes AAV capitalisation étaient amplifié par PCR à partir de foies de souris extrait d'une semaine après perfusion périphérique bibliothèque, à l'aide paire d'amorces SAF / R (Fig. 2). La ligne 1 montre la bande 2,2 kilobases attendu, tandis que la piste 2 est un contrôle non-modèle. À la suite Pac I et Asc I restriction, les fragments amplifiés sont re-cloné dans le plasmide destinataire initial (fig. 2) pour la production subséquente et ré-perfusion d'une bibliothèque secondaire. Tailles de bandes de marqueur d'ADN dans M voies sont indiqués en kilobases.

Figure 10. Exemple pour une performance supérieure d'un AAV enrichi chimère dans les cellules cultivées. Clone AAV-DJ a été rapporté par nous avant le 3, il représente surtout un hybride entre les sérotypes d'AAV 2, 8 et 9, et a été sélectionné à partir d'une biblio y contenant ces trois plus cinq sérotypes supplémentaires dans les cellules hépatiques humaines en présence d'antisérums humains mis en commun. Pour comparer son efficacité à huit sérotypes d'AAV naturelles (indiqué par 1-6, 8 et 9 sur le dessus), tous les gènes capitalisation ont été utilisés pour produire des purifié auto-complémentaires GFP-exprimant les vecteurs 8,19. Elles ont été normalisées pour contenir 2x10 9 génomes vecteurs par ml, puis utilisés pour transduire des lignées cellulaires indiquées à dix-dilutions. Trois jours plus tard, Gfp cellules exprimant ont été comptés et titres infectieux ont été déterminés en prenant en compte le facteur de dilution. En revanche le code dans la figure. 5, les couleurs plus foncées indiquent les plus élevés infectivités par le nombre de particules. Comme on le voit, l'sélectionnée AAV-DJ chimère surpasse tous les wildtypes AAV naturelles, ce qui illustre le succès de la méthode de sélection appliqués. Fibr, les fibroblastes; ha, le hamster; Hu, de l'homme; mu, murins, Si, simiens.

Figure 11. Exemple pour l'analyse d'un AAV sélectionné chimère dans le foie de souris. Clone AAV-DN a été sélectionné sur des cellules d'hépatome murin et ensuite utilisé pour produire des vecteurs recombinants exprimant la luciférase-8. Montré sont représentatives des souris (trois par groupe) une semaine après la perfusion périphérique de doses égales de ce vecteur ou un contrôle basé sur type sauvage AAV8, l'un des plus puissants connus des isolats naturels de 9 foie de souris. Notez que bien que le clone AAV-DN donne une expression un peu moins globale dans le foie (le panneau (I)), il est plus spécifique pour cet organe, car il présente beaucoup moins hors de ciblage dans les tissus hépatiques non une fois que les niveaux d'expression du foie ont été ajustée par l'intermédiaire du logiciel d'imagerie (panneau (II)).
Discussion
Ici, nous avons esquissé essentiels étapes expérimentales et les lignes directrices pour AAV capside d'ingénierie via la famille réarrangement d'ADN et de l'évolution dans les cellules ou chez l'animal. En substance, ces protocoles sont des versions normalisées des procédures nous avons d'abord signalés dans le domaine de l'AAV en 2008 3. Alors qu'une vague de suivi des études par d'autres ont rapporté de nombreux ex modifications, 10-13, nos vers...
Déclarations de divulgation
Tous les auteurs déclarent qu'ils n'ont rien à révéler.
Remerciements
Les auteurs tiennent à remercier le soutien exceptionnel de leur laboratoire, membres de l'équipe et le travail par le Pôle d'excellence de CellNetworks à l'Université de Heidelberg ainsi que par le Chica et Heinz Schaller (CHS) de la Fondation. Nous apprécions que l'évolution moléculaire AAV par l'ADN de la famille brassage est devenue un domaine très actif depuis notre publication initiale il ya trois ans et, par conséquent présenter des excuses à tous les auteurs de publications pertinentes, dont le travail ne pouvait pas être cité ici en raison de contraintes d'espace.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Entreprise | Numéro de catalogue | |
| DNase I | Invitrogen | 18068-015 | |
| Polyéthylènimine (PEI) | Sigma-Aldrich | 408727 | |
| Les enzymes de restriction | ONÉ | Divers | |
| ADN ligase T4 | ONÉ | M0202T | |
| Kit d'extraction de gel | Qiagen | 28704 | |
| Kit Phusion polymérase II | Finnzymes (ONÉ) | F-540S | |
| Kit Hotstar polymérase Hifi | Qiagen | 202602 | |
| DMSO | Finnzymes (ONÉ) | F-540S (partie de la trousse) | |
| L'EDTA (25 mM) | Invitrogen | 18068-015 (partie de la trousse) | |
| Tris | Roth | 4855,2 | |
| Sel de sodium ampicilline | Roth | K029.2 | |
| dNTP (10 mM, 100 pl) | Invitrogen | 18427013 | |
| Iodixanol (OptiPrep) | Axis-Shield | 1114739 | |
| PHENOLRED | Merck | 107241 | |
| Plasmide kit de préparation méga | Qiagen | 12181 | |
| Ultracentrifugeuse | Beckman-Coulter | Optima L90K | |
| Tubes à centrifuger Quick-Seal | Beckman-Coulter | 342414 | |
| Unité d'électroporation | Bio-Rad | GenePulserXcell | |
| Thermocycleur | Eppendorf | Vapo Protect | |
| Bloc chauffage | BIOER | Mo-102 | |
| Microscope à fluorescence | Olympe | IX81 | |
| Analyseur FACS | Beckman-Coulter | Cytomics FC500 MLP | |
| MEGAX DH10B T1R cellules | Invitrogen | C640003 | |
| Benzonase | Merck | 101695 | |
| Adenovirus-5 | ATCC | VR-5 | |
| pBluescript II KS (+) plasmide | Stratagene | 212207 | |
| cap5F (Pac I du site en jaune, cap5 séquences spécifiques en gras): GACTCTTAATTAACAGGT ATGTCTTTTGTTGATCACCCTCC | IDTDNA | Amorce personnalisé | |
| cap5R (Asc I du site en vert, cap5 séquences spécifiques en gras): GTGAGGGCGCGCC TTAAAGGGGTCGGGTAAGGTATC | IDTDNA | Amorce personnalisé |
Références
- Grimm, D., Kay, M. A. From virus evolution to vector revolution: use of naturally occurring serotypes of adeno-associated virus (AAV) as novel vectors for human gene therapy. Curr. Gene Ther. 3, 281-304 (2003).
- Grimm, D. Production methods for gene transfer vectors based on adeno-associated virus serotypes. Methods. 28, 146-157 (2002).
- Grimm, D. In vitro and in vivo gene therapy vector evolution via multispecies interbreeding and retargeting of adeno-associated viruses. J. Virol. 82, 5887-5911 (2008).
- Muller, O. J. Random peptide libraries displayed on adeno-associated virus to select for targeted gene therapy vectors. Nat. Biotechnol. 21, 1040-1046 (2003).
- Perabo, L. In vitro selection of viral vectors with modified tropism: the adeno-associated virus display. Mol. Ther. 8, 151-157 (2003).
- Zolotukhin, S., Potter, M., Hauswirth, W. W., Guy, J., Muzyczka, N. A "humanized" green fluorescent protein cDNA adapted for high-level expression in mammalian cells. J. Virol. 70, 4646-4654 (1996).
- Wobus, C. E. Monoclonal antibodies against the adeno-associated virus type 2 (AAV-2) capsid: epitope mapping and identification of capsid domains involved in AAV-2-cell interaction and neutralization of AAV-2 infection. J. Virol. 74, 9281-9293 (2000).
- Grimm, D. Fatality in mice due to oversaturation of cellular microRNA/short hairpin RNA pathways. Nature. 441, 537-541 (2006).
- Nakai, H. Unrestricted hepatocyte transduction with adeno-associated virus serotype 8 vectors in mice. J. Virol. 79, 214-224 (2005).
- Koerber, J. T., Jang, J. H., Schaffer, D. V. DNA shuffling of adeno-associated virus yields functionally diverse viral progeny. Mol. Ther. 16, 1703-1709 (2008).
- Li, W. Engineering and selection of shuffled AAV genomes: a new strategy for producing targeted biological nanoparticles. Mol. Ther. 16, 1252-1260 (2008).
- Ward, P., Walsh, C. E. Chimeric AAV Cap sequences alter gene transduction. Virology. 386, 237-248 (2009).
- Yang, L. A myocardium tropic adeno-associated virus (AAV) evolved by DNA shuffling and in vivo selection. Proc. Natl. Acad. Sci. U.S.A. 106, 3946-3951 (2009).
- Perabo, L. Combinatorial engineering of a gene therapy vector: directed evolution of adeno-associated virus. J. Gene. Med. 8, 155-162 (2006).
- Maheshri, N., Koerber, J. T., Kaspar, B. K., Schaffer, D. V. Directed evolution of adeno-associated virus yields enhanced gene delivery vectors. Nat. Biotechnol. 24, 198-204 (2006).
- Wu, Z., Asokan, A., Samulski, R. J. Adeno-associated virus serotypes: vector toolkit for human gene therapy. Mol. Ther. 14, 316-327 (2006).
- Kwon, I., Schaffer, D. V. Designer gene delivery vectors: molecular engineering and evolution of adeno-associated viral vectors for enhanced gene transfer. Pharm. Res. 25, 489-499 (2008).
- Perabo, L., Huber, A., Marsch, S., Hallek, M., Buning, H. Artificial evolution with adeno-associated viral libraries. Comb. Chem. High. Throughput. Screen. 11, 118-126 (2008).
- McCarty, D. M., Monahan, P. E., Samulski, R. J. Self-complementary recombinant adeno-associated virus (scAAV) vectors promote efficient transduction independently of DNA synthesis. Gene Ther. 8, 1248-1254 (2001).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon