É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Engenharia e Evolução da Synthetic vírus adeno-associado (AAV) Gene vetores de terapia através de DNA Família Shuffling
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Nós demonstramos a base técnica para projetar e evoluir molecularmente sintéticos virais adeno-associados (AAV) vetores de terapia genética através de DNA da família baralhar. Além disso, fornecer orientações gerais e exemplos representativos para a selecção ea análise de individuais cápsides quiméricos com propriedades melhoradas sobre células alvo em cultura ou em ratinhos.
Resumo
Virais adeno-associados (AAV) vectores representam alguns dos veículos mais potentes e promissora para a transferência de gene terapêutico humano devido a uma combinação única de propriedades benéficas 1. Estes incluem o apathogenicity dos vírus tipo selvagem subjacentes e as metodologias altamente avançadas para a produção de título elevado, de elevada pureza e grau clínico vectores recombinantes 2. Uma outra vantagem particular do sistema de AAV sobre outros vírus é a disponibilidade de uma variedade de ocorrência natural serotipos que diferem em propriedades essenciais ainda podem ser facilmente concebido como vectores utilizando um protocolo comum 1,2. Além disso, um número de grupos incluindo o nosso próprio recentemente desenvolvido estratégias para usar estes vírus naturais como modelos para a criação de vectores sintéticos que ou se combinam os activos de serotipos de entrada múltiplas, ou que melhoram as propriedades de um único isolado. As respectivas tecnologias para alcançar essas metas are ambas as famílias de DNA baralhar 3, isto é, a fragmentação de vários genes de AAV cápside, seguida da sua re-montagem com base em homologias parciais (tipicamente> 80% para a maioria dos serotipos de AAV), ou de exibição de péptidos 4,5, inserção ou seja, de geralmente sete aminoácidos na um circuito exposta da cápside viral em que o peptídeo idealmente medeia re-direccionamento para um tipo de célula desejado. Para o máximo sucesso, ambos os métodos são aplicados de uma forma de alto rendimento em que os protocolos são-up escalado para produzir bibliotecas de cerca de um milhão distintas variantes do capsídeo. Cada clone é então constituído por uma combinação única de numerosos vírus parentais (ADN abordagem baralhar) é ou contém um péptido distintivo dentro da espinha dorsal mesmo viral (abordagem de exibição de péptidos). O passo final subsequente é iterativo selecção de uma biblioteca em células alvo, a fim de enriquecer para cápsides individuais que preenchem mais ou, idealmente, todos os requisitos do processo de selecção. O último de preferência penteines pressão positiva, como o crescimento de um tipo de célula certa de interesse, com seleção negativa, para a eliminação exemplo de todos os capsídeos reagem com anticorpos anti-AAV. Esta combinação aumenta a probabilidade de que cápsides sintéticos sobreviventes a selecção correspondem às necessidades da aplicação dada de uma forma que, provavelmente, não têm sido encontrados em qualquer AAV que ocorre naturalmente isolar. Aqui, vamos nos concentrar no método DNA família baralhar como teórica e experimentalmente mais desafiador das duas tecnologias. Descrevemos e demonstrar todas as etapas essenciais para a geração e seleção de bibliotecas embaralhadas AAV (Fig. 1), e depois discutir as armadilhas e os aspectos críticos dos protocolos de que é preciso estar ciente, a fim de ter sucesso com a evolução molecular de AAV.
Protocolo
1. Preparação de conjuntos de genes que codificam Plasmideo AAV capsídeo
- Para facilitar a preparação de rotina de quantidades suficientes dos vários AAV da cápside (cap) genes para subsequente DNA shuffling, inicialmente subclone estes genes em comum uma espinha dorsal de plasmídeo. É importante para incluir idênticas sequências flanqueadoras de mais de 20 nucleótidos para uma utilização posterior como sítios de ligação do primer para a PCR aninhada e para a clonagem (Fig. 2).
- Utilizando iniciadores apropriados (ver Tabela para primers exemplares para AAV5), PCR amplificar os genes desejados tampão de plasmídeos de AAV vulgarmente disponíveis que geralmente contêm o gene rep AAV2 ao lado do gene cap de escolha. Porque o produto de PCR serão utilizados para a clonagem padrão, ~ ug 1 de produto purificado já é suficiente, e qualquer protocolo de PCR convencional pode, portanto, ser utilizado.
- Digerir o produto de PCR purificado (por exemplo, usar agarose purificação em gel ou um kit de purificação PCR padrão) edestinatário plasmídeo com enzimas de restrição cujas reconhecimento sítios estão presentes nos iniciadores utilizados para amplificação tampa (1,1), bem como no plasmídeo receptor. No nosso laboratório, usamos Pac I e sítios Asc I (Fig. 2), como eles estão ausentes na maioria dos AAV.
2. DNase baseado Fragmentação gene cap
- PCR amplificar genes tampão de escolha a partir dos plasmídeos gerados nos passos 1,1-1,3. Uma reacção conforme descrito abaixo irá produzir ~ 3 ug de produto de PCR. Dependendo do número de genes tampão para ser incluído na biblioteca, isso é suficiente para até seis reacções de baralhar.
- Para a PCR, configurar uma reacção de 50 l contendo 200 ng tampa plasmídeo, cada primer, 2 mM concentração final, 10 ul de tampão Hifi 5x e 1 polimerase Hifi ul. Começar com 5 min a 95 ° C e em seguida 40 ciclos de correr 15 seg 94 ° C, 30 seg 57 ° C e 3 min 68 ° C, seguido por uma etapa final de 10 min em72 ° C. Purificar os produtos da PCR através de gel ou kit e, em seguida, criar um ambiente controlado DNase digest para criar fragmentos de genes tampão para a montagem em quimeras.
- Portanto, igualmente misturar os vários produtos da PAC de PCR para um montante total de 4 g em 54 ul de H 2 O. Adicionar 6 tampão de reação ul DNase e 0,5 ul DNase I da reação, cuidadosamente agite por três vezes, girar rapidamente e imediatamente colocado em um bloco de aquecimento 25 ° C. Incubar entre 1 e 2 min (configurar várias reacções paralelas e encerrar-los em incrementos de 15 seg), em seguida, parar a reacção por adição de 6 uL de 25 mM de EDTA e por uma breve vórtice e incubando 10 min a 75 ° C.
- Purifica-se os fragmentos tampão sobre um padrão de gel de agarose a 1%. Idealmente, um esfregaço deve ser visível entre 100 e 500 pares de bases. Uma vez que a DNase I é uma enzima altamente potente, manuseamento e de temporização são críticos neste passo, e diversas variações no tempo de incubação na etapa 2,3 possa ser necessária para res óptimasÜLTS (Fig. 3). Purifica-se o DNA eluído utilizando um kit padrão e determinar a sua concentração.
3. Família DNA Shuffling
- Em primeiro lugar, voltar a montar os fragmentos da tampa em full-length seqüências através de um PCR em que eles se auto-prime com base em homologias parciais. Portanto, defina uma reação 50 ul com 500 ng fragmentos purificados (passo 2.4), 10 de buffer Phusion ul 10x, 1 ml de 10 mM de dNTPs, 1,5 ul DMSO e 0,5 ul Phusion polimerase II. Incubar 30 seg a 98 ° C e em seguida 40 ciclos de correr 10 seg 98 ° C, 30 seg 42 ° C e 45 seg 72 ° C, seguido por uma etapa final de 10 min a 72 ° C.
- Em um segundo PCR subsequente, amplificar os genes re-montado tampão para a clonagem subsequente, utilizando iniciadores que se ligam às sequências conservadas que flanqueiam (Fig. 2). Portanto, configurar uma reacção de 50 uL contendo 2 ul da primeira PCR (passo 3.1), cada iniciador a uma concentração final de 2 mM, 0,5 ul de MgCl2,10 ul de tampão Hifi 5x e 1 polimerase Hifi uL. Nós recomendamos a execução de 16-24 PCRs para garantir rendimentos suficientemente elevados para clonagem posterior. Piscina os PCRs e purificar a banda cap full-length (gel ou kit).
- Digerir o conjunto de genes purificados tampão com Pac I e I ASC para clonagem num competente para replicação de AAV plasmídeo portador de AAV ITR (repetições terminais invertidas; de replicação e sinais de empacotamento), bem como o gene rep AAV2. Este último deve ser seguido pelos mesmos locais para acomodar o conjunto de genes cap "no frame" (ver Fig. 2. Para detalhes). Para alcançar a digestão completa do produto da PCR, incubar durante a noite com enzima em excesso.
- Ligadura os fragmentos do tampão e do apropriadamente cortado AAV ITR backbone / rep a uma razão molar de 3:1. Fazer uma 40 ul volume total mastermix (suficiente para 20 transformações) com uma concentração de DNA final de 50 C. ng / uL e incubar durante a noite a 16 °
- Transformar reacção de ligação por mistura (em gelo) 2 ul com 30 uL electro-competente E. coli (cultivado a partir de células comerciais e fez competente, utilizando qualquer protocolo padrão). Adicionar em cuvetes de pré-arrefecidos electroporação (1 lacuna mm) em gelo. Electroporate em 1,8 kV, 200 Ω e 25 uF. A constante de tempo deve ser perto de 5 ms. Imediatamente adicionar 1 ml de pré-aquecida meio SOC e transferência para um frasco de 250 ml. 20 electroporações tais irá produzir uma biblioteca com uma diversidade de cerca de 1x10 6 clones diferentes.
- Adicionar 1 volume pré-aquecido médio SOC para as transformações reunidas e agitar a 37 ° C e 180 rpm durante 1 h. Em seguida, trazer o volume total até 800 ml com meio LB mais ampicilina (concentração final de 50 ug / ml) e incubar durante 16 h outro sob as mesmas condições. Purifica-se o ADN da biblioteca de plasmídeo usando, por exemplo. um mega kit Qiagen prep.
Opcional no passo 3.6: Para calcular a diversidade biblioteca exato, uma placa aliquot da solução de 800 ml (antes da incubação de 16 h) em placas de ampicilina-LB (por exemplo, 10 uL sobre uma placa de 10 cm) e colónias de contagem no dia seguinte. Além disso, para validar altas eficiências baralhar, por exemplo, sequência de 24 clones e alinhar-los para os genes tampão parental (ver também a fig. 8). Finalmente, para confirmar a vitalidade biblioteca e diversidade funcional alta, subclone escolhidos aleatoriamente genes tampa em um plasmídeo ajudante de AAV e usá-los para produzir e analisar vectores recombinantes em pequena escala (Figs. 4-5) (ver também o passo 5.4).
4. Produção de Biblioteca Viral
- Semente de 10 15 cm 2 pratos de células HEK293T (4.5x10 6 células / placa) e 48 horas depois transfectar com 220 mg AAV biblioteca e 220 mcg plasmídeo adenoviral (necessário para a propagação AAV). Assim, o pré-aquecimento de PEI (polietilenimina) e 300 mM de NaCl a 37 ° C. Em seguida, misturar 7,9 ml de NaCl e de DNA, e adicionar H2O para um total de volume de 15,8 ml. Num tubo separado, misturar 3,52 ml PEI, 7,9 ml de NaCl e 4,38 ml de H2O (todos os volumes para dez transfecções). Combinar as misturas (vórtex) e incubar durante 10 min à temperatura ambiente, antes de distribuir a solução uniformemente entre os pratos (3 ml por placa).
- Após 48 h, raspar as células em meio e colocá-los para baixo a 1200 rpm durante 15 min. Ressuspender o sedimento de células em 6 ml de tampão de lise (50 mM Tris-HCl pH 8,5, 50 mM de NaHCO3) e sujeito a 5 ciclos de congelamento-descongelamento (-80/37 ° C). Incubar com 50 U por ml benzonase durante 1 h, a 37 ° C, antes de girar para baixo detritos de células em 3750 rpm durante 20 min.
- Preparar 15%, 25% e 40% (diluições com 2,5 ul / phenolred ml) a partir de uma iodixanol 60% (OptiPrep em PBS-MK) estoque em PBS-MK (1x PBS, 1 mM de MgCl2, 2,5 mM de KCl).
- Configurar um gradiente para a purificação de AAV utilizando uma pipeta de Pasteur de adicionar 5 ml de suspensão de vírus para um tubo de centrífuga Beckman Quick-Seal (14x89 mm), seguido por 1,5 ml each de 15%, 25% e solução iodixanol 40%. Cubra fora do gradiente com tampão de lise.
- Ultracentrífuga em 50,000 K durante 2 h, a 4 ° C em um Ti70.1 rotor Beckman. Em seguida, limpar o exterior do tubo com etanol a 70%, enfiar uma agulha para o topo do tubo de ventilação e desenhar 1,2 ml da fracção de iodixanol 40%, usando uma agulha. Tome cuidado para evitar o desenho a partir da fração de 25%, já que contém capsídeos vazios de AAV.
5. Triagem e Seleção
- Neste ponto, pode-se quer iterativamente amplificar a biblioteca inteira em células em cultura ou em animais, até cápsides quiméricos que exibem propriedades desejadas surgiram (Figs. 6-11).
- Para seleccionar em células cultivadas, co-infectar várias alíquotas da biblioteca (por exemplo, 1, 10 e 100 uL) e adenovírus-5 para suportar o crescimento de AAV. Testando diversas variações de biblioteca e auxiliar adenoviral é essencial como infecciosidade biblioteca em um dado tipo celular não pode ser prevista. Colher océlulas após ~ 3 dias, extrair AAV amplificado através de congelamento-descongelamento, inactivar Adenovírus durante 30 min a 56 ° C e re-infectar novas células. Repita até 5 vezes até capsídeos distintos tornam-se enriquecido (validar por seqüenciamento).
- Para a selecção em animais, com a biblioteca de infectar e extrair o desejado tipo de tecido ou célula após ~ semana 1. Não co-infectar com o adenovírus como isso irá causar toxicidade adverso nos animais. Resgatar DNA viral através de PCR utilizando os mesmos iniciadores, como anteriormente (Fig. 2), re-clonar o pool de tampão, produzir uma biblioteca de fresco e repetir até cápsides distintas enriquecidos (validar por sequenciação).
- As condições exactas (volume, título, rota) para a selecção in vivo irá depender do tecido alvo de interesse. Para fígado, os ratinhos são tipicamente infectadas com 1x10 a 1x10 11 12 partículas virais em um volume total de 200 ul de PBS através da cauda injecção na veia (IV). O factor limitante geralmente é o título virai do originadorpreparação l, porque a injecção de 1x10 AAV 12 em 200 uL requer um título de pelo menos 5x10 12 ml /, não qual todos os laboratórios podem rotineiramente alcançar. Assim, enquanto um título máximo é benéfico para a primeira infecção (porque a biblioteca ainda não foi enriquecido para cápsides eficientes em um dado tecido e podem ter assim uma infecciosidade relativamente baixa global), recomendamos a utilizar pelo menos 1x10 11 partículas por mouse para seleção de fígado.
- Se os ratos estão disponíveis múltiplos, é muito útil para injectar números de partículas diferentes semelhantes para a selecção em células em cultura, e para usar vários ratos por grupo (e, em seguida, juntam os fígados recolhidos dentro de cada grupo), a fim de minimizar a variabilidade e para aumentar o sucesso as taxas de, por exemplo, 3 ratos na 1x10 11 e 3 ratos na 1x10 12 partículas.
- Uma vez que AAV é um vírus não patogénico e replicação incompetente-(sem helpervirus), não existem reacções adversas a procurar, na ausência de Adenovirus.
- Para a eutanásia, os animais foram anestesiados usando um vaporizador de isoflurano e subsequentemente sacrificados através de deslocação cervical.
- Os tecidos (fígado, neste caso) foram colhidas depois de os animais foram confirmados sacrificados. Em geral, não há necessidade também para outros tecidos para perfundir os animais ou para a colheita de AAV-infectadas órgãos antes da morte, uma vez que o ADN de AAV é estável e pode facilmente ser resgatado a partir de células congeladas / tecidos.
- Para estudar cápsides individuais (a partir da biblioteca (passo 3.6) ou após selecção (passos 5,2-5,3)), produzir vectores de AAV que codificam um gene repórter (eg, GFP 6) seguindo os passos 4,1-4,5. Portanto, clonar o gene de interesse tampa em um ajudante AAV padrão plasmídeo 2. No passo 4.1, triple-transfectar as células com 14,7 ug cada de AAV vector (que codifica o repórter), ajudante de AAV e auxiliar adenoviral. Infectar células cultivadas ou animais com o vírus purificado em diferentes quantidades e determinaçãone por exemplo, eficiência de transdução via FACS ou microscopia.
6. Os resultados representativos
Utilização do protocolo descrito aqui geralmente resulta em bibliotecas virais com uma diversidade de cerca de 1x10 6 cápsides únicos que podem então ser testadas para partículas individuais mais visualizar, ou todas as propriedades desejadas de uma linha dada célula ou em animais. No que se segue, iremos apresentar exemplos representativos para os resultados de tais in vitro ou in rastreios in vivo.
Antes disso, no entanto, consideramos que é importante novamente ressaltar a utilidade de analisar clones individuais da biblioteca original plasmídeo para a sua funcionalidade e diversidade (opcional no passo 3.6). Isto é porque os dois últimos parâmetros são extremos pré-requisitos críticos para o sucesso da selecção da biblioteca real viral que é feita a partir da biblioteca de plasmídeo. Portanto, pode-se escolher aleatoriamente único embaralhadasgenes tampão e utilizá-los para produzir vectores de AAV recombinantes (extractos brutos ou partículas purificadas, dependendo do grau de precisão desejado) que expressam um gene repórter facilmente detectável e quantificáveis. Um ensaio típico para comparar os diferentes variantes da cápside é, então, a microscopia de fluorescência, tal como mostrado no exemplo representativo na fig. 4.
Um método alternativo é baseado em FACS medição da expressão do gene repórter fluorescente que possui as vantagens adicionais que também permite a determinação da expressão do gene por célula, mais que ele funciona para células em suspensão. Fig. 5 mostra um resultado típico desta análise FACS um baseado em bruto lisados vector de AAV em vários tipos de células.
Porque a selecção acima descrito de quimeras tampão individuais é aleatória e restrita, isto é, naturalmente, não é útil como uma abordagem real para o enriquecimento de candidatos desejados. Em vez disso, selecção do viral biblioteca em células ou tecidos alvo em animais é mais adequado. Como as condições e os parâmetros variam de acordo com cada aplicação, vamos apenas destacar algumas orientações representativas poucos e resultados.
A maneira mais fácil de selecção é iterativo amplificação da biblioteca em linhas de células cultivadas ou células primárias (passo 5.2). Uma vez que requer AAV Adenovírus co-infecção para a sua propagação, as células alvo deve ser susceptível de adenovírus. Pode-se então crescer e infectar-los por exemplo, em placas de 6 poços que possuem os números de células por poço suficientes para assegurar a cobertura total da biblioteca. Uma segunda noção importante é que o equilíbrio do AAV e adenovírus é delicada; AAV muito irá inibir o adenovírus, enquanto um excesso deste último matará as células (ao contrário de AAV, o Adenovírus provoca infecções líticas) antes de os AAV pode replicar.
No entanto, como a infectividade biblioteca em um determinado tipo de célula é desconhecida, encontrando AAV "bom": os índices de Adenovirus emque tanto pode propagar requer paralelo teste de várias combinações de doses da biblioteca eo auxiliar adenoviral (por exemplo, 10:1, 1:1 e 1:10). Uma boa medida para a infecção por adenovirus potente é a ocorrência de efeitos citopáticos três dias após a inoculação do vírus, evidenciada pelo arredondamento celular e descolamento como pode ser visto na fig. Versa. 6-versa, um úteis leitura para fora para a infecção por AAV e amplificação é a detecção de proteínas da cápside por Western blotting, utilizando o anticorpo que reconhece um B1 altamente conservada AAV cápside epitopo (Fig. 7) 7.
Uma decisão importante é, então, que AAV extratos brutos de escolher para posterior re-infecção de células frescas (passo 5.2). Idealmente, um terá aqueles em que as bandas de AAV do capsídeo são, pelo menos notável (Fig. 7) porque estes sugerem uma ligação genótipo-fenótipo apertado. Esta última descreve a situação em que um genoma que codifica uma variante da cápside é determinadana verdade, embalados em o capsídeo correspondente. Para atingir e manter uma ligação genótipo-fenótipo apertado é a chave para selecção bem sucedida de individuais candidatos virais como, por sua vez assegura que cápsides com propriedades desejadas entregar o modelo cognato genético nas células durante rodadas de infecção sucessivas. Portanto, recomendamos escolher os extratos com expressão capsídeo moderada para re-infecção e usar quantidades mínimas. Juntas, essas duas medidas vão evitar a sobrecarga das células infectadas com diferentes capsídeo / genoma combinações que poderiam perturbar a ligação genótipo-fenótipo uma vez que os vírus re-infectados iniciar a replicação e re-embalagem em seus genomas não-cognatas capsídeos.
Além disso, é crítico para monitorar a diversidade da biblioteca durante rodadas repetidas infecções por sequenciação de ADN. Idealmente, um notará mudanças na composição dos clones individuais indicativos de selecção bem sucedida, isto é, accumlação de fragmentos sorotipo distintas e perdas de outros, como exemplificado na figura. 8. No caso ideal, apenas alguns ou mesmo um único clone será em última análise, ser detectado que pode então ser analisada como descrito acima. Se, no entanto, não houve mudanças são observados depois de quatro ou cinco passagens, deve-se quer aumentar a pressão de selecção (ver discussão) ou considerar que a linha celular utilizada pode ser inadequada, pois pode ser demasiado susceptível a muitos serótipos.
Para amplificação biblioteca in vivo (passo 5.3), as mesmas regras e considerações se aplicam, com duas diferenças importantes: Em primeiro lugar, não se faz clones de tela selecionados aleatoriamente em animais, devido aos custos associados e considerações éticas. Em segundo lugar, não se irá co-infectar com adenovírus ajudante uma vez que provoca toxicidade ou fatalidades em animais, mais o tropismo adenoviral restringiria selecção para células susceptíveis ao vírus auxiliar. Em vez disso, a biblioteca de AAV é infundida thE, os animais resgatados das células alvo / tecido por PCR (Fig. 9) e depois re-clonados e re-embalado para uma rodada nova infecção. Tal como acontece com selecção em cultura, este processo é repetido até que os candidatos individuais têm surgido.
Independentemente do processo de selecção, o passo final é a validação das variantes da cápside enriquecidos em sistemas apropriados. Figs. 10 e 11 mostram exemplos representativos de nossa própria seleção anterior de quimeras de AAV que executam excepcionalmente bem em cultura de tecidos ou em fígados de ratos. Como visto na fig. 10, um clone particular (AAV-DJ 3) na verdade supera uma recolha de oito tipos selvagens de AAV naturais de uma ampla gama de linhas celulares. Finalmente, outro clone selecionado em linhas de hepatoma cultura apresenta maior tropismo para o fígado murino e, consequentemente, menos off-alvo quando perifericamente infundido em ratos adultos do que o vetor de controle potente AAV8 testado em directo comparison (Fig. 11). Note-se que apesar de ser mais específico para o fígado, o clone de AAV-DN realmente transduz este órgão um pouco menos eficiente do que AAV8. A este respeito, AAV-DN é um exemplo representativo bom para o resultado da evolução de AAV e de selecção, onde candidatos finais exibem tipicamente um número de propriedades desejadas, mas não são necessariamente perfeita em todos os aspectos.
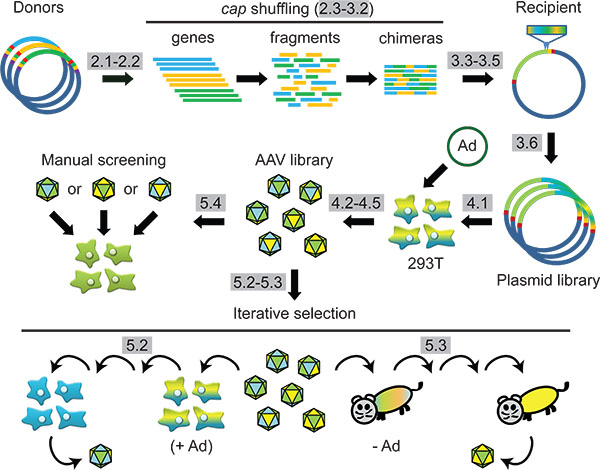
A Figura 1 Esquema:. Sintético AAV engenharia cápside através da família de DNA shuffling e subsequente selecção em células ou em animais. Etapas do protocolo são destacadas em cinza.
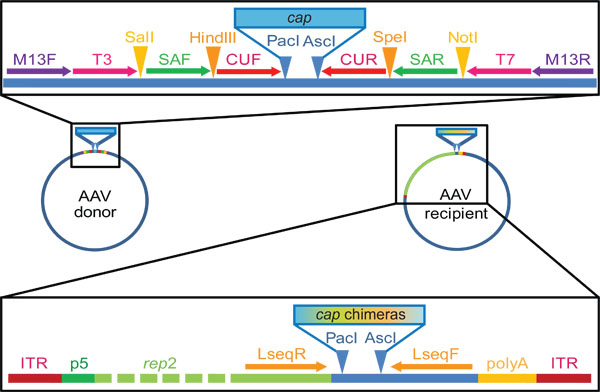
Figura 2. AAV doador gene tampa e plasmídeos recipiente utilizado em nosso laboratório para a família de DNA shuffling e geração de biblioteca. Note que estes são apenas exemplos representativos, e que os locais exatos e seqüênciaspode ser personalizado. O nosso doador de base plasmídeo é derivada da comercialmente disponível pBluescript II KS (+) vector que nós engenharia para conter a ligação iniciador mostrado, bem como locais de restrição, representado por setas ou triângulos, respectivamente. Em seguida, os genes clonados tampão de AAV serotipos 1-9 (amplificado com os iniciadores CAPF / R) em deste plasmídeo, para se tornar flanqueado por Pac I e locais de restrição Asc I. Iniciadores T3 e T7 são utilizados para o isolamento tampa no passo 2.2, enquanto que a amplificação mais tarde de re-montados sequências quiméricas (passo 3.2) pode ser realizada utilizando pares de primers quer SAF / R ou CUF / R, ou ambos, em uma PCR aninhada. O destinatário competente para replicação do plasmídeo transporta repetições terminais de AAV invertidas (ITRs, replicação e sinais de empacotamento) que flanqueiam o gene rep AAV2 sob o controlo do promotor p5 de AAV. Pac I e locais de restrição Asc I a jusante do rep permitir "em moldura" clonagem do conjunto de genes embaralhadas cap. Osítios de ligação do primer Lseq mostrados são úteis para a tampa sequenciamento genético (passo 3.6).

Figura 3. Exemplo para DNase I digerir de genes tampão (AAV2, 8 e 9). É mostrado um agarose análise de electroforese em gel dos produtos de gene cap digere utilizando os indicados diferentes tempos de incubação (em minutos: segundos). Pista U mostra o fragmento não digerido entrada piscina tampa como controle. Tamanhos de bandas de ADN marcadas em pistas M são em quilobases. Neste exemplo, ideal digere foram obtidos com tempos de incubação de 1:45 ou 2:00 min, o que originou o pico preferido predominante de cerca de 100 a 500 pares de bases (caixa amarela).

Figura 4. Exemplo para microscopia baseado análise de três linhas de células humanas infectadas com cinco diferentes recombinante ve AAV YFP-expressandoctors (nomes em cima) feitas com genes cap embaralhadas aleatoriamente selecionado de uma biblioteca original, baseada em AAV2, 8 e 9.

Figura 5. Exemplo para FACS baseado em análise de quatro tipos de células infectadas (a série de dez diluições) com 18 diferentes vetores recombinantes expressando-YFP AAV (nomes em cima, incluindo os da Fig. 4) feito com embaralhadas genes cap selecionados aleatoriamente a partir de uma biblioteca original (AAV2, 8 e 9). Expressão YFP foi codificados por cores para facilitar a visualização. Representados são percentagens de células transduzidas, em que preto sempre indica 0% e branco o maior número medido em cada tipo de célula. B2 clone (vermelho) exemplifica um clone com a eficácia global pobres, como se pode esperar a partir de cápsides não selecionadas. Note-se que a análise FACS é mais sensível e pode também ser utilizado para células em suspensão (tal como SupT1) que são menos propícios a microscopia. hu, Humano; mu, murino.
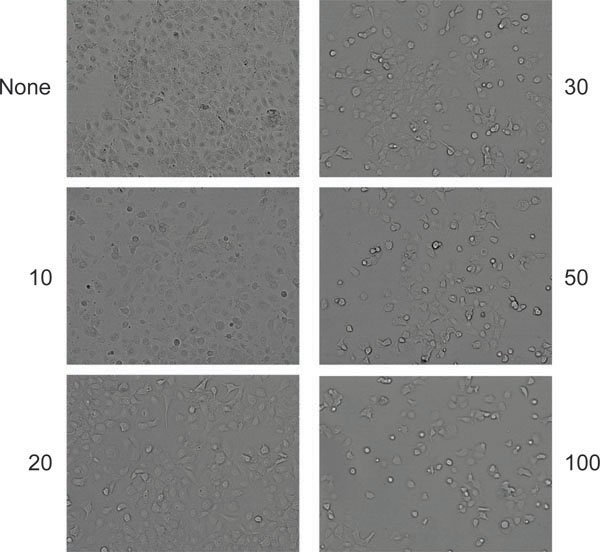
Figura 6. Aparência típica de efeitos citopáticos nas células (HeLa, neste caso) após produtivo co-infecção com AAV e adenovírus. As células foram ou não infectados esquerda (painel superior esquerdo) ou infectados com as quantidades indicadas (partículas por célula) de adenovírus.

Figura 7. Detecção de AAV a expressão da proteína da cápside por Western blotting como uma medida para a infecção biblioteca e amplificação. O blot à esquerda mostra células co-infectadas com vários volumes (em uL) de uma biblioteca de AAV e adenovírus ajudante. As duas primeiras faixas são bons exemplos para conditions originando a expressão da proteína dificilmente detectável de AAV, o que indica suficiente mas não excessiva infecção de AAV e de amplificação e, consequentemente, a desejada ligação genótipo-fenótipo apertado. Assim, 0,1, 1 ou 10 uL de estes sobrenadantes foram utilizados para re-infecção de células frescas (blot direita). As células em pista C foram infectadas com AAV sozinho como um controlo negativo (ausência de expressão detectável devido à ausência do helpervirus).
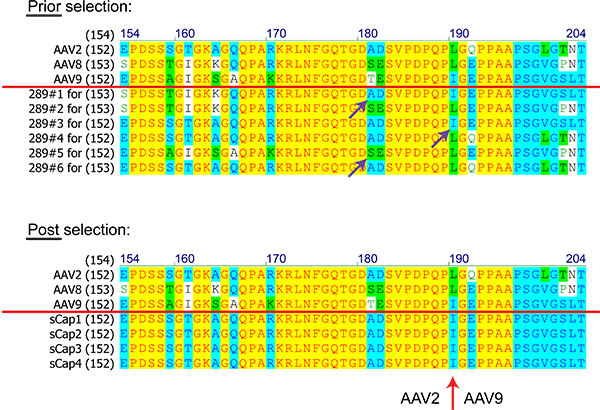
A Figura 8. Comparações de sequências de proteínas (os números são aminoácidos) de AAV clones a partir de uma biblioteca com base em AAV2, 8 e 9, antes e após a selecção. Sequências acima da linha vermelha mostra os AAV parentais. Roxo setas indicam eventos de recombinação homóloga. A seta vermelha representa um cross-over entre AAV2 e AAV9 observado em todos os clones selecionados.
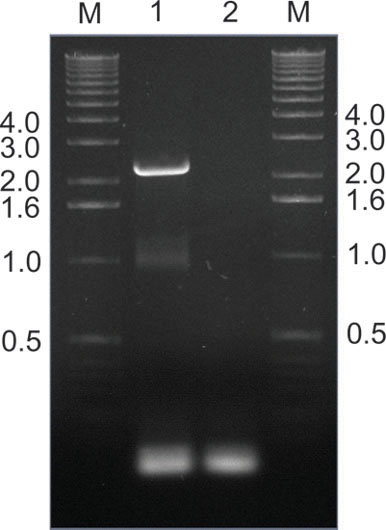
A Figura 9. </ Rescue strong> de infectado com sucesso clones de AAV de tecidos de camundongos via PCR. Neste exemplo, os genes de AAV cap foram amplificado por PCR a partir de fígados de rato extraída uma semana após a infusão da biblioteca periférica, utilizando par de primers SAF / R (Fig. 2). Pista 1 mostra a banda esperada quilobases 2,2, enquanto que a pista 2 é um controle não-molde. Após Pac I e restrição Asc I, os fragmentos amplificados foram re-clonado no plasmídeo destinatário original (Fig. 2) para a produção subsequente e re-infusão de uma biblioteca secundária. Tamanhos de bandas de ADN marcadas em M pistas são indicados em kilobases.

Figura 10. Exemplo para um desempenho superior de uma AAV enriquecido quimera em células em cultura. Clone AAV-DJ tem sido relatada por nós antes 3, mas na maior parte representa um híbrido entre serótipos de AAV 2, 8 e 9, e tem sido seleccionado de entre um librar y contendo esses três serotipos mais cinco adicionais nas células de fígado humano na presença de anti-soros reunidos humano. Para comparar a sua eficácia a oito serótipos de AAV naturais (indicado por 1-6, 8 e 9 na parte superior), todos os genes do tampão foram usados para produzir purificado auto-complementar que expressam GFP-vectores 8,19. Estes foram normalizados para conter 2x10 9 genomas vector por ml e depois usado para transduzir as linhas de células mostradas em 10 diluições em série. Três dias mais tarde, Gfp-expressando células foram contadas e os títulos infecciosos foram determinados tendo em conta o factor de diluição. Em contraste com o código na fig. 5, cores mais escuras indicam aqui infectividade mais elevados por número de partículas. Como é evidente, o selecionado AAV-DJ quimera supera todos os tipos selvagens de AAV naturais, exemplificando o êxito do sistema de seleção aplicada. fibr, fibroblastos, ha, hu, hamster, humano; mu, murinos; si, símios.

Figura 11. Exemplo para a análise de um AAV seleccionado quimera no fígado do rato. Clone AAV-DN foi seleccionada em células de hepatoma de murídeo e, em seguida, utilizado para produzir vectores de luciferase-expressando recombinantes 8. São mostrados três ratinhos representativos (por grupo) uma semana após a infusão periférica de doses iguais de este vector ou um controlo baseado tipo selvagem AAV8, um dos mais potentes conhecidos isolados naturais de fígado de rato 9. Note-se que enquanto o clone de AAV-DN dá expressão ligeiramente menos global no fígado (painel (I)), é mais específico para este órgão, uma vez que exibe substancialmente menos off-alvo em não-hepáticas tecidos uma vez que os níveis de expressão do fígado têm sido ajustado através do software de imagem (painel (II)).
Discussão
Aqui, nós descrevemos essenciais etapas experimentais e diretrizes para AAV capsídeo de engenharia através da família de DNA shuffling e para a evolução em células ou em animais. Em essência, estes protocolos são versões padronizadas de os procedimentos relatados, primeiro dentro do campo AAV em 2008 3. Enquanto uma onda de estudos de acompanhamento por outros relataram por exemplo, inúmeras modificações, 10-13, nossas versões atuais representam estratégias básicas que rend...
Divulgações
Todos os autores declaram que não têm nada a revelar.
Agradecimentos
Os autores agradecem o apoio notável de seu laboratório, os membros da equipe e do trabalho pelo Cluster de CellNetworks Excelência na Universidade de Heidelberg, bem como pela Chica e Schaller Heinz (CHS) da fundação. Nós apreciamos que a evolução molecular de AAV via DNA família baralhar tornou-se um campo muito ativo desde a nossa primeira publicação há três anos e, portanto, peço desculpas a todos os autores de publicações relevantes, cujo trabalho não pode ser citado aqui, devido a limitações de espaço.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | |
| DNase I | Invitrogen | 18068-015 | |
| Polietilenoimina (PEI) | Sigma-Aldrich | 408727 | |
| As enzimas de restrição | NEB | Vário | |
| DNA-ligase de T4 | NEB | M0202T | |
| Gel extraction kit | Qiagen | 28704 | |
| Phusion Kit polimerase II | Finnzymes (NEB) | F-540S | |
| HotStar Kit polimerase Hifi | Qiagen | 202602 | |
| DMSO | Finnzymes (NEB) | F-540S (parte do kit) | |
| EDTA (25 mM) | Invitrogen | 18068-015 (parte do kit) | |
| Tris | Roth | 4855,2 | |
| Ampicilina sal de sódio | Roth | K029.2 | |
| dNTPs (10 mM, 100 ul) | Invitrogen | 18427013 | |
| Iodixanol (OptiPrep) | Axis-Shield | 1114739 | |
| Phenolred | Merck | 107241 | |
| Kit de preparação plasmídeo mega- | Qiagen | 12181 | |
| Ultracentrífuga | Beckman-Coulter | Optima L90K | |
| Seal-Quick tubos de centrífuga | Beckman-Coulter | 342414 | |
| Unidade de Eletroporação | Bio-Rad | GenePulserXcell | |
| Termociclador | Eppendorf | Vapo Protect | |
| Bloco de aquecimento | BIOER | MB-102 | |
| Microscópio de fluorescência | Olimpo | IX81 | |
| Analisador FACS | Beckman-Coulter | Cytomics FC500 MLP | |
| MegaX DH10B células T1R | Invitrogen | C640003 | |
| Benzonase | Merck | 101695 | |
| Adenovírus-5 | ATCC | VR-5 | |
| pBluescript II KS (+) do plasmídeo | Stratagene | 212207 | |
| cap5F (Pac site que em amarelo, CAP5-sequências específicas em negrito): GACTCTTAATTAACAGGT ATGTCTTTTGTTGATCACCCTCC | IDTDNA | Cartilha personalizado | |
| cap5R (Asc site que em verde, CAP5-sequências específicas em negrito): GTGAGGGCGCGCC TTAAAGGGGTCGGGTAAGGTATC | IDTDNA | Cartilha personalizado |
Referências
- Grimm, D., Kay, M. A. From virus evolution to vector revolution: use of naturally occurring serotypes of adeno-associated virus (AAV) as novel vectors for human gene therapy. Curr. Gene Ther. 3, 281-304 (2003).
- Grimm, D. Production methods for gene transfer vectors based on adeno-associated virus serotypes. Methods. 28, 146-157 (2002).
- Grimm, D. In vitro and in vivo gene therapy vector evolution via multispecies interbreeding and retargeting of adeno-associated viruses. J. Virol. 82, 5887-5911 (2008).
- Muller, O. J. Random peptide libraries displayed on adeno-associated virus to select for targeted gene therapy vectors. Nat. Biotechnol. 21, 1040-1046 (2003).
- Perabo, L. In vitro selection of viral vectors with modified tropism: the adeno-associated virus display. Mol. Ther. 8, 151-157 (2003).
- Zolotukhin, S., Potter, M., Hauswirth, W. W., Guy, J., Muzyczka, N. A "humanized" green fluorescent protein cDNA adapted for high-level expression in mammalian cells. J. Virol. 70, 4646-4654 (1996).
- Wobus, C. E. Monoclonal antibodies against the adeno-associated virus type 2 (AAV-2) capsid: epitope mapping and identification of capsid domains involved in AAV-2-cell interaction and neutralization of AAV-2 infection. J. Virol. 74, 9281-9293 (2000).
- Grimm, D. Fatality in mice due to oversaturation of cellular microRNA/short hairpin RNA pathways. Nature. 441, 537-541 (2006).
- Nakai, H. Unrestricted hepatocyte transduction with adeno-associated virus serotype 8 vectors in mice. J. Virol. 79, 214-224 (2005).
- Koerber, J. T., Jang, J. H., Schaffer, D. V. DNA shuffling of adeno-associated virus yields functionally diverse viral progeny. Mol. Ther. 16, 1703-1709 (2008).
- Li, W. Engineering and selection of shuffled AAV genomes: a new strategy for producing targeted biological nanoparticles. Mol. Ther. 16, 1252-1260 (2008).
- Ward, P., Walsh, C. E. Chimeric AAV Cap sequences alter gene transduction. Virology. 386, 237-248 (2009).
- Yang, L. A myocardium tropic adeno-associated virus (AAV) evolved by DNA shuffling and in vivo selection. Proc. Natl. Acad. Sci. U.S.A. 106, 3946-3951 (2009).
- Perabo, L. Combinatorial engineering of a gene therapy vector: directed evolution of adeno-associated virus. J. Gene. Med. 8, 155-162 (2006).
- Maheshri, N., Koerber, J. T., Kaspar, B. K., Schaffer, D. V. Directed evolution of adeno-associated virus yields enhanced gene delivery vectors. Nat. Biotechnol. 24, 198-204 (2006).
- Wu, Z., Asokan, A., Samulski, R. J. Adeno-associated virus serotypes: vector toolkit for human gene therapy. Mol. Ther. 14, 316-327 (2006).
- Kwon, I., Schaffer, D. V. Designer gene delivery vectors: molecular engineering and evolution of adeno-associated viral vectors for enhanced gene transfer. Pharm. Res. 25, 489-499 (2008).
- Perabo, L., Huber, A., Marsch, S., Hallek, M., Buning, H. Artificial evolution with adeno-associated viral libraries. Comb. Chem. High. Throughput. Screen. 11, 118-126 (2008).
- McCarty, D. M., Monahan, P. E., Samulski, R. J. Self-complementary recombinant adeno-associated virus (scAAV) vectors promote efficient transduction independently of DNA synthesis. Gene Ther. 8, 1248-1254 (2001).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados