このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
シャッフルDNAファミリーを経由して合成アデノ随伴ウイルス(AAV)遺伝子治療ベクターのエンジニアリングと進化
* これらの著者は同等に貢献しました
要約
我々は分子DNAファミリーシャフリングを介して合成アデノ随伴ウイルス(AAV)の遺伝子治療ベクターを設計し、進化するための基本的なテクニックを示しています。さらに、一般的なガイドラインや文化やマウスの標的細胞上の強化されたプロパティを持つ個々のキメラキャプシドの選択と分析のための代表的な例を提供しています。
要約
アデノ随伴ウイルス(AAV)ベクターは、有益な特性の1のユニークな組み合わせにより、治療用ヒト遺伝子導入のための最も強力かつ有望な車両の一部を表しています。これらは、基礎となる野生型ウイルスと高力価、高純度、臨床グレードの組換えベクター2の生産のための高度な方法論のapathogenicityが含まれています。他のウイルス以上のAAVシステムの更なる特定の利点は、当然、まだすべて簡単に一般的なプロトコルの1,2を使用してベクトルとして操作することができる本質的な特性で異なる血清型に発生するの富の可用性です。また、私たち自身を含むグループの数は、最近、どちらか、複数の入力の血清型の資産、またはその分離、単一の特性を向上させるを組み合わせて合成ベクトルを作成するためのテンプレートとして、これらの天然のウイルスを使用するための戦略を考案した。これらの目標ARを実現するためにそれぞれの技術電子どちらDNA家族シャフ3、部分的相同性に基づいて再組み立てに続いて様々なAAVキャプシド遺伝子のつまり断片化(通常はほとんどのAAV血清型のために> 80%)、またはペプチドディスプレイには通常7つのアミノ酸の4,5、 すなわち挿入ペプチドは、理想的に所望の細胞型への再ターゲティングを媒介ウイルスキャプシドの露出したループ。最大の成功のために、両方の方法は、約100万の異なるカプシド変異体のライブラリを生成するためにプロトコルがアップスケーリングされることにより、高スループットな方法で適用されます。各クローンは、その後多数の親ウイルスのユニークな組み合わせ(DNAシャフリングアプローチ)から構成されるか、同じウイルスのバックボーン(ペプチドディスプレイアプローチ)内の独特のペプチドが含まれています。その後の最後のステップでは、ほとんど、または選択プロセスの理想的なすべての要件を満たす個々のカプシドを濃縮するために、標的細胞上のそのようなライブラリの選択を繰り返します。後者は、好ましくは、櫛抗AAV抗体と反応するすべてのキャプシドのインスタンスを除去するための負の選択とそのような興味のある特定の細胞型の成長としてINES正圧、、、。この組み合わせは選択を生き残った合成キャプシドは、おそらく任意の天然AAV分離で発見されなかったであろう方法で、特定のアプリケーションのニーズと一致している可能性が高くなります。ここでは、DNAのファミリー2つの技術の理論と実験のより困難なようにメソッドをシャッフルに焦点を当てています。我々は、記述し、実証するシャッフルAAVライブラリの生成と選択( 図1)の重要なすべての手順を実行し、次に1つのニーズは、分子AAVの進化を成功させるために注意すべきことのプロトコルの落とし穴や重要な側面について説明します。
プロトコル
1。 AAVキャプシド遺伝子をコードするプラスミドセットの準備
- その後のDNAシャッフリングのための様々なAAVキャプシド( キャップ )の遺伝子の十分な量の日常的な準備を容易にするために、最初に共通のプラスミドバックボーンにこれらの遺伝子をサブクローニング。 nested PCR法およびクローニングのためのプライマー結合部位( 図2)として後で使用するために> 20のヌクレオチドの同一のフランキング配列を含めることが重要です。
- (AAV5のための例示的なプライマーは、表を参照)、適切なプライマーを用いて、PCRは通常、選択肢のcap遺伝子の隣にAAV2 rep遺伝子を含む一般に入手可能なAAVプラスミドから目的のcap遺伝子を増幅する。 PCR産物は標準的なクローニングのために使用されるため、製品の精製〜1μgのは、すでに十分であり、任意の従来のPCRプロトコルが故に使用することができます。
- 精製PCR産物( 例えば 、アガロースゲル精製または標準的なPCR精製キットを使用)とダイジェスト受信者、その認識部位キャップ増幅 (1.1)のためだけでなく、プラスミドの受信に使用するプライマーに存在する制限酵素でプラスミド。彼らはほとんどのAAVのには存在しないように私たちの研究室では、 パックマン私と ASC I部位( 図2)を使用します 。
2。 DNase処理ベースのcap遺伝子断片
- PCRは、ステップ1.1から1.3で生成されたプラスミドからの選択のcap遺伝子を増幅する。下記のように一つの反応は、PCR産物の〜3μgを得られます。ライブラリに含まれるcap遺伝子の数は、最大6つのシャッフルの反応については、この十分に応じて。
- PCRのために、プラスミド200ngのキャップ 、2μMの最終濃度での各プライマー、10μlの5倍のハイファイバッファーと1μlのハイファイポリメラーゼを含む50μlの反応を設定します。 95°Cとし、15の40サイクルを実行する秒94℃、30秒57℃、3分間68℃、最後の10分のステップが続く時で5分で始まる72℃ゲルまたはキットを介したPCR産物を精製して、キメラに再アセンブリのcap遺伝子の断片を作成するために制御されたDNaseをダイジェストを設定します。
- したがって、同様に54μlのH 2 Oで4μgの合計額に様々なキャップのPCR産物を混ぜて、反応に6μlののDNase反応バッファーと0.5μlのDNase Iを加える慎重に三回をフリック、簡単にスピンし、直ちに25℃の加熱ブロックに置く。 1と2分(複数の並列反応を設定し、15秒刻みで、それらを終了する)の間インキュベートし、6μlの25mMのEDTAと簡単に75℃10分間ボルテックスし、インキュベートすることにより℃を加えることによって反応を停止
- 標準1%アガロースゲル上でキャップ断片を精製。理想的には、塗抹標本は、100と500塩基対の間に表示されるはずです。 DNaseは私は非常に強力な酵素、適切な取り扱いとタイミングであるため、この段階で重要である、ステップ2.3のインキュベーション時間内に複数のバリエーションが最適な解像度のために必要になることがありますults( 図3)。標準キットを使用して溶出したDNAを精製し、その濃度を決定します。
3。 DNAファミリーシャフリング
- 最初に、彼らは自己プライム部分的相同性に基づいたPCRによる完全長の配列にキャップの断片を組み立て直す。したがって、500 ngの精製したフラグメント(ステップ2.4)、10μlの10倍备考バッファー、1μlの10 mMのdNTPsを、1.5μlのDMSOおよび0.5μlの备考IIポリメラーゼと50μlの反応を設定します。 98℃でインキュベート30秒°Cとし、秒98℃、30秒42℃、45秒72℃、72℃最後の10分のステップに続いて10℃の40サイクルを実行します。
- その後の第二のPCRでは、保存されたフランキング配列( 図2)に結合するプライマーを使用して、その後のクローニングのために再組み立てcap遺伝子を増幅する。したがって、最初のPCR(ステップ3.1)、2μMの最終濃度での各プライマー、0.5μlのMgCl2を、2μlを含む50μlの反応を設定する10μlの5倍のハイファイバッファーと1μlのハイファイポリメラーゼ。我々は、その後のクローニングのための十分に高い歩留まりを確保するために16から24までのPCRを実行することをお勧めします。プールのPCRを、完全長キャップバンド(ゲルまたはキット)を浄化する。
- 同様にAAV2 rep遺伝子、複製コンピテントAAVプラスミドを運ぶAAV ITRを(複製とパッケージングシグナル逆方向末端反復)にクローニングするためのパック私と ASC Iで精製されたキャップの遺伝子プールを消化。後者は、(詳細については、 図2を参照してください) "フレームの" cap遺伝子プールに対応するために、同じサイトで従わなければならない。 PCR産物の完全消化を達成するために、過剰な酵素で一晩インキュベートする。
- 3時01モル比でキャップ断片と適切にカットAAV ITR / 担当者のバックボーンを連結。は50 ng /μlの最終DNA濃度で40μlの合計体積マスターミックス(20変換するのに十分な)を作ると16で一晩インキュベート℃、
- 30μlのエレクトロコンピテントE.と混合してライゲーション反応(氷上で)2μlを変換する大腸菌 (商業細胞から成長し、任意の標準プロトコルを使用して、有能な製)。氷の上でプレ冷却エレクトロポレーションキュベット(1mmのギャップ)に追加します。 1.8 kVの、200Ω、25μFでエレクトロポ。時定数は5 ms程度である必要があります。すぐに1ミリリットル予め温めておいたSOC培地および250 mlのフラスコへの転送を追加します。 20そのようなエレクトロポは6×10約の異なるクローンの多様性を持つライブラリをもたらすでしょう。
- プールされた変換に1ボリューム予め温めておいたSOC培地を追加し、37℃、1時間180rpmで振とうする。その後、LB培地に加えてアンピシリン(50μg/ mlの最終濃度)との合計容積800ミリリットルまでを起動し、同じ条件で別の16時間インキュベートします。 を使用して、例えばライブラリープラスミドDNAを精製する。キアゲンメガ準備キットです。
正確なライブラリーの多様性、プレートaliquを計算するには、次の手順3.6でオプションLB-アンピシリンプレート上で800ミリリットル溶液(前16時間のインキュベーションへ)( 例えば 、10 cmのプレートに10μl)のOTと翌日コロニーをカウントします。また、( 図も参照してください。8)高効率シャッフル、シーケンスなどの 24クローンを検証し、親のcap遺伝子にそれらを合わせます。最後に、ライブラリの活力と高機能の多様性を確認するために、サブクローンは、ランダムプラスミドAAVヘルパーにcap遺伝子を選んだと(以下の5.4ステップも参照してください)小規模( 図4-5)で組換えベクターを生産し、分析するためにそれらを使用します。
4。ウイルスライブラリーの製造
- シード10 15センチメートルHEK293T細胞の2皿(4.5x10 6細胞/皿)および48時間220μgのAAVライブラリとそれ以降のトランスフェクションおよびアデノウイルスプラスミド220μgを(AAV伝播に必要)。したがって、事前に暖かいPEI(ポリエチレンイミン)と37℃で300mMのNaCl℃、その後、7.9ミリリットルのNaClおよびDNAを混合し、合計voluにH 2 Oを追加する15.8ミリリットルの私。別のチューブに、3.52ミリリットルPEI、7.9ミリリットルのNaCl及び4.38ミリリットルH 2 O(10のトランスフェクションのすべてのボリューム)を混ぜます。ミックス(渦)を組み合わせると料理(皿当たり3 ml)を均等にソリューションを配布する前に、室温で10分間インキュベートする。
- 48時間後、培地に細胞をこすり、15分間1200rpmでそれらをスピンダウンする。 6 mlの溶解バッファー(50mMトリス-HCl pH8.5で、50mMのNaHCO 3水溶液 )と5回の凍結融解サイクル(-80/37℃)に件名に細胞ペレットを再懸濁します。 20分間3750 rpmで細胞残渣をスピンダウンする前に、37℃で1時間mlあたり50 Uベンゾナーゼ℃、インキュベートします。
- 60%のイオジキサノール(PBS-MKのOptiPrep)PBS-MKの株式(1X PBS、1mMのMgCl 2、2.5mMのKClの)から15%、25%および40%希釈液(2.5μL/ mlのphenolredで)を準備します。
- 1.5ミリリットルEAC続いて、ベックマンクイックシール遠心チューブ(14x89 mm)の中に5 mlのウイルス懸濁液を追加するには、パスツールピペットを用いてAAV精製のための勾配を設定します。15%の時間、25%および40%イオジキサノールソリューションを提供します。溶解緩衝液で勾配オフトップ。
- 4℃で2時間50.000 Kでの超遠心°ベックマンTi70.1ローターのC。その後70%エタノールでチューブの外側を清掃、換気用チューブの上に針を堅持し、針を用いて40%のイオジキサノール画分の1.2ミリリットルを描画します。それが空のAAVキャプシドが含まれている25%の画分から描画を避けるために注意してください。
5。スクリーニングおよび選択
- 所望の特性を示すキメラキャプシドが登場するまで、この時点では、1のいずれかを繰り返した( 図6-11)、培養細胞や動物のライブラリ全体を増幅することができます。
- 培養細胞で選択するには、AAVの成長をサポートするために様々なライブラリのアリコート( 例えば 、1、10、100μL)およびアデノウイルス-5を共感染させる。予測できない特定の細胞型のライブラリの感染としてライブラリおよびアデノウイルスヘルパーの複数のバリエーションをテストすることが不可欠です。収穫〜3日後の細胞は、56℃で30分間、再感染する新しい細胞にアデノウイルスを不活性化、凍結融解を介して増幅されたAAVを抽出します。異なるキャプシドは(シークエンシングにより検証する)豊かになるまで、5回まで繰り返します。
- 動物の選択については、ライブラリを使用して感染し、約1週間後に所望の組織または細胞型を抽出します。これは動物に有害な毒性の原因となりますようにアデノウイルスを共感染させないようにしてください。前と同じプライマー( 図2)を用いてPCRを介したウイルスDNAを救出する、 キャッププールを再クローニングし、別個のキャプシドは(シークエンシングにより検証する)豊かになるまで、新鮮なライブラリとリピートを生成します。
- in vivoでの選択のための正確な条件は、(ボリューム、力価、ルート)興味のある標的組織に依存します。肝臓では、マウスが一般的に尾静脈注射(IV)を介して、200μlのPBSの総量で1×10 11〜1×10 12ウイルス粒子が感染している。制限要因は、通常、原送信のウイルス力価である200μlの1×10 12 AAVの注入は、少なくとも5×10 12 / mlの力価を必要とするので、lの準備は、そのすべてのラボでは日常的に達成できない場合があります。最大の力価は、(ライブラリがまだ与えられた組織内の効率的なキャプシドが濃縮されていないため、比較的低い全体的な感染力を持っているかもしれないので)最初の感染のために有益ですがそれ故に、我々は当たり少なくとも1×10 11粒子を使用することをお勧めします肝臓選択のためのマウス。
- 複数のマウスが使用可能な場合には、ばらつきを最小限にし、成功を高めるために培養細胞での選択に似た別の粒子数を注入し、グループごとに複数のマウス(その後、各グループ内のプールに収集肝臓)を使用することは非常に便利です。例えば、レートは、1×11の3匹と1×10 12粒子で3匹。
- AAVが非病原性及び複製無能な(helpervirusなし)ウイルスであるため、アデンの不在下で探しする副作用はありませんovirus。
- 安楽死のために、動物はイソフルラン気化器を用いて麻酔し、頚椎脱臼を経由してその後安楽死させた。
- 動物は安楽死が確認された後、組織(この場合は肝臓)を採取した。一般的に、AAV DNAは安定であり、簡単に凍結した細胞/組織から救出することができるため、動物を灌流するか、または前の死へのAAV感染臓器を収穫する他の組織にも必要はありません。
- 単一のカプシドを((ステップ5.2から5.3)ライブラリ(ステップ3.6)から、または選択した後)勉強するには、次の手順に4.1から4.5でレポーター遺伝子( 例えば、GFP 6)をコードするAAVベクターを生成します。したがって、2プラスミド標準AAVヘルパーに興味のcap遺伝子をクローニングします。ステップ4.1で、14.7μgのAAVベクター(レポーターをコードする)、AAVヘルパーとアデノウイルスのヘルパーの各々のセルをトリプルトランスフェクション。異なる量で精製されたウイルスの培養細胞や動物に感染するとdetermiFACSまたは顕微鏡を経由してね伝達効率など 。
6。代表的な結果
プロトコルの使用は、与えられたセルの行または動物の大部分を表示する単一の粒子またはすべての所望の特性についてスクリーニングすることができる6×10のユニークなキャプシドの多様性を持つウイルスのライブラリ内の典型的結果ここで概説した。以下では、我々はこのようなin vitroまたは in vivoスクリーニングでから結果を得るために、代表的な例を提供します。
その前に、しかし、我々はそれが重要な再びその機能性と多様性のためにオリジナルのプラスミドライブラリー(オプションのステップが3.6)から個々のクローンを分析の有用性を指摘することを検討してください。後者の二つのパラメータは、プラスミドライブラリーから作られ、実際のウイルスライブラリの選択の成功のために最大限の重要な前提条件であるためです。したがって、1がランダムにシャッフルされ、単一選択することができますcap遺伝子と容易に検出および定量化可能なレポーター遺伝子を発現する組換えAAVベクター(粗抽出物または精製された粒子は、所望の精度の程度に応じて)を生成するためにそれらを使用します。 図の代表例に示すように、異なるカプシド変異体を比較する典型的なアッセイは、その後、蛍光顕微鏡です。 4。
別の方法としては、浮遊細胞のために動作していることそれはまた、細胞あたりの遺伝子発現の決定のために許可されている余分な利点を保持している蛍光レポーター遺伝子の発現のFACSベースの測定を加えたものです。 図。 5は、種々の細胞型における原油AAVベクター溶解物のようなFACSベースの分析から、典型的な結果を示しています。
個々のキャップキメラの上記の選択はランダムで制限されているため、それはもちろん、希望する候補者の濃縮のための実際的なアプローチとして有用ではありません。 viの代わりに、選択動物における標的細胞または組織中のRALライブラリは、より適切です。条件とパラメータは、各アプリケーションによって異なりますので、我々はいくつかの代表的なガイドラインとの結果を強調表示されます。
最も簡単な選択は、培養細胞や初代培養細胞(ステップ5.2)上でライブラリーの増幅を繰り返しです。 AAVは、その伝播のアデノウイルスの同時感染を必要とするので、標的細胞は、アデノウイルスのために敏感でなければなりません。一つは、ライブラリの完全なカバレッジを確保するためにウェルあたり十分な細胞数を保持し、6ウェルプレート中で成長し、それらは、 例えば感染することができます。第二に重要な概念は、AAVおよびアデノウイルスのバランスが繊細であるということです。AAVの複製ができる前に、後者の過剰細胞(AAVとは異なり、アデノウイルスは溶解感染症を引き起こす)殺してしまうしながらあまりにも多くのAAVは、アデノウイルスを抑制します。
アデノウイルスの比率で:特定の細胞型のライブラリの感染が不明であるので、しかし、 "良い" AAVを見つけるその両方が伝播することができ、ライブラリの用量およびアデノウイルスヘルパー( 例えば 、10:1、1:1と1:10)の様々な組合せの並列テストをする必要があります。強力なアデノウイルス感染症のためのよい指標は、三日間ウイルス接種後の細胞変性効果、細胞の丸めによって証明されると図に見られるように取り外しの発生です。 6。 逆に 、AAV感染の読み出し有用と増幅が高度に保存されたAAVキャプシドエピトープ( 図7)7 を認識B1抗体を用いて、ウェスタンブロッティングによってキャプシドタンパク質の検出である。
重要な決定は、原油AAVは、新鮮な細胞のその後の再感染(ステップ5.2)のために迎えに抽出した後です。理想的には、これらのタイトな遺伝子型と表現型のリンケージを示唆しているので、AAVキャプシドのバンドが( 図7)は、少なくとも顕著であるところ、それらがかかります。後者は、特定のキャプシド変異体をコードするゲノムである状況について説明します。実際に対応するカプシドにパッケージされています。それは順番に所望の特性を有するキャプシドは、連続した感染症のラウンド中に細胞内に同種の遺伝子テンプレートを提供することを保証するようにタイトな遺伝子型と表現型のリンケージを達成し、維持するには、個々のウイルスの候補者の成功した選択の鍵となります。したがって、我々は、再感染の中等度のカプシド式で粗抽出物を選択すると、最小限の量を使用することをお勧めします。これら2つの措置は、再感染したウイルスは、非同族のキャプシドに自分のゲノムを複製、再パッケージ化し始めると、そうでなければ遺伝子型と表現型のリンケージを混乱させることができる異なったカプシド/ゲノムの組み合わせで新たに感染した細胞の過負荷を防ぐことができます。
また、DNAシーケンシングにより繰り返される感染症のラウンド中にライブラリの多様性を監視することが重要です。理想的には、成功した選択を示す個々のクローンの組成のシフト、 すなわち 、ACCUMに気づくでしょう図に例示した他の異なる血清型の断片と損失のulation。 8。完璧なケースでも、ほんの数または単一のクローンは、最終的には上述のように分析することができる検出されます。しかし、全くシフトは4または5継代後に観察されていない場合は、1つのいずれかの選択圧(説明を参照してください)増やすか、それがあまりにも多くの血清型にも影響されやすいかもしれませんので、使用細胞株は不適切である可能性があることを考慮する必要があります。
in vivoでのライブラリの増幅(ステップ5.3)については、同一のルールおよび注意事項には、2つの重要な違いは、適用されます:まず、1は、関連するコストと倫理的配慮のために動物の画面ランダムに選択されたクローンをしません。それは動物に毒性または死亡の原因に加えて、アデノウイルスの向性は、ヘルパーウイルスの感受性細胞に選択を制限すると同じように第二に、人はヘルパーアデノウイルスと共感染しません。代わりに、AAVライブラリが目に注入されるeの動物は、再クローン化し、新たな感染ラウンド用に再パッケージ化し、PCRによる標的細胞/組織( 図9)から救出します。個々の候補者が浮上しているまで、文化の中で選択と同様、このプロセスが繰り返されます。
かかわらず、選択手順の、最後のステップは、適切なシステムの豊富なキャプシド変異体の検証図である。 10と11は組織培養やマウスの肝臓に非常によく実行するAAVキメラの私たち自身の以前の選択範囲から代表的な例を示します。 図に見られる。 10、一つの特定のクローン(AAV-DJ 3)は実際に細胞株の広い範囲で8天然のAAV wildtypesのコレクションよりも優れています。最後に、培養肝細胞ラインで選択されている他のクローンは、マウス肝臓に高い親和性を示し、末梢直接COMでテストされ強力なAAV8コントロールベクターよりも成体マウスに注入したときにそれに応じて以下のオフターゲティングパリソン( 図11)。肝臓のためのより具体的であるにもかかわらず、クローンAAV-DNは、実際にAAV8よりも少し少ない効率的なこの器官を伝達することに注意してください。この点では、AAV-DNは、最終候補者は、通常、所望の特性の数を示すが、すべての面で必ずしも完璧ではありませんAAVの進化と選択の結果の良い代表的な例です。
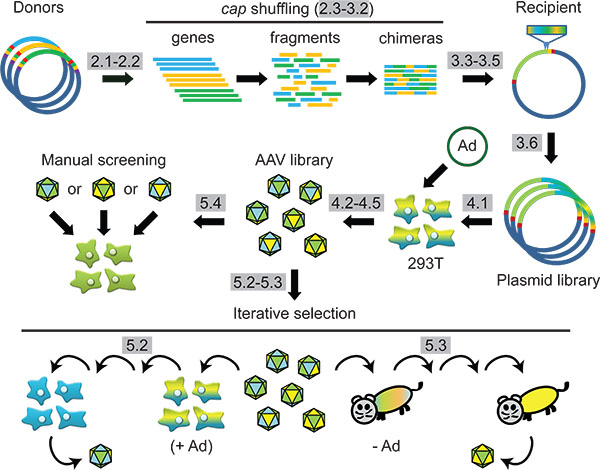
図1方式:セル内または動物のDNAファミリーシャフリングおよびその後の選択によって合成AAVキャプシドエンジニアリング。プロトコルステップは灰色で強調表示されます。
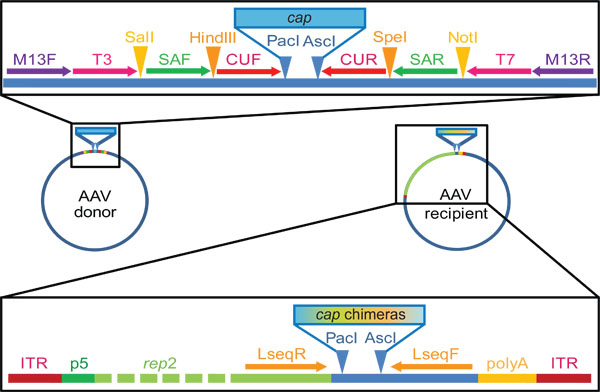
DNAシャッフリング家族とライブラリの生成のために私たちのラボで使用されている図2のAAV cap遺伝子のドナーとレシピエントプラスミド。これらは唯一の代表例であることに注意して、正確なサイトとシーケンスすることカスタマイズすることができます。プラスミド私たちの基本的なドナーは、それぞれの矢印または三角形で示されている我々が示したプライマーの結合と同様に制限酵素部位を含むように設計された市販したpBluescript II KS(+)ベクター、から派生しています。その後、 パックマン私と ASC I制限部位に隣接になるために、このプラスミドにAAV血清型1-9(プライマーCAPF / Rで増幅)のcap遺伝子をクローニングした。 T3およびT7プライマーは、ステップ2.2のキャップを分離するために使用され、再組み立てキメラ配列(ステップ3.2)のしばらくして増幅プライマー対SAF / RまたはCUF / R、またはネストされたPCRの両方のいずれかを使用して行うことができます。プラスミド複製コンピテント受信者は、AAVのp5プロモーターの制御下にAAV2 rep遺伝子に隣接するAAV逆方向末端反復配列(ITR、複製およびパッケージングシグナル)を運びます。下流のrepのパックマンIおよび ASC I制限部位は、 "フレームの"クローニングを可能にするシャッフルcap遺伝子のプール。ザ示さLseqプライマー結合部位は、 キャップ遺伝子配列(ステップ3.6)のために有用である。

図3。cap遺伝子のDNase Iをダイジェスト(AAV2、8、9)の例。 cap遺伝子の産物のアガロースゲル電気泳動分析が示すように指示された別のインキュベーション時間(分:秒)を使用してダイジェスト。レーンUコントロールとして未消化の入力コンデンサの断片プールを示しています。レーンMにおけるDNAマーカーのバンドのサイズはキロにある。この例では、理想的なダイジェスト(黄色のボックス)100から500塩基対の周りに優先する支配的なピークが得られた1時45分、または2:00分のインキュベーション時間で得られた。

図4 5つの異なる組換えYFPを発現するAAVのVEに感染した3つのヒト細胞株の顕微鏡ベースの分析の例ctors(上の名前)シャッフルcap遺伝子で作られた、ランダムにAAV2、図8及び図9に基づいて、元のライブラリから選択します。

図5 18の異なる組換えYFPを発現するAAVベクター( 図4からも含めて上の名前、)で感染した4種類の細胞のFACSベースの分析(シリアル10倍希釈時)の例ランダムに選択されたシャッフルcap遺伝子で作られた元のライブラリから(AAV2、8、9)。 YFPの発現は、容易に可視化するために色分けされました。黒は常に0%と白のそれぞれの細胞型で測定した最大数を示します。それによって現在のものです描か割合は、形質導入細胞。選択されていないカプシドから期待されることができるようにクローンB2(赤)は、貧しい人々の全体的な効果を持つクローンを例示している。 FACS分析はより敏感であり、また、顕微鏡にあまり適している懸濁細胞(例えば、SupT1など)に使用されることに注意してください。フー、ヒト;ムー、ネズミ。
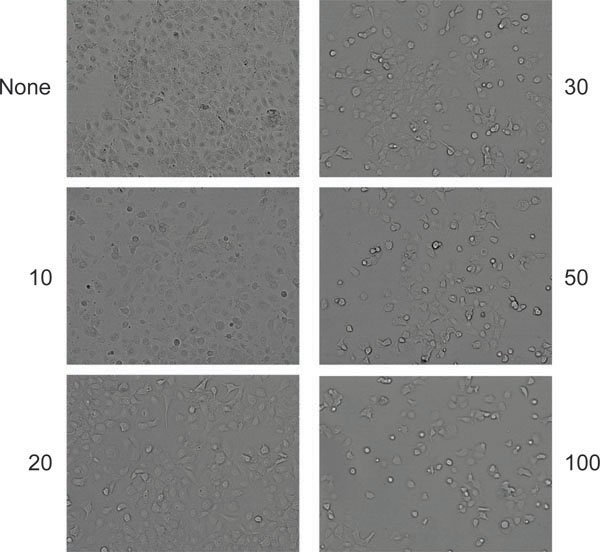
図6。AAVおよびアデノウイルスとの共感染の生産後の細胞で細胞変性効果の典型的な外観(このケースではHeLa細胞)。細胞は、どちらか感染していない左(左上パネル)またはアデノウイルスの指示量(細胞あたりの粒子)を感染させた。

図7。図書館感染と増幅のための尺度としてウエスタンブロット法によるAAVキャプシドタンパク質の発現の検出。左のブロットは、AAVライブラリとヘルパーアデノウイルスの様々な量(μl中)と共感染した細胞を示しています。最初の二つのレーンは、共同のためのよい例です。十分ではなく過度のAAV感染と増幅、したがって、希望するタイトな遺伝子型と表現型のリンケージを示す、かろうじて検出可能なAAV蛋白質の発現をもたらすnditions。したがって、これらの上清の0.1、1または10μlの新鮮な細胞の再感染(右ブロット)を使用しました。レーンCの細胞をネガティブコントロール(helpervirusの欠如のために検出可能な表現)として単独でAAVを感染させた。
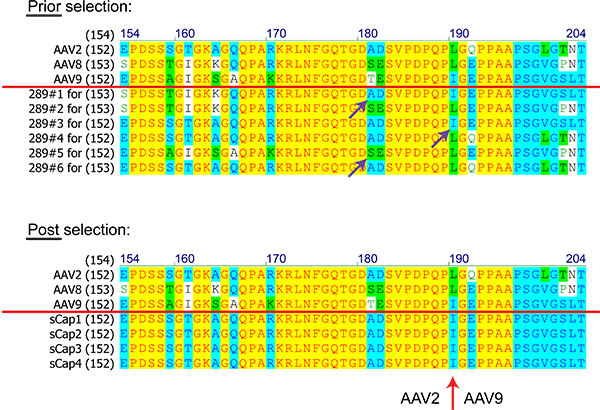
図8。選択前と後のAAV2、図8及び図9に基づいてライブラリからAAVクローンのタンパク質配列の比較(数字はアミノ酸である)。赤い線は上記の配列は親のAAVのを示しています。紫色の矢印は、相同組換え事象を示しています。赤い矢印がAAV2と、選択したすべてのクローンに記載されAAV9間のクロスオーバーをマークします。
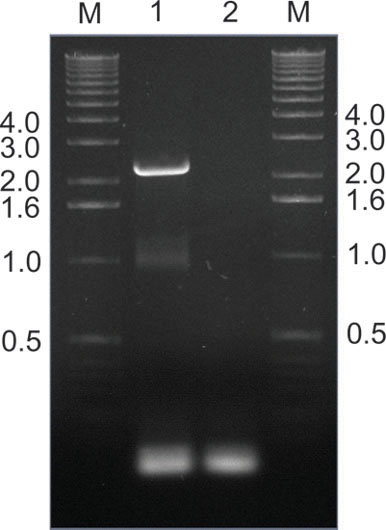
図9。<PCRによるマウス組織から正常に感染したAAVクローン/ strong>のレスキュー。この例では、AAV cap遺伝子は、マウスの肝臓からPCR増幅されたプライマー対SAF / Rを( 図2)を使用して、一週間の周辺ライブラリ注入した後に抽出した。レーン2は、非テンプレートコントロールである間、レーン1は、期待される2.2キロベースのバンドを示しています。 パックマン私と ASC I制限後、増幅した断片は、二次ライブラリのその後の生産と再注入のためのプラスミド、元の受信者( 図2)に再クローニングした。レーンMにおけるDNAマーカーのバンドのサイズはキロで表示されます。

図10培養細胞におけるキメラ濃縮AAVの優れた性能の例。クローンAAV-DJは、3の前に私達によって報告されている、それはほとんどのAAV血清型2、8と9の間のハイブリッドを表しており、館から選択されている yは、プールしたヒト血清の存在下でヒト肝細胞ではこれらの3つに加え、さらに5種の血清型を含む。 8自然のAAV血清型(1-6、上に8と9で示される)にその効率を比較するには、すべてのcap遺伝子は、自己相補的なGFP発現ベクター8,19を精製生成するために使用されました。これらはmlあたり2×10 9ベクターのゲノムを含むように正規化し、10倍希釈系列で示した細胞株を形質導入するために使用された。三日後、GFP発現細胞を計数し、感染価は考慮希釈係数をとることによって決定した。 図内のコードとは対照的である。ここでは5、暗い色は、粒子数当たりの感染性を示しています。明らかなように、選択されたAAV-DJキメラは適用された選択法の成功の一例を示す、すべての自然のAAV wildtypesよりも優れています。 fibr、線維芽細胞、HA、ハムスター、胡、ヒト;ムー、ネズミ、SI、サル。

図11マウス肝臓におけるキメラ選択されたAAVの分析のための例。クローンAAV-DNは、マウス肝癌細胞で選択して、 ルシフェラーゼを発現する組換えベクター8を生成するために使用されていました。示すように、代表的なマウス(グループあたり3つ)は、このベクトルまたは野生型AAV8、マウスの肝臓9の最も強力な既知の自然分離株のいずれかに基づいてコントロールの等しい用量の末梢投与後一週間です。 AAV-DNクローンは肝臓でわずかに全体の式(パネル(I))を与えながら、肝臓の発現レベルがされた後は、非肝組織では実質的に少ないから標的を示すので、それはこのオルガンのためのより具体的であることに注意してくださいイメージングソフトウェア(パネル(II))を介して調整されます。
ディスカッション
ここでは、DNAシャフリングを介して家族や細胞や動物の進化に不可欠な実験の手順およびAAVキャプシドエンジニアリングのためのガイドラインを概説しています。本質的には、これらのプロトコルは、2008年3我々は最初のAAVのフィールド内で報告手順の標準化バージョンです。他の人の研究のフォローアップの突風は、多くの変更、 例えば 、10から13を報告し?...
開示事項
すべての著者は、開示することは何もないことを宣言します。
謝辞
作者は感謝してハイデルベルク大学で優秀CellNetworksのクラスタで同様にチカとハインツシャラー(CHS)の基礎によって彼らのラボは、チームメンバーとの仕事の優れたサポートを認める。我々はDNAファミリーシャフリングを介して分子のAAVの進化は、三年前に我々の最初の出版以来、非常にアクティブなフィールドになるので、仕事に関連する出版物のすべての著者に謝罪しました感謝は、スペースの制約からここでは引用できませんでした。
資料
| Name | Company | Catalog Number | Comments |
| 試薬の名前 | 会社 | カタログ番号 | |
| DNアーゼI | インビトロジェン | 18068-015 | |
| ポリエチレンイミン(PEI) | Sigma-Aldrich社 | 408727 | |
| 制限酵素 | NEB | さまざまな | |
| T4 DNAリガーゼ | NEB | M0202T | |
| ゲル抽出キット | キアゲン | 28704 | |
| 备考IIポリメラーゼキット | Finnzymes(NEB) | F-540S | |
| HotStarハイファイポリメラーゼキット | キアゲン | 202602 | |
| DMSO | Finnzymes(NEB) | F-540S(キットの一部) | |
| EDTA(25mM)の | インビトロジェン | 18068-015(キットの一部) | |
| トリス | ロート | 4855.2 | |
| アンピシリンナトリウム塩 | ロート | K029.2 | |
| のdNTP(10mMの、100μl)を | インビトロジェン | 18427013 | |
| イオジキサノール(OptiPrep) | 軸シールド | 1114739 | |
| Phenolred | メルク | 107241 | |
| プラスミドメガ準備キット | キアゲン | 12181 | |
| 超遠心 | Beckman-Coulter社 | オプティマL90K | |
| クイックシールの遠心チューブ | Beckman-Coulter社 | 342414 | |
| エレクトロ·ユニット | バイオ·ラッド | GenePulserXcell | |
| サーマルサイクラー | エッペンドルフ | Vapo保護 | |
| 加熱ブロック | BIOER | MB-102 | |
| 蛍光顕微鏡 | オリンポス | IX81 | |
| FACSアナライザ | Beckman-Coulter社 | Cytomics FC500 MLP | |
| MegaX DH10B T1R細胞 | インビトロジェン | C640003 | |
| ベンゾナーゼ | メルク | 101695 | |
| アデノウイルス-5 | ATCC | VR-5 | |
| プラスミドpBluescript II KS(+) | ストラタジーン | 212207 | |
| cap5F(太字の黄色、cap5特異的配列のパック私のサイト): GACTCTTAATTAACAGGT ATGTCTTTTGTTGATCACCCTCC | IDTDNA | カスタムプライマー | |
| cap5R(緑、太字でcap5特異的配列のASC Iサイト): GTGAGGGCGCGCC TTAAAGGGGTCGGGTAAGGTATC | IDTDNA | カスタムプライマー |
参考文献
- Grimm, D., Kay, M. A. From virus evolution to vector revolution: use of naturally occurring serotypes of adeno-associated virus (AAV) as novel vectors for human gene therapy. Curr. Gene Ther. 3, 281-304 (2003).
- Grimm, D. Production methods for gene transfer vectors based on adeno-associated virus serotypes. Methods. 28, 146-157 (2002).
- Grimm, D. In vitro and in vivo gene therapy vector evolution via multispecies interbreeding and retargeting of adeno-associated viruses. J. Virol. 82, 5887-5911 (2008).
- Muller, O. J. Random peptide libraries displayed on adeno-associated virus to select for targeted gene therapy vectors. Nat. Biotechnol. 21, 1040-1046 (2003).
- Perabo, L. In vitro selection of viral vectors with modified tropism: the adeno-associated virus display. Mol. Ther. 8, 151-157 (2003).
- Zolotukhin, S., Potter, M., Hauswirth, W. W., Guy, J., Muzyczka, N. A "humanized" green fluorescent protein cDNA adapted for high-level expression in mammalian cells. J. Virol. 70, 4646-4654 (1996).
- Wobus, C. E. Monoclonal antibodies against the adeno-associated virus type 2 (AAV-2) capsid: epitope mapping and identification of capsid domains involved in AAV-2-cell interaction and neutralization of AAV-2 infection. J. Virol. 74, 9281-9293 (2000).
- Grimm, D. Fatality in mice due to oversaturation of cellular microRNA/short hairpin RNA pathways. Nature. 441, 537-541 (2006).
- Nakai, H. Unrestricted hepatocyte transduction with adeno-associated virus serotype 8 vectors in mice. J. Virol. 79, 214-224 (2005).
- Koerber, J. T., Jang, J. H., Schaffer, D. V. DNA shuffling of adeno-associated virus yields functionally diverse viral progeny. Mol. Ther. 16, 1703-1709 (2008).
- Li, W. Engineering and selection of shuffled AAV genomes: a new strategy for producing targeted biological nanoparticles. Mol. Ther. 16, 1252-1260 (2008).
- Ward, P., Walsh, C. E. Chimeric AAV Cap sequences alter gene transduction. Virology. 386, 237-248 (2009).
- Yang, L. A myocardium tropic adeno-associated virus (AAV) evolved by DNA shuffling and in vivo selection. Proc. Natl. Acad. Sci. U.S.A. 106, 3946-3951 (2009).
- Perabo, L. Combinatorial engineering of a gene therapy vector: directed evolution of adeno-associated virus. J. Gene. Med. 8, 155-162 (2006).
- Maheshri, N., Koerber, J. T., Kaspar, B. K., Schaffer, D. V. Directed evolution of adeno-associated virus yields enhanced gene delivery vectors. Nat. Biotechnol. 24, 198-204 (2006).
- Wu, Z., Asokan, A., Samulski, R. J. Adeno-associated virus serotypes: vector toolkit for human gene therapy. Mol. Ther. 14, 316-327 (2006).
- Kwon, I., Schaffer, D. V. Designer gene delivery vectors: molecular engineering and evolution of adeno-associated viral vectors for enhanced gene transfer. Pharm. Res. 25, 489-499 (2008).
- Perabo, L., Huber, A., Marsch, S., Hallek, M., Buning, H. Artificial evolution with adeno-associated viral libraries. Comb. Chem. High. Throughput. Screen. 11, 118-126 (2008).
- McCarty, D. M., Monahan, P. E., Samulski, R. J. Self-complementary recombinant adeno-associated virus (scAAV) vectors promote efficient transduction independently of DNA synthesis. Gene Ther. 8, 1248-1254 (2001).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved