Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
In Vitro Culture de l’ovule pour vivre-cellule d’imagerie du Zygote polarisation et structuration d’embryon chez Arabidopsis thaliana
Dans cet article
Résumé
Ce manuscrit décrit une in vitro ovule méthode de culture qui permet l’imagerie de cellules vivantes des Arabidopsis zygotes et des embryons. Cette méthode est utilisée pour visualiser la dynamique intracellulaire au cours de la polarisation du zygote et la spécification des cellules sort dans le développement des embryons.
Résumé
Dans la plupart des usines fleurissantes, le zygote et les embryons sont cachés profondément dans les tissus de la mère, et donc il a longtemps été un mystère de comment ils se développent dynamiquement ; par exemple, comment le zygote se polarise à établir l’axe du corps et comment l’embryon spécifie divers destins cellulaires au cours de la formation des organes. Ce manuscrit décrit une méthode in vitro ovule culture pour effectuer l’imagerie de cellules vivantes du développement des zygotes et des embryons d’Arabidopsis thaliana. Le milieu de culture optimisé permet de zygotes ou jeunes embryons d’évoluer vers des plantes fertiles. En le combinant avec un dispositif de tableau de micropillar de poly(dimethylsiloxane) (PDMS), l’ovule se tient dans le milieu liquide dans la même position. Cette fixation est cruciale d’observer l’ovule même sous un microscope pour plusieurs jours de la division zygotique à la fin du stade embryonnaire. L’imagerie de cellules vivantes qui en résulte peut être utilisé pour surveiller la dynamique en temps réel de la polarisation du zygote, tels que la migration nucléaire et réarrangement du cytosquelette et aussi le calendrier de la division cellulaire et la spécification sort des cellules au cours de la structuration de l’embryon. En outre, ce système de culture ovule peut être combiné avec des traitements d’inhibiteur pour analyser les effets de divers facteurs sur le développement de l’embryon et avec les manipulations optiques telles que des perturbations de laser d’examiner le rôle de la communication de cellule-cellule.
Introduction
Le plan de base du corps d’un organisme se développe à partir un zygote unicellulaire. Dans la plupart des usines fleurissantes, division zygotique génère une apicale et un baso-cellulaire, qui se développent dans les pousses et des racines, respectivement1. Par conséquent, il est important de comprendre comment le corps de la plante est formé au cours de l’embryogenèse, mais il n’a pas été un outil efficace d’observer directement la dynamique de vie zygotes et des embryons parce qu’ils développent profondément dans la fleur. Chez plusieurs espèces de monocotylédones, telles que le maïs et le riz, une méthode de fécondation in vitro a été établie2,3. Dans cette méthode, isolé des spermatozoïdes et ovules sont soudés électriquement ou chimiquement, et la cellule générée peut évoluer vers une plante fertile. Cependant, chez les plantes dicotylédones, il n’y a aucune méthode de fécondation in vitro qui peut produire des embryons correcte, sans doute en raison de l’état non synchronisé le cycle cellulaire des gamètes mâles et femelles4,5. En outre, le tissu qui entoure l’embryon (endosperme) joue un rôle important dans le développement embryonnaire6.
Dans une espèce de dicotylédones modèle, Arabidopsis, une méthode de culture in vitro a été développée en se concentrant sur l’ovule entier, qui contient l’embryon et l’endosperme7. Ce système a été utilisé avec succès pour analyser les effets de divers réactifs chimiques sur l’embryogenèse, mais il n’est pas adapté pour Time-lapse imagerie parce qu’il a un faible taux de survie. Par conséquent, un système d’élevage de l’ovule roman in vitro a été développé afin de commencer dès le stade du zygote et de produire des plantes fertiles à un ratio élevé8. Après divers essais, on a trouvé ce moyen de Nitsch et tréhalose a considérablement amélioré le taux de survie des ovules8. En outre, parce que l’ovule se développe comme il se développe et donc souvent s’éloigne du champ d’observation du microscope, un dispositif PDMS a été développé pour corriger l’ovule dans la moyenne de9. Le dispositif PDMS a permis l’imagerie à long terme pour les 3-4 jours, qui est suffisant pour suivre le développement d’un zygote à un embryon de coeur-scène. En utilisant cette méthode, il devient possible de visualiser la dynamique de la polarisation du zygote et de l’embryon le patterning, non seulement dans des conditions normales, mais également en présence d’inhibiteurs chimiques ou dans divers milieux mutant8,10 ,11.
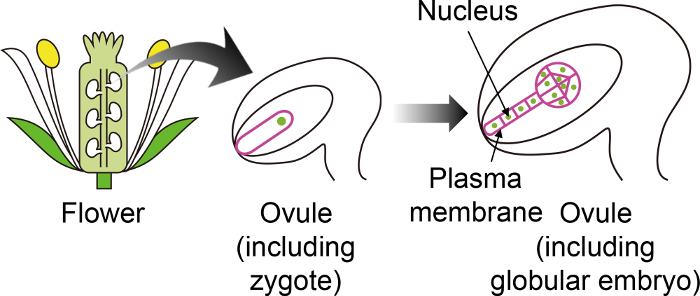
Figure 1 : Schéma des marqueurs fluorescents spécifiques permettant de visualiser des Zygotes et des embryons dans l’Ovule.
L’Arabidopsis zygote se développe en embryon dans l’ovule, qui est généré à l’intérieur de la fleur. Dans ce système de culture in vitro , le zygote et les embryons sont observés par le biais de l’ovule, et il est donc important d’utiliser des marqueurs fluorescents spécifiques qui ne sont pas exprimées dans d’autres tissus de l’ovule. Veuillez cliquer ici pour visionner une version agrandie de cette figure.
Protocole
1. préparation du milieu de Culture In Vitro Ovule
- faire le milieu liquide pour l’in vitro culture ovule (" N5T moyenne ") contenant 1 x Nitsch basale mélange de sel, 5 % (p/v) dihydrate de tréhalose, 0,05 % (p/v) 2-(N-morpholino) ethanesulfonic acide (MES)-KOH (pH 5,8) et 1 x Gamborg ' solution de vitamines de s.
- Ajuster le pH à 5,8 avec KOH.
- Stériliser le milieu autoclavage (121 ° C, 20 min) ou filtrer à travers un filtre de 0,22 µm.
Remarque : Le milieu stérilisé peut être stocké à 4 ° C pendant 2 à 3 mois.
2. Préparation de l’appareil tableau de PDMS Micropillar
- coupe l’appareil de micropillar PDMS par un couteau, une lame de rasoir ou de ciseaux pour s’insérer dans la partie en verre (14 mm φ) d’un plat de fond en verre de 35 mm.
Remarque : La procédure complète de la construction du dispositif PDMS est décrit dans les précédents documents 8 , 9, et donc les détails sont omises ici. Des plats avec des volumes plus petits, tels que des plaques de 4 puits ou 96 puits, fond en verre peuvent être utilisés pour l’imagerie à court terme sans le dispositif de. - Stériliser le dessus de l’appareil PDMS sous lumière UV pendant 15 min.
- Tourner le dispositif PDMS par à l’aide de la pince à épiler pointe carrée et gardez-le sous exposition aux UV lumière pendant 15 min stériliser bas.
- Transférer le dispositif stérilisé de PDMS dans une boîte de Petri de 35 mm et ajouter le support de N5T jusqu'à ce que l’appareil est complètement trempée dans le milieu (environ 5-7 mL).
- Mettre le plat dans une chambre à vide et réduire la pression pour dégazer. Gardez passer l’aspirateur pendant 3 h pour la nuit jusqu'à ce que l’air dans l’appareil est remplacé par le milieu.
Remarque : L’appareil peut être enlevé par des bulles d’air sous un vide fort. - Prendre le périphérique de la capsule en la tenant avec une pince à épiler pointe carrée et mettez-le sur du papier absorbant pour enlever le support supplémentaire sur le côté et le bas.
NOTE : le support sur le tableau de micropillar (surface supérieure) ne doit pas être supprimé car il sera utilisé pour la culture de l’ovule. - L’appareil sur un verre de lame 76 x 26 mm, veiller à préserver la partie micropillar dans la partie supérieure, de transfert et de recouvrir l’appareil avec 35 mm culture plat couvercle afin d’empêcher le milieu ne se dessèchent pas lors de l’extraction d’ovule suivant.
3. Silique Dissection et Extraction d’ovules
- stériliser une aiguille (0,40 mm G) et des pinces fines en l’essuyant avec l’éthanol à 70 %. L’aiguille est facilement manipulé en l’attachant à un bâton de bois ou de seringue.
- Vérifier l’inflorescence de la plante, puis sélectionnez les siliques appropriées pour l’expérience. Les siliques mm environ 5 contiennent des zygotes et des siliques de 8-10 mm comprennent jeunes embryons globulaires.
- D’accise les siliques à l’aide de pinces à épiler et placez-les sur le ruban adhésif double-face sur un verre de lame 76 x 26 mm. Une silique contient environ 40-60 ovules et 3-4 siliques (c.-à-d., les ovules de 120-240) sont suffisants pour un seul appareil.
- Ouvrir la silique (paroi de l’ovaire) pour voir les ovules à l’intérieur à l’aide de l’aiguille stérilisée et pince à épiler sous un stéréomicroscope. Couper seulement la paroi de l’ovaire afin que les ovules ne sont pas endommagés.
- Transférer la silique ouvert dans le milieu de la N5T sur le périphérique PDMS (préparé à l’étape 2.7) et libérer les ovules dans le milieu avec l’aiguille ou pincettes.
- , Mettez une petite lamelle couvre-objet (18 x 18 mm) sur le dispositif PDMS pour pousser les ovules dans les espaces dans le tableau micropillar.
- Enlever le couvercle en verre en le tirant horizontalement à l’aide de pince à épiler ou avec les doigts pour éliminer le milieu extra.
- Retournez l’appareil PDMS, placez-le dans un plat de fond en verre de 35 mm et appuyez légèrement sur l’appareil en poussant à l’aide de la pince à épiler pointe carrée pour le coller sur la vitre.
- Milieu N5T versez doucement dans le fond en verre plat par décantation la bouteille moyenne jusqu'à ce que le dispositif PDMS est complètement trempé dans le milieu et sceller le plat avec le film de paraffine.
Remarque : Le dispositif PDMS peut être recyclé, mais les dispositifs qui sont trop vieux sont facilement enlevés le fond du verre. En outre, le périphérique peut flotter si elle n’est pas complètement dégazé dans étape 2.5 ou le milieu est coulé trop vite.
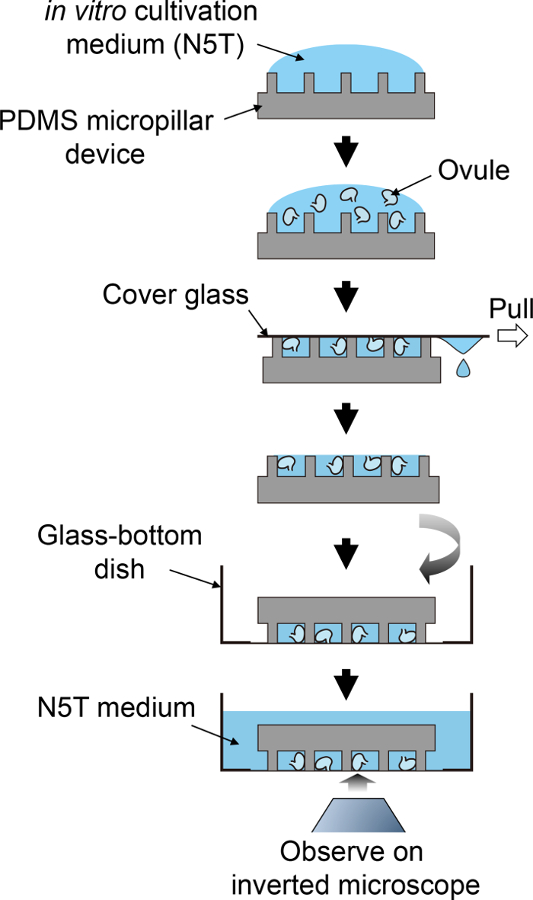
figure 2 : Schéma procédure de préparation de l’échantillon.
Ce flux schématique correspond aux étapes 3,5 à 4,1. s’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
4. décomposée Imaging
- Placer le plat de fond en verre qui a été préparé à l’étape 3.9 sur un microscope inversé et sélectionner les ovules convenables de se concentrer sur.
NOTE : Parce que les ovules varient dans le stade, la position et la direction, ovules adaptés devraient être trouvées en vérifiant leur fluorescence marqueur. - Commencer le live d’image selon le fabricant ' s directives du système microscope.
Remarque : Les paramètres et le microscope appareils sont diverses, et ainsi, les utilisateurs doivent choisir les paramètres appropriés qui conviennent à l’objectif de l’expérience. À titre d’exemple, les paramètres utilisés dans ce manuscrit sont énumérés au tableau 1.
| équipement/réglage | Figure 3 (A) et 1 de vidéos supplémentaires et 4 | Figure 3 (B) et 2 vidéo supplémentaire | Supplemental vidéo 3 |
| Microscope | à balayage laser microscope (A1R MP) inversé | tourne disque confocal microscope inversé (CSU-W1) | caisson inversé système de microscope confocal avec une chambre d’incubation stable (CV1000) |
| Laser | TI : Sapphire laser à impulsions femtosecondes | 488 nm et 561 nm laser LD | 488 nm et 561 nm laser LD |
| O bjectif lentille | 40 X objectif à immersion dans l’eau (NA = 1.15) avec le milieu d’immersion | 60 X objectif d’immersion de l’huile silicone (NA = 1,30), monté sur un lecteur de focus Piezo | 40 X objectif (NA = 0,95) |
| Detect ou | externes non-descanned PMT GaAsP détecteur | caméra EMCCD | caméra EMCCD |
| miroir dichroïque | DM495 et DM560 | DM488/561 | DM400-410/488/561 |
| filtre | filtres passe-bande ; 520/35 nm et 593/46 nm | filtres passe-bande ; 520/50 nm et 617/73 nm | |
| tranche le long de l’axe z | z 31 - cheminées avec des intervalles de 1 µm | inter 17 z-piles avec 3 µm Vals | 7 z-piles avec des intervalles de 5 µm |
| intervalle de temps | 20 min | 5 min | 10 min |
tableau 1 : les Systèmes de microscope et paramètres utilisés dans ce manuscrit.
Les microscopes et les paramètres sont diverses, et ainsi, chaque utilisateur doit choisir le système adéquat pour l’expérience.
- Vérifier la séquence d’images acquises et le convertir au format film générales, tels que .avi ou .mov pour présentation.
Remarque : Si le logiciel de microscope ne peut pas sortie les images comme un format de film, il est possible d’enregistrer les images sous .tif et puis ouvrez-les dans ImageJ, un programme open source inspiré par les NIH Image, changer le type de fichier. ImageJ est disponible à : https://imagej.nih.gov/ij/. ImageJ peut également être utilisé pour faire un Z-sTuck film, tels que la projection de l’intensité maximale, tel que décrit au : https://imagej.net/Z-functions.
Résultats
Grâce à ce système de culture ovule, cette méthode peut suivre la dynamique de la vie de polarisation du zygote et de structuration de l’embryon. Il s’agit d’une réalisation parce qu’auparavant il n’y avait aucune technique pour visualiser le comportement en temps réel le zygote et de l’embryon, qui sont cachés profondément dans les tissus de la mère. Figure 3 a et supplémentaire 1 vidéo montrent que les microtubules (M...
Discussion
Ce manuscrit présente un simple in vitro ovule culture protocole qui est efficace pour une utilisation dans l’imagerie de cellules vivantes du développement des zygotes et des embryons.
La conception de l’appareil PDMS peut être nécessaire d’optimisation en fonction du stade de l’embryon. Le premier appareil développé était un tableau de microcage pour régler l’orientation et de fixer la position des ovules9, et ensuite un dispositif de microp...
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Microscopie dans ce travail a été menée à l’Institut de transformatrices biomolécules (WPI-ITbM) de l’Université de Nagoya et soutenue par le Japon Advanced Plant Science Network. Ce travail a été soutenu par les bourses de la Japan Science and Technology Agency (projet ERATO T.H. et M.U.) et la société japonaise pour la Promotion de la Science : une subvention pour la recherche scientifique sur les domaines innovants (Nos. JP24113514, JP26113710, JP15H05962 et JP15H05955 pour M.U. et amendements. JP16H06465, JP16H06464 et JP16K21727 pour T.H), une subvention pour les jeunes scientifiques (B, Nos. JP24770045 et JP26840093 pour M.U.) et une subvention de recherche exploratoire difficiles (no JP16K14753 pour M.U.).
matériels
| Name | Company | Catalog Number | Comments |
| Nitsch basal salt mixture | Duchefa | N0223 | |
| trehalose dihydrate | Wako Pure Chemical | 206-18455 | |
| MES | Dojindo | 345-01625 | |
| Gamborg’s vitamin solution | Sigma-Aldrich | G1019 | |
| 35-mm glass-bottom dish | Matsunami Glass | D111300 | |
| 35 mm culture dish | Corning | 430588 | |
| PDMS | Dow Corning Co. | Sylgard184 | |
| 76 × 26 mm slide glass | Matsunami Glass | S1225 | |
| 18 × 18 mm slide glass | Matsunami Glass | C018181 | |
| needle (gauge 0.40 mm) | Terumo | NN-2719S | |
| Immersion medium Immersol W 2010 | Zeiss | 444969-0000-000 | |
| A1R MP | Nikon | A1RsiMP(1080) Ti-E-TIRF | |
| CSU-W1 | Yokogawa Electric | It is a customized equipment, and thus Catalog Number is not avairable. | |
| CV1000 | Yokogawa Electric | CV1000-SP84 |
Références
- Natesh, S., Rau, M. A., Johri, B. M. . Embryology of Angiosperms. , 377-443 (1984).
- Kranz, E., Lorz, H. In vitro fertilisation of maize by single egg and sperm cell protoplast fusion mediated by high calcium and high pH. Zygote. 2 (2), 125-128 (1994).
- Uchiumi, T., Uemura, I., Okamoto, T. Establishment of an in vitro fertilization system in rice (Oryza sativa L.). Planta. 226 (3), 581-589 (2007).
- Sun, M. X., Moscatelli, A., Yang, H. Y., Cresti, M. In vitro double fertilization in Nicotiana tabacum (L.): the role of cell volume in cell fusion. Sex Plant Rep. 13 (4), 225-229 (2001).
- Tian, H. Q., Yuan, T., Russell, S. D. Relationship between double fertilization and the cell cycle in male and female gametes of tobacco. Sex Plant Rep. 17 (5), 243-252 (2004).
- Costa, L. M., et al. Central cell-derived peptides regulate early embryo patterning in flowering plants. Science. 344 (6180), 168-172 (2014).
- Sauer, M., Friml, J. In vitro culture of Arabidopsis embryos within their ovules. Plant J. 40 (5), 835-843 (2004).
- Gooh, K., et al. Live-cell imaging and optical manipulation of Arabidopsis early embryogenesis. Dev Cell. 34 (2), 242-251 (2015).
- Park, J., Kurihara, D., Higashiyama, T., Arata, H. Fabrication of microcage arrays to fix plant ovules for long-term live imaging and observation. Sens Act B-Chem. 191, 178-185 (2014).
- Kimata, Y., et al. Cytoskeleton dynamics control the first asymmetric cell division in Arabidopsis zygote. Proc Natl Acad Sci U S A. 113 (49), 14157-14162 (2016).
- Nambo, M., et al. Combination of Synthetic Chemistry and Live-Cell Imaging Identified a Rapid Cell Division Inhibitor in Tobacco and Arabidopsis thaliana. Plant Cell Physiol. 57 (11), 2255-2268 (2016).
- Mizuta, Y., Kurihara, D., Higashiyama, T. Two-photon imaging with longer wavelength excitation in intact Arabidopsis tissues. Protoplasma. 252 (5), 1231-1240 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon