È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
In Vitro Coltivazione dell'ovulo per Live cell Imaging di zigote polarizzazione e Patterning di embrione in Arabidopsis thaliana
In questo articolo
Riepilogo
Questo manoscritto descrive un in vitro dell'ovulo metodo di coltivazione che permette l'imaging di cellule vive di Arabidopsis zigoti ed embrioni. Questo metodo viene utilizzato per visualizzare le dinamiche intracellulare durante la polarizzazione di zigote e la specifica di destino delle cellule nello sviluppo di embrioni.
Abstract
Nella maggior parte delle piante da fiore, lo zigote ed embrione sono nascosti nel profondo del tessuto di madre, e così è stato a lungo un mistero di come si sviluppano in modo dinamico; ad esempio, come lo zigote polarizza per stabilire l'asse del corpo e come l'embrione specifica vari destini delle cellule durante la formazione dell'organo. Questo manoscritto descrive un in vitro dell'ovulo cultura metodo per eseguire la formazione immagine di cellule vive di sviluppare zigoti ed embrioni di Arabidopsis thaliana. Il medium di coltivazione ottimizzato permette di zigoti o embrioni in anticipo a crescere nelle piante fertili. Attraverso la combinazione con un dispositivo di matrice micropillar poly(dimethylsiloxane) (PDMS), l'ovulo si svolge nel liquido di coltura nella stessa posizione. Questo sistema di fissaggio è fondamentale per osservare l'ovulo stesso sotto un microscopio per diversi giorni dalla divisione zygotic per la fine della fase embrionale. L'imaging di vivere-cella risultante consente di monitorare le dinamiche in tempo reale di polarizzazione di zigote, quali migrazione nucleare e riarrangiamento del citoscheletro e anche i tempi di divisione cellulare e specifica di destino delle cellule durante il patterning di embrione. Inoltre, questo sistema di coltivazione dell'ovulo possa essere combinato con trattamenti inibitore per analizzare gli effetti di vari fattori per lo sviluppo dell'embrione e con ottiche manipolazioni come rottura di laser per esaminare il ruolo della comunicazione cellula-cellula.
Introduzione
Il piano di base del corpo di un organismo si sviluppa da uno zigote unicellulare. Nella maggior parte delle piante da fiore, divisione zygotic genera un apicale e delle cellule basali, che si sviluppano nella radice, rispettivamente1e sparare. Pertanto, è importante capire come il corpo di pianta si forma durante l'embriogenesi, ma non c'è stato uno strumento efficace per osservare direttamente le dinamiche della vita zigoti ed embrioni perché si sviluppano nel profondo il fiore. In varie specie di monocotiledoni, come mais e riso, un metodo di fecondazione in vitro è stato stabilito2,3. In questo metodo, le cellule uovo e dello sperma isolato sono fuse elettricamente o chimicamente, e la cella generata può svilupparsi in una pianta fertile. Tuttavia, nelle piante dicotiledoni, non c'è nessun in vitro fertilizzazione metodo che può produrre embrioni corretta, presumibilmente a causa dello stato non sincronizzate ciclo cellulare di gameti maschili e femminili4,5. Inoltre, il tessuto che circonda embrione (endosperma) svolge i ruoli importanti nell'embrione di sviluppo6.
In una specie di modello dicotilene, a. thaliana, un metodo di coltivazione in vitro è stato sviluppato focalizzando l'attenzione sull'ovulo intero, che contiene l'embrione e l'endosperma7. Questo sistema è stato usato con successo per analizzare gli effetti di vari agenti chimici su embriogenesi, ma non è adatto per l'imaging di time-lapse perché ha un basso tasso di sopravvivenza. Di conseguenza, un sistema di coltivazione del ovulo romanzo in vitro è stato sviluppato al fine di avviare più presto la stadio di zigote e producono piante fertili in un alto rapporto8. Dopo varie prove, è stato trovato quel mezzo di Nitsch e trealosio ha migliorato significativamente il tasso di sopravvivenza di ovuli8. Inoltre, perché l'ovulo si espande come cresce e così spesso si sposta lontano dal campo di osservazione del microscopio, un dispositivo PDMS è stato sviluppato per risolvere l'ovulo in media9. Il dispositivo PDMS abilitato l'imaging a lungo termine per 3-4 giorni, che è sufficiente per tracciare lo sviluppo da uno zigote, un embrione di cuore-fase. Utilizzando questo metodo, diventa possibile visualizzare le dinamiche di polarizzazione di zigote ed embrione patterning, non solo in condizioni normali, ma anche in presenza di inibitori chimici o in vari ambiti di provenienza mutante8,10 ,11.
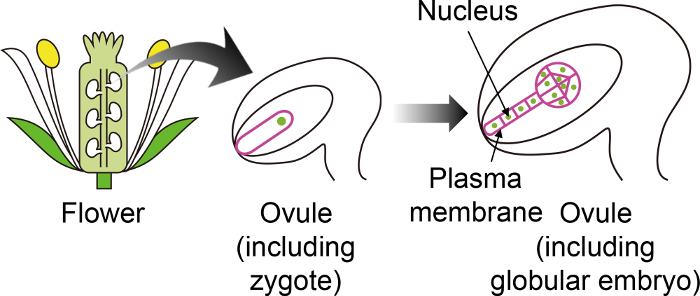
Figura 1: Diagramma schematico dei marcatori fluorescenti specifici utilizzati per visualizzare zigoti ed embrioni attraverso l'ovulo.
Lo zigote Arabidopsis si sviluppa in un embrione nell'ovulo, che viene generato profondo all'interno del fiore. In questo sistema in vitro coltura, lo zigote ed embrione sono osservati attraverso l'ovulo, e quindi è importante utilizzare marcatori fluorescenti specifici che non sono espressi in altri tessuti dell'ovulo. Clicca qui per visualizzare una versione più grande di questa figura.
Protocollo
1. preparazione del terreno di coltura In Vitro dell'ovulo
- rendono il mezzo liquido per la coltura in vitro di ovulo (" N5T medio ") contenenti 1 x Nitsch tecnica basale miscela di sale, 5% (p/v) trealosio diidrato, 0.05 % (p/v) 2-(N-morfolino) ethanesulfonic acido (MES)-KOH (pH 5,8) e 1 x Gamborg ' soluzione di vitamine di s.
- Aggiustare il pH a 5,8 con KOH.
- Sterilizzare il mezzo in autoclave (121 ° C, 20 min) o filtrandola attraverso un filtro da 0,22 µm.
Nota: Il mezzo sterilizzato può essere memorizzato a 4 ° C per 2-3 mesi.
2. Preparazione del dispositivo matrice PDMS Micropillar
- tagliare il dispositivo di micropillar PDMS di un coltello, lama di rasoio o forbici per adattarsi con la parte di vetro (14mm φ) di un piatto di vetro-fondo di 35 mm.
Nota: La procedura completa della costruzione dispositivo PDMS è descritto in precedenti documenti 8 , 9, e così i dettagli sono omessi qui. Piatti con fondo di vetro con volumi più piccoli, ad esempio 4-pozzetti o 96 pozzetti piastre, possono essere utilizzati per l'imaging a breve termine senza il dispositivo. - Sterilizzare il lato superiore del dispositivo PDMS sotto luce UV per 15 min.
- Accendere l'apparecchio PDMS su utilizzando una pinzetta punta quadrata e tenerlo sotto UV luce per 15 min sterilizzare fondo.
- Trasferire il dispositivo PDMS sterilizzato in una piastra di coltura di 35 mm e aggiungere il supporto di N5T fino a quando il dispositivo è completamente imbevuto nel medio (circa 5-7 mL).
- Mettere il piatto in una camera a vuoto e ridurre la pressione a degas. Mantenere l'aspirazione per 3 h a tutta la notte fino a quando l'aria nel dispositivo viene sostituita dal mezzo.
Nota: Il dispositivo può essere staccato da bolle d'aria sotto una forte depressione. - Prendere il dispositivo dal piatto, tenendola con le pinzette a punta quadrata e metterlo su un tovagliolo di carta per rimuovere il mezzo supplementare sulla parte laterale e inferiore.
Nota: il mezzo sulla matrice micropillar (superficie superiore) non deve essere rimosso perché questo sarà utilizzato per la coltivazione di ovulo. - Trasferire il dispositivo su un vetro da diapositiva 76 x 26 mm, garantendo per mantenere la parte di micropillar come il lato superiore e coprire l'apparecchio con il coperchio di piatto di cultura 35 mm per evitare il terreno dall'asciugarsi durante la successiva estrazione dell'ovulo.
3. Anemofila dissezione ed estrazione dell'ovulo
- sterilizzare un ago (mm 0,40 G) e pinzette bene strofinando con etanolo al 70%. L'ago è facilmente gestibile collegandolo a un bastone di legno o siringa.
- Verifica l'infiorescenza della pianta e selezionare i corretto silique per l'esperimento. Le silique circa 5 mm contengono zigoti, e le silique di 8-10 mm includono giovani embrioni globulari.
- Accise i silique utilizzando una pinzetta e metterli su nastro biadesivo su un vetro di diapositiva 76 x 26 mm. Un anemofila contiene circa 40-60 ovuli e 3-4 silique (cioè, 120-240 ovuli) sono sufficienti per un unico dispositivo.
- Aprire l'anemofila (parete dell'ovaia) per vedere gli ovuli all'interno utilizzando l'ago sterilizzato e pinzette sotto un microscopio stereoscopico. Tagliare solo la parete dell'ovaia, in modo che gli ovuli non siano danneggiati.
- Trasferire l'anemofila aperto nel mezzo di N5T sul dispositivo PDMS (preparato al punto 2.7) e rilasciare gli ovuli nel mezzo con l'ago o una pinzetta.
- Mettere una piccola copertura vetrata (18 x 18 mm) sul dispositivo PDMS per spingere gli ovuli negli spazi nella matrice micropillar.
- Togliere il vetro di copertura tirandolo orizzontalmente con una pinzetta o le dita per rimuovere medium extra.
- Capovolgere il dispositivo PDMS, metterlo in un piatto fondo di vetro 35mm e premere leggermente il dispositivo spingendo con le pinzette punta quadrata per attaccarlo al vetro.
- Medie N5T versare delicatamente in vetro-fondo piatto di decantazione la bottiglia fino a quando il dispositivo PDMS è completamente imbevuto nel mezzo e sigillare il piatto con la pellicola di paraffina.
Nota: Il dispositivo PDMS può essere riciclato, ma dispositivi che sono troppo vecchi si staccano facilmente dalla parte inferiore del vetro. Inoltre, il dispositivo può galleggiare se esso non è completamente degassato nel passaggio 2.5 o il mezzo viene versato troppo in fretta.
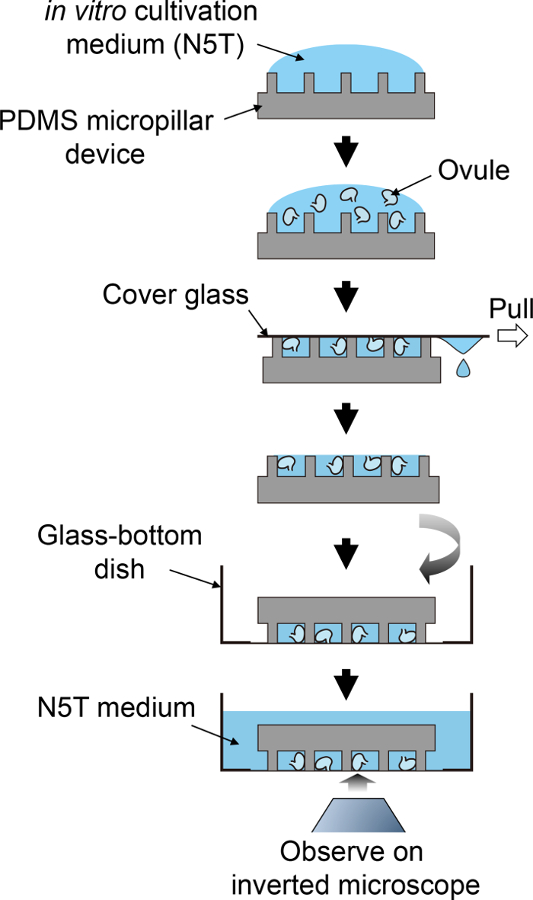
Figura 2: procedura schematica per la preparazione del campione.
Questo flusso schematico corrisponde ai passaggi 3.5 a 4.1. Clicca qui per visualizzare una versione più grande di questa figura.
4. Time-lapse Imaging
- posizionare il piatto con fondo di vetro che è stato preparato in passaggio 3.9 su un microscopio invertito e selezionare adatti ovuli di concentrarsi su.
Nota: Perché gli ovuli variano in fase, la posizione e la direzione, ovuli adatti dovrebbero essere trovati controllando loro fluorescenza marcatore. - Avviare la live-imaging secondo il produttore ' s istruzioni del sistema microscopio.
Nota: La strumentazione del microscopio e i parametri sono diversi, e così gli utenti devono scegliere le impostazioni corrette che corrispondono allo scopo di esperimento. Ad esempio, le impostazioni utilizzate in questo manoscritto sono elencate nella tabella 1.
| attrezzature/impostazione | supplementare video 1, figura 3 (A) e 4 | Figura 3 (B) e supplementare Video 2 | supplementare Video 3 |
| microscopio | microscopio (A1R MP) invertito a scansione laser | disco rotante invertito il microscopio confocale (CSU-W1) | casella-tipo invertito sistema di microscopio confocale con una camera di incubazione stabile (CV1000) |
| Laser | ti femtosecond laser di impulso | laser LD 488 nm e 561 nm | 488 nm e 561 nm laser LD |
| O biettivi lente | 40 X obiettivo di immersione in acqua (NA = 1.15) con mezzo immersione | 60 X obiettivo obiettivo ad immersione in silicone (NA = 1.30), montato su un'unità di messa a fuoco Piezo | 40 X Diametro obiettivo (NA = 0.95) |
| Detect o | esterne non descanned GaAsP PMT rivelatore | telecamera EMCCD | fotocamera EMCCD |
| dicroiche specchio | DM495 e DM560 | DM488/561 | DM400-410/488/561 |
| filtro | filtri passa-banda; 520/35 nm e 593/46 nm | filtri passa-banda; 520/50 nm e 617/73 nm | |
| fetta lungo asse z | 31 z-stack con intervalli da 1 µm | inter 17 z-stack con 3 µm Vals | 7 z-stack con intervalli di 5-µm |
| intervallo di tempo | 20 min | 5 min | 10 min |
tabella 1: il Sistemi di microscopia e le impostazioni utilizzate in questo manoscritto.
I microscopi e i parametri sono diversi, e così ogni utente dovrebbe scegliere il sistema adatto per l'esperimento.
- controllare la sequenza di immagini acquisite e convertirlo nel formato di film generale, come. avi o. MOV per presentazione.
Nota: Se il software di microscopio non può emettere le immagini come un film in formato, è possibile salvare le immagini come TIF e quindi aprirli in ImageJ, un programma open source ispirato da NIH Image, per modificare il tipo di file. ImageJ è disponibile presso: https://imagej.nih.gov/ij/. ImageJ può essere utilizzato anche per fare un Z-sInfilare il film, come la proiezione di massima intensità, come descritto in: https://imagej.net/Z-functions.
Risultati
Utilizzando questo sistema di coltivazione dell'ovulo, questo metodo può tracciare la dinamica vita di polarizzazione di zigote e patterning di embrione. Questo è un risultato, perché in precedenza non c'era nessuna tecnica per visualizzare il comportamento in tempo reale di zigote ed embrione, che sono nascosti nel profondo del tessuto di madre. Figura 3A e supplementare Video 1 mostrano che i microtubuli (MTs) nello zigote giovane si acc...
Discussione
Questo manoscritto presenta un semplice in vitro dell'ovulo coltivazione protocollo che è efficiente per l'uso nell'imaging di cellule vive di sviluppare zigoti e degli embrioni.
Il design del dispositivo PDMS potrebbe bisogno di ottimizzazione secondo allo stadio embrionale. Il primo dispositivo sviluppato era una gamma di microcage per regolare l'orientamento e per fissare la posizione di ovuli9, e quindi un dispositivo di micropillar è stato costruito per ...
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Microscopia in questo lavoro è stato condotto presso l'Istituto di Transformative Bio-molecole (WPI-ITbM) dell'Università di Nagoya e supportata dal Giappone Advanced Plant Science Network. Questo lavoro è stato sostenuto da sovvenzioni dal Japan Science and Technology Agency (progetto ERATO T.H. e M.U.) e la Japan Society per la promozione della scienza: una sovvenzione per la ricerca scientifica in settori innovativi (Nos. JP24113514, JP26113710, JP15H05962 e JP15H05955 per M.U. e Nos. JP16H06465, JP16H06464 e JP16K21727 per Tartaglia), una sovvenzione per giovani scienziati (B, Nos. JP24770045 e JP26840093 per M.U.) e una sovvenzione per impegnativa ricerca esplorativa (No. JP16K14753 per M.U.).
Materiali
| Name | Company | Catalog Number | Comments |
| Nitsch basal salt mixture | Duchefa | N0223 | |
| trehalose dihydrate | Wako Pure Chemical | 206-18455 | |
| MES | Dojindo | 345-01625 | |
| Gamborg’s vitamin solution | Sigma-Aldrich | G1019 | |
| 35-mm glass-bottom dish | Matsunami Glass | D111300 | |
| 35 mm culture dish | Corning | 430588 | |
| PDMS | Dow Corning Co. | Sylgard184 | |
| 76 × 26 mm slide glass | Matsunami Glass | S1225 | |
| 18 × 18 mm slide glass | Matsunami Glass | C018181 | |
| needle (gauge 0.40 mm) | Terumo | NN-2719S | |
| Immersion medium Immersol W 2010 | Zeiss | 444969-0000-000 | |
| A1R MP | Nikon | A1RsiMP(1080) Ti-E-TIRF | |
| CSU-W1 | Yokogawa Electric | It is a customized equipment, and thus Catalog Number is not avairable. | |
| CV1000 | Yokogawa Electric | CV1000-SP84 |
Riferimenti
- Natesh, S., Rau, M. A., Johri, B. M. . Embryology of Angiosperms. , 377-443 (1984).
- Kranz, E., Lorz, H. In vitro fertilisation of maize by single egg and sperm cell protoplast fusion mediated by high calcium and high pH. Zygote. 2 (2), 125-128 (1994).
- Uchiumi, T., Uemura, I., Okamoto, T. Establishment of an in vitro fertilization system in rice (Oryza sativa L.). Planta. 226 (3), 581-589 (2007).
- Sun, M. X., Moscatelli, A., Yang, H. Y., Cresti, M. In vitro double fertilization in Nicotiana tabacum (L.): the role of cell volume in cell fusion. Sex Plant Rep. 13 (4), 225-229 (2001).
- Tian, H. Q., Yuan, T., Russell, S. D. Relationship between double fertilization and the cell cycle in male and female gametes of tobacco. Sex Plant Rep. 17 (5), 243-252 (2004).
- Costa, L. M., et al. Central cell-derived peptides regulate early embryo patterning in flowering plants. Science. 344 (6180), 168-172 (2014).
- Sauer, M., Friml, J. In vitro culture of Arabidopsis embryos within their ovules. Plant J. 40 (5), 835-843 (2004).
- Gooh, K., et al. Live-cell imaging and optical manipulation of Arabidopsis early embryogenesis. Dev Cell. 34 (2), 242-251 (2015).
- Park, J., Kurihara, D., Higashiyama, T., Arata, H. Fabrication of microcage arrays to fix plant ovules for long-term live imaging and observation. Sens Act B-Chem. 191, 178-185 (2014).
- Kimata, Y., et al. Cytoskeleton dynamics control the first asymmetric cell division in Arabidopsis zygote. Proc Natl Acad Sci U S A. 113 (49), 14157-14162 (2016).
- Nambo, M., et al. Combination of Synthetic Chemistry and Live-Cell Imaging Identified a Rapid Cell Division Inhibitor in Tobacco and Arabidopsis thaliana. Plant Cell Physiol. 57 (11), 2255-2268 (2016).
- Mizuta, Y., Kurihara, D., Higashiyama, T. Two-photon imaging with longer wavelength excitation in intact Arabidopsis tissues. Protoplasma. 252 (5), 1231-1240 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon