Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Fermentations contrôlées par la lumière pour la production de produits chimiques microbiens et de protéines
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Le contrôle optogénétique du métabolisme microbien offre un contrôle dynamique flexible des processus de fermentation. Le protocole montre ici comment mettre en place des fermentations régulées par la lumière bleue pour la production de produits chimiques et de protéines à différentes échelles volumétriques.
Résumé
Les usines de cellules microbiennes offrent une alternative durable pour la production de produits chimiques et de protéines recombinantes à partir de matières premières renouvelables. Cependant, surcharger un micro-organisme avec des modifications génétiques peut réduire la condition physique et la productivité de l’hôte. Ce problème peut être surmonté en utilisant un contrôle dynamique: expression inductible des enzymes et des voies, généralement à l’aide d’additifs chimiques ou nutritifs, pour équilibrer la croissance et la production cellulaires. L’optogénétique offre une méthode non invasive, hautement accordable et réversible de régulation dynamique de l’expression des gènes. Ici, nous décrivons comment mettre en place des fermentations contrôlées par la lumière d’Escherichia coli et de Saccharomyces cerevisiae pour la production de produits chimiques ou de protéines recombinantes. Nous discutons de la façon d’appliquer la lumière à des moments et des dosages sélectionnés pour découpler la croissance et la production microbiennes afin d’améliorer le contrôle de la fermentation et la productivité, ainsi que des principales considérations d’optimisation pour de meilleurs résultats. De plus, nous décrivons comment mettre en œuvre des contrôles de lumière pour des expériences de bioréacteur à l’échelle du laboratoire. Ces protocoles facilitent l’adoption de contrôles optogénétiques dans les micro-organismes modifiés pour améliorer les performances de fermentation.
Introduction
L’optogénétique, le contrôle des processus biologiques avec des protéines sensibles à la lumière, offre une nouvelle stratégie pour contrôler dynamiquement les fermentations microbiennes pour la production chimique et protéique1,2. Le fardeau des voies métaboliques modifiées et la toxicité de certains intermédiaires et produits nuisent souvent à la croissance cellulaire3. De tels stress peuvent entraîner une mauvaise accumulation de biomasse et une productivité réduite3. Ce défi peut être relevé en divisant temporellement les fermentations en une phase de croissance et de production, qui consacrent des ressources métaboliques à l’accumulation de biomasse ou à la synthèse de produits respectivement4. Nous avons récemment montré que la transition de la croissance à la production dans cette fermentation en deux phases peut être induite par des changements dans les conditions d’éclairage5,6,7. La haute accordabilité, la réversibilité et l’orthogonalité des apports lumineux8 offrent des avantages uniques aux fermentations contrôlées par la lumière qui sont difficiles ou impossibles à reproduire avec des inducteurs chimiques utilisés dans le contrôle dynamique des fermentations biphasées conventionnelles4,9,10,11.
La protéine EL222 sensible à la lumière bleue dérivée d’Erythrobacter litoralis a été utilisée pour développer plusieurs circuits optogénétiques pour l’ingénierie métabolique chez Saccharomyces cerevisiae5,7,12,13. EL222 contient un domaine de capteur lumière-oxygène-tension (LOV) qui subit un décalage conformationnel lors de l’activation de la lumière bleue (465 nm), ce qui lui permet de se lier à sa séquence d’ADN apparenté (C120)13. La fusion d’EL222 au domaine d’activation viral VP16 (VP16-EL222) donne un facteur de transcription sensible à la lumière bleue qui peut activer de manière réversible l’expression génique chez S. cerevisiae7 et d’autres organismes14 à partir du promoteur synthétique PC120. Plusieurs circuits basés sur EL222 ont été développés et utilisés pour la production chimique chez S. cerevisiae, tels que le système OptoEXP de base activé par la lumière7, dans lequel le gène d’intérêt est directement exprimé à partir de PC120 (Figure 1A). Cependant, les préoccupations de pénétration de la lumière aux densités cellulaires élevées généralement rencontrées dans la phase de production des fermentations nous ont motivés à développer des circuits inversés induits dans l’obscurité, tels que les circuits OptoINVRT et OptoQ-INVRT (Figure 1B)5,7,13. Ces systèmes exploitent les régulons de galactose (GAL) ou d’acide quinique (Q) de S. cerevisiae et N. crassa, respectivement, contrôlant leurs répresseurs correspondants (GAL80 et QS) avec VP16-EL222, pour réprimer l’expression des gènes dans la lumière et l’induire fortement dans l’obscurité. La combinaison des circuits OptoEXP et OptoINVRT permet un contrôle bidirectionnel de l’expression des gènes, permettant des fermentations en deux phases dans lesquelles la phase de croissance est induite par la lumière bleue et la phase de production par l’obscurité (Figure 2A)5,7.
L’utilisation de la lumière au lieu de l’obscurité pour induire l’expression des gènes pendant la phase de production élargirait considérablement les capacités des contrôles optogénétiques, mais nécessiterait également de surmonter les limites de pénétration de la lumière des densités cellulaires élevées généralement rencontrées dans cette phase de fermentation. À cette fin, nous avons développé des circuits, connus sous le nom d’OptoAMP et OptoQ-AMP, qui amplifient la réponse transcriptionnelle à la stimulation par la lumière bleue. Ces circuits utilisent des mutants de type sauvage ou hypersensibles de VP16-EL222 pour contrôler la production des activateurs transcriptionnels Gal4p ou QF2 des régulons GAL ou Q, respectivement, obtenant une sensibilité accrue et une expression génique plus forte avec la lumière12,13 (Figure 1C). Les circuits OptoAMP peuvent obtenir une induction lumineuse complète et homogène dans des bioréacteurs de 5 L à une densité optique (mesurée à 600 nm; OD600) valeurs d’au moins 40 avec seulement ~ 0,35% d’éclairage (dose de lumière de 5% sur seulement ~ 7% de la surface en vrac). Cela démontre un degré de sensibilité plus élevé par rapport à OptoEXP, qui nécessite un éclairage proche de 100 %12. La capacité d’induire efficacement l’expression des gènes avec de la lumière à des densités cellulaires élevées ouvre de nouvelles possibilités de contrôle dynamique des fermentations. Cela inclut l’exploitation de fermentations dans plus de deux phases temporelles, telles que les fermentations triphasées, dans lesquelles les phases de croissance, d’induction et de production sont établies avec des calendriers d’éclairage uniques pour optimiser la production chimique (Figure 2B)12.

Figure 1 : Circuits optogénétiques pour le contrôle dynamique de S. cerevisiae. Les circuits OptoEXP, OptoINVRT et OptoAMP sont basés sur le système sensible à la lumière VP16-EL222. (A) Dans le circuit OptoEXP, l’exposition à la lumière bleue provoque un changement conformationnel et une dimérisation de VP16-EL222, ce qui expose un domaine de liaison à l’ADN et permet la transcription à partir de PC120. La figure a été modifiée à partir de Zhao et al.7. (B) Les circuits OptoINVRT exploitent les régulons GAL (illustrés) ou Q pour induire une expression dans l’obscurité. Dans les circuits à base de GAL, VP16-EL222 et GAL4 sont exprimés de manière constitutive, tandis que le PC120 entraîne l’expression du répresseur GAL80 (dans les circuits à base de Q, GAL4 et GAL80 sont remplacés par QF2 et QS, respectivement, et un promoteur synthétique contenant DU QUAS est utilisé à la place d’un promoteur GAL). À la lumière, Gal80p empêche l’activation du gène d’intérêt de PGAL1. Dans l’obscurité, GAL80 n’est pas exprimé et se dégrade rapidement en le fusionnant en un domaine degron constitutif (petit domaine brun), ce qui permet l’activation de PGAL1 par Gal4p. La figure a été modifiée à partir de Zhao et al.5. (C) Les circuits OptoAMP utilisent également VP16-EL222 pour contrôler les régulons GAL (illustrés) ou Q. Dans ces circuits, le répresseur GAL80 (ou QS) est exprimé de manière constitutive et fusionné à un degron photosensible (petit domaine bleu) assurant une répression stricte dans l’obscurité. PC120 et un mutant hypersensible VP16-EL222 contrôlent l’expression de GAL4 (ou QF2) avec la lumière, ce qui active fortement PGAL1 (ou un promoteur contenant QUAS) dans la lumière. Les circuits dérivés de GAL peuvent utiliser des formes techniques de PGAL1, telles que PGAL1-M ou PGAL1-S, qui ont une activité accrue, ainsi que des promoteurs de type sauvage contrôlés par le régulateur GAL (PGAL1, PGAL10, PGAL2, PGAL7). La figure a été modifiée à partir de Zhao et al.12. Veuillez cliquer ici pour voir une version plus grande de cette figure.

Figure 2: Fermentations biphasées et triphasées dans le temps. (A) Les fermentations biphasées opérées avec des circuits inversés se composent d’une phase de croissance entraînée par la lumière et d’une phase de production sombre. Dans la phase de croissance, la biomasse s’accumule à mesure que la voie de production reste réprimée. Lorsqu’elles atteignent l’OD600 souhaitée, les cellules sont déplacées vers l’obscurité pour s’ajuster métaboliquement avant d’être remises en suspension dans des milieux frais pour la phase de production. (B) Dans un processus en trois phases, les phases de croissance, d’incubation et de production sont définies par des horaires lumineux uniques, qui peuvent consister en une période de croissance sombre, une incubation pulsée et une phase de production entièrement éclairée. Figure créée avec Biorender. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Des circuits optogénétiques ont également été développés pour le contrôle dynamique de la production chimique et protéique chez E. coli. Les circuits OptoLAC contrôlent le répresseur LacI bactérien à l’aide du circuit pDawn sensible à la lumière, basé sur le système à deux composants YF1/FixJ6 (Figure 3). Semblables à OptoINVRT5, les circuits OptoLAC sont conçus pour réprimer l’expression des gènes dans la lumière et l’induire dans l’obscurité. Les niveaux d’expression utilisant les circuits OptoLAC peuvent égaler ou dépasser ceux atteints avec l’induction standard de l’isopropyle β-d-1-thiogalactopyranoside (IPTG), maintenant ainsi la force de l’induction chimique tout en offrant une accordabilité et une réversibilité améliorées6. Par conséquent, les circuits OptoLAC permettent un contrôle optogénétique efficace pour l’ingénierie métabolique chez E. coli.
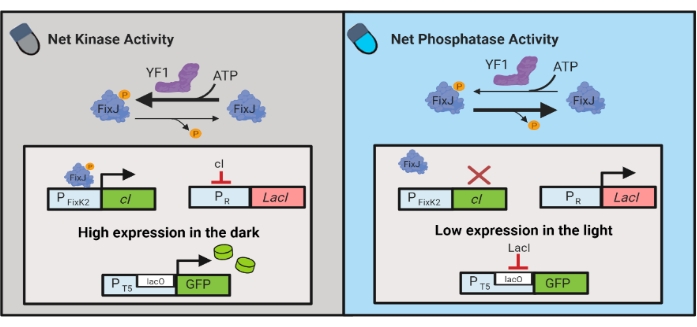
Figure 3 : Circuits OptoLAC pour le contrôle dynamique d’E. coli. Les circuits OptoLAC adaptent le système pDawn et l’opéron lac pour réaliser l’activation dans l’obscurité et la répression dans la lumière. Dans l’obscurité, YF1 phosphoryle FixJ, qui active ensuite le promoteur PFixK2 pour exprimer le répresseur cI. Le répresseur cI empêche l’expression du répresseur lacI à partir du promoteur PR, ce qui permet la transcription du gène d’intérêt à partir d’un promoteur contenant du lacO. Inversement, la lumière bleue réduit l’activité de la kinase nette YF1, inversant la phosphorylation fixJ et donc l’expression cI, ce qui déprime l’expression de lacI et empêche l’expression du promoteur contenant du lacO. La figure a été modifiée à partir de Lalwani et al.6. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Nous décrivons ici les protocoles de base pour les fermentations contrôlées par la lumière de S. cerevisiae et E. coli pour la production chimique ou protéique. Pour les levures et les bactéries, nous nous concentrons d’abord sur les fermentations avec une phase de croissance entraînée par la lumière et une phase de production induite par l’obscurité rendue possible par les circuits OptoINVRT et OptoLAC. Par la suite, nous décrivons un protocole pour une fermentation triphasée (croissance, induction, production) contrôlée par la lumière rendue possible par les circuits OptoAMP. En outre, nous décrivons comment étendre les fermentations contrôlées optogénétiquement des microplaques aux bioréacteurs à l’échelle du laboratoire. Avec ce protocole, nous visons à fournir un guide complet et facilement reproductible pour effectuer des fermentations contrôlées par la lumière pour la production de produits chimiques ou de protéines.
Protocole
1. Production chimique contrôlée par la lumière à l’aide du circuit S. cerevisiae OptoINVRT7
- Construction de la souche
- Obtenir une souche avec l’auxotrophie his3 , car ce marqueur est nécessaire pour la plupart des plasmides OptoINVRT existants5. Si vous cherchez une régulation optogénétique d’un gène natif de S. cerevisiae, construisez une souche dans laquelle toute copie endogène du gène est supprimée.
- Linéariser le plasmide contenant le circuit OptoINVRT7, tel que EZ-L4395, et l’intégrer dans le his3-locus de la souche auxotrophe en utilisant des méthodes de transformation lithium-acétate standard15. Si vous utilisez le plasmide EZ-L439, qui contient les composants pour réprimer PGAL1 à la lumière et l’activer dans l’obscurité, linéariser au site de restriction PmeI.
- Après la transformation, centrifuger les cellules à 150 x g pendant 1 min et remettre doucement en suspension dans 200 μL de milieu complet synthétique (SC-His) frais d’histidine.
- Plaquer tout le volume cellulaire sur des plaques de gélose SC-His et incuber à 30 °C pendant 2-3 jours jusqu’à l’apparition de colonies.
- Préparez des cellules compétentes à partir de cette souche à l’aide de protocoles de transformation de l’acétate de lithium standard et transformez-les avec un plasmide contenant le ou les gènes à contrôler optogénétiquement en aval du promoteur PGAL1-M ou PGAL1-S5.
REMARQUE: L’utilisation d’un plasmide qui s’intègre à δ sites (YARCdelta5) et sélectionne avec Zeocin permet une intégration multicopique stable7,16,17,18. - Après transformation, centrifuger la culture à 150 x g pendant 1 min et remettre doucement en suspension dans 200 μL de milieu frais de chute SC.
REMARQUE: Le promoteur PGAL1-M est une version synthétique du promoteur PGAL1 avec les sites de répression Mig1p supprimés, tandis que PGAL1-S est une version technique de PGAL1-M, qui a des sites de liaison d’activateurs Gal4p supplémentaires. Le promoteur PGAL1 régulier peut être utilisé pour contrôler l’expression; cependant, la force d’expression sera inférieure à celle de ces promoteurs techniques. - Plaquer tout le volume cellulaire sur une plaque de gélose peptone dextrose (YPD) extrait de levure si elle s’intègre dans des sites δ, ou sur une plaque de décrochage SC si elle se transforme avec un plasmide contenant un marqueur de sélection. Incuber à 30 °C pendant 16 h sous une lumière bleue constante pour maintenir le gène contrôlé optogénétiquement réprimé.
REMARQUE : Pour certaines souches, les colonies peuvent croître plus rapidement lorsqu’elles sont incubées dans des impulsions de lumière bleue (p. ex., 1 s marche/79 s éteint, 5 s marche/75 s éteint, 10 s marche/70 s éteint, etc.) plutôt que dans un éclairage constant, qui doit être déterminé expérimentalement pour chaque souche, si nécessaire. - Utilisez n’importe quelle source de lumière de 465 nm et placez un panneau LED ~ 40 cm au-dessus de la plaque de sorte que l’intensité lumineuse soit d’environ 80-110 μmol / m2 / s. Mesurez l’intensité à l’aide d’un compteur quantique (voir Tableau des matériaux).
- Si vous intégrez des sites δ, créez une réplique de la plaque sur des plaques YPD contenant une plage de concentrations de Zeocin comprise entre 400 μg / mL et 1 200 μg / mL pour sélectionner une variété de numéros de copie d’intégration5,7,12,16,17,18. Incuber les plaques de réplique à 30 °C sous une lumière bleue constante ou pulsée pendant 2-3 jours jusqu’à l’apparition des colonies.
- Sélection préliminaire des meilleures colonies
- Sélectionnez huit colonies dans chaque plaque et utilisez-les pour inoculer 1 mL de milieu SC-His complété par 2% de glucose dans des puits individuels d’une plaque de 24 puits. Croître dans des plaques de 24 puits sous les cellules pendant la nuit (16-20 h) à 30 °C avec des secousses à 200 tr/min (diamètre orbital de 19 mm) sous un éclairage constant de lumière bleue.
- Le lendemain matin, diluer chaque culture dans 1 mL d’un milieu SC-His frais avec 2 % de glucose à des valeurs OD600 allant de 0,01 à 0,3 et croître dans des plaques de 24 puits sous une lumière constante ou pulsée à 30 °C avec des secousses à 200 tr/min jusqu’à ce qu’elles atteignent des densités cellulaires comprises entre 2 et 9 valeurs OD600 (Figure 4A). Le temps nécessaire pour cette phase de croissance dépendra de la souche.
- Incuber les plaques dans l’obscurité pendant 4 h à 30 °C avec une secousse à 200 tr/min en éteignant le panneau lumineux et en enveloppant les plaques dans du papier d’aluminium.
REMARQUE: Cette étape permet aux cellules de passer métaboliquement à la phase de production avant la remise en suspension dans le milieu de production. - Pour commencer la phase de production, centrifugez les cultures dans la plaque de 24 puits à 234 x g pendant 5 min et remettez en suspension les cellules dans 1 mL de milieu de chute SC frais avec 2% de glucose. Scellez les plaques pour éviter l’évaporation du produit souhaité en utilisant du ruban d’étanchéité stérile à microplaques.
- Fermenter les plaques scellées à l’obscurité pendant 48 h à 30 °C en agitant à 200 tr/min. Assurez-vous que les plaques sont enveloppées dans du papier d’aluminium pour éviter toute exposition à la lumière.
REMARQUE: L’emballage des plaques dans du papier d’aluminium ne limite pas la disponibilité de l’oxygène ou du gaz dans les fermentations; cependant, le ruban d’étanchéité stérile limite le transfert de gaz. De petits trous peuvent être percés dans le ruban adhésif pour introduire de l’oxygène, si nécessaire.
- Récolte et analyse
- Pour récolter les fermentations, centrifuger les plaques pendant 5 min à 234 x g et transférer 800 μL du surnageant dans des tubes de microcentrifugation de 1,5 mL.
- Selon le produit chimique d’intérêt, analysez en utilisant la chromatographie liquide à haute performance (CLHP), la chromatographie en phase gazeuse-spectrométrie de masse (GC-MS) ou une autre méthode analytique utilisant la technique de préparation de l’échantillon la mieux adaptée à l’instrument utilisé.
2. Production de protéines contrôlée par la lumière à l’aide du système OptoLAC d’E. coli
- Construction de la souche
- Co-transformez l’électrocompétent BL21 DE3 ΔlacI ΔlacI-DE3 avec un plasmide contenant le circuit OptoLAC1B ou OptoLAC2B6 et un plasmide qui exprime la protéine recombinante d’intérêt du promoteur PT719.
- Après transformation, récupérer les cellules pendant 1 h dans 1 mL de bouillon super optimal avec répression catabolisante (SOC ; 2 % de tryptone, 0,5 % d’extrait de levure, 10 mM de NaCl, 2,5 mM de KCl, 10 mM MgCl2, 10 mM de MgSO4, 20 mM de glucose) à 37 °C avec rotation ou agitation.
REMARQUE : Le plasmide contenant la protéine d’intérêt doit être compatible avec le plasmide OptoLAC (c.-à-d. différent marqueur de résistance et origine de la réplication) et ne doit pas contenir de copie de lacI. - Centrifuger les cellules à 4845 x g pendant 3 min et concentrer la pastille dans 200 μL de bouillon de lysogénie (LB). Plaquez les cellules concentrées entières sur une plaque de gélose LB avec des antibiotiques appropriés et poussez à 37 ° C pendant la nuit sous une lumière bleue constante pour maintenir l’expression des protéines réprimée.
- Dépistage initial pour vérifier l’expression
- Prenez trois colonies individuelles et utilisez-les pour inoculer 1 mL de milieu LB avec des antibiotiques appropriés dans des puits individuels d’une plaque de 24 puits. Croître pendant la nuit (16-20 h) à 37 °C avec des secousses à 200 tr/min sous un éclairage constant de lumière bleue (Figure 4A).
- Le lendemain, utilisez 1,5 μL de culture pour mesurer l’OD600 dans un spectrophotomètre avec une mesure en microvolume. Diluer les cultures en 1 mL de LB frais dans des plaques de 24 puits à des valeurs OD600 allant de 0,01 à 0,1.
- Cultivez les cultures à 37 °C avec 200 tr/min en secouant sous la lumière bleue pendant 2-3 h. À partir de la deuxième heure, prenez des mesures OD600 toutes les 15 minutes pour vous assurer que les cultures ne dépassent pas la plage OD600 de 0,1 à 1,5.
- Une fois que les cultures sont à l’OD600 souhaité, éteignez le panneau lumineux et enveloppez la plaque dans du papier d’aluminium pour lancer la phase de production. Gardez la plaque dans l’obscurité pendant 8 h (37 °C), 20 h (30 °C) ou 48 h (18 °C), avec une secousse à 200 tr/min.
- Mesurez et enregistrez la valeur OD600 finale de chaque culture.
- Récolte et analyse
- Transférer 800 μL de chaque culture dans un tube de microcentrifugation de 1,5 mL et centrifuger pendant 5 min à 17 000 x g.
- Remettre en suspension la pastille de la cellule dans 200 μL de tampon de remise en suspension (Tris 50 mM, pH 8,0, NaCl 300 mM).
REMARQUE: La concentration de NaCl peut être ajustée en fonction de la protéine recombinante analysée. - Ajouter 50 μL de tampon d’échantillon de dodécylsulfate de sodium (SDS) 6x (Tris 375 mM, pH 6,8, FDS 9 %, glycérol 50 %, bleu de bromophénol 0,03 %, TNT 9 %). Incuber à 100 °C pendant 10 min en agitant à 700 tr/min dans un thermomixeur.
- Chargez ~3-20 μL de la culture dans un gel SDS-PAGE à 12%. En utilisant la mesure finale de l’OD600 comme guide, chargez approximativement la même quantité de protéines pour chaque échantillon (équivalent à 10 μL de l’échantillon correspondant à une valeur finale de l’OD600 de 1). Utilisez une alimentation pour exécuter l’électrophorèse à 100 V jusqu’à ce que le gel soit complètement résolu.
- Tachez le gel avec la solution de G-250 bleu brillant coomassie en chauffant pendant 30 à 40 s dans un four à micro-ondes, puis en incubant sur un rotateur de plate-forme pendant au moins 15 minutes.
- Rincez à l’eau désionisée deux fois et détachez-vous sur un rotateur de plate-forme pendant au moins 30 minutes (ou toute la nuit), en ajoutant deux lingettes nettoyantes attachées dans un nœud pour aider à absorber la tache. Faire bouillir le gel dans une quantité suffisante d’eau dans le four à micro-ondes pendant 15 minutes pour accélérer le processus de conservation.
3. Fermentation triphasée à l’aide du système OptoAMP de S. cerevisiae
- Construction de la souche
- Obtenir une souche avec un marqueur auxotrophique his3 , car ce marqueur est nécessaire pour utiliser les plasmides OptoAMP existants5. Si vous cherchez une régulation optogénétique d’un gène originaire de S. cerevisiae, construisez une souche dans laquelle la copie endogène de ce gène est supprimée.
- Linéariser un plasmide contenant le circuit OptoAMP4, tel que EZ-L58012, et l’intégrer dans le locus his3 de la souche auxotrophique en utilisant des méthodes standard de transformation lithium-acétate15. Si vous utilisez EZ-L580, linéariser le plasmide au site de restriction PmeI.
- Après la transformation, centrifuger les cellules à 150 x g pendant 1 min et remettre doucement en suspension dans 200 μL de milieu FRAIS SC-His.
- Plaquer tout le volume cellulaire sur un milieu sélectif (SC-His-agar) et incuber à 30 °C pendant 2-3 jours jusqu’à l’apparition de colonies.
- Préparez des cellules compétentes à partir de cette souche et transformez-les avec un plasmide contenant le(s) gène(s) à contrôler optogénétiquement en aval du promoteur PGAL1-S12.
REMARQUE: L’utilisation d’un plasmide qui s’intègre à δ sites et sélectionne avec Zeocin permet une intégration et une sélection multicopiques stables. - Après transformation, centrifuger la culture à 150 x g pendant 1 min et remettre doucement en suspension dans 200 μL de milieu frais de chute SC.
REMARQUE: Le promoteur PGAL1-S est une version synthétique du promoteur PGAL1 dans laquelle les sites de répression Mig1p sont supprimés et des sites de liaison d’activateurs Gal4p supplémentaires sont ajoutés. Le promoteur PGAL1 régulier peut être utilisé; cependant, la force d’expression sera inférieure à celle de ce promoteur conçu. - Plaquer tout le volume de la cellule sur une plaque de gélose YPD ou SC-dropout et incuber à 30 °C pendant 16 h dans l’obscurité (enveloppé dans du papier d’aluminium). L’incubation dans l’obscurité maintient le gène contrôlé optogénétiquement réprimé, ce qui permet aux cellules de diriger leurs ressources métaboliques vers la croissance cellulaire plutôt que vers la production chimique.
- En cas d’intégration dans δ-sites, répliquer la plaque sur des plaques YPD contenant une gamme de concentrations de Zeocin à sélectionner pour une variété de numéros de copie d’intégration. Incuber les plaques à 30 °C dans l’obscurité (enveloppées dans du papier d’aluminium) pendant 2-3 jours jusqu’à l’apparition de colonies.
- Sélection préliminaire des meilleures colonies
- Sélectionnez huit colonies dans chaque plaque et utilisez-les pour inoculer 1 mL de milieu SC-His avec 2% de glucose dans des puits individuels d’une plaque de 24 puits. Cultivez les cellules pendant la nuit (16-20 h) dans l’obscurité à 30 °C avec des secousses à 200 tr/min.
- Le lendemain matin, diluer chaque culture dans 1 mL de milieu FRAIS SC-His avec 2% de glucose à 0,1 OD600 et pousser dans l’obscurité à 30 °C avec une agitation de 200 tr/min jusqu’à ce qu’ils atteignent un OD600 de 3. Enveloppez les plaques dans du papier d’aluminium pour éviter toute exposition à la lumière. Le temps nécessaire pour cette phase de croissance dépendra de la souche.
- Pour démarrer la phase d’induction, incuber les plaques sous une lumière pulsée (par exemple, 5 s marche/95 s éteint) pendant 12 h à 30 °C avec une secousse de 200 tr/min. Utilisez n’importe quelle source lumineuse de 465 nm et placez un panneau LED au-dessus de la plaque de manière à ce que l’intensité lumineuse soit d’environ 80-110 μmol/m2/s pour des résultats optimaux (Figure 4A).
REMARQUE: La durée optimale de l’impulsion lumineuse pour cette incubation variera en fonction du produit chimique produit. Il est recommandé de filtrer une gamme d’horaires de lumière allant de 0,1% (par exemple, 1 s sur 999 s off) à 100% (pleine lumière). - Pour commencer la phase de production, centrifugez les cultures à 234 x g pendant 5 min et remettez-les en suspension dans un milieu SC-His frais avec 2% de glucose. Scellez les plaques pour éviter l’évaporation du produit souhaité à l’aide d’un ruban d’étanchéité stérile à microplaques.
- Fermenter les plaques scellées à la lumière pendant 48 h à 30 °C en agitant à 200 tr/min. Optimisez le programme d’éclairage pendant cette étape, car certains produits chimiques bénéficient d’une phase de production pulsée plutôt que d’une pleine lumière.
- Récolte et analyse
- Récolter les fermentations en centrifugant les plaques pendant 5 min à 234 x g et en transférant 800 μL du surnageant dans des tubes de microcentrifugation de 1,5 mL.
- Selon le produit chimique d’intérêt, analysez à l’aide de la CLHP, de la GC-MS ou d’une autre méthode analytique utilisant la technique de préparation de l’échantillon la mieux adaptée à l’instrument utilisé.
4. Production chimique (mévalonate) à partir d’E. coli dans un bioréacteur contrôlé par la lumière
- Inoculation initiale et configuration du bioréacteur
- Inoculer une colonie d’une souche d’E. coli conçue avec une production chimique contrôlée par la lumière en 5 mL de sels minimaux M9 (3,37 mM Na2HPO4, 2,2 mM KH2PO4, 0,855 mM NaCl, 0,935 mM NH4Cl) complétés par 0,2 % p/v d’acides casaminés, 5 % p/v de glucose et mélange de métaux traces20 (0,0084 g/L EDTA, 0,0025 g/L coCl2, 0,015 g/L MnCl2, 0,0015 g/L CuCl2, 0,003 g/L H3BO3, 0,0025 g/L Na2MoO4, 0,008 g/L ZnCl2, 0,06 g/L de citrate Fe (III), 0,0045 g/L de thiamine, 1,3 g/L MgSO4) dans un tube conique de 50 mL.
- Cultivez la culture pendant la nuit à 30 °C avec des secousses à 200 tr/min sous un éclairage de lumière bleue.
- Installez la plaque de tête du réservoir du bioréacteur, en vous assurant que les orifices suivants sont installés: insert pour sonde thermique; sonde d’oxygène dissous (OD); sparger de gaz - se connecter à la source d’air à travers un filtre de 0,2 μm; roue; condenseur de gaz - se connecter à un filtre de 0,2 μm; ligne de refroidissement (x2); lignes d’alimentation (x2) - une pour l’ajout de milieu, une pour le contrôle du pH; ligne d’échantillonnage - s’assurer qu’elle atteint le fond du navire; port vide; Sonde de pH - calibrer la sonde avec des étalons de pH = 4 et pH = 7 avant l’installation, soit autoclaver avec le reste du récipient, soit stériliser avec de l’éthanol à 95% et insérer aseptiquement avant la configuration.
- Remplissez le récipient avec 1 L d’eau filtrée, fixez la plaque de tête et serrez-la en vous assurant que le joint torique s’adapte et se scelle parfaitement.
- Couvrez toutes les ouvertures dans le réacteur avec du papier d’aluminium.
- Préparez trois bandes de tubes: une pour enlever l’eau, une pour insérer l’aliment et une pour contrôler le pH. Couvrez les extrémités avec du papier d’aluminium et enveloppez tous les tubes dans du papier d’aluminium.
REMARQUE: NH4OH, qui est utilisé pour l’ajustement du pH, ne s’écoule pas correctement dans les tubes en silicone, ce qui peut entraîner des débits inexacts et une sur-basification de la culture. Pour éviter ce problème, utilisez des tubes de pompe biocompatibles (BPT) pour l’alimentation NH4OH. - Autoclavez le bioréacteur et le tube, en utilisant un cycle liquide de 30 minutes.
- Retirez les matériaux de l’autoclave. Une fois suffisamment froid pour être manipulé, connectez la roue, les sondes de pH et d’OD, la source d’air, l’entrée et la sortie du condenseur, ainsi que l’entrée et la sortie de refroidissement à la station de contrôle.
- Insérez la sonde thermique et couvrez le récipient avec une gaine chauffante. Fixez la veste vers le haut du récipient pour éviter de bloquer la culture de l’exposition à la lumière.
- Connectez l’un des tubes stériles à la conduite d’échantillonnage et fixez-le à travers la pompe d’échantillonnage. Placez l’autre extrémité de manière à ce qu’elle s’écoule dans un récipient vide pouvant contenir au moins 1 L. Drainez l’eau à l’intérieur du récipient.
- Connectez un autre tube stérile à l’une des conduites d’alimentation et fixez-le à travers l’une des pompes d’alimentation. Connectez l’autre extrémité du tube à une bouteille de support M9. Introduisez le média dans le réacteur.
- Connectez un autre tube stérile à l’une des conduites d’alimentation et fixez-le à travers l’une des pompes d’alimentation. Connectez l’autre extrémité du tube à la bouteille contenant 28% -30% nh4OH.
ATTENTION : Le NH4OH est corrosif. Travaillez dans une hotte pendant le transfert dans un biberon et assurez-vous que le biberon est placé dans un confinement secondaire. - Placez trois panneaux lumineux dans une formation triangulaire à environ 20 cm du réacteur, en vérifiant que les intensités lumineuses à la surface de la cuve atteignent environ 80 à 110 μmol/m2/s de chaque côté (figure 4B).
- Le lendemain, allumez le système de bioréacteur et le refroidisseur. Réglez le point de consigne de la température du réacteur à 37 °C, le point de consigne du pH à 7,0 et l’agitation à 200 tr/min. La veste chauffante doit s’allumer.
- Étalonnez la sonde DO en attendant d’abord que la température et les mesures DO deviennent constantes (ce sera le point de consigne à 100%). Ensuite, déconnectez la sonde du système (ce sera le point de consigne de 0%). Répétez l’opération jusqu’à ce que la mesure DO se stabilise à 100 % lorsque la sonde est connectée, puis réglez le point de consigne DO sur 20 %.
- Production chimique contrôlée par la lumière
- Inoculer le bioréacteur à une OD600 initiale de 0,001-0,1. Allumez les panneaux lumineux pour lancer la croissance.
- Après 3 h, commencez à prélever des échantillons de la ligne d’échantillonnage pour prendre des mesures OD600 afin d’éviter de dépasser la densité cellulaire optimale d’induction (ρs). Une fois que le ρs optimal est atteint (la valeur optimale pour la production de mévalonate est de 0,17), éteignez les panneaux lumineux, recouvrez le réacteur de papier d’aluminium et enveloppez l’installation dans un tissu noir pour lancer la phase de production sombre.
- Ajouter 50 μL d’antimousse, 8 h après le passage à l’obscurité. Dévissez l’orifice vide et pipetez l’antimousse directement dans le réacteur.
- Utilisez le port d’échantillonnage pour prélever périodiquement des échantillons pour l’analyse HPLC ou GC.
- Démontage et analyse
- Une fois l’expérience terminée, éteignez le système. Dévissez soigneusement les sondes d’OD et de pH et lavez-les à l’eau et au savon. Dévissez la plaque de tête et lavez-la avec de l’eau et du savon à l’aide d’une brosse.
- Transférer la culture dans un récipient vide et ajouter de l’eau de Javel à une concentration finale de 10 % v/v. Placer dans une hotte et jeter après 30 min.
- Lavez la cuve du réacteur avec de l’eau et du savon à l’aide d’une brosse.
- Préparer les échantillons pour l’analyse en fonction du produit d’intérêt. Pour la production de mévalonate, mélanger 560 μL de culture avec 140 μL de 0,5 M HCl et vortex à grande vitesse pendant 1 min. Cela convertit le mévalonate en (±)-mévalonolactone.
ATTENTION : HCl est un danger pour la santé. Manipulez avec un EPI approprié et assurez-vous que les tubes d’échantillon sont correctement bouchés avant le vortex. - Centrifuger à 17 000 x g pendant 45 min à 4 °C. Transférer 250 μL du surnageant dans un flacon HPLC.
- Pour le mévalonate, analysez les échantillons à l’aide d’une colonne d’échange d’ions d’acides organiques. Quantifier la production à l’aide d’un détecteur à indice de réfraction (RID), en comparant les zones de crête à une norme de (±)-mévalonolactone.
Résultats
La régulation optogénétique du métabolisme microbien a été mise en œuvre avec succès pour produire une variété de produits, y compris des biocarburants, des produits chimiques en vrac, des protéines et des produits naturels5,6,7,12,13. La plupart de ces processus sont conçus pour que la croissance cellulaire se produise dans la lumière (lorsque la...
Discussion
Le contrôle dynamique est appliqué depuis longtemps pour améliorer les rendements pour l’ingénierie métabolique et la production de protéines recombinantes4. Les changements dans l’expression enzymatique sont le plus souvent mis en œuvre à l’aide d’inducteurs chimiques tels que IPTG21, galactose22 et tétracycline23, mais ont également été médiés en utilisant des conditions de processus telles que la temp?...
Déclarations de divulgation
Les auteurs ont déposé plusieurs brevets pour les circuits et méthodes optogénétiques décrits dans cet article.
Remerciements
Cette recherche a été soutenue par le département de l’Énergie des États-Unis, l’Office of Science, le numéro de prix DE-SC0019363 du bureau de la recherche biologique et environnementale, le prix NSF CAREER Award CBET-1751840, The Pew Charitable Trusts et le prix Camille Dreyfus Teacher-Scholar.
matériels
| Name | Company | Catalog Number | Comments |
| Light-controlled chemical production using S. cerevisiae | |||
| 24-well culture plate | USA Scientific | CC7672-7524 | |
| Agar powder | Thermo Fisher Scientific | 303991049 | |
| Aluminum foil | Reynolds | B004NG90YO | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Blue LED panel | HQRP | 884667106091218 | |
| EZ-L439 OptoINVRT7 Plasmid | N/A | N/A | See Reference 1 |
| Glucose | Thermo Fisher Scientific | 501879892 (G8270-5KG) | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Petri dish | Celltreat | 229656 | |
| PmeI | New England Biolabs | R0560L | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| Replica-plating device | Thomas Scientific | F37848-0000 | |
| Replica-plating pads | Sunrise Science Products | 3005-012 | |
| SC-His powder | Sunrise Science Products | 1303-030 | |
| SC Complete powder | Sunrise Science Products | 1459-100 | |
| Sterile sealing film | Excel Scientific | STR-SEAL-PLT | |
| YPD agar plates | VWR | 100217-054 | |
| Zeocin | Thermo Fisher Scientific | R25005 | |
| Light-controlled protein production using E. coli | |||
| 6X SDS Sample Buffer | Cepham Life Sciences | 10502 | |
| 12% Acrylamide protein gels | Thermo Fisher Scientific | NP0341BOX | |
| 24-well culture plate | USA Scientific | CC7672-7524 | |
| Aluminum foil | Reynolds | B004NG90YO | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Blue LED panel | HQRP | 884667106091218 | |
| Coomassie Brilliant Blue G-250 | Thermo Fisher Scientific | 20279 | |
| Electrophoresis cell | Bio-Rad | 1658004 | |
| Electrophoresis power supply | Bio-Rad | 1645050 | |
| LB broth (Miller) | Fisher Scientific | BP97235 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| NaCl | Thomas Scientific | SX0425-1 | |
| OptoLAC plasmids | N/A | N/A | See Reference 2 |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Petri dish | Celltreat | 229656 | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| SOC medium | Thermo Fisher Scientific | 15544034 | |
| Thermomixer | Eppendorf | 5382000015 | |
| Tris base | Fisher Scientific | BP1521 | |
| Three-phase fermentation using S. cerevisiae | |||
| Same materials as "Light-controlled chemical production using S. cerevisiae" protocol plus the following: | |||
| EZ-L580 OptoAMP4 Plasmid | N/A | N/A | See Reference 10 |
| Chemical production in a light-controlled bioreactor | |||
| Aluminum foil | Reynolds | B004NG90YO | |
| Antifoam | Sigma-Aldrich | A8311 | |
| Bioreactor with control station | Eppendorf | B120110001 | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Bleach | VWR Scientific | 89501-620 (CS) | |
| Blue LED panel | HQRP | 884667106091218 | |
| BPT tubing | Fisher Scientific | 14-170-15 | |
| Glucose | Thermo Fisher Scientific | 501879892 (G8270-5KG) | |
| Hydrochloric acid (HCl) | Fisher Scientific | 7647-01-0 | |
| M9 Minimal Salts | Thermo Fisher Scientific | A1374401 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| NH4OH Solution | Sigma-Aldrich | I0503-1VL | |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| SC Complete powder | Sunrise Science Products | 1459-100 |
Références
- Figueroa, D., Rojas, V., Romero, A., Larrondo, L. F., Salinas, F. The rise and shine of yeast optogenetics. Yeast. 38 (2), 131-146 (2021).
- Pouzet, S., et al. The promise of optogenetics for bioproduction: Dynamic control strategies and scale-up instruments. Bioengineering. 7 (4), 151 (2020).
- Venayak, N., Anesiadis, N., Cluett, W. R., Mahadevan, R. Engineering metabolism through dynamic control. Current Opinion in Biotechnology. 34, 142-152 (2015).
- Lalwani, M. A., Zhao, E. M., Avalos, J. L. Current and future modalities of dynamic control in metabolic engineering. Current Opinion in Biotechnology. 52, 56-65 (2018).
- Zhao, E. M., et al. Design and characterization of rapid optogenetic circuits for dynamic control in yeast metabolic engineering. ACS Synthetic Biology. 9 (12), 3254-3266 (2020).
- Lalwani, M. A., et al. Optogenetic control of the lac operon for bacterial chemical and protein production. Nature Chemical Biology. 17 (1), 71-79 (2021).
- Zhao, E. M., et al. Optogenetic regulation of engineered cellular metabolism for microbial chemical production. Nature. 555 (7698), 683-687 (2018).
- Baumschlager, A., Khammash, M. Synthetic biological approaches for optogenetics and tools for transcriptional light-control in bacteria. Advanced Biology. 5 (5), 2000256 (2021).
- Dvorak, P., et al. Exacerbation of substrate toxicity by IPTG in Escherichia coli BL21(DE3) carrying a synthetic metabolic pathway. Microbial Cell Factories. 14, 201 (2015).
- Hartline, C. J., Schmitz, A. C., Han, Y., Zhang, F. Dynamic control in metabolic engineering: Theories, tools, and applications. Metabolic Engineering. 63, 126-140 (2021).
- Ni, C., Dinh, C. V., Prather, K. L. J. Dynamic control of metabolism. Annual Review of Chemical and Biomolecular Engineering. 12, 519-560 (2021).
- Zhao, E. M., et al. Optogenetic amplification circuits for light-induced metabolic control. ACS Synthetic Biology. 10 (5), 1143-1154 (2021).
- Lalwani, M. A., Zhao, E. M., Wegner, S. A., Avalos, J. L. The Neurospora crassa Inducible Q System Enables Simultaneous Optogenetic Amplification and Inversion in Saccharomyces cerevisiae for Bidirectional Control of Gene Expression. ACS Synthetic Biology. 10 (8), 2060-2075 (2021).
- Motta-Mena, L. B., et al. An optogenetic gene expression system with rapid activation and deactivation kinetics. Nature Chemical Biology. 10 (3), 196-202 (2014).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Marx, H., Mecklenbräuker, A., Gasser, B., Sauer, M., Mattanovich, D. Directed gene copy number amplification in Pichia pastoris by vector integration into the ribosomal DNA locus. FEMS Yeast Research. 9 (8), 1260-1270 (2009).
- Nordén, K., et al. Increasing gene dosage greatly enhances recombinant expression of aquaporins in Pichia pastoris. BMC Biotechnology. 11, 47 (2011).
- Zhao, E. M., et al. Light-based control of metabolic flux through assembly of synthetic organelles. Nature Chemical Biology. 15 (6), 589-597 (2019).
- Dowee, W. J., Miller, J. F., Ragsdale, C. W. High efficiency transformation of E. coli by high voltage electroporation. Nucleic Acids Research. 16 (13), 6127-6145 (1988).
- Zhou, K., Edgar, S., Stephanopoulos, G. Engineering microbes to synthesize plant isoprenoids. Methods in Enzymology. 575, 225-245 (2016).
- Arfman, N., Worrell, V., Ingram, L. O. Use of the tac promoter and lacI(q) for the controlled expression of Zymomonas mobilis fermentative genes in Escherichia coli and Zymomonas mobilis. Journal of Bacteriology. 174 (22), 7370-7378 (1992).
- Steen, E. J., et al. Metabolic engineering of Saccharomyces cerevisiae for the production of n-butanol. Microbial Cell Factories. 7 (1), 1-8 (2008).
- Tan, S. Z., Manchester, S., Prather, K. L. J. Controlling central carbon metabolism for improved pathway yields in Saccharomyces cerevisiae. ACS Synthetic Biology. 5 (2), 116-124 (2015).
- Jayaraman, P., et al. Blue light-mediated transcriptional activation and repression of gene expression in bacteria. Nucleic Acids Research. 44 (14), 6994 (2016).
- Fernandez-Rodriguez, J., Moser, F., Song, M., Voigt, C. A. Engineering RGB color vision into Escherichia coli. Nature Chemical Biology. 13 (7), 706-708 (2017).
- Ding, Q., et al. Light-powered Escherichia coli cell division for chemical production. Nature Communications. 11 (1), 1-14 (2020).
- Senoo, S., Tandar, S. T., Kitamura, S., Toya, Y., Shimizu, H. Light-inducible flux control of triosephosphate isomerase on glycolysis in Escherichia coli. Biotechnology and Bioengineering. 116 (12), 3292-3300 (2019).
- Ramakrishnan, P., Tabor, J. J. Repurposing synechocystis PCC6803 UirS-UirR as a UV-violet/green photoreversible transcriptional regulatory tool in E. Coli. ACS Synthetic Biology. 5 (7), 733-740 (2016).
- Tabor, J. J., Levskaya, A., Voigt, C. A. Multichromatic control of gene expression in escherichia coli. Journal of Molecular Biology. 405 (2), 315-324 (2011).
- Stewart, C. J., McClean, M. N. Design and implementation of an automated illuminating, culturing, and sampling system for microbial optogenetic applications. Journal of Visualized Experiments:JoVE. (120), e54894 (2017).
- Grødem, E. O. S., Sweeney, K., McClean, M. N. Automated calibration of optoPlate LEDs to reduce light dose variation in optogenetic experiments. BioTechniques. 69 (4), 313-316 (2020).
- Gerhardt, K. P., et al. An open-hardware platform for optogenetics and photobiology. Scientific Reports. 6, (2016).
- Bugaj, L. J., Lim, W. A. High-throughput multicolor optogenetics in microwell plates. Nature Protocols. 14 (7), 2205-2228 (2019).
- Steel, H., Habgood, R., Kelly, C., Papachristodoulou, A. In situ characterisation and manipulation of biological systems with Chi.Bio. PLoS Biology. 18 (7), (2020).
- Carrasco-López, C., García-Echauri, S. A., Kichuk, T., Avalos, J. L. Optogenetics and biosensors set the stage for metabolic cybergenetics. Current Opinion in Biotechnology. 65, 296-309 (2020).
- Milias-Argeitis, A., Rullan, M., Aoki, S. K., Buchmann, P., Khammash, M. Automated optogenetic feedback control for precise and robust regulation of gene expression and cell growth. Nature Communications. 7 (1), 1-11 (2016).
- Melendez, J., et al. Real-time optogenetic control of intracellular protein concentration in microbial cell cultures. Integrative Biology: Quantitative Biosciences From Nano to Macro. 6 (3), 366-372 (2014).
- Milias-Argeitis, A., et al. In silico feedback for in vivo regulation of a gene expression circuit. Nature Biotechnology. 29 (12), 1114-1116 (2011).
- Castillo-Hair, S. M., Baerman, E. A., Fujita, M., Igoshin, O. A., Tabor, J. J. Optogenetic control of Bacillus subtilis gene expression. Nature Communications. 10 (1), 1-11 (2019).
- Xia, A., et al. Optogenetic modification of pseudomonas aeruginosa enables controllable twitching motility and host infection. ACS Synthetic Biology. 10 (3), 531-541 (2021).
- Pu, L., Yang, S., Xia, A., Jin, F. Optogenetics manipulation enables prevention of biofilm formation of engineered pseudomonas aeruginosa on surfaces. ACS Synthetic Biology. 7 (1), 200-208 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon