È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Fermentazioni a controllo leggero per la produzione chimica microbica e proteica
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Il controllo optogenetico del metabolismo microbico offre un controllo dinamico flessibile sui processi di fermentazione. Il protocollo qui mostra come impostare fermentazioni regolate dalla luce blu per la produzione chimica e proteica a diverse scale volumetriche.
Abstract
Le fabbriche di cellule microbiche offrono un'alternativa sostenibile per la produzione di sostanze chimiche e proteine ricombinanti da materie prime rinnovabili. Tuttavia, sovraccaricare un microrganismo con modificazioni genetiche può ridurre la forma fisica e la produttività dell'ospite. Questo problema può essere superato utilizzando il controllo dinamico: espressione inducibile di enzimi e percorsi, in genere utilizzando additivi chimici o nutrizionali, per bilanciare la crescita e la produzione cellulare. L'optogenetica offre un metodo non invasivo, altamente sintonizzabile e reversibile per regolare dinamicamente l'espressione genica. Qui, descriviamo come impostare fermentazioni controllate dalla luce di Escherichia coli ingegnerizzato e Saccharomyces cerevisiae per la produzione di sostanze chimiche o proteine ricombinanti. Discutiamo di come applicare la luce in momenti e dosaggi selezionati per disaccoppiare la crescita e la produzione microbica per migliorare il controllo e la produttività della fermentazione, nonché le considerazioni chiave di ottimizzazione per i migliori risultati. Inoltre, descriviamo come implementare i controlli della luce per esperimenti di bioreattori su scala di laboratorio. Questi protocolli facilitano l'adozione di controlli optogenetici in microrganismi ingegnerizzati per migliorare le prestazioni di fermentazione.
Introduzione
L'optogenetica, il controllo dei processi biologici con proteine sensibili alla luce, offre una nuova strategia per controllare dinamicamente le fermentazioni microbiche per la produzione chimica e proteica1,2. L'onere delle vie metaboliche ingegnerizzate e la tossicità di alcuni intermedi e prodotti spesso compromettono la crescita cellulare3. Tali sollecitazioni possono portare a uno scarso accumulo di biomassa e a una ridotta produttività3. Questa sfida può essere affrontata dividendo temporalmente le fermentazioni in una fase di crescita e produzione, che dedicano risorse metaboliche rispettivamente all'accumulo di biomassa o alla sintesi del prodotto4. Recentemente abbiamo dimostrato che il passaggio dalla crescita alla produzione in questa fermentazione bifase può essere indotto con cambiamenti nelle condizioni di illuminazione5,6,7. L'elevata sintonizzazione, reversibilità e ortogonalità degli ingressi luminosi8 offre vantaggi unici alle fermentazioni controllate dalla luce che sono difficili o impossibili da replicare con induttori chimici utilizzati nel controllo dinamico delle fermentazioni bifase convenzionali4,9,10,11.
La proteina EL222 sensibile alla luce blu derivata da Erythrobacter litoralis è stata utilizzata per sviluppare diversi circuiti optogenetici per l'ingegneria metabolica in Saccharomyces cerevisiae5,7,12,13. EL222 contiene un dominio LOV (Light-Oxygen-Voltage Sensor) che subisce uno spostamento conformazionale all'attivazione della luce blu (465 nm), che gli consente di legarsi alla sua sequenza di DNA affine (C120)13. La fusione di EL222 con il dominio di attivazione virale VP16 (VP16-EL222) si traduce in un fattore di trascrizione sensibile alla luce blu che può attivare in modo reversibile l'espressione genica in S. cerevisiae7 e altri organismi14 dal promotore sintetico PC120. Diversi circuiti basati su EL222 sono stati sviluppati e utilizzati per la produzione chimica in S. cerevisiae, come il sistema optoEXP di base attivato dalla luce7, in cui il gene di interesse è espresso direttamente da PC120 (Figura 1A). Tuttavia, le preoccupazioni di penetrazione della luce alle alte densità cellulari tipicamente incontrate nella fase di produzione delle fermentazioni ci hanno motivato a sviluppare circuiti invertiti indotti al buio, come i circuiti OptoINVRT e OptoQ-INVRT (Figura 1B)5,7,13. Questi sistemi sfruttano i regolaloni di galattosio (GAL) o acido chinico (Q) di S. cerevisiae e N. crassa, rispettivamente, controllando i loro corrispondenti repressori (GAL80 e QS) con VP16-EL222, per reprimere l'espressione genica alla luce e indurla fortemente al buio. La combinazione dei circuiti OptoEXP e OptoINVRT si traduce in un controllo bidirezionale dell'espressione genica, consentendo fermentazioni bifase in cui la fase di crescita è indotta con luce blu e la fase di produzione con oscurità (Figura 2A)5,7.
L'uso della luce al posto dell'oscurità per indurre l'espressione genica durante la fase di produzione espanderebbe notevolmente le capacità dei controlli optogenetici, ma richiederebbe anche il superamento dei limiti di penetrazione della luce delle alte densità cellulari tipicamente incontrate in questa fase di fermentazione. A tal fine, abbiamo sviluppato circuiti, noti come OptoAMP e OptoQ-AMP, che amplificano la risposta trascrizionale alla stimolazione della luce blu. Questi circuiti utilizzano mutanti wild-type o ipersensibili di VP16-EL222 per controllare la produzione degli attivatori trascrizionali Gal4p o QF2 dei reguloni GAL o Q, rispettivamente, ottenendo una maggiore sensibilità e una maggiore espressione genica con la luce12,13 (Figura 1C). I circuiti OptoAMP possono ottenere un'induzione luminosa completa e omogenea in bioreattori da 5 L a densità ottica (misurata a 600 nm; OD600) valori di almeno 40 con solo ~0,35% di illuminazione (5% di dose di luce solo su ~7% della superficie di massa). Ciò dimostra un grado di sensibilità più elevato rispetto a OptoEXP, che richiede un'illuminazione vicina al 100%12. La capacità di indurre efficacemente l'espressione genica con la luce ad alte densità cellulari apre nuove opportunità per il controllo dinamico delle fermentazioni. Ciò include fermentazioni operative in più di due fasi temporali, come le fermentazioni trifase, in cui le fasi di crescita, induzione e produzione sono stabilite con programmi leggeri unici per ottimizzare la produzione chimica (Figura 2B)12.

Figura 1: Circuiti optogenetici per il controllo dinamico di S. cerevisiae. I circuiti OptoEXP, OptoINVRT e OptoAMP si basano sul sistema VP16-EL222 sensibile alla luce. (A) Nel circuito OptoEXP, l'esposizione alla luce blu provoca un cambiamento conformazionale e la dimerizzazione di VP16-EL222, che espone un dominio legante il DNA e consente la trascrizione da PC120. La figura è stata modificata da Zhao et al.7. (B) I circuiti OptoINVRT sfruttano i regolagni GAL (mostrati) o Q per indurre l'espressione al buio. Nei circuiti basati su GAL, VP16-EL222 e GAL4 sono espressi costitutivamente, mentre PC120 guida l'espressione del repressore GAL80 (nei circuiti basati su Q, GAL4 e GAL80 sono sostituiti rispettivamente da QF2 e QS e viene utilizzato un promotore sintetico contenente QUAS al posto di un promotore GAL). Alla luce, Gal80p impedisce l'attivazione del gene di interesse da PGAL1. Al buio, GAL80 non è espresso e rapidamente degradato fondendolo in un dominio costitutivo di degron (piccolo dominio marrone), che consente l'attivazione di PGAL1 da parte di Gal4p. La figura è stata modificata da Zhao et al.5. (C) I circuiti OptoAMP utilizzano anche VP16-EL222 per controllare i reguloni GAL (mostrato) o Q. In questi circuiti, il repressore GAL80 (o QS) è costitutivamente espresso e fuso in un degron fotosensibile (piccolo dominio blu) che garantisce una stretta repressione al buio. PC120 e un'espressione di controllo mutante ipersensibile VP16-EL222 di GAL4 (o QF2) con la luce, che attiva fortemente PGAL1 (o un promotore contenente QUAS) nella luce. I circuiti derivati da GAL possono utilizzare forme ingegnerizzate di PGAL1, come PGAL1-M o PGAL1-S, che hanno aumentato l'attività, così come promotori wild-type controllati dal regulon GAL (PGAL1, PGAL10, PGAL2, PGAL7). La figura è stata modificata da Zhao et al.12. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Fermentazioni a due e tre fasi nel tempo. (A) Le fermentazioni bifase operate con circuiti invertiti consistono in una fase di crescita guidata dalla luce e una fase di produzione scura. Nella fase di crescita, la biomassa si accumula mentre il percorso di produzione rimane represso. Al raggiungimento dell'OD600 desiderato, le cellule vengono spostate al buio per regolarsi metabolicamente prima di essere risospese in mezzi freschi per la fase di produzione. (B) In un processo in tre fasi, le fasi di crescita, incubazione e produzione sono definite da programmi di luce unici, che possono consistere in un periodo di crescita scuro, incubazione pulsata e fase di produzione completamente illuminata. Figura creata con Biorender. Fare clic qui per visualizzare una versione più grande di questa figura.
Sono stati inoltre sviluppati circuiti optogenetici per il controllo dinamico della produzione chimica e proteica in E. coli. I circuiti OptoLAC controllano il repressore batterico LacI utilizzando il circuito pDawn sensibile alla luce, basato sul sistema bicomponente YF1/FixJ6 (Figura 3). Simile a OptoINVRT5, i circuiti OptoLAC sono progettati per reprimere l'espressione genica nella luce e indurla al buio. I livelli di espressione che utilizzano circuiti OptoLAC possono eguagliare o superare quelli raggiunti con l'induzione standard isopropil β-d-1-tiogalattopoliranoside (IPTG), mantenendo così la forza dell'induzione chimica offrendo al contempo una maggiore sintonizzazione e reversibilità6. Pertanto, i circuiti OptoLAC consentono un efficace controllo optogenetico per l'ingegneria metabolica in E. coli.
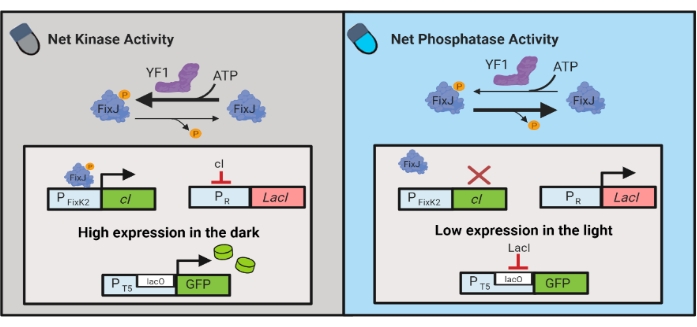
Figura 3: Circuiti OptoLAC per il controllo dinamico di E. coli. I circuiti OptoLAC adattano il sistema pDawn e l'operone lac per ottenere l'attivazione al buio e la repressione alla luce. Al buio, YF1 fosforila FixJ, che quindi attiva il promotore PFixK2 per esprimere il repressore cI . Il repressore cI impedisce l'espressione del repressore lacI dal promotore PR , che consente la trascrizione del gene di interesse da un promotore contenente lacO. Al contrario, la luce blu riduce l'attività della chinasi netta YF1, invertendo la fosforilazione di FixJ e quindi l'espressione di cI , che dereprime l'espressione di lacI e impedisce l'espressione dal promotore contenente lacO. La figura è stata modificata da Lalwani et al.6. Fare clic qui per visualizzare una versione più grande di questa figura.
Descriviamo qui i protocolli di base per le fermentazioni controllate dalla luce di S. cerevisiae ed E. coli per la produzione chimica o proteica. Sia per i lieviti che per i batteri, ci concentriamo innanzitutto sulle fermentazioni con una fase di crescita guidata dalla luce e una fase di produzione indotta dall'oscurità abilitata dai circuiti OptoINVRT e OptoLAC. Successivamente, descriviamo un protocollo per una fermentazione a tre fasi (crescita, induzione, produzione) controllata dalla luce abilitata dai circuiti OptoAMP. Inoltre, descriviamo come aumentare le fermentazioni optogeneticamente controllate dalle micropiastre ai bioreattori su scala di laboratorio. Con questo protocollo, miriamo a fornire una guida completa e facilmente riproducibile per eseguire fermentazioni controllate dalla luce per la produzione chimica o proteica.
Protocollo
1. Produzione chimica controllata dalla luce utilizzando il circuito S. cerevisiae OptoINVRT7
- Costruzione della deformazione
- Ottenere un ceppo con l'auxotrofia his3 , poiché questo marcatore è necessario per la maggior parte dei plasmidi OptoINVRT esistenti5. Se cerchi la regolazione optogenetica di un gene nativo di S. cerevisiae, costruisci un ceppo in cui qualsiasi copia endogena del gene viene eliminata.
- Linearizzare il plasmide contenente il circuito OptoINVRT7, come EZ-L4395, e integrarlo nell'his3-locus del ceppo auxotrofico utilizzando metodi standard di trasformazione litio-acetato15. Se si utilizza il plasmide EZ-L439, che contiene i componenti per reprimere PGAL1 alla luce e attivarlo al buio, linearizzare nel sito di restrizione PmeI.
- Dopo la trasformazione, centrifugare le cellule a 150 x g per 1 minuto e risospese delicatamente in 200 μL di mezzo sintetico completo (SC-His) istidina-dropout fresco.
- Placcare l'intero volume cellulare su piastre di agar SC-His e incubare a 30 °C per 2-3 giorni fino alla comparsa delle colonie.
- Preparare cellule competenti da questo ceppo utilizzando protocolli standard di trasformazione dell'acetato di litio e trasformarle con un plasmide contenente il gene o i geni da controllare optogeneticamente a valle del promotore PGAL1-M o PGAL1-S5.
NOTA: L'utilizzo di un plasmide che si integra in siti δ (YARCdelta5) e seleziona con Zeocin consente un'integrazione multicopia stabile7,16,17,18. - Dopo la trasformazione, centrifugare la coltura a 150 x g per 1 minuto e risospesare delicatamente in 200 μL di terreno fresco SC-dropout.
NOTA: Il promotore PGAL1-M è una versione sintetica del promotore PGAL1 con i siti di repressione Mig1p eliminati, mentre PGAL1-S è una versione ingegnerizzata di PGAL1-M, che ha siti di legame dell'attivatore Gal4p extra. Il promotore PGAL1 regolare può essere utilizzato per controllare l'espressione; tuttavia, la forza di espressione sarà inferiore a quella di questi promotori ingegnerizzati. - Placcare l'intero volume cellulare su una piastra di agar di peptone destrosio (YPD) estratto di lievito se si integra in siti δ o una piastra SC-dropout se si trasforma con un plasmide contenente un marcatore di selezione. Incubare a 30 °C per 16 ore sotto luce blu costante per mantenere represso il gene optogeneticamente controllato.
NOTA: per alcuni ceppi, le colonie potrebbero crescere più velocemente se incubate in impulsi di luce blu (ad esempio, 1 s on/79 s off, 5 s on/75 s off, 10 s on/70 s off, ecc.) piuttosto che in illuminazione costante, che deve essere determinata sperimentalmente per ogni ceppo, se necessario. - Utilizzare qualsiasi sorgente luminosa a 465 nm e posizionare un pannello LED ~ 40 cm sopra la piastra in modo tale che l'intensità della luce sia ~ 80-110 μmol / m2 / s. Misurare l'intensità utilizzando un misuratore quantistico (vedi Tabella dei materiali).
- Se si esegue l'integrazione in siti δ, creare una replica della piastra su piastre YPD contenenti un intervallo di concentrazioni di Zeocin tra 400 μg/mL e 1.200 μg/mL da selezionare per una varietà di numeri di copia di integrazione5,7,12,16,17,18. Incubare le piastre replica a 30 °C sotto luce blu costante o pulsata per 2-3 giorni fino alla comparsa delle colonie.
- Screening preliminare per le migliori colonie
- Selezionare otto colonie da ogni piastra e usarle per inoculare 1 mL di SC-Il suo mezzo integrato con il 2% di glucosio nei singoli pozzetti di una piastra a 24 pozzetti. Crescere in piastre a 24 pozzetti sotto le celle durante la notte (16-20 h) a 30 °C con 200 rpm (diametro orbitale 19 mm) che scuotono sotto un'illuminazione a luce blu costante.
- La mattina successiva, diluire ogni coltura in 1 mL di un mezzo SC-His fresco con il 2% di glucosio a valori di OD600 che vanno da 0,01-0,3 e crescere in piastre a 24 pozzetti sotto luce costante o pulsata a 30 °C con 200 rpm scuotendo fino a raggiungere densità cellulari comprese tra 2 e 9 valori di OD600 (Figura 4A). La quantità di tempo necessaria per questa fase di crescita dipenderà dalla tensione.
- Incubare le piastre al buio per 4 ore a 30 °C con 200 giri/min scuotendo il pannello luminoso e avvolgendo le piastre in un foglio di alluminio.
NOTA: Questo passaggio consente alle cellule di passare metabolicamente alla fase di produzione prima della risospensione nei mezzi di produzione. - Per iniziare la fase di produzione, centrifugare le colture nella piastra a 24 pozzetti a 234 x g per 5 minuti e risospese le cellule in 1 mL di terreno fresco SC-dropout con il 2% di glucosio. Sigillare le piastre per evitare l'evaporazione del prodotto desiderato utilizzando nastro sigillante micropiastra sterile.
- Fermentare le piastre sigillate al buio per 48 ore a 30 °C con agitazione a 200 giri/min. Assicurarsi che le piastre siano avvolte in un foglio di alluminio per evitare qualsiasi esposizione alla luce.
NOTA: Avvolgere le piastre in un foglio non limita la disponibilità di ossigeno o gas nelle fermentazioni; tuttavia, il nastro sigillante sterile limita il trasferimento di gas. Piccoli fori possono essere infilati nel nastro per introdurre ossigeno, se necessario.
- Raccolta e analisi
- Per raccogliere le fermentazioni, centrifugare le piastre per 5 minuti a 234 x g e trasferire 800 μL del surnatante in tubi microcentrifuga da 1,5 mL.
- A seconda della sostanza chimica di interesse, analizzare utilizzando cromatografia liquida ad alte prestazioni (HPLC), gascromatografia-spettrometria di massa (GC-MS) o un altro metodo analitico utilizzando la tecnica di preparazione del campione più adatta allo strumento utilizzato.
2. Produzione di proteine controllate dalla luce utilizzando il sistema E. coli OptoLAC
- Costruzione della deformazione
- Co-trasformare l'elettrocompetente BL21 DE3 ΔlacI ΔlacI-DE3 con un plasmide contenente il circuito OptoLAC1B o OptoLAC2B6 e un plasmide che esprime la proteina ricombinante di interesse dal promotore PT719.
- Dopo la trasformazione, recuperare le cellule per 1 ora in 1 mL di brodo super ottimale con repressione della catabolite (SOC; 2% triptone, 0,5% estratto di lievito, 10 mM NaCl, 2,5 mM KCl, 10 mM MgCl2, 10 mM MgSO4, 20 mM glucosio) a 37 °C con rotazione o agitazione.
NOTA: Il plasmide contenente la proteina di interesse deve essere compatibile con il plasmide OptoLAC (cioè diverso marcatore di resistenza e origine di replicazione) e non deve contenere una copia di lacI. - Centrifugare le celle a 4845 x g per 3 min e concentrare il pellet in 200 μL di brodo di lisogenia (LB). Placcare le intere cellule concentrate su una piastra di agar LB con antibiotici appropriati e crescere a 37 ° C durante la notte sotto una luce blu costante per mantenere repressa l'espressione proteica.
- Screening iniziale per verificare l'espressione
- Prendere tre colonie singole e usarle per inoculare 1 mL di lb media con antibiotici appropriati in singoli pozzetti di una piastra a 24 pozzetti. Crescere durante la notte (16-20 h) a 37 °C con 200 giri/min che scuotono sotto illuminazione a luce blu costante (Figura 4A).
- Il giorno successivo, utilizzare 1,5 μL di coltura per misurare OD600 in uno spettrofotometro con una misurazione del microvolume. Diluire le colture in 1 mL di LB fresco in piastre a 24 pozzetti a valori di OD600 compresi tra 0,01 e 0,1.
- Coltivare le colture a 37 °C con 200 giri/min scuotendo sotto la luce blu per 2-3 ore. A partire dalla seconda ora, effettuare misurazioni OD600 ogni 15 minuti per garantire che le colture non superino l'intervallo OD600 di 0,1-1,5.
- Una volta che le colture sono all'OD600 desiderato, spegnere il pannello luminoso e avvolgere la piastra in un foglio di alluminio per avviare la fase di produzione. Tenere la piastra al buio per 8 ore (37 °C), 20 ore (30 °C) o 48 ore (18 °C), con 200 giri/min di agitazione.
- Misurare e registrare il valore OD600 finale di ogni lingua.
- Raccolta e analisi
- Trasferire 800 μL di ogni coltura in un tubo microcentrifuga da 1,5 mL e centrifuga per 5 minuti a 17.000 x g.
- Risospesso il pellet cellulare in 200 μL di tampone di risospensione (Tris 50 mM, pH 8,0, NaCl 300 mM).
NOTA: La concentrazione di NaCl può essere regolata in base alla proteina ricombinante analizzata. - Aggiungere 50 μL di tampone campione 6x sodio dodecil solfato (SDS) (Tris 375 mM, pH 6,8, SDS 9%, glicerolo 50%, blu bromofenolo 0,03%, DTT 9%). Incubare a 100 °C per 10 minuti con agitazione a 700 giri/min in un bimby.
- Caricare ~3-20 μL della coltura in un gel SDS-PAGE al 12%. Utilizzando la misurazione finale OD600 come guida, caricare approssimativamente la stessa quantità di proteine per ciascun campione (equivalente a 10 μL del campione corrispondente a un valore OD600 finale di 1). Utilizzare un alimentatore per eseguire l'elettroforesi a 100 V fino a quando il gel non è completamente risolto.
- Macchiare il gel con la soluzione blu brillante di Coomassie G-250 riscaldando per 30-40 s in un forno a microonde e quindi incubando su un rotatore a piattaforma per almeno 15 minuti.
- Risciacquare con acqua deionizzata due volte e detonare su un rotatore a piattaforma per almeno 30 minuti (o durante la notte), aggiungendo due salviette detergenti legate in un nodo per aiutare ad assorbire la macchia. Far bollire il gel in quantità sufficiente di acqua nel forno a microonde per 15 minuti per accelerare il processo di detaining.
3. Fermentazione trifase con il sistema S. cerevisiae OptoAMP
- Costruzione della deformazione
- Ottenere un ceppo con un marcatore auxotrofico his3 , poiché questo marcatore è necessario per utilizzare i plasmidi OptoAMP esistenti5. Se cerchi la regolazione optogenetica di un gene nativo di S. cerevisiae, costruisci un ceppo in cui la copia endogena di questo gene viene eliminata.
- Linearizzare un plasmide contenente il circuito OptoAMP4, come EZ-L58012, e integrarlo nel locus his3 del ceppo auxotrofico utilizzando metodi standard di trasformazione litio-acetato15. Se si utilizza EZ-L580, linearizzare il plasmide nel sito di restrizione PmeI.
- Dopo la trasformazione, centrifugare le cellule a 150 x g per 1 minuto e risospese delicatamente in 200 μL di mezzo SC-His fresco.
- Placcare l'intero volume cellulare su un mezzo selettivo (SC-His-agar) e incubare a 30 °C per 2-3 giorni fino alla comparsa delle colonie.
- Preparare cellule competenti da questo ceppo e trasformarle con un plasmide contenente il gene o i geni da controllare optogeneticamente a valle del promotore PGAL1-S12.
NOTA: L'utilizzo di un plasmide che si integra in δ-siti e seleziona con Zeocin consente un'integrazione e una selezione multicopia stabili. - Dopo la trasformazione, centrifugare la coltura a 150 x g per 1 minuto e risospenare delicatamente in 200 μL di terreno fresco SC-dropout.
NOTA: Il promotore PGAL1-S è una versione sintetica del promotore PGAL1 in cui i siti di repressione Mig1p vengono eliminati e vengono aggiunti ulteriori siti di legame dell'attivatore Gal4p. È possibile utilizzare il normale promotore PGAL1 ; tuttavia, la forza di espressione sarà inferiore a quella di questo promotore ingegnerizzato. - Placcare l'intero volume della cella su una piastra di agar YPD o SC-dropout e incubare a 30 °C per 16 ore al buio (avvolto in un foglio di alluminio). L'incubazione al buio mantiene represso il gene optogeneticamente controllato, il che consente alle cellule di dirigere le loro risorse metaboliche verso la crescita cellulare piuttosto che verso la produzione chimica.
- Se si esegue l'integrazione in siti δ, replicare la piastra su piastre YPD contenenti un intervallo di concentrazioni di Zeocin da selezionare per una varietà di numeri di copia di integrazione. Incubare le piastre a 30 °C al buio (avvolte in un foglio di alluminio) per 2-3 giorni fino alla comparsa delle colonie.
- Screening preliminare per le migliori colonie
- Selezionare otto colonie da ogni piastra e utilizzarle per inoculare 1 mL di SC-His mezzo con il 2% di glucosio nei singoli pozzetti di una piastra a 24 pozzetti. Far crescere le cellule durante la notte (16-20 h) al buio a 30 °C con 200 giri/min scuotendo.
- La mattina successiva, diluire ogni coltura in 1 mL di terreno fresco SC-His con il 2% di glucosio a 0,1 OD600 e crescere al buio a 30 °C con 200 rpm scuotendo fino a raggiungere un OD600 di 3. Avvolgere le piastre in un foglio di alluminio per evitare l'esposizione alla luce. La quantità di tempo necessaria per questa fase di crescita dipenderà dalla tensione.
- Per avviare la fase di induzione, incubare le piastre sotto luce pulsata (ad esempio, 5 s on/95 s off) per 12 h a 30 °C con 200 rpm di agitazione. Utilizzare qualsiasi sorgente luminosa a 465 nm e posizionare un pannello LED sopra la piastra in modo tale che l'intensità luminosa sia ~ 80-110 μmol / m2 / s per risultati ottimali (Figura 4A).
NOTA: la durata ottimale dell'impulso luminoso per questa incubazione varierà in base alla sostanza chimica prodotta. Si consiglia di schermare una gamma di programmi di illuminazione dallo 0,1% (ad esempio, 1 s su 999 s spento) al 100% (piena luce). - Per iniziare la fase di produzione, centrifugare le colture a 234 x g per 5 minuti e risospendare in mezzo SC-His fresco con il 2% di glucosio. Sigillare le piastre per evitare l'evaporazione del prodotto desiderato utilizzando nastro sigillante sterile a micropiastre.
- Fermentare le piastre sigillate alla luce per 48 ore a 30 °C con agitazione a 200 giri/min. Ottimizza il programma di luce durante questa fase poiché alcune sostanze chimiche beneficiano di una fase di produzione pulsata piuttosto che di luce piena.
- Raccolta e analisi
- Raccogliere le fermentazioni centrifugando le piastre per 5 minuti a 234 x g e trasferendo 800 μL del surnatante in tubi microcentrifuga da 1,5 mL.
- A seconda della sostanza chimica di interesse, analizzare utilizzando HPLC, GC-MS o un altro metodo analitico utilizzando la tecnica di preparazione del campione più adatta allo strumento utilizzato.
4. Produzione chimica (mevalonato) da E. coli in un bioreattore a controllo leggero
- Inoculazione iniziale e configurazione del bioreattore
- Inoculare una colonia di un ceppo di E. coli ingegnerizzato con produzione chimica controllata dalla luce in 5 mL di sali minimi M9 (3,37 mM Na2HPO4, 2,2 mM KH2PO4, 0,855 mM NaCl, 0,935 mM NH4Cl) integrati con 0,2% p/v casaminoacidi, 5% p/v glucosio e miscela di metalli in traccia20 (0,0084 g/L EDTA, 0,0025 g/L CoCl2, 0,015 g/L MnCl2, 0,0015 g/L CuCl2, 0,003 g/L H3BO3, 0,0025 g/L Na2MoO4, 0,008 g/L ZnCl2, 0,06 g/L Fe (III) citrato, 0,0045 g/L tiamina, 1,3 g/L MgSO4) in un tubo conico da 50 mL.
- Coltiva la coltura durante la notte a 30 °C con 200 giri/min scuotendo sotto l'illuminazione a luce blu.
- Impostare la piastra di testa del serbatoio del bioreattore, assicurandosi che siano installate le seguenti porte: inserto per sonda termica; sonda di ossigeno disciolto (DO); sparger di gas - collegarsi alla fonte d'aria attraverso un filtro da 0,2 μm; girante; condensatore di gas - collegare a un filtro da 0,2 μm; linea di raffreddamento (x2); linee di alimentazione (x2) - una per l'aggiunta di supporti, una per il controllo del pH; linea di campionamento - assicurarsi che raggiunga il fondo della nave; porta vuota; Sonda di pH - calibrare la sonda con standard di pH = 4 e pH = 7 prima dell'installazione, in autoclave con il resto del recipiente o sterilizzare con etanolo al 95% e inserire asetticamente prima dell'installazione.
- Riempire il recipiente con 1 L di acqua filtrata e fissare la piastra di testa e stringere, assicurandosi che l'O-ring si adatti perfettamente e sigilli.
- Coprire eventuali aperture nel reattore con un foglio di alluminio.
- Preparare tre strisce di tubi: una per rimuovere l'acqua, una per l'inserimento del mangime e una per il controllo del pH. Coprire le estremità con un foglio di alluminio e avvolgere tutto il tubo in un foglio di alluminio.
NOTA: NH4OH, che viene utilizzato per la regolazione del pH, non scorre senza intoppi nei tubi in silicone, il che può portare a portate imprecise e sovra-basificazione della coltura. Per evitare questo problema, utilizzare tubi della pompa biocompatibili (BPT) per l'alimentazione NH4OH. - Autoclave del bioreattore e del tubo, utilizzando un ciclo di 30 minuti del liquido.
- Rimuovere i materiali dall'autoclave. Una volta raffreddato abbastanza da gestire, collegare la girante, le sonde di pH e DO, la sorgente d'aria, l'ingresso e l'uscita del condensatore e l'ingresso e l'uscita del raffreddamento alla stazione di controllo.
- Inserire la sonda termica e coprire la nave con una camicia riscaldante. Fissare la giacca verso la parte superiore del recipiente per evitare di bloccare la coltura dall'esposizione alla luce.
- Collegare uno dei tubi sterili alla linea di campionamento e fissarlo attraverso la pompa di campionamento. Posizionare l'altra estremità in modo tale che scorra in un contenitore vuoto che può contenere almeno 1 L. Scaricare l'acqua all'interno della nave.
- Collegare un altro tubo sterile a una delle linee di alimentazione e fissarlo attraverso una delle pompe di alimentazione. Collegare l'altra estremità del tubo a una bottiglia di supporto M9. Alimentare i media nel reattore.
- Collegare un altro tubo sterile a una delle linee di alimentazione e fissarlo attraverso una delle pompe di alimentazione. Collegare l'altra estremità del tubo al flacone contenente il 28%-30% di NH4OH.
ATTENZIONE: NH4OH è corrosivo. Lavorare in una cappa aspirante durante il trasferimento in un biberon e assicurarsi che il biberon sia posto in contenimento secondario. - Posizionare tre pannelli luminosi in una formazione triangolare a circa 20 cm di distanza dal reattore, controllando che le intensità luminose sulla superficie del recipiente raggiungano ~ 80-110 μmol / m2 / s da ciascun lato (Figura 4B).
- Il giorno successivo, accendere il sistema di bioreattore e il refrigeratore. Impostare il setpoint della temperatura del reattore a 37 °C, il setpoint del pH a 7,0 e l'agitazione a 200 giri/min. La giacca riscaldante dovrebbe accendersi.
- Calibrare la sonda DO aspettando prima che la temperatura e le misurazioni DO diventino costanti (questo sarà il setpoint al 100%). Quindi, scollegare la sonda dal sistema (questo sarà il setpoint dello 0%). Ripetere l'operazione fino a quando la misurazione DO non si stabilizza al 100% quando la sonda è collegata, quindi impostare il setpoint DO su 20%.
- Produzione chimica controllata dalla luce
- Inoculare il bioreattore ad un OD600 iniziale di 0,001-0,1. Accendi i pannelli luminosi per avviare la crescita.
- Dopo 3 ore, iniziare a prelevare campioni dalla linea di campionamento per effettuare misurazioni OD600 per evitare di aumentare eccessivamente la densità cellulare ottimale di induzione (ρs). Una volta raggiunto il ρs ottimale (il valore ottimale per la produzione di mevalonato è 0,17), spegnere i pannelli luminosi, coprire il reattore con un foglio di alluminio e avvolgere il setup in un panno nero per avviare la fase di produzione scura.
- Aggiungere 50 μL di antischiuma, 8 ore dopo il passaggio all'oscurità. Svitare la porta vuota e pipettare l'antischiuma direttamente nel reattore.
- Utilizzare la porta di campionamento per prelevare periodicamente campioni per l'analisi HPLC o GC.
- Smontaggio e analisi
- Al termine dell'esperimento, spegnere il sistema. Svitare con cura le sonde DO e pH e lavarle con acqua e sapone. Svitare la piastra della testa e lavarla con acqua e sapone usando un pennello.
- Trasferire la coltura in un contenitore vuoto e aggiungere candeggina ad una concentrazione finale del 10% v/v. Mettere in una cappa aspirante e smaltire dopo 30 min.
- Lavare il recipiente del reattore con acqua e sapone usando un pennello.
- Preparare i campioni per l'analisi in base al prodotto di interesse. Per la produzione di mevalonato, mescolare 560 μL di coltura con 140 μL di 0,5 M HCl e vortice ad alta velocità per 1 min. Questo converte il mevalonato in (±)-mevalonolactone.
ATTENZIONE: HCl è un pericolo per la salute. Maneggiare con DPI adeguati e assicurarsi che le provette del campione siano adeguatamente tappate prima del vortice. - Centrifugare a 17.000 x g per 45 min a 4 °C. Trasferire 250 μL del surnatante in un flaconcino HPLC.
- Per il mevalonato, analizzare i campioni utilizzando una colonna di scambio ionico di acidi organici. Quantificare la produzione utilizzando un rivelatore di indice di rifrazione (RID), confrontando le aree di picco con uno standard di (±)-mevalonolattone.
Risultati
La regolazione optogenetica del metabolismo microbico è stata implementata con successo per produrre una varietà di prodotti, tra cui biocarburanti, sostanze chimiche sfuse, proteine e prodotti naturali5,6,7,12,13. La maggior parte di questi processi sono progettati per la crescita cellulare che avviene alla luce (quando la bassa densità cellulare pone sfid...
Discussione
Il controllo dinamico è stato a lungo applicato per migliorare le rese per l'ingegneria metabolica e la produzione di proteine ricombinanti4. I cambiamenti nell'espressione enzimatica sono più tipicamente implementati utilizzando induttori chimici come IPTG21, galattosio22 e tetraciclina23, ma sono stati anche mediati utilizzando condizioni di processo come temperatura e pH. Il controllo optogenetico dell'espressione genica ...
Divulgazioni
Gli autori hanno richiesto diversi brevetti per i circuiti e i metodi optogenetici descritti in questo articolo.
Riconoscimenti
Questa ricerca è stata sostenuta dal Dipartimento dell'Energia degli Stati Uniti, dall'Office of Science, dall'Office of Biological and Environmental Research Award Number DE-SC0019363, dal NSF CAREER Award CBET-1751840, dal Pew Charitable Trusts e dal Camille Dreyfus Teacher-Scholar Award.
Materiali
| Name | Company | Catalog Number | Comments |
| Light-controlled chemical production using S. cerevisiae | |||
| 24-well culture plate | USA Scientific | CC7672-7524 | |
| Agar powder | Thermo Fisher Scientific | 303991049 | |
| Aluminum foil | Reynolds | B004NG90YO | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Blue LED panel | HQRP | 884667106091218 | |
| EZ-L439 OptoINVRT7 Plasmid | N/A | N/A | See Reference 1 |
| Glucose | Thermo Fisher Scientific | 501879892 (G8270-5KG) | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Petri dish | Celltreat | 229656 | |
| PmeI | New England Biolabs | R0560L | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| Replica-plating device | Thomas Scientific | F37848-0000 | |
| Replica-plating pads | Sunrise Science Products | 3005-012 | |
| SC-His powder | Sunrise Science Products | 1303-030 | |
| SC Complete powder | Sunrise Science Products | 1459-100 | |
| Sterile sealing film | Excel Scientific | STR-SEAL-PLT | |
| YPD agar plates | VWR | 100217-054 | |
| Zeocin | Thermo Fisher Scientific | R25005 | |
| Light-controlled protein production using E. coli | |||
| 6X SDS Sample Buffer | Cepham Life Sciences | 10502 | |
| 12% Acrylamide protein gels | Thermo Fisher Scientific | NP0341BOX | |
| 24-well culture plate | USA Scientific | CC7672-7524 | |
| Aluminum foil | Reynolds | B004NG90YO | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Blue LED panel | HQRP | 884667106091218 | |
| Coomassie Brilliant Blue G-250 | Thermo Fisher Scientific | 20279 | |
| Electrophoresis cell | Bio-Rad | 1658004 | |
| Electrophoresis power supply | Bio-Rad | 1645050 | |
| LB broth (Miller) | Fisher Scientific | BP97235 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| NaCl | Thomas Scientific | SX0425-1 | |
| OptoLAC plasmids | N/A | N/A | See Reference 2 |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Petri dish | Celltreat | 229656 | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| SOC medium | Thermo Fisher Scientific | 15544034 | |
| Thermomixer | Eppendorf | 5382000015 | |
| Tris base | Fisher Scientific | BP1521 | |
| Three-phase fermentation using S. cerevisiae | |||
| Same materials as "Light-controlled chemical production using S. cerevisiae" protocol plus the following: | |||
| EZ-L580 OptoAMP4 Plasmid | N/A | N/A | See Reference 10 |
| Chemical production in a light-controlled bioreactor | |||
| Aluminum foil | Reynolds | B004NG90YO | |
| Antifoam | Sigma-Aldrich | A8311 | |
| Bioreactor with control station | Eppendorf | B120110001 | |
| BioSpectrometer with μcuvette | Eppendorf | 6135000923 | |
| Bleach | VWR Scientific | 89501-620 (CS) | |
| Blue LED panel | HQRP | 884667106091218 | |
| BPT tubing | Fisher Scientific | 14-170-15 | |
| Glucose | Thermo Fisher Scientific | 501879892 (G8270-5KG) | |
| Hydrochloric acid (HCl) | Fisher Scientific | 7647-01-0 | |
| M9 Minimal Salts | Thermo Fisher Scientific | A1374401 | |
| Microcentrifuge | Thermo Fisher Scientific | 75002403 | |
| Microcentrifuge tubes | USA Scientific | 1615-5510 | |
| NH4OH Solution | Sigma-Aldrich | I0503-1VL | |
| Orbital Shaker | Yamato Scientific America | SOU-300 | |
| Quantum meter | Apogee Instruments | MQ-510 | |
| SC Complete powder | Sunrise Science Products | 1459-100 |
Riferimenti
- Figueroa, D., Rojas, V., Romero, A., Larrondo, L. F., Salinas, F. The rise and shine of yeast optogenetics. Yeast. 38 (2), 131-146 (2021).
- Pouzet, S., et al. The promise of optogenetics for bioproduction: Dynamic control strategies and scale-up instruments. Bioengineering. 7 (4), 151 (2020).
- Venayak, N., Anesiadis, N., Cluett, W. R., Mahadevan, R. Engineering metabolism through dynamic control. Current Opinion in Biotechnology. 34, 142-152 (2015).
- Lalwani, M. A., Zhao, E. M., Avalos, J. L. Current and future modalities of dynamic control in metabolic engineering. Current Opinion in Biotechnology. 52, 56-65 (2018).
- Zhao, E. M., et al. Design and characterization of rapid optogenetic circuits for dynamic control in yeast metabolic engineering. ACS Synthetic Biology. 9 (12), 3254-3266 (2020).
- Lalwani, M. A., et al. Optogenetic control of the lac operon for bacterial chemical and protein production. Nature Chemical Biology. 17 (1), 71-79 (2021).
- Zhao, E. M., et al. Optogenetic regulation of engineered cellular metabolism for microbial chemical production. Nature. 555 (7698), 683-687 (2018).
- Baumschlager, A., Khammash, M. Synthetic biological approaches for optogenetics and tools for transcriptional light-control in bacteria. Advanced Biology. 5 (5), 2000256 (2021).
- Dvorak, P., et al. Exacerbation of substrate toxicity by IPTG in Escherichia coli BL21(DE3) carrying a synthetic metabolic pathway. Microbial Cell Factories. 14, 201 (2015).
- Hartline, C. J., Schmitz, A. C., Han, Y., Zhang, F. Dynamic control in metabolic engineering: Theories, tools, and applications. Metabolic Engineering. 63, 126-140 (2021).
- Ni, C., Dinh, C. V., Prather, K. L. J. Dynamic control of metabolism. Annual Review of Chemical and Biomolecular Engineering. 12, 519-560 (2021).
- Zhao, E. M., et al. Optogenetic amplification circuits for light-induced metabolic control. ACS Synthetic Biology. 10 (5), 1143-1154 (2021).
- Lalwani, M. A., Zhao, E. M., Wegner, S. A., Avalos, J. L. The Neurospora crassa Inducible Q System Enables Simultaneous Optogenetic Amplification and Inversion in Saccharomyces cerevisiae for Bidirectional Control of Gene Expression. ACS Synthetic Biology. 10 (8), 2060-2075 (2021).
- Motta-Mena, L. B., et al. An optogenetic gene expression system with rapid activation and deactivation kinetics. Nature Chemical Biology. 10 (3), 196-202 (2014).
- Gietz, R. D., Woods, R. A. Transformation of yeast by lithium acetate/single-stranded carrier DNA/polyethylene glycol method. Methods in Enzymology. 350, 87-96 (2002).
- Marx, H., Mecklenbräuker, A., Gasser, B., Sauer, M., Mattanovich, D. Directed gene copy number amplification in Pichia pastoris by vector integration into the ribosomal DNA locus. FEMS Yeast Research. 9 (8), 1260-1270 (2009).
- Nordén, K., et al. Increasing gene dosage greatly enhances recombinant expression of aquaporins in Pichia pastoris. BMC Biotechnology. 11, 47 (2011).
- Zhao, E. M., et al. Light-based control of metabolic flux through assembly of synthetic organelles. Nature Chemical Biology. 15 (6), 589-597 (2019).
- Dowee, W. J., Miller, J. F., Ragsdale, C. W. High efficiency transformation of E. coli by high voltage electroporation. Nucleic Acids Research. 16 (13), 6127-6145 (1988).
- Zhou, K., Edgar, S., Stephanopoulos, G. Engineering microbes to synthesize plant isoprenoids. Methods in Enzymology. 575, 225-245 (2016).
- Arfman, N., Worrell, V., Ingram, L. O. Use of the tac promoter and lacI(q) for the controlled expression of Zymomonas mobilis fermentative genes in Escherichia coli and Zymomonas mobilis. Journal of Bacteriology. 174 (22), 7370-7378 (1992).
- Steen, E. J., et al. Metabolic engineering of Saccharomyces cerevisiae for the production of n-butanol. Microbial Cell Factories. 7 (1), 1-8 (2008).
- Tan, S. Z., Manchester, S., Prather, K. L. J. Controlling central carbon metabolism for improved pathway yields in Saccharomyces cerevisiae. ACS Synthetic Biology. 5 (2), 116-124 (2015).
- Jayaraman, P., et al. Blue light-mediated transcriptional activation and repression of gene expression in bacteria. Nucleic Acids Research. 44 (14), 6994 (2016).
- Fernandez-Rodriguez, J., Moser, F., Song, M., Voigt, C. A. Engineering RGB color vision into Escherichia coli. Nature Chemical Biology. 13 (7), 706-708 (2017).
- Ding, Q., et al. Light-powered Escherichia coli cell division for chemical production. Nature Communications. 11 (1), 1-14 (2020).
- Senoo, S., Tandar, S. T., Kitamura, S., Toya, Y., Shimizu, H. Light-inducible flux control of triosephosphate isomerase on glycolysis in Escherichia coli. Biotechnology and Bioengineering. 116 (12), 3292-3300 (2019).
- Ramakrishnan, P., Tabor, J. J. Repurposing synechocystis PCC6803 UirS-UirR as a UV-violet/green photoreversible transcriptional regulatory tool in E. Coli. ACS Synthetic Biology. 5 (7), 733-740 (2016).
- Tabor, J. J., Levskaya, A., Voigt, C. A. Multichromatic control of gene expression in escherichia coli. Journal of Molecular Biology. 405 (2), 315-324 (2011).
- Stewart, C. J., McClean, M. N. Design and implementation of an automated illuminating, culturing, and sampling system for microbial optogenetic applications. Journal of Visualized Experiments:JoVE. (120), e54894 (2017).
- Grødem, E. O. S., Sweeney, K., McClean, M. N. Automated calibration of optoPlate LEDs to reduce light dose variation in optogenetic experiments. BioTechniques. 69 (4), 313-316 (2020).
- Gerhardt, K. P., et al. An open-hardware platform for optogenetics and photobiology. Scientific Reports. 6, (2016).
- Bugaj, L. J., Lim, W. A. High-throughput multicolor optogenetics in microwell plates. Nature Protocols. 14 (7), 2205-2228 (2019).
- Steel, H., Habgood, R., Kelly, C., Papachristodoulou, A. In situ characterisation and manipulation of biological systems with Chi.Bio. PLoS Biology. 18 (7), (2020).
- Carrasco-López, C., García-Echauri, S. A., Kichuk, T., Avalos, J. L. Optogenetics and biosensors set the stage for metabolic cybergenetics. Current Opinion in Biotechnology. 65, 296-309 (2020).
- Milias-Argeitis, A., Rullan, M., Aoki, S. K., Buchmann, P., Khammash, M. Automated optogenetic feedback control for precise and robust regulation of gene expression and cell growth. Nature Communications. 7 (1), 1-11 (2016).
- Melendez, J., et al. Real-time optogenetic control of intracellular protein concentration in microbial cell cultures. Integrative Biology: Quantitative Biosciences From Nano to Macro. 6 (3), 366-372 (2014).
- Milias-Argeitis, A., et al. In silico feedback for in vivo regulation of a gene expression circuit. Nature Biotechnology. 29 (12), 1114-1116 (2011).
- Castillo-Hair, S. M., Baerman, E. A., Fujita, M., Igoshin, O. A., Tabor, J. J. Optogenetic control of Bacillus subtilis gene expression. Nature Communications. 10 (1), 1-11 (2019).
- Xia, A., et al. Optogenetic modification of pseudomonas aeruginosa enables controllable twitching motility and host infection. ACS Synthetic Biology. 10 (3), 531-541 (2021).
- Pu, L., Yang, S., Xia, A., Jin, F. Optogenetics manipulation enables prevention of biofilm formation of engineered pseudomonas aeruginosa on surfaces. ACS Synthetic Biology. 7 (1), 200-208 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon