Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Analyse du type de fibre et de la teneur en gouttelettes lipidiques dans le muscle squelettique
Dans cet article
Résumé
De plus en plus de preuves indiquent qu’une infiltration excessive de lipides à l’intérieur du muscle squelettique entraîne une lipotoxicité et un diabète. Ici, nous présentons un protocole complet, y compris le traitement des tissus, la coloration avec Bodipy, l’acquisition d’images et l’analyse, pour quantifier la taille, la densité et la distribution subcellulaire des gouttelettes lipidiques d’une manière spécifique au type de fibre.
Résumé
L’infiltration lipidique des muscles squelettiques, connue sous le nom de myostéatose, augmente avec l’obésité et le vieillissement. La myostéatose a également été récemment découverte comme facteur pronostique négatif pour plusieurs autres troubles tels que les maladies cardiovasculaires et le cancer. Une infiltration lipidique excessive diminue la masse musculaire et la force. Il en résulte également une lipotoxicité et une résistance à l’insuline en fonction de la teneur totale en lipides intramyocellulaires, de la morphologie des gouttelettes lipidiques (LD) et de la distribution subcellulaire. Le type de fibre (oxydatif vs glycolytique) est également important, car les fibres oxydatives ont une plus grande capacité à utiliser les lipides. En raison de leurs implications cruciales en physiopathologie, des études approfondies sur la dynamique et la fonction des TA d’une manière spécifique au type de fibre sont justifiées.
Ici, un protocole complet est présenté pour la quantification de la teneur en lipides intramyocellulaires et l’analyse de la morphologie de la LD et de la distribution subcellulaire d’une manière spécifique au type de fibre. À cette fin, des cryosections musculaires en série ont été colorées avec le colorant fluorescent Bodipy et des anticorps contre les isoformes de chaîne lourde de myosine. Ce protocole permet le traitement simultané de différents muscles, ce qui permet de gagner du temps et d’éviter d’éventuels artefacts et, grâce à une macro personnalisée créée aux Fidji, l’automatisation de l’analyse LD est également possible.
Introduction
L’infiltration lipidique des muscles squelettiques, connue sous le nom de myostéatose, augmente avec l’obésité et le vieillissement. La myostéatose est négativement corrélée à la masse musculaire et à la force et à la sensibilité à l’insuline1. De plus, des études récentes indiquent que le degré de myostéatose pourrait être utilisé comme facteur pronostique pour d’autres affections telles que lesmaladies cardiovasculaires 2, la stéatose hépatique non alcoolique3 ou le cancer4. Les lipides peuvent s’accumuler dans le muscle squelettique entre les fibres musculaires sous forme de lipides extramyocellulaires ou dans les fibres, sous forme de lipides intramyocellulaires (IMCL). Les IMCL sont principalement stockés sous forme de triglycérides dans des gouttelettes lipidiques (LD) qui sont utilisées comme carburant métabolique pendant l’exercice physique 5,6. Cependant, lorsque l’offre de lipides dépasse la demande ou lorsque les mitochondries deviennent dysfonctionnelles, les IMCL seront impliqués dans la résistance musculaire à l’insuline, comme on le voit chez les personnes métaboliquement malsaines, obèses et chez les patients diabétiques de type 27. Curieusement, les athlètes d’endurance ont des niveaux similaires, sinon plus élevés, de MCMC que ceux trouvés chez les patients obèses atteints de diabète sucré de type 2, tout en maintenant une sensibilité élevée à l’insuline. Ce phénomène est décrit comme le « paradoxe de l’athlète »8,9, et s’explique par une évaluation plus nuancée des TA musculaires, liée à leur taille, leur densité, leur localisation, leur dynamique et la composition des espèces lipidiques.
Tout d’abord, la taille de la LD est inversement corrélée à la sensibilité à l’insuline et à la forme physique10,11. En fait, les LD plus petites présentent une surface relativement plus grande pour l’action de la lipase et, par conséquent, ont potentiellement une plus grande capacité à mobiliser les lipides12. Deuxièmement, la densité LD (nombre/surface) joue un rôle controversé dans l’actionde l’insuline 8,10; pourtant, il semble être augmenté chez les athlètes. Troisièmement, la localisation subcellulaire des LD est importante, car les LD situées juste sous la membrane superficielle (sous-sarmateuse ou périphérique) exercent un effet plus délétère sur la sensibilité à l’insuline que les LD centrales 8,9,13. Ces dernières fournissent du carburant aux mitochondries centrales, qui ont une plus grande activité respiratoire et sont plus spécialisées pour répondre à la forte demande d’énergie requise pour la contraction14. En revanche, les TA périphériques fournissent des mitochondries sous-sarmateuses, qui sont impliquées dans les processus liés à la membrane8. Enfin, au-delà des triglycérides, des lipides complexes spécifiques dans le muscle peuvent être plus délétères que d’autres. Par exemple, le diacylglycérol, l’acyl-CoA à longue chaîne et les céramides peuvent s’accumuler dans les muscles lorsque le taux de renouvellement des triglycérides est faible, altérant ainsi la signalisation de l’insuline 9,15. Pour en revenir au « paradoxe de l’athlète », les athlètes d’endurance ont un nombre élevé de TA centrales plus petites avec des taux de renouvellement élevés dans les fibres de type I (oxydatives), tandis que les patients obèses et diabétiques ont des TA périphériques plus importants avec de faibles taux de renouvellement dans les fibres de type II (glycolytiques) 8,15,16. En plus de leur rôle dans le stockage et la libération de l’énergie, les LD via des acides gras dérivés (FA) et une protéine de pelage (périlamine 5) pourraient également fonctionner comme des acteurs critiques impliqués dans la régulation transcriptionnelle de l’oxydation de l’AF et de la biogenèse mitochondriale8. En raison de leurs implications cruciales en physiologie et en physiopathologie, des études approfondies sur la dynamique et les fonctions des TA sont justifiées.
Bien qu’il existe plusieurs techniques pour étudier les IMCL, elles ne sont pas toutes adaptées pour quantifier avec précision la taille, la densité et la distribution des DA DL d’une manière spécifique à la fibre. Par exemple, l’évaluation des NML par spectroscopie par résonance magnétique, tout en étant non invasive, offre un niveau de résolution qui n’est pas suffisant pour étudier la taille et l’emplacement précis des LD dans la fibre, et ce n’est pas spécifique au type de fibre17,18. De même, les techniques biochimiques effectuées sur des homogénats musculaires entiers19 ne peuvent pas évaluer l’emplacement et la taille des lipides. Par conséquent, la méthode la plus adéquate pour analyser la morphologie et l’emplacement de la LD est la microscopie électronique à transmission quantitative13, mais cette technique est coûteuse et prend beaucoup de temps. Par conséquent, l’imagerie par fluorescence confocale sur des préparations avec des colorants tels que Oil Red O (ORO)20,21, monodansylpentane (MDH)22 ou Bodipy 23,24,25, est apparue comme le meilleur outil pour ces études.
Ici, un protocole complet est décrit, y compris l’échantillonnage et le traitement des tissus, la coloration corporelle et l’acquisition et l’analyse d’images confocales pour quantifier la taille, le nombre et la localisation des TA dans les cryosections musculaires de souris. Étant donné que les IMCL ne sont pas répartis uniformément entre les fibres oxydatives et glycolytiques, et que chaque type de fibre régule différemment la dynamique de la LD, l’étude des IMCL doit être spécifique au type de fibre 16,25,26,27. Par conséquent, ce protocole utilise l’immunofluorescence sur des sections en série pour identifier les isoformes de chaîne lourde de myosine (MyHC) exprimées par chaque fibre. Un autre avantage de ce protocole est le traitement simultané d’un muscle glycolytique (extenseur digitorum longus, EDL) et d’un muscle oxydatif (soléaire) placé côte à côte avant la congélation (Figure 1). Ce traitement simultané permet non seulement de gagner du temps, mais aussi d’éviter la variabilité due au traitement séparé des échantillons.
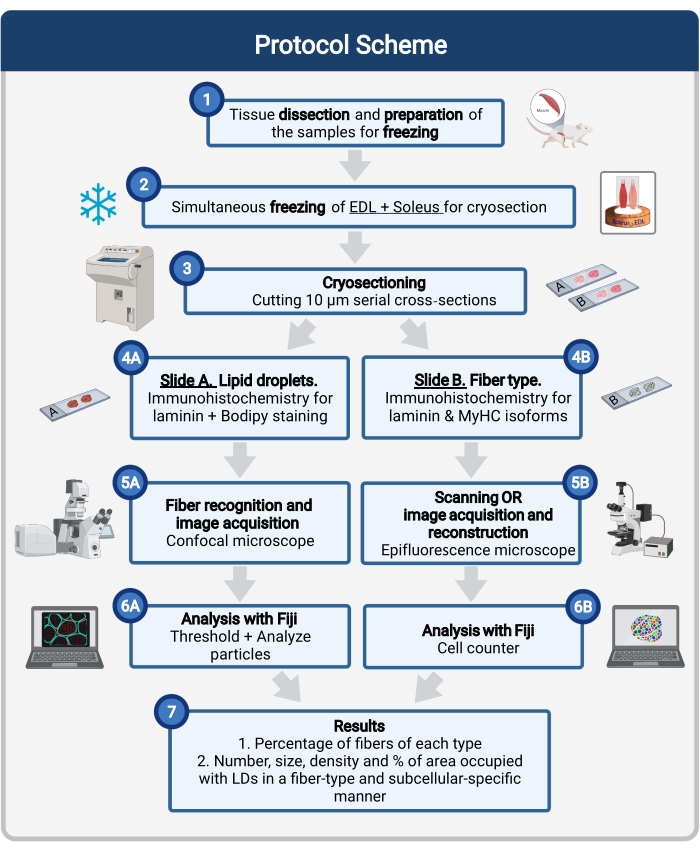
Figure 1 : Vue d’ensemble schématique de la procédure. Après la dissection musculaire (1), des muscles sélectionnés de taille similaire sont préparés et congelés ensemble (2). Les sections transversales en série de 10 μm sont obtenues à l’aide d’un cryostat et directement montées sur des lames d’adhérence (3). À partir de deux diapositives en série, la première (4A) est immunomarquée pour la laminine et colorée avec Bodipy pour reconnaître les LD et la seconde (4B) est immunocolorée avec des anticorps contre les MyHC pour la reconnaissance des types de fibres musculaires. Les images sont acquises à l’aide d’un microscope confocal pour Bodipy (5A) et d’un microscope à épifluorescence pour les types de fibres musculaires (5B). Les images sont analysées aux Fidji en appliquant un seuil et en quantifiant les particules (6A) pour obtenir le nombre, la taille moyenne, la densité et le pourcentage de la surface totale occupée par les TA (7) ou en comptant les cellules (6B) pour obtenir le pourcentage de fibres de chaque type dans la section (7). Abréviations : LD = gouttelettes lipidiques ; EDL = extenseur digitorum longus; MyHC = isoformes de chaîne lourde de myosine. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Access restricted. Please log in or start a trial to view this content.
Protocole
Toutes les procédures menées sur des souris ont été approuvées par le Comité d’éthique pour l’expérimentation animale du secteur médical de l’Université catholique de Louvain (2019/UCL/MD/013).
1. Dissection et préparation des échantillons pour la congélation
- Étiquetez un morceau de liège de 3 mm d’épaisseur pour chaque paire de muscles.
- À travers une petite incision faite avec une lame au centre du liège, insérez perpendiculairement un morceau rectangulaire de plastique rigide (0,5 cm W, 1 cm H) qui servira de support (Figure 2B).
REMARQUE: La taille de la pièce en plastique rectangulaire dépendra de la taille du muscle. Ici, les dimensions décrites sont adaptées à la taille du soléaire (~9 mg, 1 cm L, 2-3 mm L) et EDL (~5 mg, 1 cm L, 2-3 mm L) d’une souris mâle C57BL/6J de 3 mois. - Au moment de la dissection, retirez le soléaire et l’EDL du membre postérieur de la souris. Pour éviter que les échantillons ne se dessèchent pendant la dissection, placez-les sur une compresse légèrement humidifiée avec une solution saline dans une boîte de Pétri placée sur de la glace.
NOTE: Voir Wang et al.28 pour des explications sur la façon de disséquer ces deux muscles des membres. - Placez une petite goutte de composé de température de coupe optimale (OCT) à la jonction liège/plastique, en évitant les bulles d’air.
- Éliminez l’excès d’humidité des échantillons en les séchant doucement avec une serviette en papier (Figure 2A) et placez les deux muscles sur le plastique perpendiculairement au liège (Figure 2C).
- Vérifiez l’orientation des myofibres musculaires au stéréomicroscope (Figure 2D).
REMARQUE: Il est important de ne pas couvrir le muscle avec de l’OCT, car son effet isolant empêcherait le gel rapide et produirait des artefacts de congélation.
2. Congélation d’échantillons de muscles squelettiques pour la cryosection
ATTENTION : La congélation du muscle doit se faire sous une cagoule chimique, en portant l’équipement de protection individuelle approprié (voir le Tableau des matériaux).
- Utilisez un gobelet en acier inoxydable (~8 cm H, 6 cm Ø) avec deux sangles latérales attachées à lui d’au moins 25 cm de long (Figure 2F), et remplissez le gobelet jusqu’à 2/3 de sa capacité avec de l’isopentane.
- En saisissant le gobelet par les sangles, immergez-le doucement dans une boîte en polystyrène remplie d’azote liquide afin que le niveau d’azote à l’extérieur du récipient soit supérieur au niveau d’isopentane à l’intérieur (Figure 2F, G).
ATTENTION : Lorsque le gobelet entre en contact avec l’azote, le choc thermique peut provoquer des tourbillons. Assurez-vous que le gobelet est suffisamment immergé mais évitez l’entrée d’azote car cela provoquerait des précipitations d’isopentane. Si cela se produit, laissez l’isopentane refroidir, remplissez le gobelet avec un nouvel isopentane et recommencez. - Lorsque l’intérieur du gobelet est complètement recouvert d’une couche solide blanche d’isopentane, sortez-le de la boîte d’azote liquide (Figure 2H).
REMARQUE: La température de fusion de l’isopentane étant de -159 ° C, les bords du gobelet deviendront blancs lorsqu’il fera assez froid. - Remuez doucement les morceaux d’isopentane solide dans l’isopentane liquide restant avec une pince jusqu’à ce que tout le volume redevienne liquide.
- Réassurez le gobelet dans l’azote liquide jusqu’à ce que l’isopentane forme des cailloux blancs sur tout le fond et les bords du gobelet (Figure 2G,H).
REMARQUE: Cette deuxième étape de refroidissement assure la température de congélation appropriée de l’isopentane. - Retirez le gobelet de l’azote liquide et plongez rapidement les muscles dans le fond du gobelet, en tenant le bouchon avec une pince à dents de rat. Faire tourbillonner le liège pendant 15 s dans l’isopentane et le conserver à -80 °C jusqu’au traitement (Figure 2I).
REMARQUE: Pour un stockage à court terme, les échantillons peuvent être conservés dans un congélateur à -20 ° C. Le protocole complet pour la congélation rapide du muscle squelettique a déjà été publié ailleurs. Pour des références détaillées et le dépannage, voir: Meng et al.29, Kumar et al.30, et Leiva-Cepas et al.31.
3. Cryosection
- Apportez les échantillons dans la chambre d’un cryostat préalablement refroidi à -20 °C et la température de la lame réglée à -25 °C.
REMARQUE: Transporter les échantillons du congélateur de -20 °C/-80 °C au cryostat dans une boîte en polystyrène remplie de glace carbonique et permettre aux échantillons de s’équilibrer pendant au moins 20 à 30 minutes à la température de la chambre avant la coupe. - Retirez le plastique du liège avec une pince à épiler fine et placez le disque d’échantillon de cryostat sur la plaque de congélation rapide pour refroidir. Une fois que la plaque a atteint -50 °C, placez un peu d’OCT sur le disque et placez rapidement le liège sur le dessus du disque en appuyant fermement. Attendez que l’OCT se solidifie et que le bouchon soit bien fixé sur le disque.
- Placez le disque sur la tête de l’objet dans l’orientation de coupe souhaitée (Figure 2J, K) et coupez le bloc musculaire au-delà d’au moins 1/3 de la longueur des muscles et jusqu’à ce que les deux sections transversales des muscles soient visibles.
- Réglez l’épaisseur de coupe à 10 μm et placez une section sur une lame d’adhérence pour vérifier l’orientation correcte des fibres sur un microscope à fond clair.
REMARQUE: Il est essentiel de vérifier l’orientation transversale des fibres. Si les fibres ne sont pas correctement orientées, ajustez l’angle de la tête de l’objet, coupez une autre section et vérifiez à nouveau. - Placez deux coupes transversales en série sur deux lames d’adhérence prémarquées : une lame pour déterminer les types de fibres, l’autre pour quantifier la teneur en lipides (Figure 2L).
REMARQUE: Des coupes transversales en série supplémentaires peuvent être obtenues et maintenues à -80 °C pour d’autres études histologiques. Cependant, pour éviter de modifier la teneur en lipides et la morphologie intracellulaire24, il est essentiel de traiter les deux premières lames immédiatement après la coupe pour éviter le séchage à l’air. La congélation et la décongélation des lames pour la quantification de la TA auraient le même effet et sont donc très déconseillées.
4. Typage des fibres et coloration corporelle
- Détection immunohistochimique du type de fibre musculaire
REMARQUE: Pour le protocole suivant, un volume de solution totale de 250 μL est suffisant pour couvrir toute la section musculaire entourée d’un cercle dessiné avec un stylo hydrophobe de la taille approximative d’une pièce de 1 cent.- Entourez les sections d’un contour dessiné avec un stylo hydrophobe et rincez avec une solution saline tamponnée au phosphate (PBS) glacée de 0,1 M pendant 1 min à température ambiante (RT). Placer la lame dans une chambre humide et bloquer pendant 90 min à 37 °C dans une solution bloquante (sérum de chèvre normal à 10 % (NGS) et réactif bloquant 1:30 souris sur souris (MOM) dans PBS).
- Retirer la solution bloquante et incuber les lames pendant 90 min à 37 °C avec la solution contenant les anticorps primaires (5 % NGS, réactif bloquant MOM 1:30, les anticorps primaires de souris pour reconnaître les fibres de type I (IgG2b, à 1:10), de type IIa (IgG1, à 1:10) et de type IIx (IgM, à 1:5) et une anti-laminine de rat (chaîne α2, 1:1 000) dans PBS.
- Lavez les diapositives avec PBS 3 x 5 min à RT.
- Incuber les lames dans l’obscurité pendant 1 h à RT avec la solution contenant les anticorps secondaires (chèvre anti-IgG2b AF405 (1:500), chèvre anti-IgG1 AF488 (1:500), chèvre anti-IgM AF568 (1:1 000) et chèvre anti-laminine AF647 (1:500) dans PBS).
ATTENTION : Pour le reste du protocole, assurez-vous de garder les lames à l’écart de la lumière pour préserver la fluorescence. - Laver à nouveau 3 x 5 min dans du PBS, rincer dans duH2O double distillé, éliminer l’excès d’eau et monter avec un réactif antifade.
REMARQUE: Stockez les diapositives à 4 ° C, à l’abri de la lumière pour préserver la fluorescence jusqu’à l’acquisition de l’image. Comme les lames ne sont pas fixes, il est recommandé d’utiliser des solutions fraîchement préparées pour chaque expérience, d’autoclaver le PBS et d’acquérir les images dès que possible pour éviter la contamination des sections.
- Coloration des TA avec Bodipy
REMARQUE: Semblable à l’étape 4.1., un volume de solution totale de 250 μL est suffisant pour couvrir toute la section musculaire entourée d’un cercle dessiné avec un stylo hydrophobe de la taille approximative d’une pièce de 1 cent.- Entourez les sections d’un contour dessiné par un stylo hydrophobe et rincez avec du PBS glacé de 0,1 M pendant 10 min à TA. Utilisez du PBS glacé pour tous les rinçages et lavages.
- Fixer avec du paraformaldéhyde (PFA) froid à 4% sans méthanol pendant 10 min à TA. Après un premier rinçage rapide, laver les lames avec PBS 3 x 5 min à RT.
ATTENTION : Effectuez cette étape sous une hotte chimique. - Placez la glissière dans une chambre humide et bloquez pendant 1 h à RT avec 5% de NGS en PBS.
- Incuber les lames pendant 90 min à 37 °C avec la solution de l’anticorps primaire (2 % de NGS et anti-laminine de rat (chaîne α2, 1:1 000) dans du PBS). Lavez les diapositives avec PBS 3 x 5 min à RT.
ATTENTION : Pour le reste du protocole, assurez-vous de garder les lames à l’écart de la lumière pour préserver la fluorescence. - Incuber pendant 1 h à TA avec la solution d’anticorps secondaire contenant l’anticorps anti-rat de chèvre AF647 (1:500) dans le PBS. Lavez les diapositives avec PBS 3 x 5 min à RT.
- Incuber pendant 20 min à RT avec une solution de 4',6-diamidino-2-phénylindole (DAPI, 0,5 μg/mL) et de BODIPY (1 μg/mL) dans du PBS.
REMARQUE: Pour préparer la solution mère BODIPY, dissoudre dans le DMSO à une concentration de 1 mg / mL. Différentes formulations Bodipy sont disponibles dans le commerce pour la coloration LD. Selon le choix effectué, la méthode de coloration est la même (mêmes étapes, concentration et temps d’incubation); cependant, la méthode d’acquisition sera légèrement différente. - Après un premier rinçage rapide, laver les lames avec PBS 3 x 5 min à TA, rincer dans H2O double distillé, éliminer l’excès d’eau et monter avec un réactif antifade.
REMARQUE: Stockez les lames à 4 °C, à l’abri de la lumière, pour préserver la fluorescence jusqu’à l’acquisition de l’image.
5. Acquisition d’images
REMARQUE: Une fois les protocoles de coloration terminés, il est important de procéder immédiatement à l’acquisition d’images (dans les 24 heures suivantes), non seulement pour éviter la contamination, mais aussi pour préserver la morphologie, la taille et le nombre de LD.
- Acquisition d’images pour évaluer les types de fibres de chaque muscle échantillonné
REMARQUE: Cette étape pourrait être réalisée avec un microscope à balayage à fluorescence à lames entières ou avec un microscope à épifluorescence conventionnel. Avec ce dernier, l’assemblage manuel ou automatisé des images doit être effectué pour reconstruire la section.- Pour la reconnaissance de type fibre, utilisez un microscope à épifluorescence avec un objectif 10x/0.3. Sélectionnez des filtres d’excitation pour DAPI (405 nm), FITC, TRITC et Cy5 pour détecter les fibres de type I, IIa, IIx et la laminine, respectivement.
REMARQUE: Les fibres de type IIb ne seront pas immunomarquées. Ils seront reconnus comme des fibres colorées par la laminine aux limites du sarcolemme avec un sarcoplasme noir. - Ajustez le temps d’exposition approprié pour chaque canal.
- Lorsque vous utilisez un microscope à épifluorescence conventionnel, acquérez toujours les images de l’ensemble du muscle en suivant le même ordre pour faciliter la reconstruction musculaire. Assurez-vous que les fibres sur le bord droit d’une image apparaissent également sur le bord gauche de l’image suivante. Il en va de même pour les parties supérieure et inférieure des images (Figure 3A).
REMARQUE: À titre de référence, pour une section de l’EDL ou du soléaire disséqué d’une souris de 3 mois, une moyenne de six et huit images, respectivement, couvrira toute la section transversale musculaire. - Une fois le muscle scanné, téléchargez les images numériques capturées dans n’importe quel logiciel de traitement d’image pour la reconstruction (couture), en fonction de la morphologie de la fibre (laminine) et de l’histologie de la section musculaire, et enregistrez-les en tant que fichier TIFF, PNG ou JPG avec tous les canaux (couleurs) fusionnés (Figure 3A).
- Pour la reconnaissance de type fibre, utilisez un microscope à épifluorescence avec un objectif 10x/0.3. Sélectionnez des filtres d’excitation pour DAPI (405 nm), FITC, TRITC et Cy5 pour détecter les fibres de type I, IIa, IIx et la laminine, respectivement.
- Acquisition d’images avec laminine et co-coloration Bodipy
REMARQUE: Pour reconnaître le type de fibre et avoir une estimation du nombre de fibres pour chaque type capturé, il est essentiel d’avoir la section de type fibre déjà scannée et les muscles reconstruits avant de commencer l’acquisition d’image de Bodipy-laminin (Figure 3B).- Pour l’observation et l’acquisition d’images corporelles, utilisez un microscope confocal avec un objectif à immersion dans l’huile 40x avec une ouverture numérique de 1,4.
- Utilisez les paramètres suivants : sténopé à 1 UA, résolution de 2 048 pixels x 2 048 pixels, taille des pixels à 0,08 μm, mode unidirectionnel, vitesse de numérisation à 4 (~ 4 μs/pixel), moyenne des lignes réglée sur 4x et zoom numérique réglé sur 1.
- Pour éviter la diaphonie entre Bodipy-558/568 et laminin-AF647, utilisez le mode de balayage séquentiel sur le logiciel confocal.
REMARQUE: Lorsque le colorant choisi est Bodipy-493/503, le balayage laser confocal simultané est possible sans diaphonie entre le canal Bodipy et le canal laminine-AF647. Cela accélérera l’acquisition d’images. - Excitez Bodipy-493/503 à l’aide de la ligne laser 488 nm ou de la ligne laser argon, et excitez Bodipy-555/568 à l’aide de la ligne laser à diode 561 nm. Enfin, détectez la laminine-AF647 avec une ligne laser à diode de 640 nm.
ATTENTION: Gardez à l’esprit que les molécules Bodipy sont très sensibles au photoblanchiment, évitez donc le balayage laser inutile. Pour reconnaître les fibres, utilisez uniquement le laser pour la laminine. - Selon le colorant choisi, réglez les plages d’émission à 570-650 nm pour Bodipy-493/50324 et à 565-620 nm pour Bodipy-558/568. Réglez la plage d’émission de la laminine à 656-700 nm.
- Réglez le gain et le gain numérique de manière appropriée afin qu’aucun pixel saturé ne soit détecté sur l’indicateur de plage. Corrigez le signal d’arrière-plan en ajustant le décalage.
REMARQUE: La sélection du filtre et les autres paramètres de balayage mentionnés ci-dessus doivent être optimisés pour chaque microscope confocal. Il est important que tous les paramètres mentionnés ci-dessus soient maintenus constants pour que toutes les images capturées à partir d’échantillons soient comparées. - Pour identifier le type de fibre parmi celles visualisées au microscope confocal, utilisez un ordinateur portable personnel sur lequel l’image de la section reconstruite après une immunodétection de type fibre est vérifiée (Figure 3B).
- Une fois qu’un groupe de fibres est correctement identifié, acquérez l’image avec les canaux Bodipy et Laminin.
REMARQUE: Il est recommandé de noter le nom de l’image Bodipy-laminin sur la région du muscle où ces fibres sont situées sur l’image acquise pour la reconnaissance MyHC afin de faciliter l’analyse ultérieure des LD spécifiques aux fibres.
6. Analyse des images
- Analyse des types de fibres sur chaque échantillon musculaire
- Dans Fidji (ou ImageJ)32, ouvrez le fichier TIFF, PNG ou JPG avec le muscle reconstruit obtenu à partir de la fusion de tous les canaux utilisés pour détecter les isoformes de fibres.
- Pour démarrer l’outil Cell Counting , cliquez sur Plugins | Analyser | Compteur de cellules | Compteur de cellules.
- Dans la fenêtre Compteur de cellules , cliquez sur Actions | Initialiser.
- Sous Compteurs dans la même fenêtre, sélectionnez Type 1.
- Dans la fenêtre principale de Fidji, sélectionnez l’outil Baguette .
- Pour quantifier le nombre de fibres de chaque type, cliquez sur chaque fibre du même type, de sorte que le programme enregistre le nombre de fibres cliquées.
- Une fois terminé, sélectionnez le type de fibre suivant et répétez les mêmes étapes.
- Lorsque toutes les fibres de l’image ont été affectées à un type de fibre donné, cliquez sur Résultats dans la fenêtre Compteur de cellules pour afficher les résultats dans un tableau.
- Enregistrez ce tableau en tant que table de feuille de calcul en cliquant sur Fichier | Enregistrer sous dans la fenêtre Résultats .
- Enregistrez et rechargez les sélections sur la même image à tout moment en cliquant sur Enregistrer les marqueurs ou Charger les marqueurs, respectivement, dans la fenêtre Comptage des cellules .
- Analyse des gouttelettes lipidiques en fonction du type de fibre
- Analyser les images de Bodipy et de laminine obtenues sur le confocal en utilisant Fidji pour la quantification des TA.
REMARQUE : Les auteurs ont conçu une macro personnalisée pour automatiser l’analyse. Cette macro, ainsi qu’une explication étape par étape sur la façon de l’utiliser, est disponible en tant que fichier supplémentaire 1 et fichier supplémentaire 2, respectivement. - Ouvrez chaque image à l’aide de l’importateur de bioformats des Fidji. Sous l’option Afficher la pile avec , sélectionnez Hyperstack | Mode couleur, par défaut. Assurez-vous que la fenêtre Mise à l’échelle automatique est sélectionnée.
REMARQUE: Les étapes suivantes décriront le protocole d’analyse d’une fibre sur l’image, mais il doit être répété autant de fois que le nombre de fibres entières apparaît sur l’image. - Utilisez l’outil de sélection à main levée pour sélectionner manuellement le sarcolemme de la fibre en fonction du canal de laminine (Figure 4A) et appuyez sur T sur le clavier pour enregistrer la sélection ou la région d’intérêt (ROI) dans la fenêtre ROI .
- Allez dans la fenêtre principale de Fidji et cliquez sur Analyser | Définissez Mesures, puis dans la fenêtre contextuelle, sélectionnez Zone et diamètre de Feret. Laissez les cases restantes décochées et les autres paramètres tels qu’ils apparaissent par défaut.
- Cliquez sur Mesurer dans la fenêtre ROI pour obtenir la surface et le diamètre minimal de la férète (MF) de la fibre sélectionnée, et notez-les pour une utilisation ultérieure.
REMARQUE: Lors de l’analyse des LD, il est important de garder à l’esprit que leur taille et leur densité varient entre le centre et la périphérie (région sous-sarpolymale-SS) de la fibre. Par conséquent, l’analyse doit être effectuée séparément. - Calculez la valeur de 1/6 du MF pour délimiter la partie centrale de la fibre.
REMARQUE : Dans la macro, la valeur MF par défaut est définie sur 6, ce qui signifie que la réduction appliquée sera définie sur 1/6 de la MF. Cette valeur a été choisie sur la base des données empiriques obtenues à partir de la soleus d’animaux nourris avec un régime riche en graisses. Cependant, chaque chercheur doit modifier ce nombre en fonction des données empiriques et du muscle analysé, du type de fibre et de l’état de l’animal. - Dans la fenêtre ROI , cliquez sur Ajouter pour avoir un duplicata du premier ROI et sélectionnez le deuxième ROI qui apparaît sur la fenêtre.
- Dans la fenêtre principale des Fidji, cliquez sur Modifier | Sélection | Agrandissez et introduisez la valeur précédemment calculée (à partir de l’étape 6.2.6.) avec un signe moins avant le nombre et cliquez sur OK. Dans la fenêtre ROI, cliquez sur Ajouter[t] (un troisième ROI doit apparaître) et Supprimer, pour supprimer le deuxième ROI.
REMARQUE: Le chercheur peut vérifier les résultats en cliquant sur la case Afficher tout dans la fenêtre ROI . À ce stade, deux ROI doivent apparaître, l’un qui entoure tout le périmètre de la fibre (Figure 4B) et l’autre qui est placé au centre (Figure 4C). - Sélectionnez les deux ROI dans la fenêtre ROI et cliquez sur Plus | | XOR Ajouter[t]. Attendez qu’un troisième ROI apparaisse, qui correspond à la périphérie de la fibre (Figure 4D).
- Enregistrez le retour sur investissement en cliquant sur Plus | Enregistrer pour enregistrer les rois au cas où une réanalyse ultérieure des mêmes fibres serait nécessaire.
- Sélectionnez le canal Bodipy et ouvrez l’outil Seuil en cliquant sur Image | Ajuster | Seuil sur la fenêtre principale des Fidji.
- Dans la fenêtre contextuelle Seuil , définissez les valeurs sur 70/255, sélectionnez Yen | Méthode N&B, et cliquez sur Fond sombre | Postulez.
REMARQUE : Les valeurs appliquées sur le seuil peuvent varier en fonction des conditions de l’expérience et le seuil doit être défini de manière appropriée pour optimiser l’analyse. Une fenêtre N&B avec le signal Bodipy au-dessus de la limite de seuil affichée en blanc et l’arrière-plan en noir doit apparaître (comparez l’image Bodipy originale de la Figure 4E avec celle de la Figure 4F). - Allez dans la fenêtre principale de Fidji et cliquez sur Analyser | Définissez Mesures et, dans la fenêtre contextuelle, sélectionnez Zone, Fraction d’aire et Limite au seuil. Laissez les cases restantes décochées et les autres paramètres tels qu’ils apparaissent par défaut.
REMARQUE: Si le chercheur veut analyser la « circularité » des LD, qui est un index de la morphologie sphérique allant de 1 pour une sphère parfaite à 0 pour une ligne, cliquez sur la boîte descripteurs de forme de la fenêtre contextuelle Définir les mesures . - Accédez à la fenêtre ROI et sélectionnez le premier ROI. Dans la fenêtre principale des Fidji, cliquez sur Analyser | Outil Analyser les particules.
REMARQUE: Cet outil quantifie le nombre, la taille, la zone couverte et le pourcentage de la surface totale couverte par les particules à chaque sélection et enregistre les résultats sous forme de fichier de feuille de calcul. - Définissez les valeurs de 2 à Infinity (2-Infinity) dans la fenêtre Analyser les particules , cochez la case Pixels , conservez les valeurs de circularité par défaut, sélectionnez Résumer, puis cliquez sur OK.
REMARQUE: Pour vérifier les résultats sur la fibre, sous l’option Afficher , sélectionnez l’une des options disponibles. Pour que les informations de chaque LD soient reconnues sur la sélection d’un tableau, cochez l’option Afficher les résultats dans la fenêtre Analyser les particules . Les résultats de l’analyse de la surface totale de la fibre sont moyennés et résumés dans un tableau à plusieurs colonnes (Nombre, Superficie totale, Taille moyenne, % Superficie ; celles-ci correspondent au nombre de particules [LD], à la surface occupée par ces particules, à leur taille moyenne, et au pourcentage de la surface totale de la sélection occupée par les particules, respectivement). Pour calculer la densité, divisez le nombre de particules par la surface totale de chaque sélection. - Pour obtenir les valeurs du centre et de la périphérie de la fibre, répétez les étapes 6.2.14 et 6.2.15, en sélectionnant le deuxième (centre) et le troisième ROI (périphérie) à chaque fois.
- Enregistrez les résultats en cliquant sur Fichier | Enregistrer sous dans la fenêtre Résumé .
REMARQUE: Incluez le type de fibre, la condition et le nom de l’image sur le nom attribué des résultats pour faciliter l’unification ultérieure et l’analyse statistique des données. Pour analyser le reste des fibres dans la même image, répétez les étapes 6.2.3 à 6.2.17. Pour l’analyse statistique, au moins 10 à 15 fibres de chaque type doivent être analysées par animal.
- Analyser les images de Bodipy et de laminine obtenues sur le confocal en utilisant Fidji pour la quantification des TA.
Access restricted. Please log in or start a trial to view this content.
Résultats
Le protocole décrit ici fournit une méthode efficace pour quantifier facilement les LD d’une manière spécifique à un type de fibre et à un niveau subcellulaire. Il montre comment, en congelant ensemble deux muscles de taille similaire, tels que l’EDL et le soleus, le temps et les ressources consacrés aux étapes suivantes sont réduits de moitié.
Un protocole complet est fourni pour l’immunocoloration, l’acquisition d’images et l’analyse des différentes isoformes de MyHC e...
Access restricted. Please log in or start a trial to view this content.
Discussion
Le protocole détaillé ici décrit une méthode efficace pour quantifier les LD marquées avec Bodipy sur une base spécifique au type de fibre et au subcellulaire. Au cours des dernières années, les colorants lipidiques classiques, tels que l’ORO ou le Sudan Black B, ont été remplacés par une nouvelle gamme de colorants fluorescents lipophiles perméables aux cellules qui se lient aux lipides neutres (par exemple, Bodipy). Disponible sous différents conjugués, Bodipy s’est avéré très efficace pour marquer...
Access restricted. Please log in or start a trial to view this content.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à déclarer.
Remerciements
Ce travail a été soutenu par des subventions du Fonds National de la Recherche Scientifique (FNRS-Crédit de Recherche J.0022.20) et de la Société Francophone du Diabète (SFD-Roche Diabetes Care).C.M.S. est titulaire d’une bourse de doctorat du FRIA (FNRS). M.A.D.-L.d.C. a reçu une bourse du Programme d’Excellence Internationale Wallonie-Bruxelles.
Les auteurs remercient Alice Monnier pour sa contribution au développement de ce protocole et Caroline Bouzin pour son expertise et son aide technique dans le processus d’acquisition d’images. Nous remercions également la plateforme d’imagerie 2IP-IREC pour l’accès au cryostat et aux microscopes (2IP-IREC Imaging Platform, Institut de recherche expérimentale et clinique, Université Catholique de Louvain, 1200 Bruxelles, Belgique). Enfin, les auteurs tiennent à remercier Nicolas Dubuisson, Romain Versele et Michel Abou-Samra pour leur critique constructive du manuscrit. Certaines des figures de cet article ont été créées avec BioRender.com.
Access restricted. Please log in or start a trial to view this content.
matériels
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| AxioCam 506 mono 6 Mpix camera | Zeiss | ||
| AxioCam MRm 1.4MPix CCD camera | Zeiss | ||
| Chemical hood | Potteau Labo | EN-14175 | |
| Confocal microscope | Zeiss | LSM800 | |
| Cork discs (ø 20 mm, 3 mm thick) | Electron Microscopy Sciences | 63305 | |
| Cryo-Gloves | Tempshield | 16072252 | |
| Cryostat | Thermo Scientific | Microm Cryo Star HM 560 | |
| Dissecting Stereo Microscope SMZ745 | Nikon | ||
| Dry Ice | |||
| Dumont Forceps | F.S.T | 11295-10 | |
| Epifluorescence microscope | Zeiss | AxioImage-Apotome Z1 | |
| Extra Fine Bonn Scissors | F.S.T | 14084-08 | |
| FisherBrand Disposable Base Molds (0.7 x 0.7 cm) | ThermoFisher | 22-363-552 | Used to cut a piece to hold the muscle on the cork for freezing |
| Glass petri dish (H 25 mm, ø 150 mm) | BRAND Petri dish, MERK | BR455751 | Used to place the muscles on ice during dissection |
| ImmEdge Hydrophobic barrier PAP Pen | Vector Labs | H-4000 | Used to create an hidrophobic barrier around the muscle sections |
| Incubator | MMM Medcenter | Incucell 707 | |
| Microscope Cover Glasses (24x50 mm) | Assistent | 40990151 | |
| Microscope Slide Boxes | Kartell | 278 | Used as humid chambers for immunohistochemistry |
| Neck holder | Linie zwo | SB-035X-02 | Used as strap to hold the stainless steel tumbler |
| No 15 Sterile Carbon Steel Scalpel Blade | Swann-Morton | 0205 | |
| Paint brushes | Van Bleiswijck | Amazon B07W7KJQ2X | Used to handle cryosections |
| Permanent Marker Pen Black | Klinipath/VWR | 98307-R | Used to label slides |
| Pierce Fixation Forceps | F.S.T | 18155-13 | |
| Polystyrene Box | H 12 cm x L 25 cm x W 18 cm, used as a liquid nitrogen container and to transport the samples to the cryostat | ||
| Scalpel Handle, 125 mm (5"), No. 3 | Aesculap | BB073R | |
| Stainless Steel Cup 10oz | Eboxer | B07GFCBPFH | Tumbler to fill with isopentene for muscle freezing |
| Superfrost Ultra Plus slides | ThermoFisher | J1800AMNZ | |
| Surgical tweezers 1/2 teeth | Medische Vakhandel | 1303152 | Also called "Rat teeth tweezers" |
| Vannas Spring Scissors - 3 mm Cutting Edge | F.S.T | 15000-00 | |
| Weighing boats | VWR international | 611-2249 | |
| Whole-Slide Scanner for Fluorescence | Zeiss | Axio Scan.Z1 | |
| Reagents | |||
| Alexa Fluor 405 Goat Anti-Mouse IgG2b | Sigma-Aldrich | SAB4600477 | Used at a final concentration of 1:500 |
| Alexa Fluor 488 Goat Anti-Mouse IgG1 | ThermoFisher | A-21121 | Used at a final concentration of 1:500 |
| Alexa Fluor 568 Goat Anti-Mouse IgM | Abcam | ab175702 | Used at a final concentration of 1:1,000 |
| Alexa Fluor 647 goat anti rat-IgG (H+L) secondary antibody | ThermoFisher | A-21247 | Used at a final concentration of 1:500 |
| BODIPY-493/503 (4,4-difluoro-1,3,5,7,8-pentametil-4-bora-3a,4a-diaza-s-indaceno) | ThermoFisher | D3922 | Used at a final concentration of 1 µg/mL |
| BODIPY-558/568 C12 (4,4-Difluoro-5-(2-Thienyl)-4-Bora-3a,4a-Diaza-s-Indacene-3-Dodecanoic Acid) | ThermoFisher | D3835 | Used at a final concentration of 1 µg/mL |
| DAPI (4',6-diamidino-2-phenylindole) | ThermoFisher | D1306 | Used at a final concentration of 0.5 µg/mL |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich | D-8418 | Used to solve Bodipy for the 1 mg/mL stock solution. CAUTION: Toxic and flammable. Vapors may cause irritation. Manipulate in a fume hood. Avoid direct contact with skin. Wear rubber gloves, protective eye goggles. |
| Formaldehyde solution 4%, buffered, pH 6.9 | Sigma-Aldrich | 1004969011 | CAUTION: May cause an allergic skin reaction. Suspected of causing genetic defects. May cause cancer. Manipulate in a fume hood. Avoid direct contact with skin. Wear rubber gloves, protective eye goggles. |
| Isopentane GPR RectaPur | VWR international | 24872.298 | CAUTION: Extremely flammable liquid and vapor. May be fatal if swallowed and enters airways. May cause drowsiness or dizziness. Repeated exposure may cause skin dryness or cracking. Wear protective gloves/protective clothing/eye protection/face protection. |
| Liquid Nitrogen | CAUTION: Extremely cold. Wear gloves. Handle slowly to minimize boiling and splashing and in well ventilated areas. Use containers designed for low-temperature liquids. | ||
| Mouse on mouse Blocking Reagent | Vector Labs | MKB-2213-1 | Used at concentration of 1:30 |
| Myosin heavy chain Type I (BA-D5-s Primary Antibody) Gene: MYH7, monoclonal bovine anti mouse IgG2b | DSHB University of Iowa | BA-D5-supernatant | Used at a final concentration of 1:10 |
| Myosin heavy chain Type IIA (SC-71-s Primary Antibody) Gene: MYH2, Monoclonal bovine anti mouse IgG1 | DSHB University of Iowa | SC-71-supernatant | Used at a final concentration of 1:10 |
| Myosin heavy chain Type IIX (6H1-s Primary Antibody), Gene: MYH1, Monoclonal rabbit anti mouse IgM | Developmental Studies Hybridoma Bank, University of Iowa | 6H1-supernatant | Used at a final concentration of 1:5 |
| Normal Goat Serum (NGS) | Vector Labs | S-1000 | |
| PBS 0.1 M | Commonly used on histology laboratories | ||
| ProLong Gold Antifade Mountant | Invitrogen | P36930 | |
| Rat anti-Laminin-2 (α-2 Chain) primary antibody (monoclonal) | Sigma-Aldrich | L0663 | Used at a final concentration of 1:1,000 |
| Tissue-Tek O.C.T compound | Sakura | 4583 | |
| Software | |||
| Adobe Illustrator CC | Adobe Inc. | Used to design the figures | |
| Adobe Photoshop | Adobe Inc. | Confocal software | |
| BioRender | https://biorender.com/ | Used to design the figures | |
| Fiji/ImageJ | https://imagej.net/software/fiji/ | Used to analyse the acquired images | |
| Microsoft PowerPoint | Microsoft | Used to reconstruct the histology of the whole muscle after scanning the fiber types | |
| Zen Blue 2.6 | Zeiss | Used to reconstruct the histology of the whole muscle after scanning the fiber types |
Références
- Correa-de-Araujo, R., et al. Myosteatosis in the context of skeletal muscle function deficit: an interdisciplinary workshop at the National Institute on Aging. Frontiers in Physiology. 11, 963(2020).
- Miljkovic, I., et al. Greater skeletal muscle fat infiltration is associated with higher all-cause and cardiovascular mortality in older men. Journals of Gerontology Series A: Biomedical Sciences and Medical Sciences. 70 (9), 1133-1140 (2015).
- Nachit, M., et al. Myosteatosis rather than sarcopenia associates with non-alcoholic steatohepatitis in non-alcoholic fatty liver disease preclinical models. Journal of Cachexia, Sarcopenia, and Muscle. 12 (1), 144-158 (2021).
- Aleixo, G. F. P., et al. Myosteatosis and prognosis in cancer: Systematic review and meta-analysis. Critical Reviews in Oncology/Hematolgoy. 145, 102839(2020).
- Gemmink, A., Schrauwen, P., Hesselink, M. K. C. Exercising your fat (metabolism) into shape: a muscle-centred view. Diabetologia. 63 (8), 1453-1463 (2020).
- van Loon, L. J. Use of intramuscular triacylglycerol as a substrate source during exercise in humans. Journal of Applied Physiology. 97 (4), 1170-1187 (2004).
- Coen, P. M., Goodpaster, B. H. Role of intramyocelluar lipids in human health. Trends in Endocrinology and Metabolism. 23 (8), 391-398 (2012).
- Seibert, J. T., Najt, C. P., Heden, T. D., Mashek, D. G., Chow, L. S. Muscle lipid droplets: cellular signaling to exercise physiology and beyond. Trends in Endocrinology and Metabolism. 31 (12), 928-938 (2020).
- Bergman, B. C., Goodpaster, B. H. Exercise and muscle lipid content, composition, and localization: influence on muscle insulin sensitivity. Diabetes. 69 (5), 848-858 (2020).
- Nielsen, J., Christensen, A. E., Nellemann, B., Christensen, B. Lipid droplet size and location in human skeletal muscle fibers are associated with insulin sensitivity. American Journal of Physiology-Endocrinology and Metabolism. 313 (6), 721-730 (2017).
- Covington, J. D., et al. Intramyocellular lipid droplet size rather than total lipid content is related to insulin sensitivity after 8 weeks of overfeeding. Obesity (Silver Spring). 25 (12), 2079-2087 (2017).
- Bosma, M. Lipid droplet dynamics in skeletal muscle). Experimental Cell Research. 340 (2), 180-186 (2016).
- Nielsen, J., et al. Increased subsarcolemmal lipids in type 2 diabetes: effect of training on localization of lipids, mitochondria, and glycogen in sedentary human skeletal muscle. American Journal of Physiology-Endocrinology and Metabolism. 298 (3), 706-713 (2010).
- Ferreira, R., et al. Subsarcolemmal and intermyofibrillar mitochondria proteome differences disclose functional specializations in skeletal muscle. Proteomics. 10 (17), 3142-3154 (2010).
- Barrett, J. S., Whytock, K. L., Strauss, J. A., Wagenmakers, A. J. M., Shepherd, S. O. High intramuscular triglyceride turnover rates and the link to insulin sensitivity: influence of obesity, type 2 diabetes and physical activity. Applied Physiology, Nutrition and Metabolism. , 1-14 (2022).
- Daemen, S., et al. Distinct lipid droplet characteristics and distribution unmask the apparent contradiction of the athlete's paradox. Molecular Metabolism. 17, 71-81 (2018).
- Bredella, M. A., Ghomi, R. H., Thomas, B. J., Miller, K. K., Torriani, M. Comparison of 3.0 T proton magnetic resonance spectroscopy short and long echo-time measures of intramyocellular lipids in obese and normal-weight women. Journal of Magnetic Resonance Imaging. 32 (2), 388-393 (2010).
- Schrauwen-Hinderling, V. B., Hesselink, M. K., Schrauwen, P., Kooi, M. E. Intramyocellular lipid content in human skeletal muscle. Obesity (Silver Spring). 14 (3), 357-367 (2006).
- De Bock, K., et al. Evaluation of intramyocellular lipid breakdown during exercise by biochemical assay, NMR spectroscopy, and Oil Red O staining. American Journal of Physiology-Endocrinology and Metabolism. 293 (1), 428-434 (2007).
- Koopman, R., Schaart, G., Hesselink, M. K. Optimisation of oil red O staining permits combination with immunofluorescence and automated quantification of lipids. Histochemistry and Cell Biology. 116 (1), 63-68 (2001).
- Gueugneau, M., et al. Skeletal muscle lipid content and oxidative activity in relation to muscle fiber type in aging and metabolic syndrome. Journal of Gerontology Series A: Biomedical Sciences and Medical Sciences. 70 (5), 566-576 (2015).
- Gemmink, A., et al. Super-resolution microscopy localizes perilipin 5 at lipid droplet-mitochondria interaction sites and at lipid droplets juxtaposing to perilipin 2. Biochimica et Biophysica Acta-Molecular and Cell Biology of Lipids. 1863 (11), 1423-1432 (2018).
- Spangenburg, E. E., Pratt, S. J. P., Wohlers, L. M., Lovering, R. M. Use of BODIPY (493/503) to visualize intramuscular lipid droplets in skeletal muscle. Journal of Biomedicine and Biotechnology. 598358, (2011).
- Prats, C., et al. An optimized histochemical method to assess skeletal muscle glycogen and lipid stores reveals two metabolically distinct populations of type I muscle fibers. PLoS One. 8 (10), 77774(2013).
- Strauss, J. A., Shepherd, D. A., Macey, M., Jevons, E. F. P., Shepherd, S. O. Divergence exists in the subcellular distribution of intramuscular triglyceride in human skeletal muscle dependent on the choice of lipid dye. Histochemistry and Cell Biology. 154 (4), 369-382 (2020).
- Shepherd, S. O., et al. Sprint interval and traditional endurance training increase net intramuscular triglyceride breakdown and expression of perilipin 2 and 5. Journal of Physiology. 591 (3), 657-675 (2013).
- Whytock, K. L., et al. A 7-day high-fat, high-calorie diet induces fibre-specific increases in intramuscular triglyceride and perilipin protein expression in human skeletal muscle. Journal of Physiology. 598 (6), 1151-1167 (2020).
- Wang, C., Yue, F., Kuang, S. Muscle histology characterization using h&e staining and muscle fiber type classification using immunofluorescence staining. Bio-Protocol. 7 (10), (2017).
- Meng, H., et al. Tissue triage and freezing for models of skeletal muscle disease. Journal of Visualized Experiments: JoVE. (89), e51586(2014).
- Kumar, A., Accorsi, A., Rhee, Y., Girgenrath, M. Do's and don'ts in the preparation of muscle cryosections for histological analysis. Journal of Visualized Experiments: JoVE. (99), e52793(2015).
- Leiva-Cepas, F., et al. Laboratory methodology for the histological study of skeletal muscle. Archivos de Medicina del Deporte. 35 (186), 254-262 (2018).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Schiaffino, S., et al. Three myosin heavy chain isoforms in type 2 skeletal muscle fibres. Journal of Muscle Research & Cell Motility. 10 (3), 197-205 (1989).
- Komiya, Y., et al. Mouse soleus (slow) muscle shows greater intramyocellular lipid droplet accumulation than EDL (fast) muscle: fiber type-specific analysis. Journal of Muscle Research & Cell Motility. 38 (2), 163-173 (2017).
- Andrich, D. E., et al. Altered lipid metabolism impairs skeletal muscle force in young rats submitted to a short-term high-fat diet. Frontiers in Physiology. 9, 1327(2018).
- Schiaffino, S. Fibre types in skeletal muscle: a personal account. Acta Physiologica. 199 (4), 451-463 (2010).
- Bloemberg, D., Quadrilatero, J. Rapid determination of myosin heavy chain expression in rat, mouse, and human skeletal muscle using multicolor immunofluorescence analysis. PLoS One. 7 (4), 35273(2012).
- Gemmink, A., et al. Decoration of intramyocellular lipid droplets with PLIN5 modulates fasting-induced insulin resistance and lipotoxicity in humans. Diabetologia. 59 (5), 1040-1048 (2016).
- Askinas, C., et al. Biophotonics Congress: Biomedical Optics Congress 2018 (Microscopy/Translational/Brain/OTS). , JTu3A.4 (Optical Society of America) (2018).
- Morén, B., et al. EHD2 regulates adipocyte function and is enriched at cell surface-associated lipid droplets in primary human adipocytes. Molecular Biology of the Cell. 30 (10), 1147-1159 (2019).
- Benador, I. Y., et al. Mitochondria bound to lipid droplets have unique bioenergetics, composition, and dynamics that support lipid droplet expansion. Cell Metabolism. 27 (4), 869-885 (2018).
- de la Rosa Rodriguez, M. A., et al. Hypoxia-inducible lipid droplet-associated induces DGAT1 and promotes lipid storage in hepatocytes. Molecular Metabolism. 47, 101168(2021).
- Jevons, E. F. P., Gejl, K. D., Strauss, J. A., Ørtenblad, N., Shepherd, S. O. Skeletal muscle lipid droplets are resynthesized before being coated with perilipin proteins following prolonged exercise in elite male triathletes. American Journal of Physiology-Endocrinology and Metabolism. 318 (3), 357-370 (2020).
- Ohsaki, Y., Maeda, T., Fujimoto, T. Fixation and permeabilization protocol is critical for the immunolabeling of lipid droplet proteins. Histochemistry and Cell Biology. 124 (5), 445-452 (2005).
- Prats, C., et al. Decrease in intramuscular lipid droplets and translocation of HSL in response to muscle contraction and epinephrine. Journal of Lipid Research. 47 (11), 2392-2399 (2006).
- Listenberger, L. L., Brown, D. A. Fluorescent detection of lipid droplets and associated proteins. Current Protocols in Cell Biology. , Chapter 24 (Unit 24.22) (2007).
- Xue, Y., Lim, S., Bråkenhielm, E., Cao, Y. Adipose angiogenesis: quantitative methods to study microvessel growth, regression and remodeling in vivo. Nature Protocols. 5 (5), 912-920 (2010).
- Muliyil, S., et al. ADAM17-triggered TNF signalling protects the ageing Drosophila retina from lipid droplet-mediated degeneration. The EMBO Journal. 39 (17), 104415(2020).
- Yan, Q., et al. Autophagy activation contributes to lipid accumulation in tubular epithelial cells during kidney fibrosis. Cell Death Discovery. 4, 2(2018).
- Coassin, S., et al. Investigation and functional characterization of rare genetic variants in the adipose triglyceride lipase in a large healthy working population. PLoS Genetics. 6 (12), 1001239(2010).
- Daemen, S., van Zandvoort, M., Parekh, S. H., Hesselink, M. K. C. Microscopy tools for the investigation of intracellular lipid storage and dynamics. Molecular Metabolism. 5 (3), 153-163 (2016).
- Chen, Q., et al. Rab8a deficiency in skeletal muscle causes hyperlipidemia and hepatosteatosis by impairing muscle lipid uptake and storage. Diabetes. 66 (9), 2387-2399 (2017).
- Gemmink, A., et al. Decoration of myocellular lipid droplets with perilipins as a marker for in vivo lipid droplet dynamics: A super-resolution microscopy study in trained athletes and insulin resistant individuals. Biochimica et Biophysica Acta (BBA)-Molecular and Cell Biology of Lipids. 1866 (2), 158852(2021).
- Bergman, B. C., Hunerdosse, D. M., Kerege, A., Playdon, M. C., Perreault, L. Localisation and composition of skeletal muscle diacylglycerol predicts insulin resistance in humans. Diabetologia. 55 (4), 1140-1150 (2012).
Access restricted. Please log in or start a trial to view this content.
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon