È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Tipo di fibra e analisi subcellulare specifica del contenuto di goccioline lipidiche nel muscolo scheletrico
In questo articolo
Riepilogo
Prove crescenti indicano che l'eccessiva infiltrazione di lipidi all'interno del muscolo scheletrico provoca lipotossicità e diabete. Qui, presentiamo un protocollo completo, che include l'elaborazione dei tessuti, la colorazione con Bodipy, l'acquisizione di immagini e l'analisi, per quantificare le dimensioni, la densità e la distribuzione subcellulare delle goccioline lipidiche in un modo specifico per il tipo di fibra.
Abstract
L'infiltrazione lipidica del muscolo scheletrico, nota come miosteatosi, aumenta con l'obesità e l'invecchiamento. La miosteatosi è stata anche recentemente scoperta come fattore prognostico negativo per molti altri disturbi come le malattie cardiovascolari e il cancro. L'eccessiva infiltrazione lipidica diminuisce la massa muscolare e la forza. Si traduce anche in lipotossicità e insulino-resistenza a seconda del contenuto lipidico intramiocellulare totale, della morfologia delle goccioline lipidiche (LD) e della distribuzione subcellulare. Anche il tipo di fibra (ossidativo vs glicolitico) è importante, poiché le fibre ossidative hanno una maggiore capacità di utilizzare i lipidi. A causa delle loro implicazioni cruciali in fisiopatologia, sono giustificati studi approfonditi sulla dinamica e la funzione della LD in un modo specifico per tipo di fibra.
Qui viene presentato un protocollo completo per la quantificazione del contenuto lipidico intramiocellulare e l'analisi della morfologia della LD e della distribuzione subcellulare in modo specifico per tipo di fibra. A tal fine, le criosezioni muscolari seriali sono state colorate con il colorante fluorescente Bodipy e anticorpi contro le isoforme a catena pesante della miosina. Questo protocollo consente l'elaborazione simultanea di diversi muscoli, risparmiando tempo ed evitando possibili artefatti e, grazie a una macro personalizzata creata nelle Fiji, è anche possibile l'automatizzazione dell'analisi LD.
Introduzione
L'infiltrazione lipidica del muscolo scheletrico, nota come miosteatosi, aumenta con l'obesità e l'invecchiamento. La miosteatosi è correlata negativamente con la massa e la forza muscolare e con la sensibilità all'insulina1. Inoltre, studi recenti indicano che il grado di miosteatosi potrebbe essere utilizzato come fattore prognostico per altre condizioni come la malattia cardiovascolare2, la steatosi epatica non alcolica3 o il cancro4. I lipidi possono accumularsi nel muscolo scheletrico tra le fibre muscolari come lipidi extramiocellulari o all'interno delle fibre, come lipidi intramiocellulari (IMCL). Gli IMCL sono prevalentemente immagazzinati come trigliceridi in goccioline lipidiche (LD) che vengono utilizzate come combustibile metabolico durante l'esercizio fisico 5,6. Tuttavia, quando l'offerta lipidica supera la domanda, o quando i mitocondri diventano disfunzionali, gli IMCL saranno implicati nella resistenza all'insulina muscolare, come si vede in individui metabolicamente malsani e obesi e nei pazienti con diabete di tipo 27. Curiosamente, gli atleti di resistenza hanno livelli simili, se non più alti, di IMCL a quelli trovati nei pazienti obesi con diabete mellito di tipo 2, pur mantenendo un'elevata sensibilità all'insulina. Questo fenomeno è descritto come il "paradosso dell'atleta"8,9, ed è spiegato da una valutazione più sfumata degli LCD muscolari, relativi alle loro dimensioni, densità, localizzazione, dinamica e composizione delle specie lipidiche.
In primo luogo, la dimensione della LD è inversamente correlata alla sensibilità all'insulina e alla forma fisica10,11. In effetti, le LD più piccole mostrano una superficie relativamente maggiore per l'azione della lipasi e, quindi, potenzialmente hanno una maggiore capacità di mobilitare i lipidi12. In secondo luogo, la densità LD (numero/superficie) svolge un ruolo controverso nell'azione dell'insulina 8,10; tuttavia, sembra essere aumentato negli atleti. In terzo luogo, la localizzazione subcellulare delle LD è importante, poiché le LD situate appena sotto la membrana superficiale (subsarcolemmale o periferica) esercitano un effetto più deleterio sulla sensibilità all'insulina rispetto a quelle centrali 8,9,13. Questi ultimi forniscono carburante ai mitocondri centrali, che hanno una maggiore attività respiratoria e sono più specializzati per soddisfare l'elevata domanda di energia richiesta per la contrazione14. Al contrario, le LD periferiche forniscono mitocondri subsarcolemmali, che sono coinvolti nei processi correlati alla membrana8. Infine, oltre ai trigliceridi, specifici lipidi complessi all'interno del muscolo possono essere più deleteri di altri. Ad esempio, il diacilglicerolo, l'acil-CoA a catena lunga e le ceramidi possono accumularsi nel muscolo quando il tasso di turnover dei trigliceridi è basso, compromettendo così la segnalazione dell'insulina 9,15. Tornando al "paradosso dell'atleta", gli atleti di resistenza hanno un numero elevato di LD centrali più piccoli con elevati tassi di turnover nelle fibre di tipo I (ossidative), mentre i pazienti obesi e diabetici hanno LD periferici più grandi con bassi tassi di turnover nelle fibre di tipo II (glicolitiche) 8,15,16. Oltre al loro ruolo nell'immagazzinamento e nel rilascio di energia, gli LCD tramite acidi grassi derivati (FA) e una proteina del mantello (perilipina 5) potrebbero anche funzionare come attori critici coinvolti nella regolazione trascrizionale dell'ossidazione fa e della biogenesi mitocondriale8. A causa delle loro implicazioni cruciali in fisiologia e fisiopatologia, sono giustificati studi approfonditi sulla dinamica e le funzioni dei LD.
Sebbene esistano diverse tecniche per studiare le IMCL, non sono tutte adatte a quantificare con precisione le dimensioni, la densità e la distribuzione dei LD in modo specifico per le fibre. Ad esempio, la valutazione delle IMCL mediante spettroscopia di risonanza magnetica, pur essendo non invasiva, offre un livello di risoluzione che non è sufficiente per studiare le dimensioni e la posizione precisa degli LCD all'interno della fibra, e non è specifico per il tipo di fibra17,18. Allo stesso modo, le tecniche biochimiche eseguite su omogeneizzati muscolari interi19 non possono valutare la posizione e le dimensioni dei lipidi. Di conseguenza, il metodo più adeguato per analizzare la morfologia e la posizione della LD è la microscopia elettronica a trasmissione quantitativa13, ma questa tecnica è costosa e richiede tempo. Pertanto, l'imaging a fluorescenza confocale su preparati con coloranti come Oil Red O (ORO)20,21, monodansylpentane (MDH)22 o Bodipy 23,24,25, è emerso come lo strumento migliore per questi studi.
Qui viene descritto un protocollo completo, che include il campionamento e l'elaborazione dei tessuti, la colorazione di Bodipy e l'acquisizione e l'analisi di immagini confocali per quantificare le dimensioni, il numero e la localizzazione di LD nelle criosezioni muscolari del topo. Poiché gli IMCL non sono distribuiti uniformemente tra fibre ossidative e glicolitiche e ogni tipo di fibra regola la dinamica LD in modo diverso, lo studio delle IMML deve essere specifico per il tipo di fibra 16,25,26,27. Pertanto, questo protocollo utilizza l'immunofluorescenza su sezioni seriali per identificare le isoforme a catena pesante della miosina (MyHC) espresse da ciascuna fibra. Un altro vantaggio di questo protocollo è l'elaborazione simultanea di un muscolo glicolitico (estensore digitorum longus, EDL) e di un muscolo ossidativo (soleo) posti fianco a fianco prima del congelamento (Figura 1). Questa elaborazione simultanea non solo consente di risparmiare tempo, ma evita anche la variabilità dovuta all'elaborazione separata dei campioni.
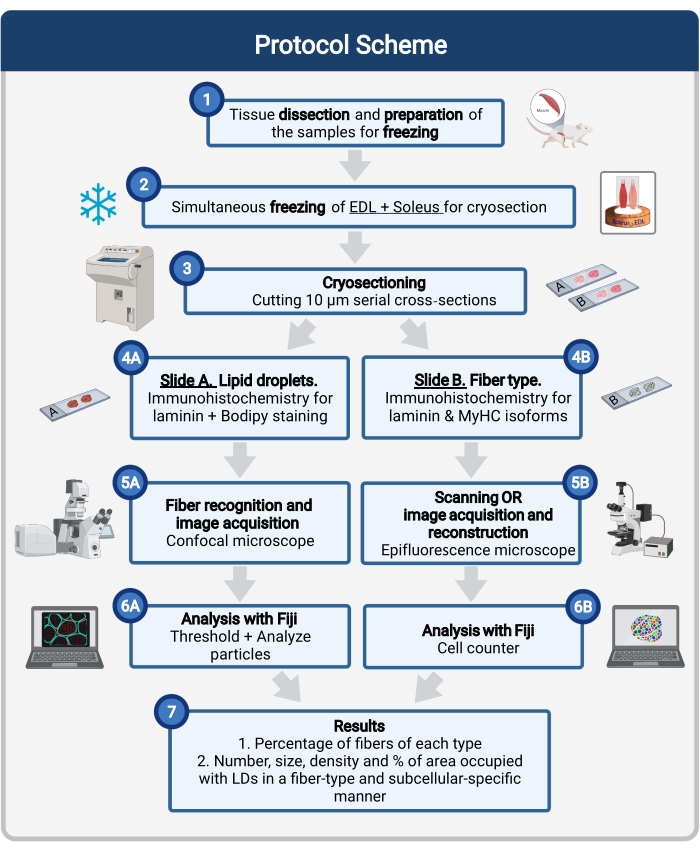
Figura 1: Panoramica schematica della procedura. Dopo la dissezione muscolare (1), i muscoli selezionati di dimensioni simili vengono preparati e congelati insieme (2). Le sezioni trasversali seriali di 10 μm sono ottenute utilizzando un criostato e montate direttamente su vetrini di adesione (3). Da due vetrini seriali, il primo (4A) è immunomarcato per la laminina e colorato con Bodipy per riconoscere gli LD e il secondo (4B) è immunocolorato con anticorpi contro i MyHC per il riconoscimento dei tipi di fibre muscolari. Le immagini vengono acquisite utilizzando un microscopio confocale per Bodipy (5A) e un microscopio a epifluorescenza per i tipi di fibre muscolari (5B). Le immagini vengono analizzate nelle Figi applicando una soglia e quantificando le particelle (6A) per ottenere il numero, la dimensione media, la densità e la percentuale dell'area totale occupata da LD (7) o celle di conteggio (6B) per ottenere la percentuale di fibre di ciascun tipo nella sezione (7). Abbreviazioni: LDs = goccioline lipidiche; EDL = estensore digitorum longus; MyHC = isoforme a catena pesante della miosina. Fare clic qui per visualizzare una versione più grande di questa figura.
Protocollo
Tutte le procedure condotte sui topi sono state approvate dal Comitato etico per la sperimentazione animale del settore medico dell'Université Catholique de Louvain (2019 / UCL / MD / 013).
1. Dissezione e preparazione dei campioni per il congelamento
- Etichettare un pezzo di sughero di 3 mm di spessore per ogni paio di muscoli.
- Attraverso una piccola incisione realizzata con una lama al centro del tappo, inserire perpendicolarmente un pezzo rettangolare di plastica rigida (0,5 cm W, 1 cm H) che servirà da supporto (Figura 2B).
NOTA: La dimensione del pezzo di plastica rettangolare dipenderà dalla dimensione del muscolo. Qui, le dimensioni descritte sono adattate alle dimensioni del soleo (~ 9 mg, 1 cm L, 2-3 mm W) e EDL (~ 5 mg, 1 cm L, 2-3 mm W) di un topo maschio C57BL / 6J di 3 mesi. - Al momento della dissezione, rimuovere il soleo e l'EDL dell'arto posteriore del topo. Per evitare che i campioni si secchino durante la dissezione, metterli su un impacco leggermente inumidito con soluzione salina in una capsula di Petri posta su ghiaccio.
NOTA: Vedi Wang et al.28 per le spiegazioni su come sezionare questi due muscoli degli arti. - Posizionare una piccola goccia di composto OCT (Optimal Cutting Temperature) sulla giunzione di sughero/plastica, evitando bolle d'aria.
- Eliminare l'umidità in eccesso dai campioni asciugandoli delicatamente con un tovagliolo di carta (Figura 2A) e posizionare entrambi i muscoli sulla plastica perpendicolare al tappo (Figura 2C).
- Controllare l'orientamento delle miofibre muscolari al microscopio stereo (Figura 2D).
NOTA: È importante non coprire il muscolo con OCT, poiché il suo effetto isolante impedirebbe il congelamento rapido e produrrà artefatti di congelamento.
2. Congelamento di campioni di muscolo scheletrico per la criosezione
ATTENZIONE: Il congelamento del muscolo deve essere effettuato sotto un cappuccio chimico, indossando adeguati dispositivi di protezione individuale (vedere la Tabella dei materiali).
- Utilizzare un tumbler in acciaio inossidabile (~8 cm H, 6 cm Ø) con due cinghie laterali attaccate ad esso lunghe almeno 25 cm (Figura 2F) e riempire il tumbler fino a 2/3 della sua capacità con isopentano.
- Afferrando il tumbler per le cinghie, immergerlo delicatamente in una scatola di polistirolo riempita con azoto liquido in modo che il livello di azoto all'esterno del contenitore sia superiore al livello di isopentano all'interno (Figura 2F,G).
ATTENZIONE: Quando il tumbler entra in contatto con l'azoto, lo shock termico può causare vortici. Assicurarsi che il tumbler sia sufficientemente immerso, ma evitare l'ingresso di azoto in quanto ciò causerebbe precipitazioni isopentane. Se ciò accade, lasciare raffreddare l'isopentano, riempire il tumbler con nuovo isopentano e ricominciare da capo. - Quando l'interno del tumbler è completamente coperto da uno strato solido bianco di isopentano, estrarlo dalla scatola di azoto liquido (Figura 2H).
NOTA: La temperatura di fusione dell'isopentano è -159 °C, i bordi del tumbler diventeranno bianchi quando fa abbastanza freddo. - Mescolare delicatamente i pezzi di isopentano solido nell'isopentano liquido rimanente con una pinza fino a quando l'intero volume diventa di nuovo liquido.
- Immergere nuovamente il tumbler nell'azoto liquido fino a formare ciottoli bianchi su tutto il fondo e sui bordi del tumbler (Figura 2G,H).
NOTA: Questa seconda fase di raffreddamento garantisce la temperatura di congelamento appropriata dell'isopentano. - Rimuovere il tumbler dall'azoto liquido e immergere rapidamente i muscoli nella parte inferiore del tumbler, tenendo il tappo con una pinzetta a denti di ratto. Ruotare il tappo per 15 s nell'isopentano e conservarlo a -80 °C fino alla lavorazione (Figura 2I).
NOTA: per la conservazione a breve termine, i campioni possono essere conservati in un congelatore a -20 °C. Il protocollo completo per il congelamento rapido del muscolo scheletrico è già stato pubblicato altrove. Per riferimenti dettagliati e risoluzione dei problemi, vedere: Meng et al.29, Kumar et al.30 e Leiva-Cepas et al.31.
3. Criosezione
- Portare i campioni nella camera di un criostato precedentemente raffreddato a -20 °C e la temperatura della lama impostata a -25 °C.
NOTA: Trasportare i campioni dal congelatore -20 °C/-80 °C al criostato in una scatola di polistirolo riempita con ghiaccio secco e lasciare che i campioni si equilibrino per almeno 20-30 minuti alla temperatura della camera prima del taglio. - Rimuovere la plastica dal tappo con una pinzetta fine e posizionare il disco del campione di criostato sulla piastra di congelamento rapido per raffreddare. Una volta che la piastra ha raggiunto -50 °C, posizionare un po' di OCT sul disco e posizionare rapidamente il tappo sopra il disco, premendo con decisione. Attendere che l'OCT si solidifichi e che il tappo sia ben fissato sul disco.
- Posizionare il disco sulla testa dell'oggetto nell'orientamento di taglio desiderato (Figura 2J, K) e tagliare il blocco muscolare oltre almeno 1/3 della lunghezza dei muscoli e fino a quando entrambe le sezioni trasversali dei muscoli sono visibili.
- Impostare lo spessore di taglio a 10 μm e posizionare una sezione su un vetrino di adesione per verificare il corretto orientamento delle fibre su un microscopio a campo luminoso.
NOTA: Il controllo dell'orientamento trasversale delle fibre è fondamentale. Se le fibre non sono adeguatamente orientate, regolare l'angolo della testa dell'oggetto, tagliare un'altra sezione e controllare di nuovo. - Posizionare due sezioni trasversali seriali su due vetrini di adesione premarcati: un vetrino per determinare i tipi di fibre, l'altro per quantificare il contenuto lipidico (Figura 2L).
NOTA: Ulteriori sezioni trasversali seriali possono essere ottenute e mantenute a -80 °C per altri studi istologici. Tuttavia, per evitare di alterare il contenuto lipidico e la morfologia intracellulare24, è essenziale elaborare i primi due vetrini subito dopo il taglio per evitare l'essiccazione all'aria. Il congelamento e lo scongelamento dei vetrini per la quantificazione LD avrebbero lo stesso effetto ed è quindi altamente sconsigliabile.
4. Tipizzazione delle fibre e colorazione Bodipy
- Rilevazione immunoistochimica del tipo di fibra muscolare
NOTA: Per il seguente protocollo, un volume totale di soluzione di 250 μL è sufficiente a coprire l'intera sezione muscolare circondata da un cerchio disegnato con una penna idrofobica delle dimensioni approssimative di una moneta da 1 centesimo.- Circondare le sezioni con un contorno disegnato con una penna idrofoba e risciacquare con soluzione salina tamponata con fosfato (PBS) da 0,1 M ghiacciata per 1 minuto a temperatura ambiente (RT). Posizionare il vetrino in una camera umida e bloccare per 90 minuti a 37 °C in soluzione bloccante (10% di siero di capra normale (NGS) e 1:30 di reagente bloccante topo su topo (MOM) in PBS).
- Rimuovere la soluzione bloccante e incubare i vetrini per 90 minuti a 37 °C con la soluzione contenente gli anticorpi primari (5% NGS, reagente bloccante MOM 1:30, gli anticorpi primari del topo per riconoscere le fibre di tipo I (IgG2b, a 1:10), di tipo IIa (IgG1, a 1:10) e di tipo IIx (IgM, a 1:5) e un antilaminina di ratto (catena α2, 1:1.000) in PBS.
- Lavare le diapositive con PBS 3 x 5 min a RT.
- Incubare i vetrini al buio per 1 ora a RT con la soluzione contenente gli anticorpi secondari (anti-IgG2b AF405 di capra (1:500), anti-IgG1 AF488 di capra (1:500), anti-IgM di capra AF568 (1:1.000) e anti-laminina di capra AF647 (1:500) in PBS).
ATTENZIONE: Per il resto del protocollo, assicurarsi di tenere le diapositive lontane dalla luce per preservare la fluorescenza. - Lavare nuovamente 3 x 5 minuti in PBS, risciacquare in H2O a doppia distillazione, rimuovere l'acqua in eccesso e montare con un reagente antifalda.
NOTA: Conservare le diapositive a 4 °C, al riparo dalla luce per preservare la fluorescenza fino all'acquisizione dell'immagine. Poiché le diapositive non sono fisse, si consiglia di utilizzare soluzioni appena fatte per ogni esperimento, autoclave il PBS e acquisire le immagini il prima possibile per evitare la contaminazione delle sezioni.
- Colorazione di LD con Bodipy
NOTA: Simile al passaggio 4.1., un volume totale di soluzione di 250 μL è sufficiente per coprire l'intera sezione muscolare circondata da un cerchio disegnato con una penna idrofoba delle dimensioni approssimative di una moneta da 1 centesimo.- Circondare le sezioni con un contorno disegnato da una penna idrofoba e risciacquare con PBS 0,1 M ghiacciato per 10 minuti a RT. Utilizzare PBS ghiacciato per tutti i risciacqui e lavaggi.
- Fissare con paraformaldeide fredda al 4% (PFA) senza metanolo per 10 minuti a RT. Dopo un primo risciacquo rapido, lavare i vetrini con PBS 3 x 5 min a RT.
ATTENZIONE: eseguire questo passaggio sotto un cappuccio chimico. - Posizionare la slitta in una camera umida e bloccare per 1 ora a RT con il 5% di NGS in PBS.
- Incubare i vetrini per 90 min a 37 °C con la soluzione dell'anticorpo primario (2% NGS e antilaminina di ratto (catena α2, 1:1.000) in PBS). Lavare le diapositive con PBS 3 x 5 min a RT.
ATTENZIONE: Per il resto del protocollo, assicurarsi di tenere le diapositive lontane dalla luce per preservare la fluorescenza. - Incubare per 1 ora a RT con la soluzione anticorpale secondaria contenente anticorpo anti-ratto-AF647 di capra (1:500) in PBS. Lavare le diapositive con PBS 3 x 5 min a RT.
- Incubare per 20 minuti a RT con una soluzione di 4',6-diamidino-2-fenilindolo (DAPI, 0,5 μg/mL) e BODIPY (1 μg/mL) in PBS.
NOTA: Per preparare la soluzione madre BODIPY, scioglierla in DMSO ad una concentrazione di 1 mg/mL. Diverse formulazioni di Bodipy sono disponibili in commercio per la colorazione LD. A seconda della scelta effettuata, il metodo di colorazione è lo stesso (stessi passaggi, concentrazione e tempo di incubazione); tuttavia, il metodo di acquisizione sarà leggermente diverso. - Dopo un primo risciacquo rapido, lavare i vetrini con PBS 3 x 5 min a RT, risciacquare in H2O a doppio distillato, rimuovere l'acqua in eccesso e montare con un reagente antideflagrante.
NOTA: Conservare le diapositive a 4 °C, al riparo dalla luce, per preservare la fluorescenza fino all'acquisizione dell'immagine.
5. Acquisizione di immagini
NOTA: Una volta completati i protocolli di colorazione, è importante procedere immediatamente all'acquisizione delle immagini (entro le successive 24 ore), non solo per evitare la contaminazione ma anche per preservare la morfologia, le dimensioni e il numero di LD.
- Acquisizione di immagini per valutare i tipi di fibre di ciascun muscolo campionato
NOTA: Questo passaggio potrebbe essere ottenuto con un microscopio a scansione a fluorescenza a vetrino intero o con un microscopio a epifluorescenza convenzionale. Con quest'ultimo, la cucitura manuale o automatizzata delle immagini deve essere fatta per ricostruire la sezione.- Per il riconoscimento del tipo di fibra, utilizzare un microscopio a epifluorescenza con un obiettivo 10x/0,3. Selezionare i filtri di eccitazione per DAPI (405 nm), FITC, TRITC e Cy5 per rilevare rispettivamente le fibre di tipo I, IIa, IIx e laminina.
NOTA: le fibre di tipo IIb non saranno immunoamarcate. Saranno riconosciute come fibre macchiate dalla laminina sui limiti del sarcolemma con un sarcoplasma nero. - Regolare il tempo di esposizione appropriato per ciascun canale.
- Quando si utilizza un microscopio a epifluorescenza convenzionale, acquisire sempre le immagini dell'intero muscolo seguendo lo stesso ordine per facilitare la ricostruzione muscolare. Assicurarsi che le fibre sul bordo destro di un'immagine vengano visualizzate anche sul bordo sinistro dell'immagine seguente. Lo stesso vale per le parti superiore e inferiore delle immagini (Figura 3A).
NOTA: Come riferimento, per una sezione dell'EDL o del soleo sezionato da un topo di 3 mesi, una media di sei e otto immagini, rispettivamente, coprirà l'intera sezione trasversale muscolare. - Dopo che il muscolo è stato scansionato, caricare le immagini digitali acquisite in qualsiasi software di elaborazione delle immagini per la ricostruzione (cucitura), in base alla morfologia della fibra (laminina) e all'istologia della sezione muscolare, e salvarlo come file TIFF, PNG o JPG con tutti i canali (colori) uniti (Figura 3A).
- Per il riconoscimento del tipo di fibra, utilizzare un microscopio a epifluorescenza con un obiettivo 10x/0,3. Selezionare i filtri di eccitazione per DAPI (405 nm), FITC, TRITC e Cy5 per rilevare rispettivamente le fibre di tipo I, IIa, IIx e laminina.
- Acquisizione di immagini con laminina e co-colorazione Bodipy
NOTA: Per riconoscere il tipo di fibra e avere una stima del numero di fibre per ogni tipo catturato, è essenziale avere la sezione del tipo di fibra già scansionata e i muscoli ricostruiti prima di iniziare l'acquisizione dell'immagine di Bodipy-laminina (Figura 3B).- Per l'osservazione e l'acquisizione di immagini Bodipy, utilizzare un microscopio confocale con un obiettivo ad immersione in olio 40x con un'apertura numerica di 1,4.
- Utilizzare le seguenti impostazioni: foro stenopeico a 1 UA, risoluzione di 2.048 pixel x 2.048 pixel, dimensione dei pixel a 0,08 μm, modalità unidirezionale, velocità di scansione a 4 (~ 4 μs / pixel), media della linea impostata su 4x e zoom digitale impostato su 1.
- Per evitare il cross-talk tra Bodipy-558/568 e laminin-AF647, utilizzare la modalità di scansione sequenziale sul software confocale.
NOTA: Quando il colorante scelto è Bodipy-493/503, è possibile la scansione laser confocale simultanea senza diafonia tra il canale Bodipy e il canale laminina-AF647. Ciò accelererà l'acquisizione delle immagini. - Eccita Bodipy-493/503 utilizzando la linea laser a 488 nm o la linea laser ad argon ed eccita Bodipy-555/568 utilizzando la linea laser a diodi da 561 nm. Infine, rileva la laminina-AF647 con una linea laser a diodi da 640 nm.
ATTENZIONE: Tieni presente che le molecole di Bodipy sono molto sensibili al fotosbiancamento, quindi evita inutili scansioni laser. Per riconoscere le fibre, utilizzare solo il laser per la laminina. - A seconda del colorante scelto, impostare gli intervalli di emissione a 570-650 nm per Bodipy-493/50324 e a 565-620 nm per Bodipy-558/568. Impostare l'intervallo di emissione per la laminina a 656-700 nm.
- Impostare il guadagno e il guadagno digitale in modo appropriato in modo che non vengano rilevati pixel saturi sull'indicatore dell'intervallo. Correggere il segnale di sfondo regolando l'offset.
NOTA: la selezione del filtro e altri parametri di scansione sopra menzionati devono essere ottimizzati per ciascun microscopio confocale. È importante che tutte le impostazioni sopra menzionate siano mantenute costanti per tutte le immagini acquisite dai campioni da confrontare. - Per identificare il tipo di fibra tra quelle visualizzate al microscopio confocale, utilizzare un laptop personale su cui viene controllata l'immagine della sezione ricostruita dopo l'immunodetezione di tipo fibra (Figura 3B).
- Una volta identificato correttamente un gruppo di fibre, acquisire l'immagine con i canali Bodipy e laminina.
NOTA: Si consiglia di notare il nome dell'immagine Bodipy-laminin sulla regione del muscolo in cui si trovano queste fibre sull'immagine acquisita per il riconoscimento MyHC per facilitare la successiva analisi specifica della fibra di LDs.
6. Analisi delle immagini
- Analisi dei tipi di fibre su ciascun campione muscolare
- In Fiji (o ImageJ)32, apri il file TIFF, PNG o JPG con il muscolo ricostruito ottenuto dalla fusione di tutti i canali utilizzati per rilevare le isoforme della fibra.
- Per avviare lo strumento Conteggio celle , fare clic su Plugin | Analizza | Contatore di celle | Contatore di celle.
- Nella finestra Contatore di celle , fare clic su Azioni | Inizializza.
- In Contatori nella stessa finestra selezionare Tipo 1.
- Nella finestra principale delle Figi, seleziona lo strumento Bacchetta .
- Per quantificare il numero di fibre di ciascun tipo, fare clic su ciascuna fibra dello stesso tipo, in modo che il programma registri il numero di fibre cliccate.
- Una volta terminato, selezionare il prossimo tipo di fibra e ripetere gli stessi passaggi.
- Quando tutte le fibre dell'immagine sono state assegnate a un determinato tipo di fibra, fare clic su Risultati nella finestra Contatore celle per visualizzare i risultati in una tabella.
- Salva questa tabella come tabella di foglio di calcolo facendo clic su File | Salva con nome nella finestra Risultati .
- Salva e ricarica le selezioni sulla stessa immagine in qualsiasi momento facendo clic su Salva marcatori o Carica marcatori, rispettivamente, nella finestra Conteggio celle .
- Analisi delle goccioline lipidiche in modo dipendente dal tipo di fibra
- Analizza le immagini di Bodipy e laminina ottenute sulla confocale usando Fiji per la quantificazione degli LD.
NOTA: gli autori hanno progettato una macro personalizzata per automatizzare l'analisi. Questa macro, insieme a una spiegazione dettagliata su come utilizzarla, è disponibile rispettivamente come file supplementare 1 e file supplementare 2. - Apri ogni immagine con l'aiuto dell'importatore di bioformati dalle Figi. Sotto l'opzione Visualizza stack con , selezionare Hyperstack | Modalità colore, predefinito. Assicurarsi che la finestra Ridimensiona automatica sia selezionata.
NOTA: i seguenti passaggi descriveranno il protocollo per l'analisi di una fibra sull'immagine, ma deve essere ripetuto tutte le volte che il numero di fibre intere appare sull'immagine. - Utilizzare lo strumento di selezione a mano libera per selezionare manualmente il sarcolemma della fibra in base al canale della laminina (Figura 4A) e premere T sulla tastiera per registrare la selezione o la regione di interesse (ROI) nella finestra ROI .
- Vai alla finestra principale delle Figi e fai clic su Analizza | Impostare Misure e, nella finestra pop-up, selezionare Area e diametro di Feret. Lasciare deselezionate le caselle rimanenti e altri parametri come appaiono per impostazione predefinita.
- Fare clic su Misura nella finestra ROI per ottenere l'area e il diametro minimo di Feret (MF) della fibra selezionata e annotarli per un uso successivo.
NOTA: Quando si analizzano gli LCD, è importante tenere presente che le loro dimensioni e densità variano tra il centro e la periferia (regione subsarcolemmale-SS) della fibra. Pertanto, l'analisi deve essere eseguita separatamente. - Calcola il valore di 1/6 del MF per delimitare la parte centrale della fibra.
Nota : nella macro, il valore MF predefinito è impostato su 6, il che significa che la riduzione applicata verrà impostata come 1/6 della MF. Questo valore è stato scelto sulla base dei dati empirici ottenuti dal soleo di animali alimentati con una dieta ricca di grassi. Tuttavia, ogni ricercatore deve modificare questo numero in base ai dati empirici e al muscolo analizzato, al tipo di fibra e alle condizioni dell'animale. - Nella finestra ROI , fai clic su Aggiungi per avere un duplicato del primo ROI e seleziona il secondo ROI che appare nella finestra.
- Nella finestra principale delle Figi, fai clic su Modifica | | di selezione Ingrandire e introdurre il valore calcolato in precedenza (dal passaggio 6.2.6.) con un segno meno prima del numero e fare clic su OK. Nella finestra ROI, fai clic su Aggiungi[t] (deve apparire un terzo ROI) ed Elimina, per eliminare il secondo ROI.
NOTA: il ricercatore può controllare i risultati facendo clic sulla casella Mostra tutto nella finestra ROI . A questo punto devono apparire due ROI, uno che circonda l'intero perimetro della fibra (Figura 4B) e un altro che è posto al centro (Figura 4C). - Seleziona entrambi i ROI nella finestra ROI e fai clic su Altro | | XOR Aggiungi[t]. Attendere che appaia un terzo ROI, che corrisponde alla periferia della fibra (Figura 4D).
- Salva il ROI facendo clic su Altre | Salva per salvare i ROI nel caso in cui sia necessaria una successiva rianalisi delle stesse fibre.
- Seleziona il canale Bodipy e apri lo strumento Soglia facendo clic su Immagine | Regolare | Soglia sulla finestra principale delle Figi.
- Nella finestra pop-up Soglia , imposta i valori su 70/255, seleziona Yen | Metodo B&W e fare clic su Sfondo scuro | Applica.
NOTA: I valori applicati sulla Soglia possono variare a seconda delle condizioni dell'esperimento e la soglia deve essere opportunamente impostata per ottimizzare l'analisi. Deve apparire una finestra in bianco e nero con il segnale Bodipy al di sopra del limite di soglia mostrato in bianco e lo sfondo in nero (confrontare l'immagine originale di Bodipy nella Figura 4E con quella nella Figura 4F). - Vai alla finestra principale delle Figi e fai clic su Analizza | Impostare Misure e nella finestra pop-up selezionare Area, Frazione di area e Limita alla soglia. Lasciare deselezionate le caselle rimanenti e altri parametri come appaiono per impostazione predefinita.
NOTA: Se il ricercatore vuole analizzare la "circolarità" dei LD, che è un indice della morfologia sferica che va da 1 per una sfera perfetta a 0 per una linea, fare clic sulla casella Descrittori forma della finestra pop-up Imposta misure . - Vai alla finestra ROI e seleziona il primo ROI. Nella finestra principale delle Figi, fai clic su Analizza | Strumento Analizza particelle.
NOTA: questo strumento quantifica il numero, le dimensioni, l'area coperta e la percentuale dell'area totale coperta dalle particelle in ogni selezione e salva i risultati come file di foglio di calcolo. - Impostare i valori da 2 a Infinito (2-Infinito) nella finestra Analizza particelle, selezionare la casella Pixel , mantenere i valori di circolarità predefiniti, selezionare Riepilogo e fare clic su OK.
NOTA: per controllare i risultati sulla fibra, sotto l'opzione Mostra selezionare una delle opzioni disponibili. Per far riconoscere le informazioni di ogni LD sulla selezione in una tabella, selezionare l'opzione Visualizza risultati nella finestra Analizza particelle . I risultati dell'analisi dell'area totale della fibra sono mediati e riassunti in una tabella con più colonne (Conteggio, Area totale, Dimensione media, % Area; queste corrispondono al numero di particelle [LD], all'area occupata da queste particelle, alla loro dimensione media e alla percentuale dell'area totale della selezione occupata dalle particelle, rispettivamente). Per calcolare la densità, dividere il numero di particelle per l'area totale di ciascuna selezione. - Per ottenere i valori del centro e della periferia della fibra, ripetere i passaggi 6.2.14 e 6.2.15, selezionando ogni volta il secondo (centro) e il terzo ROI (periferia).
- Salva i risultati facendo clic su File | Salva con nome nella finestra Riepilogo .
NOTA: includere il tipo di fibra, la condizione e il nome dell'immagine sul nome assegnato dei risultati per facilitare la successiva unificazione e l'analisi statistica dei dati. Per analizzare il resto delle fibre nella stessa immagine, ripetere i passaggi da 6.2.3 a 6.2.17. Per l'analisi statistica, devono essere analizzate almeno 10-15 fibre di ciascun tipo per animale.
- Analizza le immagini di Bodipy e laminina ottenute sulla confocale usando Fiji per la quantificazione degli LD.
Risultati
Il protocollo qui descritto fornisce un metodo efficiente per quantificare facilmente gli LCD in un tipo di fibra e in modo specifico subcellulare. Mostra come, congelando insieme due muscoli di dimensioni simili, come l'EDL e il soleo, il tempo e le risorse spesi per i passaggi successivi si riducono della metà.
Viene fornito un protocollo completo per l'immunocolorazione, l'acquisizione di immagini e l'analisi delle diverse isoforme di MyHC espresse nei muscoli del topo adulto. Questo proto...
Discussione
Il protocollo qui dettagliato descrive un metodo efficiente per quantificare gli LD etichettati con Bodipy su una base specifica per tipo di fibra e subcellulare. Negli ultimi anni, i coloranti lipidici classici, come ORO o Sudan Black B, sono stati sostituiti con una nuova serie di coloranti fluorescenti permeabili alle cellule, lipofili e che si legano ai lipidi neutri (ad esempio, Bodipy). Disponibile come diversi coniugati, Bodipy si è dimostrato molto efficace nel taggare gli LD per studiare la loro morfologia, din...
Divulgazioni
Gli autori non hanno conflitti di interesse da dichiarare.
Riconoscimenti
Questo lavoro è stato sostenuto da sovvenzioni del Fonds National de la Recherche Scientifique (FNRS-Crédit de Recherche J.0022.20) e della Société Francophone du Diabète (SFD-Roche Diabetes Care).C.M.S. è il destinatario di una borsa di dottorato dalla FRIA (FNRS). M.A.D.-L.d.C. ha ricevuto una borsa di studio dal Wallonie-Bruxelles International Excellence Program.
Gli autori ringraziano Alice Monnier per il suo contributo allo sviluppo di questo protocollo e Caroline Bouzin per la sua esperienza e l'aiuto tecnico nel processo di acquisizione delle immagini. Ringraziamo anche la piattaforma di imaging 2IP-IREC per l'accesso al criostato e ai microscopi (2IP-IREC Imaging Platform, Institute of Experimental and Clinical Research, Université Catholique de Louvain, 1200 Bruxelles, Belgio). Infine, gli autori desiderano ringraziare Nicolas Dubuisson, Romain Versele e Michel Abou-Samra per la critica costruttiva del manoscritto. Alcune delle figure di questo articolo sono state create con BioRender.com.
Materiali
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| AxioCam 506 mono 6 Mpix camera | Zeiss | ||
| AxioCam MRm 1.4MPix CCD camera | Zeiss | ||
| Chemical hood | Potteau Labo | EN-14175 | |
| Confocal microscope | Zeiss | LSM800 | |
| Cork discs (ø 20 mm, 3 mm thick) | Electron Microscopy Sciences | 63305 | |
| Cryo-Gloves | Tempshield | 16072252 | |
| Cryostat | Thermo Scientific | Microm Cryo Star HM 560 | |
| Dissecting Stereo Microscope SMZ745 | Nikon | ||
| Dry Ice | |||
| Dumont Forceps | F.S.T | 11295-10 | |
| Epifluorescence microscope | Zeiss | AxioImage-Apotome Z1 | |
| Extra Fine Bonn Scissors | F.S.T | 14084-08 | |
| FisherBrand Disposable Base Molds (0.7 x 0.7 cm) | ThermoFisher | 22-363-552 | Used to cut a piece to hold the muscle on the cork for freezing |
| Glass petri dish (H 25 mm, ø 150 mm) | BRAND Petri dish, MERK | BR455751 | Used to place the muscles on ice during dissection |
| ImmEdge Hydrophobic barrier PAP Pen | Vector Labs | H-4000 | Used to create an hidrophobic barrier around the muscle sections |
| Incubator | MMM Medcenter | Incucell 707 | |
| Microscope Cover Glasses (24x50 mm) | Assistent | 40990151 | |
| Microscope Slide Boxes | Kartell | 278 | Used as humid chambers for immunohistochemistry |
| Neck holder | Linie zwo | SB-035X-02 | Used as strap to hold the stainless steel tumbler |
| No 15 Sterile Carbon Steel Scalpel Blade | Swann-Morton | 0205 | |
| Paint brushes | Van Bleiswijck | Amazon B07W7KJQ2X | Used to handle cryosections |
| Permanent Marker Pen Black | Klinipath/VWR | 98307-R | Used to label slides |
| Pierce Fixation Forceps | F.S.T | 18155-13 | |
| Polystyrene Box | H 12 cm x L 25 cm x W 18 cm, used as a liquid nitrogen container and to transport the samples to the cryostat | ||
| Scalpel Handle, 125 mm (5"), No. 3 | Aesculap | BB073R | |
| Stainless Steel Cup 10oz | Eboxer | B07GFCBPFH | Tumbler to fill with isopentene for muscle freezing |
| Superfrost Ultra Plus slides | ThermoFisher | J1800AMNZ | |
| Surgical tweezers 1/2 teeth | Medische Vakhandel | 1303152 | Also called "Rat teeth tweezers" |
| Vannas Spring Scissors - 3 mm Cutting Edge | F.S.T | 15000-00 | |
| Weighing boats | VWR international | 611-2249 | |
| Whole-Slide Scanner for Fluorescence | Zeiss | Axio Scan.Z1 | |
| Reagents | |||
| Alexa Fluor 405 Goat Anti-Mouse IgG2b | Sigma-Aldrich | SAB4600477 | Used at a final concentration of 1:500 |
| Alexa Fluor 488 Goat Anti-Mouse IgG1 | ThermoFisher | A-21121 | Used at a final concentration of 1:500 |
| Alexa Fluor 568 Goat Anti-Mouse IgM | Abcam | ab175702 | Used at a final concentration of 1:1,000 |
| Alexa Fluor 647 goat anti rat-IgG (H+L) secondary antibody | ThermoFisher | A-21247 | Used at a final concentration of 1:500 |
| BODIPY-493/503 (4,4-difluoro-1,3,5,7,8-pentametil-4-bora-3a,4a-diaza-s-indaceno) | ThermoFisher | D3922 | Used at a final concentration of 1 µg/mL |
| BODIPY-558/568 C12 (4,4-Difluoro-5-(2-Thienyl)-4-Bora-3a,4a-Diaza-s-Indacene-3-Dodecanoic Acid) | ThermoFisher | D3835 | Used at a final concentration of 1 µg/mL |
| DAPI (4',6-diamidino-2-phenylindole) | ThermoFisher | D1306 | Used at a final concentration of 0.5 µg/mL |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich | D-8418 | Used to solve Bodipy for the 1 mg/mL stock solution. CAUTION: Toxic and flammable. Vapors may cause irritation. Manipulate in a fume hood. Avoid direct contact with skin. Wear rubber gloves, protective eye goggles. |
| Formaldehyde solution 4%, buffered, pH 6.9 | Sigma-Aldrich | 1004969011 | CAUTION: May cause an allergic skin reaction. Suspected of causing genetic defects. May cause cancer. Manipulate in a fume hood. Avoid direct contact with skin. Wear rubber gloves, protective eye goggles. |
| Isopentane GPR RectaPur | VWR international | 24872.298 | CAUTION: Extremely flammable liquid and vapor. May be fatal if swallowed and enters airways. May cause drowsiness or dizziness. Repeated exposure may cause skin dryness or cracking. Wear protective gloves/protective clothing/eye protection/face protection. |
| Liquid Nitrogen | CAUTION: Extremely cold. Wear gloves. Handle slowly to minimize boiling and splashing and in well ventilated areas. Use containers designed for low-temperature liquids. | ||
| Mouse on mouse Blocking Reagent | Vector Labs | MKB-2213-1 | Used at concentration of 1:30 |
| Myosin heavy chain Type I (BA-D5-s Primary Antibody) Gene: MYH7, monoclonal bovine anti mouse IgG2b | DSHB University of Iowa | BA-D5-supernatant | Used at a final concentration of 1:10 |
| Myosin heavy chain Type IIA (SC-71-s Primary Antibody) Gene: MYH2, Monoclonal bovine anti mouse IgG1 | DSHB University of Iowa | SC-71-supernatant | Used at a final concentration of 1:10 |
| Myosin heavy chain Type IIX (6H1-s Primary Antibody), Gene: MYH1, Monoclonal rabbit anti mouse IgM | Developmental Studies Hybridoma Bank, University of Iowa | 6H1-supernatant | Used at a final concentration of 1:5 |
| Normal Goat Serum (NGS) | Vector Labs | S-1000 | |
| PBS 0.1 M | Commonly used on histology laboratories | ||
| ProLong Gold Antifade Mountant | Invitrogen | P36930 | |
| Rat anti-Laminin-2 (α-2 Chain) primary antibody (monoclonal) | Sigma-Aldrich | L0663 | Used at a final concentration of 1:1,000 |
| Tissue-Tek O.C.T compound | Sakura | 4583 | |
| Software | |||
| Adobe Illustrator CC | Adobe Inc. | Used to design the figures | |
| Adobe Photoshop | Adobe Inc. | Confocal software | |
| BioRender | https://biorender.com/ | Used to design the figures | |
| Fiji/ImageJ | https://imagej.net/software/fiji/ | Used to analyse the acquired images | |
| Microsoft PowerPoint | Microsoft | Used to reconstruct the histology of the whole muscle after scanning the fiber types | |
| Zen Blue 2.6 | Zeiss | Used to reconstruct the histology of the whole muscle after scanning the fiber types |
Riferimenti
- Correa-de-Araujo, R., et al. Myosteatosis in the context of skeletal muscle function deficit: an interdisciplinary workshop at the National Institute on Aging. Frontiers in Physiology. 11, 963 (2020).
- Miljkovic, I., et al. Greater skeletal muscle fat infiltration is associated with higher all-cause and cardiovascular mortality in older men. Journals of Gerontology Series A: Biomedical Sciences and Medical Sciences. 70 (9), 1133-1140 (2015).
- Nachit, M., et al. Myosteatosis rather than sarcopenia associates with non-alcoholic steatohepatitis in non-alcoholic fatty liver disease preclinical models. Journal of Cachexia, Sarcopenia, and Muscle. 12 (1), 144-158 (2021).
- Aleixo, G. F. P., et al. Myosteatosis and prognosis in cancer: Systematic review and meta-analysis. Critical Reviews in Oncology/Hematolgoy. 145, 102839 (2020).
- Gemmink, A., Schrauwen, P., Hesselink, M. K. C. Exercising your fat (metabolism) into shape: a muscle-centred view. Diabetologia. 63 (8), 1453-1463 (2020).
- van Loon, L. J. Use of intramuscular triacylglycerol as a substrate source during exercise in humans. Journal of Applied Physiology. 97 (4), 1170-1187 (2004).
- Coen, P. M., Goodpaster, B. H. Role of intramyocelluar lipids in human health. Trends in Endocrinology and Metabolism. 23 (8), 391-398 (2012).
- Seibert, J. T., Najt, C. P., Heden, T. D., Mashek, D. G., Chow, L. S. Muscle lipid droplets: cellular signaling to exercise physiology and beyond. Trends in Endocrinology and Metabolism. 31 (12), 928-938 (2020).
- Bergman, B. C., Goodpaster, B. H. Exercise and muscle lipid content, composition, and localization: influence on muscle insulin sensitivity. Diabetes. 69 (5), 848-858 (2020).
- Nielsen, J., Christensen, A. E., Nellemann, B., Christensen, B. Lipid droplet size and location in human skeletal muscle fibers are associated with insulin sensitivity. American Journal of Physiology-Endocrinology and Metabolism. 313 (6), 721-730 (2017).
- Covington, J. D., et al. Intramyocellular lipid droplet size rather than total lipid content is related to insulin sensitivity after 8 weeks of overfeeding. Obesity (Silver Spring). 25 (12), 2079-2087 (2017).
- Bosma, M. Lipid droplet dynamics in skeletal muscle). Experimental Cell Research. 340 (2), 180-186 (2016).
- Nielsen, J., et al. Increased subsarcolemmal lipids in type 2 diabetes: effect of training on localization of lipids, mitochondria, and glycogen in sedentary human skeletal muscle. American Journal of Physiology-Endocrinology and Metabolism. 298 (3), 706-713 (2010).
- Ferreira, R., et al. Subsarcolemmal and intermyofibrillar mitochondria proteome differences disclose functional specializations in skeletal muscle. Proteomics. 10 (17), 3142-3154 (2010).
- Barrett, J. S., Whytock, K. L., Strauss, J. A., Wagenmakers, A. J. M., Shepherd, S. O. High intramuscular triglyceride turnover rates and the link to insulin sensitivity: influence of obesity, type 2 diabetes and physical activity. Applied Physiology, Nutrition and Metabolism. , 1-14 (2022).
- Daemen, S., et al. Distinct lipid droplet characteristics and distribution unmask the apparent contradiction of the athlete's paradox. Molecular Metabolism. 17, 71-81 (2018).
- Bredella, M. A., Ghomi, R. H., Thomas, B. J., Miller, K. K., Torriani, M. Comparison of 3.0 T proton magnetic resonance spectroscopy short and long echo-time measures of intramyocellular lipids in obese and normal-weight women. Journal of Magnetic Resonance Imaging. 32 (2), 388-393 (2010).
- Schrauwen-Hinderling, V. B., Hesselink, M. K., Schrauwen, P., Kooi, M. E. Intramyocellular lipid content in human skeletal muscle. Obesity (Silver Spring). 14 (3), 357-367 (2006).
- De Bock, K., et al. Evaluation of intramyocellular lipid breakdown during exercise by biochemical assay, NMR spectroscopy, and Oil Red O staining. American Journal of Physiology-Endocrinology and Metabolism. 293 (1), 428-434 (2007).
- Koopman, R., Schaart, G., Hesselink, M. K. Optimisation of oil red O staining permits combination with immunofluorescence and automated quantification of lipids. Histochemistry and Cell Biology. 116 (1), 63-68 (2001).
- Gueugneau, M., et al. Skeletal muscle lipid content and oxidative activity in relation to muscle fiber type in aging and metabolic syndrome. Journal of Gerontology Series A: Biomedical Sciences and Medical Sciences. 70 (5), 566-576 (2015).
- Gemmink, A., et al. Super-resolution microscopy localizes perilipin 5 at lipid droplet-mitochondria interaction sites and at lipid droplets juxtaposing to perilipin 2. Biochimica et Biophysica Acta-Molecular and Cell Biology of Lipids. 1863 (11), 1423-1432 (2018).
- Spangenburg, E. E., Pratt, S. J. P., Wohlers, L. M., Lovering, R. M. Use of BODIPY (493/503) to visualize intramuscular lipid droplets in skeletal muscle. Journal of Biomedicine and Biotechnology. 598358, (2011).
- Prats, C., et al. An optimized histochemical method to assess skeletal muscle glycogen and lipid stores reveals two metabolically distinct populations of type I muscle fibers. PLoS One. 8 (10), 77774 (2013).
- Strauss, J. A., Shepherd, D. A., Macey, M., Jevons, E. F. P., Shepherd, S. O. Divergence exists in the subcellular distribution of intramuscular triglyceride in human skeletal muscle dependent on the choice of lipid dye. Histochemistry and Cell Biology. 154 (4), 369-382 (2020).
- Shepherd, S. O., et al. Sprint interval and traditional endurance training increase net intramuscular triglyceride breakdown and expression of perilipin 2 and 5. Journal of Physiology. 591 (3), 657-675 (2013).
- Whytock, K. L., et al. A 7-day high-fat, high-calorie diet induces fibre-specific increases in intramuscular triglyceride and perilipin protein expression in human skeletal muscle. Journal of Physiology. 598 (6), 1151-1167 (2020).
- Wang, C., Yue, F., Kuang, S. Muscle histology characterization using h&e staining and muscle fiber type classification using immunofluorescence staining. Bio-Protocol. 7 (10), (2017).
- Meng, H., et al. Tissue triage and freezing for models of skeletal muscle disease. Journal of Visualized Experiments: JoVE. (89), e51586 (2014).
- Kumar, A., Accorsi, A., Rhee, Y., Girgenrath, M. Do's and don'ts in the preparation of muscle cryosections for histological analysis. Journal of Visualized Experiments: JoVE. (99), e52793 (2015).
- Leiva-Cepas, F., et al. Laboratory methodology for the histological study of skeletal muscle. Archivos de Medicina del Deporte. 35 (186), 254-262 (2018).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Schiaffino, S., et al. Three myosin heavy chain isoforms in type 2 skeletal muscle fibres. Journal of Muscle Research & Cell Motility. 10 (3), 197-205 (1989).
- Komiya, Y., et al. Mouse soleus (slow) muscle shows greater intramyocellular lipid droplet accumulation than EDL (fast) muscle: fiber type-specific analysis. Journal of Muscle Research & Cell Motility. 38 (2), 163-173 (2017).
- Andrich, D. E., et al. Altered lipid metabolism impairs skeletal muscle force in young rats submitted to a short-term high-fat diet. Frontiers in Physiology. 9, 1327 (2018).
- Schiaffino, S. Fibre types in skeletal muscle: a personal account. Acta Physiologica. 199 (4), 451-463 (2010).
- Bloemberg, D., Quadrilatero, J. Rapid determination of myosin heavy chain expression in rat, mouse, and human skeletal muscle using multicolor immunofluorescence analysis. PLoS One. 7 (4), 35273 (2012).
- Gemmink, A., et al. Decoration of intramyocellular lipid droplets with PLIN5 modulates fasting-induced insulin resistance and lipotoxicity in humans. Diabetologia. 59 (5), 1040-1048 (2016).
- Askinas, C., et al. . Biophotonics Congress: Biomedical Optics Congress 2018 (Microscopy/Translational/Brain/OTS). , (2018).
- Morén, B., et al. EHD2 regulates adipocyte function and is enriched at cell surface-associated lipid droplets in primary human adipocytes. Molecular Biology of the Cell. 30 (10), 1147-1159 (2019).
- Benador, I. Y., et al. Mitochondria bound to lipid droplets have unique bioenergetics, composition, and dynamics that support lipid droplet expansion. Cell Metabolism. 27 (4), 869-885 (2018).
- de la Rosa Rodriguez, M. A., et al. Hypoxia-inducible lipid droplet-associated induces DGAT1 and promotes lipid storage in hepatocytes. Molecular Metabolism. 47, 101168 (2021).
- Jevons, E. F. P., Gejl, K. D., Strauss, J. A., Ørtenblad, N., Shepherd, S. O. Skeletal muscle lipid droplets are resynthesized before being coated with perilipin proteins following prolonged exercise in elite male triathletes. American Journal of Physiology-Endocrinology and Metabolism. 318 (3), 357-370 (2020).
- Ohsaki, Y., Maeda, T., Fujimoto, T. Fixation and permeabilization protocol is critical for the immunolabeling of lipid droplet proteins. Histochemistry and Cell Biology. 124 (5), 445-452 (2005).
- Prats, C., et al. Decrease in intramuscular lipid droplets and translocation of HSL in response to muscle contraction and epinephrine. Journal of Lipid Research. 47 (11), 2392-2399 (2006).
- Listenberger, L. L., Brown, D. A. Fluorescent detection of lipid droplets and associated proteins. Current Protocols in Cell Biology. , (2007).
- Xue, Y., Lim, S., Bråkenhielm, E., Cao, Y. Adipose angiogenesis: quantitative methods to study microvessel growth, regression and remodeling in vivo. Nature Protocols. 5 (5), 912-920 (2010).
- Muliyil, S., et al. ADAM17-triggered TNF signalling protects the ageing Drosophila retina from lipid droplet-mediated degeneration. The EMBO Journal. 39 (17), 104415 (2020).
- Yan, Q., et al. Autophagy activation contributes to lipid accumulation in tubular epithelial cells during kidney fibrosis. Cell Death Discovery. 4, 2 (2018).
- Coassin, S., et al. Investigation and functional characterization of rare genetic variants in the adipose triglyceride lipase in a large healthy working population. PLoS Genetics. 6 (12), 1001239 (2010).
- Daemen, S., van Zandvoort, M., Parekh, S. H., Hesselink, M. K. C. Microscopy tools for the investigation of intracellular lipid storage and dynamics. Molecular Metabolism. 5 (3), 153-163 (2016).
- Chen, Q., et al. Rab8a deficiency in skeletal muscle causes hyperlipidemia and hepatosteatosis by impairing muscle lipid uptake and storage. Diabetes. 66 (9), 2387-2399 (2017).
- Gemmink, A., et al. Decoration of myocellular lipid droplets with perilipins as a marker for in vivo lipid droplet dynamics: A super-resolution microscopy study in trained athletes and insulin resistant individuals. Biochimica et Biophysica Acta (BBA)-Molecular and Cell Biology of Lipids. 1866 (2), 158852 (2021).
- Bergman, B. C., Hunerdosse, D. M., Kerege, A., Playdon, M. C., Perreault, L. Localisation and composition of skeletal muscle diacylglycerol predicts insulin resistance in humans. Diabetologia. 55 (4), 1140-1150 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon