É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Tipo de fibra e análise subcelular-específica do conteúdo de gotícula lipídica no músculo esquelético
Neste Artigo
Resumo
Evidências crescentes indicam que a infiltração excessiva de lipídios dentro do músculo esquelético resulta em lipotoxicidade e diabetes. Aqui, apresentamos um protocolo completo, incluindo processamento de tecidos, coloração com Bodipy, aquisição de imagens e análise, para quantificar o tamanho, densidade e distribuição subcelular de gotículas lipídicas de forma específica do tipo fibra.
Resumo
A infiltração de lipídios musculares esqueléticos, conhecida como micosteatose, aumenta com obesidade e envelhecimento. A miostetose também foi descoberta recentemente como um fator prognóstico negativo para vários outros transtornos, como doenças cardiovasculares e câncer. A infiltração excessiva de lipídios diminui a massa e a força muscular. Também resulta em lipotoxicidade e resistência à insulina, dependendo do teor total de lipídio intramialcelular, morfologia de gotícula lipídica (LD) e distribuição subcelular. O tipo de fibra (oxidativo versus glicolítico) também é importante, uma vez que as fibras oxidativas têm maior capacidade de utilizar lipídios. Devido às suas implicações cruciais na fisiopatologia, estudos aprofundados sobre a dinâmica ld e função de forma específica do tipo de fibra são justificados.
Aqui, é apresentado um protocolo completo para a quantificação do conteúdo lipídudo intramyocelular e análise da morfologia LD e distribuição subcelular de forma específica do tipo de fibra. Para isso, as crioseções musculares em série foram manchadas com o corante fluorescente Bodipy e anticorpos contra isoformas de cadeia pesada de miosina. Este protocolo permite o processamento simultâneo de diferentes músculos, economizando tempo e evitando possíveis artefatos e, graças a uma macro personalizada criada em Fiji, a automatização da análise LD também é possível.
Introdução
A infiltração de lipídios musculares esqueléticos, conhecida como micosteatose, aumenta com obesidade e envelhecimento. A miostetose está negativamente correlacionada com massa muscular e força e com sensibilidade à insulina1. Além disso, estudos recentes indicam que o grau de mostetose poderia ser usado como fator prognóstico para outras condições como doença cardiovascular2, doença hepática gordurosa não alcoólica3 ou câncer4. Lipídios podem se acumular no músculo esquelético entre as fibras musculares como lipídios extramyocelulares ou dentro das fibras, como lipídios intramyocelulares (IMCLs). Os IMCLs são predominantemente armazenados como triglicérides em gotículas lipídicas (LDs) que são usadas como combustível metabólico durante o exercício físico 5,6. No entanto, quando a oferta lipídica excede a demanda, ou quando as mitocôndrias se tornarem disfuncionais, os IMCLs serão implicados na resistência à insulina muscular, como visto em indivíduos metabolicamente insalubres, obesos e em pacientes com diabetes tipo 27. Curiosamente, os atletas de resistência têm níveis semelhantes, se não mais elevados, de IMCLs aos encontrados em pacientes obesos com diabetes mellitus tipo 2, mantendo alta sensibilidade à insulina. Esse fenômeno é descrito como o "paradoxo do atleta"8,9, e é explicado por uma avaliação mais matizada de LDs musculares, relacionadas ao seu tamanho, densidade, localização, dinâmica e composição de espécies lipídicas.
Primeiro, o tamanho do LD está inversamente correlacionado à sensibilidade à insulina e ao condicionamento físico10,11. De fato, LDs menores exibem uma área de superfície relativamente maior para a ação de lipase e, portanto, potencialmente têm maior capacidade de mobilizar lipídios12. Em segundo lugar, a densidade LD (número/superfície) desempenha um papel controverso na ação de insulina 8,10; ainda, parece ser aumentado em atletas. Em terceiro lugar, a localização subcelular de LDs é importante, uma vez que os LDs localizados logo abaixo da membrana superficial (subsarcolemmal ou periférico) exercem um efeito mais deletério sobre a sensibilidade à insulina do que os centrais 8,9,13. Estas últimas fornecem combustível para mitocôndrias centrais, que possuem maior atividade respiratória e são mais especializadas para atender à alta demanda energética necessária para a contração14. Em contrapartida, os LDs periféricos fornecem mitocôndrias subsarcolemmal, que estão envolvidas nos processos relacionados à membrana8. Finalmente, além dos triglicérides, lipídios complexos específicos dentro do músculo podem ser mais deletérios do que outros. Por exemplo, diaciglicerol, acicl-CoA de cadeia longa e ceramidas podem se acumular em músculos quando a taxa de rotatividade de triglicérides é baixa, prejudicando assim a sinalização de insulina 9,15. Voltando ao "paradoxo do atleta", os atletas de resistência têm um alto número de LDs centrais menores com taxas de rotatividade elevadas nas fibras tipo I (oxidativa), enquanto pacientes obesos e diabéticos têm LDs periféricos maiores com baixas taxas de rotatividade nas fibras tipo II (glicolítico) 8,15,16. Além de seu papel no armazenamento e liberação de energia, os LDs via ácidos graxos derivados (FA) e uma proteína de casaco (perilipin 5) também poderiam funcionar como atores críticos envolvidos na regulação transcricional da oxidação da FA e biogênese mitocondrial8. Devido às suas implicações cruciais na fisiologia e na fisiopatologia, estudos aprofundados sobre dinâmicas e funções de LDs são justificados.
Embora existam várias técnicas para estudar IMCLs, elas não são todas adequadas para quantificar com precisão o tamanho, a densidade e a distribuição de LD de forma específica. Por exemplo, a avaliação de IMCLs por espectroscopia de ressonância magnética, embora não invasiva, oferece um nível de resolução que não é suficiente para estudar o tamanho e a localização precisa de LDs dentro da fibra, e não é específica do tipo fibra 17,18. Da mesma forma, técnicas bioquímicas realizadas em homogeneizadores muscularesinteiros 19 não podem avaliar a localização e o tamanho dos lipídios. Consequentemente, o método mais adequado para analisar a morfologia e localização de LD é a microscopia eletrônica de transmissão quantitativa13, mas essa técnica é cara e demorada. Portanto, a fluorescência confocal em preparações com corantes como Oil Red O (ORO)20,21, monodansylpentane (MDH)22, ou Bodipy 23,24,25, emergiu como a melhor ferramenta para esses estudos.
Aqui, é descrito um protocolo completo, incluindo amostragem e processamento de tecidos, coloração bodipy e aquisição e análise de imagem confocal para quantificar o tamanho, o número e a localização de LD em crioseções musculares de camundongos. Uma vez que as IMCLs não são distribuídas uniformemente entre fibras oxidativas e glicolíticas, e cada tipo de fibra regula a dinâmica LD de forma diferente, o estudo das IMCLs deve ser específico do tipo fibra 16,25,26,27. Portanto, este protocolo utiliza imunofluorescência em seções seriais para identificar isoform(s) de cadeia pesada de miosina (MyHC) expressa por cada fibra. Outra vantagem deste protocolo é o processamento simultâneo de um músculo glicolítico (extensor digitorum longus, EDL) e um músculo oxidativo (soleus) colocado lado a lado antes de congelar (Figura 1). Esse processamento simultâneo não só economiza tempo, mas também evita a variabilidade devido ao processamento separado das amostras.
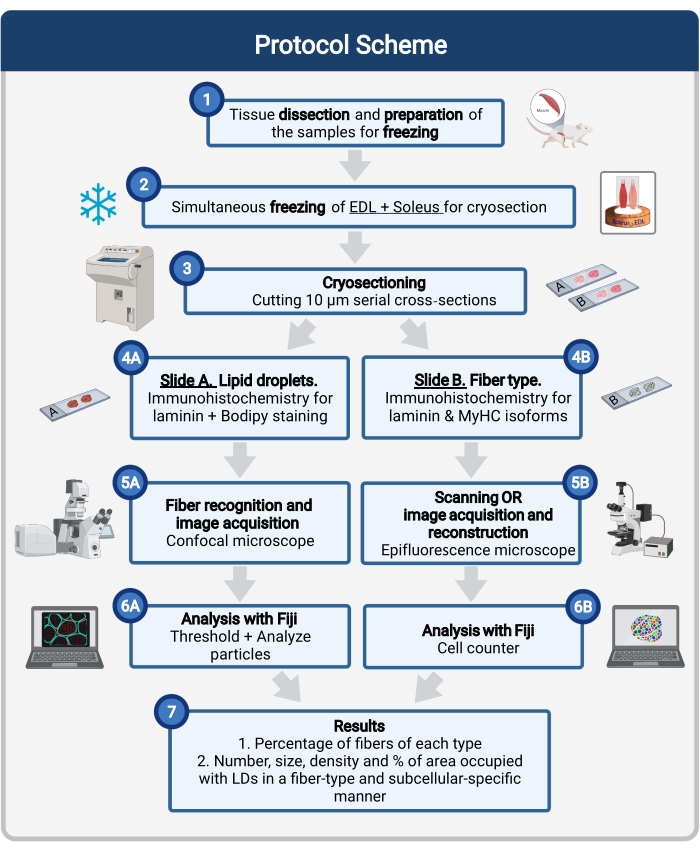
Figura 1: Visão geral do procedimento. Após dissecção muscular (1), músculos selecionados de tamanho semelhante são preparados e congelados juntos (2). Seções transversais em série de 10 μm são obtidas usando um criostat e montadas diretamente em slides de adesão (3). A partir de dois slides seriais, o primeiro (4A) é imunolabeled para laminina e manchado com Bodipy para reconhecer LDs e o segundo (4B) é imunossumado com anticorpos contra MyHCs para o reconhecimento de tipos de fibras musculares. As imagens são adquiridas usando um microscópio confocal para Bodipy (5A) e um microscópio de epifluorescência para tipos de fibras musculares (5B). As imagens são analisadas em Fiji aplicando um limiar e quantificando partículas (6A) para obter o número, tamanho médio, densidade e percentual da área total ocupada por LDs (7) ou células de contagem (6B) para obter a porcentagem de fibras de cada tipo na seção (7). Abreviaturas: LDs = gotículas lipídicas; EDL = extensor digitorum longus; MyHCs = isoforms de cadeia pesada de miosina. Clique aqui para ver uma versão maior desta figura.
Access restricted. Please log in or start a trial to view this content.
Protocolo
Todos os procedimentos realizados em camundongos foram aprovados pelo Comitê de Ética para Experimentação Animal do Setor Médico da Université Catholique de Louvain (2019/UCL/MD/013).
1. Dissecção e preparação das amostras para congelamento
- Rotule um pedaço de rolha de 3 mm de espessura para cada par de músculos.
- Através de uma pequena incisão feita com uma lâmina no centro da rolha, insira perpendicularmente uma peça retangular de plástico rígido (0,5 cm W, 1 cm H) que servirá como suporte (Figura 2B).
NOTA: O tamanho da peça de plástico retangular dependerá do tamanho do músculo. Aqui, as dimensões descritas são adaptadas ao tamanho do soleus (~9 mgs, 1 cm L, 2-3 mm W) e EDL (~5 mgs, 1 cm L, 2-3 mm W) de um rato masculino C57BL/6J de 3 meses de idade. - No momento da dissecção, remova o sola e o EDL do membro traseiro do rato. Para evitar que as amostras sequem durante a dissecção, coloque-as em uma compressa levemente umedecida com solução salina em uma placa de Petri colocada no gelo.
NOTA: Consulte Wang et al.28 para obter explicações sobre como dissecar esses dois músculos dos membros. - Coloque uma pequena gota do composto de temperatura de corte ideal (OCT) na junção de cortiça/plástico, evitando bolhas de ar.
- Elimine o excesso de umidade das amostras secando-as suavemente com uma toalha de papel (Figura 2A) e coloque ambos os músculos no perpendicular plástico à rolha (Figura 2C).
- Verifique a orientação dos myofibers musculares sob um microscópio estéreo (Figura 2D).
NOTA: É importante não cobrir o músculo com OCT, uma vez que seu efeito isolante evitaria o congelamento rápido e produziria artefatos congelantes.
2. Congelar amostras de músculo esquelético para criosectioning
ATENÇÃO: O congelamento do músculo deve ser feito sob uma capa química, usando equipamentos de proteção individual adequados (veja a Tabela de Materiais).
- Use um tumbler de aço inoxidável (~8 cm H, 6 cm Ø) com duas correias laterais presas a ele com pelo menos 25 cm de comprimento (Figura 2F), e encha o tumbler até 2/3 de sua capacidade com isopia.
- Agarrando o tumbler pelas correias, mergulhe-o suavemente em uma caixa de poliestireno cheia de nitrogênio líquido para que o nível de nitrogênio fora do recipiente esteja acima do nível de isopentano dentro (Figura 2F,G).
ATENÇÃO: Quando o tumbler entra em contato com o nitrogênio, o choque térmico pode causar rodírio. Certifique-se de que o tumbler está suficientemente imerso, mas evite a entrada de nitrogênio, pois isso causaria precipitação isofentane. Se isso acontecer, deixe a isopentane esfriar, encha o tumbler com novo isopentane e comece de novo. - Quando o interior do tumbler estiver completamente coberto com uma camada branca sólida de isopentane, tire-o da caixa de nitrogênio líquido (Figura 2H).
NOTA: A temperatura de fusão da isopentane é de -159 °C, as bordas do tumbler ficarão brancas quando estiver fria o suficiente. - Mexa suavemente as peças de isopentane sólidas no isopentane líquido restante com fórceps até que todo o volume se torne líquido novamente.
- Remerque o tumbler no nitrogênio líquido até que a isopentane forme seixos brancos por todo o fundo e bordas do tumbler (Figura 2G,H).
NOTA: Esta segunda etapa de resfriamento garante a temperatura de congelamento adequada da isopentane. - Remova o tumbler do nitrogênio líquido e mergulhe rapidamente os músculos na parte inferior do tumbler, segurando a rolha com pinça de dente de rato. Gire a rolha por 15 s na isopentane e armazene-a a -80 °C até o processamento (Figura 2I).
NOTA: Para armazenamento a curto prazo, as amostras podem ser mantidas em um congelador de -20 °C. O protocolo completo para o congelamento rápido do músculo esquelético já foi publicado em outros lugares. Para obter referências detalhadas e solução de problemas, consulte: Meng et al.29, Kumar et al.30 e Leiva-Cepas et al.31.
3. Criosectioning
- Leve as amostras para a câmara de um criostat previamente resfriado a -20 °C e a temperatura da lâmina está definida para -25 °C.
NOTA: Transportar amostras do congelador -20 °C/-80 °C até o criostat em uma caixa de poliestireno cheia de gelo seco e permitir que as amostras se equilibrem por pelo menos 20-30 minutos até a temperatura da câmara antes de cortar. - Retire o plástico da rolha com pinças finas e coloque o disco do espécime criostat na placa de congelamento rápido para esfriar. Uma vez que a placa tenha atingido -50 °C, coloque algum OCT no disco e coloque rapidamente a rolha em cima do disco, pressionando firmemente. Aguarde até que o OCT se solidifique e a rolha esteja bem fixada no disco.
- Coloque o disco sobre a cabeça do objeto na orientação de corte desejada (Figura 2J,K) e corte o bloco muscular além de pelo menos 1/3 do comprimento dos músculos e até que ambas as seções transversais dos músculos sejam visíveis.
- Coloque a espessura de corte em 10 μm e coloque uma seção em um slide de adesão para verificar a orientação correta das fibras em um microscópio de campo brilhante.
NOTA: Verificar a orientação transversal das fibras é fundamental. Se as fibras não estiverem adequadamente orientadas, ajuste o ângulo da cabeça do objeto, corte outra seção e verifique novamente. - Coloque duas seções transversais em série em dois slides de adesão pré-rotulados: um slide para determinar tipos de fibra, o outro para quantificar o conteúdo lipídico (Figura 2L).
NOTA: Podem ser obtidas seções transversais adicionais e mantidas a -80 °C para outros estudos histológicos. No entanto, para evitar alterar o conteúdo lipídudo e a morfologia intracelular24, é essencial processar os dois primeiros slides imediatamente após o corte para evitar a secagem do ar. Congelar e descongelar os slides para quantificação LD teria o mesmo efeito e, portanto, é altamente desaconselhável.
4. Digitação de fibras e coloração bodipy
- Detecção imunohistoquímica do tipo de fibra muscular
NOTA: Para o protocolo a seguir, um volume total de solução de 250 μL é suficiente para cobrir toda a seção muscular cercada por um círculo desenhado com uma caneta hidrofóbica do tamanho aproximado de uma moeda de 1 centavo.- Cerque as seções com um contorno desenhado com uma caneta hidrofóbica e enxágue com soro fisiológico com fosfato de 0,1 M (PBS) por 1 min a temperatura ambiente (RT). Coloque o slide em uma câmara úmida e bloqueie por 90 min a 37 °C na solução de bloqueio (soro de cabra 10% normal (NGS) e 1:30 mouse on mouse (MOM) bloqueando reagente em PBS).
- Remova a solução de bloqueio e incuba os slides por 90 min a 37 °C com a solução contendo os anticorpos primários (5% de NGS, 1:30 MOM bloqueador reagente, os anticorpos primários do rato para reconhecer o tipo I (IgG2b, à 1:10), tipo IIa (IgG1, às 1:10), e tipo IIx (IgM, em 1:5) fibras e um anti-laminina de rato (cadeia α2, 1:1.000) na PBS.
- Lave os slides com PBS 3 x 5 min no RT.
- Incubar os slides no escuro por 1 h no RT com a solução contendo os anticorpos secundários (anti-IgG2b AF405 (1:500), anti-IgG1 AF488 (1:500), anti-IgM AF568 (1:1.000) e anti-laminina de cabra AF647 (1:500) na PBS).
ATENÇÃO: Para o resto do protocolo, certifique-se de manter os slides longe da luz para preservar a fluorescência. - Lave novamente 3 x 5 min em PBS, enxágue em H2O duplamente destilado, remova o excesso de água e monte com um reagente antifade.
NOTA: Armazene os slides a 4 °C, protegidos da luz para preservar a fluorescência até a aquisição da imagem. Como os slides não são fixos, recomenda-se usar soluções recém-feitas para cada experimento, autoclave o PBS e adquira as imagens o mais rápido possível para evitar a contaminação das seções.
- Mancha de LDs com Bodipy
NOTA: Semelhante ao passo 4.1., um volume total de solução de 250 μL é suficiente para cobrir toda a seção muscular cercada por um círculo desenhado com uma caneta hidrofóbica do tamanho aproximado de uma moeda de 1 centavo.- Cerque as seções com um contorno desenhado por uma caneta hidrofóbica e enxágue com pbs gelado de 0,1 M por 10 min na RT. Use PBS gelado para todas as lavagens e lavagens.
- Corrija com paraformaldeído frio de 4% (PFA) sem metanol por 10 minutos no RT. Depois de uma primeira lavagem rápida, lave os slides com PBS 3 x 5 min no RT.
ATENÇÃO: Execute esta etapa sob um capô químico. - Coloque o slide em uma câmara úmida e bloqueie por 1h no RT com 5% de NGS em PBS.
- Incubar os slides por 90 min a 37 °C com a solução do anticorpo primário (2% NGS e anti-laminina de rato (cadeia α2, 1:1.000) em PBS). Lave os slides com PBS 3 x 5 min no RT.
ATENÇÃO: Para o resto do protocolo, certifique-se de manter os slides longe da luz para preservar a fluorescência. - Incubar por 1 h na RT com a solução secundária de anticorpos contendo anticorpo anti-rato-AF647 (1:500) em PBS. Lave os slides com PBS 3 x 5 min no RT.
- Incubar por 20 min em RT com uma solução de 4',6-diamidino-2-fenilôndole (DAPI, 0,5 μg/mL) e BODIPY (1 μg/mL) em PBS.
NOTA: Para preparar a solução de estoque BODIPY, dissolva-a em DMSO a uma concentração de 1 mg/mL. Diferentes formulações bodipy estão disponíveis comercialmente para coloração LD. Dependendo da escolha feita, o método de coloração é o mesmo (mesmos passos, concentração e tempo de incubação); no entanto, o método de aquisição será ligeiramente diferente. - Depois de uma primeira lavagem rápida, lave os slides com PBS 3 x 5 min no RT, enxágue em H2O duplamente destilado, remova o excesso de água e monte com um reagente antifado.
NOTA: Armazene os slides a 4 °C, protegidos da luz, para preservar a fluorescência até a aquisição da imagem.
5. Aquisição de imagens
NOTA: Uma vez concluídos os protocolos de coloração, é importante proceder imediatamente à aquisição de imagens (dentro das seguintes 24h), não apenas para evitar contaminação, mas também para preservar a morfologia, tamanho e número de LDs.
- Aquisição de imagens para avaliar os tipos de fibras de cada músculo amostrado
NOTA: Esta etapa pode ser alcançada com um microscópio de varredura de fluorescência de lâmina total ou com um microscópio de epifluorescência convencional. Com esta última, a costura manual ou automatizada das imagens deve ser feita para reconstruir a seção.- Para reconhecimento do tipo de fibra, utilize um microscópio de epifluorescência com um objetivo de 10x/0.3. Selecione filtros de excitação para DAPI (405 nm), FITC, TRITC e Cy5 para detectar as fibras tipo I, IIa, IIx e laminina, respectivamente.
NOTA: As fibras IIb tipo não serão imunolabeladas. Eles serão reconhecidos como fibras manchadas por laminina nos limites do sarcolemma com um sarcoplasmo preto. - Ajuste o tempo de exposição adequado para cada canal.
- Ao usar um microscópio de epifluorescência convencional, sempre adquira as imagens de todo o músculo seguindo a mesma ordem para facilitar a reconstrução muscular. Certifique-se de que as fibras na borda direita de uma imagem também apareçam na borda esquerda da imagem a seguir. O mesmo se aplica às partes superior e inferior das imagens (Figura 3A).
NOTA: Como referência, para uma seção do EDL ou do soleus dissecado de um rato de 3 meses de idade, uma média de seis e oito imagens, respectivamente, cobrirá toda a seção transversal muscular. - Depois que o músculo é escaneado, carregue as imagens digitais capturadas em qualquer software de processamento de imagem para reconstrução (costura), com base na morfologia de fibras (laminina) e na histologia da seção muscular, e salve-a como um arquivo TIFF, PNG ou JPG com todos os canais (cores) mesclados (Figura 3A).
- Para reconhecimento do tipo de fibra, utilize um microscópio de epifluorescência com um objetivo de 10x/0.3. Selecione filtros de excitação para DAPI (405 nm), FITC, TRITC e Cy5 para detectar as fibras tipo I, IIa, IIx e laminina, respectivamente.
- Aquisição de imagens com co-coloração de laminina e Bodipy
NOTA: Para reconhecer o tipo de fibra e ter uma estimativa do número de fibras para cada tipo capturado, é essencial ter a seção do tipo fibra já escaneada e os músculos reconstruídos antes de iniciar a aquisição de imagem de Bodipy-laminin (Figura 3B).- Para observação e aquisição de imagem Bodipy, use um microscópio confocal com uma lente objetiva de imersão de óleo de 40x com uma abertura numérica de 1,4.
- Use as seguintes configurações: pinhole a 1 UA, resolução de 2.048 pixels x 2.048 pixels, tamanho do pixel a 0,08 μm, modo unidirecional, velocidade de varredura em 4 (~4 μs/pixel), linha de média definida para 4x e zoom digital definido para 1.
- Para evitar conversas cruzadas entre Bodipy-558/568 e laminin-AF647, use o modo de varredura sequencial no software confocal.
NOTA: Quando o corante escolhido é Bodipy-493/503, a varredura simultânea de laser confocal é possível sem conversa cruzada entre o canal Bodipy e o canal laminin-AF647. Isso acelerará a aquisição de imagens. - Excite Bodipy-493/503 usando a linha laser de 488 nm ou linha laser de argônio, e excite Bodipy-555/568 usando a linha laser de diodo de 561 nm. Finalmente, detecte laminin-AF647 com uma linha laser de diodo de 640 nm.
ATENÇÃO: Tenha em mente que as moléculas bodipy são muito sensíveis ao fotobleaching, por isso evite a varredura desnecessária a laser. Para reconhecer fibras, use apenas o laser para laminina. - Dependendo do corante escolhido, defina as faixas de emissão em 570-650 nm para Bodipy-493/50324 e em 565-620 nm para Bodipy-558/568. Defina a faixa de emissão para laminina em 656-700 nm.
- Defina o ganho e o ganho digital adequadamente para que nenhum pixel saturado seja detectado no indicador de intervalo. Corrija o sinal de fundo ajustando o deslocamento.
NOTA: A seleção do filtro e outros parâmetros de varredura acima mencionados devem ser otimizados para cada microscópio confocal. É importante que todas as configurações acima mencionadas sejam mantidas constantes para que todas as imagens capturadas a partir de amostras sejam comparadas. - Para identificar o tipo de fibra entre os visualizados no microscópio confocal, utilize um laptop pessoal no qual a imagem da seção reconstruída após a imunodedoca do tipo fibra seja verificada (Figura 3B).
- Uma vez que um grupo de fibras é corretamente identificado, adquira a imagem com os canais Bodipy e laminin.
NOTA: Recomenda-se observar o nome da imagem Bodipy-laminin na região do músculo onde essas fibras estão localizadas na imagem adquirida para o reconhecimento myHC para facilitar a análise posterior de fibras específicas de LDs.
6. Análise de imagens
- Análise dos tipos de fibras em cada amostra muscular
- Em Fiji (ou ImageJ)32, abra o arquivo TIFF, PNG ou JPG com o músculo reconstruído obtido a partir da fusão de todos os canais utilizados para detectar as isoformas de fibra.
- Para iniciar a ferramenta Contagem de células , clique em Plugins | Analisar | Contador de células | Contador de células.
- Na janela do contador Celular , clique em Ações | Inicialize.
- Em Contadores na mesma janela, selecione Tipo 1.
- Na janela principal de Fiji, selecione a ferramenta Varinha .
- Para quantificar o número de fibras de cada tipo, clique em cada fibra do mesmo tipo, para que o programa registra o número de fibras clicadas.
- Uma vez terminado, selecione o próximo tipo de fibra e repita os mesmos passos.
- Quando todas as fibras da imagem tiverem sido atribuídas a um determinado tipo de fibra, clique em Resultados na janela Contador de células para exibir os resultados em uma tabela.
- Salve esta tabela como uma tabela de planilha clicando em Arquivo | Salve Como na janela Resultados .
- Salve e recarregue as seleções na mesma imagem a qualquer momento clicando em Salvar marcadores ou marcadores de carga, respectivamente, na janela Contagem de células .
- Análise de gotículas lipídicas de forma dependente de fibras
- Analise as imagens de Bodipy e laminina obtidas no confocal usando Fiji para quantificação de LDs.
NOTA: Os autores criaram uma macro personalizada para automatizar a análise. Esta macro, juntamente com uma explicação passo a passo sobre como usá-la, está disponível como Arquivo Suplementar 1 e Arquivo Suplementar 2, respectivamente. - Abra cada imagem com o auxílio do Importador de Bioformitos de Fiji. Na opção Exibir pilha com opção, selecione Hyperstack | Modo de cor, Padrão. Certifique-se de que a janela Escala automática esteja selecionada.
NOTA: As seguintes etapas descreverão o protocolo para a análise de uma fibra na imagem, mas deve ser repetido tantas vezes quanto o número de fibras inteiras aparecem na imagem. - Use a ferramenta de seleção à mão livre para selecionar manualmente o sarcolemma da fibra com base no canal laminin (Figura 4A) e pressione T no teclado para gravar a seleção ou Região de Interesse (ROI) na janela ROI .
- Vá até a janela principal de Fiji e clique em Analisar | Defina medidas e na janela pop-up, selecione Área e diâmetro de Feret. Deixe as caixas restantes desmarcadas e outros parâmetros à medida que aparecem por padrão.
- Clique em Medir na janela ROI para obter a Área e o Diâmetro Mínimo do Feret (MF) da fibra selecionada e observe-as para uso posterior.
NOTA: Ao analisar LDs, é importante ter em mente que seu tamanho e densidade variam entre o centro e a periferia (região subsarcolemmal-SS) da fibra. Portanto, a análise deve ser feita separadamente. - Calcule o valor de 1/6 do MF para delimitar a parte central da fibra.
NOTA: Na macro, o valor mf padrão é definido para 6, o que significa que a redução aplicada será definida como 1/6 do MF. Esse valor foi escolhido com base nos dados empíricos obtidos do soleus dos animais alimentados com uma dieta rica em gordura. No entanto, cada pesquisador deve alterar esse número com base nos dados empíricos e nos músculos analisados, no tipo de fibra e na condição animal. - Na janela ROI , clique em Adicionar para ter uma duplicata do primeiro ROI e selecione o segundo ROI que aparece na janela.
- Na janela principal de Fiji, clique em Editar | | de seleção Amplie e introduza o valor calculado anteriormente (a partir da etapa 6.2.6.) com um sinal de menos antes do número e clique em OK. Na janela ROI, clique em Adicionar[t] (um terceiro ROI deve aparecer) e Excluir, para excluir o segundo ROI.
NOTA: O pesquisador pode conferir os resultados clicando na caixa Mostrar Tudo na janela roi . Neste ponto, devem aparecer dois ROIs, um que circunda todo o perímetro da fibra (Figura 4B) e outro que é colocado no centro (Figura 4C). - Selecione ambos os ROIs na janela ROI e clique em Mais | XOR | Adicione[t]. Aguarde que um terceiro ROI apareça, o que corresponde à periferia da fibra (Figura 4D).
- Salve os ROIs clicando em Mais | Economize para salvar os ROIs no caso de uma reanálise posterior das mesmas fibras ser necessária.
- Selecione o canal Bodipy e abra a ferramenta Limiar clicando em Imagem | Ajuste | Limiar na janela principal de Fiji.
- Na janela pop-up Threshold , defina os valores para 70/255, selecione Yen | Método B&W e clique em | de fundo escuro Inscreva-se.
NOTA: Os valores aplicados no Limiar podem variar dependendo das condições do experimento e o limiar deve ser adequadamente definido para otimizar a análise. Uma janela B&W com o sinal Bodipy acima do limite de limiar mostrado em branco e o fundo em preto deve aparecer (compare a imagem Bodipy original na Figura 4E com a da Figura 4F). - Vá até a janela principal de Fiji e clique em Analisar | Defina medidas e na janela pop-up, selecione Área, Fração de área e Limite ao limite. Deixe as caixas restantes desmarcadas e outros parâmetros à medida que aparecem por padrão.
NOTA: Se o pesquisador quiser analisar a "circularidade" dos LDs, que é um índice da morfologia esférica que varia de 1 para uma esfera perfeita a 0 para uma linha, clique na caixa de descritores de forma da janela pop-up Set Measurements . - Vá para a janela do ROI e selecione o primeiro ROI. Na janela principal de Fiji, clique em Analisar | Analise a ferramenta de partículas.
NOTA: Esta ferramenta quantifica o número, tamanho, área coberta e porcentagem da área total coberta por partículas em cada seleção e salva os resultados como um arquivo de planilha. - Defina os valores de 2 a Infinity (2-Infinity) na janela Analisar partículas , verifique a caixa Pixels , mantenha os valores de circularidade padrão, selecione Resumo e clique em OK.
NOTA: Para verificar os resultados da fibra, na opção Mostrar , selecione qualquer uma das opções disponíveis. Para ter as informações de cada LD reconhecidas na seleção em uma tabela, verifique a opção Resultados de Exibição na janela Analisar partículas . Os resultados da análise da área total da fibra são mediados e resumidos em uma tabela com várias colunas (Contagem, Área Total, Tamanho Médio, % Área; estas correspondem ao número de partículas [LDs], a área ocupada por essas partículas, seu tamanho médio, e a porcentagem da área total da seleção ocupada por partículas, respectivamente). Para calcular a densidade, divida o número de partículas pela área total de cada seleção. - Para obter os valores do centro e da periferia da fibra, repita as etapas 6.2.14 e 6.2.15, selecionando o segundo (centro) e o terceiro ROI (periferia) de cada vez.
- Salve os resultados clicando em Arquivo | Guarde como na janela Resumo .
NOTA: Inclua o tipo de fibra, a condição e o nome da imagem no nome atribuído dos resultados para facilitar a unificação posterior e a análise estatística dos dados. Para analisar o resto das fibras na mesma imagem, repita as etapas 6.2.3 a 6.2.17. Para a análise estatística, pelo menos 10-15 fibras de cada tipo devem ser analisadas por animal.
- Analise as imagens de Bodipy e laminina obtidas no confocal usando Fiji para quantificação de LDs.
Access restricted. Please log in or start a trial to view this content.
Resultados
O protocolo descrito aqui fornece um método eficiente para quantificar facilmente os LDs de forma subcelular e subcelular. Mostra como, congelando juntos dois músculos de tamanho semelhante, como o EDL e o soleus, o tempo e os recursos gastos nas etapas seguintes são reduzidos pela metade.
Um protocolo completo é fornecido para imunostaining, aquisição de imagem e análise dos diferentes isoformes MyHC expressos em músculos de camundongos adultos. Este protocolo é baseado no primeiro p...
Access restricted. Please log in or start a trial to view this content.
Discussão
O protocolo aqui detalhado descreve um método eficiente para quantificar LDs marcados com Bodipy em uma base tipo de fibra e subcelular específico. Nos últimos anos, corantes lipídicos clássicos, como ORO ou Sudan Black B, foram substituídos por uma nova matriz de corantes celulares permeáveis, lipofílicos, fluorescentes que se ligam a lipídios neutros (por exemplo, Bodipy). Disponível como diferentes conjugados, Bodipy tem se mostrado muito eficaz na marcação de LDs para estudar sua morfologia, dinâmica e i...
Access restricted. Please log in or start a trial to view this content.
Divulgações
Os autores não têm conflitos de interesse para declarar.
Agradecimentos
Este trabalho foi apoiado por subvenções da Fonds National de la Recherche Scientifique (FNRS-Crédit de Recherche J.0022.20) e da Société Francophone du Diabète (SFD-Roche Diabetes Care).C.M.S. é o beneficiário de uma bolsa de doutorado da FRIA (FNRS). M.A.D.-L.d.C. recebeu uma bolsa do Programa internacional de Excelência Wallonie-Bruxelles.
Os autores agradecem a Alice Monnier por sua contribuição para o desenvolvimento deste protocolo e Caroline Bouzin por sua expertise e ajuda técnica no processo de aquisição de imagens. Agradecemos também à plataforma de imagem 2IP-IREC pelo acesso ao criostat e aos microscópios (2IP-IREC Imaging Platform, Institute of Experimental and Clinical Research, Université Catholique de Louvain, 1200 Bruxelas, Bélgica). Por fim, os autores gostariam de agradecer Nicolas Dubuisson, Romain Versele e Michel Abou-Samra pela crítica construtiva ao manuscrito. Algumas das figuras deste artigo foram criadas com BioRender.com.
Access restricted. Please log in or start a trial to view this content.
Materiais
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| AxioCam 506 mono 6 Mpix camera | Zeiss | ||
| AxioCam MRm 1.4MPix CCD camera | Zeiss | ||
| Chemical hood | Potteau Labo | EN-14175 | |
| Confocal microscope | Zeiss | LSM800 | |
| Cork discs (ø 20 mm, 3 mm thick) | Electron Microscopy Sciences | 63305 | |
| Cryo-Gloves | Tempshield | 16072252 | |
| Cryostat | Thermo Scientific | Microm Cryo Star HM 560 | |
| Dissecting Stereo Microscope SMZ745 | Nikon | ||
| Dry Ice | |||
| Dumont Forceps | F.S.T | 11295-10 | |
| Epifluorescence microscope | Zeiss | AxioImage-Apotome Z1 | |
| Extra Fine Bonn Scissors | F.S.T | 14084-08 | |
| FisherBrand Disposable Base Molds (0.7 x 0.7 cm) | ThermoFisher | 22-363-552 | Used to cut a piece to hold the muscle on the cork for freezing |
| Glass petri dish (H 25 mm, ø 150 mm) | BRAND Petri dish, MERK | BR455751 | Used to place the muscles on ice during dissection |
| ImmEdge Hydrophobic barrier PAP Pen | Vector Labs | H-4000 | Used to create an hidrophobic barrier around the muscle sections |
| Incubator | MMM Medcenter | Incucell 707 | |
| Microscope Cover Glasses (24x50 mm) | Assistent | 40990151 | |
| Microscope Slide Boxes | Kartell | 278 | Used as humid chambers for immunohistochemistry |
| Neck holder | Linie zwo | SB-035X-02 | Used as strap to hold the stainless steel tumbler |
| No 15 Sterile Carbon Steel Scalpel Blade | Swann-Morton | 0205 | |
| Paint brushes | Van Bleiswijck | Amazon B07W7KJQ2X | Used to handle cryosections |
| Permanent Marker Pen Black | Klinipath/VWR | 98307-R | Used to label slides |
| Pierce Fixation Forceps | F.S.T | 18155-13 | |
| Polystyrene Box | H 12 cm x L 25 cm x W 18 cm, used as a liquid nitrogen container and to transport the samples to the cryostat | ||
| Scalpel Handle, 125 mm (5"), No. 3 | Aesculap | BB073R | |
| Stainless Steel Cup 10oz | Eboxer | B07GFCBPFH | Tumbler to fill with isopentene for muscle freezing |
| Superfrost Ultra Plus slides | ThermoFisher | J1800AMNZ | |
| Surgical tweezers 1/2 teeth | Medische Vakhandel | 1303152 | Also called "Rat teeth tweezers" |
| Vannas Spring Scissors - 3 mm Cutting Edge | F.S.T | 15000-00 | |
| Weighing boats | VWR international | 611-2249 | |
| Whole-Slide Scanner for Fluorescence | Zeiss | Axio Scan.Z1 | |
| Reagents | |||
| Alexa Fluor 405 Goat Anti-Mouse IgG2b | Sigma-Aldrich | SAB4600477 | Used at a final concentration of 1:500 |
| Alexa Fluor 488 Goat Anti-Mouse IgG1 | ThermoFisher | A-21121 | Used at a final concentration of 1:500 |
| Alexa Fluor 568 Goat Anti-Mouse IgM | Abcam | ab175702 | Used at a final concentration of 1:1,000 |
| Alexa Fluor 647 goat anti rat-IgG (H+L) secondary antibody | ThermoFisher | A-21247 | Used at a final concentration of 1:500 |
| BODIPY-493/503 (4,4-difluoro-1,3,5,7,8-pentametil-4-bora-3a,4a-diaza-s-indaceno) | ThermoFisher | D3922 | Used at a final concentration of 1 µg/mL |
| BODIPY-558/568 C12 (4,4-Difluoro-5-(2-Thienyl)-4-Bora-3a,4a-Diaza-s-Indacene-3-Dodecanoic Acid) | ThermoFisher | D3835 | Used at a final concentration of 1 µg/mL |
| DAPI (4',6-diamidino-2-phenylindole) | ThermoFisher | D1306 | Used at a final concentration of 0.5 µg/mL |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich | D-8418 | Used to solve Bodipy for the 1 mg/mL stock solution. CAUTION: Toxic and flammable. Vapors may cause irritation. Manipulate in a fume hood. Avoid direct contact with skin. Wear rubber gloves, protective eye goggles. |
| Formaldehyde solution 4%, buffered, pH 6.9 | Sigma-Aldrich | 1004969011 | CAUTION: May cause an allergic skin reaction. Suspected of causing genetic defects. May cause cancer. Manipulate in a fume hood. Avoid direct contact with skin. Wear rubber gloves, protective eye goggles. |
| Isopentane GPR RectaPur | VWR international | 24872.298 | CAUTION: Extremely flammable liquid and vapor. May be fatal if swallowed and enters airways. May cause drowsiness or dizziness. Repeated exposure may cause skin dryness or cracking. Wear protective gloves/protective clothing/eye protection/face protection. |
| Liquid Nitrogen | CAUTION: Extremely cold. Wear gloves. Handle slowly to minimize boiling and splashing and in well ventilated areas. Use containers designed for low-temperature liquids. | ||
| Mouse on mouse Blocking Reagent | Vector Labs | MKB-2213-1 | Used at concentration of 1:30 |
| Myosin heavy chain Type I (BA-D5-s Primary Antibody) Gene: MYH7, monoclonal bovine anti mouse IgG2b | DSHB University of Iowa | BA-D5-supernatant | Used at a final concentration of 1:10 |
| Myosin heavy chain Type IIA (SC-71-s Primary Antibody) Gene: MYH2, Monoclonal bovine anti mouse IgG1 | DSHB University of Iowa | SC-71-supernatant | Used at a final concentration of 1:10 |
| Myosin heavy chain Type IIX (6H1-s Primary Antibody), Gene: MYH1, Monoclonal rabbit anti mouse IgM | Developmental Studies Hybridoma Bank, University of Iowa | 6H1-supernatant | Used at a final concentration of 1:5 |
| Normal Goat Serum (NGS) | Vector Labs | S-1000 | |
| PBS 0.1 M | Commonly used on histology laboratories | ||
| ProLong Gold Antifade Mountant | Invitrogen | P36930 | |
| Rat anti-Laminin-2 (α-2 Chain) primary antibody (monoclonal) | Sigma-Aldrich | L0663 | Used at a final concentration of 1:1,000 |
| Tissue-Tek O.C.T compound | Sakura | 4583 | |
| Software | |||
| Adobe Illustrator CC | Adobe Inc. | Used to design the figures | |
| Adobe Photoshop | Adobe Inc. | Confocal software | |
| BioRender | https://biorender.com/ | Used to design the figures | |
| Fiji/ImageJ | https://imagej.net/software/fiji/ | Used to analyse the acquired images | |
| Microsoft PowerPoint | Microsoft | Used to reconstruct the histology of the whole muscle after scanning the fiber types | |
| Zen Blue 2.6 | Zeiss | Used to reconstruct the histology of the whole muscle after scanning the fiber types |
Referências
- Correa-de-Araujo, R., et al. Myosteatosis in the context of skeletal muscle function deficit: an interdisciplinary workshop at the National Institute on Aging. Frontiers in Physiology. 11, 963(2020).
- Miljkovic, I., et al. Greater skeletal muscle fat infiltration is associated with higher all-cause and cardiovascular mortality in older men. Journals of Gerontology Series A: Biomedical Sciences and Medical Sciences. 70 (9), 1133-1140 (2015).
- Nachit, M., et al. Myosteatosis rather than sarcopenia associates with non-alcoholic steatohepatitis in non-alcoholic fatty liver disease preclinical models. Journal of Cachexia, Sarcopenia, and Muscle. 12 (1), 144-158 (2021).
- Aleixo, G. F. P., et al. Myosteatosis and prognosis in cancer: Systematic review and meta-analysis. Critical Reviews in Oncology/Hematolgoy. 145, 102839(2020).
- Gemmink, A., Schrauwen, P., Hesselink, M. K. C. Exercising your fat (metabolism) into shape: a muscle-centred view. Diabetologia. 63 (8), 1453-1463 (2020).
- van Loon, L. J. Use of intramuscular triacylglycerol as a substrate source during exercise in humans. Journal of Applied Physiology. 97 (4), 1170-1187 (2004).
- Coen, P. M., Goodpaster, B. H. Role of intramyocelluar lipids in human health. Trends in Endocrinology and Metabolism. 23 (8), 391-398 (2012).
- Seibert, J. T., Najt, C. P., Heden, T. D., Mashek, D. G., Chow, L. S. Muscle lipid droplets: cellular signaling to exercise physiology and beyond. Trends in Endocrinology and Metabolism. 31 (12), 928-938 (2020).
- Bergman, B. C., Goodpaster, B. H. Exercise and muscle lipid content, composition, and localization: influence on muscle insulin sensitivity. Diabetes. 69 (5), 848-858 (2020).
- Nielsen, J., Christensen, A. E., Nellemann, B., Christensen, B. Lipid droplet size and location in human skeletal muscle fibers are associated with insulin sensitivity. American Journal of Physiology-Endocrinology and Metabolism. 313 (6), 721-730 (2017).
- Covington, J. D., et al. Intramyocellular lipid droplet size rather than total lipid content is related to insulin sensitivity after 8 weeks of overfeeding. Obesity (Silver Spring). 25 (12), 2079-2087 (2017).
- Bosma, M. Lipid droplet dynamics in skeletal muscle). Experimental Cell Research. 340 (2), 180-186 (2016).
- Nielsen, J., et al. Increased subsarcolemmal lipids in type 2 diabetes: effect of training on localization of lipids, mitochondria, and glycogen in sedentary human skeletal muscle. American Journal of Physiology-Endocrinology and Metabolism. 298 (3), 706-713 (2010).
- Ferreira, R., et al. Subsarcolemmal and intermyofibrillar mitochondria proteome differences disclose functional specializations in skeletal muscle. Proteomics. 10 (17), 3142-3154 (2010).
- Barrett, J. S., Whytock, K. L., Strauss, J. A., Wagenmakers, A. J. M., Shepherd, S. O. High intramuscular triglyceride turnover rates and the link to insulin sensitivity: influence of obesity, type 2 diabetes and physical activity. Applied Physiology, Nutrition and Metabolism. , 1-14 (2022).
- Daemen, S., et al. Distinct lipid droplet characteristics and distribution unmask the apparent contradiction of the athlete's paradox. Molecular Metabolism. 17, 71-81 (2018).
- Bredella, M. A., Ghomi, R. H., Thomas, B. J., Miller, K. K., Torriani, M. Comparison of 3.0 T proton magnetic resonance spectroscopy short and long echo-time measures of intramyocellular lipids in obese and normal-weight women. Journal of Magnetic Resonance Imaging. 32 (2), 388-393 (2010).
- Schrauwen-Hinderling, V. B., Hesselink, M. K., Schrauwen, P., Kooi, M. E. Intramyocellular lipid content in human skeletal muscle. Obesity (Silver Spring). 14 (3), 357-367 (2006).
- De Bock, K., et al. Evaluation of intramyocellular lipid breakdown during exercise by biochemical assay, NMR spectroscopy, and Oil Red O staining. American Journal of Physiology-Endocrinology and Metabolism. 293 (1), 428-434 (2007).
- Koopman, R., Schaart, G., Hesselink, M. K. Optimisation of oil red O staining permits combination with immunofluorescence and automated quantification of lipids. Histochemistry and Cell Biology. 116 (1), 63-68 (2001).
- Gueugneau, M., et al. Skeletal muscle lipid content and oxidative activity in relation to muscle fiber type in aging and metabolic syndrome. Journal of Gerontology Series A: Biomedical Sciences and Medical Sciences. 70 (5), 566-576 (2015).
- Gemmink, A., et al. Super-resolution microscopy localizes perilipin 5 at lipid droplet-mitochondria interaction sites and at lipid droplets juxtaposing to perilipin 2. Biochimica et Biophysica Acta-Molecular and Cell Biology of Lipids. 1863 (11), 1423-1432 (2018).
- Spangenburg, E. E., Pratt, S. J. P., Wohlers, L. M., Lovering, R. M. Use of BODIPY (493/503) to visualize intramuscular lipid droplets in skeletal muscle. Journal of Biomedicine and Biotechnology. 598358, (2011).
- Prats, C., et al. An optimized histochemical method to assess skeletal muscle glycogen and lipid stores reveals two metabolically distinct populations of type I muscle fibers. PLoS One. 8 (10), 77774(2013).
- Strauss, J. A., Shepherd, D. A., Macey, M., Jevons, E. F. P., Shepherd, S. O. Divergence exists in the subcellular distribution of intramuscular triglyceride in human skeletal muscle dependent on the choice of lipid dye. Histochemistry and Cell Biology. 154 (4), 369-382 (2020).
- Shepherd, S. O., et al. Sprint interval and traditional endurance training increase net intramuscular triglyceride breakdown and expression of perilipin 2 and 5. Journal of Physiology. 591 (3), 657-675 (2013).
- Whytock, K. L., et al. A 7-day high-fat, high-calorie diet induces fibre-specific increases in intramuscular triglyceride and perilipin protein expression in human skeletal muscle. Journal of Physiology. 598 (6), 1151-1167 (2020).
- Wang, C., Yue, F., Kuang, S. Muscle histology characterization using h&e staining and muscle fiber type classification using immunofluorescence staining. Bio-Protocol. 7 (10), (2017).
- Meng, H., et al. Tissue triage and freezing for models of skeletal muscle disease. Journal of Visualized Experiments: JoVE. (89), e51586(2014).
- Kumar, A., Accorsi, A., Rhee, Y., Girgenrath, M. Do's and don'ts in the preparation of muscle cryosections for histological analysis. Journal of Visualized Experiments: JoVE. (99), e52793(2015).
- Leiva-Cepas, F., et al. Laboratory methodology for the histological study of skeletal muscle. Archivos de Medicina del Deporte. 35 (186), 254-262 (2018).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Schiaffino, S., et al. Three myosin heavy chain isoforms in type 2 skeletal muscle fibres. Journal of Muscle Research & Cell Motility. 10 (3), 197-205 (1989).
- Komiya, Y., et al. Mouse soleus (slow) muscle shows greater intramyocellular lipid droplet accumulation than EDL (fast) muscle: fiber type-specific analysis. Journal of Muscle Research & Cell Motility. 38 (2), 163-173 (2017).
- Andrich, D. E., et al. Altered lipid metabolism impairs skeletal muscle force in young rats submitted to a short-term high-fat diet. Frontiers in Physiology. 9, 1327(2018).
- Schiaffino, S. Fibre types in skeletal muscle: a personal account. Acta Physiologica. 199 (4), 451-463 (2010).
- Bloemberg, D., Quadrilatero, J. Rapid determination of myosin heavy chain expression in rat, mouse, and human skeletal muscle using multicolor immunofluorescence analysis. PLoS One. 7 (4), 35273(2012).
- Gemmink, A., et al. Decoration of intramyocellular lipid droplets with PLIN5 modulates fasting-induced insulin resistance and lipotoxicity in humans. Diabetologia. 59 (5), 1040-1048 (2016).
- Askinas, C., et al. Biophotonics Congress: Biomedical Optics Congress 2018 (Microscopy/Translational/Brain/OTS). , JTu3A.4 (Optical Society of America) (2018).
- Morén, B., et al. EHD2 regulates adipocyte function and is enriched at cell surface-associated lipid droplets in primary human adipocytes. Molecular Biology of the Cell. 30 (10), 1147-1159 (2019).
- Benador, I. Y., et al. Mitochondria bound to lipid droplets have unique bioenergetics, composition, and dynamics that support lipid droplet expansion. Cell Metabolism. 27 (4), 869-885 (2018).
- de la Rosa Rodriguez, M. A., et al. Hypoxia-inducible lipid droplet-associated induces DGAT1 and promotes lipid storage in hepatocytes. Molecular Metabolism. 47, 101168(2021).
- Jevons, E. F. P., Gejl, K. D., Strauss, J. A., Ørtenblad, N., Shepherd, S. O. Skeletal muscle lipid droplets are resynthesized before being coated with perilipin proteins following prolonged exercise in elite male triathletes. American Journal of Physiology-Endocrinology and Metabolism. 318 (3), 357-370 (2020).
- Ohsaki, Y., Maeda, T., Fujimoto, T. Fixation and permeabilization protocol is critical for the immunolabeling of lipid droplet proteins. Histochemistry and Cell Biology. 124 (5), 445-452 (2005).
- Prats, C., et al. Decrease in intramuscular lipid droplets and translocation of HSL in response to muscle contraction and epinephrine. Journal of Lipid Research. 47 (11), 2392-2399 (2006).
- Listenberger, L. L., Brown, D. A. Fluorescent detection of lipid droplets and associated proteins. Current Protocols in Cell Biology. , Chapter 24 (Unit 24.22) (2007).
- Xue, Y., Lim, S., Bråkenhielm, E., Cao, Y. Adipose angiogenesis: quantitative methods to study microvessel growth, regression and remodeling in vivo. Nature Protocols. 5 (5), 912-920 (2010).
- Muliyil, S., et al. ADAM17-triggered TNF signalling protects the ageing Drosophila retina from lipid droplet-mediated degeneration. The EMBO Journal. 39 (17), 104415(2020).
- Yan, Q., et al. Autophagy activation contributes to lipid accumulation in tubular epithelial cells during kidney fibrosis. Cell Death Discovery. 4, 2(2018).
- Coassin, S., et al. Investigation and functional characterization of rare genetic variants in the adipose triglyceride lipase in a large healthy working population. PLoS Genetics. 6 (12), 1001239(2010).
- Daemen, S., van Zandvoort, M., Parekh, S. H., Hesselink, M. K. C. Microscopy tools for the investigation of intracellular lipid storage and dynamics. Molecular Metabolism. 5 (3), 153-163 (2016).
- Chen, Q., et al. Rab8a deficiency in skeletal muscle causes hyperlipidemia and hepatosteatosis by impairing muscle lipid uptake and storage. Diabetes. 66 (9), 2387-2399 (2017).
- Gemmink, A., et al. Decoration of myocellular lipid droplets with perilipins as a marker for in vivo lipid droplet dynamics: A super-resolution microscopy study in trained athletes and insulin resistant individuals. Biochimica et Biophysica Acta (BBA)-Molecular and Cell Biology of Lipids. 1866 (2), 158852(2021).
- Bergman, B. C., Hunerdosse, D. M., Kerege, A., Playdon, M. C., Perreault, L. Localisation and composition of skeletal muscle diacylglycerol predicts insulin resistance in humans. Diabetologia. 55 (4), 1140-1150 (2012).
Access restricted. Please log in or start a trial to view this content.
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados