Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
L’imagerie par feuillet de lumière révèle la structure cardiaque du cœur des rongeurs
Dans cet article
Résumé
Le protocole utilise la microscopie à feuillet de lumière avancée ainsi que des méthodes de nettoyage tissulaire adaptées pour étudier les structures cardiaques complexes dans les cœurs de rongeurs, ce qui présente un grand potentiel pour la compréhension de la morphogenèse et du remodelage cardiaques.
Résumé
La microscopie à feuillet de lumière (LSM) joue un rôle central dans la compréhension de la structure tridimensionnelle complexe (3D) du cœur, fournissant des informations cruciales sur la physiologie cardiaque fondamentale et les réponses pathologiques. Nous nous penchons ici sur le développement et la mise en œuvre de la technique LSM pour élucider la micro-architecture du cœur dans des modèles murins. La méthodologie intègre un système LSM personnalisé avec des techniques de nettoyage des tissus, atténuant la diffusion de la lumière dans les tissus cardiaques pour l’imagerie volumétrique. La combinaison d’un LSM conventionnel avec des approches d’assemblage d’images et de déconvolution multivues permet de capturer l’ensemble du cœur. Pour résoudre le compromis inhérent entre la résolution axiale et le champ de vision (FOV), nous introduisons une méthode de microscopie à feuillet de lumière à balayage axial (ASLM) pour minimiser la lumière floue et éclairer uniformément le cœur dans le sens de la propagation. Entre-temps, les méthodes de nettoyage des tissus telles que iDISCO améliorent la pénétration de la lumière, facilitant la visualisation des structures profondes et assurant un examen complet du myocarde dans l’ensemble du cœur. La combinaison du LSM proposé et des méthodes de nettoyage des tissus présente une plate-forme prometteuse pour les chercheurs dans la résolution des structures cardiaques dans les cœurs de rongeurs, avec un grand potentiel pour la compréhension de la morphogenèse et du remodelage cardiaques.
Introduction
L’insuffisance cardiaque reste la principale cause de mortalité dans le monde, principalement en raison du manque de capacité de régénération des cardiomyocytes matures1. L’architecture complexe du cœur joue un rôle crucial dans sa fonction et donne un aperçu des processus de développement. Une compréhension approfondie de la structure cardiaque est essentielle pour élucider les processus fondamentaux de la morphogenèse et du remodelage cardiaques en réponse à l’infarctus du myocarde. Des progrès récents ont démontré que les souris néonatales peuvent restaurer la fonction cardiaque après une blessure, alors que les souris adultes n’ont pas une telle capacité de régénération2. Cela établit une base pour l’étude des indices associés aux anomalies structurelles et fonctionnelles dans les modèles murins. Les méthodes d’imagerie traditionnelles, telles que la microscopie confocale, présentent des limites techniques, notamment une profondeur de pénétration limitée, un schéma de balayage ponctuel lent et des dommages photographiques dus à une exposition prolongée à la lumière laser. Ceux-ci entravent l’imagerie tridimensionnelle (3D) complète du cœur intact. Dans ce contexte, la microscopie à feuillet de lumière (LSM) apparaît comme une solution puissante, offrant les avantages d’une imagerie à grande vitesse, d’une réduction des dommages photographiques et de capacités exceptionnelles de sectionnement optique 3,4,5. Les caractéristiques uniques de la LSM en font une méthode prometteuse pour surmonter les limites des techniques conventionnelles, fournissant des informations sans précédent sur les processus de développement et de remodelage cardiaques 6,7,8.
Dans ce protocole, nous introduisons une stratégie d’imagerie qui combine une LSM avancée avec des approches de clarification tissulaire adaptées9, permettant l’imagerie de cœurs de souris entiers sans avoir besoin d’un marquage spécifique et d’une section mécanique. Nous proposons en outre que l’imagerie LSM conventionnelle puisse être améliorée par des techniques de déconvolutionmultivue 10 ou de microscopie à feuillet de lumière à balayage axial (ASLM) 11,12,13,14,15 pour améliorer la résolution axiale. De plus, l’intégration de l’assemblage d’images avec l’une ou l’autre de ces méthodes peut surmonter efficacement le compromis entre la résolution spatiale et le champ de vision (FOV), faisant ainsi progresser l’imagerie des cœurs de souris adultes. L’incorporation de nombreuses approches de nettoyage des tissus, y compris les méthodes hydrophobes, hydrophiles et à base d’hydrogel, permet une pénétration plus profonde de la lumière pour capturer la morphologie de l’ensemble du cœur 16,17,18,19.
Bien que plusieurs méthodes de clarification soient compatibles avec les systèmes LSM actuels, l’objectif est de minimiser la diffusion des photons et d’améliorer la pénétration de la lumière dans les tissus, comme le cœur, en remplaçant les lipides par un milieu qui correspond étroitement à son indice de réfraction. iDISCO a été choisi comme représentant20,21 et adapté à l’imagerie par autofluorescence dans ce protocole en raison de son traitement rapide et de sa grande transparence (Figure 1A). Collectivement, l’intégration de l’approche LSM avancée avec des techniques de nettoyage des tissus offre un cadre prometteur pour démêler l’anatomie cardiaque complexe dans les cœurs de rongeurs, avec un potentiel important pour faire progresser notre compréhension de la morphogenèse et de la pathogenèse cardiaques.
Protocole
Les protocoles et les expériences sur les animaux ont été approuvés et menés sous la supervision du Comité institutionnel de soin et d’utilisation des animaux de l’Université du Texas à Dallas (IACUC #21-03). Des souris C57BL6, y compris des nouveau-nés au jour 1 postnatal (P1) et des adultes de 8 semaines, ont été utilisées dans cette étude. Aucune différence n’a été observée entre les mâles et les femelles. L’ensemble de l’acquisition des données et du post-traitement des images a été réalisé à l’aide de logiciels open source ou de plateformes disposant de licences de recherche ou d’enseignement. Les ressources sont disponibles auprès des auteurs sur demande raisonnable.
1. Préparation de l’échantillon et nettoyage des tissus (6 à 10 jours)
- Préparez toutes les solutions chimiques de la table des matériaux requises pour la méthode d’élimination des tissus avant d’amorcer la procédure d’élimination des tissus.
REMARQUE : Effectuez toutes les procédures dans une hotte tout en portant un équipement de protection individuelle approprié, en particulier lors de la manipulation de produits chimiques toxiques. - Euthanasier les animaux contenant du CO2 en les plaçant dans une chambre de CO2 aux points temporels souhaités, tels que P1 et 8 semaines, pour le prélèvement du cœur entier et immerger le cœur fraîchement isolé à température ambiante (RT) dans 0,2 M KCl pour l’arrêter en diastole16.
- Fixez le cœur en l’immergeant dans du paraformaldéhyde à 4 % (PFA) dans une solution saline tamponnée 1x phosphate (PBS) avec une légère agitation sur un rocker pendant la nuit pour préserver l’anatomie cardiaque à 4 °C, voir tableau 1.
ATTENTION : Le PFA est un produit chimique toxique et doit être effectué sous une hotte. - Lavez le cœur avec 1x PBS à RT pendant 30 min, 3x.
REMARQUE : Facultatif : Intégrez le cœur dans du gel d’agarose pour créer un assemblage de cœur pour le système LSM personnalisé à l’étape 2, comme décrit ci-dessous.- Préparez la solution d’agarose à 1 % en dissolvant l’agarose dans du PBS et chauffez le mélange au micro-ondes jusqu’à dissolution complète. Versez la solution dans un moule pour créer une couche inférieure jusqu’à ce que la solution refroidisse à 40 °C.
- Placez le cœur dans le moule et remplissez le moule de gel d’agarose jusqu’à ce qu’il se solidifie. Éliminez toutes les bulles dans le gel d’agarose pour minimiser les artefacts dans l’imagerie.
REMARQUE : L’intégration de l’échantillon réduit le risque de dommages potentiels à l’anatomie cardiaque lors du montage.
ATTENTION : Utilisez de l’agarose à bas point de fusion et évitez de placer le cœur directement dans de l’agarose chauffée pour atténuer le risque d’exposer l’échantillon à des températures élevées.
- Déshydrater le cœur à l’aide de gradients de méthanol mélangé à de l’eau désionisée (figure 1B), en utilisant des concentrations séquentielles de 20 %, 40 %, 60 %, 80 % et 100 %, comme indiqué dans le tableau 1. Répétez l’opération avec du méthanol frais à 100 % 1x pour assurer une déshydratation complète. Déshydrater les cœurs de souris néonatales pendant 1 h dans chaque mélange de méthanol en secouant. Ajustez le temps d’incubation à 2 h pour les cœurs de souris et de rats de 8 semaines.
ATTENTION : Le méthanol est un produit chimique volatil, irritant et inflammable. - Remplacez et refroidissez le cœur avec du méthanol frais à 100 % pendant 10 min à 4 °C.
- Remplacer la solution et blanchir le cœur avec du peroxyde d’hydrogène à 5 % (H2O2) dans du méthanol en utilisant un rapport volumique de 1:5 (30 % H2O2 à 100 % méthanol) pendant une nuit à 4 °C pour éliminer les pigments.
REMARQUE : L’élimination des pigments permet de minimiser l’absorption des photons, ce qui améliore la qualité de l’image avec une réduction des artefacts. - Remplacer la solution et incuber le cœur dans du méthanol à 100% pendant 1 h à RT.
- Délipider le cœur avec la solution contenant 66 % de dichlorométhane (DCM) et 33 % de méthanol pendant 3 h en agitant. Assurez-vous que le cœur coule au fond de la fiole à la fin de cette étape. Sinon, renouveler la solution (66 % DCM / 33 % méthanol) et prolonger le temps d’incubation (par exemple, 1 heure de plus) pour obtenir une délipidation complète. Transférez rapidement le cœur dans une solution fraîche pour éviter la dessiccation, car le DCM est très volatil.
ATTENTION : Utilisez du polypropylène ou de la verrerie pour l’expérience, car le DCM n’est pas compatible avec le polystyrène. - Utilisez de l’éther dibenzylique (DBE) pour l’adaptation de l’indice de réfraction (RI = 1,56). Pour les cœurs de souris néonatales, le processus d’appariement de l’indice de réfraction prend 2 jours, tandis que pour les cœurs de souris de 8 semaines, il prend 5 jours avec de légères secousses. Remplacez-le par du DBE frais et prolongez le temps d’incubation jusqu’à ce que le cœur atteigne une transparence complète.
ATTENTION : Le DBE est dangereux. Évitez tout contact avec la peau.
2. Montage de l’échantillon (1 jour)
REMARQUE : Dans le cas où un système LSM commercial est utilisé, suivez son protocole spécifique fourni par la société pour réparer le cœur et sautez les étapes 2.1 à 2.9.
- Personnaliser une chambre et un support résistants à la solution d’adaptation de l’indice de réfraction (par exemple, DBE dans l’approche iDISCO adaptée) à l’aide d’une imprimante 3D et de matériaux Onyx (Figure 2A)16.
- Fixez un aimant au fond de la chambre pour faciliter une connexion rapide et une localisation précise sous le système LSM (Figure 2B).
- Fixez des verres de protection de 25 mm x 50 mm x 0,17 mm à la chambre à l’aide d’une colle silicone transparente et vérifiez l’intégrité de l’étanchéité de la chambre en la remplissant de DBE et en vérifiant les fuites après 1 jour.
- Personnalisez un support de pince à l’aide d’une imprimante 3D et fixez un aimant pour monter l’ensemble cœur à l’intérieur de la chambre (Figure 2C) ou insérez une aiguille dans le gel d’agarose pour monter l’ensemble cœur.
- Réaliser le développement d’un système LSM interne à l’aide d’une lentille cylindrique et de lasers à semi-conducteurs à diode à ondes continues (DPSS), comme indiqué précédemment16,22. Utilisez la longueur d’onde d’excitation de 532 nm pour capturer l’autofluorescence du myocarde (Figure 2D).
- Montez la chambre sur une platine à 6 dimensions et localisez la position optimale où la chambre est perpendiculaire à la direction de propagation du laser.
- À l’aide d’un support de pince magnétique, positionnez l’échantillon au milieu de la chambre et connectez le support à une platine motorisée en 4 dimensions (X, Y, Z et lacet ; Figure 2D).
- Immergez l’ensemble cardiaque dans la chambre remplie de DBE à l’aide de la platine motorisée et déterminez la portée de balayage le long de l’axe de détection (direction Z).
- Déterminez la vitesse de balayage (Vz) du moteur le long de la direction Z en fonction de la taille du pas (S) et du temps d’acquisition d’une image (T) à l’aide de l’équation suivante :
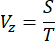
REMARQUE : La relation entre la taille du pas et la résolution axiale du système d’imagerie est conforme au théorème d’échantillonnage de Nyquist-Shannon. Par exemple, la taille du pas a été fixée à 1 μm, étant donné que la résolution axiale était de 2,91 μm, comme indiqué dans le rapport précédent16. - Pour vérifier la résolution spatiale et minimiser les problèmes d’opacité potentiels à différentes profondeurs, mesurez la fonction d’étalement ponctuel (PSF) du système en imageant des billes fluorescentes d’un diamètre de 0,53 μm diluées dans un gel d’agarose à faible point de fusion à 1 % avec une concentration de 1:1,5 x 105. Calculez la PSF sur l’ensemble de l’échantillon en mesurant la largeur totale à mi-hauteur (FWHM)16.
3. Assemblage d’images (4-8 h)
- Calculez le nombre de tuiles nécessaires pour couvrir l’ensemble du cœur de la souris, en tenant compte du champ de vision limité. Pour chaque tuile, les dimensions correspondent au FOV du système LSM. Par exemple, un cœur de souris nouveau-né d’une section transversale de 3 x 3,6 mm2 nécessite 2 x 2 tuiles pour être couvert dans le LSM personnalisé (figure 3A). En revanche, un cœur de souris de 8 semaines mesurant 5 x 8 mm2 nécessite 3 x 4 tuiles pour couvrir toute la structure cardiaque (figure 3B).
REMARQUE : L’assemblage d’images est nécessaire si le LSM conventionnel a un FOV limité pour l’imagerie de l’ensemble du cœur. - Capturez des images du cœur à partir de la tuile 1, c’est-à-dire la tuile du coin supérieur gauche du cœur (Figure 3A-B).
- Capturez séquentiellement des images des tuiles restantes avec un chevauchement de 10 % entre les tuiles consécutives jusqu’à ce que tout le cœur soit couvert, comme illustré à la figure 3C.
- Téléchargez et installez le logiciel open-source Fiji23 . Téléchargez et installez le plugin Fiji, BigStitcher24.
- Accédez au menu Aide, sélectionnez Mettre à jour, Gérer le site de mise à jour, puis choisissez BigStitcher. Ensuite, cliquez sur Fermer, puis sur Appliquer les modifications. Une fois le téléchargement du plugin terminé, redémarrez le logiciel Fidji.
- Ouvrez le plug-in BigStitcher et importez toutes les tuiles nécessaires dans le répertoire du fichier image. Enregistrez le jeu de données en tant que fichier HDF5.
- Organisez les tuiles en choisissant Déplacer la tuile selon la grille régulière, sélectionnez le motif utilisé pour déplacer le cœur et sélectionnez un chevauchement de 10 % entre chaque tuile. Brodez les carreaux à l’aide de l’option Assistant d’assemblage.
- Exportez les données assemblées à l’aide d’Image Fusion pour générer un fichier tiff.
4. Déconvolution multivue (5 jours)
- Utilisez des billes fluorescentes pour l’enregistrement d’images de reconstruction multivues le long du cœur (Figure 3D).
- Préparez la résine25 en mélangeant de l’éther diglycidylique de bisphénol-A (D.E.R. 332), de l’isophorone diamine, de la 5-amino-1,3,3-triméthylcyclohexaneméthylamine (IPDA) et de l’éther diglycidylique de polypropylène glycol (D.E.R 736) à un rapport de volume de 4:1:1.
- Centrifuger 10 μL de billes de fluorescence à 161 x g pendant 5 min et ajouter 20 μL de méthanol pour remplacer la solution de stockage des billes. Répétez l’opération 3 fois.
- Préparez une solution de 10 mL avec une concentration de billes de 1:1 x 105 en mélangeant la solution de billes à base de méthanol dans la résine préparée à l’étape 4.1.1.
- Dégazez la solution dans une chambre à vide pendant 3 h pour éliminer les poches d’air et les bulles.
- Versez la solution dans un moule en silicone ou une paille en plastique et attendez 2 à 3 jours jusqu’à ce qu’elle se solidifie. Après 1 jour avant que la résine ne devienne solide, placez le tube de verre à l’intérieur du moule et attendez que le tube se fixe à la résine. Retirez la résine du moule à l’aide du tube en verre (Figure 3D).
- Placez le cœur dans le tube de verre, remplissez-le de DBE et montez le tube de verre dans la chambre remplie de DBE.
- Capturez des images séquentielles du cœur de la souris avec les perles de la section éclairée en balayant l’ensemble du cœur sur l’axe de détection (axe Z ; Figures 3E-F).
REMARQUE : Implémentez l’assemblage d’images à l’étape 3 pour générer des piles individuelles d’images si la taille du cœur dépasse le FOV. - Faites pivoter le cœur de la souris de 60° le long de l’axe Y pour capturer les piles suivantes sous plusieurs angles, c’est-à-dire 0°, 60°, 120°, 180°, 240° et 300° (Figure 3E-F).
- Ouvrez le plug-in BigStitcher et importez toutes les images via le répertoire du fichier image. Enregistrez le jeu de données en tant que fichier HDF5.
- Choisissez Détecter les points d’intérêt et sélectionnez manuellement les perles d’intérêt pour l’enregistrement.
- Sélectionnez Modèle de transformation affine pour l’enregistrement. Choisissez Fonction d’étalement de points et attribuez PSF à toutes les vues.
- Cliquez sur Multiview Deconvolution et choisissez le type d’itération Efficient Bayesian. Définissez le nombre d’itérations sur 10 et exécutez-lesur 26.
- Cliquez sur Image Fusion pour exporter un fichier tiff .
REMARQUE : Des informations plus détaillées sur la déconvolution multivue peuvent être trouvées dans d’autres rapports ou protocoles 24,27,28.
5. Quincaillerie du système de feuille d’éclairage à balayage axial (1 jour)
- Installez une lentille électrique accordable (ETL) sur le plan de Fourier de la lentille cylindrique (CL)27 (Figure 2D) pour balayer le faisceau laser axialement14. Assurez-vous que l’interface liquide dans l’ETL est horizontale pour éviter la distorsion du front d’onde causée par la gravité. Alignez l’ETL avec précision pour minimiser le décentrement du faisceau et les aberrations optiques d’ordre supérieur30.
- Installez une carte DAQ sur le poste de travail et connectez un câble BNC à la carte DAQ pour synchroniser l’exposition de la caméra et le balayage ETL.
- Ouvrez le boîtier du pilote ETL et retirez le capot (Figure 4).
- Soudez le fil de signal du câble BNC à la broche Analog in B, comme indiqué au bas de la carte PCB (Figure 4).
- Soudez le fil de terre du câble BNC à la broche GND ; connectez ensuite l’autre extrémité du câble BNC à l’AO (sortie analogique) de la carte DAQ (Figure 4).
- Branchez le pilote ETL sur le port USB de la station de travail, connectez-le à l’ETL à l’aide d’un câble Hirose et installez le logiciel Lens Driver Controller.
- Changez le mode de fonctionnement dans le logiciel Lens Driver Controller Software sur Analog pour contrôler l’ETL avec un déclencheur généré à partir de la carte DAQ.
- Dans le logiciel de la caméra sCMOS, basculez le mode de capture sur Externe (feuille de lumière). Assurez-vous que la direction de balayage des pixels actifs est perpendiculaire à la direction de balayage laser pour faciliter la synchronisation.
- Connectez le port de déclenchement externe situé à l’arrière de la caméra sCMOS à l’AO de la carte DAQ à l’aide d’un câble BNC (Figure 4).
6. Synchronisation du système de feuille lumineuse à balayage axial (7 jours)
- Définir le temps d’exposition en fonction du rapport signal/bruit de fond (SBR) acceptable qui, pour l’étude proposée, est d’au moins 10. Augmentez le temps d’exposition si le SBR est inférieur à 10.
REMARQUE : Le temps d’exposition allant de 10 à 50 ms fournit un SBR acceptable dans ce projet. - Définissez le temps d’acquisition d’une image (T) et l’intervalle de ligne (L) en fonction du temps d’exposition (E) de l’appareil photo et de la fréquence ETL (f) à l’aide des équations suivantes :
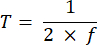
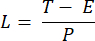
Où P indique le nombre de colonnes de pixels sur le capteur de l’appareil photo. Les paramètres représentatifs de la référence sont respectivement f = 1 Hz, P = 2048, E = 10 ms et L = 243 μs. - Dans le programme LabVIEW, Trigger Generator.vi génère des déclencheurs carrés et triangulaires pour la synchronisation (Figure 5).
REMARQUE : Le programme LabVIEW personnalisé est disponible auprès des auteurs sur demande raisonnable. - Montez des billes fluorescentes diluées dans du geld’agarose 16 à 1 % à faible point de fusion avec une concentration de 1:1 x 103 pour localiser la position de la taille du faisceau dans l’image (figure 6A).
- Dans le programme, identifiez les points de départ et d’arrivée du faisceau laser sous le FOV en modifiant la tension le long de la direction de balayage.
- Pour synchroniser la caméra sCMOS et l’ETL, balayez le faisceau laser le long de l’axe X et les pixels actifs le long de l’axe Y pour générer une image 2D (Figure 6B). Les perles plus lumineuses et plus nettes le long de la diagonale de l’image indiquent la synchronisation précise entre le sCMOS et l’ETL.
REMARQUE : La synchronisation de la vitesse de balayage de l’activation des pixels ETL et sCMOS est essentielle pour la qualité de l’image. Diverses erreurs asynchrones courantes sont résumées à la figure 6C-F. - Après avoir déterminé les paramètres de synchronisation optimaux pour la synchronisation, faites pivoter la caméra sCMOS de 90° autour de l’axe Z pour aligner la direction du pixel actif avec la direction du balayage laser.
- Répéter l’étape 2.10 et mesurer la FWHM de la PSF. Utiliser la même méthode que celle indiquée dans le rapport précédent16, avec les résolutions latérales et axiales moyennes de l’ASLM à 2,18 μm et 2,88 μm, respectivement (figure 7).
REMARQUE : Un éclairage et une résolution uniformes sur l’ensemble du champ de vision sont possibles dans des conditions idéales (Figure 7A-B). Le fonctionnement asynchrone de l’ETL avec la caméra sCMOS, ainsi qu’un désalignement de l’éclairage et de la détection, peuvent entraîner une résolution spatiale non uniforme (Figure 7C-D). - Pour l’imagerie cardiaque, après avoir aligné le système et déterminé les paramètres optimaux pour la synchronisation du système, passez aux étapes 2.6 à 2.9.
Résultats
Il a été démontré que la LSM favorise les études cardiaques 31,32,33,34,35,36,37 en raison du risque minimal de photodommages, de la haute résolution spatiale et du sectionnement optique par rapport à d’autres méthodes d’imagerie optique telles que les techniques de balayage en fond clair et en point <...
Discussion
Les progrès de l’imagerie, du calcul et des méthodes d’élimination des tissus ont fourni une occasion sans précédent d’étudier en profondeur la structure et la fonction cardiaques. Cela présente un grand potentiel pour approfondir notre compréhension de la morphogenèse et de la pathogenèse cardiaques à l’aide d’un modèle de cœur de rongeur intact. Contrairement aux études in vivo du cœur de poisson-zèbre utilisant une approche similaire 40,41,42,43, l’intégration de techniques LSM av...
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à divulguer.
Remerciements
Nous exprimons notre gratitude au groupe du Dr Eric Olson du UT Southwestern Medical Center pour avoir généreusement partagé les modèles animaux. Nous apprécions tous les commentaires constructifs fournis par les membres de D-incubator à UT Dallas. Ce travail a été soutenu par le programme NIH R00HL148493 (Y.D.), R01HL162635 (Y.D.) et UT Dallas STARS (Y.D.).
matériels
| Name | Company | Catalog Number | Comments |
| 1% Agarose | |||
| Low melting point agarose | Thermo Fisher | 16520050 | |
| Deionized water | - | - | |
| Chemicals for tissue clearing | |||
| 5-Amino-1,3,3-trimethylcyclohexanemethylamine, mixture of cis and trans | Sigma-Aldrich | 118184 | |
| D.E.R.™ 332 | Sigma-Aldrich | 31185 | |
| D.E.R.™ 736 | Sigma-Aldrich | 31191 | |
| Dibenzyl ether (DBE) | Sigma-Aldrich | 33630 | |
| Dichloromethane (DCM) | Sigma-Aldrich | 270997 | |
| Fluorescent beads | Spherotech | FP-0556-2 | |
| Hydrogen peroxide (H2O2) | Sigma-Aldrich | 216736 | |
| Methanol | Sigma-Aldrich | 439193 | |
| Paraformaldehyde (PFA) | Thermo Fisher | 47392 | |
| Phosphate Buffered Saline (PBS) | Sigma-Aldrich | 79383 | |
| Potassium Chloride (KCl) | Sigma-Aldrich | P3911 | |
| Software and algorithms | |||
| Amira | Thermo Fisher Scientific | 2021.2 | |
| BigStitcher | Hörl et al.22 | ||
| Fiji-ImageJ | Schindelin et al.20 | 1.54f | |
| HCImage Live | Hamamatsu Photonics | 4.6.1.2 | |
| LabVIEW | National Instruments Corporation | 2017 SP1 | |
| Key components of the customized light-sheet system | |||
| 0.63 - 6.3X Zoom body | Olympus | MVX-ZB10 | |
| 10X Illumination objective | Nikon | MRH00105 | |
| 1X detection objective | Olympus | MV PLAPO 1X/0.25 | |
| 473nm DPSS Laser | Laserglow Technologies | LRS-0473-PFM-00100-05 | |
| 532nm DPSS laser | Laserglow Technologies | LRS-0532-PFM-00100-05 | |
| 589 nm DPSS laser | Laserglow Technologies | LRS-0589-GFF-00100-05 | |
| BNC connector | National Instrument | BNC-2110 | |
| Cylindrical lens | Thorlabs | ACY254-050-A | |
| DC-Motor Controller, 4 axes | Physik Instrumente | C-884.4DC | |
| ETL | Optotune | EL-16-40-TC-VIS-5D-1-C | |
| ETL Cable | Optotune | CAB-6-300 | |
| ETL Lens Driver | Optotune | EL-E-4i | |
| Filter | Chroma | ET525/30 | |
| Filter | Chroma | ET585-40 | |
| Filter | Chroma | ET645-75 | |
| Filter wheel | Shutter Instrument | LAMBDA 10-B | |
| Motorized translation stage | Physik Instrumente | L-406.20DG10 | |
| Motorized translation stage | Physik Instrumente | L-406.40DG10 | |
| Motorized translation stage | Physik Instrumente | M-403.4PD | |
| NI multifunction I/O | National Instrument | PCIe-6363 | |
| sCMOS camera | Hamamatsu | C13440-20CU | |
| Stepper motor | Pololu | 1474 | |
| Tube lens | Olympus | MVX-TLU |
Références
- Sadek, H., Olson, E. N. Toward the goal of human heart regeneration. Cell Stem Cell. 26, 7-16 (2020).
- Porrello, E. R., et al. Transient regenerative potential of the neonatal mouse heart. Science. 331, 1078-1080 (2011).
- Stelzer, E. H. K. K., et al. Light sheet fluorescence microscopy. Nat Rev Methods Prim. 1, 73 (2021).
- Girkin, J. M., Carvalho, M. T. The light-sheet microscopy revolution. J Opt. 20, 053002 (2018).
- Power, R. M., Huisken, J. A guide to light-sheet fluorescence microscopy for multiscale imaging. Nat Methods. 14, 360-373 (2017).
- Ding, Y., et al. Multiscale light-sheet for rapid imaging of cardiopulmonary system. JCI Insight. 3, e121396 (2018).
- Ding, Y., et al. Light-sheet fluorescence imaging to localize cardiac lineage and protein distribution. Sci Rep. 7, 42209 (2017).
- Fei, P., et al. Cardiac light-sheet fluorescent microscopy for multi-scale and rapid imaging of architecture and function. Sci Rep. 6, 1-12 (2016).
- Richardson, D. S., Lichtman, J. W. Clarifying tissue clearing. Cell. 162, 246-257 (2015).
- Stelzer, E. H. K., Huisken, J., Swoger, J., Greger, K., Verveer, P. Multi-view image fusion improves resolution in three-dimensional microscopy. Opt Express. 15 (13), 8029-8042 (2007).
- Dean, K. M., Roudot, P., Welf, E. S., Danuser, G., Fiolka, R. Deconvolution-free subcellular imaging with axially swept light sheet microscopy. Biophys J. 108, 2807-2815 (2015).
- Dean, K. M., et al. Isotropic imaging across spatial scales with axially swept light-sheet microscopy. Nat Protoc. 17, 2025-2053 (2022).
- Hedde, P. N., Gratton, E. Selective plane illumination microscopy with a light sheet of uniform thickness formed by an electrically tunable lens. Microsc Res Tech. 81, 924 (2018).
- Voigt, F. F., et al. The mesoSPIM initiative: open-source light-sheet microscopes for imaging cleared tissue. Nat Meth. 16, 1105-1108 (2019).
- Giardini, F., et al. Mesoscopic optical imaging of whole mouse heart. J Vis Exp. (176), e62795 (2021).
- Sodimu, O., et al. Light sheet imaging and interactive analysis of the cardiac structure in neonatal mice. J Biophotonics. 16, e202200278 (2023).
- Ariel, P. A beginner's guide to tissue clearing. Int J Biochem Cell Biol. 84, 35-39 (2017).
- Richardson, D. S., et al. Tissue clearing. Nat Rev Methods Prim. 1, 1-24 (2021).
- Ueda, H. R., et al. Tissue clearing and its applications in neuroscience. Nat Rev Neurosci. 21, 61-79 (2020).
- Renier, N., et al. iDISCO: a simple, rapid method to immunolabel large tissue samples for volume imaging. Cell. 159, 896-910 (2014).
- Kirchner, K. N., et al. A hydrophobic tissue clearing method for rat brain tissue. J Vis Exp. (166), e61821 (2020).
- Ding, Y., et al. Light-sheet fluorescence microscopy for the study of the murine heart. J Vis Exp. (139), e57769 (2018).
- Schindelin, J., Rueden, C. T., Hiner, M. C., Eliceiri, K. W. The ImageJ ecosystem: An open platform for biomedical image analysis. Mol Reprod Dev. 82, 518-529 (2015).
- Hörl, D., et al. BigStitcher: reconstructing high-resolution image datasets of cleared and expanded samples. Nat Methods. 16, 870-874 (2019).
- Becker, K., et al. Reduction of Photo Bleaching and Long Term Archiving of Chemically Cleared GFP-Expressing Mouse Brains. PLoS One. 9, e114149 (2014).
- Preibisch, S., et al. Efficient Bayesian-based multiview deconvolution. Nat. Methods. 11, 645-648 (2014).
- Guo, M., et al. Rapid image deconvolution and multiview fusion for optical microscopy. Nat. Biotechnol. 38, 1337-1346 (2020).
- Tomer, R., Khairy, K., Amat, F., Keller, P. J. Quantitative high-speed imaging of entire developing embryos with simultaneous multiview light-sheet microscopy. Nat Methods. 9, 755-763 (2012).
- Fahrbach, F. O., Voigt, F. F., Schmid, B., Helmchen, F., Huisken, J. Rapid 3D light-sheet microscopy with a tunable lens. Opt Express. 21, 21010-21026 (2013).
- Liu, Y., Rollins, A. M., Jenkins, M. W. CompassLSM: axially swept light-sheet microscopy made simple. Biomed Opt Express. 12, 6571-6589 (2021).
- Kolesová, H., Olejníčková, V., Kvasilová, A., Gregorovičová, M., Sedmera, D. Tissue clearing and imaging methods for cardiovascular development. iScience. 24 (4), 102387 (2021).
- Sands, G. B., et al. It's clearly the heart! Optical transparency, cardiac tissue imaging, and computer modelling. Prog Biophys Mol Biol. 168, 18-32 (2022).
- Wilson, A. J., Sands, G. B., LeGrice, I. J., Young, A. A., Ennis, D. B. Muscle mechanics and ventricular function: Myocardial mesostructure and mesofunction. Am J Physiol - Hear Circ Physiol. 323, H257 (2022).
- Lee, S. E., et al. Three-dimensional cardiomyocytes structure revealed by diffusion tensor imaging and its validation using a tissue-clearing technique. Sci. Reports. 8, 1-11 (2018).
- Sereti, K. I., et al. Analysis of cardiomyocyte clonal expansion during mouse heart development and injury. Nat Commun. 9, 754 (2018).
- Olianti, C., et al. 3D imaging and morphometry of the heart capillary system in spontaneously hypertensive rats and normotensive controls. Sci. Reports. 10, 1-9 (2020).
- Olianti, C., et al. Optical clearing in cardiac imaging: A comparative study. Prog Biophys Mol Biol. 168, 10-17 (2022).
- Baek, K. I., et al. Advanced microscopy to elucidate cardiovascular injury and regeneration: 4D light-sheet imaging. Prog Biophys Mol Biol. 138, 105-115 (2018).
- Merz, S. F., et al. Contemporaneous 3D characterization of acute and chronic myocardial I/R injury and response. Nat Commun. 10, 1-14 (2019).
- Zhang, X., et al. 4D Light-sheet imaging and interactive analysis of cardiac contractility in zebrafish larvae. APL Bioeng. 7, 26112 (2023).
- Lee, J., et al. 4-Dimensional light-sheet microscopy to elucidate shear stress modulation of cardiac trabeculation. J Clin Invest. 126, 1679-1690 (2016).
- Zhang, X., Alexander, R. V., Yuan, J., Ding, Y. Computational Analysis of Cardiac Contractile Function. Curr Cardiol Rep. 24, 1983-1994 (2022).
- Zhang, X., et al. 4D Light-sheet Imaging of Zebrafish Cardiac Contraction. J Vis Exp. (203), e66263 (2024).
- Huisken, J., Swoger, J., Del Bene, F., Wittbrodt, J., Stelzer, E. H. K. K. Optical sectioning deep inside live embryos by selective plane illumination microscopy. Science. 305, 1007-1009 (2004).
- Molbay, M., Kolabas, Z. I., Todorov, M. I., Ohn, T., Ertürk, A. A guidebook for DISCO tissue clearing. Mol Syst Biol. 17, 9807 (2021).
- Chi, J., Crane, A., Wu, Z., Cohen, P. Adipo-clear: a tissue clearing method for three-dimensional imaging of adipose tissue. J Vis Exp. (137), e58271 (2018).
- Wan, Y., McDole, K., Keller, P. J. Light-sheet microscopy and its potential for understanding developmental processes. Ann Rev Cell Dev Biol. 35, 655-681 (2019).
- Yuan, J., et al. Extended reality for biomedicine. Nat Rev Methods Prim. 3, 1-1 (2023).
- Ding, Y., et al. Saak transform-based machine learning for light-sheet imaging of cardiac trabeculation. IEEE Trans Biomed Eng. 68, 225-235 (2020).
- Buffinton, C. M., Benjamin, A. K., Firment, A. N., Moon, A. M. Myocardial wall stiffening in a mouse model of persistent truncus arteriosus. PLoS One. 12 (9), e0184678 (2017).
- Trincot, C. E., et al. Adrenomedullin induces cardiac lymphangiogenesis after myocardial infarction and regulates cardiac edema via Cx43. Circ Res. 124, 101 (2019).
- Yokoyama, T., et al. Quantification of sympathetic hyperinnervation and denervation after myocardial infarction by three-dimensional assessment of the cardiac sympathetic network in cleared transparent murine hearts. PLoS One. 12, e0182072 (2017).
- Coram, R. J., et al. Muscleblind-like 1 is required for normal heart valve development in vivo. BMC Dev Biol. 15, 36 (2015).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon