É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
Imagem de folha de luz para revelar estrutura cardíaca em corações de roedores
Neste Artigo
Resumo
O protocolo utiliza microscopia avançada de folha de luz junto com métodos adaptados de limpeza de tecidos para investigar estruturas cardíacas intrincadas em corações de roedores, com grande potencial para a compreensão da morfogênese e remodelação cardíaca.
Resumo
A microscopia de folha de luz (LSM) desempenha um papel fundamental na compreensão da intrincada estrutura tridimensional (3D) do coração, fornecendo informações cruciais sobre a fisiologia cardíaca fundamental e as respostas patológicas. Por meio deste, investigamos o desenvolvimento e a implementação da técnica LSM para elucidar a microarquitetura do coração em modelos de camundongos. A metodologia integra um sistema LSM personalizado com técnicas de limpeza de tecidos, mitigando a dispersão de luz dentro dos tecidos cardíacos para imagens volumétricas. A combinação de LSM convencional com costura de imagem e abordagens de deconvolução multiview permite a captura de todo o coração. Para abordar o trade-off inerente entre resolução axial e campo de visão (FOV), introduzimos ainda um método de microscopia de folha de luz varrida axialmente (ASLM) para minimizar a luz fora de foco e iluminar uniformemente o coração em toda a direção de propagação. Enquanto isso, os métodos de limpeza de tecidos, como o iDISCO, aumentam a penetração da luz, facilitando a visualização de estruturas profundas e garantindo um exame abrangente do miocárdio em todo o coração. A combinação dos métodos propostos de LSM e limpeza de tecidos apresenta uma plataforma promissora para pesquisadores na resolução de estruturas cardíacas em corações de roedores, com grande potencial para a compreensão da morfogênese e remodelação cardíaca.
Introdução
A insuficiência cardíaca continua sendo a principal causa de mortalidade em todo o mundo, principalmente devido à falta de capacidade regenerativa dos cardiomiócitos maduros1. A intrincada arquitetura do coração desempenha um papel crucial em sua função e fornece informações sobre os processos de desenvolvimento. Uma compreensão profunda da estrutura cardíaca é essencial para elucidar os processos fundamentais da morfogênese e remodelamento cardíaco em resposta ao infarto do miocárdio. Progressos recentes demonstraram que camundongos recém-nascidos podem restaurar a função cardíaca após lesão, enquanto camundongos adultos não têm essa capacidade regenerativa2. Isso estabelece uma base para investigar pistas associadas a anormalidades estruturais e funcionais em modelos de camundongos. Os métodos tradicionais de imagem, como a microscopia confocal, têm limitações técnicas, incluindo profundidade de penetração restrita, esquema de varredura de ponto lento e danos fotográficos causados pela exposição prolongada à luz laser. Isso dificulta a aquisição de imagens tridimensionais (3D) abrangentes do coração intacto. Nesse contexto, a microscopia de folha de luz (LSM) surge como uma solução poderosa, oferecendo as vantagens da imagem de alta velocidade, danos fotográficos reduzidos e recursos excepcionais de seccionamento óptico 3,4,5. As características únicas do LSM o posicionam como um método promissor para superar as limitações das técnicas convencionais, fornecendo insights sem precedentes sobre os processos de desenvolvimento e remodelação cardíaca 6,7,8.
Neste protocolo, introduzimos uma estratégia de imagem que combina LSM avançado com abordagens adaptadas de limpeza de tecidos9, permitindo a imagem de corações inteiros de camundongos sem a necessidade de marcação específica e seccionamento mecânico. Propomos ainda que a imagem LSM convencional pode ser aprimorada por meio de técnicas de deconvolução multiview10 ou microscopia de folha de luz varrida axialmente (ASLM) 11,12,13,14,15 para melhorar a resolução axial. Além disso, a integração da costura de imagens com qualquer um desses métodos pode efetivamente superar o trade-off entre resolução espacial e campo de visão (FOV), avançando assim a imagem de corações de camundongos adultos. A incorporação de inúmeras abordagens de limpeza de tecidos, incluindo métodos hidrofóbicos, hidrofílicos e baseados em hidrogel, permite uma penetração de luz mais profunda para capturar a morfologia de todo o coração 16,17,18,19.
Embora vários métodos de limpeza sejam compatíveis com os sistemas LSM atuais, o objetivo é minimizar a dispersão de fótons e aumentar a penetração da luz nos tecidos, como o coração, substituindo os lipídios por um meio que corresponda ao seu índice de refração. O iDISCO foi escolhido como o representante 20,21 e adaptado para imagens de autofluorescência neste protocolo devido ao seu rápido processamento e alta transparência (Figura 1A). Coletivamente, a integração da abordagem avançada do LSM com técnicas de limpeza de tecidos oferece uma estrutura promissora para desvendar a intrincada anatomia cardíaca em corações de roedores, com potencial significativo para avançar nossa compreensão da morfogênese e patogênese cardíaca.
Protocolo
Protocolos e experimentos com animais foram aprovados e conduzidos sob a supervisão do Comitê Institucional de Cuidados e Uso de Animais da Universidade do Texas em Dallas (IACUC # 21-03). Camundongos C57BL6, incluindo neonatos no dia 1 pós-natal (P1) e adultos com 8 semanas de idade, foram usados neste estudo. Não foi observada diferença entre homens e mulheres. Toda a aquisição de dados e pós-processamento de imagens foram realizados por meio de software ou plataformas de código aberto com licenças de pesquisa ou educacionais. Os recursos estão disponíveis com os autores mediante solicitação razoável.
1. Preparação da amostra e limpeza do tecido (6 - 10 dias)
- Prepare todas as soluções químicas na Tabela de Materiais necessários para o método de limpeza de tecidos antes de iniciar o procedimento de limpeza de tecidos.
NOTA: Execute todos os procedimentos em uma hotte usando equipamento de proteção individual adequado, especialmente ao manusear produtos químicos tóxicos. - Eutanasiar animais com CO2 colocando-os em uma câmara de CO2 nos pontos de tempo desejados, como P1 e 8 semanas, para coleta de coração inteiro e mergulhar o coração recém-isolado à temperatura ambiente (RT) em 0,2 M KCl para parar na diástole16.
- Fixe o coração imergindo em paraformaldeído a 4% (PFA) em solução salina tamponada com fosfato 1x (PBS) com agitação suave em um balancim durante a noite para preservar a anatomia cardíaca a 4 ° C, consulte a Tabela 1.
CUIDADO: O PFA é um produto químico tóxico e deve ser realizado sob um exaustor. - Lave o coração com 1x PBS em RT por 30 min, 3x.
NOTA: Opcional: Incorpore o coração em gel de agarose para criar um conjunto de coração para o sistema LSM personalizado na etapa 2, conforme descrito abaixo.- Prepare a solução de agarose a 1% dissolvendo a agarose em PBS e aqueça a mistura no microondas até a dissolução completa. Despeje a solução em um molde para criar uma camada inferior até que a solução esfrie até 40 °C.
- Coloque o coração na forma e encha a forma com gel de agarose até solidificar. Elimine quaisquer bolhas no gel de agarose para minimizar artefatos na imagem.
NOTA: A incorporação da amostra reduz o risco de danos potenciais à anatomia do coração durante a montagem.
CUIDADO: Utilize agarose de baixo ponto de fusão e evite colocar o coração diretamente na agarose aquecida para mitigar o risco de expor a amostra a temperaturas elevadas.
- Desidrate o coração usando gradientes de metanol misturado com água deionizada (Figura 1B), processando por meio de concentrações sequenciais de 20%, 40%, 60%, 80% e 100%, conforme descrito na Tabela 1. Repita com 100% de metanol fresco 1x para garantir a desidratação completa. Desidrate corações de camundongos neonatais por 1 h em cada mistura de metanol com agitação. Ajuste o tempo de incubação para 2 h para corações de camundongos e corações de ratos com 8 semanas de idade.
CUIDADO: O metanol é um produto químico volátil, irritante e inflamável. - Substitua e resfrie o coração por metanol 100% fresco por 10 min a 4 °C.
- Substitua a solução e branqueie o coração com peróxido de hidrogênio a 5% (H2O2) em metanol usando uma proporção de volume de 1:5 (30% H2O2 a 100% de metanol) durante a noite a 4 ° C para remover os pigmentos.
NOTA: A remoção de pigmentos permite minimizar a absorção de fótons, levando a uma melhor qualidade de imagem com artefatos reduzidos. - Substitua a solução e incube o coração em metanol 100% por 1 h em RT.
- Deslipidar o coração com a solução contendo 66% de diclorometano (DCM) e 33% de metanol por 3 h com agitação. Certifique-se de que o coração afunda no fundo do frasco até o final desta etapa. Caso contrário, renove a solução (66% DCM / 33% metanol) e estenda o tempo de incubação (por exemplo, mais 1 hora) para obter a deslipidação completa. Transfira o coração para uma solução nova rapidamente para evitar a dessecação, pois o DCM é altamente volátil.
CUIDADO: Use polipropileno ou vidraria para o experimento, pois o DCM não é compatível com o poliestireno. - Utilize éter dibenzílico (DBE) para correspondência do índice de refração (RI = 1,56). Para corações de camundongos neonatais, o processo de correspondência do índice de refração leva 2 dias, enquanto para corações de camundongos de 8 semanas, leva 5 dias com tremores leves. Substitua-o por DBE fresco e estenda o tempo de incubação até que o coração atinja total transparência.
CUIDADO: O DBE é perigoso. Evite qualquer contato com a pele.
2. Montagem da amostra (1 dia)
NOTA: Caso seja usado um sistema LSM comercial, siga seu protocolo específico fornecido pela empresa para consertar o coração e pule as etapas 2.1 - 2.9.
- Personalize uma câmara e um suporte resistentes à solução de correspondência de índice de refração (por exemplo, DBE na abordagem iDISCO adaptada) usando uma impressora 3D e materiais Onyx (Figura 2A)16.
- Fixe um ímã na parte inferior da câmara para facilitar a conexão rápida e a localização precisa no sistema LSM (Figura 2B).
- Prenda os vidros de cobertura de 25 mm x 50 mm x 0.17 mm na câmara usando cola de silicone transparente e verifique a integridade à prova d'água da câmara enchendo-a com DBE e verificando se há vazamentos após 1 dia.
- Personalize um clamp suporte usando uma impressora 3D e fixe um ímã para montar o conjunto do coração dentro da câmara (Figura 2C) ou insira uma agulha no gel de agarose para montar o conjunto do coração.
- Realizar o desenvolvimento de um sistema LSM interno usando uma lente cilíndrica e lasers de estado sólido bombeados por diodo de onda contínua (DPSS), conforme relatado anteriormente16,22. Use o comprimento de onda de excitação de 532 nm para capturar a autofluorescência do miocárdio (Figura 2D).
- Monte a câmara em um estágio de 6 dimensões e localize a posição ideal onde a câmara é perpendicular à direção de propagação do laser.
- Use um suporte de grampo magnético para posicionar a amostra no meio da câmara e conecte o suporte a um estágio motorizado de 4 dimensões (X, Y, Z e Yaw; Figura 2D).
- Mergulhe o conjunto do coração na câmara cheia de DBE usando o estágio motorizado e determine a faixa de varredura ao longo do eixo de detecção (direção Z).
- Determine a velocidade de varredura (Vz) do motor ao longo da direção Z com base no tamanho do passo (S) e no tempo de aquisição de uma imagem (T) usando a seguinte equação:
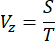
NOTA: A relação entre o tamanho do passo e a resolução axial do sistema de imagem segue o teorema de amostragem de Nyquist-Shannon. Por exemplo, o tamanho do passo foi fixado em 1 μm, uma vez que a resolução axial foi de 2,91 μm, conforme indicado no relatório anterior16. - Para verificar a resolução espacial e minimizar possíveis problemas de opacidade em diferentes profundidades, meça a função de propagação de pontos (PSF) do sistema por meio de imagens de esferas fluorescentes com diâmetro de 0,53 μm diluídas em gel de agarose de baixo ponto de fusão a 1% com concentração de 1:1,5 x 105. Calcule o PSF em toda a amostra medindo a largura total na metade máxima (FWHM)16.
3. Costura de imagem (4-8 h)
- Calcule o número de blocos necessários para cobrir todo o coração do rato, considerando o FOV limitado. Para cada bloco, as dimensões correspondem ao FOV do sistema LSM. Por exemplo, um coração de camundongo neonatal com uma medida transversal de 3 x 3,6 mm2 necessita de 2 x 2 ladrilhos para cobertura no LSM personalizado (Figura 3A). Em contraste, um coração de camundongo de 8 semanas medindo 5 x 8 mm2 requer 3 x 4 ladrilhos para cobrir toda a estrutura cardíaca (Figura 3B).
NOTA: A costura da imagem é necessária se o LSM convencional tiver FOV limitado para imagens de todo o coração. - Capture imagens do coração a partir do bloco 1, ou seja, o bloco do canto superior esquerdo do coração (Figura 3A-B).
- Capture sequencialmente imagens dos ladrilhos restantes com uma sobreposição de 10% entre ladrilhos consecutivos até que todo o coração seja coberto, conforme ilustrado na Figura 3C.
- Baixe e instale o software de código aberto Fiji23 . Baixe e instale o plugin Fiji, BigStitcher24.
- Navegue até o menu Ajuda, selecione Atualizar, escolha Gerenciar site de atualização e opte por BigStitcher. Em seguida, clique em Fechar, seguido de Aplicar alterações. Ao concluir o download do plug-in, reinicie o software Fiji.
- Abra o plug-in BigStitcher e importe todos os blocos necessários por meio do diretório do arquivo de imagem. Salve o conjunto de dados como um arquivo HDF5.
- Organize os blocos escolhendo Mover bloco por grade regular, selecione o padrão usado para mover o coração e selecione uma sobreposição de 10% entre cada bloco. Costure os ladrilhos usando a opção Assistente de costura.
- Exporte os dados compilados usando o Image Fusion para gerar um arquivo tiff.
4. Deconvolução Multiview (5 dias)
- Utilize esferas fluorescentes para registro de imagem da reconstrução multiview ao longo do coração (Figura 3D).
- Prepare a resina25 misturando éter diglicidílico de bisfenol-A (DER 332), isoforona diamina, 5-amino-1,3,3-trimetilciclohexanometilamina (IPDA) e éter diglicidílico de polipropilenoglicol (DER 736) em uma proporção de volume de 4:1:1.
- Centrifugue 10 μL de grânulos de fluorescência a 161 x g por 5 min e adicione 20 μL de metanol para substituir a solução de armazenamento dos grânulos. Repita 3x.
- Preparar uma solução de 10 ml com uma concentração de 1:1 x 105 grânulos misturando a solução de grânulos à base de metanol na resina preparada no passo 4.1.1.
- Desgaseifique a solução em uma câmara de vácuo por 3 h para remover bolsas de ar e bolhas.
- Despeje a solução em um molde de silicone ou canudo de plástico e aguarde de 2 a 3 dias até solidificar. Após 1 dia antes que a resina fique sólida, coloque o tubo de vidro dentro do molde e espere até que o tubo se prenda à resina. Remova a resina do molde com o tubo de vidro (Figura 3D).
- Coloque o coração no tubo de vidro, encha-o com DBE e monte o tubo de vidro na câmara cheia de DBE.
- Capture imagens sequenciais do coração do camundongo junto com as contas da seção iluminada digitalizando o conjunto do coração no eixo de detecção (eixo Z; Figura 3E-F).
NOTA: Implemente a costura de imagens na etapa 3 para gerar pilhas individuais de imagens se o tamanho do coração exceder o FOV. - Gire o coração do mouse 60° ao longo do eixo Y para capturar pilhas subsequentes de vários ângulos, ou seja, 0°, 60°, 120°, 180°, 240° e 300° (Figura 3E-F).
- Abra o plug-in BigStitcher e importe todas as imagens através do diretório do arquivo de imagem. Salve o conjunto de dados como um arquivo HDF5.
- Escolha Detectar pontos de interesse e selecione manualmente os grânulos de interesse para registro.
- Selecione Modelo de transformação afim para registro. Escolha Função de difusão de pontos e atribua PSF a todas as vistas.
- Clique em Multiview Deconvolution e escolha o tipo de iteração como Efficient Bayesian. Defina Número de iterações como 10 e execute-o26.
- Clique em Image Fusion para exportar um arquivo tiff .
NOTA: Informações mais detalhadas sobre a deconvolução multiview podem ser encontradas em outros relatórios ou protocolos 24,27,28.
5. Hardware do sistema de folha de luz varrido axialmente (1 dia)
- Instale uma lente elétrica sintonizável (ETL) no plano de Fourier da lente cilíndrica (CL)27 (Figura 2D) para escanear o feixe de laser axialmente14. Certifique-se de que a interface líquida no ETL esteja horizontal para evitar a distorção da frente de onda causada pela gravidade. Alinhe o ETL com precisão para minimizar o descentramento do feixe e as aberrações ópticas de ordem superior30.
- Instale uma placa DAQ na estação de trabalho e conecte um cabo BNC à placa DAQ para sincronizar a exposição da câmera e a varredura ETL.
- Abra a caixa do driver ETL e remova a tampa (Figura 4).
- Solde o fio de sinal do cabo BNC ao pino Analog in B, conforme identificado na parte inferior da placa PCB (Figura 4).
- Solde o fio terra do cabo BNC ao pino GND; depois disso, conecte a outra extremidade do cabo BNC à AO (Saída Analógica) na placa DAQ (Figura 4).
- Conecte o driver ETL à porta USB da estação de trabalho, conecte o driver ao ETL usando um cabo hirose e instale o software Lens Driver Controller.
- Altere o modo de operação no software do controlador do driver da lente para analógico para controlar o ETL com um gatilho gerado a partir da placa DAQ.
- No software da câmera sCMOS, alterne o modo de captura para Externo (folha de luz). Certifique-se de que a direção de varredura de pixel ativa seja perpendicular à direção de varredura a laser para auxiliar na sincronização.
- Conecte a porta de disparo externa na parte traseira da câmera sCMOS ao AO da placa DAQ usando um cabo BNC (Figura 4).
6. Sincronização do sistema de folha de luz varrida axialmente (7 dias)
- Defina o tempo de exposição com base na relação sinal-fundo (SBR) aceitável que, para o estudo proposto, é de pelo menos 10. Aumente o tempo de exposição se o SBR for inferior a 10.
NOTA: O tempo de exposição variando de 10 a 50 ms fornece um SBR aceitável neste projeto. - Defina o tempo de aquisição de uma imagem (T) e o intervalo de linha (L) com base no tempo de exposição (E) da câmera e na frequência ETL (f) usando as seguintes equações:
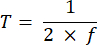
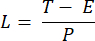
Onde P indica o número de colunas de pixels no sensor da câmera. Os parâmetros representativos para a referência são f = 1 Hz, P = 2048, E = 10 ms e L = 243 μs, respectivamente. - No programa LabVIEW, o Trigger Generator.vi gera triggers quadrados e triangulares para sincronização (Figura 5).
NOTA: O programa LabVIEW personalizado está disponível com os autores mediante solicitação razoável. - Monte grânulos fluorescentes diluídos em gel de agarose de baixo ponto de fusãoa 1% 16 com uma concentração de 1:1 x 103 para localizar a posição da cintura do feixe na imagem (Figura 6A).
- No programa, identifique os pontos inicial e final do feixe de laser sob o FOV alterando a tensão ao longo da direção de varredura.
- Para sincronizar a câmera sCMOS e o ETL, digitalize o feixe de laser ao longo do eixo X e os pixels ativos ao longo do eixo Y para gerar uma imagem 2D (Figura 6B). As contas mais brilhantes e nítidas ao longo da diagonal na imagem indicam a sincronização precisa entre o sCMOS e o ETL.
NOTA: A sincronização da velocidade de varredura da ativação de pixel ETL e sCMOS é crítica para a qualidade da imagem. Vários erros assíncronos comuns estão resumidos na Figura 6C-F. - Depois de determinar os parâmetros de sincronização ideais para sincronização, gire a câmera sCMOS 90° em torno do eixo Z para alinhar a direção do pixel ativo com a direção da varredura a laser.
- Repita a etapa 2.10 e meça o FWHM do PSF. Use o mesmo método indicado no relatório anterior16, com as resoluções laterais e axiais médias do ASLM como 2,18 μm e 2,88 μm, respectivamente (Figura 7).
NOTA: Iluminação e resolução uniformes em todo o FOV são viáveis em condições ideais (Figura 7A-B). A operação assíncrona do ETL com a câmera sCMOS, bem como o desalinhamento da iluminação e da detecção, podem resultar em resolução espacial não uniforme (Figura 7C-D). - Para imagens cardíacas, depois de alinhar o sistema e determinar os parâmetros ideais para sincronização do sistema, prossiga com as etapas 2.6 a 2.9.
Resultados
Foi demonstrado que o LSM promove estudos cardíacos 31,32,33,34,35,36,37 devido ao risco mínimo de dano fotográfico, alta resolução espacial e seccionamento óptico, em oposição a outros métodos de imagem óptica, como técnicas de campo claro e varredura pontual <...
Discussão
O avanço dos métodos de imagem, computação e limpeza de tecidos proporcionou uma oportunidade incomparável para investigar extensivamente a estrutura e a função cardíaca. Isso tem um grande potencial para aprofundar nossa compreensão da morfogênese e patogênese cardíaca usando um modelo de coração de roedor intacto. Em contraste com estudos in vivo de coração de peixe-zebra usando uma abordagem semelhante 40,41,42,43, a integração de técnicas avançadas de LSM e métodos de limpeza de tecidos ...
Divulgações
Os autores não têm conflito de interesses a divulgar.
Agradecimentos
Expressamos nossa gratidão ao grupo do Dr. Eric Olson no UT Southwestern Medical Center por compartilhar generosamente os modelos animais. Agradecemos todos os comentários construtivos fornecidos pelos membros da incubadora D na UT Dallas. Este trabalho foi apoiado pelo NIH R00HL148493 (YD), R01HL162635 (YD) e UT Dallas STARS program (YD).
Materiais
| Name | Company | Catalog Number | Comments |
| 1% Agarose | |||
| Low melting point agarose | Thermo Fisher | 16520050 | |
| Deionized water | - | - | |
| Chemicals for tissue clearing | |||
| 5-Amino-1,3,3-trimethylcyclohexanemethylamine, mixture of cis and trans | Sigma-Aldrich | 118184 | |
| D.E.R.™ 332 | Sigma-Aldrich | 31185 | |
| D.E.R.™ 736 | Sigma-Aldrich | 31191 | |
| Dibenzyl ether (DBE) | Sigma-Aldrich | 33630 | |
| Dichloromethane (DCM) | Sigma-Aldrich | 270997 | |
| Fluorescent beads | Spherotech | FP-0556-2 | |
| Hydrogen peroxide (H2O2) | Sigma-Aldrich | 216736 | |
| Methanol | Sigma-Aldrich | 439193 | |
| Paraformaldehyde (PFA) | Thermo Fisher | 47392 | |
| Phosphate Buffered Saline (PBS) | Sigma-Aldrich | 79383 | |
| Potassium Chloride (KCl) | Sigma-Aldrich | P3911 | |
| Software and algorithms | |||
| Amira | Thermo Fisher Scientific | 2021.2 | |
| BigStitcher | Hörl et al.22 | ||
| Fiji-ImageJ | Schindelin et al.20 | 1.54f | |
| HCImage Live | Hamamatsu Photonics | 4.6.1.2 | |
| LabVIEW | National Instruments Corporation | 2017 SP1 | |
| Key components of the customized light-sheet system | |||
| 0.63 - 6.3X Zoom body | Olympus | MVX-ZB10 | |
| 10X Illumination objective | Nikon | MRH00105 | |
| 1X detection objective | Olympus | MV PLAPO 1X/0.25 | |
| 473nm DPSS Laser | Laserglow Technologies | LRS-0473-PFM-00100-05 | |
| 532nm DPSS laser | Laserglow Technologies | LRS-0532-PFM-00100-05 | |
| 589 nm DPSS laser | Laserglow Technologies | LRS-0589-GFF-00100-05 | |
| BNC connector | National Instrument | BNC-2110 | |
| Cylindrical lens | Thorlabs | ACY254-050-A | |
| DC-Motor Controller, 4 axes | Physik Instrumente | C-884.4DC | |
| ETL | Optotune | EL-16-40-TC-VIS-5D-1-C | |
| ETL Cable | Optotune | CAB-6-300 | |
| ETL Lens Driver | Optotune | EL-E-4i | |
| Filter | Chroma | ET525/30 | |
| Filter | Chroma | ET585-40 | |
| Filter | Chroma | ET645-75 | |
| Filter wheel | Shutter Instrument | LAMBDA 10-B | |
| Motorized translation stage | Physik Instrumente | L-406.20DG10 | |
| Motorized translation stage | Physik Instrumente | L-406.40DG10 | |
| Motorized translation stage | Physik Instrumente | M-403.4PD | |
| NI multifunction I/O | National Instrument | PCIe-6363 | |
| sCMOS camera | Hamamatsu | C13440-20CU | |
| Stepper motor | Pololu | 1474 | |
| Tube lens | Olympus | MVX-TLU |
Referências
- Sadek, H., Olson, E. N. Toward the goal of human heart regeneration. Cell Stem Cell. 26, 7-16 (2020).
- Porrello, E. R., et al. Transient regenerative potential of the neonatal mouse heart. Science. 331, 1078-1080 (2011).
- Stelzer, E. H. K. K., et al. Light sheet fluorescence microscopy. Nat Rev Methods Prim. 1, 73 (2021).
- Girkin, J. M., Carvalho, M. T. The light-sheet microscopy revolution. J Opt. 20, 053002 (2018).
- Power, R. M., Huisken, J. A guide to light-sheet fluorescence microscopy for multiscale imaging. Nat Methods. 14, 360-373 (2017).
- Ding, Y., et al. Multiscale light-sheet for rapid imaging of cardiopulmonary system. JCI Insight. 3, e121396 (2018).
- Ding, Y., et al. Light-sheet fluorescence imaging to localize cardiac lineage and protein distribution. Sci Rep. 7, 42209 (2017).
- Fei, P., et al. Cardiac light-sheet fluorescent microscopy for multi-scale and rapid imaging of architecture and function. Sci Rep. 6, 1-12 (2016).
- Richardson, D. S., Lichtman, J. W. Clarifying tissue clearing. Cell. 162, 246-257 (2015).
- Stelzer, E. H. K., Huisken, J., Swoger, J., Greger, K., Verveer, P. Multi-view image fusion improves resolution in three-dimensional microscopy. Opt Express. 15 (13), 8029-8042 (2007).
- Dean, K. M., Roudot, P., Welf, E. S., Danuser, G., Fiolka, R. Deconvolution-free subcellular imaging with axially swept light sheet microscopy. Biophys J. 108, 2807-2815 (2015).
- Dean, K. M., et al. Isotropic imaging across spatial scales with axially swept light-sheet microscopy. Nat Protoc. 17, 2025-2053 (2022).
- Hedde, P. N., Gratton, E. Selective plane illumination microscopy with a light sheet of uniform thickness formed by an electrically tunable lens. Microsc Res Tech. 81, 924 (2018).
- Voigt, F. F., et al. The mesoSPIM initiative: open-source light-sheet microscopes for imaging cleared tissue. Nat Meth. 16, 1105-1108 (2019).
- Giardini, F., et al. Mesoscopic optical imaging of whole mouse heart. J Vis Exp. (176), e62795 (2021).
- Sodimu, O., et al. Light sheet imaging and interactive analysis of the cardiac structure in neonatal mice. J Biophotonics. 16, e202200278 (2023).
- Ariel, P. A beginner's guide to tissue clearing. Int J Biochem Cell Biol. 84, 35-39 (2017).
- Richardson, D. S., et al. Tissue clearing. Nat Rev Methods Prim. 1, 1-24 (2021).
- Ueda, H. R., et al. Tissue clearing and its applications in neuroscience. Nat Rev Neurosci. 21, 61-79 (2020).
- Renier, N., et al. iDISCO: a simple, rapid method to immunolabel large tissue samples for volume imaging. Cell. 159, 896-910 (2014).
- Kirchner, K. N., et al. A hydrophobic tissue clearing method for rat brain tissue. J Vis Exp. (166), e61821 (2020).
- Ding, Y., et al. Light-sheet fluorescence microscopy for the study of the murine heart. J Vis Exp. (139), e57769 (2018).
- Schindelin, J., Rueden, C. T., Hiner, M. C., Eliceiri, K. W. The ImageJ ecosystem: An open platform for biomedical image analysis. Mol Reprod Dev. 82, 518-529 (2015).
- Hörl, D., et al. BigStitcher: reconstructing high-resolution image datasets of cleared and expanded samples. Nat Methods. 16, 870-874 (2019).
- Becker, K., et al. Reduction of Photo Bleaching and Long Term Archiving of Chemically Cleared GFP-Expressing Mouse Brains. PLoS One. 9, e114149 (2014).
- Preibisch, S., et al. Efficient Bayesian-based multiview deconvolution. Nat. Methods. 11, 645-648 (2014).
- Guo, M., et al. Rapid image deconvolution and multiview fusion for optical microscopy. Nat. Biotechnol. 38, 1337-1346 (2020).
- Tomer, R., Khairy, K., Amat, F., Keller, P. J. Quantitative high-speed imaging of entire developing embryos with simultaneous multiview light-sheet microscopy. Nat Methods. 9, 755-763 (2012).
- Fahrbach, F. O., Voigt, F. F., Schmid, B., Helmchen, F., Huisken, J. Rapid 3D light-sheet microscopy with a tunable lens. Opt Express. 21, 21010-21026 (2013).
- Liu, Y., Rollins, A. M., Jenkins, M. W. CompassLSM: axially swept light-sheet microscopy made simple. Biomed Opt Express. 12, 6571-6589 (2021).
- Kolesová, H., Olejníčková, V., Kvasilová, A., Gregorovičová, M., Sedmera, D. Tissue clearing and imaging methods for cardiovascular development. iScience. 24 (4), 102387 (2021).
- Sands, G. B., et al. It's clearly the heart! Optical transparency, cardiac tissue imaging, and computer modelling. Prog Biophys Mol Biol. 168, 18-32 (2022).
- Wilson, A. J., Sands, G. B., LeGrice, I. J., Young, A. A., Ennis, D. B. Muscle mechanics and ventricular function: Myocardial mesostructure and mesofunction. Am J Physiol - Hear Circ Physiol. 323, H257 (2022).
- Lee, S. E., et al. Three-dimensional cardiomyocytes structure revealed by diffusion tensor imaging and its validation using a tissue-clearing technique. Sci. Reports. 8, 1-11 (2018).
- Sereti, K. I., et al. Analysis of cardiomyocyte clonal expansion during mouse heart development and injury. Nat Commun. 9, 754 (2018).
- Olianti, C., et al. 3D imaging and morphometry of the heart capillary system in spontaneously hypertensive rats and normotensive controls. Sci. Reports. 10, 1-9 (2020).
- Olianti, C., et al. Optical clearing in cardiac imaging: A comparative study. Prog Biophys Mol Biol. 168, 10-17 (2022).
- Baek, K. I., et al. Advanced microscopy to elucidate cardiovascular injury and regeneration: 4D light-sheet imaging. Prog Biophys Mol Biol. 138, 105-115 (2018).
- Merz, S. F., et al. Contemporaneous 3D characterization of acute and chronic myocardial I/R injury and response. Nat Commun. 10, 1-14 (2019).
- Zhang, X., et al. 4D Light-sheet imaging and interactive analysis of cardiac contractility in zebrafish larvae. APL Bioeng. 7, 26112 (2023).
- Lee, J., et al. 4-Dimensional light-sheet microscopy to elucidate shear stress modulation of cardiac trabeculation. J Clin Invest. 126, 1679-1690 (2016).
- Zhang, X., Alexander, R. V., Yuan, J., Ding, Y. Computational Analysis of Cardiac Contractile Function. Curr Cardiol Rep. 24, 1983-1994 (2022).
- Zhang, X., et al. 4D Light-sheet Imaging of Zebrafish Cardiac Contraction. J Vis Exp. (203), e66263 (2024).
- Huisken, J., Swoger, J., Del Bene, F., Wittbrodt, J., Stelzer, E. H. K. K. Optical sectioning deep inside live embryos by selective plane illumination microscopy. Science. 305, 1007-1009 (2004).
- Molbay, M., Kolabas, Z. I., Todorov, M. I., Ohn, T., Ertürk, A. A guidebook for DISCO tissue clearing. Mol Syst Biol. 17, 9807 (2021).
- Chi, J., Crane, A., Wu, Z., Cohen, P. Adipo-clear: a tissue clearing method for three-dimensional imaging of adipose tissue. J Vis Exp. (137), e58271 (2018).
- Wan, Y., McDole, K., Keller, P. J. Light-sheet microscopy and its potential for understanding developmental processes. Ann Rev Cell Dev Biol. 35, 655-681 (2019).
- Yuan, J., et al. Extended reality for biomedicine. Nat Rev Methods Prim. 3, 1-1 (2023).
- Ding, Y., et al. Saak transform-based machine learning for light-sheet imaging of cardiac trabeculation. IEEE Trans Biomed Eng. 68, 225-235 (2020).
- Buffinton, C. M., Benjamin, A. K., Firment, A. N., Moon, A. M. Myocardial wall stiffening in a mouse model of persistent truncus arteriosus. PLoS One. 12 (9), e0184678 (2017).
- Trincot, C. E., et al. Adrenomedullin induces cardiac lymphangiogenesis after myocardial infarction and regulates cardiac edema via Cx43. Circ Res. 124, 101 (2019).
- Yokoyama, T., et al. Quantification of sympathetic hyperinnervation and denervation after myocardial infarction by three-dimensional assessment of the cardiac sympathetic network in cleared transparent murine hearts. PLoS One. 12, e0182072 (2017).
- Coram, R. J., et al. Muscleblind-like 1 is required for normal heart valve development in vivo. BMC Dev Biol. 15, 36 (2015).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados