Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Визуализация светового листа для выявления структуры сердца в сердцах грызунов
В этой статье
Резюме
В протоколе используется передовая световая микроскопия наряду с адаптированными методами очистки тканей для исследования сложных сердечных структур в сердцах грызунов, что имеет большой потенциал для понимания морфогенеза и ремоделирования сердца.
Аннотация
Световая микроскопия (LSM) играет ключевую роль в понимании сложной трехмерной (3D) структуры сердца, обеспечивая решающее понимание фундаментальной физиологии сердца и патологических реакций. Мы углубляемся в разработку и внедрение метода LSM для выяснения микроархитектуры сердца на мышиных моделях. Методология объединяет индивидуальную систему LSM с методами очистки тканей, уменьшая рассеяние света в тканях сердца для объемной визуализации. Сочетание традиционного LSM со сшивкой изображений и многоракурсной деконволюцией позволяет захватывать все сердце. Чтобы устранить неизбежный компромисс между осевым разрешением и полем зрения (FOV), мы также представляем метод осевой световой листовой микроскопии (ASLM) для минимизации расфокусированного света и равномерного освещения сердца в направлении распространения. В то же время методы очистки тканей, такие как iDISCO, усиливают проникновение света, облегчая визуализацию глубоких структур и обеспечивая всестороннее обследование миокарда по всему сердцу. Комбинация предложенных методов LSM и очистки тканей представляет собой многообещающую платформу для исследователей в разрешении сердечных структур в сердцах грызунов, обладающую большим потенциалом для понимания морфогенеза и ремоделирования сердца.
Введение
Сердечная недостаточность остается основной причиной смертности во всем мире, в первую очередь из-за отсутствия регенеративной способности зрелых кардиомиоцитов1. Сложная архитектура сердца играет решающую роль в его функционировании и дает представление о процессах развития. Глубокое понимание структуры сердца необходимо для выяснения фундаментальных процессов морфогенеза сердца и ремоделирования в ответ на инфаркт миокарда. Недавний прогресс показал, что неонатальные мыши могут восстанавливать сердечную функцию после травмы, в то время как взрослые мыши не имеют такой регенеративной способности2. Это создает основу для исследования сигналов, связанных со структурными и функциональными аномалиями в мышиных моделях. Традиционные методы визуализации, такие как конфокальная микроскопия, имеют технические ограничения, включая ограниченную глубину проникновения, медленную схему точечного сканирования и фотоповреждения при длительном воздействии лазерного света. Они препятствуют всестороннему трехмерному (3D) изображению неповрежденного сердца. В этом контексте светолистовая микроскопия (LSM) становится мощным решением, предлагающим преимущества высокоскоростной визуализации, снижения фотоповреждений и исключительных возможностей оптического среза 3,4,5. Уникальные особенности LSM позиционируют его как многообещающий метод преодоления ограничений традиционных методов, обеспечивающий беспрецедентное понимание процессов развития сердца и ремоделирования 6,7,8.
В этом протоколе мы представляем стратегию визуализации, которая сочетает в себе усовершенствованную LSM с адаптированными подходами к очищению тканей9, что позволяет визуализировать целые сердца мышей без необходимости специальной маркировки и механического среза. Мы также предполагаем, что традиционная визуализация LSM может быть улучшена с помощью методов многоракурсной деконволюции10 или аксиальной светолистовой микроскопии (ASLM) 11,12,13,14,15 для улучшения аксиального разрешения. Кроме того, интеграция сшивки изображений с любым из этих методов может эффективно преодолеть компромисс между пространственным разрешением и полем зрения (FOV), тем самым улучшая визуализацию сердец взрослых мышей. Включение многочисленных подходов к очищению тканей, включая гидрофобные, гидрофильные и гидрогелевые методы, обеспечивает более глубокое проникновение света для захвата морфологии всего сердца 16,17,18,19.
В то время как несколько методов очистки совместимы с существующими системами LSM, цель состоит в том, чтобы свести к минимуму рассеяние фотонов и улучшить проникновение света в ткани, такие как сердце, путем замены липидов средой, которая близко соответствует его показателю преломления. iDISCO был выбран в качестве репрезентативноговарианта 20,21 и адаптирован для автофлуоресцентной визуализации в этом протоколе благодаря его быстрой обработке и высокой прозрачности (рис. 1A). В совокупности интеграция передового подхода LSM с методами очистки тканей предлагает многообещающую основу для разгадки сложной анатомии сердца в сердцах грызунов, что имеет значительный потенциал для улучшения нашего понимания морфогенеза и патогенеза сердца.
протокол
Протоколы и эксперименты на животных были одобрены и проведены под надзором Комитета по уходу и использованию животных Техасского университета в Далласе (IACUC #21-03). В этом исследовании использовались мыши C57BL6, включая новорожденных на 1-й день после рождения (P1) и 8-недельных взрослых. Различий между самцами и самками не наблюдалось. Весь сбор данных и постобработка изображений осуществлялись с использованием программного обеспечения с открытым исходным кодом или платформ с исследовательскими или образовательными лицензиями. Ресурсы предоставляются авторами по обоснованному запросу.
1. Подготовка образцов и очистка тканей (6 - 10 дней)
- Перед началом процедуры очищения тканей приготовьте все химические растворы, указанные в таблице материалов , необходимых для метода очищения тканей.
ПРИМЕЧАНИЕ: Выполняйте все процедуры в вытяжном шкафу, надев надлежащие средства индивидуальной защиты, особенно при работе с токсичными химическими веществами. - Усыпляют животных сСО2 , помещая их в камеруСО2 в желаемые моменты времени, такие как Р1 и 8 недель, для сбора всего сердца и погружают только что выделенное сердце при комнатной температуре (ОТ) в 0,2 М KCl для остановки в диастоле16.
- Зафиксируйте сердце, погрузив его в 4% параформальдегид (PFA) в 1x фосфатно-солевой буфер (PBS) с легким перемешиванием на коромысле на ночь, чтобы сохранить анатомию сердца при 4 °C, см. Таблицу 1.
ВНИМАНИЕ: PFA является токсичным химическим веществом и должен выполняться под вытяжным шкафом. - Промыть сердце 1x PBS в RT в течение 30 мин, 3x.
ПРИМЕЧАНИЕ: Дополнительно: Вставьте сердце в агарозный гель, чтобы создать сердечную сборку для специально созданной системы LSM на шаге 2, как описано ниже.- Приготовьте 1% раствор агарозы, растворив агарозу в PBS, и нагрейте смесь в микроволновой печи до полного растворения. Перелейте раствор в форму, чтобы создать нижний слой, пока раствор не остынет до 40 °C.
- Поместите сердечко в форму и заполните форму агарозным гелем до застывания. Удалите все пузырьки в агарозном геле, чтобы свести к минимуму артефакты при визуализации.
ПРИМЕЧАНИЕ: Встраивание образца снижает риск потенциального повреждения анатомии сердца во время монтажа.
ВНИМАНИЕ: Используйте агарозу с низкой температурой плавления и избегайте помещения сердца непосредственно в нагретую агарозу, чтобы снизить риск воздействия на образец повышенных температур.
- Обезвоживайте сердце с помощью градиентов метанола, смешанного с деионизированной водой (рисунок 1B), обрабатывая последовательными концентрациями 20%, 40%, 60%, 80% и 100%, как показано в таблице 1. Повторите со свежим 100% метанолом 1 раз, чтобы обеспечить полное обезвоживание. Обезвоживайте сердца новорожденных мышей в течение 1 ч в каждой смеси метанола с встряхиванием. Отрегулируйте время инкубации до 2 часов для 8-недельных мышей и крысиных сердец.
ВНИМАНИЕ: Метанол является летучим, раздражающим и легковоспламеняющимся химическим веществом. - Замените и охладите сердце свежим 100% метанолом в течение 10 минут при температуре 4 °C.
- Замените раствор и отбеливайте сердце 5% перекисью водорода (H2O2) в метаноле с объемным соотношением 1:5 (30% H2O2 до 100% метанола) в течение ночи при 4 °C для удаления пигментов.
ПРИМЕЧАНИЕ: Удаление пигментов позволяет свести к минимуму поглощение фотонов, что приводит к улучшению качества изображения с уменьшением артефактов. - Замените раствор и инкубируйте сердце в 100% метаноле в течение 1 ч при РТ.
- Делипидатировать сердце раствором, содержащим 66% дихлорметана (ДКМП) и 33% метанола, в течение 3 ч с встряхиванием. Убедитесь, что к концу этого шага сердце опустится на дно флакона. Если нет, замените раствор (66% DCM / 33% метанола) и увеличьте время инкубации (например, еще на 1 час) для достижения полного удаления липидов. Быстро переведите сердце в свежий раствор, чтобы предотвратить высыхание, так как ДКМП очень летуч.
ВНИМАНИЕ: Для эксперимента используйте полипропилен или стеклянную посуду, так как DCM несовместим с полистиролом. - Используйте дибензиловый эфир (DBE) для согласования показателей преломления (RI = 1,56). Для неонатальных мышиных сердец процесс сопоставления показателя преломления занимает 2 дня, в то время как для 8-недельных мышиных сердец он занимает 5 дней с легким встряхиванием. Замените его свежим ДБЭ и продлите время инкубации до тех пор, пока сердце не достигнет полной прозрачности.
ВНИМАНИЕ: ДБЭ опасен. Избегайте любого контакта с кожей.
2. Монтаж образца (1 день)
ПРИМЕЧАНИЕ: Если используется коммерческая LSM-система, следуйте ее специальному протоколу, предоставленному компанией, чтобы исправить сердце, и пропустите шаги 2.1 - 2.9.
- Настройте камеру и держатель, устойчивые к решению для согласования показателя преломления (например, DBE в адаптированном подходе iDISCO) с помощью 3D-принтера и материалов из оникса (рис. 2A)16.
- Прикрепите магнит к нижней части камеры, чтобы облегчить быстрое подключение и точную локализацию под системой LSM (рис. 2B).
- Прикрепите защитные стекла размером 25 мм x 50 мм x 0,17 мм к камере с помощью прозрачного силиконового клея и проверьте водонепроницаемость камеры, заполнив ее DBE и проверив на герметичность через 1 день.
- Настройте держатель зажима с помощью 3D-принтера и прикрепите магнит для крепления блока сердца внутри камеры (рис. 2C) или вставьте иглу в агарозный гель для монтажа блока сердца.
- Осуществить разработку собственной системы LSM с использованием цилиндрической линзы и твердотельных лазеров с непрерывной диодной накачкой (DPSS), как сообщалось ранее16,22. Используйте длину волны возбуждения 532 нм, чтобы захватить автофлуоресценцию из миокарда (рис. 2D).
- Установите камеру на 6-мерный столик и найдите оптимальное положение, где камера перпендикулярна направлению распространения лазера.
- Используйте магнитный держатель зажима, чтобы поместить образец в середину камеры и подключить держатель к 4-мерному моторизованному столику (X, Y, Z и Yaw; Рисунок 2D).
- Погрузите сердечный блок в камеру, заполненную ДБЭ, с помощью моторизованного столика и определите дальность сканирования по оси обнаружения (направление Z).
- Определите скорость сканирования (Vz) двигателя по оси Z на основе размера шага (S) и времени получения изображения (T) с помощью следующего уравнения:
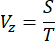
ПРИМЕЧАНИЕ: Соотношение между размером шага и осевым разрешением системы визуализации соответствует теореме Найквиста-Шеннона. Например, размер шага был установлен на 1 мкм, учитывая, что осевое разрешение составляло 2,91 мкм, как указано в предыдущем отчете16. - Чтобы проверить пространственное разрешение и свести к минимуму потенциальные проблемы с непрозрачностью на разных глубинах, измерьте функцию рассеяния точки (PSF) системы, визуализируя флуоресцентные шарики диаметром 0,53 мкм, разбавленные в 1% агарозном геле с низкой температурой плавления с концентрацией 1:1,5 x 105. Рассчитайте PSF по всему образцу, измерив полную ширину при половине максимума (FWHM)16.
3. Сшивка изображений (4-8 часов)
- Рассчитайте количество плиток, необходимых для покрытия всего сердца мыши, с учетом ограниченного поля зрения. Для каждой плитки размеры соответствуют полю зрения системы LSM. Например, для неонатального сердца мыши с поперечным сечением 3 x 3,6мм2 требуется 2 x 2 плитки для покрытия в индивидуальном LSM (рис. 3A). Напротив, 8-недельное сердце мыши размером 5 x 8мм2 требует 3 x 4 плиток, чтобы покрыть всю сердечную структуру (рисунок 3B).
ПРИМЕЧАНИЕ: Сшивание изображений требуется, если обычный LSM имеет ограниченное поле зрения для визуализации всего сердца. - Сделайте снимки сердца, начиная с плитки 1, т. е. верхней левой угловой плитки сердца (рисунок 3A-B).
- Последовательно захватывайте изображения оставшихся плиток с 10-процентным перекрытием между последовательными плитками, пока не будет покрыто все сердцевина, как показано на рисунке 3C.
- Загрузите и установите программное обеспечение с открытым исходным кодом Fiji23 . Скачайте и установите плагин для Фиджи, BigStitcher24.
- Перейдите в меню «Справка», выберите «Обновить», выберите «Управление сайтом обновлений» и выберите BigStitcher. Затем нажмите кнопку Закрыть, а затем Применить изменения. После завершения загрузки плагина перезапустите программное обеспечение Fiji.
- Откройте плагин BigStitcher и импортируйте все необходимые тайлы через каталог с файлами изображений. Сохраните набор данных в виде файла HDF5.
- Организуйте плитки, выбрав «Переместить плитку по обычной сетке», выберите узор, который используется для перемещения сердца, и выберите 10-процентное перекрытие между каждой плиткой. Сшейте плитки с помощью параметра Мастер сшивки.
- Экспортируйте сшитые данные с помощью Image Fusion, чтобы создать tiff-файл.
4. Многоракурсная деконволюция (5 дней)
- Используйте флуоресцентные шарики для регистрации изображений многоракурсной реконструкции вдоль сердца (Рисунок 3D).
- Смолу25 готовят путем смешивания диглицидилового эфира бисфенола-А (D.E.R. 332), изофорона-дамина, 5-амино-1,3,3-триметилциклогексанметиламина (IPDA) и диглицидилового эфира полипропиленгликоля (D.E.R. 736) в объемном соотношении 4:1:1.
- Центрифугируйте 10 мкл флуоресцентных шариков при 161 x g в течение 5 мин и добавьте 20 мкл метанола для замены раствора для хранения гранул. Повторите это 3 раза.
- Приготовьте раствор объемом 10 мл с концентрацией гранул 1:1 x 105 , смешав раствор гранул на основе метанола со смолой, приготовленной на шаге 4.1.1.
- Дегазация раствора в вакуумной камере в течение 3 ч для удаления воздушных карманов и пузырьков.
- Налейте раствор в силиконовую форму или пластиковую соломинку для питья и подождите 2-3 дня, пока он не застынет. Через 1 день, прежде чем смола станет твердой, поместите стеклянную трубку внутрь формы и подождите, пока трубка прилипнет к смоле. Извлеките смолу из формы с помощью стеклянной трубки (рисунок 3D).
- Поместите сердце в стеклянную трубку, наполните ее DBE и установите стеклянную трубку в камеру, заполненную DBE.
- Получение последовательных изображений сердца мыши вместе с шариками из освещенной части путем сканирования блока сердца по оси обнаружения (ось Z; Рисунок 3E-F).
ПРИМЕЧАНИЕ: Реализуйте сшивку изображений на шаге 3 для создания отдельных стопок изображений, если размер сердца превышает поле зрения. - Поверните сердце мыши на 60° вдоль оси Y, чтобы захватить последующие стопки под разными углами, т. е. 0°, 60°, 120°, 180°, 240° и 300° (рис. 3E-F).
- Откройте плагин BigStitcher и импортируйте все изображения через каталог файлов изображений. Сохраните набор данных в виде файла HDF5.
- Выберите «Определить точки интереса » и вручную выберите интересующие вас бусины для регистрации.
- Выберите Модель аффинного преобразования для регистрации. Выберите «Функция разброса точек» и назначьте кадр PSF всем видам.
- Нажмите Multiview Deconvolution и выберите тип итерации Efficient Bayesian. Определите Число итераций равным 10 и выполнитеего 26.
- Нажмите на Image Fusion , чтобы экспортировать файл tiff .
ПРИМЕЧАНИЕ: Более подробную информацию о многоракурсной деконволюции можно найти в других отчетах или протоколах 24,27,28.
5. Аппаратное обеспечение системы светового листа с осевой стреловидностью (1 день)
- Установите электрическую перестраиваемую линзу (ETL) на плоскость Фурье цилиндрической линзы (CL)27 (рис. 2D) для сканирования лазерного луча в осевом направлении14. Убедитесь, что граница раздела жидкости в ETL находится горизонтально, чтобы предотвратить искажение волнового фронта, вызванное гравитацией. Точное выравнивание ETL, чтобы свести к минимуму децентрацию луча и оптические аберрации более высокого порядка30.
- Установите плату сбора данных на рабочую станцию и подключите кабель BNC к плате сбора данных для синхронизации экспозиции камеры и сканирования ETL.
- Откройте корпус ETL-драйвера и снимите крышку (рис. 4).
- Припаяйте сигнальный провод кабеля BNC к выводу Analog in B, как указано на нижней части печатной платы (рис. 4).
- Припаяйте провод заземления кабеля BNC к выводу GND; после этого подключите другой конец кабеля BNC к AO (аналоговому выходу) на плате сбора данных (рис. 4).
- Подключите драйвер ETL к USB-порту рабочей станции, подключите драйвер к ETL с помощью кабеля Hirose и установите программное обеспечение контроллера драйвера объектива.
- Измените режим работы в программном обеспечении контроллера драйвера объектива на аналоговый, чтобы управлять ETL с помощью триггера, сгенерированного картой сбора данных.
- В программном обеспечении sCMOS камеры переключите режим съемки на Внешний (Light-sheet). Убедитесь, что активное направление сканирования пикселей перпендикулярно направлению лазерного сканирования, чтобы облегчить синхронизацию.
- Подключите внешний триггерный порт на задней панели камеры sCMOS к AO платы сбора данных с помощью кабеля BNC (рис. 4).
6. Синхронизация системы светового листа с осевой стреловидностью (7 дней)
- Определите время экспозиции на основе приемлемого отношения сигнал/фон (SBR), которое для предлагаемого исследования составляет не менее 10. Увеличьте время экспозиции, если SBR меньше 10.
ПРИМЕЧАНИЕ: Время экспозиции в диапазоне от 10 до 50 мс обеспечивает приемлемый SBR в этом проекте. - Определите время получения изображения (T) и интервал линии (L) на основе времени экспозиции (E) для камеры и частоты ETL (f), используя следующие уравнения:
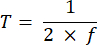
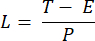
Где P обозначает количество столбцов пикселей на сенсоре камеры. Репрезентативные параметры для опорного сигнала: f = 1 Гц, P = 2048, E = 10 мс и L = 243 μс соответственно. - В программе LabVIEW Trigger Generator.vi генерирует как квадратные, так и треугольные триггеры для синхронизации (рисунок 5).
ПРИМЕЧАНИЕ: Индивидуальная программа LabVIEW доступна у авторов по обоснованному запросу. - Установите флуоресцентные шарики, разведенные в 1% агарозном геле16 с низкой температурой плавления с концентрацией 1:1 x 103 , чтобы определить положение талии пучка на изображении (фиг.6А).
- В программе определите начальную и конечную точки лазерного луча под полем зрения, изменяя напряжение по направлению сканирования.
- Чтобы синхронизировать sCMOS-камеру и ETL, отсканируйте лазерный луч вдоль оси X и активные пиксели вдоль оси Y, чтобы получить 2D-изображение (рис. 6B). Более яркие и четкие бусины по диагонали на изображении указывают на точную синхронизацию между sCMOS и ETL.
ПРИМЕЧАНИЕ: Синхронизация скорости сканирования ETL и активации пикселей sCMOS имеет решающее значение для качества изображения. Различные распространенные асинхронные ошибки обобщены на рисунке 6C-F. - После определения оптимальных параметров синхронизации для синхронизации поверните sCMOS-камеру на 90° вокруг оси Z, чтобы выровнять направление активного пикселя с направлением лазерного сканирования.
- Повторите шаг 2.10 и измерьте FWHM PSF. Используйте тот же метод, что и в предыдущем отчете16, со усредненным боковым и осевым разрешениями ASLM 2,18 мкм и 2,88 мкм, соответственно (рис. 7).
ПРИМЕЧАНИЕ: Равномерное освещение и разрешение по всему полю зрения возможны в идеальных условиях (Рисунок 7A-B). Асинхронная работа ETL с камерой sCMOS, а также несогласованность освещения и обнаружения могут привести к неравномерному пространственному разрешению (рис. 7C-D). - Для визуализации сердца, после выравнивания системы и определения оптимальных параметров для синхронизации системы, перейдите к шагам 2.6–2.9.
Результаты
Было продемонстрировано, что LSM способствует кардиологическим исследованиям 31,32,33,34,35,36,37 из-за минимального риска фотоповреждения, высокого пространственного разрешения и оптического среза по сравнению с другими методами оптической визуализации, такими как светлопольные
Обсуждение
Развитие методов визуализации, вычислений и очистки тканей предоставило беспрецедентную возможность для обширного исследования структуры и функции сердца. Это имеет большой потенциал для углубления нашего понимания морфогенеза и патогенеза сердца с использованием модели сердца ин?...
Раскрытие информации
У авторов нет конфликта интересов, который можно было бы раскрыть.
Благодарности
Мы выражаем благодарность группе доктора Эрика Олсона в Юго-западном медицинском центре Техасского университета за щедрое предоставление моделей животных. Мы ценим все конструктивные комментарии, предоставленные членами D-инкубатора в Техасском университете в Далласе. Эта работа была поддержана NIH R00HL148493 (Y.D.), R01HL162635 (Y.D.) и программой UT Dallas STARS (Y.D.).
Материалы
| Name | Company | Catalog Number | Comments |
| 1% Agarose | |||
| Low melting point agarose | Thermo Fisher | 16520050 | |
| Deionized water | - | - | |
| Chemicals for tissue clearing | |||
| 5-Amino-1,3,3-trimethylcyclohexanemethylamine, mixture of cis and trans | Sigma-Aldrich | 118184 | |
| D.E.R.™ 332 | Sigma-Aldrich | 31185 | |
| D.E.R.™ 736 | Sigma-Aldrich | 31191 | |
| Dibenzyl ether (DBE) | Sigma-Aldrich | 33630 | |
| Dichloromethane (DCM) | Sigma-Aldrich | 270997 | |
| Fluorescent beads | Spherotech | FP-0556-2 | |
| Hydrogen peroxide (H2O2) | Sigma-Aldrich | 216736 | |
| Methanol | Sigma-Aldrich | 439193 | |
| Paraformaldehyde (PFA) | Thermo Fisher | 47392 | |
| Phosphate Buffered Saline (PBS) | Sigma-Aldrich | 79383 | |
| Potassium Chloride (KCl) | Sigma-Aldrich | P3911 | |
| Software and algorithms | |||
| Amira | Thermo Fisher Scientific | 2021.2 | |
| BigStitcher | Hörl et al.22 | ||
| Fiji-ImageJ | Schindelin et al.20 | 1.54f | |
| HCImage Live | Hamamatsu Photonics | 4.6.1.2 | |
| LabVIEW | National Instruments Corporation | 2017 SP1 | |
| Key components of the customized light-sheet system | |||
| 0.63 - 6.3X Zoom body | Olympus | MVX-ZB10 | |
| 10X Illumination objective | Nikon | MRH00105 | |
| 1X detection objective | Olympus | MV PLAPO 1X/0.25 | |
| 473nm DPSS Laser | Laserglow Technologies | LRS-0473-PFM-00100-05 | |
| 532nm DPSS laser | Laserglow Technologies | LRS-0532-PFM-00100-05 | |
| 589 nm DPSS laser | Laserglow Technologies | LRS-0589-GFF-00100-05 | |
| BNC connector | National Instrument | BNC-2110 | |
| Cylindrical lens | Thorlabs | ACY254-050-A | |
| DC-Motor Controller, 4 axes | Physik Instrumente | C-884.4DC | |
| ETL | Optotune | EL-16-40-TC-VIS-5D-1-C | |
| ETL Cable | Optotune | CAB-6-300 | |
| ETL Lens Driver | Optotune | EL-E-4i | |
| Filter | Chroma | ET525/30 | |
| Filter | Chroma | ET585-40 | |
| Filter | Chroma | ET645-75 | |
| Filter wheel | Shutter Instrument | LAMBDA 10-B | |
| Motorized translation stage | Physik Instrumente | L-406.20DG10 | |
| Motorized translation stage | Physik Instrumente | L-406.40DG10 | |
| Motorized translation stage | Physik Instrumente | M-403.4PD | |
| NI multifunction I/O | National Instrument | PCIe-6363 | |
| sCMOS camera | Hamamatsu | C13440-20CU | |
| Stepper motor | Pololu | 1474 | |
| Tube lens | Olympus | MVX-TLU |
Ссылки
- Sadek, H., Olson, E. N. Toward the goal of human heart regeneration. Cell Stem Cell. 26, 7-16 (2020).
- Porrello, E. R., et al. Transient regenerative potential of the neonatal mouse heart. Science. 331, 1078-1080 (2011).
- Stelzer, E. H. K. K., et al. Light sheet fluorescence microscopy. Nat Rev Methods Prim. 1, 73 (2021).
- Girkin, J. M., Carvalho, M. T. The light-sheet microscopy revolution. J Opt. 20, 053002 (2018).
- Power, R. M., Huisken, J. A guide to light-sheet fluorescence microscopy for multiscale imaging. Nat Methods. 14, 360-373 (2017).
- Ding, Y., et al. Multiscale light-sheet for rapid imaging of cardiopulmonary system. JCI Insight. 3, e121396 (2018).
- Ding, Y., et al. Light-sheet fluorescence imaging to localize cardiac lineage and protein distribution. Sci Rep. 7, 42209 (2017).
- Fei, P., et al. Cardiac light-sheet fluorescent microscopy for multi-scale and rapid imaging of architecture and function. Sci Rep. 6, 1-12 (2016).
- Richardson, D. S., Lichtman, J. W. Clarifying tissue clearing. Cell. 162, 246-257 (2015).
- Stelzer, E. H. K., Huisken, J., Swoger, J., Greger, K., Verveer, P. Multi-view image fusion improves resolution in three-dimensional microscopy. Opt Express. 15 (13), 8029-8042 (2007).
- Dean, K. M., Roudot, P., Welf, E. S., Danuser, G., Fiolka, R. Deconvolution-free subcellular imaging with axially swept light sheet microscopy. Biophys J. 108, 2807-2815 (2015).
- Dean, K. M., et al. Isotropic imaging across spatial scales with axially swept light-sheet microscopy. Nat Protoc. 17, 2025-2053 (2022).
- Hedde, P. N., Gratton, E. Selective plane illumination microscopy with a light sheet of uniform thickness formed by an electrically tunable lens. Microsc Res Tech. 81, 924 (2018).
- Voigt, F. F., et al. The mesoSPIM initiative: open-source light-sheet microscopes for imaging cleared tissue. Nat Meth. 16, 1105-1108 (2019).
- Giardini, F., et al. Mesoscopic optical imaging of whole mouse heart. J Vis Exp. (176), e62795 (2021).
- Sodimu, O., et al. Light sheet imaging and interactive analysis of the cardiac structure in neonatal mice. J Biophotonics. 16, e202200278 (2023).
- Ariel, P. A beginner's guide to tissue clearing. Int J Biochem Cell Biol. 84, 35-39 (2017).
- Richardson, D. S., et al. Tissue clearing. Nat Rev Methods Prim. 1, 1-24 (2021).
- Ueda, H. R., et al. Tissue clearing and its applications in neuroscience. Nat Rev Neurosci. 21, 61-79 (2020).
- Renier, N., et al. iDISCO: a simple, rapid method to immunolabel large tissue samples for volume imaging. Cell. 159, 896-910 (2014).
- Kirchner, K. N., et al. A hydrophobic tissue clearing method for rat brain tissue. J Vis Exp. (166), e61821 (2020).
- Ding, Y., et al. Light-sheet fluorescence microscopy for the study of the murine heart. J Vis Exp. (139), e57769 (2018).
- Schindelin, J., Rueden, C. T., Hiner, M. C., Eliceiri, K. W. The ImageJ ecosystem: An open platform for biomedical image analysis. Mol Reprod Dev. 82, 518-529 (2015).
- Hörl, D., et al. BigStitcher: reconstructing high-resolution image datasets of cleared and expanded samples. Nat Methods. 16, 870-874 (2019).
- Becker, K., et al. Reduction of Photo Bleaching and Long Term Archiving of Chemically Cleared GFP-Expressing Mouse Brains. PLoS One. 9, e114149 (2014).
- Preibisch, S., et al. Efficient Bayesian-based multiview deconvolution. Nat. Methods. 11, 645-648 (2014).
- Guo, M., et al. Rapid image deconvolution and multiview fusion for optical microscopy. Nat. Biotechnol. 38, 1337-1346 (2020).
- Tomer, R., Khairy, K., Amat, F., Keller, P. J. Quantitative high-speed imaging of entire developing embryos with simultaneous multiview light-sheet microscopy. Nat Methods. 9, 755-763 (2012).
- Fahrbach, F. O., Voigt, F. F., Schmid, B., Helmchen, F., Huisken, J. Rapid 3D light-sheet microscopy with a tunable lens. Opt Express. 21, 21010-21026 (2013).
- Liu, Y., Rollins, A. M., Jenkins, M. W. CompassLSM: axially swept light-sheet microscopy made simple. Biomed Opt Express. 12, 6571-6589 (2021).
- Kolesová, H., Olejníčková, V., Kvasilová, A., Gregorovičová, M., Sedmera, D. Tissue clearing and imaging methods for cardiovascular development. iScience. 24 (4), 102387 (2021).
- Sands, G. B., et al. It's clearly the heart! Optical transparency, cardiac tissue imaging, and computer modelling. Prog Biophys Mol Biol. 168, 18-32 (2022).
- Wilson, A. J., Sands, G. B., LeGrice, I. J., Young, A. A., Ennis, D. B. Muscle mechanics and ventricular function: Myocardial mesostructure and mesofunction. Am J Physiol - Hear Circ Physiol. 323, H257 (2022).
- Lee, S. E., et al. Three-dimensional cardiomyocytes structure revealed by diffusion tensor imaging and its validation using a tissue-clearing technique. Sci. Reports. 8, 1-11 (2018).
- Sereti, K. I., et al. Analysis of cardiomyocyte clonal expansion during mouse heart development and injury. Nat Commun. 9, 754 (2018).
- Olianti, C., et al. 3D imaging and morphometry of the heart capillary system in spontaneously hypertensive rats and normotensive controls. Sci. Reports. 10, 1-9 (2020).
- Olianti, C., et al. Optical clearing in cardiac imaging: A comparative study. Prog Biophys Mol Biol. 168, 10-17 (2022).
- Baek, K. I., et al. Advanced microscopy to elucidate cardiovascular injury and regeneration: 4D light-sheet imaging. Prog Biophys Mol Biol. 138, 105-115 (2018).
- Merz, S. F., et al. Contemporaneous 3D characterization of acute and chronic myocardial I/R injury and response. Nat Commun. 10, 1-14 (2019).
- Zhang, X., et al. 4D Light-sheet imaging and interactive analysis of cardiac contractility in zebrafish larvae. APL Bioeng. 7, 26112 (2023).
- Lee, J., et al. 4-Dimensional light-sheet microscopy to elucidate shear stress modulation of cardiac trabeculation. J Clin Invest. 126, 1679-1690 (2016).
- Zhang, X., Alexander, R. V., Yuan, J., Ding, Y. Computational Analysis of Cardiac Contractile Function. Curr Cardiol Rep. 24, 1983-1994 (2022).
- Zhang, X., et al. 4D Light-sheet Imaging of Zebrafish Cardiac Contraction. J Vis Exp. (203), e66263 (2024).
- Huisken, J., Swoger, J., Del Bene, F., Wittbrodt, J., Stelzer, E. H. K. K. Optical sectioning deep inside live embryos by selective plane illumination microscopy. Science. 305, 1007-1009 (2004).
- Molbay, M., Kolabas, Z. I., Todorov, M. I., Ohn, T., Ertürk, A. A guidebook for DISCO tissue clearing. Mol Syst Biol. 17, 9807 (2021).
- Chi, J., Crane, A., Wu, Z., Cohen, P. Adipo-clear: a tissue clearing method for three-dimensional imaging of adipose tissue. J Vis Exp. (137), e58271 (2018).
- Wan, Y., McDole, K., Keller, P. J. Light-sheet microscopy and its potential for understanding developmental processes. Ann Rev Cell Dev Biol. 35, 655-681 (2019).
- Yuan, J., et al. Extended reality for biomedicine. Nat Rev Methods Prim. 3, 1-1 (2023).
- Ding, Y., et al. Saak transform-based machine learning for light-sheet imaging of cardiac trabeculation. IEEE Trans Biomed Eng. 68, 225-235 (2020).
- Buffinton, C. M., Benjamin, A. K., Firment, A. N., Moon, A. M. Myocardial wall stiffening in a mouse model of persistent truncus arteriosus. PLoS One. 12 (9), e0184678 (2017).
- Trincot, C. E., et al. Adrenomedullin induces cardiac lymphangiogenesis after myocardial infarction and regulates cardiac edema via Cx43. Circ Res. 124, 101 (2019).
- Yokoyama, T., et al. Quantification of sympathetic hyperinnervation and denervation after myocardial infarction by three-dimensional assessment of the cardiac sympathetic network in cleared transparent murine hearts. PLoS One. 12, e0182072 (2017).
- Coram, R. J., et al. Muscleblind-like 1 is required for normal heart valve development in vivo. BMC Dev Biol. 15, 36 (2015).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены