A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
קביעת הגנום כולו של תזמון שכפול יונק ידי מדידת תוכן DNA
In This Article
Summary
We describe here a relatively fast and simple approach for mapping genome-wide mammalian replication timing, from cell isolation to the basic analysis of the sequencing results. A genomic map of a representative replication program will be provided following the protocol.
Abstract
שכפול של הגנום מתרחש בשלב S של מחזור התא בתהליך פיקוח הדוק שיבטיח את הנאמנות של שכפול ה- DNA. אזור גנומי כל משוכפל בכל פעם ברורה בשלב S דרך ההפעלה בו הזמנית של מקורות מרובים של שכפול. זמן של שכפול (TOR) עולה בקנה אחד עם תכונות רבות גנומי אפיגנטיים והיא צמודה קצב מוטציות וסרטן. להבין את לעיני הגנומי של התכנית שכפול, בבריאות ובחולי הוא מטרה בעתיד גדולה ואתגר.
מאמר זה מתאר בפרוטרוט את "יחס מספר עותק של S / G1 למיפוי זמן הגנומי של שכפול" השיטה (להלן בשם: CNR-תור), גישה פשוטה כדי למפות את הגנום רחב טור של בתאי יונקים. השיטה מבוססת על ההבדלים מספר עותק בין תאים בשלב S ותאי בשלב G1. שיטת CNR-התור מתבצעת 6 שלבים: 1. הכנת תאים מכתים עם יודיד propidium (PI); 2. סורG1 טינג ו- S תאי שלב באמצעות תא מופעל קרינת מיון (FACS); טיהור DNA 3.; 4. Sonication; 5. הכנת הספרייה וסדר; ו 6. ניתוח bioinformatic. שיטת CNR-התור היא גישה מהירה וקלה כי תוצאות מפות שכפולות מפורטות.
Introduction
שכפול הדנ"א יונקים קיים רגולציה הדוקה כדי להבטיח את השכפול המדויק של כל כרומוזום בדיוק פעם אחת במהלך מחזור התא. שכפול מתרחש על פי צו פיקוח הדוק - מספר אזורים גנומית גדולים (~ Mb) לשכפל בתחילת שלב S (מוקדם משכפלי תחומים) ואילו אזורים גנומית אחרים לשכפל מאוחר יותר באמצע או בשלב מאוחר (באמצע המשכפלים מאוחר תחומים) 1. רוב הגנום משכפל בעת ובעונה אחת בכל הרקמות (דומיינים TOR מכוננים), ואילו 30% - 50% של הגנום, משנה TOR שלה בין רקמות 2, במהלך התמיינות 3, 4, ובמידה פחותה גם במהלך הטרנספורמציה סרטן 5 . יתר על כן, אזורים הגנומי מסוימים לשכפל אסינכרוני 6, 7, 8, כלומר יש הבדלב TOR בין שני אללים.
TOR בקורלציה עם תכונות רבות גנומי epigenomic כולל רמות שעתוק, תוכן GC, המדינה הכרומטין, צפיפות הגן, וכו '1, 9. TOR קשורה גם קצב מוטציות וסוגים 10, 11 ולכן לא מפתיע, הפרעות של התכנית שהכפולה קשורות לסרטן 12, 13. הקשר הסיבתי בין TOR ומבנה הכרומטין אינו מובן עדיין. יתכן כי הכרומטין פתוח הופך לפשוט שכפול מוקדם. עם זאת, מודל אלטרנטיבי עולה כי הכרומטין הוא כינס במהלך שכפול ואת הרגולטורים הכרומטין שונים שנכחו את ההתחלה ואת הסוף של עופרת S שלב דיפרנציאלי אריזה של אזורים מעתיק מוקדמים ומאוחרים 1, 14 . הראינו לאחרונה כי TOR מעצבת את התוכן GC על ידי המשפיע על סוג של מוטציות המתרחשות באזורים גנומי שונים 11.
קרינת הכלאה באתרו (FISH) היא השיטה העיקרית למדידת TOR ב לוקוסי פרט. הדבר מתבצע פשוט על ידי ספירת אחוז התאים בשלב S כי תערוכת אותות דג בודדים לעומת אחוז הכפילויות עבור נתון אלל 15, 16. שיטה חלופית, מורכב הדופק תיוג DNA עם BrdU, מיון תאים על פי תוכן DNA שלהם לנקודות זמן מרובים לאורך S, immunoprecipitating DNA המכיל BrdU, ובדיקת שפע של DNA זירז עם qPCR 17.
ניתן להשיג מיפוי הגנום TOR על ידי שתי שיטות. השיטה הראשונה היא גרסה הגנומי של שיטה המבוססת BrdU-IP שתואר לעיל, שבו כימות של סכוםDNA של זירז בכל שבריר נעשה בו זמנית עבור הגנום כולו באמצעות הכלאה כדי microarrays או רצף עמוק. השיטה השנייה, CNR-תור, מבוססת על מדידת מספר עותק של כל אזור גנומי של תאים בשלב S ו נרמול ידי תוכן DNA בתאים G1. בשיטה זו, תאים מסודרים על ידי FACS אל הלא מעתיק (שלב G1) משכפלים (שלב S) קבוצות (איור 1). תאים ב- G1 יש את אותו מספר עותק בכל האזורים הגנומי ובכך תוכן DNA שלהם צריך להיות זהה. מצד השני, מספר עותק דנ"א S תלוי TOR, מאז אזורים מעתיקים מוקדם עברו שכפולים ברוב התאים ולכן תוכן DNA שלהם מוכפל, ואילו האזורים מעתיקים מאוחר לא משוכפלים עדיין ברוב התאים ולכן תוכן DNA שלהם יהיה להיות דומה לזה של תאי G1. מכאן S יחס G1 של תוכן DNA מעיד על TOR. כמות ה- DNA לכל איזור גנומי נמדדה באמצעות הכלאהmicroarrays או רצף עמוק 2, 8. יתרונותיה של שיטת CNR-התור יידונו גם.
מאמר זה מתאר את שיטת CNR-תור למיפוי הגנום TOR כמתואר באיור 2. הנייר דן את הפרטים הקטנים של התהליך כולו מגביית תאים עד הניתוח הבסיסי של התוצאות ויצירת מפות גנומי TOR. הפרוטוקול המתואר במאמר זה שבוצע בהצלחה על סוגי תאים שונים הגדלים בתרבית. שיפורים עתידיים של פרוטוקול זה יכול להוביל את המיפוי של TOR in vivo ו סוגי תאים נדירים.
Protocol
הערה: TOR יכול להימדד רק לגדול, תאים לא מסונכרנים. ההליך צריך להתחיל עם לפחות 1 - 2 x 10 6 מהירי תאים גדלים, אשר בדרך כלל יגרמו ~ 1 x 10 5 תאים בשלב S (צעד הגבלת השיעור). מומלץ לנהל כל ניסוי באמצעות שניים או שלושה משכפל. התהליך כולו של CNR-תור ניתן להשלים בתוך שבוע - ימים אמורים להיות מוקדשים לכל השלבים עד כנת ספרייה, יום או ימים נדרשים עבור סידור ויום נוסף הוא הכרחי עבור ניתוח נתונים הראשוני.
1. איסוף של תאים מן התרבות
הערה: הפרוטוקול נכתב עבור התאים הגדלים בתרבית 10 צלחות ס"מ (המכיל כ 2 - 5 x 10 6 תאים), אך ניתן להתאימו בקלות לפלטפורמות אחרות.
- עבור תאים שגודלו השעיה, המשך קיבעון (סעיף 2).
- עבור תאים חסידים, לשאוב ולשטוף את plמ"ל PBS אכל עם 3 ללא Ca 2 + ו Mg 2+.
- בטל PBS ו דגירה התאים למשך 5 דקות ב 37 מעלות צלזיוס עם 1-EDTA טריפסין מסחרי מ"ל עד תאים לנתק.
הערה: משך הטיפול טריפסין צריך להיות מותאם לכל סוג תא. - הוסף 3 התקשורת והתרבות מ"ל לנטרל את טריפסין ולאסוף תאים צינור חרוטי 15 מ"ל או צינור קלקר 5 מ"ל. שמור על הקרח.
קיבוע 2.
הערה: עבור חלק זה, כל השלבים צריכים להיעשות על 4 מעלות צלזיוס.
- צנטריפוגה התאים ב XG 300 במשך 5 דקות ב 4 ° C..
- לשאוב ולשטוף תאים פעמיים עם 1 מ"ל קר PBS.
- תאים Resuspend ב 250 μL (סה"כ) PBS קר.
- בעוד vortexing את הצינור בעדינות, לאט להוסיף dropwise 800 μL של -20 ° C אתנול 100%. זה מוביל לריכוז אתנול סופי של 70 - 80%.
הערה: אתנול טוהר גבוה מומלץ בשלב זה. - דגירת תאים על קרח FOr 30 דקות.
הערה: בשעה תאים בשלב זה יכול להישמר במשך כמה ימים על 4 מעלות צלזיוס או במשך כמה חודשים ב -20 ° C.
3. PI מכתים
- צנטריפוגה התאים ב XG 500 עבור 10 דקות ב 4 °.
- לשאוב supernatant בזהירות לשטוף תאים פעמיים עם 1 מ"ל קר PBS.
- לשאוב ו resuspend כל המדגם עם תערובת הבאים: 1 מ"ל PBS, 5 μL 10 מ"ג / מ"ל RNaseA, 50 μL 1 מ"ג / מ"ל יודיד propidium (PI; לערבב הבקבוק לפני השימוש). התאם ריכוז עד ~ 2 x 10 6 תאים / מ"ל).
הערה: יש להרחיק מהישג אור - PI הוא רגיש לאור. - סינון דרך 35 מיקרומטר רשת לצינור קלקר 5 מ"ל, וקרוב עם Parafilm.
- לדגור על RT בחושך, במשך 15 - 30 דקות.
הערה: תאים מוכנים כעת לניתוח FACS. במידת הצורך, ניתן לאחסן את התאים מוכתם עבור 24 שעות לפחות ב 4 ° C בחושך.
מיין 4.
- תאי מיין באמצעות מכונת FACS. לָנוּדואר הלייזר 561 ננומטר להבדיל תאים בהתבסס על עוצמת PI שלהם. לייזרים אחרים לקראת מקסימום עירור 535 ננומטר כמו 488 ננומטר או 532 ניתן להשתמש בהתאם לתצורת מכשיר FACS.
- לקבלת תוצאות אופטימליות, השתמש הזרבובית הקטנה מומלצת בגודל התאים הספציפי (לרוב תאי 85 מיקרומטר). תיעדוף מצבים טוהר מעל התשואה. השתמש זרימה איטית יציבה, בדרך כלל עד 300-500 אירועים / s, עם לחץ נדן של 45 psi.
- באמצעות gating, להפלות תאים מתים ופסולת subcellular (FCS נמוך וגבוה SSC) על ידי התוויית FCS vs SSC. מן התאים קיימא להפלות כפילויות על ידי התוויית SSC-רוחב (W) לעומת SSC-גובה (H) ואחריו FSC-W לעומת העלילה FSC-H ועל ידי נרדף למלק W vs PI-H (כפילויות יהיה אותו ערך H אבל W-ערך גדול). עבור תאים בודדים קיימא לצייר היסטוגרמה של עוצמת PI-שטח (א) המייצגת את תוכן ה- DNA של התאים.
- תאים מיין לשלבים G1 ו- S, כפי שמוצג באיור 1. Gating עבור S צריך להיות רחב להפריע לשלבים G1 ו G2, בעוד gating G1 צריך להיות צר וככל מ S ככל האפשר.
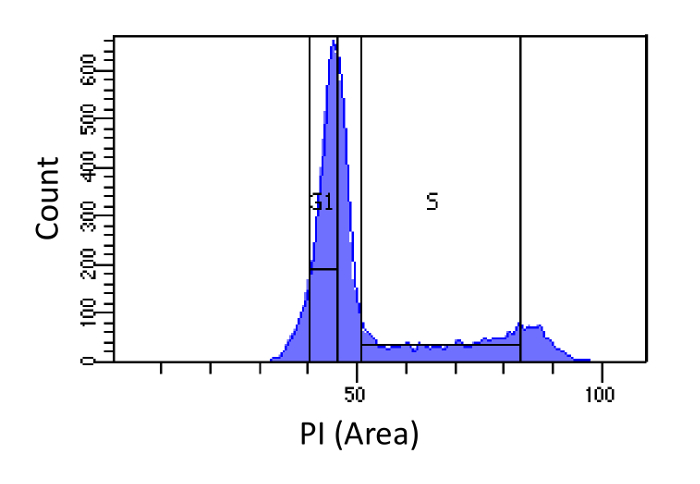
נחישות באיור 1. שלב מחזור התא מבוסס על עוצמת PI. היסטוגרמה המציגה את התפלגות תוכן DNA התאי (נמדד על ידי PI-Area) של האוכלוסייה פיברובלסטים עכבר עובריים (MEF). תוכן DNA משמש כדי למיין את האוכלוסייה לשתי תאי G1 תת אוכלוסיות i) (תוכן 2N DNA) ו- II) תאים בשלב S (2N - 4N תוכן DNA), באמצעות האזורים המסומנים. אנא לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
הערה: מטרת האוסף של תאי G1 היא לתת דין וחשבון על הטיות היעילות הרצף בין אזורים גנומית השונים. אפליקציה חלופיתמקק הוא להשתמש בתאי נעצר G1 מאותו סוג התא. גישה זו נותנת תוצאות נקיות (כי יקטין זיהום שלב S) אבל זה עשוי להציג הטיות נובעות הבדלים גנטיים בין התאים נעצרו התאים נמדדים.
- מיין בתנאים קרים ולאסוף תאים ממוינים לתוך 1.5 / 2 צינורות מ"ל. שמור צינורות על קרח בעקבות המין.
הערה: כדי לשפר את התאוששות DNA, עדיף להשתמש צינורות מחייב נמוכים או להשתמש צינורות מצופים 4 - 5% BSA 1 - 3 שעות ב 4 ° C 18.
5. ה- DNA טיהור
- עבור כל דגימה (G1 ו- S) לטהר DNA באמצעות ערכת טיהור DNA.
הערה: בעת שימוש בערכה המסחרית, elute עם 400 μL חיץ elution לתוך צינור 2 מיליליטר עבור תשואה גבוהה, כפי שהומלץ על ידי היצרן. - בדוק ריכוז ה- DNA באמצעות fluorometer.
הערה: החל מ 100,000 בתאי יונקים שגבתה FACS, אחד צריך לקבל ~ 1 מיקרוגרם של ה- DNA. אt זה DNA בשלב יכול להישמר במשך כמה ימים על 4 מעלות צלזיוס או ב -20 ° C לאחסון ארוך.
6. Sonication
- העבר DNA לתוך צינור 1.7 מיליליטר התואם את המעמד המגנטי המשמש.
- תתרכז DNA באמצעות חרוזים 2x SPRI פי הוראות היצרן, תוך שימוש לעמוד מגנטי, ו elute במאגר elution 50 μL.
- DNA Shear עם ultrasonicator ממוקד לגודל שיא היעד ממוצע של 250 נ"ב. השתמש בהגדרות הבאות עבור דגימת DNA 50 μL: 50 W, גורם Duty 20%, 200 מחזורים פרץ, 20 ° C, 120 s.
- בדוק את הגודל של ה- DNA טעון על ידי אלקטרופורזה. התפלגות הגודל המומלצת היא 200 - 700 נ"ב עם שיא ב ~ 250 נ"ב.
הערה: בשעה DNA בשלב זה יכול להישמר במשך כמה ימים על 4 מעלות צלזיוס או ב -20 ° C לאחסון ארוך.
כנת הספרייה 7., סידור
הערה: הכנת ערכות ספרייה רבות ושונהפלטפורמות רצף ent אמורות לעבוד, זהה לאלו המשמשים אותנו ואת שהוזכרו בסעיף החומרים. בפועל בעבר, מפות TOR נוצרו בשיטה דומה מאוד עם פלטפורמות microarray 2.
- הכן ספריות באמצעות כל ערכת כנת הספרייה מסחרית.
- בסוף הכנה הספרייה, בחר גודל באמצעות חרוזים מגנטיים עבור 300 - 800 נ"ב.
- לאחר הכנת ספריות, למדוד ריכוז ה- DNA באמצעות fluorometer.
- למדוד את גודל ה- DNA באמצעות אלקטרופורזה.
- בצע רצף על כל פלטפורמה.
הערה: רצף לפחות 10 M קוראים לדגימה מומלצת. עומק זה שווה לקרוא בערך כל 300 נ"ב (עבור ~ 3 הגנום גודל Gb), והוא מספיק למדידות TOR ברזולוציה של 50 - 100 kb. הגדלת העומק יגרום לירידת גודל החלון ובכך תאפשר הגדלת רזולוציה בודאות גבוהה. רצף סוף לזווג אין צורך בבפרוטוקול זה משום שמידע כיסוי רק נאסף. זה, לעומת זאת, עשוי לעזור בפתרון המיקום של קורא המכילים רצפים חוזרים.
ניתוח 8.
הערה: ניתוח נתונים מבוסס על השיטה על ידי אל א קורן et. 19.
- נתונים רצפו מפת הגנום התואם באמצעות bowtie2 או כל aligner קריאה קצרה אמינה. הגדר בגודל משתנה, חלונות כרומוזומליות שווה-כיסוי כמגזרים מכוסה על ידי 200 קורא בשבריר G1 ולספור שלב S קורא באותה חלונות.
- חשב את יחס S / G1 לכל חלון. זה אמור ליצור מפה עם תנודות גדולות יחס S / G1 לאורך הגנום (איור 3). שליטה טובה על האמינות של מדידות TOR היא להשוות את המפה הזאת ליחס G1 / G1 (משתי מדידות נפרדות של G1) שאמורה להיות להחמיא הרבה.
- לנרמל את הנתונים ל -0 מתכוון 1 SD על ידי הפחתת כל נalue הערך הממוצע של כל החלונות (למעט כרומוזום X) ו בחלוקת תוצאות לפי סטיית התקן של ה- G1 S / של כל החלונות. הדבר נעשה על מנת להמיר ציוני z ולאפשר השוואה בין ניסויים שונים.
- הסר כל אזורי הפער הרשומים על ידי דפדפן הגנום UCSC וכן כל שבר פער בין נותרים המכילים פחות מ -15 חלונות נתונים.
- חלק מה את השאריות עם שגם החלקה מעוקבת דרך csaps פונקצית Matlab עם פרמטר 10 - 16 ו לשרבב בנקודות סט כל 100 kb.
הערה: פרמטרים של החלקה אינטרפולציה צריך להיות מותאם בהתאם לעומק של הנתונים. שיטות ופונקציות החלקה מתאימות אחרות קיימות וניתן להשתמש בו. - לאחר חזותי המאשר את האמינות של כל לשכפל, למזג את כל הקורא ולחשב פרופיל ברזולוציה עמוק על ידי ביצוע אותו התהליך שתואר לעיל על נתונים אלה.
תוצאות
מפת TOR טיפוסית מוצגת באיור 3 עבור פיברובלסטים עובריים בעכבר (MEFs). נתון זה ממחיש את תהליך הניתוח שכן הוא מראה גם את הנקודות, אשר יחס S / G1 המנורמל עבור חלונות בודדים (שלב 8.3), כמו גם את הקו נובע החלקה מהעוקבת אינטרפולציה (שלב 8.5).
Discussion
CNR-תור יכול להתבצע באופן עקרוני על כל אוכלוסיית תאים מתרבים האיקריוטים כי ניתן לחלק ידי FACS ל S ושלבי G1 (נבדק על ידי Rhind נ וגילברט DM 20). השיטה המתוארת כאן הותאמה בתאי יונקים עם גודל הגנום של ~ 3 Gb כגון אדם ועכבר. שינויים קטנים בפרוטוקול CNR-התור (בהכנת תא ועומק רצ?...
Disclosures
No conflicts of interest declared.
Acknowledgements
אנו מודים אוריה ורד לסיוע ביצירת דמויות. עבודה בקבוצה היא נתמכה על ידי הקרן הלאומית למדע (567/10 מס מענק) ואת המועצה האירופית למחקר החל גרנט (# 281,306).
Materials
| Name | Company | Catalog Number | Comments |
| PBS | BI (Biological Industries) | 02-023-1A | |
| Trypsin-EDTA | BI (Biological Industries) | 03-052-1B | |
| 15 mL conical tube | Corning | 430790 | |
| 5 mL Polystyrene round Bottom tube with cell strainer cap | BD-Falcon | 352235 | |
| Ethanol | Gadot | 64-17-5 | |
| RNAse-A 10 mg/mL | Sigma | R4875 | |
| Propidiom iodide 1 mg/mL | Sigma | P4170 | |
| Parafilm | Parafilm | PM-996 | |
| 1.5 mL DNA LoBind Eppendorf tubes | Eppendorf | 22431021 | |
| BSA | Sigma | A7906 | |
| 1.7 mL MaxyClear tube | Axygen | MCT-175-C | |
| magnetic beads - Agencourt AMPure XP | Beckman Coulter | A63881 | |
| Ultrasonicator | Covaris | M-series -530092 | |
| 50 µL microTUBE AFA Fiber Screw-Cap 6 x 16 mm | Covaris | 520096 | |
| Qubit fluorometer | Invitrogen | ||
| Qubit dsDNA High Sensitivity (HS) Assay Kit | Invitrogen | Q32854 | |
| Electrophoresis 2200 Tape station system | Agilent | D1000 ScreenTape | |
| Seqeuncing - Illumina NextSeq system | Illumina | SY-415-1001 | |
| Dneasy kit for DNA purification | Qiagen | 69504 | |
| PureProteom Magnetic Stand | Millipore | LSKMAGS08 | |
| Anti-BrdU/FITC | DAKO | F7210 | |
| FACS sorter | BD | FACSARIA III | |
| FACS software | BD | FACSDiva v 8.0.1 |
References
- Farkash-Amar, S., Simon, I. Genome-wide analysis of the replication program in mammals. Chromosome Res. 18 (1), 115-125 (2010).
- Yaffe, E., et al. Comparative analysis of DNA replication timing reveals conserved large-scale chromosomal architecture. PLoS Genet. 6 (7), e1001011 (2010).
- Hiratani, I., et al. Global reorganization of replication domains during embryonic stem cell differentiation. PLoS Biol. 6 (10), (2008).
- Rivera-Mulia, J. C., et al. Dynamic changes in replication timing and gene expression during lineage specification of human pluripotent stem cells. Genome Res. 25 (8), 1091-1103 (2015).
- Ryba, T., et al. Abnormal developmental control of replication-timing domains in pediatric acute lymphoblastic leukemia. Genome Res. 22 (10), 1833-1844 (2012).
- Farkash-Amar, S., et al. Global organization of replication time zones of the mouse genome. Genome Res. 18 (10), 1562-1570 (2008).
- Koren, A., McCarroll, S. A. Random replication of the inactive X chromosome. Genome Res. 24 (1), 64-69 (2014).
- Mukhopadhyay, R., et al. Allele-specific genome-wide profiling in human primary erythroblasts reveal replication program organization. PLoS Genet. 10 (5), e1004319 (2014).
- McNairn, A. J., Gilbert, D. M. Epigenomic replication: linking epigenetics to DNA replication. Bioessays. 25 (7), 647-656 (2003).
- Sima, J., Gilbert, D. M. Complex correlations: replication timing and mutational landscapes during cancer and genome evolution. Curr Opin Genet Dev. 25, 93-100 (2014).
- Kenigsberg, E., et al. The mutation spectrum in genomic late replication domains shapes mammalian GC content. Nucleic Acids Res. 44 (9), 4222-4232 (2016).
- Woo, Y. H., Li, W. H. DNA replication timing and selection shape the landscape of nucleotide variation in cancer genomes. Nat Commun. 3, 1004 (2012).
- Liu, L., De, S., Michor, F. DNA replication timing and higher-order nuclear organization determine single-nucleotide substitution patterns in cancer genomes. Nat Commun. 4, 1502 (2013).
- Goren, A., Cedar, H. Replicating by the clock. Nat Rev Mol Cell Biol. 4 (1), 25-32 (2003).
- Selig, S., Okumura, K., Ward, D. C., Cedar, H. Delineation of DNA replication time zones by fluorescence in situ hybridization. EMBO J. 11 (3), 1217-1225 (1992).
- Smith, L., Thayer, M. Chromosome replicating timing combined with fluorescent in situ hybridization. J Vis Exp. (70), e4400 (2012).
- Simon, I., et al. Asynchronous replication of imprinted genes is established in the gametes and maintained during development. Nature. 401 (6756), 929-932 (1999).
- Phi-Wilson, J. T., Recktenwald, D. J. Coating agents for cell recovery. Google Patents. , (1993).
- Koren, A., et al. Differential relationship of DNA replication timing to different forms of human mutation and variation. Am J Hum Genet. 91 (6), 1033-1040 (2012).
- Rhind, N., Gilbert, D. M. DNA replication timing. Cold Spring Harb Perspect Biol. 5 (8), a010132 (2013).
- Koren, A., et al. Genetic variation in human DNA replication timing. Cell. 159 (5), 1015-1026 (2014).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved