A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
דנ א של מיקרולוווין הגנוזות וזרימה Cy, בדיקות פלוטודיה של פורמאלין-קבוע פרפין-מוטבע רקמות טוחנת
In This Article
Summary
שומות hydatidiform הם הריונות אנושיים חריגים עם aetiologies הטרוגנית שניתן סיווג על פי תכונות מורפולוגיות שלהם תרומת הורים הגנום הטוחנת. כאן, פרוטוקולים של הננו-DNA מיקרולוווין של דנ א וזרימה cy, הרקמה הקבועה של formalin-מוטבע הרקמות הטוחנת מתוארים בפרוטרוט, יחד עם התוצאות ' פרשנות ואינטגרציה.
Abstract
Hydatidiform שומה (HM) הוא הריון אנושי נורמלי מאופיין התפשטות trophoblastic מוגזמת ופיתוח עובריים חריגים. ישנם שני סוגים של HM מבוסס על הערכה מורפולוגית מיקרוסקופית, להשלים HM (CHM) חלקי HM (PHM). אלה יכולים להיות מחולקת עוד יותר על בסיס תרומת הורים הגנום הטוחנת. אפיון כזה של HM, לפי מורפולוגיה וניתוחי גנוטיפ, הוא חיוני לניהול המטופלים ולהבנה הבסיסית של הפתולוגיה הזאת מעניינת. זה מתועד היטב כי ניתוח מורפולוגית של HM הוא כפוף השונות הבינמתבונן רחב והוא לא מספיק על עצמו כדי לסווג במדויק HM לתוך CHM ו PHM ולהבדיל אותם מפני הפלות הלא טוחנת הידרופית. ניתוח הקלדה מבוצע בעיקר ב-DNA ורקמות מ-formalin קבוע פרפין-מוטבע (FFPE) מוצרים של תפיסה, אשר יש פחות מאשר איכות אופטימלית ולכן עלול להוביל למסקנות שגויות. במאמר זה, פרוטוקולים מפורטים עבור מולטיטפלקס והזרמת cy, ניתוח הרקמות הטוחנת של FFPE מסופקים, יחד עם הפרשנות של תוצאות שיטות אלה, פתרון בעיות שלהם, אינטגרציה עם הערכה מורפולוגית , p57KIP2 אימונוהיסטוכימיה, ו זריחה באתרו היברידיזציה (דג) כדי להגיע לאבחנה נכונה ואיתנה. כאן, המחברים חולקים את השיטות והלקחים שנלמדו במהלך 10 השנים האחרונות מהניתוח של כ 400 מוצרים של ההתעברות.
Introduction
שומה מולה (HM) הוא הריון אנושי נורמלי המאופיינת התפתחות עובריים חריגים, היפרהפצה של trophoblast פיצוץ, ו הידרוpic ניוון של villi כוריוני (CV). מבחינה היסטורית, HM היה מחולק לשני סוגים, להשלים HM (CHM) חלקי HM (PHM) מבוסס רק על הערכה מורפולוגית1. עם זאת, הוכח כי הערכה מורפולוגית לבד אינה מספיקה כדי לסווג HM לתוך שני סוגי המשנה (CHM ו-phm) ולהבדיל אותם מפני הפלות לא-טוחנת2,3,4.
בגלל CHM ו PHM יש נטיות שונים כדי ממאירות, ולכן חשוב לקבוע במדויק את סוג גנוטיפ של HM כדי לספק מעקב מתאים וניהול לחולים. כתוצאה מכך, בעשורים האחרונים פותחו מספר מתודולוגיות והתפתחו לצורך זיהוי התרומה הורית לרקמות הטוחנת ולהשגת סיווג נכון של HM. אלה כוללים ניתוח קריוטיפ, פולימורפיזם פסים כרומוזומלית, אנטיגן לוקיציט אנושי (hla) סרולוגית הקלדה, מגבלת אורך קטע ההגבלה, מספר משתנה של הטנדם חוזר, גנוטיפ מיקרולוווין, הזרמת cy, ו p57 KIP2 אימונוהיסטוכימיה. זה איפשר חלוקה מדויקת של התפיסות HM מבוסס על תרומת הורים כדי genomes שלהם, כדלקמן: CHM, אשר דיפלואידי דמוי מונוספרמיק או דיפלואידי אנדרוגנטי, ו phm, אשר הם triploid, מפזרים ב 99% ו מונוספרמיק ב 1% מהמקרים5,6,7,8. יתר על כן, יש עוד סוג של גנוטיפ של HM אשר התפתחה בשני העשורים האחרונים, אשר הוא דיפלואידי biparental ורים. האחרון הוא בעיקר וחוזר ועלול להשפיע על בן משפחה בודד (במקרים משפחתיים) או לפחות שני בני משפחה. אלה דיפלואידי הורים שומות נגרמות בעיקר על ידי מוטציות רצסיבי ב NLRP7 או KHDC3L בחולים9,10,11,12. Diploid ביורית HM בחולים עם מוטציות רצוניות ב NLRP7 ניתן לאבחן כמו CHM או phm על ידי ניתוח מורפולוגית וזה נראה קשור לחומרת המוטציות בחולים13,14. בנוסף לסיווג של HM על פי הגנוטיפים שלהם, המבוא והשימוש של מספר שיטות גנוסטי מותר את ההבחנה של ישויות טוחנת שונות מפני הפלות שאינן מולרי, כגון תפיסות הורים ו תפיסות מסוגים אחרים5,15. תפיסות כאלה אולי יש כמה התפשטות trophoblast פיצוץ ומורפולוגיה villous חריגים המחקים, במידה מסוימת, כמה תכונות מורפולוגיות של HM.
המטרה של מאמר זה היא לספק פרוטוקולים מפורטים עבור מולטיפלקס וזרימה cy, לזרום עם הרקמה הקבועה של הפרפין-מוטבע (FFPE) רקמות, וניתוחים מקיפים של תוצאות שיטות אלה ושילובם עם שיטות אחרות עבור אבחנה נכונה וחותכת של רקמות טוחנת.
Protocol
מחקר זה אושרה על ידי הלוח סקירה מוסדית מקגיל. כל המטופלים סיפקו הסכמה בכתב להשתתף במחקר ולקבל מוצרים FFPE שלהם של תפיסה (POCs) אוחזרו ממחלקות פתולוגיה שונים.
הערה: בעוד ישנן מספר שיטות לקביעת הגנוזות ולהגדרה פלויידי על ידי הזרימה cy, הפרוטוקולים המסופקים כאן לתאר שיטה אחת של ניתוח באמצעות פלטפורמה אחת לכל.
1. הקלדת הגנוזה
-
בחירה של בלוק FFPE הטוב ביותר
- עבור כל מוצר FFPE של תפיסה (POC), להכין 4 μm-המטאוקסילין עבה ו אאוזין (H & E) סעיפים מוכתם כמתואר סעיפים 1.2 ו 1.3, אחד עבור כל בלוק זמין, עבור הערכה מורפולוגית על ידי מיקרוסקופ.
- באמצעות שקופיות H & E ומיקרוסקופ אור, בחר את בלוק FFPE כי יש את הכמות הגדולה ביותר של villi כוריוני (CV), ואם אפשרי, את הבלוק כי יש קורות חיים נפרד, ולא התערבבו עם, רקמות אימהי.
-
חלוקתה
- מניחים את הבלוק הנבחר על הקרח 15 דקות כדי להקל על החסימה.
- להתאים את המיקרוטומה כדי לגזור סעיפים כי הם 4 יקרומטר עבה עבור הערכה מורפולוגית מיקרוסקופית 10 יקרומטר עבה עבור חילוץ DNA.
- מניחים את הגוש הקר במיקרוטומה וחותכים קטע אחד מכל בלוק לצביעת H & E ו -10-30 סעיפים מהבלוק הנבחר, בהתאם לכמות קורות החיים בבלוק, להפקת דנ א.
הערה: עבור בלוקים מלאים קורות חיים, 10 סעיפים מספיקים להפקת DNA. אם רק כ 10% של הבלוק מכיל קורות חיים בעוד השאר הם רקמות אימהי, אז 20 על 30 סעיפים נדרשים כדי להבטיח כמויות מספיקות של דנ א. - באמצעות מלקחיים, להעביר כל קטע לאמבט מים 45 ° c. הרם את החלק ממרחץ המים עם שקופית טעונה באופן חיובי (טבלת חומרים) שמסומנת בעבר עם מספר הזיהוי לדוגמה באמצעות עיפרון.
- הצב את השקופיות המכילות את המקטעים בתנור ב-65 ° צ' כדי לאפשר למקטעים לדבוק בשקופיות. שמור את השקופיות עבור H & E בתנור במשך 25 דקות. שמור את השקופיות להפקת דנ א בתנור במשך 20 דקות.
הערה: זמן הדגירה קצר יותר הופך את הרקמות מעט פחות מחסיד לשקופיות וכתוצאה מכך מקל על הסרת רקמות אימהי.
-
כתמים של H & E
- אפשר לשקופיות להתקרר לטמפרטורת החדר (10 דקות).
- הכנה מגיב
- להכין פתרון עבודה אאוזין Y (0.25%) כלפי שולחן 1. מערבבים היטב ומאחסנים בטמפרטורת החדר.
- הכנת פתרון המטאוקסילין על ידי דילול תמיסת מניות של המטאוקסילין 5x במים (כלומר, מערבבים 80 מ ל של מים עם 20 מ ל של המטאוקסילין).
הערה: לעטוף את מלאי התמיסה של המטאוקסילין בנייר כסף לאחסון.
- הכינו צנצנות מכתים עם הריאגנטים הנכון מתחת למכסה המנוע לפי שולחן 2.
- בצע את הצביעת ה-H & E על-ידי מיזוג השקופיות לתוך צנצנות המכתים המתאימות לתקופת הזמן הנכונה בהתאם לטבלה 2.
- הר 4 יקרומטר סעיפים עבור ניתוח מורפולוגית עם הרכבה בינונית ו coverslip עם שמיכות זכוכית (טבלת חומרים).
הערה: הסעיפים 10 יקרומטר עבור הקלדה לא צריך להיות שמיכות. - השאירו את הסעיפים 10 יקרומטר תחת מכסה המנוע עבור מינימום של 3 h על מנת להפיג את הריחות הרעילים כדי להתפזר.
זהירות: כל צעדי הצביעה צריכים. להתבצע תחת מכסה המנוע מוצרי קסילן צריך להישמר מתחת למכסה המנוע כל הזמן, כי קסילן ריחות רעילים. כמו כן, יש להיפטר מקסילין וממטאוקסילין במכולות מיוחדות. ברגע שהמכולות הללו מלאות, יש להיפטר ממנו כפי שמומלץ על ידי ארגון הבטיחות של המעבדה.
| ריאגנט | כמות |
| פתרון מניות אאוזין Y (1%) | 250 מ ל |
| 80% אתנול | 750 מ ל |
| חומצה אצטית קרחוני (מרוכז) | 5 מ ל |
טבלה 1: אאוזין Y פתרון עבודה (0.25%) כנה.
| מגיב בשימוש (100 mL לכל סל) | משך |
| 1) קסילן | 5 דקות |
| 2) קסילן | 5 דקות |
| 3) 100% אתנול | 2 דקות |
| 4) 95% אתנול | 2 דקות |
| 5) 70% אתנול | 2 דקות |
| 6) 50% אתנול | 2 דקות |
| 7) מים מזוקקים | 5 דקות |
| 8) המטאוקסילין | 4 דקות |
| 9) מים מזוקקים | 5 דקות |
| 10) אאוזין | 1 דקות |
| 11) 95% אתנול | 5 דקות |
| 12) 100% אתנול | 5 דקות |
| 13) קסילן | 5 דקות |
| 14) קסילן | 5 דקות |
שולחן 2: ריאגנטים ומשכים עבור פרוטוקול הצביעת H & E.
-
בידוד קורות חיים
- תחת stereomicroscope אור, השתמש מלקחיים וחתיכות קטנות של מגבונים נייר הלחלח (לוח חומרים) כדי לגרד רקמות אימהי לא רצויות מ-H & E-מוכתם 10 יקרומטר חלקים עבים.
הערה: המטרה הסופית היא לשמור רק על קורות חיים או ממברנות עוברי (כאשר קיים) על השקופיות ובכך להסיר את כל הרקמות האחרות. שלב זה עשוי להזדקק להרבה זמן וסבלנות, בהתאם לבלוק, כפי שהוא דורש תשומת לב קפדנית לפרטים. - יש אדם שני לבדוק את השקופיות לאחר ניקוי כדי לוודא שהם חופשיים של רקמות אימהי.
- צלם תמונות של השקופיות הנקיות או תעד את הפרטים הבאים כדי לסייע בפענוח נתונים: 1) אם הרקמה הייתה קשה לניקוי, דימום או ניקוי מאוד, 2) מספר הסעיפים המשמשים ו-3) הכמות המשוערת של רקמות נקיות.
הערה: איור 1 מספק דוגמה לשקופית קלה לניקוי. עבור גוש המכיל בערך כמות זו של קורות חיים, 10 סעיפים מספיקים להפקת DNA. השקופית באיור 2 יש מעט מאוד קורות חיים כי הם התערבבו עם רקמות אימהי, מה שהופך אותו קשה מאוד זמן רב לנקות. עבור גוש המכיל בערך כמות זו של קורות חיים, 30 סעיפים נחוצים להפקת ה-DNA. - לאסוף את קורות החיים באמצעות פיסות מגבונים קטנים לחלח. באמצעות מלקחיים, לקרוע חתיכה קטנה מתוך מגבונים לחלח ולהשתמש בו כדי לאסוף את קורות החיים.
- מניחים את פיסות הנייר עם קורות החיים המצורפים שלהם לתוך שפופרת מתויג 1.5 mL.
- מזער את כמות מגבונים הנייר המשמשים בשלב זה כמו יותר מדי עשוי לסתום את עמודת החילוץ DNA וכתוצאה מכך להקטין את הסכום הסופי של ה-DNA שנאסף. בממוצע, המטרה היא להשתמש בפחות משבעה פיסות נייר קטנות לכל מדגם. אם זה לא אפשרי עקב נוכחות של כמויות גדולות של קורות חיים, לפצל את המדגם בין שתי שפופרות כדי להקל על החילוץ.
- תחת stereomicroscope אור, השתמש מלקחיים וחתיכות קטנות של מגבונים נייר הלחלח (לוח חומרים) כדי לגרד רקמות אימהי לא רצויות מ-H & E-מוכתם 10 יקרומטר חלקים עבים.

איור 1: שקופית ייצוגית להקלדת הגנוזה. למעלה: שקופית שצריך "לנקות" כדי להיות חופשי של רקמות אימהי. בתחתית: אותה שקופית המוצגת לאחר ניקוי וכעת היא מכילה רק קורות חיים עבור חילוץ ה-DNA. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
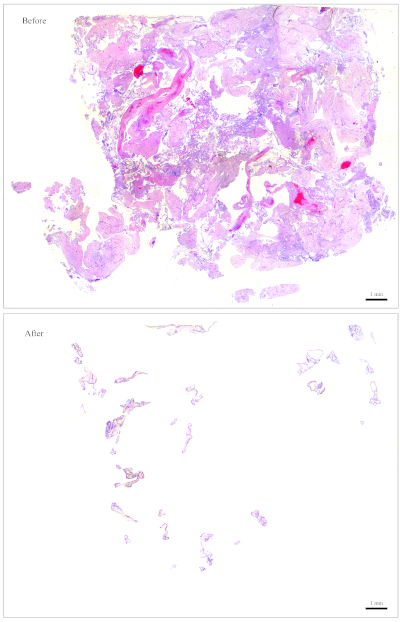
איור 2: שקופית ייצוגית להקלדת הגנוזה. למעלה: שקופית שצריך "לנקות" כדי להיות חופשי של רקמות אימהי. בתחתית: אותה שקופית המוצגת לאחר ניקוי וכעת היא מכילה רק קורות חיים עבור חילוץ ה-DNA. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
- בצע את הפרוטוקול של חילוץ ה-DNA מערכת FFPE (לוח חומרים) כדי לבצע חילוץ dna.
הערה: ערכות מסוימות ממליצים על שימוש ב-15 עד 20 μL של מאגר הימנעות עבור האחרון. מניסיון, הימנעות עם 15 μL של מאגר הימנעות עובד היטב עבור רוב הדגימות. דילול עשוי להיות מוכן DNA מלאי לפי הצורך.
-
כימות דנ א
- באמצעות מכשיר ספקטרוסקופיה מעבדה, טען 1 μL של DNA ולמדוד ספיגת bance ב 260 nm עבור כימות.
- טען 1 μL של ה-DNA על 2% העלה ג'ל והפעל אלקטרופורזה ג'ל במתח של 80-100 V להערכה איכותית.
- בהתבסס על התוצאות של שלבים 1.6.1 ו 1.6.2, לבחור את הנפח של ה-DNA כדי לשמש במגה-בתים קצרים החוזר המקביל (STR) פולימראז תגובת שרשרת (PCR) הגברה. המטרה להשתמש לפחות 1000 ng של ה-DNA בהגברה הPCR שלהלן.
הערה: איור 3 מדגים את הדוגמאות הנציגים של ג'ל יחד עם ריכוזי ה-dna (על בסיס התוצאות של הספקטרוסקופיה), ואת נפח של פתרון ה-dna כי הוא מומלץ עבור מולטיפלקס ה-PCR העוקב.
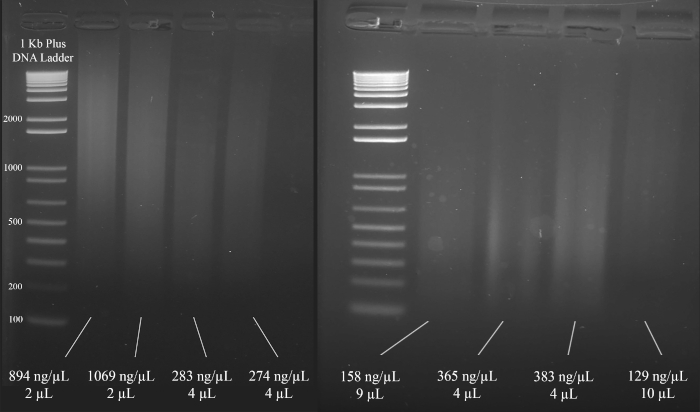
איור 3: ג'ל מייצג לקוונפיקציה של דנ א. כלולים הם ריכוזים של כל דנ א, כפי שנמדד באמצעות ספקטרוסקופיה, ואת הכמויות המשמשות את ה-PCR הקולנוע. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
-
הגברה הPCR
- ביצוע שימוש במיקרו-לווין פלורסצנט באמצעות מערכת STR מולטיקלבית (טבלת חומרים).
- השתמש בתנאי ה-PCR המוצגים באיור 4 עבור הגברה ה-pcr באמצעות מערכת ה-STR (טבלת חומרים).
הערה: התחל הבאים משמשים במערכת הקולנוע הזאת STR: D18S51, D21S11, TH01, D3S1358, פנטאה E, FGA, TPOX, D8S1179, vWA, Amelogenin, CSF1PO, D16S539, D7S820, D13S317, D5S818, ופנטאה D.

איור 4: התנאים של מחזור ה-PCR עבור מערכת STR. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
-
לפתור את מוצרי ה-PCR על ידי אלקטרופורזה קפילר.
- השהה 1 μL של כל מדגם מוגבר ב-0.5 μL של נתיב התקן הפנימי של המערכת במגה-בוני ו9.5 μL של המערך הגבוה ביותר (טבלת חומרים).
- הפעל דגימות דרך מכשיר אלקטרופורזה בכלי (לוח חומרים) באמצעות מטריצת הפרדה מתאימה (טבלת חומרים) לכלי הנגינה ולערכת הצבע של המערכת.
- ניתוח מידע
- לנתח את הנתונים עם התוכנה ניתוח קטע ה-DNA ולהשוות את POC אללים לאלס הורים כדי לקבוע את מקורם.
- התקן גודל.
הערה: הדבר מאפשר לתוכנה לזהות את הסולם המשמש במערכת ה-STR, ולהקצות צמדי בסיס להמגברה המבוססת על הסולם. השלבים הבאים מוגדרים עבור תוכנה ספציפית אחת (טבלת חומרים) אך עשויה לסייע להקמת סוגי תוכנה אחרים גם כן.- פתח את התוכנה. לחץ על התחל פרוייקט חדש ולאחר מכן בתקן גודל חדש.
- תן לתקן הגודל שם (לדוגמה, ABI_600).
- בתיבה בשם הזן הגדרה סטנדרטית בגודל חדש: הזן את הפרטים הבאים: 60, 80, 100, 120, 140, 160, 180, 200, 225, 250, 275, 300, 325, 350, 375, 400, 425, 450, 475, 500, 550, 600. לאחר מכן לחץ על הוסף גודל (ים).
הערה: המספרים שהוזנו יופיעו מתחת לתיבה שבצד ימין, הנקרא הגדרת תקן סטנדרטי (ראה איור 5). - לחץ על שמור.
- לייבוא ולניתוח קובץ, לחצו על ' הוסף קבצים' ובחרו בקובץ fsa לניתוח. לחץ על הוסף קבצים נבחרים ולאחר מכן על אישור. לאחר מכן בצע את השלבים הבאים:
- אתר את העמודה גודל רגיל ובחר באפשרות ABI_600 (או באיזה שם שניתן לתקן הגודל).
- תחת שיטת ניתוח, לחץ על גודל ברירת מחדל-npp ולאחר מכן לחץ על כפתור ירוק לנתח .
- הקובץ מוכן כעת לתצוגה. כוונן את אפשרויות הצפייה כדי להציג את הנתונים כרצונך.
- שיטת פתרון בעיות
הערה: התוכנה עלולה לעתים להיכשל בזיהוי פסגות וליישר אותן כראוי. זה קורה כאשר הפסגות נמוכות מדי או גבוהות מדי. שתי שיטות הניתוח הבאות יכולות לתקן זאת ויש לנסות לפני שמדגם נבדק מחדש.- שיטת ניתוח 1 לפסגות גבוהות:
- לחץ על שיטת ניתוח חדשה והשם אותו פסגות גבוה (או שם אחר לפי העדפה אישית).
- לחץ על טווח ולאחר מכן על טווח חלקי עבור ניתוח ושינוי גודל. לאחר מכן הקלד ב- 100 עבור נקודת ההתחלה וגודל ההתחלה.
- עבור נקודת העצירה, הזן 10,000. עבור גודל העצירה, הזן 1000.
- לאחר מכן לחץ על מינימום שיא גבהים ולשנות את המספרים כגון סף שיא עבור הצבעים הוא כדלקמן: כחול: 50; ירוק: 50; צהוב: 20; אדום: 100; כתום: 5000.
- שמור את שיטת הניתוח החדשה.
- שיטת ניתוח 2 לפסגות נמוכות:
- לחצו על ' שיטת ניתוח חדשה ' והשם לו ' פסגות נמוכות ' (או שם אחר לפי העדפה אישית).
- לחץ על טווח ולאחר מכן על טווח חלקי עבור ניתוח ושינוי גודל. לאחר מכן הקלד ב- 100 עבור נקודת ההתחלה וגודל ההתחלה.
- עבור נקודת העצירה, הזן 10,000. עבור גודל העצירה, הזן 1000.
- לאחר מכן לחץ על דגלי איכות ולשנות את טווח המעבר כך שהוא קורא מ 0.5 ל 1. שנה את טווח האיכות הנמוכה כך שהיא קוראת מ 0.0 עד 0.0. שינוי להניח יניאריות להלן: מ (bp) 100.0 (bp) 800.0.
- שמור את שיטת הניתוח החדשה.
הערה: עכשיו אפשר לנתח מחדש את הקובץ על ידי בחירת פסגות נמוך או פסגות גבוהה תחת שיטת ניתוח ולאחר מכן לחיצה על כפתור ירוק לנתח .
- שיטת ניתוח 1 לפסגות גבוהות:

איור 5: מסך המציג את העורך הסטנדרטי גודל. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
2. הזרמה הציקיתיים
-
בחירת בלוק האידיאל FFPE
- באמצעות שקופיות H & E ומיקרוסקופ קל, בחר בלוק FFPE בעל כ-50-70% מרקמות המורכב מקורות חיים.
הערה: איור 6 הוא דוגמה מייצגת של בלוק המתאים לניתוח cy, שהוא מורכב מ50% קורות חיים (המחצית הימנית של המקטע) ו 50% רקמות אימהי (שמאל חצי). הנוכחות של רקמות אימהי חשוב כי הם משמשים כשליטה פנימית לפסגת דיפלואידי. - עבור בלוקים שאין להם את הכמות האידיאלית של קורות חיים, להעשיר עבור קורות חיים כמו הציבור מבוצע. כדי לעשות זאת, לזהות איזה צד של סעיפים לחתוך טרי מכיל יותר קורות חיים על פי השקופית המתאימה H & E. בהתבסס על זה, להשתמש להב כדי לחתוך את החצי השני צריך להיות מושלך כדי להעשיר עבור קורות חיים.
הערה: איור 7 מציג בלוק שאין לו קורות חיים מספיקים עבור הזרימה cy, try. עבור בלוקים כגון זה, הסעיפים צריכים להיות גזור כזה כי החצי המכיל פחות קורות חיים מקבל נמחק על מנת להגדיל את כמויות של קורות חיים ביחס לרקמות אימהי, כפי שמוצג בדמות. הקפד לגזור חלקים נוספים כדי לפצות על מה שנמחק.
- באמצעות שקופיות H & E ומיקרוסקופ קל, בחר בלוק FFPE בעל כ-50-70% מרקמות המורכב מקורות חיים.
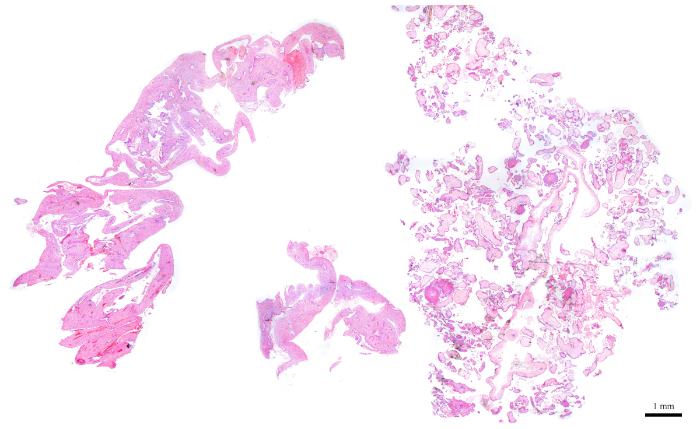
איור 6: H &Amp; E מקטע המייצג בלוק POC כי הוא אידיאלי עבור cy try זרימה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
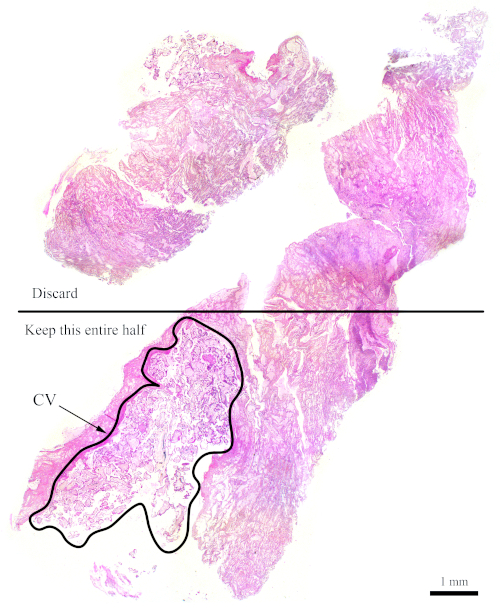
איור 7: H &Amp; E סעיף המייצג בלוק קשה יותר עבור cy, הזרימה הציטונסה. מקטע H & E של נציג זה מראה שרק במחצית התחתונה של סעיף זה יש להשתמש עבור הניתוח cy, לצורך זרימה, במטרה להעשיר את קורות החיים. אזור החלוקה לאזורים, המסומנת על-ידי קורות חיים, מורכב ברובו מקורות חיים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
- חלוקתה
- להשאיר את הבלוקים על הקרח 15 דקות כדי להקל על המחסומים.
- באמצעות לחסום ffpe הטוב ביותר, לחתוך ארבעה סעיפים כי הם 50 יקרומטר עבה (או 2 100 יקרומטר סעיפים עבים) באמצעות microtome.
הערה: כדי להזרים cy, עדיף להיות חלקים עבים יותר. - במקרה שבלוק FFPE אידיאלי אינו זמין, המטרה לשמור על היחס של קורות חיים לרקמות אימהי בכל זאת. לדוגמה, אם רק 30% של הבלוק מורכב קורות חיים בעוד השאר יש רקמות אימהי, ולאחר מכן להסיר לפחות חצי מהמקטע המכיל את רקמות אימהי ולהשתמש בסעיפים נוספים כדי לפצות (ראה איור 7).
- מניחים את המקטעים בשפופרות 15 מ ל.
הערה: הקפד להקליט מעל התוויות מכיוון שהריאגנטים האורגני המשמש בשלב הבא יכול להמיס ולהסיר דיו.
- הפרוטוקול הציטוטרי של זרימה מרקמות FFPE
- לחות והתחדשות
- בצע את השטף הבא (שולחן 3) תחת מכסה המנוע.
- ממלאים את הצינור 15 מ"ל עם 6 מ ל של הכימית המתאימה, בעקבות ההזמנה שהוצגה בטבלה 3, להשאיר את הסעיפים הריאגנטים עבור הזמן המתאים, ולאחר מכן להסיר את החומרים מגיב באמצעות שאיבה ואקום זכוכית פסטר.
- בין כל שלב, טבול את הפיפטה הפסטר הראשון ב-70% אתנול, ואז במים מזוקקים, ולאחר מכן המשך לשלב הבא.
- להיזהר מאוד לא להסיר פיסות רקמה יחד עם הכימית. להטות את השפופרת 15 מ ל לזווית 60 מעלות כדי להקל על שאיבה של מגיב נוזלי ללא רקמות הציור.
זהירות: הנוזלים הנמחקים מכילים קסילן ויש להיפטר ממיכלי פסולת קסילן.
- הכנה לפתרון
- הכינו את התמיסה הציטראט על ידי המסת 2 גרם של חומצת לימון ב-1 ל של מים מזוקקים כפולים. . הביאי את ה-pH ל -6 חנות ב -4 ° c.
- להכין פתרון פפסין על ידי המסת 0.01 g של פפסין ב 2 מ ל 9 חלקים לאלף הנאל, pH 1.64. . זה לדוגמא אחת
זהירות: פפסין רעיל ויכול בקלות להתפזר ולהיות מוטס. לחבוש מסכה בעת טיפול פפסין בצורת אבקה ולנגב את כל אזור העבודה לאחר השימוש בו. - Propidium יודיד (PI)-ribonuclease הכנה פתרון למדגם אחד.
- מערבבים 50 μL של PI עם 450 μL של PBS (כדי לדלל 10x).
- הוסף 50 μl של ריבונוקלאז A (1 מ"ג/mL) לתערובת. המשיכו לעטוף בנייר כסף כל הזמן.
- עיכול וכתמים
- הוסיפו מענה של 4 ° c מעלות ציטראט ל-15 הצינורות mL ומשם באמבט מים בגובה 80 ° c במשך 2 שעות.
- תנו לפתרון להתקרר לטמפרטורת החדר (15 דקות). הסר את התמיסה הציטראט.
- הוסף 6 מ ל של 1 x PBS, ומערבולת, והמתן 1 כדי 2 דקות כדי לאפשר לרקמות להתיישב בתחתית. הסירו את ה-PBS 1x באמצעות שאיבה ואקום וזכוכית פסטר.
- הוסף 1 מ ל של פתרון פפסין (מחומם ל37 ° c) ומניחים באמבט יבש בגודל 37 ° c למשך 30 דקות.. מערבולת כל 10 דקות הכן את ה-PI-ribonuclease פתרון ב -10 הדקות האחרונות של הדגירה.
- הוסף 6 מ ל של 1 x PBS, ומערבולת, והמתן 1 כדי 2 דקות כדי לאפשר לרקמות להתיישב בתחתית. הסירו את ה-PBS 1x באמצעות שאיבה ואקום וזכוכית פסטר.
- הוסף 550 μL של PI-ribonuclease פתרון ומניחים את הדגימות באמבטיה יבש 37 ° c עבור 30 דקות.
הערה: בשלב זה, ניתן לעטוף את הדגימות בנייר כסף ולהשאיר את הלילה בארבע מעלות צלזיוס עד הבוקר שלמחרת. - סנן את הפתרון באמצעות רשת סינון של 48-μm. לאסוף את פילטרט פוליסטירן התחתון שפופרות עגול, אשר ניתן להשתמש עם cytometer זרם. השתמש מלקחיים למקום 5 ס"מ על 5 ס מ פיסת סינון של רשת בחלק העליון של הצינור, כך הנוזל יכול להיות מצינור דרך הרשת לתוך הצינור.
הערה: הדגימות מוכנות כעת לפעול. עם הציטומטר הזורם השאר אותם עטופים בנייר כסף עד שהם מוכנים לפעול.
- הפעל דגימות עם cytometer זרם עם העזרה של הארגון cy, לנסות את טכנאי פלטפורמת.
הערה: ערוץ PE משמש כדי לזהות את ה-DNA מוכתם PI ואת קצב הזרימה צריך להיות איטי במהלך הרכישה. ודא כי המתח נבחר כך השיא דיפלואידי הוא בערך ב 200 לאורך הציר PE-A-x כדי להקל על ניתוח ופרשנות. המטרה להקליט מינימום של 20,000 אירועים לכל מדגם.
- לחות והתחדשות
| מגיב בשימוש (6 מ ל כל אחד) | משך |
| 1) קסילן | 2 x 10 דקות |
| 2) 100% אתנול | 2 x 10 דקות |
| 3) 95% אתנול | 10 דקות |
| 4) 70% אתנול | 10 דקות |
| 5) 50% אתנול | 10 דקות |
| 6) מים מזוקקים | 2 x 10 דקות |
שולחן 3: ריאגנטים ומשכי זמן לדפסיזציה ולחות מחדש.
-
הזרמת מידע
- ניתוח נתונים בעזרת התוכנה לניתוח cy,שולחן חומרים.
הערה: השלבים הבאים מוגדרים עבור תוכנה ספציפית אחת (טבלת חומרים) אך עשויה לסייע להקמת סוגי תוכנה אחרים גם כן.- לאחר הפעלת דגימות על cytometer זרימה, להוריד FCS 2.0 קבצים לניתוח.
- פתח את זרימת תוכנת ניתוח cy, לחץ על הקובץ | מסמך חדש.
- לחץ על סמל ההיסטוגרמה (
 ) ולאחר מכן גרור את המצביע כדי ליצור מלבן.
) ולאחר מכן גרור את המצביע כדי ליצור מלבן. - אתר את הקובץ FCS ולאחר מכן לחץ על פתוח. לאורך ציר ה-x, לחץ על Fcs-a ולאחר מכן בחר PE-a.
- לחץ על הסמל נקודה התוויה (
 ) ולאחר מכן גרור את המצביע כדי ליצור מלבן אחר מתחת להתוויה של ההיסטוגרמה. לאחר מכן אתר את אותו קובץ FCS שנבחר עבור ההיסטוגרמה.
) ולאחר מכן גרור את המצביע כדי ליצור מלבן אחר מתחת להתוויה של ההיסטוגרמה. לאחר מכן אתר את אותו קובץ FCS שנבחר עבור ההיסטוגרמה. - שנה את ציר ה-x של מגרש הנקודה ל- pe-A ואת ציר ה-y ל- pe-W.
הערה: איור 8A ממחיש את המראה של המגרשים בשלב זה. - לחץ על סמל האזור (
 ) וצייר תיבה על העלילה נקודה שמתחילה לפני שיא דיפלואידי (סביב 100 על ציר ה-x באיור 8ב) וזה מסתיים סביב 700 על ציר ה-x, כפי שמוצג בהתוויה נקודה באיור 8 ב.
) וצייר תיבה על העלילה נקודה שמתחילה לפני שיא דיפלואידי (סביב 100 על ציר ה-x באיור 8ב) וזה מסתיים סביב 700 על ציר ה-x, כפי שמוצג בהתוויה נקודה באיור 8 ב.
הערה: שיא דיפלואידי באיור 8 הוא בערך ב 200 על ציר ה-x. זה נבחר באופן שרירותי כפי הדגימות מוקלטות דרך cytometer הזרימה, פשוט כדי להקל על ניתוח ופרשנות של התוצאות. - לחץ על העלילה | ערוך אזורים/שערים, ולאחר מכן הקלד R0 בתא הנמצא ליד התא G0 תחת אסטרטגיה. לאחר מכן לחץ על סגור.
- לחץ במקום כלשהו על ההיסטוגרמה, ולאחר מכן על העלילה | עיצוב מגרש/שכבת-על. תחת שער, בחר G0 = R0 ולאחר מכן לחץ על אישור.
הערה: זהו הצעד המאפשר לאדם להמחיש טוב יותר את פסגות הפלואידיות. כעת ההיסטוגרמה תיראה כמו ההיסטוגרמה באיור 8B. ניתן לשחק עם השער שנוצר (על ידי הזזת התיבה שצוירה בשלב 2.4.1.7) כדי להתמקד באזורים מסוימים של העלילה נקודה. - כדי לתייג את החלקות, לחץ על סמל אזור הטקסט (
 ) ולאחר מכן גרור את המצביע כדי ליצור תיבה בחלק העליון של המסמך ולאחר מכן הקלד את המידע הבא: מזהה מטופל, POC id והבלוק המשמש (כיוון שייתכן שיהיו מספר בלוקים עבור POC אחד) , אחוז קורות חיים נוכח בבלוק, מתח המשמש להפעלת המדגם, ואת התאריך.
) ולאחר מכן גרור את המצביע כדי ליצור תיבה בחלק העליון של המסמך ולאחר מכן הקלד את המידע הבא: מזהה מטופל, POC id והבלוק המשמש (כיוון שייתכן שיהיו מספר בלוקים עבור POC אחד) , אחוז קורות חיים נוכח בבלוק, מתח המשמש להפעלת המדגם, ואת התאריך.
- ניתוח נתונים בעזרת התוכנה לניתוח cy,שולחן חומרים.
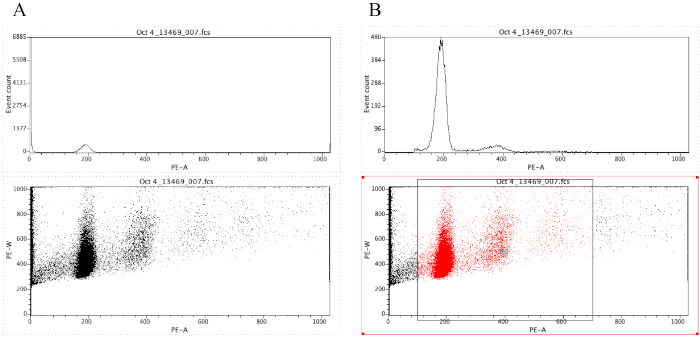
איור 8: תמונת מסך המציגה היסטוגרמה ומגרש נקודה של מדגם מייצג שאינו מגודר (a) ומגודרת (ב). אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
תוצאות
המורכבות של רקמות טוחנת והעובדה כי יש להם גנוטיפים שונים מחייב ניתוח קפדני ושימוש בשיטות מסוימות כגון הערכה מורפולוגית, p57 אימונוהיסטוכימיה, גנוזה מיקרולווין, זרימה cy, ודגים. לדוגמה, מטופל אחד (1790) הופנה עם שני PHM שנמצאו להיות triploid על ידי ניתוח microarray של POCs בלבד. המטופל אובח?...
Discussion
HM הם הריונות אנושיים חריגים עם חבלות הטרוגנית יש סוגים שונים היסטולוגית ו-genotypic, מה שהופך סיווג מדויק שלהם ואבחון מאתגרת. הערכה מורפולוגית היפואוולוגית הוכחה לעתים קרובות מדויק, ולכן לא אמין על עצמו לסווג HM לתוך CHM ו PHM ולהבדיל אותם מפני שאינם טוחנת הפלות. לכן, אבחנה מדויקת של HM דורש שימוש בש...
Disclosures
. למחברים אין מה לגלות
Acknowledgements
המחברים מודים סופי Patrier מריאן Parésy עבור שיתוף את הפרוטוקול המקורי cy, לנסות הפרוטוקולים, פרונגה ו Qiagen לספק אספקה וריאגנטים. עבודה זו נתמכת על ידי הרבייה והמכון הקנדי לחקר בריאות (מגב-130364) לR.S.
Materials
| Name | Company | Catalog Number | Comments |
| BD FACS Canto II | BD BioSciences | 338960 | |
| Capillary electrophoresis instrument: Genomes Applied Biosystems 3730xl DNA Analyzer | Applied biosystems | 313001R | Service offered by the Centre for Applied Genomics (http://www.tcag.ca) |
| Citric acid | Sigma | 251275 | |
| Cytoseal 60, histopathology mounting medium | Fisher | 23244257 | |
| Eosin Y stock solution (1%) | Fisher | SE23-500D | |
| FCSalyzer - flow cytometry analysis software | SourceForge | - | https://sourceforge.net/projects/fcsalyzer/ |
| FFPE Qiagen kit | Qiagen | 80234 | |
| Forceps | Fine Science Tools | 11295-51 | For sectioning and for the cleaning process |
| Glacial Acetic Acid (Concentrated) | Sigma | A6283-500mL | |
| Glass coverslips: Cover Glass | Fisher | 12-541a | |
| Hematoxylin | Fisher | CS401-1D | |
| Highly deionized formamide: Hi-Di Formamide | Thermofisher | 4311320 | |
| IHC platform: Benchmark Ultra | Roche | - | |
| Kimwipes | Ultident | 30-34120 | |
| Microtome | Leica | RM2135 | |
| Microtome blades | Fisher | 12-634-1C | |
| Nitex filtering mesh, 48 microns | Filmar | 74011 | http://www.filmar.qc.ca/index.php?filet=produits&id=51&lang=en ; any other filter is suitable, but this is an inexpensive and effective option from a non-research company |
| p57 antibody | Cell Marque | 457M | |
| Pasteur pipette | VWR | 53499-632 | |
| PCR machine | Perkin Elmer, Applied Biosystems | GeneAmp PCR System 9700 | |
| PeakScanner 1.0 | Applied Biosystems | 4381867 | Software for genotyping analysis. |
| Pepsin from porcine gastric mucosa | Sigma | P7012 | |
| Polystyrene round-bottom tubes | BD Falcon | 352058 | |
| Positively charged slides: Superfrost Plus 25x75mm | Fisher | 1255015 | |
| PowerPlex 16 HS System | Promega Corporation | DC2102 | |
| Propidium Iodide | Sigma | P4864 | |
| Ribonuclease A from bovine pancreas | Sigma | R4875 | |
| Separation matrix: POP-7 Polymer | Thermofisher | 4352759 | |
| UltraPure Agarose | Fisher | 16500-500 | |
| Xylene | Fisher | X3P1GAL |
References
- Szulman, A. E., Surti, U. The syndromes of hydatidiform mole. II. Morphologic evolution of the complete and partial mole. American Journal of Obstetrics & Gynecology. 132 (1), 20-27 (1978).
- Fukunaga, M., et al. Interobserver and intraobserver variability in the diagnosis of hydatidiform mole. The American Journal of Surgical Pathology. 29 (7), 942-947 (2005).
- Gupta, M., et al. Diagnostic reproducibility of hydatidiform moles: ancillary techniques (p57 immunohistochemistry and molecular genotyping) improve morphologic diagnosis for both recently trained and experienced gynecologic pathologists. The American Journal of Surgical Pathology. 36 (12), 1747-1760 (2012).
- Howat, A. J., et al. Can histopathologists reliably diagnose molar pregnancy?. Journal of Clinical Pathology. 46 (7), 599-602 (1993).
- Banet, N., et al. Characteristics of hydatidiform moles: analysis of a prospective series with p57 immunohistochemistry and molecular genotyping. Modern Pathology. 27 (2), 238-254 (2014).
- Lipata, F., et al. Precise DNA genotyping diagnosis of hydatidiform mole. Obstetrics & Gynecology. 115 (4), 784-794 (2010).
- Buza, N., Hui, P. Partial hydatidiform mole: histologic parameters in correlation with DNA genotyping. International Journal of Gynecologic Pathology. 32 (3), 307-315 (2013).
- Fisher, R. A., et al. Frequency of heterozygous complete hydatidiform moles, estimated by locus-specific minisatellite and Y chromosome-specific probes. Human Genetics. 82 (3), 259-263 (1989).
- Murdoch, S., et al. Mutations in NALP7 cause recurrent hydatidiform moles and reproductive wastage in humans. Nature Genetics. 38 (3), 300-302 (2006).
- Parry, D. A., et al. Mutations causing familial biparental hydatidiform mole implicate c6orf221 as a possible regulator of genomic imprinting in the human oocyte. American Journal of Human Genetics. 89 (3), 451-458 (2011).
- Nguyen, N. M., Slim, R. Genetics and Epigenetics of Recurrent Hydatidiform Moles: Basic Science and Genetic Counselling. Current Obstetrics and Gynecology Reports. 3, 55-64 (2014).
- Sebire, N. J., Savage, P. M., Seckl, M. J., Fisher, R. A. Histopathological features of biparental complete hydatidiform moles in women with NLRP7 mutations. Placenta. 34 (1), 50-56 (2013).
- Nguyen, N. M., et al. Comprehensive genotype-phenotype correlations between NLRP7 mutations and the balance between embryonic tissue differentiation and trophoblastic proliferation. Journal of Medical Genetics. 51 (9), 623-634 (2014).
- Brown, L., et al. Recurrent pregnancy loss in a woman with NLRP7 mutation: not all molar pregnancies can be easily classified as either "partial" or "complete" hydatidiform moles. International Journal of Gynecologic Pathology. 32 (4), 399-405 (2013).
- Colgan, T. J., Chang, M. C., Nanji, S., Kolomietz, E. A Reappraisal of the Incidence of Placental Hydatidiform Mole Using Selective Molecular Genotyping. The International Journal of Gynecological Cancer. 26 (7), 1345-1350 (2016).
- Murphy, K. M., McConnell, T. G., Hafez, M. J., Vang, R., Ronnett, B. M. Molecular genotyping of hydatidiform moles: analytic validation of a multiplex short tandem repeat assay. The Journal of Molecular Diagnostics. 11 (6), 598-605 (2009).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved