Bu içeriği görüntülemek için JoVE aboneliği gereklidir. Oturum açın veya ücretsiz deneme sürümünü başlatın.
Method Article
Formalin-sabit Parafin gömülü Hydatidiform Molar Dokuların Mikrouydu DNA Genotipleme ve Akış Sitometri Ploidy Analizleri
Bu Makalede
Özet
Hydatidiform benler, morfolojik özelliklerine ve molar genomlara ebeveyn katkısına göre sınıflandırılabilen heterojen etiyolojileri olan anormal insan gebelikleridir. Burada formalin-sabit parafin gömülü azı laksamalarının multipleks mikrouydu DNA genotipleme ve akış sitometrisi protokolleri, sonuçların yorumlanması ve entegrasyonu ile birlikte ayrıntılı olarak açıklanmıştır.
Özet
Hidatidiform mol (HM), aşırı trofoblastik proliferasyon ve anormal embriyonik gelişim ile karakterize anormal bir insan gebeliğidir. Mikroskobik morfolojik değerlendirmeye dayalı iki tip HM vardır: komple HM (CHM) ve kısmi HM (PHM). Bunlar, molar genomlarına ebeveyn katkısına göre daha fazla alt bölümlere ayrılabilir. HM'nin morfoloji ve genotip analizleri ile böyle bir karakterizasyonu, hasta yönetimi ve bu ilginç patolojinin temel anlayışı için çok önemlidir. HM'nin morfolojik analizinin geniş bir gözlemci ler arası değişkenliğe tabi olduğu ve HM'yi CHM ve PHM'ye doğru bir şekilde sınıflandırmak ve bunları hidropik molar olmayan düşüklerden ayırmak için tek başına yeterli olmadığı belgelenmiştir. Genotipleme analizi çoğunlukla optimal kaliteden daha az olan ve sonuç olarak yanlış sonuçlara yol açabilecek formalin-sabit parafin gömülü (FFPE) döllenme ürünlerinden DNA ve dokular üzerinde yapılır. Bu makalede FFPE molar dokularının çokk genotipleme ve akış sitometri analizleri için ayrıntılı protokoller, bu yöntemlerin sonuçlarının yorumlanması, bunların sorun giderme ve morfolojik değerlendirme ile entegrasyon ile birlikte sağlanmaktadır. , p57KIP2 immünohistokimya, ve floresan yerinde hibridizasyon (FISH) doğru ve sağlam bir tanı ulaşmak için. Burada yazarlar, son 10 yılda yaklaşık 400 conception ürününün analizinden öğrenilen yöntem ve dersleri paylaşıyorlar.
Giriş
Hydatidiform mol (HM), anormal embriyonik gelişim, trofoblastın hiperproproproproproproprobu ve koryonik villinin hidropik dejenerasyonu (CV) ile karakterize anormal bir insan gebeliğidir. Tarihsel olarak, HM iki tip, tam HM (CHM) ve kısmi HM (PHM) sadece morfolojik değerlendirme1dayalı bölünmüş olması için kullanılır. Ancak, morfolojik değerlendirmenin tek başına HM'yi iki alt tipe (CHM ve PHM) ayırmak ve bunları molar olmayan düşüklerden ayırmak için yeterli olmadığı gösterilmiştir2,3,4.
CHM ve PHM'nin malignitelere göre farklı eğilimleri olduğundan, hastalara uygun takip ve yönetimi sağlamak için genotik HM tipini doğru bir şekilde belirlemek önemlidir. Sonuç olarak, son yıllarda, azı laklarına ebeveyn katkısını belirlemek ve HM'nin doğru bir sınıflandırmasına ulaşmak amacıyla çeşitli metodolojiler geliştirilmiş ve geliştirilmiştir. Bunlar arasında karyotip analizi, kromozom bantlama polimorfizmi, insan lökosit antijeni (HLA) serolojik yazma, restriksiyon parça uzunluğu polimorfizmi, değişken tandem tekrarı sayısı, mikrouydu genotipleme, akış sitometrisi ve p57 KIP2 immünohistokimya. Bu, hm gebelerinin genomlarına ebeveyn katkısına dayalı doğru bir alt bölüme sahip olmasına olanak sağlamıştır: Dploid androgenetik monospermik veya diploid androgenetik dispermik olan CHM ve triploid olan PHM, %99 ve olguların %1'inde monospermik5,6,7,8. Ayrıca, son yirmi yılda ortaya çıkan başka bir genotypic HM tipi vardır, hangi diploid biparental olduğunu. İkincisi çoğunlukla tekrarlayan ve tek bir aile üyesi etkileyebilir (simpleks durumlarda) veya en az iki aile üyesi (ailesel durumlarda). Bu diploid biebeveyn benler çoğunlukla NLRP7 veya KHDC3L hastalarda9,10,11,12resesif mutasyonlar neden olur. NLRP7'de resesif mutasyonu olan hastalarda diploid biebeveynLI HM morfolojik analiz ile CHM veya PHM tanısı konabilir ve bu hastalardamutasyonların şiddeti ile ilişkili gibi görüner13,14. HM'nin genotiplerine göre sınıflandırılmasına ek olarak, çeşitli genotipleme yöntemlerinin tanıtımı ve kullanımı, çeşitli molar varlıkların molar olmayan düşüklerden, örneğin aneuploid diploid biebeveyn kavramları ndan ayırt edilebilmiştir ve diğer kavram türleri5,15. Bu tür kavramlar bazı trofoblast proliferasyonu ve bir dereceye kadar HM bazı morfolojik özellikleri taklit anormal villous morfolojisi olabilir.
Bu makalenin amacı, formalin-sabit parafin gömülü (FFPE) dokuların çokkatlı genotipleme ve akış sitometrisi için ayrıntılı protokoller sağlamak ve bu yöntemlerin sonuçlarının kapsamlı analizleri ve bunların diğer yöntemlerle bütünleştirilmesi azı laklarının doğru ve kesin tanısı.
Access restricted. Please log in or start a trial to view this content.
Protokol
Bu araştırma McGill Kurumsal İnceleme Kurulu tarafından onaylanmıştır. Tüm hastalar çalışmaya katılmak ve ffpe ürünlerinin çeşitli patoloji bölümlerinden alınması için yazılı izin verilmiştir.
NOT: Akış sitometrisi ile genotipleme ve ploidi tayini için çeşitli yöntemler olmakla birlikte, burada sağlanan protokoller her biri için bir platform kullanarak bir analiz yöntemini tanımlar.
1. Genotipleme
-
En iyi FFPE bloğunun seçimi
- Gebeliğin her FF ÜRÜNÜ (POC) için, mikroskopi ile morfolojik değerlendirme için, mevcut her blok için bir tane olan 1.2 ve 1.3 bölümlerinde açıklandığı şekilde 4 μm kalınlığında hematoksilin ve eozin (H&E) lekeli kesitler hazırlayın.
- H&E slaytları ve hafif mikroskobu kullanarak, en fazla koroyonik villi (CV) miktarına sahip FFPE bloğunu ve mümkünse, CV'si anne dokularından ayrı ve iç içe olmayan bloğu seçin.
-
Kesit
- Kesiti kolaylaştırmak için seçilen bloğu 15 dakika boyunca buzüzerine yerleştirin.
- Mikrotomu mikroskobik morfolojik değerlendirme için 4 μm kalınlığında ve DNA ekstraksiyonu için 10 μm kalınlığında kesecek şekilde ayarlayın.
- Soğuk bloğu mikrotome yerleştirin ve DNA ekstraksiyonu için bloktaki CV miktarına bağlı olarak H&E boyama için her bloktan bir bölüm ve seçilen bloktan 10−30 kesit kesin.
NOT: CV dolu bloklar için DNA ekstraksiyonu için 10 bölüm yeterlidir. Bloğun sadece %10'u CV, geri kalanı ise anne dokuları ise yeterli miktarda DNA sağlamak için 20−30 kesite ihtiyaç duyulabilmektedir. - Forceps kullanarak, her bölümü 45 °C su banyosuna aktarın. Daha önce bir kalem kullanarak örnek kimlik numarası ile etiketlenmiş bir pozitif yüklü slayt(Malzeme Tablosu)ile su banyosundan bölümü alın.
- Bölümleri içeren slaytları 65 °C'de bir fırına yerleştirin ve bölümlerin slaytlara yapışmasını bekleyin. H&E için slaytları 25 dakika fırında tutun.
NOT: Daha kısa kuluçka süresi dokuları slaytlara biraz daha az yapışır ve dolayısıyla anne dokularının çıkarılmasını kolaylaştırır.
-
H&E boyama
- Slaytların oda sıcaklığına (10 dk) kadar soğumasını bekleyin.
- Reaktif hazırlama
- Eosin Y çalışma solüsyonu hazırlayın (%0.25) Tablo 1'egöre . İyi bir şekilde karıştırın ve oda sıcaklığında saklayın.
- Hematoksilin 5x'in stok çözeltisini suda seyrelterek hematoksilin çözeltisini hazırlayın (yani 80 mL suyu 20 mL hematoksilin ile karıştırın).
NOT: Depolama için folyo hematoksilin stok çözeltisi sarın.
- Tablo 2'yegöre bir duman kaputunun altında doğru reaktiflerle boyama kavanozları hazırlayın.
- Slaytları Tablo 2'yegöre doğru zaman dilimi için uygun boyama kavanozlarına batırarak H&E boyama gerçekleştirin.
- Morfolojik analiz için 4 μm kesitleri montaj ortamı ve cam kapaklı kapaklı kapaklı(Malzeme Tablosu) monte edin.
NOT: Genotipleme için 10 μm kesitler örtülmemelidir. - Zehirli ksilen kokularının dağılabilmesi için duman kaputunun altındaki 10 μm'lik kesitleri en az 3 saat bekletin.
DİkKAT: Tüm boyama adımları bir duman başlık altında yapılmalıdır. Ksilen kokuları toksik olduğu için ksilen ürünleri her zaman kaputun altında tutulmalıdır. Ayrıca, ksilen ve hematoksilin özel kaplarda atılması gerekir. Bu konteynerler dolduğunda, laboratuvarın güvenlik organizasyonu tarafından tavsiye edildiği şekilde atılmalıdır.
| Reaktif | Miktar |
| Eosin Y stok çözeltisi (%1) | 250 mL |
| %80 Etanol | 750 mL |
| Buzul Asetik Asit (Konsantre) | 5 mL |
Tablo 1: Eozin Y çalışma çözeltisi (%0.25) Hazırlık.
| Kullanılan reaktif (depo başına 100 mL) | Süre |
| 1) Ksilen | 5 dk |
| 2) Ksilen | 5 dk |
| 3) %100 Etanol | 2 dk |
| 4) %95 Etanol | 2 dk |
| 5) %70 Etanol | 2 dk |
| 6) %50 Etanol | 2 dk |
| 7) Distile su | 5 dk |
| 8) Hematoksilin | 4 dk |
| 9) Distile su | 5 dk |
| 10) Eozin | 1 dk |
| 11) %95 Etanol | 5 dk |
| 12) %100 Etanol | 5 dk |
| 13) Ksilen | 5 dk |
| 14) Ksilen | 5 dk |
Tablo 2: H&E boyama protokolü için reaktifler ve süreler.
-
CV Yalıtımı
- Hafif bir stereomikroskop altında, H&E-lekeli 10 μm kalınlığındaki kesitlerden istenmeyen anne dokularını kazımak için forseps ve suyla nemlendirilmiş kağıt mendiller(Malzeme Tablosu)kullanın.
NOT: Nihai amaç slaytlar üzerinde CV veya fetal membranlar (mevcut) başka bir şey tutmak ve böylece diğer tüm dokuları kaldırmaktır. Detaylara titizlikle dikkat gerektirdiğinden, bu adım, blota bağlı olarak çok fazla zaman alabilirsiniz. - Anne dokularından arınmış olduğundan emin olmak için temizlikten sonra slaytları ikinci bir kişiye iki kez kontrol edin.
- Temizlenmiş slaytların fotoğraflarını çekin veya veri yorumuna yardımcı olmak için aşağıdakileri belgeleyin: 1) dokunun temizlenmesinin zor olup olmadığı, hemorajik veya çok temiz, 2) kullanılan kesit sayısı ve 3) temizlenmiş dokuların yaklaşık miktarı.
NOT: Şekil 1, temizlenmesi kolay bir slayt örneği sağlar. Kabaca bu miktarda CV içeren bir blok için DNA ekstraksiyonu için 10 bölüm yeterlidir. Şekil 2'deki slaytta anne dokuları ile iç içe olan çok az CV'si vardır, bu da temizlemeyi çok zor ve zaman alıcı hale getirir. Kabaca bu miktarda CV içeren bir blok için DNA çıkarma için 30 bölüm gereklidir. - Küçük nemlendirilmiş kağıt mendiller kullanarak CV toplayın. Forceps kullanarak, nemlendirilmiş kağıt mendil küçük bir parça gözyaşı ve CV toplamak için kullanabilirsiniz.
- Kağıt mendil parçalarını ekli CV'si ile 1,5 mL etiketli bir tüpe yerleştirin.
- Bu adımda kullanılan kağıt mendil miktarını en aza indirin, çünkü DNA çıkarma sütununu çok fazla tıkayabilir ve dolayısıyla toplanan DNA'nın son miktarını azaltabilir. Ortalama olarak, numune başına yedi küçük kağıt mendilden daha az kullanmayı hedefleyin. Büyük miktarda CV varlığı nedeniyle bu mümkün değilse, ekstraksiyonu kolaylaştırmak için numuneyi iki tüpe bölün.
- Hafif bir stereomikroskop altında, H&E-lekeli 10 μm kalınlığındaki kesitlerden istenmeyen anne dokularını kazımak için forseps ve suyla nemlendirilmiş kağıt mendiller(Malzeme Tablosu)kullanın.

Şekil 1: Genotipleme için temsili slayt. Üst: Anne dokularından arınmak için "temizlenmesi" gereken bir slayt. Alt: Temizlendikten sonra gösterilen aynı slayt ve şimdi DNA çıkarma için CV'den başka bir şey içermez. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
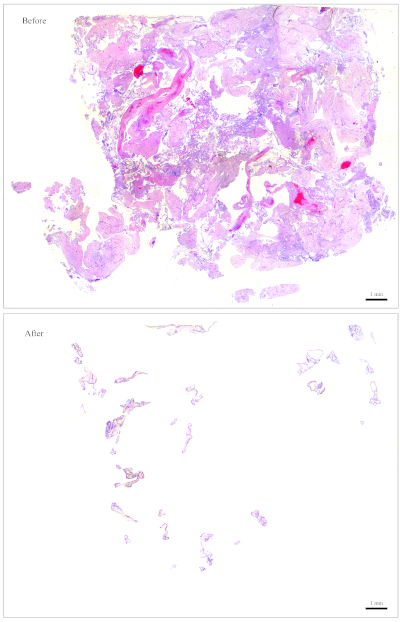
Şekil 2: Genotipleme için temsili slayt. Üst: Anne dokularından arınmak için "temizlenmesi" gereken bir slayt. Alt: Temizlendikten sonra gösterilen aynı slayt ve şimdi DNA çıkarma için CV'den başka bir şey içermez. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
- DNA ekstraksiyonu gerçekleştirmek için FFPE kitinden(Malzeme Tablosu)DNA çıkarma protokolünü uygulayın.
NOT: Bazı kitler son elüsyon için 15−20 μL elüsyon arabelleği kullanmanızı önerir. Deneyimlerinden, 15 μL elütion tamponu ile elüsasyon çoğu örnek için iyi çalışır. Seyreltmeler gerektiğinde stok DNA'dan hazırlanabilir.
-
DNA nicelleştirme
- Laboratuvar spektrofotometre cihazı kullanarak, 1 μL DNA yükleyin ve ölçüm ölçümü için 260 nm'de absorbans ölçün.
- %2'lik agarose jelüzerine 1 μL DNA yükleyin ve nitel değerlendirme için 80−100 V voltajda jel elektroforezini çalıştırın.
- 1.6.1 ve 1.6.2 adımlarının sonuçlarına göre, çokkatlı kısa tandem tekrarı (STR) polimeraz zincir reaksiyonu (PCR) amplifikasyonunda kullanılacak DNA hacmini seçin. Aşağıdaki PCR amplifikasyonunda en az 1000 ng DNA kullanmayı amaçlayın.
NOT: Şekil 3, DNA konsantrasyonları (spektrofotometre sonuçlarına göre) ve aşağıdaki çok katlı STR PCR için önerilen DNA çözeltisinin hacmi ile birlikte jellerin temsili örneklerini göstermektedir.
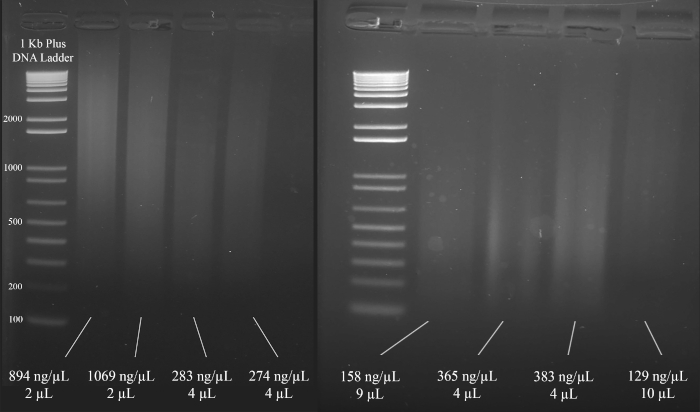
Şekil 3: DNA niceliği için temsili jel. Spektrofotometre kullanılarak ölçülen her DNA'nın konsantrasyonları ve multipleks PCR için kullanılan miktarlar dahildir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
-
PCR amplifikasyonu
- Çok katlı STR sistemi(Tablo Malzemeler)kullanarak floresan mikrouydu genotipleme gerçekleştirin.
- Çok katlı STR sistemi(Tablo Malzemeler)kullanarak PCR amplifikasyonu için Şekil 4'te gösterilen PCR koşullarını kullanın.
NOT: Bu çok katlı STR sisteminde aşağıdaki astarlar kullanılmaktadır: D18S51, D21S11, TH01, D3S1358, Penta E, FGA, TPOX, D8S1179, vWA, Amelogenin, CSF1PO, D16S539, D7S820, D13S317, D5S817, D5S817, D5S817.

Şekil 4: Multipleks STR sistemi için PCR çevrim koşulları. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
-
PCR ürünlerini kapiller elektroforez ile çözün.
- Çok katlı sistemin iç standart şeridinin 0,5 l'lik ve yüksek deiyonize formamidin 9,5 μL 'lik(Malzeme Tablosu)her güçlendirilmiş numunenin 1 0,5 l'sini askıya alın.
- Numuneleri, alet ve multipleks sisteminin boya seti için uygun bir ayırma matrisi(Malzeme Tablosu)kullanarak bir kapiller elektroforez aleti(Malzeme Tablosu)ile çalıştırın.
- Veri analizi
- Verileri bir DNA parçası analiz yazılımıyla analiz edin ve kökenini belirlemek için POC alellerini ebeveyn alelleriyle karşılaştırın.
- Boyut standardı ayarlayın.
NOT: Bu, yazılımın multipleks STR sisteminde kullanılan merdiveni tanımasına ve merdivene dayalı olarak amplikonlara baz çiftleri atamasına olanak tanır. Aşağıdaki adımlar belirli bir yazılım içindir(Malzeme Tablosu)ancak diğer yazılım türlerinin de ayarlanmasında yardımcı olabilir.- Yazılımı açın. Yeni Projeyi Başlat'a ve ardından Yeni Boyut Standardı'natıklayın.
- Boyut standardına bir ad verin (örn. ABI_600).
- Yeni Boyut Standart ını Girin adlıkutuya: aşağıdakileri girin: 60, 80, 100, 120, 140, 160, 180, 200, 225, 250, 275, 300, 325, 350, 375, 400, 425, 450, 475, 500, 550, 600. Ardından Boyut Ekle(ler)'etıklayın.
NOT: Girilen sayılar sağdaki kutunun altında görünür ve bu kutuya Current Size Standard Definition (bkz. Şekil 5). - Kaydet'etıklayın.
- Bir dosyayı almak ve çözümlemek için Dosya Ekle'yitıklatın ve çözümlenecek fsa dosyasını seçin. Seçili Dosyaları Ekle'ye ve ardından Tamam'atıklayın. Ardından şu adımları izleyin:
- Boyut Standart sütununa bulun ve ABI_600'ü (veya boyut standardına hangi ad verildiyonu) seçin.
- Analiz Yöntemialtında, Boyutlandırma Varsayılanı - NPP'yi tıklatın ve ardından yeşil Analiz düğmesine tıklayın.
- Dosya artık görüntülenmeye hazır. Verileri istenilen şekilde görüntülemek için görüntüleme seçeneklerini ayarlayın.
- Sorun giderme - analiz yöntemi
NOT: Yazılım bazen zirveleri tanımlamak ve doğru hizalamak için başarısız olabilir. Bu zirveleri ya çok düşük ya da çok yüksek olduğunda olur. Aşağıdaki iki analiz yöntemi bunun için düzeltilebilir ve bir örnek yeniden test edilmeden önce denenmelidir.- Yüksek zirveler için analiz yöntemi 1:
- Yeni Analiz Yöntemi'ni tıklatın ve yüksek tepelerle (veya kişisel tercihe göre başka bir ad) adını adlandırın.
- Analiz ve boyutlandırma için Aralık'a ve ardından Kısmi Aralık'a tıklayın. Daha sonra Başlangıç Noktası ve Başlangıç Boyutuiçin 100 yazın.
- Dur Noktasıiçin 10.000 girin. Stop Boyutuiçin 1000 girin.
- Sonra Minimum tepe yükseklikleri tıklayın ve renkler için tepe eşik aşağıdaki gibi numaraları değiştirin: Mavi: 50; Yeşil: 50; Sarı: 20; Kırmızı: 100; Turuncu: 5000.
- Yeni çözümleme yöntemini kaydedin.
- Düşük zirveler için analiz yöntemi 2:
- Yeni Analiz Yöntemi'ni tıklatın ve düşük tepelerle (veya kişisel tercihe göre başka bir ad) adını adlandırın.
- Analiz ve boyutlandırma için Aralık'a ve ardından Kısmi Aralık'a tıklayın. Daha sonra Başlangıç Noktası ve Başlangıç Boyutuiçin 100 yazın.
- Dur Noktasıiçin 10.000 girin. Stop Boyutuiçin 1000 girin.
- Ardından Kalite Bayrakları'na tıklayın ve 0,5'ten 1'ekadar okunacak şekilde Geçiş Aralığını değiştirin. 0,0'dan 0,0'akadar okunacak şekilde Düşük Kalite Aralığını değiştirin. Doğrusallığı aşağıdakilere değiştirin: (bp) 100,0'dan (bp) 800,0'akadar.
- Yeni çözümleme yöntemini kaydedin.
NOT: Artık Analiz Yöntemi altında Düşük Tepeler veya Yüksek Tepeler'i seçip yeşil Analiz düğmesine tıklayarak bir dosyayı yeniden çözümlemek mümkündür.
- Yüksek zirveler için analiz yöntemi 1:

Şekil 5: Boyut Standart Düzenleyicisini gösteren ekran görüntüsü. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
2. Akış Sitometrisi
-
İdeal FFPE bloğunu seçme
- H&E slaytları ve hafif bir mikroskop kullanarak, dokularının yaklaşık %50-70'ine sahip bir FFPE bloğu seçin.
NOT: Şekil 6, kabaca %50 CV (bölümün sağ yarısı) ve %50 maternal dokulardan (sol yarısı) oluştuğundan, akış sitometrisi analizi için uygun bir bloğun temsili bir örneğidir. Onlar diploid tepe için bir iç kontrol olarak hizmet çünkü maternal dokuların varlığı önemlidir. - İdeal cv miktarına sahip olmayan bloklar için, kesit işlemi gerçekleştirilirken CV için zenginleştirin. Bunu yapmak için, yeni kesilmiş bölümlerin hangi tarafında ilgili H&E slaytına göre daha fazla CV içerdiğini belirleyin. Buna dayanarak, CV için zenginleştirmek için atılması gereken diğer yarısını kesmek için bir bıçak kullanın.
NOT: Şekil 7 akış sitometri analizi için yeterli CV'si olmayan bir bloğu göstermektedir. Bu gibi bloklar için, daha az CV içeren yarısı, anne dokularına göre CV miktarlarını artırmak için, şekilde gösterildiği gibi atılır gibi kesilmelidir. Atılanları telafi etmek için daha fazla kesit kestiğinden emin olun.
- H&E slaytları ve hafif bir mikroskop kullanarak, dokularının yaklaşık %50-70'ine sahip bir FFPE bloğu seçin.
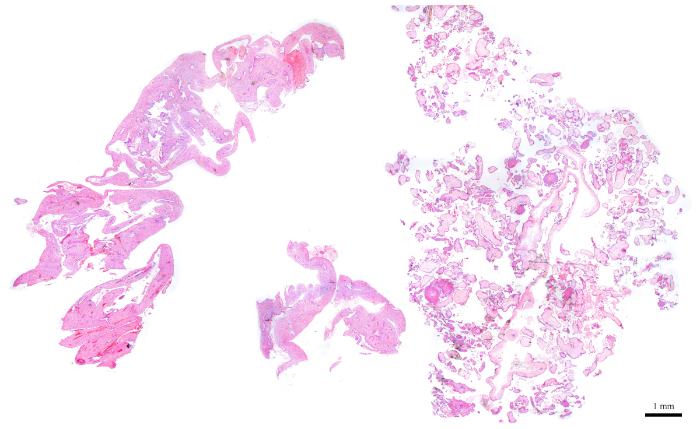
Şekil 6: Akış sitometrisi için ideal olan bir POC bloğunu temsil eden H&E bölümü. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
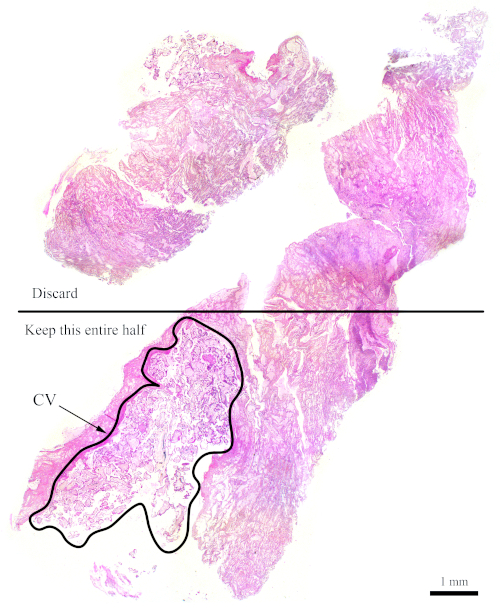
Şekil 7: Akış sitometrisi için daha zor bir bloğu temsil eden H&E bölümü. Bu temsilci H&E bölümü, CV için zenginleştirme amacıyla, akış sitometrisi analizi için bu bölümün sadece alt yarısının kullanılması gerektiğini göstermektedir. "CV" etiketli ana hatlar çoğunlukla CV'den oluşur.
- Kesit
- Kesiti kolaylaştırmak için blokları 15 dk buz üzerinde bırakın.
- Mümkün olan en iyi FFPE bloğu kullanarak, bir mikrotom kullanarak 50 μm kalınlığında (veya iki 100 μm kalınlığında kesin) dört bölüm kesin.
NOT: Akış sitometrisi için daha kalın kesitlere sahip olmak tercih edilir. - İdeal bir FFPE bloğu mevcut olmaması durumunda, yine de CV'nin anne dokusuna oranını korumayı amaçlamaktadır. Örneğin, bloğun yalnızca %30'u CV'den oluşurken, geri kalanı anne dokularını içeren bölümün en az yarısını çıkarın ve telafi etmek için daha fazla kesit kullanın (Bkz. Şekil 7).
- Bölümleri etiketli 15 mL tüplere yerleştirin.
NOT: Bir sonraki adımda kullanılan organik reaktifler mürekbete çözülüp çıkarabildiği için etiketlerin üzerine bantladığından emin olun.
- FFPE dokularından akış sitometri protokolü
- Deparafinizasyon ve rehidrasyon
- Bir duman başlığı altında aşağıdaki yıkıntıları(Tablo 3)gerçekleştirin.
- Tablo 3'te verilen sırayı izleyerek 15 mL'lik tüpü uygun reaktifin 6 mL'si ile doldurun, reaktifteki bölümleri ilgili süre boyunca bırakın ve vakum emme ve cam Pasteur pipetiyle reaktifi çıkarın.
- Her adım arasında Pasteur pipetini önce %70 etanol, sonra distile suya batırın ve bir sonraki adıma geçin.
- Reaktif ile birlikte doku parçaları kaldırmak için çok dikkatli olun. 15 mL'lik tüpü, sıvı reaktifinin dokuları çizmeden emmesini kolaylaştırmak için 60 derecelik bir açıya yatırın.
DİkKAT: Atılan sıvılar ksilen içerir ve ksilen atık kaplarına atılmalıdır.
- Çözüm hazırlama
- Çift distile su 1 L sitrik asit 2 g eriterek sitrat çözeltisi hazırlayın. pH'ı 6'ya getirin. 4 °C'de saklayın.
- Bin DaK başına 9 parçadan 2 mL pepsin 0.01 g eriterek pepsin çözeltisini hazırlayın, pH 1.64. Bu bir örnek için.
DİkKAT: Pepsin toksik ve kolayca dağıtmak ve hava yoluyla olabilir. Toz formunda pepsin kullanırken maske takın ve kullandıktan sonra çalışma alanının tamamını silin. - Propidium Iodide (PI)-ribonukle bir örnek için bir çözelti hazırlığı.
- 50 μL PI ile 450 μL PBS'yi karıştırın (10x'i seyreltmek için).
- Karışıma 50 μL ribonükle a (1 mg/mL) ekleyin. Her zaman folyoya sarılı tutun.
- Sindirim ve boyama
- 15 mL tüplere 4 °C sitrat çözeltisi ekleyin ve 2 saat boyunca 80 °C'lik bir su banyosuna yerleştirin.
- Çözeltioda sıcaklığına (15 dk) kadar soğumaya bırakın. Sitrat çözeltisini çıkarın.
- 1x PBS, girdap 6 mL ekleyin ve dokuların dibe yerleşmek için 1−2 dk bekleyin. Vakum emme ve cam Pasteur pipet kullanarak 1x PBS çıkarın.
- 1 mL pepsin çözeltisi (önceden ısıtılmış 37 °C)'a ekleyin ve 37 °C'lik kuru bir banyoya 30 dk. Girdap her 10 dakikada bir yerleştirin. Bu kuluçka nın son 10 dakikasında PI-ribonükle a çözeltisini hazırlayın.
- 1x PBS, girdap 6 mL ekleyin ve dokuların dibe yerleşmek için 1−2 dk bekleyin. Vakum emme ve cam Pasteur pipet kullanarak 1x PBS çıkarın.
- PI-ribonüklea 550 μL bir çözelti ekleyin ve numuneleri 30 dakika boyunca 37 °C'lik kuru bir banyoya yerleştirin.
NOT: Bu noktada numuneler folyoya sarılır ve bir gecede 4 °C'de ertesi sabaha kadar bırakılabilir. - Çözeltiyi 48 μm'lik bir filtrasyon örgüsünden filtreleyin. Akış sitometresi ile kullanılabilir polistiren yuvarlak alt tüpler, filtrat toplamak. Tüpün üst kısmına 5 cm'ye 5 cm'lik filtrasyon örgüsünü yerleştirmek için, sıvının meshten ve tüpe boruyla boruya aktarılabildiği şekilde forceps kullanın.
NOT: Örnekler akış sitometresi ile çalıştırılacak hazırdır. Onlar çalıştırılmak için hazır olana kadar folyo sarılmış tutun.
- Kuruluşun akış sitometri sitometrisi platform teknisyeni yardımıyla bir akış sitometre ile örnekleri çalıştırın.
NOT: PE kanalı PI lekeli DNA'yı algılamak için kullanılır ve akış hızı satın alma sırasında Yavaş olarak ayarlanmalıdır. Analiz ve yorumlamayı kolaylaştırmak için diploid tepenin PE-A x ekseni boyunca kabaca 200 seviyesinde olmasını sağlayacak şekilde gerilimin seçildiğinden emin olun. Örnek başına en az 20.000 olay kaydetmeyi hedefliyoruz.
- Deparafinizasyon ve rehidrasyon
| Kullanılan reaktif (her biri 6 mL) | Süre |
| 1) Ksilen | 2 x 10 dk |
| 2) %100 Etanol | 2 x 10 dk |
| 3) %95 Etanol | 10 dk |
| 4) %70 Etanol | 10 dk |
| 5) %50 Etanol | 10 dk |
| 6) Distile su | 2 x 10 dk |
Tablo 3: Deparafinizasyon ve rehidrasyon için reaktifler ve süreler.
-
Akış sitometri veri analizi
- Akış sitometri analiz yazılımı(Malzeme Tablosu)ile verileri analiz edin.
NOT: Aşağıdaki adımlar belirli bir yazılım içindir(Malzeme Tablosu)ancak diğer yazılım türlerinin de ayarlanmasında yardımcı olabilir.- Örnekleri akış sitometreüzerinde çalıştırdıktan sonra, analiz için FCS 2.0 dosyalarını indirin.
- Akış sitometri analiz yazılımını açın, Dosya'ya tıklayın | Yeni Belge.
- Histogram simgesine ()
 tıklayın ve dikdörtgen yapmak için işaretçiyi sürükleyin.
tıklayın ve dikdörtgen yapmak için işaretçiyi sürükleyin. - FCS dosyasına göz atın ve ardından Aç'atıklayın. X ekseni boyunca FCS-A'yı tıklatın ve ardından PE-A'yıseçin.
- Nokta Çizimi simgesine
 () tıklayın ve ardından işaretçiyi sürükleyip histogram çiziminin altında başka bir dikdörtgen oluşturun. Ardından histogram için seçilen aynı FCS dosyasına göz atın.
() tıklayın ve ardından işaretçiyi sürükleyip histogram çiziminin altında başka bir dikdörtgen oluşturun. Ardından histogram için seçilen aynı FCS dosyasına göz atın. - Nokta çiziminin x eksenini PE-A ve y eksenini PE-Wolarak değiştirin.
NOT: Şekil 8A bu noktada çizerlerin görünümünü gösterir. - Bölge simgesine tıklayın
 ( ) ve şekil 8'deki nokta çiziminde gösterildiği gibi, diploid tepeden önce başlayan nokta çizimine bir kutu çizin (Şekil 8B'dekix ekseninde yaklaşık 100 ) ve x ekseninde 700 civarında sona eren B.
( ) ve şekil 8'deki nokta çiziminde gösterildiği gibi, diploid tepeden önce başlayan nokta çizimine bir kutu çizin (Şekil 8B'dekix ekseninde yaklaşık 100 ) ve x ekseninde 700 civarında sona eren B.
NOT: Şekil 8'deki diploid tepe kabaca x ekseninde 200'dedir. Bu, örnekler akış sitometresi aracılığıyla kaydedilirken, sadece sonuçların analizini ve yorumlanmasını kolaylaştırmak için rasgele seçilir. - Arsa üzerine tıklayın | Bölgeleri/Kapıları Edin,ardından Stratejialtında G0 hücresinin yanındaki hücreye R0 yazın. Ardından Kapat'atıklayın.
- Histogram her yerde tıklayın, sonra Arsa | Çizim/Yer Kaplamayı Biçimlendir. Geçitaltında, G0 = R0 seçin ve sonra Tamamtıklayın.
NOT: Bu bir ploidy zirveleri daha iyi görselleştirmek için izin veren gating adımdır. Histogram şimdi Şekil 8Bhistogram gibi görünmelidir. Nokta çiziminin belirli bölgelerine odaklanmak için oluşturulan kapıyla (2.4.1.7 adımda çizilen kutuyu hareket ettirerek) oynamak mümkündür. - Çizimleri etiketlemek için Metin Alanı simgesine (),
 sonra belgenin üst kısmında bir kutu oluşturmak için işaretçiyi sürükleyin ve ardından aşağıdaki bilgileri yazın: Hasta Kimliği, POC Kimliği ve kullanılan blok (bir POC için birkaç blok olabileceğinden) , blokta bulunan yüzde CV, numuneyi çalıştırmak için kullanılan gerilim ve tarih.
sonra belgenin üst kısmında bir kutu oluşturmak için işaretçiyi sürükleyin ve ardından aşağıdaki bilgileri yazın: Hasta Kimliği, POC Kimliği ve kullanılan blok (bir POC için birkaç blok olabileceğinden) , blokta bulunan yüzde CV, numuneyi çalıştırmak için kullanılan gerilim ve tarih.
- Akış sitometri analiz yazılımı(Malzeme Tablosu)ile verileri analiz edin.
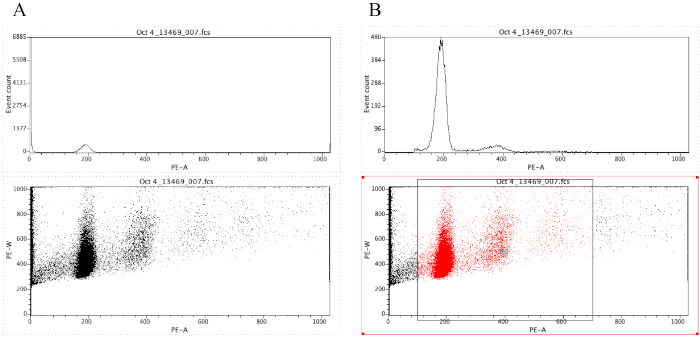
Şekil 8: Bir histogram ı ve temsili bir örneğin (A) ve (B) geçitli nokta çizimini görüntüleyen ekran görüntüsü. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Access restricted. Please log in or start a trial to view this content.
Sonuçlar
Azı laklarının karmaşıklığı ve çeşitli genotiplere sahip olmaları, morfolojik değerlendirme, p57 immünohistokimyası, mikrouydu genotipleme, akış sitometrisi ve FISH gibi çeşitli yöntemlerin kullanılmasını gerektirir. Örneğin, bir hastaya (1790) sadece POC'ların mikrodizi analizi ile triploid olduğu tespit edilen iki PHM ile sevk edildi. Bu nedenle hastaya tekrarlayan PHM tanısı konuldu. Hastanın dna'sı ve partneri ile birlikte iki "PHM"in mikrouydu genotiplem...
Access restricted. Please log in or start a trial to view this content.
Tartışmalar
HM heterojen etiyolojileri ile anormal insan gebelikleri ve farklı histolojik ve genotik türleri vardır, hangi onların doğru sınıflandırma ve tanı zor hale getirir. Histopatolojik morfolojik değerlendirme genellikle yanlış kanıtlanmıştır ve bu nedenle HM'yi CHM ve PHM olarak sınıflandırmak ve bunları molar olmayan düşüklerden ayırt etmek için tek başına güvenilmezdir. Bu nedenle, HM doğru bir tanı multiks mikrouydu DNA genotipleme, akış sitometri sitometri sitometri sitomi ile ploidi anali...
Access restricted. Please log in or start a trial to view this content.
Açıklamalar
Yazarların açıklayacak bir şeyi yok.
Teşekkürler
Yazarlar sophie Patrier ve Marianne Parésy orijinal akış sitometri protokolü paylaşımı için teşekkür ve Promega ve Qiagen malzemeleri ve reaktifler sağlamak için. Bu çalışma Réseau Québécois en Reprodüksiyon ve Kanada Sağlık Araştırmaları Enstitüsü (MOP-130364) tarafından R.S.'ye destek lenmiştir.
Access restricted. Please log in or start a trial to view this content.
Malzemeler
| Name | Company | Catalog Number | Comments |
| BD FACS Canto II | BD BioSciences | 338960 | |
| Capillary electrophoresis instrument: Genomes Applied Biosystems 3730xl DNA Analyzer | Applied biosystems | 313001R | Service offered by the Centre for Applied Genomics (http://www.tcag.ca) |
| Citric acid | Sigma | 251275 | |
| Cytoseal 60, histopathology mounting medium | Fisher | 23244257 | |
| Eosin Y stock solution (1%) | Fisher | SE23-500D | |
| FCSalyzer - flow cytometry analysis software | SourceForge | - | https://sourceforge.net/projects/fcsalyzer/ |
| FFPE Qiagen kit | Qiagen | 80234 | |
| Forceps | Fine Science Tools | 11295-51 | For sectioning and for the cleaning process |
| Glacial Acetic Acid (Concentrated) | Sigma | A6283-500mL | |
| Glass coverslips: Cover Glass | Fisher | 12-541a | |
| Hematoxylin | Fisher | CS401-1D | |
| Highly deionized formamide: Hi-Di Formamide | Thermofisher | 4311320 | |
| IHC platform: Benchmark Ultra | Roche | - | |
| Kimwipes | Ultident | 30-34120 | |
| Microtome | Leica | RM2135 | |
| Microtome blades | Fisher | 12-634-1C | |
| Nitex filtering mesh, 48 μm | Filmar | 74011 | http://www.filmar.qc.ca/index.php?filet=produits&id=51&lang=en ; any other filter is suitable, but this is an inexpensive and effective option from a non-research company |
| p57 antibody | Cell Marque | 457M | |
| Pasteur pipette | VWR | 53499-632 | |
| PCR machine | Perkin Elmer, Applied Biosystems | GeneAmp PCR System 9700 | |
| PeakScanner 1.0 | Applied Biosystems | 4381867 | Software for genotyping analysis. |
| Pepsin from porcine gastric mucosa | Sigma | P7012 | |
| Polystyrene round-bottom tubes | BD Falcon | 352058 | |
| Positively charged slides: Superfrost Plus 25x75mm | Fisher | 1255015 | |
| PowerPlex 16 HS System | Promega Corporation | DC2102 | |
| Propidium Iodide | Sigma | P4864 | |
| Ribonuclease A from bovine pancreas | Sigma | R4875 | |
| Separation matrix: POP-7 Polymer | Thermofisher | 4352759 | |
| UltraPure Agarose | Fisher | 16500-500 | |
| Xylene | Fisher | X3P1GAL |
Referanslar
- Szulman, A. E., Surti, U. The syndromes of hydatidiform mole. II. Morphologic evolution of the complete and partial mole. American Journal of Obstetrics & Gynecology. 132 (1), 20-27 (1978).
- Fukunaga, M., et al. Interobserver and intraobserver variability in the diagnosis of hydatidiform mole. The American Journal of Surgical Pathology. 29 (7), 942-947 (2005).
- Gupta, M., et al. Diagnostic reproducibility of hydatidiform moles: ancillary techniques (p57 immunohistochemistry and molecular genotyping) improve morphologic diagnosis for both recently trained and experienced gynecologic pathologists. The American Journal of Surgical Pathology. 36 (12), 1747-1760 (2012).
- Howat, A. J., et al. Can histopathologists reliably diagnose molar pregnancy? Journal of Clinical Pathology. 46 (7), 599-602 (1993).
- Banet, N., et al. Characteristics of hydatidiform moles: analysis of a prospective series with p57 immunohistochemistry and molecular genotyping. Modern Pathology. 27 (2), 238-254 (2014).
- Lipata, F., et al. Precise DNA genotyping diagnosis of hydatidiform mole. Obstetrics & Gynecology. 115 (4), 784-794 (2010).
- Buza, N., Hui, P. Partial hydatidiform mole: histologic parameters in correlation with DNA genotyping. International Journal of Gynecologic Pathology. 32 (3), 307-315 (2013).
- Fisher, R. A., et al. Frequency of heterozygous complete hydatidiform moles, estimated by locus-specific minisatellite and Y chromosome-specific probes. Human Genetics. 82 (3), 259-263 (1989).
- Murdoch, S., et al. Mutations in NALP7 cause recurrent hydatidiform moles and reproductive wastage in humans. Nature Genetics. 38 (3), 300-302 (2006).
- Parry, D. A., et al. Mutations causing familial biparental hydatidiform mole implicate c6orf221 as a possible regulator of genomic imprinting in the human oocyte. American Journal of Human Genetics. 89 (3), 451-458 (2011).
- Nguyen, N. M., Slim, R. Genetics and Epigenetics of Recurrent Hydatidiform Moles: Basic Science and Genetic Counselling. Current Obstetrics and Gynecology Reports. 3, 55-64 (2014).
- Sebire, N. J., Savage, P. M., Seckl, M. J., Fisher, R. A. Histopathological features of biparental complete hydatidiform moles in women with NLRP7 mutations. Placenta. 34 (1), 50-56 (2013).
- Nguyen, N. M., et al. Comprehensive genotype-phenotype correlations between NLRP7 mutations and the balance between embryonic tissue differentiation and trophoblastic proliferation. Journal of Medical Genetics. 51 (9), 623-634 (2014).
- Brown, L., et al. Recurrent pregnancy loss in a woman with NLRP7 mutation: not all molar pregnancies can be easily classified as either "partial" or "complete" hydatidiform moles. International Journal of Gynecologic Pathology. 32 (4), 399-405 (2013).
- Colgan, T. J., Chang, M. C., Nanji, S., Kolomietz, E. A Reappraisal of the Incidence of Placental Hydatidiform Mole Using Selective Molecular Genotyping. The International Journal of Gynecological Cancer. 26 (7), 1345-1350 (2016).
- Murphy, K. M., McConnell, T. G., Hafez, M. J., Vang, R., Ronnett, B. M. Molecular genotyping of hydatidiform moles: analytic validation of a multiplex short tandem repeat assay. The Journal of Molecular Diagnostics. 11 (6), 598-605 (2009).
Access restricted. Please log in or start a trial to view this content.
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır