Для просмотра этого контента требуется подписка на Jove Войдите в систему или начните бесплатную пробную версию.
Method Article
Микроспутниковая ДНК Генотипирование и поток Цитометрия Плоиди Анализ формалин фиксированной Парафин-встроенный Hydatidiform Моляровые ткани
В этой статье
Резюме
Hydatidiform моли являются ненормальные беременности человека с разнородными этиологиями, которые могут быть классифицированы в соответствии с их морфологических особенностей и родительского вклада в молярового геномов. Здесь подробно описаны протоколы мультиплексной микроспутниковой ДНК генотипирования и цитометрии потока формалино-фиксированных парафин-встроенных моляровских тканей, а также интерпретации результатов и интеграции.
Аннотация
Hydatidiform моль (HM) является ненормальной человеческой беременности характеризуется чрезмерным трофиобластным пролиферацией и аномальным эмбриональным развитием. Существует два типа HM на основе микроскопической морфологической оценки, полный HM (CHM) и частичный HM (PHM). Они могут быть дополнительно подразделены на основе родительского вклада в моляровские геномы. Такая характеристика HM, морфологии и генотипа анализов, имеет решающее значение для управления пациентом и для фундаментального понимания этой интригующей патологии. Хорошо задокументировано, что морфологический анализ Hm подвержен широкой изменчивости межнаблюдателей и не является достаточным сам по себе, чтобы точно классифицировать HM в CHM и PHM и отличить их от гидропиковых немолярных абортов. Генотипный анализ в основном проводится на ДНК и тканях из формалино-фиксированных парафин-встроенных (FFPE) продуктов зачатия, которые имеют меньше, чем оптимальное качество и, следовательно, может привести к неправильным выводам. В этой статье предоставляются подробные протоколы для мультиплексного генотипирования и цитометрии потока, а также интерпретация результатов этих методов, их устранение неполадок и интеграция с морфологической оценкой , p57KIP2 иммуногистохимия, и флуоресценция на месте гибридизации (FISH) для достижения правильной и надежной диагностики. Здесь авторы делятся методами и уроками, извлеченные за последние 10 лет из анализа примерно 400 продуктов зачатия.
Введение
Гиматидиформная родинка (Hm) является ненормальной человеческой беременности характеризуется аномальным эмбриональным развитием, гиперраспространением трофобласта и гидропикной дегенерацией хорионического вилли (CV). Исторически сложилось так, что HM раньше был разделен на два типа, полный HM (CHM) и частичный HM (PHM) на основе только морфологической оценки1. Тем не менее, было показано, что морфологическая оценка сама по себе не является достаточной, чтобы классифицировать HM в два подтипа (CHM и PHM) и отличить их от немолярных выкидышей2,3,4.
Поскольку CHM и PHM имеют различные склонности к злокачественным новообразованиям, поэтому важно точно определить генотипический тип HM, чтобы обеспечить надлежащее наблюдение и управление пациентами. Следовательно, в последние десятилетия было разработано и разработано несколько методологий с целью выявления вклада родителей в моляровые ткани и достижения правильной классификации НМ. К ним относятся кариотипный анализ, хромосомный полостальный полиморфизм, антиген лейкоцита человека (HLA) серологический типизон, полиморфизм длины фрагмента ограничения, переменное количество тандемных повторов, микроспутниковая генотипирование, цитометрия потока и p57 KIP2 иммуногистохимия. Это позволило точное разделение HM концепций на основе родительского вклада в их геномы, а именно: CHM, которые являются диплоидных андрогенетических моноспермных или диплоидных андрогенетических диспермических, и PHM, которые являются триплоид, диспермические в 99% и моноспермный в 1% случаев5,6,7,8. Кроме того, есть еще один генотипический тип HM, который появился в последние два десятилетия, который является диплоидный biparental. Последнее в основном повторяются и может повлиять на одного члена семьи (простые случаи) или, по крайней мере, двух членов семьи (семейные случаи). Эти диплоидные дьюдетские родинки в основном вызваны рецессивными мутациями в NLRP7 или KHDC3L у пациентов9,10,11,12. Диплоид ный родитель HM у пациентов с рецессивными мутациями в NLRP7 может быть диагностирован как CHM или PHM морфологическим анализом, и это, как представляется, связано с тяжестью мутаций у пациентов13,14. В дополнение к классификации HM в соответствии с их генотипами, введение и использование нескольких методов генотипирования позволило различать различные молярые сущности от немолянных выкидышей, таких как анеплодоидные диплоидные двухродительские концепции и другие типы зачатий5,15. Такие концепции могут иметь некоторое распространение трофобластов и ненормальную злобную морфологию, которые в некоторой степени имитируют некоторые морфологические особенности Hm.
Целью данной статьи является предоставление подробных протоколов для мультиплексной генотипирования и цитометрии сфиксированными парафиновыми (FFPE) тканями, а также комплексный анализ результатов этих методов и их интеграция с другими методами правильный и убедительный диагноз моляровских тканей.
протокол
Это исследование было одобрено Советом по институциональному обзору McGill. Все пациенты дали письменное согласие на участие в исследовании и на получение их продуктов FFPE зачатия (POCs) из различных патологических отделений.
ПРИМЕЧАНИЕ: Хотя существует несколько методов определения генотипирования и плоиды с помощью цитометрии потока, представленные здесь протоколы описывают один метод анализа с использованием одной платформы для каждого из них.
1. Генотипирование
-
Выбор лучшего блока FFPE
- Для каждого продукта FFPE зачатия (POC), подготовить 4 мкм толщиной гематоксилин и эозин (H и E) окрашенных разделов, как описано в разделах 1.2 и 1.3, по одному для каждого доступного блока, для морфологической оценки с помощью микроскопии.
- Используя h и E слайды и легкий микроскоп, выберите блок FFPE, который имеет наибольшее количество хорионических вилли (CV), и, если возможно, блок, который имеет резюме отдельно от, а не переплетемые с материнскими тканями.
-
Сечения
- Поместите выбранный блок на льду в течение 15 минут, чтобы облегчить секцию.
- Отрегулируйте микротом, чтобы сократить разделы, которые 4 мкм толщиной для микроскопической морфологической оценки и 10 мкм толщиной для извлечения ДНК.
- Поместите холодный блок в микротоме и вырежьте по одному разделу из каждого блока для окрашивания Н и Е и 10-30 секций из выбранного блока, в зависимости от количества резюме в блоке, для извлечения ДНК.
ПРИМЕЧАНИЕ: Для блоков, которые полны cv, 10 разделов достаточно для извлечения ДНК. Если только около 10% блока содержит CV, в то время как остальные материнские ткани, то 20-30 разделов необходимы для обеспечения достаточного количества ДНК. - Используя щипчи, перенесите каждую секцию на водяную ванну 45 градусов по Цельсию. Возьмите раздел из водяной ванны с положительно заряженной слайд (Таблица материалов), который ранее помечены с образцом идентификационного номера с помощью карандаша.
- Поместите слайды, содержащие секции, в духовку при температуре 65 градусов по Цельсию, чтобы разделы должны были придерживаться слайдов. Держите слайды для Н И Е в духовке в течение 25 минут. Держите слайды для извлечения ДНК в духовке в течение 20 минут.
ПРИМЕЧАНИЕ: Более короткое время инкубации делает ткани чуть менее приверженными слайдам и, следовательно, облегчает удаление материнских тканей.
-
Н И E окрашивания
- Дайте слайдам остыть до комнатной температуры (10 мин).
- Подготовка реагента
- Подготовка рабочего решения Eosin Y (0.25%) в таблице 1. Хорошо перемешать и хранить при комнатной температуре.
- Приготовьте рабочий раствор гематоксилина, разбавив в воде бульонный раствор гематоксилина 5x (т.е. смешайте 80 мл воды с 20 мл гематоксилина).
ПРИМЕЧАНИЕ: Заверните бульонный раствор гематоксилина в фольгу для хранения.
- Подготовка окрашивания банки с правильными реагентами под дымом капот в соответствии с таблицей 2.
- Выполните H и E окрашивания путем погружения слайдов в соответствующие окрашивающие банки для правильного периода времени в соответствии с таблицей 2.
- Смонтировать 4 мкм разделы для морфологического анализа с монтажом среды и покрывало со стеклянными крышками(Таблица материалов).
ПРИМЕЧАНИЕ: 10 мкм разделы для генотипирования не должны быть coverslipped. - Оставьте 10 мкм разделов под дымом капот как минимум 3 ч для того, чтобы токсичные запахи ксилена рассеять.
ПРЕДЕКТО: Все шаги окрашивания должны быть выполнены под капотом дыма. Ксиленовые продукты должны храниться под капотом во все времена, потому что запахи ксилена токсичны. Кроме того, ксилен и гематоксилин необходимо отбросить в специальных контейнерах. После того, как эти контейнеры заполнены, они должны быть удалены в соответствии с рекомендациями организации по безопасности лаборатории.
| Реагента | Количество |
| Акционерное решение Eosin Y (1%) | 250 мл |
| 80% Этанол | 750 мл |
| Ледниковая ацетическая кислота (концентрированная) | 5 мл |
Таблица 1: Рабочее решение Eosin Y (0.25%) Подготовка.
| Используется реагент (100 мл на бен) | Длительность |
| 1) Ксилен | 5 мин. |
| 2) Ксилен | 5 мин. |
| 3) 100% этанол | 2 мин. |
| 4) 95% этанол | 2 мин. |
| 5) 70% этанол | 2 мин. |
| 6) 50% этанол | 2 мин. |
| 7) Дистиллированная вода | 5 мин. |
| 8) Гематоксилин | 4 мин. |
| 9) Дистиллированная вода | 5 мин. |
| 10) Эосин | 1 мин. |
| 11) 95% этанол | 5 мин. |
| 12) 100% этанол | 5 мин. |
| 13) Ксилен | 5 мин. |
| 14) Ксилен | 5 мин. |
Таблица 2: Реагенты и длительность для протокола окрашивания Н и Е.
-
Изоляция резюме
- Под легким стереомикроскопом, используйте щипцы и небольшие кусочки воды смачивают бумажные салфетки(Таблица материалов), чтобы соскребать нежелательные материнские ткани из H и E-окрашенные 10 мкм толщиной разделов.
ПРИМЕЧАНИЕ: Конечная цель состоит в том, чтобы держать ничего, кроме резюме или плода мембраны (при настоящем) на слайдах и, таким образом, чтобы удалить все другие ткани. Этот шаг может потребовать сятвения и терпения, в зависимости от блока, так как он требует тщательного внимания к деталям. - У второго лица перепроверить слайды после очистки, чтобы убедиться, что они свободны от материнских тканей.
- Сфотографируйте очищенные слайды или задокументируйте следующее, чтобы помочь с интерпретацией данных: 1) было ли ткани трудно чистить, геморрагические, или очень чистые, 2) количество используемых разделов, и 3) приблизительное количество очищенных тканей.
ПРИМЕЧАНИЕ: Рисунок 1 является примером слайда, который легко чистить. Для блока, содержащего примерно такое количество резюме, достаточно 10 секций для извлечения ДНК. Слайд на рисунке 2 имеет очень мало резюме, которые перепходят с материнскими тканями, что делает его очень трудным и трудоемким для очистки. Для блока, содержащего примерно такое количество резюме, для извлечения ДНК требуется 30 секций. - Соберите резюме с помощью небольших увлажненных кусочков бумаги салфетки. Используя щипцы, разорвать крошечный кусок из увлажненной бумаги салфетки и использовать его для сбора резюме.
- Поместите кусочки бумаги салфетки с их прилагается CV в помечены 1,5 мл трубки.
- Свести к минимуму количество бумажных салфеток, используемых в этом шаге, как слишком много может засорить колонку извлечения ДНК и, следовательно, уменьшить окончательное количество собранной ДНК. В среднем, цель использовать менее семи небольших кусочков бумаги салфетки на образец. Если это невозможно из-за наличия большого количества cv, разделить образец между двумя трубками для облегчения извлечения.
- Под легким стереомикроскопом, используйте щипцы и небольшие кусочки воды смачивают бумажные салфетки(Таблица материалов), чтобы соскребать нежелательные материнские ткани из H и E-окрашенные 10 мкм толщиной разделов.

Рисунок 1: Представитель слайд для генотипирования. Вверху: слайд, который должен быть "очищен", чтобы освободиться от материнских тканей. Внизу: тот же слайд, показанный после того, как он был очищен и теперь содержит только резюме для извлечения ДНК. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
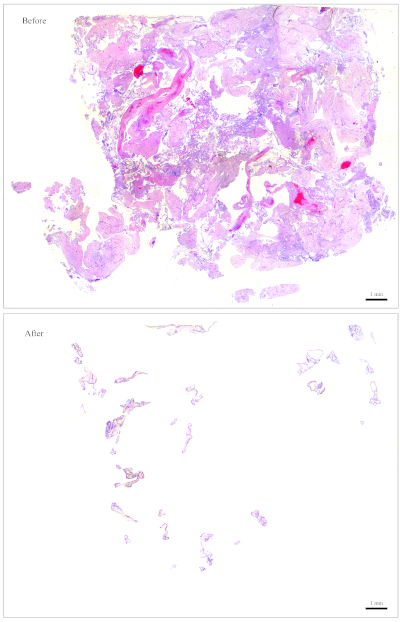
Рисунок 2: Представитель слайд для генотипирования. Вверху: слайд, который должен быть "очищен", чтобы освободиться от материнских тканей. Внизу: тот же слайд, показанный после того, как он был очищен и теперь содержит только резюме для извлечения ДНК. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
- Следуйте протоколу извлечения ДНК из комплекта FFPE (Таблица материалов) для выполнения экстракции ДНК.
ПРИМЕЧАНИЕ: Некоторые комплекты рекомендуют использовать буфер elution 15–20 л для окончательного элюционного буфера. Из опыта, elution с 15 злиционным буфером elution хорошо работает для большинства образцов. Разбавления могут быть подготовлены из запасов ДНК по мере необходимости.
-
Количественная оценка ДНК
- Используя лабораторно-спектрофотометрическое устройство, загрузите 1 кВ л ДНК и измерьте абсорбцию на уровне 260 нм для количественной оценки.
- Загрузите 1 зл ДНК на 2% агарозный гель и запустите электрофорез геля при напряжении 80–100 В для качественной оценки.
- На основе результатов шагов 1.6.1 и 1.6.2, выберите объем ДНК, который будет использоваться в мультиплексе короткого тандемного повтора (STR) полимеразной цепной реакции (ПЦР). Цель использовать как минимум 1000 нг ДНК в усилитель ПЦР, что следует.
ПРИМЕЧАНИЕ: На рисунке 3 показаны репрезентативные примеры гелей наряду с концентрациями ДНК (на основе результатов спектрофотометра) и объемом решения ДНК, которое рекомендуется для пЦР мультиплекса STR, который следует за этим.
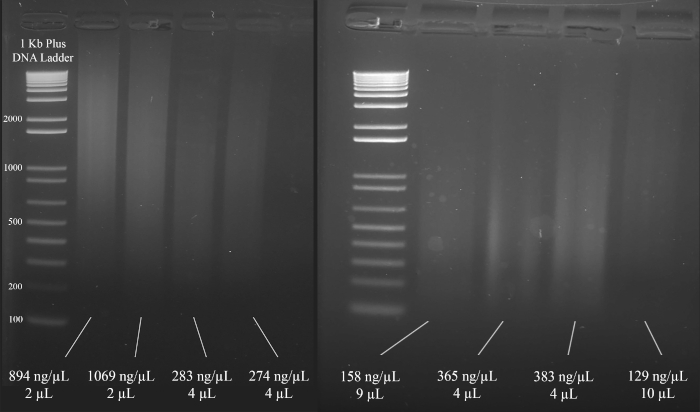
Рисунок 3: Представительгель для количественной оценки ДНК. Включены концентрации каждой ДНК, как измеряется с помощью спектрофотометра, и количества, используемые для мультиплекса ПЦР. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
-
Усиление ПЦР
- Выполните флуоресцентный микроспутниковый генотипирование с помощью мультиплексной системы STR(Таблица материалов).
- Используйте условия ПЦР, показанные на рисунке 4, для усиления ПЦР с помощью мультиплексной системы STR(Таблица материалов).
ПРИМЕЧАНИЕ: Следующие праймеры используются в этой мультиплексной системе STR: D18S51, D21S11, TH01, D3S1358, Penta E, FGA, TPOX, D8S1179, vWA, Amelogenin, CSF1PO, D16S539, D7S820, D13S317, D5S818 и Penta D.

Рисунок 4: Условия цикла ПЦР для мультиплексной системы STR. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
-
Разрешить пЦР продукты капиллярным электрофорасом.
- Приостановить 1 зл каждого усиленного образца в 0,5 л внутренней стандартной полосы мультиплексной системы и 9,5 зла высокодейонизированного формамида(Таблица материалов).
- Запуск образцов через капиллярный электрофорекс инструмент(Таблица материалов) с использованием соответствующей матрицы разделения (Таблица материалов) для инструмента и набор красителя мультиплексной системы.
- Анализ данных
- Проанализируйте данные с помощью программного обеспечения для анализа фрагментов ДНК и сравните аллели POC с родительскими аллелями, чтобы определить их происхождение.
- Настройка стандарта размера.
ПРИМЕЧАНИЕ: Это позволяет программному обеспечению распознавать лестницу, которая используется в мультиплексной системе STR, и назначать базовые пары ампронов на основе лестницы. Следующие шаги для одного конкретного программного обеспечения(Таблица материалов),но может быть поможет для создания других типов программного обеспечения, а также.- Откройте программное обеспечение. Нажмите на Начало нового проекта, а затем на новый стандарт размера.
- Дайте стандарту размера имя (например, ABI-600).
- В поле под названием Введите новый размер стандартного определения: введите следующее: 60, 80, 100, 120, 140, 160, 180, 200, 225, 250, 300, 325, 350, 375, 400, 425, 450, 450. Затем нажмите на Добавить размер (ы).
ПРИМЕЧАНИЕ: Введенные номера будут отображаться под полем справа, который называется Текущее стандартное определение размера (см. Рисунок 5). - Нажмите на Сохранить.
- Чтобы импортировать и анализировать файл, нажмите на Добавить файлыи выбрать fsa файл для анализа. Нажмите на Добавить выбранные файлы, а затем на OK. Затем выполните следующие действия:
- Найдите столбец Standard size и выберите ABI-600 (или какое бы имя ни было дано стандарту размера).
- При методе анализанажмите на Sizing Default - NPP, а затем нажмите на зеленую кнопку анализа.
- Файл готов к просмотру. Отрегулируйте параметры просмотра, чтобы просматривать данные по желанию.
- Устранение неполадок - метод анализа
ПРИМЕЧАНИЕ: Программное обеспечение может иногда не определить пики и выровнять их правильно. Это происходит, когда пики либо слишком низки, либо слишком высоки. Следующие два метода анализа могут исправить это и должны быть опробованы до повторного тестирования образца.- Метод анализа 1 для высоких пиков:
- Нажмите на новый метод анализа и назовите его High Peaks (или другое имя в зависимости от личных предпочтений).
- Нажмите на диапазон, а затем на частичный диапазон для анализа и размеров. Затем введите 100 для стартовой точки и размера старта.
- Для стоп-пойнтавведите 10 000. Для стоп-размеравведите 1000.
- Затем нажмите на минимальные пиковые высоты и измените цифры таким образом, чтобы пиковый порог для цветов был следующим: Синий: 50; Зеленый: 50; желтый: 20; Красный: 100; Оранжевый: 5000.
- Сохранить новый метод анализа.
- Метод анализа 2 для низких пиков:
- Нажмите на новый метод анализа и назовите его Low Peaks (или другое имя в зависимости от личных предпочтений).
- Нажмите на диапазон, а затем на частичный диапазон для анализа и размеров. Затем введите 100 для стартовой точки и размера старта.
- Для стоп-пойнтавведите 10 000. Для стоп-размеравведите 1000.
- Затем нажмите на флаги качества и измените диапазон Pass таким образом, чтобы он читает от 0,5 до 1. Измените диапазон низкого качества так, что он читает от 0,0 до 0,0. Изменение Допустим линейность на следующее: от (bp) 100.0 до (bp) 800.0.
- Сохранить новый метод анализа.
ПРИМЕЧАНИЕ: Теперь можно повторно проанализировать файл, выбрав Низкие пики или высокие пики в рамках метода анализа, а затем нажав на зеленую кнопку анализа.
- Метод анализа 1 для высоких пиков:

Рисунок 5: Скриншот, показывающий стандартный редактор размера. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
2. Цитометрия потока
-
Выбор идеального блока FFPE
- Используя слайды H и E и легкий микроскоп, выберите блок FFPE, который имеет около 50–70% его тканей, состоящих из резюме.
ПРИМЕЧАНИЕ: Рисунок 6 является репрезентативным примером соответствующего блока для анализа цитометрии потока, так как он состоит примерно из 50% CV (правая половина раздела) и 50% материнских тканей (левая половина). Наличие материнских тканей имеет важное значение, поскольку они служат в качестве внутреннего контроля для диплоидного пика. - Для блоков, которые не имеют идеального количества резюме, обогащают для резюме, как секция выполняется. Для этого определите, какая сторона свежесрезаемых секций содержит больше резюме в соответствии с соответствующим слайдом H и E. Исходя из этого, используйте лезвие, чтобы отрезать другую половину, которая должна быть отброшена для того, чтобы обогатить для резюме.
ПРИМЕЧАНИЕ: На рисунке 7 показан блок, который не имеет достаточного CV для анализа цитометрии потока. Для блоков, таких как этот, разделы должны быть сокращены таким образом, что половина, которая содержит меньше резюме получает отбрасываются для того, чтобы увеличить количество резюме в отношении материнских тканей, как показано на рисунке. Не забудьте сократить больше разделов, чтобы компенсировать то, что отбрасывается.
- Используя слайды H и E и легкий микроскоп, выберите блок FFPE, который имеет около 50–70% его тканей, состоящих из резюме.
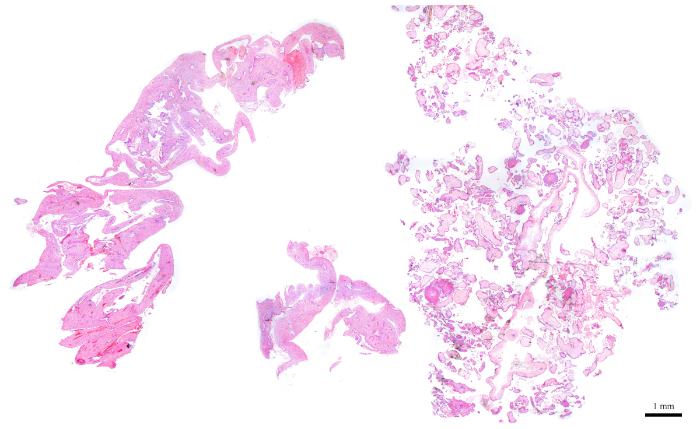
Рисунок 6: Н И Е раздел, представляющий блок POC, который идеально подходит для цитометрии потока. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
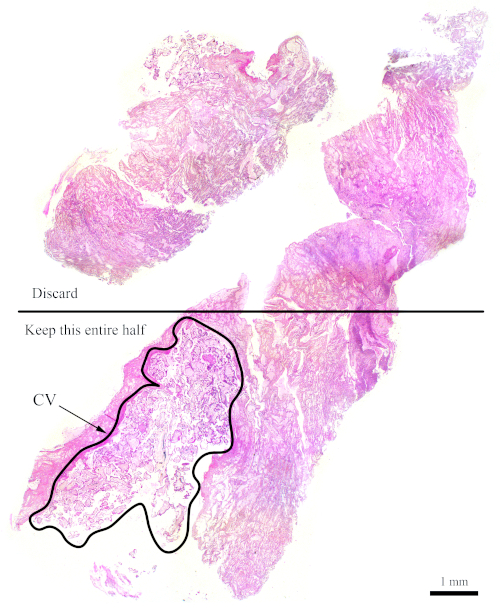
Рисунок 7: Н И Е раздел, представляющий более сложный блок для цитометрии потока. Этот репрезентативный раздел Н И Е показывает, что только нижняя половина этого раздела должна использоваться для анализа цитометрии потока, с целью обогащения для резюме. Изложенная область, помеченная как "CV", в основном состоит из резюме.
- Сечения
- Оставьте блоки на льду в течение 15 минут, чтобы облегчить секцию.
- Используя наилучший блок FFPE, вырежьте четыре секции толщиной 50 мкм (или два участка толщиной 100 мкм) с помощью микротома.
ПРИМЕЧАНИЕ: Для цитометрии потока предпочтительнее иметь более толстые секции. - В случае, если идеальный блок FFPE не доступен, цель сохранить соотношение CV к материнской ткани, тем не менее. Например, если только 30% блока состоит из резюме, в то время как остальные материнские ткани, а затем удалить по крайней мере половину раздела, который содержит материнские ткани и использовать больше разделов для компенсации (см. Рисунок 7).
- Поместите секции в маркированные трубы 15 мл.
ПРИМЕЧАНИЕ: Не забудьте ленту над этикетками, потому что органические реагенты, используемые в следующем шаге, могут растворяться и удалять чернила.
- Протокол цитометрии потока из тканей FFPE
- Депараффинизация и регидратация
- Выполните следующие стирания (Таблица 3) под дымом капот.
- Заполните трубку 15 мл с 6 мл соответствующего реагента, следуя приказу, представленному в таблице 3, оставьте секции в реагентах на соответствующую продолжительность, а затем удалите реагент с помощью вакуумного всасывания и стеклянной пипетки Pasteur.
- Между каждым шагом, окунуть Пастер пипетка сначала в 70% этанола, затем в дистиллированной воде, а затем перейти к следующему шагу.
- Будьте очень осторожны, чтобы не удалить куски ткани вместе с реагентом. Наклоните трубку 15 мл под углом 60 градусов, чтобы облегчить всасывание жидкого реагента без нанесения тканей.
ПРЕДЕКТО: Выброшенные жидкости содержат ксилен и должны быть утилизированы в контейнерах для отходов ксилена.
- Подготовка решения
- Приготовьте раствор цитрата, растворив 2 г лимонной кислоты в 1 л двойной дистиллированной воды. Доведите рН до 6. Хранить при 4 градусах по Цельсию.
- Приготовьте пепсиновый раствор путем растворения 0,01 г пепсина в 2 мл из 9 частей на тысячу NaCl, рН 1,64. Это для одного образца.
ПРЕДЕКТО: Пепсин токсичен и может легко рассеяться и стать воздушно-капельным путем. Носите маску при обработке пепсина в виде порошка и протрите вниз всю рабочую зону после его использования. - Propidium Iodide (PI)-рибонуклеаза Подготовка раствора для одного образца.
- Смешайте 50 л ИП с 450 Л ПБС (для разбавления 10x).
- Добавьте в смесь 50 л рибонуклеаза А (1 мг/мл). Держите завернутые в фольгу во все времена.
- Пищеварение и окрашивание
- Добавьте раствор цитрата 4 градуса к трубкам 15 мл, затем поместите в водяную ванну мощностью 80 градусов по Цельсию в течение 2 ч.
- Дайте раствору остыть до комнатной температуры (15 мин). Удалите раствор цитрата.
- Добавьте 6 мл 1x PBS, вихрь, и подождите 1'2 мин, чтобы ткани, чтобы осесть на дно. Удалите 1x PBS с помощью вакуумного всасывания и стеклянной пипетки Pasteur.
- Добавьте 1 мл пепсина раствора (разогретого до 37 градусов по Цельсию) и поместите в сухую ванну 37 градусов по Цельсию в течение 30 мин. Vortex каждые 10 минут. Подготовьте PI-рибонуклизуа улейся раствор в последние 10 минут этой инкубации.
- Добавьте 6 мл 1x PBS, вихрь, и подождите 1'2 мин, чтобы ткани, чтобы осесть на дно. Удалите 1x PBS с помощью вакуумного всасывания и стеклянной пипетки Pasteur.
- Добавьте 550 qL PI-рибонуклеаза Раствор и поместите образцы в сухую ванну 37 градусов по Цельсию в течение 30 минут.
ПРИМЕЧАНИЕ: В этот момент образцы можно завернуть в фольгу и оставить на ночь при 4 градусах Цельсия до следующего утра. - Фильтр раствора через 48-мм фильтрационную сетку. Соберите фильтрат в полистирол круглого дна трубки, которые могут быть использованы с потоком цитометра. Используйте щипцы, чтобы поместить 5 см на 5 см кусок фильтрации сетки в верхней части трубки, так что жидкость может быть трубчата через сетку и в трубку.
ПРИМЕЧАНИЕ: Образцы теперь готовы к запуску с цитометром потока. Держите их завернутые в фольгу, пока они не готовы к запуску.
- Запуск образцов с цитометром потока с помощью организации потока цитометрии платформы техник.
ПРИМЕЧАНИЕ: Канал PE используется для обнаружения PI-окрашенных ДНК и скорость потока должна быть установлена, чтобы замедлить во время приобретения. Убедитесь, что напряжение выбрано таким образом, что пик диплоида находится примерно на уровне 200 вдоль оси PE-A x для облегчения анализа и интерпретации. Цель зафиксировать как минимум 20 000 событий на выборку.
- Депараффинизация и регидратация
| Используется реагент (по 6 мл) | Длительность |
| 1) Ксилен | 2 x 10 мин |
| 2) 100% этанол | 2 x 10 мин |
| 3) 95% Этанол | 10 мин. |
| 4) 70% этанол | 10 мин. |
| 5) 50% этанол | 10 мин. |
| 6) Дистиллированная вода | 2 x 10 мин |
Таблица 3: Реагенты и длительность депарафинизации и регидратации.
-
Анализ цитометрии потока
- Анализ данных с помощью программного обеспечения для анализа цитометрии потока(Таблица материалов).
ПРИМЕЧАНИЕ: Следующие шаги для одного конкретного программного обеспечения(Таблица материалов),но может быть поможет для создания других типов программного обеспечения, а также.- После запуска образцов на цитометр потока, скачать FCS 2.0 файлы для анализа.
- Откройте программное обеспечение для анализа цитометрии потока, нажмите на файл Новый документ.
- Нажмите на значок
 Histogram (), а затем перетащите указатель, чтобы сделать прямоугольник.
Histogram (), а затем перетащите указатель, чтобы сделать прямоугольник. - Просмотрите файл FCS, а затем нажмите на Open. Вдоль x-оси нажмите на FCS-A, а затем выберите PE-A.
- Нажмите на значок Dot Plot (),
 а затем перетащите указатель, чтобы создать еще один прямоугольник под сюжетом гистограммы. Затем просмотрите тот же файл FCS, который был выбран для гистограммы.
а затем перетащите указатель, чтобы создать еще один прямоугольник под сюжетом гистограммы. Затем просмотрите тот же файл FCS, который был выбран для гистограммы. - Измените x-оси точечного участка на PE-A и y-оси на PE-W.
ПРИМЕЧАНИЕ: Рисунок 8А демонстрирует внешний вид участков на данный момент. - Нажмите на значок региона
 () и нарисуйте поле на точечном участке, который начинается до пика диплоида (около 100 на оси Х на рисунке 8B)и который заканчивается около 700 на оси x, как показано на точечном участке на рисунке 8 B.
() и нарисуйте поле на точечном участке, который начинается до пика диплоида (около 100 на оси Х на рисунке 8B)и который заканчивается около 700 на оси x, как показано на точечном участке на рисунке 8 B.
ПРИМЕЧАНИЕ: Пик диплоида на рисунке 8 составляет примерно 200 на оси х. Это выбрано произвольно, как образцы регистрируются через цитометр потока, просто для облегчения анализа и интерпретации результатов. - Нажмите на участок (ru) Отображать регионы/ворота,затем введите R0 в ячейке, которая находится рядом с ячейкой G0 в соответствии со стратегией. Затем нажмите на Закрыть.
- Нажмите в любом месте на гистограмму, а затем на участок (ru) Формат Участок / Оверлай. Под воротами,выберите G0 и R0, а затем нажмите на OK.
ПРИМЕЧАНИЕ: Это шаг gating который позволяет одно более лучше визуализировать пики ploidy. Гистограмма должна теперь выглядеть как гистограмма на рисунке 8B. Можно поиграть с воротами, созданными (перемещая поле, нарисованное в шаге 2.4.1.7), чтобы сосредоточиться на определенных областях точечного сюжета. - Чтобы обозначить сюжеты, нажмите
 на значок text Area (), затем перетащите указатель, чтобы создать окно в верхней части документа, а затем введите следующую информацию: Идентификатор пациента, POC ID и используемый блок (поскольку может быть несколько блоков для одного POC) , процент cv присутствует на блоке, напряжение используется для запуска образца, и дата.
на значок text Area (), затем перетащите указатель, чтобы создать окно в верхней части документа, а затем введите следующую информацию: Идентификатор пациента, POC ID и используемый блок (поскольку может быть несколько блоков для одного POC) , процент cv присутствует на блоке, напряжение используется для запуска образца, и дата.
- Анализ данных с помощью программного обеспечения для анализа цитометрии потока(Таблица материалов).
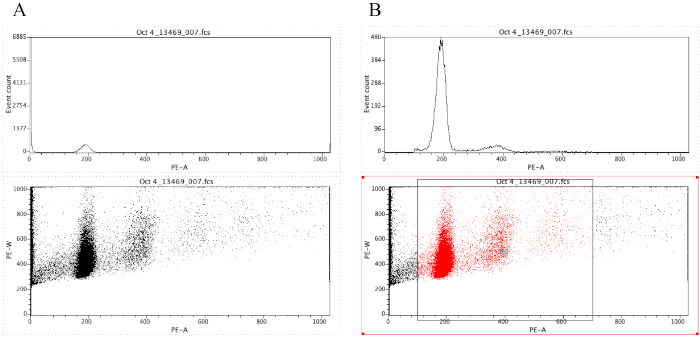
Рисунок 8: Скриншот, отображающий гистограмму и точечный участок репрезентативного образца, который неопрозрачен (A) и закрыто (B). Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
Результаты
Сложность моляровских тканей и тот факт, что они могут иметь различные генотипы, требуют строгого анализа и использования нескольких методов, таких как морфологическая оценка, иммуногистохимия p57, генотипирование микроспутников, цитометрия потока и FISH. Например, один...
Обсуждение
HM являются ненормальные человеческие беременности с разнородными этиологии и имеют различные гистологические и генотипические типы, что делает их точной классификации и диагностики сложной задачей. Гистопатологическая морфологическая оценка часто оказывалась неточной и поэтому са...
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Авторы благодарят Софи Патриер и Марианну Пареси за совместное использование оригинального протокола цитометрии потока, а также Promega и Qiagen за поставки материалов и реагентов. Эта работа была поддержана Репродукцией Репродукции Репродукции Ребикуи и Канадским институтом исследований в области здравоохранения (MOP-130364) в Р.С.
Материалы
| Name | Company | Catalog Number | Comments |
| BD FACS Canto II | BD BioSciences | 338960 | |
| Capillary electrophoresis instrument: Genomes Applied Biosystems 3730xl DNA Analyzer | Applied biosystems | 313001R | Service offered by the Centre for Applied Genomics (http://www.tcag.ca) |
| Citric acid | Sigma | 251275 | |
| Cytoseal 60, histopathology mounting medium | Fisher | 23244257 | |
| Eosin Y stock solution (1%) | Fisher | SE23-500D | |
| FCSalyzer - flow cytometry analysis software | SourceForge | - | https://sourceforge.net/projects/fcsalyzer/ |
| FFPE Qiagen kit | Qiagen | 80234 | |
| Forceps | Fine Science Tools | 11295-51 | For sectioning and for the cleaning process |
| Glacial Acetic Acid (Concentrated) | Sigma | A6283-500mL | |
| Glass coverslips: Cover Glass | Fisher | 12-541a | |
| Hematoxylin | Fisher | CS401-1D | |
| Highly deionized formamide: Hi-Di Formamide | Thermofisher | 4311320 | |
| IHC platform: Benchmark Ultra | Roche | - | |
| Kimwipes | Ultident | 30-34120 | |
| Microtome | Leica | RM2135 | |
| Microtome blades | Fisher | 12-634-1C | |
| Nitex filtering mesh, 48 microns | Filmar | 74011 | http://www.filmar.qc.ca/index.php?filet=produits&id=51&lang=en ; any other filter is suitable, but this is an inexpensive and effective option from a non-research company |
| p57 antibody | Cell Marque | 457M | |
| Pasteur pipette | VWR | 53499-632 | |
| PCR machine | Perkin Elmer, Applied Biosystems | GeneAmp PCR System 9700 | |
| PeakScanner 1.0 | Applied Biosystems | 4381867 | Software for genotyping analysis. |
| Pepsin from porcine gastric mucosa | Sigma | P7012 | |
| Polystyrene round-bottom tubes | BD Falcon | 352058 | |
| Positively charged slides: Superfrost Plus 25x75mm | Fisher | 1255015 | |
| PowerPlex 16 HS System | Promega Corporation | DC2102 | |
| Propidium Iodide | Sigma | P4864 | |
| Ribonuclease A from bovine pancreas | Sigma | R4875 | |
| Separation matrix: POP-7 Polymer | Thermofisher | 4352759 | |
| UltraPure Agarose | Fisher | 16500-500 | |
| Xylene | Fisher | X3P1GAL |
Ссылки
- Szulman, A. E., Surti, U. The syndromes of hydatidiform mole. II. Morphologic evolution of the complete and partial mole. American Journal of Obstetrics & Gynecology. 132 (1), 20-27 (1978).
- Fukunaga, M., et al. Interobserver and intraobserver variability in the diagnosis of hydatidiform mole. The American Journal of Surgical Pathology. 29 (7), 942-947 (2005).
- Gupta, M., et al. Diagnostic reproducibility of hydatidiform moles: ancillary techniques (p57 immunohistochemistry and molecular genotyping) improve morphologic diagnosis for both recently trained and experienced gynecologic pathologists. The American Journal of Surgical Pathology. 36 (12), 1747-1760 (2012).
- Howat, A. J., et al. Can histopathologists reliably diagnose molar pregnancy?. Journal of Clinical Pathology. 46 (7), 599-602 (1993).
- Banet, N., et al. Characteristics of hydatidiform moles: analysis of a prospective series with p57 immunohistochemistry and molecular genotyping. Modern Pathology. 27 (2), 238-254 (2014).
- Lipata, F., et al. Precise DNA genotyping diagnosis of hydatidiform mole. Obstetrics & Gynecology. 115 (4), 784-794 (2010).
- Buza, N., Hui, P. Partial hydatidiform mole: histologic parameters in correlation with DNA genotyping. International Journal of Gynecologic Pathology. 32 (3), 307-315 (2013).
- Fisher, R. A., et al. Frequency of heterozygous complete hydatidiform moles, estimated by locus-specific minisatellite and Y chromosome-specific probes. Human Genetics. 82 (3), 259-263 (1989).
- Murdoch, S., et al. Mutations in NALP7 cause recurrent hydatidiform moles and reproductive wastage in humans. Nature Genetics. 38 (3), 300-302 (2006).
- Parry, D. A., et al. Mutations causing familial biparental hydatidiform mole implicate c6orf221 as a possible regulator of genomic imprinting in the human oocyte. American Journal of Human Genetics. 89 (3), 451-458 (2011).
- Nguyen, N. M., Slim, R. Genetics and Epigenetics of Recurrent Hydatidiform Moles: Basic Science and Genetic Counselling. Current Obstetrics and Gynecology Reports. 3, 55-64 (2014).
- Sebire, N. J., Savage, P. M., Seckl, M. J., Fisher, R. A. Histopathological features of biparental complete hydatidiform moles in women with NLRP7 mutations. Placenta. 34 (1), 50-56 (2013).
- Nguyen, N. M., et al. Comprehensive genotype-phenotype correlations between NLRP7 mutations and the balance between embryonic tissue differentiation and trophoblastic proliferation. Journal of Medical Genetics. 51 (9), 623-634 (2014).
- Brown, L., et al. Recurrent pregnancy loss in a woman with NLRP7 mutation: not all molar pregnancies can be easily classified as either "partial" or "complete" hydatidiform moles. International Journal of Gynecologic Pathology. 32 (4), 399-405 (2013).
- Colgan, T. J., Chang, M. C., Nanji, S., Kolomietz, E. A Reappraisal of the Incidence of Placental Hydatidiform Mole Using Selective Molecular Genotyping. The International Journal of Gynecological Cancer. 26 (7), 1345-1350 (2016).
- Murphy, K. M., McConnell, T. G., Hafez, M. J., Vang, R., Ronnett, B. M. Molecular genotyping of hydatidiform moles: analytic validation of a multiplex short tandem repeat assay. The Journal of Molecular Diagnostics. 11 (6), 598-605 (2009).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеThis article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены